Method Article
Protokoll zur Differenzierung von human induzierten pluripotenten Stammzellen in Mischkulturen von Neuronen und Glia für Neurotoxizitätstests
In diesem Artikel
Zusammenfassung
Menschliche induzierte pluripotenten Stammzellen (hiPSCs) gelten als ein leistungsfähiges Werkzeug für die Arzneimittel- und Chemikalien-Screening und für die Entwicklung neuer in vitro- Modelle für die Toxizitätstests, einschließlich Neurotoxizität. Hier wird ein detailliertes Protokoll zur Differenzierung von hiPSCs in Neuronen und Glia beschrieben.
Zusammenfassung
Menschliche pluripotenten Stammzellen können in verschiedene Zelltypen differenzieren, die auf human- in-vitro- Toxizitäts-Assays angewendet werden können. Ein wichtiger Vorteil ist, dass die Neuprogrammierung von somatischen Zellen zur Erzeugung von humanen induzierten pluripotenten Stammzellen (hiPSCs) die ethischen und legislativen Fragen im Zusammenhang mit der Verwendung von menschlichen embryonalen Stammzellen (hESCs) vermeidet. HiPSCs können erweitert und effizient in verschiedene Arten von neuronalen und glialen Zellen differenziert werden, die als Testsysteme für die Toxizitätstests dienen und insbesondere für die Bewertung verschiedener Wege, die an der Neurotoxizität beteiligt sind. Diese Arbeit beschreibt ein Protokoll für die Differenzierung von hiPSCs in gemischte Kulturen von neuronalen und gliazellen. Die Signalisierungswege, die durch neuronale Differenzierung reguliert und / oder aktiviert werden, werden definiert. Diese Information ist entscheidend für die Anwendung des Zellmodells auf das neue Toxizitätstestparadigma, in dem Chemikalien auf der Grundlage ihrer Fähigkeit, peBiologische Wege umgehen. Als Nachweis des Konzepts wurde Rotenon, ein Inhibitor des mitochondrialen Atmungskomplexes I, zur Aktivierung des Nrf2-Signalweges, eines Schlüsselregulators des Antioxidans-Response-Element- (ARE) -getriebenen zellulären Abwehrmechanismus gegen oxidativen Stress, eingesetzt .
Einleitung
Der US-National Research Council Report 1 stellte ein neues Toxizitätstest-Paradigma vor, in dem die regulatorische Toxizitätstests von einem Ansatz verlagert würden, der sich auf phänotypische Veränderungen bezieht, die bei Tieren beobachtet wurden, auf einen Ansatz, der sich auf mechanistische in vitro- Assays unter Verwendung menschlicher Zellen konzentriert. Pluripotenten Stammzellen (PSC) -Derivate können Alternativen zu Krebszellmodellen darstellen, da die erhaltenen Zellen den physiologischen Zuständen menschlicher Gewebe genauer ähneln und relevantere Instrumente liefern können, um chemisch induzierte Nebenwirkungen zu untersuchen. Die beiden wichtigsten Arten von PSC-Kulturen, die für Toxizitätstests am häufigsten sind, sind menschliche embryonale Stammzellen (hESCs) und menschlich induzierte pluripotenten Stammzellen (hiPSCs), die derzeit in den Bereichen Grundlagenforschung und regenerative Medizin weit verbreitet sind 2 , 3 . Diese Expertise kann nun für die Entwicklung einer neuen Klasse von Toxicolo genutzt werdenIn-vitro- Tests, die darauf abzielen, die gestörten physiologischen Wege zu identifizieren, die mit der Entwicklung von nachteiligen Wirkungen in vivo verbunden sind. Allerdings wäre es unwahrscheinlich, dass Prüfverfahren für regulatorische Sicherheitsbewertungen auf der Grundlage von HESCs von allen EU-Mitgliedstaaten und von Ländern weltweit aufgrund möglicher ethischer Bedenken und verschiedener nationaler Gesetzgebungspolitik, die die Verwendung von Embryo-abgeleiteten Zellen regeln, akzeptiert werden.
HiPSCs teilen sich ähnliche Merkmale wie hESCs 4 , 5 und halten großes Potenzial für in vitro Methoden, sowohl für die Identifizierung von therapeutischen Zielen als auch für Sicherheitsbewertungen. Darüber hinaus verringert die hiPSC-Technologie die Einschränkungen eines begrenzten Spender-Pools und die ethischen Bedenken, die mit Embryo-abgeleiteten Zellen verbunden sind. Eine große Herausforderung für hiPSCs ist die Demonstration, dass diese Zellen reproduzierbar einen signifikanten Bereich von toxikologisch relevanten Zellderivaten erzeugen können,Mit Merkmalen und Reaktionen, die für menschliche Gewebe typisch sind. Vordefinierte Ebenen der ausgewählten Marker werden im Allgemeinen verwendet, um Zellpopulationen nach dem Differenzierungsprozess zu charakterisieren und Einblicke in die Stabilität des Differenzierungsprozesses zu geben.
Bisherige Arbeiten beurteilten die Eignung von hiPSCs, um gemischte Kulturen von neuronalen und gliazellen zu erzeugen und die Effekte von Rotenon, einem Inhibitor des mitochondrialen Atmungskomplexes I, auf die Aktivierung des Nrf2-Weges zu beurteilen, ein Schlüsselregulator für antioxidative Abwehrmechanismen in Viele Zelltypen 6 , 7 .
Diese Arbeit beschreibt ein Protokoll, das für die Differenzierung von hiPSCs in gemischte neuronale und gliale Kulturen verwendet wird, wobei Details über die Signalwege (Gen und Protein-Ebene) bereitgestellt werden, die bei der neuronalen / glialen Differenzierung aktiviert werden. Darüber hinaus zeigt die Arbeit repräsentative Ergebnisse, die zeigen, wie diesDas hiPSC-abgeleitete neuronale und glialzellenmodell kann verwendet werden, um die durch akute (24-h) Behandlung mit Rotenon induzierte Nrf2-Signalisierungsaktivierung zu beurteilen, was die Beurteilung der oxidativen Stressinduktion ermöglicht.
IMR90-Fibroblasten wurden bei der I-Stem (Frankreich) durch die virale Transduktion von 2 Transkriptionsfaktoren (Oct4 und Sox2) unter Verwendung von pMIG-Vektoren 6 in hiPSCs umprogrammiert. Es können auch analoge hiPSC-Modelle angewendet werden. Die nachstehend beschriebenen Protokolle fassen alle Stufen der Differenzierung von HiPSCs in neuronale Stammzellen (NSCs) und weiter in gemischte Kulturen von post-mitotischen Neuronen und Gliazellen zusammen (Schritte 1 und 2, siehe auch die EURL ECVAM DBALM Website für eine detaillierte Beschreibung von Das Protokoll) 8 .
Ein zusätzliches Protokoll für die Isolierung, Expansion, Kryokonservierung und weitere Differenzierung von NSCs in gemischte Neuronen und Gliazellen ist in den Schritten 3 und 4 beschrieben (siehe auch EURL ECVAM DBALM wirBsite für eine detaillierte Beschreibung dieses Protokolls) 9 . Schritt 5 beschreibt die Analysen, die getan werden können, um die phänotypische Identität der Zellen während der verschiedenen Stadien der Verpflichtung und Differenzierung zu beurteilen.
Protokoll
1. Mensch induzierte pluripotenten Stammzellen (hiPSC) Expansion
HINWEIS: HIPSCs können auf einem geeigneten Proteinmischungssubstrat in Gegenwart von mTeSR1-Medium, enthaltend mTeSR1 5x Ergänzungen (hergestellt nach den Anweisungen des Herstellers, Platte ~ 100 Koloniefragmente / 60-mm-Petrischale) kultiviert werden. Wenn die hiPSC-Kolonien eine geeignete Größe erreichen (siehe ein Beispiel einer Kolonie in Abbildung 2A ), passieren Sie die Zellen wie unten beschrieben (einmal wöchentlich).
- Mantelschalen mit hESC-qualifizierter Basalmembranmatrix (nachstehend "qualifizierte Matrix" genannt) oder irgendein anderes geeignetes Proteinsubstrat.
- Lagern Sie die qualifizierte Matrix (siehe Tabelle der Materialien ) bei -80 ° C in 200 μl Aliquoten in kalten 1,5 mL Röhrchen und kalten 5 oder 10 mL Pipetten.
- Vor dem Passieren 200 g eine qualifizierte Matrix auf Eis auftauen.
- 200 μl qualifizierte Matrix in 20 m verdünnenL von DMEM / F12-Medium (1: 100 Verdünnung).
- Mantel 60-mm-Petrischalen mit dieser Lösung (5 ml / Teller).
- Inkubieren Sie beschichtete Schalen bei 37 ° C für mindestens 1 h.
- HiPSC Kolonie Fragment Kryokonservierung und Auftauen
- Nach dem Schneiden der hiPSC-Kolonien (siehe Schritt 1.3 für die hiPSC-Kolonie-Schneidprozedur), sanft und langsam die hiPSC-Koloniefragmente im Stammzell-Einfriermedium, ~ 100 Fragmente / 250 μl (siehe Tabelle der Materialien) resuspendieren.
- Aliquotieren Sie die Koloniefragmente in geeigneten Fläschchen zur Kryokonservierung (250 μl / Durchstechflasche).
- Legen Sie die Fläschchen in einen mit 2-Propanol gefüllten Behälter und legen Sie den Behälter bei -80 ° C für mindestens 2 h und bis zu 2 Wochen.
- Übertragen Sie die Fläschchen in die Dampfphase eines flüssigen Stickstoffbehälters.
- Um die Kultur neu zu starten, 1 gefrorene Durchstechflasche im Wasserbad bei 37 ° C auftauen.
- Sammeln Sie die hiPSC-Kolonie-Fragmente vorsichtig in 7 ml vorgewärmtem komplettem hiPSCMedium (siehe Tabelle der Materialien ) in einem 15-ml-Röhrchen mit einer 1, 2 oder 5 ml Pipette.
- Zentrifugieren Sie die hiPSC-Koloniefragmente bei 130 xg für 3 min.
- Den Überstand entfernen und die hiPSC-Koloniefragmente in 1 ml vollständigem hiPSC-Medium mit einer 1, 2 oder 5 ml Pipette vorsichtig resuspendieren.
- Weiterhin verdünnen Sie die Zellsuspension in 3 oder 4 ml komplettem hiPSC-Medium.
- Die hiPSC-Kolonie-Fragmente in einer qualifizierten Matrix-beschichteten 60-mm-Petrischale (~ 100 Fragmente / Schale) schneiden, wie in Schritt 1.1 beschrieben.
- Inkubieren der hiPSCs bei 37 ° C und 5% CO 2 .
- Führen Sie täglich eine mittlere Veränderung durch.
- Passage der hiPSC-Kolonien
ANMERKUNG: Undifferenzierte HalloCPS sollten in Form sein, mit großen Nukleolen und ohne reichhaltiges Zytoplasma. Undifferenzierte Kolonien sollten durch eine flache und dicht verpackte Morphologie charakterisiert werden. Nur undifferenzierte Kolonien (ca. 1 mm iN Durchmesser) für weitere Passagen geschnitten werden.- Schneiden Sie die Stammzellkolonien in Quadraten von etwa 200 μm x 200 μm mit einer 1-ml-Spritze mit einer 30G-Nadel oder anderen handelsüblichen Werkzeugen (siehe Tabelle der Materialien). Verwenden Sie ein stereoskopisches Mikroskop bei 4facher Vergrößerung in einem laminaren Strömungsschrank bei Raumtemperatur.
- Lösen Sie die Koloniefragmente von der Schalenoberfläche mit einer 200-μl-Pipette, indem Sie das Medium vorsichtig pipettieren, um die Stücke zu heben.
- Übertragen Sie die Koloniefragmente (~ 100 Stück) auf eine qualifizierte Matrix-DMEM / F12-beschichtete Platte, die mit 4 ml komplettem hiPSC-Medium gefüllt ist (siehe Tabelle der Materialien, das Geschirr wie in Schritt 1.1 beschrieben).
- Inkubieren Sie die neue Platte (n) bei 37 ° C und 5% CO 2 .
- Führen Sie täglich eine mittlere Veränderung durch und untersuchen Sie die Morphologie der Kolonien mit einem Phasenkontrastmikroskop bei 4X und 10facher Vergrößerung.
2. HiPSC DiffereNtiation in gemischte Neuronen und Glia
HINWEIS: Der Vorgang dauert ca. 28 Tage, mit den Hauptschritten in Abbildung 1 (Oberteil).
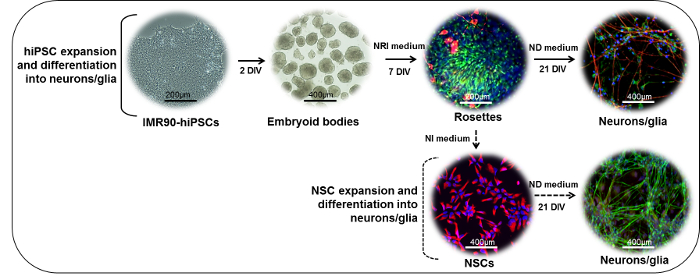
Abbildung 1: Schematische Darstellung des neuronalen Differenzierungsprotokolls. (Oberteil) IMR90-hiPSC-Kolonien können in Fragmente geschnitten werden, um Embryoidkörper (EBs) zu bilden. Nach 2 Tagen in vitro (DIV) können EBs auf Laminin- oder Standard-Matrix-beschichtete Schalen plattiert und in Gegenwart von neuroepithelialem Induktions- (NRI) -Medium kultiviert werden, um neuroectodermale Derivate zu erzeugen (Rosetten, hier für Nestin (grün) und β gefärbt -III-Tubulin (rot)). Rosetten können auf Laminin- oder Standard-Matrix-beschichteten Schalen aufgelöst, gesammelt, wieder aufgetragen und weiter in reife neuronale (NF200, rot) und glial differenziert werden(GFAP, grün) Zellen in Gegenwart von neuronaler Differenzierung (ND) Medium. (Nestin, Rot) können in Gegenwart von neuronalem Induktionsmedium (NI) -Medium expandiert, kryokonserviert oder weiter in Gegenwart von ND-Medium unter Bildung von gemischtem Neuronal (NF200, grün) und glial ( GFAP, rot) Kulturen. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
- Erzeugung von embryoidalen Körpern (EBs) (Tage 0 → 1)
HINWEIS: Dieses Verfahren erfordert gute manuelle Fertigkeiten und Präzision. HiPSC-Kolonie-Fragmente sollten von gleicher Größe sein, um homogene embryoidale Körper (EBs) in den nächsten Schritten zu erhalten. Morphologisch differenzierte Kolonien (mit großen zytoplasmatischen Fraktionen und kleinen Nukleolen) sollten verworfen werden.- Erfrischen Sie das hiPSC-Medium (3 mL / 60-mm-Petrischale) vor dem Schneiden der undifferenzierten hiPSC-Kolonien (ca. 1 mm in diamEter, siehe Abbildung 2A ) unter sterilen Bedingungen (wie in Schritt 1 beschrieben).
- Schneiden Sie die undifferenzierten Kolonien (wie in Fig. 2A und Fig. 2B gezeigt ) in Fragmente von etwa 200 um x 200 um unter Verwendung einer 1-ml-Spritze mit einer 30G-Nadel. Verwenden Sie ein stereoskopisches Mikroskop bei 4facher Vergrößerung in einem laminaren Strömungsschrank bei Raumtemperatur.
- Lösen Sie die Koloniefragmente von der Schalenoberfläche mit einer 200-μl-Pipette, indem Sie das Medium vorsichtig pipettieren, um die Stücke zu heben.
- Übertragen Sie alle abgetrennten Fragmente und das Medium auf ein 15-ml-Röhrchen mit einer 1, 2 oder 5 ml Pipette.
- Spülen Sie das Gericht mit 2 ml komplettem hiPSC-Medium, um alle Fragmente zu gewinnen.
- Zentrifuge bei 112 xg für 1 min.
- Den Überstand anspülen und die Fragmente in 5 ml vollständigem hiPSC EB-Medium (siehe Tabelle der Materialien ) vorsichtig resuspendieren .
- Plate die CoLony-Fragmente in einer 60 mm ultra-low Anlage Petrischale (5 ml / 60-mm-Petrischale).
- Die Petrischale über Nacht bei 37 ° C und 5% CO 2 inkubieren.
- Am nächsten Tag (Tag 1) sammeln Sie die EBs und ihr Medium in einem 15 mL Röhrchen mit einer 1, 2 oder 5 mL Pipette.
- Die EBs bei 112 xg für 1 min zentrifugieren.
- Den Überstand sorgfältig absaugen und die EBs in 5 ml vollständigem hiPSC EB-Medium mit einer 1, 2 oder 5 mL Pipette vorsichtig resuspendieren.
- Die EBs auf eine neue 60-mm-Ultra-Low-Attachment-Petrischale (5 ml / 60 mm Petrischale) auftragen.
- Die Petrischale über Nacht bei 37 ° C und 5% CO 2 inkubieren.
- Am Tag 1 beschichten Sie die Speisen mit Basalmembranmatrix ( z . B. Matrigel, nachfolgend als "Standardmatrix" bezeichnet) oder einem anderen geeigneten Proteinsubstrat ( zB Laminin).
- Lagern Sie die Standardmatrix (siehe Tabelle der Materialien ) bei -80 ° C200 μl Aliquots mit kalten 1,5 mL Röhrchen und kalten 5 oder 10 mL Pipetten.
- 200 μl Standardmatrix auf Eis auftauen
- 200 μl Standardmatrix in 20 ml DMEM / F12-Medium (1: 100 Verdünnung) verdünnen.
- Mantel 60-mm-Petrischalen mit dieser Lösung (5 ml / Teller).
- Inkubieren Sie die beschichteten Schalen bei 37 ° C über Nacht.
HINWEIS: Diese Gerichte werden verwendet, um die EBs (ca. 50 EBs / Schale) zu plattieren und neuroepitheliale Aggregate (Rosetten) zu erzeugen; Siehe Schritt 2.3.
- Erzeugung von neuroepithelialen Aggregaten (Rosetten) (Tage 2 → 7)
- Am Tag 2 entfernen Sie die Standard-Matrix-Beschichtungslösung aus den 60-mm-Petrischalen (keine Notwendigkeit, die Platten zu spülen) und füllen sie mit 5 ml / Schale des vollständigen neuroepithelialen Induktionsmediums (NRI); Siehe die Tabelle der Materialien .
- Übertragen Sie die schwimmenden EBs (ab Schritt 2.1.14) auf beschichtete Schalen (~ 50 EBs / Schale) mit einer 200 μl Pipette unter einer stereoskopischen miCroscope bei 4facher Vergrößerung und platziert in einem laminaren Strömungsschrank.
HINWEIS: Es ist entscheidend, homogene, mittelgroße EBs (~ 200-300 μm Durchmesser) auszuwählen. Zu kleine EBs können während der neuroektodermalen Differenzierung nicht gut überleben, während zu große EBs dazu neigen, Kernnekrose zu unterziehen. - Inkubieren Sie das Geschirr bei 37 ° C und 5% CO 2 .
- Am Tag nach (Tag 3) überprüfen Sie das Geschirr unter dem Mikroskop bei 10-facher Vergrößerung, um sicherzustellen, dass die EBs alle angeschlossen sind.
- Mit einem kompletten NRI-Medium vorsichtig einen mittleren Mittelwechsel durchführen.
- Ändern Sie das NRI-Medium jeden zweiten Tag bis zum Tag 7, wenn neuroepitheliale Aggregate (Rosetten) sichtbar sein sollten.
- Am Tag 7, Schicht-Matrix (oder Laminin), wie in Schritt 2.2 beschrieben, auf jedes erforderliche Platten- oder Tellerformat: 96-Well-Platten (100 & mgr; l / Vertiefung), 24-Well-Platten (250 & mgr; l / Vertiefung), 12- Well-Platten (500 μl / Well), MEA-Chips (für elektrische Aktivität, 1 mL / Single-Well-Chip) oder 60 mm Petri DisheS (4 ml / Teller).
- Inkubieren Sie die beschichteten Platten / Geschirr für mindestens 2 Stunden bei 37 ° C und 5% CO 2 .
- Rosettendissoziation und neuronale Differenzierung (Tage 8 → 28)
HINWEIS: Dieses Verfahren erfordert gute manuelle Fertigkeiten und Präzision. Um das Sammeln von mesodermalen und endodermalen Zellen zu vermeiden, sollten nur ektoderm rosettenartige Strukturen dissoziiert und gesammelt werden.- Am 8. Tag, schneide die rosettenähnlichen Strukturen in Fragmente unter einem stereoskopischen Mikroskop bei 10facher Vergrößerung in sterilen Bedingungen. Verwenden Sie eine 1-ml-Spritze mit einer 30G-Nadel. Beachten Sie, dass die Rosetten dazu neigen, sich leicht von der Schale zu lösen, wenn sie mit der Nadel berührt werden.
- Vervollständigen Sie die Ablösung der Rosettenfragmente mit einer 200-μl-Pipette.
- Übertragen Sie die Schale unter die laminare Strömungshaube und sammeln Sie die Rosettenfragmente und ihr Medium in ein 15 ml konisches Rohr mit einer 1, 2 oder 5 ml Pipette. Spülen Sie die Schale mit 2 ml NRI-Medium, um sich zu erholenAlle Fragmente.
- Spinne die Rosettenfragmente bei 112 xg für 2 min.
- Sauge den Überstand.
- Das Pellet vorsichtig in 1 ml 1x DPBS (ohne Calcium und Magnesium) resuspendieren und die Rosettenfragmente vorsichtig mit einer 1000 μl Pipette auf- und abpipettieren, um sie teilweise zu dissoziieren.
- Füge 4 ml komplettes NRI-Medium hinzu und zähle die Zellen mit Trypanblau und einem automatisierten Zellzähler (siehe Tabelle der Materialien )
HINWEIS: 20 μl Zellsuspension in 20 μl Trypanblau verdünnen. Dieser Schritt kann weggelassen werden, wenn die Zellen nicht in eine Einzelzellsuspension gebracht werden können. Wenn die Rosettenfragmente nicht vollständig dissoziiert sind, können Rosettenfragmente, die von etwa 50 EBs / 60-mm-Schalen abgeleitet sind, in 50 ml vollständigem NRI-Medium resuspendiert und plattiert werden, wie in Tabelle 1 angegeben. - Ansaugen der Standardmatrix (oder Laminin) Beschichtungslösung aus den Petrischalen, Platten und / oder MEA Chips (ab Schritt 2.3.7). Lass sie nicht trocknen
- Die Zellen im kompletten NRI-Medium nach dem Studienplan (ca. 15.000 Zellen / cm 2 , siehe Tabelle 1 für die Beschichtungsvolumenangaben) plattieren.
- Inkubieren Sie die Platten über Nacht bei 37 ° C und 5% CO 2 .
- Am Tag 10, führen Sie eine Gesamt-Medium-Änderung mit vollständigen neuronalen Differenzierung Medium (ND); Siehe die Tabelle der Materialien.
- Erneut das komplette ND-Medium zweimal pro Woche bis zum Tag 28.
- Charakterisieren Sie die neuronalen / Gliazellderivate, wie in Schritt 5 beschrieben (siehe Tabelle 2 für allgemeine Akzeptanzkriterien).
3. HiPSC-abgeleitete neuronale Stammzelle (NSC) Expansion und Differenzierung in gemischte Neuronen und Glia
HINWEIS: NSCs, die aus Rosettenfragmenten abgeleitet sind, können nach dem unten beschriebenen Verfahren erweitert und beibehalten werden (Abbildung 1 , Unterteil). Dies ermöglicht die Inkrementierung von thE Anzahl von Zellen für die Differenzierung und chemische Prüfung.
- Eine 60-mm-Petrischale (oder einen T-25-Kolben) mit 5 ml Standardmatrix-DMEM / F12-Beschichtungslösung beschichten und mindestens 2 h bei 37 ° C und 5% CO 2 inkubieren (wie in Schritt 2.2 beschrieben).
- Die Rosettenfragmente, die aus den Schritten 1-2 (siehe Schritt 2.4.) Gewonnen wurden, in einem konischen 15-ml-Röhrchen bei 112 xg für 2 min abreiben.
- Das Pellet vorsichtig in 5 ml des neuralen Induktionsmediums (NI) resuspendieren; Siehe die Tabelle der Materialien.
- Übertragen Sie die Zellen auf eine standardmatrixbeschichtete 60 mm Petrischale (oder T-25-Kolben).
- Kultur-Rosette-abgeleitete NSCs in Gegenwart von NI-Medium, erfrischen das Medium jeden zweiten Tag, bis die Zellen Konfluenz erreichen.
- Wenn konfluent, passieren Sie die NSCs wie in den folgenden Schritten beschrieben.
ANMERKUNG: Passage der NSCs etwa einmal pro Woche; Betrachten Sie frisch beschichtete Schalen, Flaschen oder Teller, je nach Studienplan. - Entfernen Sie das komplette NI Medium und GentlIch spüle die NSCs mit DPBS (ohne Kalzium und Magnesium).
- Fügen Sie 1,5 ml 0,05% Trypsin-EDTA vorgewärmt auf 37 ° C zu der 60-mm-Petrischale (oder T-25-Kolben), die die Zellen enthält, und legen Sie sie in den Inkubator für 1 min.
- Drücken Sie vorsichtig auf die Schale (oder den Kolben), um die Zellen abzutrennen.
- Man gibt 1,5 ml Trypsin-Inhibitor vorgewaschen auf 37 ° C und überführt die Zellen in ein 15-ml-Röhrchen.
- Spülen Sie die Petrischale (oder T-25-Kolben) mit einem gleichen Volumen NI-Medium (1,5 ml) und sammeln Sie das Volumen in der gleichen 15-ml-Röhre.
- Die Zellen bei 130 xg für 3 min zentrifugieren.
- Den Überstand entfernen und die Zellen in 1 ml vollständigem NI-Medium mit einer 1000-μl-Pipette vorsichtig resuspendieren.
- Weiterhin verdünnen Sie die Zellsuspension in 3 oder 4 ml komplettem NI-Medium und zählen Sie die Zellen mit Trypanblau und einem automatisierten Zellzähler.
- Die NSCs auf die 60-mm-Petrischale (oder T-25-Kolben) aus Schritt 3.1 mit einer Dichte von etwa 50.000 Zellen / cm 2 auftragen .
- Führen Sie einen ganzen mittleren Wechsel mit vollständigem NI-Medium jeden zweiten Tag durch.
- Charakterisieren Sie die Zellen für die Anwesenheit von neuronalen / Gliazellderivaten, wie in Schritt 5 beschrieben.
HINWEIS: NSCs können in gemischte Kulturen von Neuronen und Glia in komplettem ND-Medium (wie in den Schritten 2.4.11-2.4.13 beschrieben) unterschieden werden, wobei das vollständige ND-Medium zweimal pro Woche für 21 Tage erneuert wird.
4. HiPSC-abgeleitete NSC-Kryokonservierung und Auftauen
HINWEIS: Nach dem Passieren können NSCs nach diesem Verfahren eingefroren und nachgetaut werden.
- Zentrifuge passierte NSCs (ab Schritt 3.12) bei 130 xg für 3 min.
HINWEIS: Die Zellen sollten in Schritt 3.14 gezählt werden. - Die NSCs vorsichtig und langsam mit 3 x 10 6 / ml des Einfriermediums resuspendieren (siehe Tabelle der Materialien ).
- Die Zellen in geeigneten Fläschchen zur Kryokonservierung (ca. 0,5 mL = 1,5 x 10 6 / Durchstechflasche) aliquotieren.
- Legen Sie die Fläschchen in einen BehälterMit 2-Propanol gefüllt und den Behälter bei -80 ° C für mindestens 2 h und bis zu 2 Wochen gelegt.
- Übertragen Sie die Fläschchen in die Dampfphase eines flüssigen Stickstoffbehälters.
- Um die Zellkultur neu zu starten, tauchte 1 gefrorene Durchstechflasche in einem Wasserbad bei 37 ° C auf.
- Die Zellen in 7 ml vorgewärmtem vollständigem NI-Medium in einem 15-ml-Röhrchen unter Verwendung einer 1000-μl-Pipette vorsichtig sammeln.
- Die Zellen bei 130 xg für 3 min zentrifugieren.
- Den Überstand entfernen und die Zellen in 1 ml vollständigem NI-Medium mit einer 1000-μl-Pipette vorsichtig resuspendieren.
- Weiterhin verdünnen Sie die Zellsuspension in 3 oder 4 ml vollständigem NI-Medium und zählen Sie die Zellen mit Trypanblau und einem automatisierten Zellzähler (Anmerkung: Verdünnen Sie 20 μl Zellsuspension in 20 μl Trypanblau, die Lebensfähigkeit nach dem Auftauen sollte ≥ 80 sein %).
- Die NSCs in einer beschichteten 60-mm-Petrischale (oder T25-Kolben) mit einer Dichte von etwa 50.000 Zellen / cm 2 auftragen .
5. CharaCerisierung von hiPSC-abgeleiteten neuronalen und glialen Zellen
HINWEIS: Bei der Differenzierung können neuronale und gliale Derivate mit verschiedenen Techniken charakterisiert werden, wie sie in den folgenden Abschnitten beschrieben sind.
- Quantitative Echtzeit-PCR (qPCR) analysiert 10
- Spiessen Sie hiPSC Koloniefragmente, EBs und / oder NSCs bei 130 xg für 3 min.
- Resuspendieren des Zellpellets in 100 & mgr; l kaltem RNA-Lysepuffer, der in einem geeigneten Kit für die RNA-Extraktion bereitgestellt wird.
- Alternativ sammeln Sie neuronale / gliale Derivate direkt aus den Platten durch Absaugen des Mediums und Zugabe von kaltem RNA-Lysepuffer zu den Vertiefungen, um die Zellen zu sammeln.
- Isoliere die RNA nach der Anweisungen des Herstellers.
- Reverse-transkribieren 500 ng der gesamten RNA mit einem geeigneten Kit für RNA zu cDNA Retrotranskription.
- Führen Sie qPCR Reaktionen in doppelter Ausführung mit entsprechenden Master Mix und Primer(Siehe Tabelle der Materialien ).
- Aufzeichnung der Fluoreszenzemission in Echtzeit: 45 Zyklen mit Primern Glühen bei 60 ° C.
- Normalisieren Sie die relativen RNA-Mengen zu GAPDH und β-Actin als Referenzgene und verwenden Sie undifferenzierte hiPSCs oder unbehandelte Zellen für die Kalibrierbedingungen (ΔΔCt-Methode). Alternativ verwenden Sie eine andere geeignete Methode.
- Immunzytochemie und hochauflösende Bildgebung (HCI) 6 , 11
- Fixieren Sie die hiPSC-Kolonien, NSCs und / oder neuronalen / glialen Derivate mit kaltem 4% Paraformaldehyd für 15 min bei Raumtemperatur.
- Die Zellen vorsichtig in 1X PBS waschen und die Platten bei 4 ° C für bis zu 1 Monat aufbewahren.
- Wenn sie zur Färbung bereit sind, permeabilisieren die Zellen in Permeabilisierungspuffer (1x DPBS mit 0,1% Triton-X-100 und 3% BSA) für 15 min bei Raumtemperatur.
- Entfernen Sie die Permeabilisierung buFax und inkubieren die Zellen in Blockierungspuffer (3% BSA / 1X DPBS) für 15 min bei Raumtemperatur, um die unspezifische Bindung von Antikörpern zu verhindern.
- Entfernen Sie den Blockierungspuffer und inkubieren Sie die Zellen über Nacht bei 4 ° C in Blockierungspuffer, der geeignete primäre Antikörper enthält (siehe Tabelle der Materialien ).
- Waschen Sie die Zellen 3 mal mit 1x PBS.
- Inkubieren Sie die Zellen für 45 min bei Raumtemperatur in Blockierungspuffer, der fluorchrom-konjugierte sekundäre Antikörper enthält (siehe Tabelle der Materialien ), Gegenfärbung der Kerne mit DAPI-Farbstoff.
- Quantifizieren Sie die mittlere Fluoreszenzintensität und die relativen Prozentsätze der Zelltypen mit einer geeigneten hochauflösenden Imaging-Plattform, falls vorhanden (siehe Tabelle der Materialien ).
ANMERKUNGEN: Um den Intensitätsgrad des fluoreszierenden Hintergrundes zu bestimmen, inkubieren einige Zellen / Vertiefungen mit sekundären Antikörpern alleine. Die Durchflusszytometrische Analyse von leben (nicht fixiert), undIfferentiated hiPSCs können durchgeführt werden, um die Expression von PSC-spezifischen Markern wie SSEA4 (siehe Tabelle der Materialien ) zu bewerten . Undifferenzierte hiPSC-Kolonien können auf alkalische Phosphatase-Aktivität unter Verwendung von handelsüblichen BCIP / NBT-Kits nach den Anweisungen des Herstellers analysiert werden (siehe Tabelle der Materialien ). Zusätzlich können Reverse-Phase-Protein-Array- (RPPA-) Assays und Analysen durchgeführt werden, wie in Referenz 12 beschrieben (für eine Liste von getesteten Antikörpern siehe die Tabelle der Materialien ).
- Elektrophysiologische Messungen 13
- Stellen Sie die dissoziierten Rosettenfragmente (nach 7 DIV) oder NSCs aus Rosetten auf beschichteten Multielektroden-Arrays (MEAs, siehe Tabelle der Materialien ) in komplettem ND-Medium (~ 1 x 10 5 Zellen / Single-Well-MEA-Chip) ab.
- Differenzieren Sie die Zellen für 3 Wochen in komplettem ND-Medium, erfrischend thE mittel zweimal pro Woche
- Am Ende der Differenzierung die MEA-Chips mit einer semipermeablen Membran unter einer laminaren Strömungshaube abdichten, um die Kulturen für wiederholte Messungen steril zu halten.
- Ersetzen Sie eine der Elektroden durch eine Bodenreferenz, so dass Aufnahmen von den übrigen Elektroden möglich sind.
- Die mittlere Brennrate (MFR, Anzahl der Spikes / min) mit einem MEA-Verstärker mit der integrierten Temperaturregelung auf 37 ° C und 5% CO 2 einstellen.
- Erfassen von Peaks aus den MEA-Rohdaten unter Verwendung der Schwellengrenze von -4,7σ (σ stellt die Standardabweichung des basalen Rauschens dar).
- Verarbeiten Sie die Nachaufzeichnungsdaten mit einer geeigneten Software.
Ergebnisse
Charakterisierung von undifferenzierten hiPSCs
Zur Beurteilung des Phänotyps der hiPSCs sollte die Analyse der Kolonie / Zellmorphologie, die Bestimmung von PSC-spezifischen Markern und die Untersuchung der Genexpression und der alkalischen Phosphataktivität durchgeführt werden. Undifferenzierte hiPSCs sollten rund sein, mit großen Nukleolen und ohne reichhaltiges Zytoplasma. Die Mehrheit der Kolonien sollte durch eine flache und dicht gepackte Morphologie charakterisiert werden, die einen undifferenzierten Phänotyp anzeigt (Abbildung 2A und Abbildung 2B ). Zusätzlich sollten mehr als 80% der Kolonien für die alkalische Phosphataseaktivitätsfärbung positiv sein (Abbildung 2C ).
Etwa 80% der Zellen sollten für klassische pluripotenzbezogene Marker, wie Oct4, S, positiv seinSEA3, SSEA4 und Tra1-60 (Abbildung 2D-H ), wie durch Immunzytochemie und Durchflusszytometrie gezeigt, während Prozentsätze von Nestin + und β-III-Tubulin + Zellen signifikant niedrig sein sollten (etwa 8% bzw. 3% Wie in Fig. 2H gezeigt ). Diese Ergebnisse sollten über Passagen reproduzierbar sein.
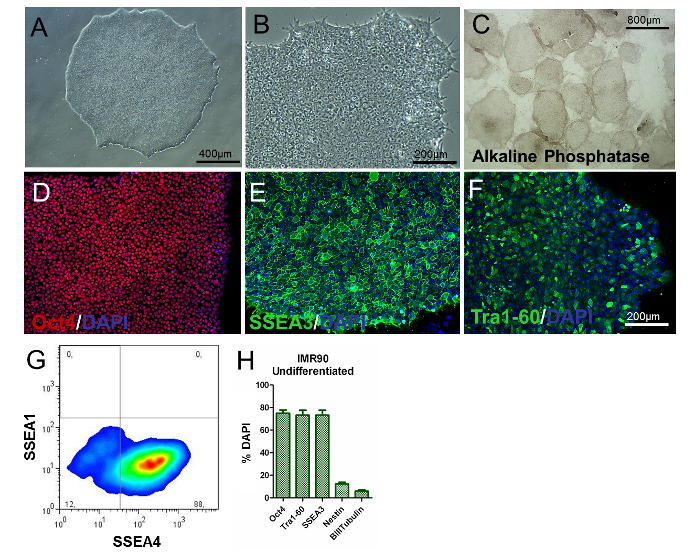
Abbildung 2. Charakterisierung von undifferenzierten IMR90-hiPSCs. (A und B) Repräsentative Phasenkontrastbilder (10X und 20fache Vergrößerungen) von undifferenzierten IMR90-hiPSC-Kolonien. (C) repräsentative Bilder von alkalischen Phosphatase-gefärbten Kolonien (4fache Vergrößerung). (DF) Repräsentative immunzytochemische Bilder von (D) Oct4 (rot), (E) SSEA3 (grün) und (F) TrA1-60 (grün). (G) Repräsentative Punktdarstellung von SSEA1 (CD15) und SSEA4-Färbung, analysiert durch Durchflusszytometrie. (H) Das Balkendiagramm zeigt die Prozentsätze von Okt4 + (~ 75 - 80%), Tra1-60 + (~ 75 - 80%), SSEA3 + (~ 75 - 80%), Nestin + (~ 10 - 15% ) Und β-III-Tubulin + (~ 3 - 7%) Zellen, mit DAPI gegengefärbt und durch HCI quantifiziert, mit einem Mittelwert von 3 bis 5 biologischen Replikaten ± der SEM (Grafik von Referenz 6 modifiziert). Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Bewertung der Pluripotenz über EB-Bildung
HiPSCs sind pluripotent, was bedeutet, dass sie drei Keimschicht-verwandte Gene unter geeigneten Bedingungen exprimieren. Um hiPSC Pluripotenz zu beurteilen, ist es möglich, eine gemeinsame anzuwendenAnsatz auf der Grundlage einer spontanen EB-Bildung, die die Bildung der drei Keimschichten 14 induziert. Analysen von Keimschicht-spezifischen Genen müssen eine zeitabhängige Erhöhung des Endoderms (α-Fetoprotein (AFP) und Cytokeratin 18 (KRT18)), Ektoderm (Nestin, SRY-Box 1 (Sox1) und gepaartes Feld 6 (Pax6) ) Und Mesoderm (natriuretisches Peptid A (NPPA) und Brachyury-T) -bezogene Genexpression ( Fig. 3A und Fig. 3B ); Siehe die Tabelle der Materialien.
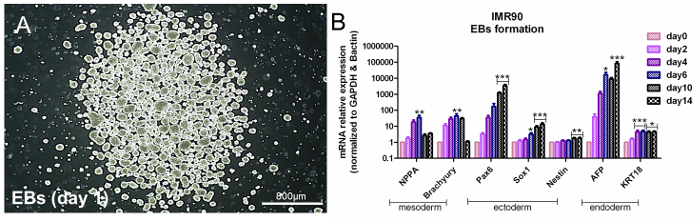
Abbildung 3. Bewertung der Pluripotenz durch EB-Bildung (A) Repräsentatives Phasenkontrastbild von EBs am Tag 1. (B) Das Balkendiagramm zeigt qPCR-Analysen von mesodermalem (NPPA und Brachyury), ektodermalem (Pax6, Sox1 und Nestin) und endodermalem (AFP und KRT18 ) Gene, die auf die Referenzgene, β-Actin und GAPDH normiert sind, und auf undifferenzierte Zellen kalibriert (Tag 0). Dies ist das ΔΔCt-Verfahren mit einem Mittelwert von 5 unabhängigen Analysen ± der SEM * p <0,05, ** p <0,01, p <0,001; Diagramm, das von Referenz 6 geändert wurde. Klicken Sie hier, um eine größere Version dieser Figur anzuzeigen .
Induktion der neuronalen und glialen Differenzierung
IMR90-iPSCs können in gemischte Kulturen von post-mitotischen Neuronen und Gliazellen nach den in Abbildung 1 und in den Protokollabschnitten zusammengefassten Schritten unterschieden werden. 5-8 Tage nach dem Plattieren der EBs auf Standardmatrix- oder Laminin-beschichtete Schalen in Gegenwart von komplettem NRI-Medium sollten rosettenartige Strukturen sichtbar werden (= "Xfig"> Abbildung 4A). Rosetten zeichnen sich durch die Anwesenheit von Nestin + -Zellen aus (neuronale Vorläufer, ~ 90%), mit wenigen β-III-Tubulin + -Zellen (engagierte neuronale Zellen, ~ 5 - 10%), die letzteren meist hauptsächlich an der Peripherie der Rosetten ( Abbildung 4B , Rosetten am Tag 12).
Bei der Rosettendissoziation und der Ablage auf Laminin- oder Standardmatrix-beschichtete Schalen oder Platten in Gegenwart eines vollständigen ND-Mediums beginnen Zellen, sich in gemischte Kulturen von Neuronen und Glia zu differenzieren, wobei sie fortschreitend Cluster von neuronalen Zellkörpern bilden, die durch Bündel von Neuriten verbunden sind ( Fig 4C und Fig. 4D ). Ähnliche Ergebnisse sollten bei der Analyse von neuronalen Populationen erhalten werden, die durch Erweitern von Rosetten-abgeleiteten NSCs erhalten wurden, und sie in Neuronen und Glia differenzieren. NSCs aus Rosetten erweitert werden sollte nEstin + (Abbildung 4E , Insert mit Nestin + Zellen).
Nach 21 Tagen der Differenzierung sollten die Zellen für β-III-Tubulin positiv sein; NF200; Tau; Und MAP2, der Spätmarker von Dendriten (Abbildung 4D , 4F und Abbildung 4H) mit mindestens 10 - 15% der für das gliale fibrilläre saure Protein (GFAP) positiven Zellen, einen astroglialen Marker (Abbildung 4G und Abbildung 4H) . Darüber hinaus sollten ~ 20 - 30% der Zellen die Expression von Nestin nach der Differenzierung beibehalten (Abbildung 4H ). Es ist wichtig zu bedenken, dass der Prozentsatz jedes Zelltyps ( dh Neuron, Astrozyt, Nestin + Zellen) über Passagen variieren kann und eine benutzerabhängige Variabilität beobachtet werden kann.
Durch die Analyse spezifischer neuronaler Subpopulationen, GABAergic Neuronen repräsentierenGesendet ~ 15 - 20% der gesamten Zellen, dopaminergen Neuronen ~ 13 - 20% und glutamatergen Neuronen ~ 35 - 42%, wie durch Immunfärbung für Gamma-Aminobuttersäure (GABA), Tyrosin-Hydroxylase (TH) und vesikulärem Glutamat gezeigt Transporter 1 (VGlut1) (siehe die repräsentative Quantifizierung in Abbildung 4H ). Die Induktion der Differenzierung kann auch durch die Analyse von Pluripotenz-verwandten Markern ( z. B. Oct4, Tra1-60 und SSEA3) beurteilt werden, die in differenzierten Zellen gegenüber unsifferenzierten hiPSCs signifikant herunterreguliert werden sollten (nicht gezeigt, siehe Referenz 6). Dies kann auch durch die Analyse der Genexpression durch qPCR bestätigt werden, was auf eine Abnahme von Oct4 und Nanog und die Hochregulierung von neuronalen Genen, wie z. B. neurales Zelladhäsionsmolekül 1 (NCAM1) und Mikrotubuli-assoziiertes Protein 2 (MAP2), hinweisen sollte; Das präsynaptische Gen, Synaptophysin (SYP); Und das post-synaptische Gen, Mikrotubuli-assoziiertes Protein Tau (MAPT), wie in Starke Klasse = "xfig"> Abbildung 4I. Des Weiteren wurden dopaminerge (TH und NR4A1), noradrenerge (PHOX2A und PHOX2B), glutamaterge (NARG2, GRIA1 und GAP43), GABAergic (GABRA1 und GABRA3), motorische Neuronen (ISL1 und LHX3) und cholinerge (SLC5A7- und SLC18A13) -bezogene Gene Zu hochregulierten neuronalen Zellen im Vergleich zu undifferenzierten Zellen führen (Abbildung 4J ).
Die Analyse der spontanen elektrischen Aktivität mittels MEA ist ein wertvolles Auslesen, um die Funktionalität des neuronalen Netzwerks in differenzierten hiPSCs zu beurteilen. Am Ende der Differenzierungsperiode sind neuronale Derivate im Allgemeinen durch eine mittlere Brennrate (MFR) von mindestens 60 Spikes / min gekennzeichnet (siehe die repräsentative Rasterkurve in Abbildung 4K ). Es werden jedoch keine Bursts beobachtet.
55702fig4.jpg "/>
Abbildung 4. Differenzierung von IMR90-hiPSCs in gemischte Kulturen von Neuronen und Glia. (A und B) Repräsentative Bilder von Rosetten nach 7 DIV (A) und nach 12 DIV (B) , gefärbt für Nestin (grün) und β-III-Tubulin (rot)). (C und D) Repräsentative Bilder von differenzierten Zellen nach 22 DIV (C) und 28 DIV (D) , gefärbt für β-III-Tubulin (rot) und NF200 (grün)). (E) Repräsentatives Bild von NSCs, die aus Rosettendissoziation und -expansion abgeleitet sind (Insert zeigt Nestin + Zellen, Rot). (F und G) Repräsentative Bilder von neuronalen Zellen ( F , gefärbt für NF200 (rot) und Tau (grün)) und Gliazellen ( G , gefärbt für GFAP (rot)), differenziert von NSCs (nach 21 DIV). (H) Quantifizierung von Nestin, MAP2, GFAP, Gamma-Aminobuttersäure (GABA), vesikulärer Glutamattransporter 1 (VGlut1) und Tyrosinhydroxylase (TH) -positiven Zellen durch HCI, Vergleich von IMR90-hiPSC-Derivaten und Zellen, die von IMR90-hiPSC-abgeleiteten NSCs unterschieden wurden (Grafik, die aus Referenz 7 modifiziert wurde). (I und J) Balkendiagramme, die qPCR-Analysen von Pluripotenzgenen (Oct4 und Nanog) und neuronalen Genen (NCAM1, MAP2, SYP und MAPT) (I) und von dopaminergen (TH und NR4A1), noradrenergen (PHOX2A und PHOX2B) (NARG2, GRIA1 und GAP43), GABAergic (GABRA1 und GABRA3), Motorneuron (ISL1 und LHX3) und cholinergische (SLC5A7 und SLC18A13) -bezogene Gene (J) . Alle Analysen wurden auf die Referenzgene, β-Actin und GAPDH normiert und auf undifferenzierte Zellen (grüne Balken) kalibriert. Dies ist das ΔΔCt-Verfahren mit einem Mittelwert von 5 unabhängigen Analysen ± der SEM * p <0,05, ** p <0,01, p <0,001. Die Graphen in I und J wurden aus Referenz 6 modifiziert. (K) Repräsentative Rasterkurve von IMR90-NSC-abgeleiteten NeurOns (die Aufzeichnung wurde für mindestens 600 s durchgeführt, die vertikalen Balken repräsentieren einzelne Spikes). Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Eine spezifische Signatur von neuronalen Markern wird in differenzierten IMR90-hiPSCs hochreguliert
In dem neuen Toxizitätstest-Paradigma ist es wichtig, die molekularen und zellulären Ereignisse, die innerhalb einer Zelle auftreten, nach der Exposition gegenüber einem gegebenen Toxizmittel zu definieren. Daher ist es wichtig zu charakterisieren, welche Signalwege in dem zu untersuchenden zellularen Modell aktiviert und / oder hochreguliert werden.
Kommerziell erhältliche Arrays für die Analysen der Proteinkinase-Genexpression können verwendet werden, um undifferenzierte HiPSCs gegenüber differenzierten Zellen zu vergleichen. UnterschiedlichDie IMR90-hiPSCs unterliegen der Hochregulierung von Genen, die an der Kontrolle von Neurotrophin-Rezeptoren beteiligt sind, Axon-Guidance-Regulation, Neurit-Auswuchsmodulation, Glial-Neurotroph-Faktor (GDNF) -Rezeptoren, Knochenmorphogenetisches Protein (BMP) / TGF-beta-Weg und Thrombozyten- Abgeleiteten Wachstumsfaktor (PDGF) -Rezeptoren ( Fig. 5A und Fig. 5B ).
Die Analyse von RPPA zeigt die Hochregulierung einer spezifischen neuronalen Signatur in differenzierten IMR90-hiPSCs. Insbesondere werden die Erk / CREB-, Akt / PDK1 / mTOR- und Notch1-Signalwege bei der Differenzierung aktiviert (Abbildung 5C ).
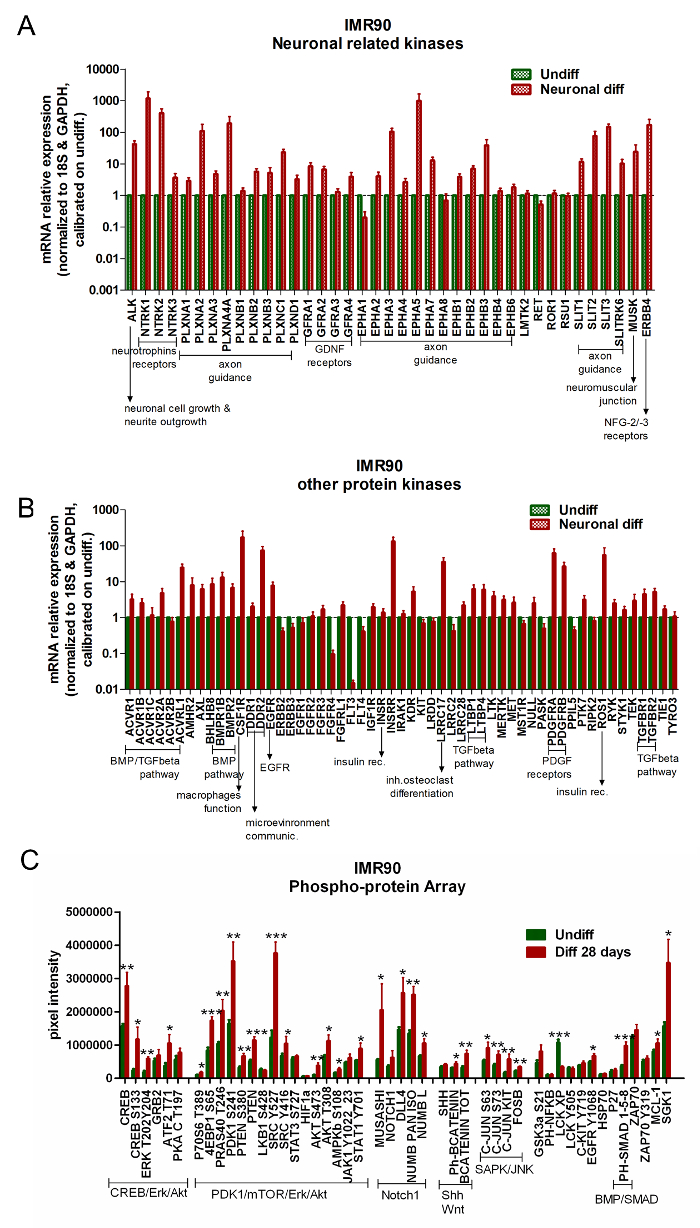
Abbildung 5. Differenzierte neuronale und gliale Zellen zeigen die Aktivierung von Neuron-bezogenen Pfaden. (EINUnd B) Balkendiagramme berichten über die qPCR-Analysen von neuronbezogenen Kinasen (A) und anderen kinasebezogenen Genen (B) . Die Genexpressionsdaten wurden auf die Referenzgene 18S und GAPDH (die in dem Array bereitgestellt wurden) normalisiert und auf undifferenzierte Zellen kalibriert. Für diese Analysen wurde ein Gen als signifikant hochreguliert angesehen, wenn seine Expression mindestens 2x höher war als in undifferenzierten Zellen (2 -ΔΔCt ≥ 2); Mittelwert von 3 unabhängigen Analysen ± der SEM). (C) Das Balkendiagramm zeigt die absoluten Proteinquantifikationen mittels RPPA-Analyse, wobei differenzierte (rote Balken) und undifferenzierte Zellen (grüne Balken) verglichen werden. Die Proteine, die zu den gleichen Signalwegskaskaden gehören, sind wie folgt zusammengefaßt: CREB / Erk / Akt, PDK1 / mTOR / Erk / Akt, Notch1, Shh und Wnt, SAPK / JNK und BMP / SMAD. Mittelwert ± der SEM von 4 unabhängigen Analysen. * P <0,05, ** p <0,01, p <0,001; Graph modifiziert fRom Referenz 6. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
IMR90-hiPSC-abgeleitete neuronale / gliale Kulturen können verwendet werden, um die Effekte von Rotenon zu beurteilen
Rotenon, ein Inhibitor des Komplexes I der mitochondrialen Atmungskette, ist bekannt, um oxidativen Stress zu verursachen, indem er die Aktivierung des Nrf2-Weges auslöst. Unter ruhigen Bedingungen ist Nrf2 im Zytoplasma von Keap1 (Kelch-ähnliches ECH-assoziiertes Protein 1), einem Nrf2-Repressor, verankert, der die Nrf2-Ubiquitinierung und Proteolyse 15 erleichtert. Bei der Induktion von oxidativem Stress translatiert sich Nrf2 in den Zellkern und aktiviert die Expression von Nrf2-ARE-Zielgenen 16 .
IMR90-hiPSC-abgeleiteten Neuronen undD Gliazellen können verwendet werden, um die Effekte von Rotenon auf die Nrf2-Aktivierung zu beurteilen, indem die Zellen für 24 h verschiedenen Konzentrationen von Rotenon ( z. B. 1, 10 und 100 nM) ausgesetzt werden. Diese Konzentrationen wurden nach früheren Studien 17 , 18 festgelegt.
Bei diesen Konzentrationen und Zeiten der Exposition führte Rotenon nicht zu einer Zytotoxizität, wie die Quantifizierung von lebenden DAPI + -Zellkernen zeigt (Abbildung 6A ). Rotenon induzierte Nrf2-Nuklear-Translokation, besonders nachdem die Zellen 100 nM Rotenon ausgesetzt wurden (Abbildung 6B und Abbildung 6E ). Bei der gleichen Konzentration wurde eine signifikante Abnahme des zytoplasmatischen Keap1 beobachtet (Abbildung 6C ), zusammen mit einer Zunahme sowohl der NAD (P) H-Chinon-Oxidoreduktase 1 (NQO1) als auch des Sulfiredoxins 1 (SRXN1), zwei Nrf2-Ziel enZymes 19 , 20 ( Fig. 6D ).
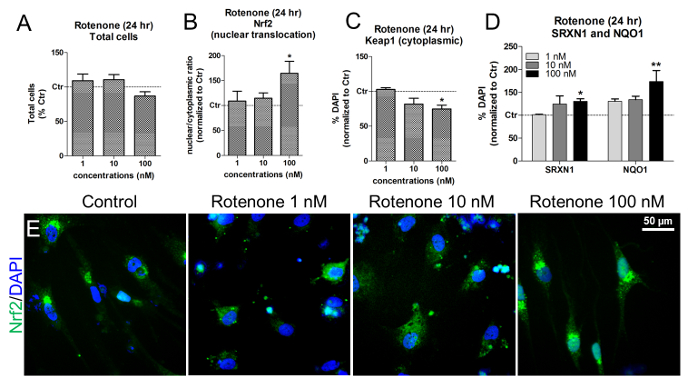
Abbildung 6. Effekte von Rotenon auf Nrf2 Nukleare Translokation, Keap1, SRXN1 und NQO1 Proteinspiegel. (A) Quantifizierung von lebenden DAPI + -Zellen ( dh nicht-pyknotischen Kernen) bei 24 h Behandlung mit 1, 10, 100 nM Rotenon und normalisiert auf unbehandelte Zellen (Kontrolle, Ctr). (B) Nrf2-Protein-Nuklear-Translokation ( dh nukleare / zytoplasmatische Verhältnisse) nach 24 h Exposition gegenüber Rotenon, beurteilt durch Messungen der Fluoreszenzintensität mittels HCI-Analyse. (C) Quantifizierung der zytoplasmatischen Keap1-Proteinspiegel bei der Rotenon-Behandlung, bewertet durch HCI-Analyse. (D) Quantifizierung der NAD (P) H-Chinon-Oxidoreduktase 1 (NQO1) und SulfiRedoxin 1 (SRXN1) mittels Immunfluoreszenz und HCI nach 24 h Behandlung mit Rotenon. (E) Repräsentative Bilder der Nrf2-Protein-Lokalisation (grün). Alle Werte sind als Mittelwert ± SEM von 3 biologischen Replikaten dargestellt. * P <0,05, ** p <0,01; Abbildung, die von Referenz 7 geändert wurde. Klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Bei diesen Konzentrationen und Behandlungszeiten hat Rotenon auch einen konzentrationsabhängigen Anstieg des astroglialen (GFAP + ) Zellprozentsatzes hervorgerufen (Abbildung 7A und Abbildung 7B ), ohne die Anteile von NSCs (Nestin + ) und Neuronen (MAP2 + ) zu beeinflussen ( Abb 7B ). Durch Betrachten der Proportionen spezifischer neuronaler Subtypen wurde die Rotenon-Behandlung (10 nM und 100 nM)Signifikant die Anzahl der dopaminergen Neuronen (TH + ) (Abbildung 7C und D ), während sich die Prozentsätze von GABAergic (GABA + ) und glutamatergen (VGlut1 + ) Neuronen nicht änderten (Abbildung 7D ). Analog haben bisherige in vivo- und in vitro- Studien einen Rotenon-abhängigen und selektiven dopaminergen neuronalen Zelltod 21 , 22 , 23 beschrieben.
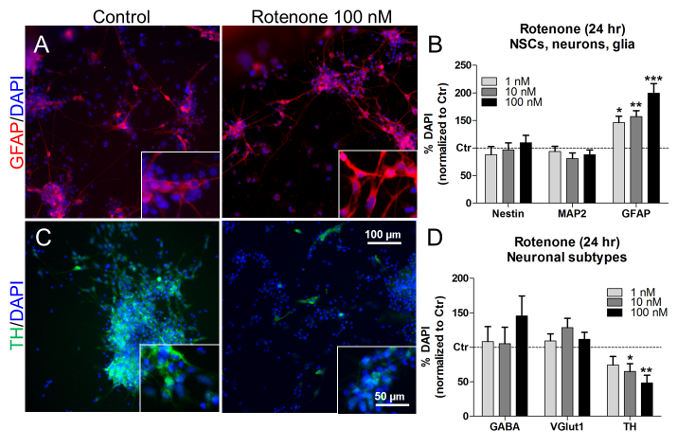
Abbildung 7. Effekte von Rotenon auf Gliazellen und dopaminergen Neuronen. (A) Repräsentative Bilder von GFAP + Zellen (rot), mit 40facher Vergrößerung in den Einsätzen, unbehandelt oder mit 100 nM Rotenon für 24 h behandelt. (B) Quantifizierung Von Nestin + , MAP2 + und GFAP + Zellen, normalisiert auf unbehandelte Zellen (Kontrolle, Ctr). (C) Repräsentative Bilder von dopaminergen TH + Neuronen (grün), mit 40facher Vergrößerung in den Einsätzen, unbehandelt oder mit 100 nM Rotenon für 24 h behandelt. (D) Quantifizierung von GABA + , VGlut1 + und TH + neuronalen Zellen, normalisiert auf unbehandelte Zellen (Ctr). Alle Werte sind als Mittelwert ± SEM von 3 biologischen Replikaten dargestellt. * P <0,05, ** p <0,01, p <0,001; Abbildung, die von Referenz 7 geändert wurde. Klicken Sie hier, um eine größere Version dieser Figur zu sehen.
Die statistische Signifikanz wurde durch eine einseitige ANOVA mit dem mehrfachen Vergleichstest von Dunnett als Post-Test (Vergleich aller Spalten gegenüber der Kontrollspalte)Xref "> 24 oder durch zwei-tailed ungepaarten oder gepaarten t-Test nach der Art der Analyse Alle Daten stellen den Durchschnitt von mindestens drei biologischen Replikaten ± der Standardfehler des Mittelwerts (SEM) dar. Ein Sternchen über einem Balken zeigt an Ein signifikanter Unterschied zur Kontrollgruppe. * P <0,05, ** p <0,01, *** p <0,001.
Tabelle 1.
Anmerkung zur dissoziierten Rosettenbeschichtungsdichte: Wenn Rosettenfragmente nicht vollständig dissoziiert aussehen, um eine Zellplattierungsdichte von etwa 15.000 Zellen / cm 2 zu erreichen, können dissoziierte Rosettenfragmente aus etwa 50 EBs / 1 x 60mm-Schale resuspendiert werden 50 mL komplettes NRI-Medium und plattiert wie folgt (je nach Plattenformat):
| Multiwell Platten / MEA | Wachstumsfläche (cm 2 / gut) | Volumen der Zellsuspension auf Platte pro Well (oder MEA-Chip) | Maximale Anzahl Platten, die plattiert werden können (mit 50 ml Zellsuspension) |
| 96 Brunnen | 0,3 | 100 ul | 5 |
| 48 Brunnen | 0,7 | 220 ul | 4 |
| 24 Brunnen | 2 | 625 ul | 3 |
| 12 Brunnen | 4 | 1,25 ml | 3 |
| 6 Brunnen | 10 | 3,125 ml | 2 |
| Single gut MEA Chip | 3.5 | 1,1 ml | 45 |
Tabelle 2: Akzeptanzkriterien
| Marker /Antikörper | Prozentsatz (auf DAPI + (lebende) Zellen) nach 28 DIV |
| B-III-Tubulin (Tuj1) | 35-45% |
| MAP2 | 50-60% |
| NF200 | 45-55% |
| GFAP | 10-25% |
| Nestin | 15-25% |
Diskussion
Diese Arbeit beschreibt ein robustes und relativ schnelles Protokoll für die Differenzierung von IMR90-hiPSCs in post-mitotische Neuronen und Gliazellen. Bisher veröffentlichte neuronale Differenzierungsprotokolle auf der Basis von hESCs und hiPSCs ergeben in der Regel hohe Prozentsätze der neuronalen Vorläufer 25 , 26 und eine signifikante Anzahl von neuronalen Zielzellen 27 , 28 , 29 , 30 , 31 , 32 , 33 . Analog dazu eignet sich das hier beschriebene Differenzierungsprotokoll zur Erzeugung heterogener Kulturen von GABAergischen, glutamatergen und dopaminergen Neuronalzellen zusammen mit Glia und einem diskreten Anteil an Nestin + Zellen. Die Anwesenheit von glutamatergen (~ 35-42%) und GABAergischen (~ 15-20%) neuralen Zellen deutet darauf hin, dassDiese Kultur besitzt Vorhirn, kortikale Merkmale und das Vorhandensein einer diskreten Anzahl von dopaminergen Neuronen (~ 13-20%) kann auch auf die Mittelhirnspezifität hindeuten. Darüber hinaus kann sich die Dauer eines bescheidenen Anteils an Nestin + Zellen als geeignet für das Studium der Neurogenese und die möglichen Auswirkungen von Chemikalien auf NSCs erweisen, die in erster Linie sowohl auf den Hippocampus als auch auf die subventrikuläre Zone (SVZ) des Vorderhirns beschränkt sind. Weitere immunzytochemische und Genexpressionsanalysen würden dazu beitragen, die regionale Spezifität der differenzierten Zellderivate besser zu definieren.
Die beiden kritischsten Schritte des in diesem Dokument beschriebenen Differenzierungsprotokolls sind: (i) das Schneiden von hiPSC-Kolonien in homogene Fragmente (die für die Erzeugung von EBs mit homogenen Größen kritisch sind) und (ii) das Schneiden von neuroektodermalen Strukturen (Rosetten) ) Für die NSC-Differenzierung, die eine beträchtliche manuelle Fertigkeit erfordertUnd Präzision, um das Sammeln von mesodermalen und endodermalen Zellen zu vermeiden, die die Proportionen von Neuronen und Gliazellen, die bei der Differenzierung erhalten werden, verringern können.
Es ist entscheidend, die Phänotypen der Zellen während der Expansion (als undifferenzierte Kolonien oder NSCs) und während aller Differenzierungsschritte zu charakterisieren. Insbesondere sollten die Gen- und Protein-Expressionsprofile der neuronalen / glialen Zellderivate eine Regulierung und Aktivierung von neuronbezogenen Signalwegen aufweisen, während die Expression von Pluripotenzmarkern verringert werden sollte.
Die Erzeugung von EBs und neuroektodermalen Derivaten (Rosetten) kann manuell anspruchsvoll und anfällig für Variabilität sein. Aus diesem Grund haben wir ein Protokoll für die Erweiterung von Rosetten-abgeleiteten NSCs und deren weitere Differenzierung in neuronale / gliale Zellen entwickelt.
Mögliche Einschränkungen dieses Differenzierungsprotokolls sind hauptsächlich (i) der relativ geringe Prozentsatz von dIfferentiierten Gliaderivaten und (ii) dem Mangel an reifen neuronalen Netzwerkfunktionen (wie durch den Mangel an Bursts gezeigt). Darüber hinaus können spezifische Subpopulationen von Astrozyten als primäre Vorläufer oder NSCs 35 fungieren. Während in dieser differenzierten Zellkultur (Daten nicht gezeigt) Nestin / GFAP-Doppel-Positiv-Zellen beobachtet wurden, wird vermutet, dass die GFAP + -Zellen in diesen Mischkulturen astrozytische Vorläufer und Astrozyten sind. Es ist plausibel, dass durch die Verlängerung der Zeit der Differenzierung die Anzahl der Astrozyten ansteigen kann und ihre Morphologie reifer werden kann, wie bereits in früheren Arbeiten von Zhangs Gruppe 36 , 37 angedeutet.
Im neuen Toxizitätstest-Paradigma ist das Wissen über chemisch-induzierte Störungen der biologischen Wege von größter Bedeutung bei der Beurteilung der chemischen Belastbarkeit. Daher sollten in vitro Testsysteme in der Lage sein,Beziehen sich auf Nebenwirkungen auf Störungen der Signalwege, nach dem Konzept des negativen Ausgangspfades (AOP). Als Proof-of-Concept kann Rotenon verwendet werden, um die Aktivierung des Nrf2-Weges zu beurteilen, der an der zellulären Abwehr gegen oxidativen oder elektrophilen Stress 38 beteiligt ist, und oxidativer Stress ist ein wichtiges und gemeinsames Schlüsselereignis in verschiedenen AOPs, Entwicklung und erwachsene Neurotoxizität 39 .
Rotenon sollte die Aktivierung des Nrf2-Weges hervorrufen, was durch die nukleare Translokation von Nrf2-Protein und eine erhöhte Expression von Nrf2-Ziel-Enzymen, einschließlich NQO1 und SRXN1, nachgewiesen werden kann. Es wurde gefunden, dass Rotenon eine dosisabhängige Erhöhung der GFAP-Proteinspiegel induziert, was auf die Astrozytenaktivierung 40 , 41 hindeutet. Rotenon verringert auch die Anzahl der dopaminergen (TH + ) - Zellen, die mit der Prävention übereinstimmtIn vitro und in vivo Studien, die den Rotenon-abhängigen dopaminergen Zelltod zeigen, da diese Art von Neuron besonders empfindlich gegenüber oxidativem Stress ist 21 , 22 , 23 .
Abschließend ist dieses hiPSC-abgeleitete neuronale und gliale Zellkulturmodell ein wertvolles Instrument, um die neurotoxischen Effekte von Chemikalien zu beurteilen, die oxidativen Stress hervorrufen, was zu einer Aktivierung von Nrf2 führt. Da dieses Differenzierungsprotokoll die Erzeugung von gemischten Kulturen von neuronalen Zellen (GABAergische, dopaminerge und glutamaterge Neuronen) und Astrozyten ermöglicht, kann es sich als geeignet erweisen, das Übersprechen zwischen Neuronen und Glia in physiologischen und pathologischen Zuständen zu untersuchen, wie bei neurodegenerativen Erkrankungen ( ZB Parkinson-Krankheit). Darüber hinaus kann die Anwesenheit eines signifikanten Anteils an NSCs dazu beitragen, die möglichen Auswirkungen von Chemikalien auf neuronales Prog zu bewertenEnitoren, von denen bekannt ist, dass sie das Hauptziel von chemisch induzierten Mutationen oder Virusinfektionen sind 42 .
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Die Autoren danken Dr. Marc Peschanski (I-Stem, Évry, Frankreich) für die Bereitstellung der IMR90-hiPSCs; Dr. Giovanna Lazzari und Dr. Silvia Colleoni (Avantea srl, Cremona, Italien); Dr. Simone Haupt (Universität Bonn, Deutschland); Dr. Tiziana Santini (Italienisches Institut für Technologie, Rom), für die Beratung bei der Immunfluoreszenzfärbung; Dr. Benedetta Accordi, Dr. Elena Rampazzo und Dr. Luca Persano (Universität Padua, Italien) für ihre Beiträge zur RPPA-Analyse und Antikörper-Validierung. Finanzierung: Diese Arbeit wurde durch das von der EU finanzierte Projekt "SCR & Tox" (Fördervereinbarung Nr. 266753) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Complete hiPSC medium: | |||
| mTeSR1 Basal Medium | Stem Cell Technologies | 05851 | (Step 1.2.6). Complete mTeSR1 is stable when stored at 2 - 8°C for up to 2 weeks. 5X Supplements can be dispensed into working aliquots and stored at -20°C. Use frozen aliquots within 3 months. |
| mTeSR1 5X Supplements | Stem Cell Technologies | 05852 | |
| Matrigel hESC-qualified Matrix | Corning | 354277 | 1:100 (Step 1.1). Thaw Matrigel on ice, prepare 200 ul aliquots and store them in -80°C. For coating, dilute 200ul aliquot in 20 ml of DMEM/F12 medium. |
| CryoStem Freezing Medium | Stemgent | 01-0013-50 | Freeze ~ 100 fragments/250 ul/vial (Step 1.2.1) |
| Name | Company | Catalog Number | Comments |
| hiPSC EB medium: | |||
| Knockout DMEM | Thermo-Fisher | 10829-018 | (Step 2.1.7) |
| Knockout Serum Replacement (KOSR) | Thermo-Fisher | 10828-028 | 20% final concentration (Step 2.1.7) |
| Non-Essential Amino Acids | Thermo-Fisher | 11140-035 | (Step 2.1.7) |
| Penicillin/Streptomycin | Thermo-Fisher | 15140-122 | 50 U/mL final concentration (Step 2.1.7) |
| L-Glutamine 200 mM Solution | Thermo-Fisher | 25030-081 | 2 mM final concentration (Step 2.1.7) |
| β-Mercaptoethanol | Thermo-Fisher | 31350-010 | 50 µM final concentration (Step 2.1.7) |
| Name | Company | Catalog Number | Comments |
| Complete neuroepithelial induction medium (NRI): | |||
| DMEM/F12 | Thermo-Fisher | 3133-038 | (Step 2.3.1) |
| Non-Essential Amino Acids | Thermo-Fisher | 11140-035 | (Step 2.3.1) |
| N2 Supplement | Thermo-Fisher | 17502-048 | (Step 2.3.1) |
| Penicillin/Streptomycin | Thermo-Fisher | 15140-122 | 50 U/mL final concentration (Step 2.3.1) |
| Heparin Grade I-A, ≥180 USP units/mg | Sigma-Aldrich | H3149-100KU | 2 µg/ml final concentration (Step 2.3.1) |
| bFGF | Thermo-Fisher | 13256-029 | 20 ng/ml final concentration added before use (Step 2.3.1) |
| Matrigel Basement Membrane Matrix | Corning | 354234 | 1:100 (Step 2.2). Thaw Matrigel on ice, prepare 200 ul aliquots and store them in -80°C. For coating, dilute 200 ul aliquot in 20 ml of cold DMEM/F12 medium. |
| Laminin | Sigma-Aldrich | L2020 | 1:100 (Step 2.2). Dilute in PBS 1X. |
| Name | Company | Catalog Number | Comments |
| Complete Neuronal Differentiation medium (ND): | |||
| Neurobasal Medium | Thermo-Fisher | 21103049 | (Step 2.4.11) |
| B-27 Supplements (50x) | Thermo-Fisher | 17504044 | (Step 2.4.11) |
| N2 Supplement | Thermo-Fisher | 17502-048 | (Step 2.4.11) |
| Penicillin/Streptomycin | Thermo-Fisher | 15140-122 | 50 U/mL final concentration (Step 2.4.11) |
| GDNF | Thermo-Fisher | PHC7045 | 1 ng/ml final concentration. Added before use. (Step 2.4.11) |
| BDNF | Thermo-Fisher | PHC7074 | 2.5 ng/ml final concentration. Added before use. (Step 2.4.11) |
| Name | Company | Catalog Number | Comments |
| Neural induction medium (NI): | |||
| DMEM/F12 | Thermo-Fisher | 3133-038 | (Step 3.3) |
| Non-Essential Amino Acids | Thermo-Fisher | 11140-035 | (Step 3.3) |
| N2 Supplement | Thermo-Fisher | 17502-048 | (Step 3.3) |
| Penicillin/Streptomycin | Thermo-Fisher | 15140-122 | 50 U/mL final concentration (Step 3.3) |
| Heparin Grade I-A, ≥180 USP units/mg | Sigma-Aldrich | H3149-100KU | 2 µg/ml final concentration (Step 3.3) |
| B-27 Supplement (50X), minus vitamin A | Thermo-Fisher | 12587010 | (Step 3.3) |
| L-Glutamine 200 mM Solution | Thermo-Fisher | 25030-081 | 2 mM final concentration (Step 3.3) |
| bFGF | Thermo-Fisher | 13256-029 | 10 ng/ml final concentration. Added before use (Step 3.3) |
| EGF | Thermo-Fisher | PHG6045 | 10 ng/ml final concentration. Added before use (Step 3.3) |
| BDNF | Thermo-Fisher | PHC7074 | 2.5 ng/ml final concentration. Added before use (Step 3.3) |
| Defined Trypsin Inhibitor (DTI) | Thermo-Fisher | R007-100 | Pre-warm at 37°C. Add an equal amount of DTI to Trypsin-EDTA (Step 3.10) |
| Trypsin-EDTA (0.5%), no phenol red | Thermo-Fisher | 15400054 | 1:10. Dilute Trypsin-EDTA in PBS 1x (without calcium and magnesium), pre-warm the solution at 37°C (Step 3.8) |
| CryoStor cell cryopreservation medium | Sigma-Aldrich | C2874-100ML | (Step 4.2) |
| Trypan Blue (0.4%) | Sigma-Aldrich | T8154-100ML | multiple manufacturers/suppliers |
| Name | Company | Catalog Number | Comments |
| TaqMan Probesets and reagents for gene expression analysis: | |||
| RNAqueous-Micro kit | Thermo-Fisher | AM1931 | (Step 5.1.6) |
| High Capacity cDNA Reverse Transcription Kits | Thermo-Fisher | 4368814 | |
| TaqMan Gene Expression Master Mix | Thermo-Fisher | 4369016 | |
| GFAP | Thermo-Fisher | Hs00909233_m1 | |
| MAP2 | Thermo-Fisher | Hs00258900_m1 | |
| NQO1 | Thermo-Fisher | Hs02512143_s1 | |
| SRXN1 | Thermo-Fisher | Hs00607800_m1 | |
| HMOX1 | Thermo-Fisher | Hs01110250_m1 | |
| GSR | Thermo-Fisher | Hs00167317_m1 | |
| PAX6 | Thermo-Fisher | Hs01088112_m1 | |
| NES | Thermo-Fisher | Hs00707120_s1 | |
| GRIA1 | Thermo-Fisher | Hs00181348_m1 | |
| GAP43 | Thermo-Fisher | Hs00967138_m1 | |
| GABRA3 | Thermo-Fisher | Hs00968132_m1 | |
| GABRA1 | Thermo-Fisher | Hs00168058_m1 | |
| NR4A2 | Thermo-Fisher | Hs00428691_m1 | |
| TH | Thermo-Fisher | Hs00165941_m1 | |
| GAPDH | Thermo-Fisher | Hs02758991_g1 | |
| ACTB | Thermo-Fisher | Hs99999903_m1 | |
| MAPT | Thermo-Fisher | Hs00902194_m1 | |
| SYP | Thermo-Fisher | Hs00300531_m1 | |
| NANOG | Thermo-Fisher | Hs04260366_g1 | |
| POU5F1 (OCT4) | Thermo-Fisher | Hs04195369_s1 | |
| SOX1 | Thermo-Fisher | Hs01057642_s1 | |
| AFP | Thermo-Fisher | Hs00173490_m1 | |
| KRT18 | Thermo-Fisher | Hs01941416_g1 | |
| NPPA | Thermo-Fisher | Hs00383230_g1 | |
| T | Thermo-Fisher | Hs00610080_m1 | |
| NCAM1 | Thermo-Fisher | Hs00941821_m1 | |
| NR4A1 | Thermo-Fisher | Hs00374226_m1 | |
| PHOX2A | Thermo-Fisher | Hs00605931_mH | |
| PHOX2B | Thermo-Fisher | Hs00243679_m1 | |
| NARG2 | Thermo-Fisher | Hs00973298_g1 | |
| SLC18A3 | Thermo-Fisher | Hs00268179_s1 | |
| SLC5A7 | Thermo-Fisher | Hs00222367_m1 | |
| ISL1 | Thermo-Fisher | Hs00158126_m1 | |
| LHX3 | Thermo-Fisher | Hs01033412_m1 | |
| TaqMan Human Protein Kinase Array | Thermo-Fisher | 4418721 | |
| Name | Company | Catalog Number | Comments |
| Antibodies and reagents for immunostaining: | |||
| B-III-tubulin (Tuj1) | Covance | MMS-435P | 1:500 (Step 5.2.5). Other antibodies may also be used. |
| MAP2 | Sigma Aldrich | M4403 | 1:500 |
| NF200 | Sigma Aldrich | N4142 | 1:1000 |
| GFAP | Acris Antibodies GmbH | AP02002SU-N | 1:500 |
| Nestin | Sigma-Aldrich | N5413 | 1:200 |
| synaptophysin (SYN) | Abcam | AB14692 | 1:200 |
| Tau | Thermo-Fisher | MA5-12808 | 1:100 |
| Nrf2 | Abcam | AB62352 | 1:200 |
| Keap1 | Abcam | AB66620 | 1:200 |
| sulfiredoxin1 (SRXN1) | Abcam | AB92298 | 1:200 |
| NAD(P)H quinone oxidoreductase 1 (NQO1) | Abcam | AB2346 | 1:200 |
| OCT4 | Millipore | MAB4401 | 1:100 |
| SSEA3 | Millipore | MAB4303 | 1:100 |
| Tra1-60 | Millipore | MAB4360 | 1:250 |
| Tyrosine hydroxylase (TH) | Millipore | AB152 | 1:200 |
| Gamma-aminobutyric acid (GABA) | Sigma-Aldrich | A0310 | 1:100 |
| Vesicular glutamate transporter 1 (VGlut1) | Abcam | AB72311 | 1:500 |
| Paraformaldehyde | Sigma-Aldrich | P6148-500G | 4% (4% formaldehyde can also be used) |
| DPBS, no calcium, no magnesium | Thermo-Fisher | 14190144 | |
| Triton-X-100 Solution | Sigma-Aldrich | 93443-100ML | 0.1% |
| BSA 35% | Sigma-Aldrich | A7979-50ML | 3.5% |
| Donkey anti-Rabbit IgG (H+L) Cross Adsorbed Secondary Antibody, DyLight 594 conjugate | Thermo-Fisher | SA5-10040 | 1:500. (Step 5.2.7) Other fluorochrome-conjugated secondary antibodies may also be used. In this case, appropriate dilutions should be tested by the enduser. |
| Donkey anti-Mouse IgG (H+L) Cross Adsorbed Secondary Antibody, DyLight 488 conjugate | Thermo-Fisher | SA5-10166 | 1:500 |
| Donkey anti-Goat IgG (H+L) Cross Adsorbed Secondary Antibody, DyLight 488 conjugate | Thermo-Fisher | SA5-10086 | 1:500 |
| DAPI Solution (1 mg/ml) | Thermo-Fisher | 62248 | 1:1000 (Step 5.2.7) |
| Name | Company | Catalog Number | Comments |
| Antibodies for Reverse Phase Protein Array (RPPA): | |||
| 4E-BP1 (S65) | Abcam | AB81297 | 1:250 (Note after step 5.2.8) |
| Akt (T308) | Cell Signaling | 9275 | 1:100 |
| Akt (S473) | Cell Signaling | 9271 | 1:100 |
| AMPKalpha (T172) | Cell Signaling | 2531 | 1:100 |
| AMPKbeta1 (S108) | Cell Signaling | 4181 | 1:100 |
| ATF-2 (T71) | Cell Signaling | 9221 | 1:100 |
| c-Jun (S63) | Cell Signaling | 9261 | 1:200 |
| c-Jun (S73) | Cell Signaling | 9164 | 1:200 |
| c-Kit (Y719) | Cell Signaling | 3391 | 1:250 |
| CREB (S133) | Cell Signaling | 9191 | 1:100 |
| EGFR (Y1068) | Cell Signaling | 2234 | 1:50 |
| ErbB2/HER2 (Y1248) | Cell Signaling | 2247 | 1:100 |
| ERK 1/2, p44/42 (T202/Y204) | Cell Signaling | 9101 | 1:2000 |
| GSK-3alpha (S21) | Cell Signaling | 9337 | 1:50 |
| Jak1 (Y1022/1023) | Cell Signaling | 3331 | 1:100 |
| Lck (Y505) | Cell Signaling | 2751 | 1:500 |
| LKB1 (S428) | Cell Signaling | 3051 | 1:100 |
| mTOR (S2448) | Cell Signaling | 5536 | 1:100 |
| NFkB p65 (S536) | Cell Signaling | 3031 | 1:50 |
| p70 S6 Kinase (T389) | Cell Signaling | 9205 | 1:200 |
| PDK1 (S241) | Cell Signaling | 3061 | 1:100 |
| PKA C (T197) | Cell Signaling | 4781 | 1:250 |
| PRAS40 (T246) | BioSource | 44-1100 | 1:2000 |
| PTEN (S380) | Cell Signaling | 9551 | 1:500 |
| Smad1 (S463/465), Smad5 (S463/465), Smad8 (S426/428) | Cell Signaling | 9511 | 1:500 |
| Src (Y527) | Cell Signaling | 2105 | 1:500 |
| Src Family (Y416) | Cell Signaling | 2101 | 1:200 |
| Stat1 (Y701) | Cell Signaling | 9171 | 1:200 |
| Stat3 (S727) | Cell Signaling | 9134 | 1:200 |
| Zap-70 (Y319) | Enogene | E011159 | 1:100 |
| βCatenin (S33/37/T41) | Cell Signaling | 9561 | 1:250 |
| CREB | Upstate Biotechnologies | 06-863 | 1:100 |
| Fos B | Cell Signaling | 2251 | 1:200 |
| GRB2 | Cell Signaling | 3972 | 1:2000 |
| HSP70 | Stressgen | SPA-810 | 1:100 |
| c-Jun | Cell Signaling | 9165 | 1:100 |
| Kip1/p27 | BD | 610241 | 1:100 |
| Lck | Cell Signaling | 2984 | 1:250 |
| Mcl-1 | Cell Signaling | 4572 | 1:80 |
| Musashi | Cell Signaling | 2154 | 1:100 |
| NOTCH1 | Cell Signaling | 3439 | 1:100 |
| PTEN | Cell Signaling | 9552 | 1:500 |
| SGK1 | Abnova | PAB4590 | 1:250 |
| Zap-70 | Cell Signaling | 2705 | 1:250 |
| β-Catenin | Abcam | AB32572 | 1:1000 |
| Dll4 | Abcam | AB7280 | 1:500 |
| Shh | Abcam | AB53281 | 1:250 |
| HIF-1α | BD | 610958 | 1:50 |
| NUMB PAN-ISO | Upstate Biotechnologies | 07-207 | 1:400 |
| NUMB-L | Chemicon | AB15145 | 1:750 |
| Cyclin B | BD | 610220 | 1:75 |
| c-Myc | Calbiochem | OP-10 | 1:100 |
| BCIP/NBT Kit | Thermo-Fisher | 002209 | (Note after step 5.2.8). Kit used to measure alkaline phosphatase activity, similar kits can be used. |
| Name | Company | Catalog Number | Comments |
| Antibodies for Flow Cytometry: | |||
| SSEA1 Antibody, Pacific Blue conjugate | Thermo-Fisher | MHCD1528 | 1:100 (Note after step 5.2.8) |
| SSEA4 Antibody (MC813-70), Alexa Fluor 647 | Thermo-Fisher | SSEA421 | 1:100 |
| Name | Company | Catalog number | Comments |
| Specific instruments, tools and softwares: | |||
| Countess Automated Cell Counter | Thermo-Fisher | C10227 | Neubauer chamber or other suitable glass hemocytometer can be used. |
| MEA1060-Inv-BC | Multichannel Systems | MEA1060-Inv-BC | (Step 5.3) |
| MEA1060-BC control software | Multichannel Systems | MEA1060-BC | (Step 5.3) |
| NeuroExplorer | Multichannel Systems | NeuroExplorer (NE) | (Step 5.3) For post-processing of MEA data |
| Multielectrode arrays (MEA) | Multichannel Systems | 60MEA100/10iR-Ti-gr | (Step 5.3) Single-well MEA chip |
| ArrayScan XTI High Content Platform | Thermo-Fisher | ASN00002P | (Step 5.2.8) Mean fluorescence can be quantified by using specific ArrayScan algorithms (e.g., Cytotoxicity V.4 and NucTrans V.4 bioapplications). It is recommended to take minimum 20 pictures/well, and have 7-8 internal replicates per condition |
| 7900HT Fast Real-Time PCR System | Thermo-Fisher | 4351405 | (Step 5.1.6) |
| BD ULTRA-FINE Needle Insulin Syringe (with 30G needle) | BD | 328280 | (Steps 1.3.1, 2.1.2, and 2.4.1) |
| StemPro EZPassage Disposable Stem Cell Passaging Tool | ThermoFisher | 23181010 | This colony cutting tool can be used as an alternative to the use of 30G needle 1 mL syringes (Step 1.3.1) |
| Ultra-Low attachment Petri dish (60 mm) | Corning | 10010582 | (Step 2.1.8) Also other brands can be used. |
| Mr. Frosty Freezing container | Sigma-Aldrich | C1562-1EA | |
Referenzen
- NRC. . Toxicity Testing in the 21st Century: A Vision and a Strategy. , (2007).
- Drews, K., Jozefczuk, J., Prigione, A., Adjaye, J. Human induced pluripotent stem cells--from mechanisms to clinical applications. J Mol Med (Berl). 90 (7), 735-745 (2012).
- Ho, P. J., Yen, M. L., Yet, S. F., Yen, B. L. Current applications of human pluripotent stem cells: possibilities and challenges. Cell Transplant. 21 (5), 801-814 (2012).
- Krueger, W. H., Swanson, L. C., Tanasijevic, B., Rasmussen, T. P. Natural and artificial routes to pluripotency. Int J Dev Biol. 54 (11-12), 1545-1564 (2010).
- Pistollato, F., Bremer-Hoffmann, S., Healy, L., Young, L., Stacey, G. Standardization of pluripotent stem cell cultures for toxicity testing. Expert Opin Drug Metab Toxicol. 8 (2), 239-257 (2012).
- Pistollato, F., et al. Development of a pluripotent stem cell derived neuronal model to identify chemically induced pathway perturbations in relation to neurotoxicity: effects of CREB pathway inhibition. Toxicol Appl Pharmacol. 280 (2), 378-388 (2014).
- Zagoura, D., Canovas-Jorda, D., Pistollato, F., Bremer-Hoffmann, S., Bal-Price, A. Evaluation of the rotenone-induced activation of the Nrf2 pathway in a neuronal model derived from human induced pluripotent stem cells. Neurochem Int. , (2016).
- Standard operating procedure for differentiation of human induced pluripotent stem cells into post-mitotic neurons and glial cells. EURL ECVAM Available from: https://ecvam-dbalm.jrc.ec.europa.eu/methods-and-protocols/protocol/standard-operating-procedure-for-differentiation-of-human-induced-pluripotent-stem-cells-into-post-mitotic-neurons-and-glial-cells-%28mixed-culture%29-protocol-no.-165/key/p_1570 (2016)
- Standard operating procedure for expansion of rosette-derived neural stem cells. EURL ECVAM Available from: https://ecvam-dbalm.jrc.ec.europa.eu/methods-and-protocols/protocol/standard-operating-procedure-for-expansion-of-rosette-derived-neural-stem-cells-protocol-no.-166/key/p_1571 (2016)
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nat Protoc. 3 (6), 1101-1108 (2008).
- Brien, P. J., et al. High concordance of drug-induced human hepatotoxicity with in vitro cytotoxicity measured in a novel cell-based model using high content screening. Arch Toxicol. 80 (9), 580-604 (2006).
- Accordi, B., et al. Functional protein network activation mapping reveals new potential molecular drug targets for poor prognosis pediatric BCP-ALL. PLoS One. 5 (10), e13552 (2010).
- Vassallo, A., et al. A multi-laboratory evaluation of microelectrode array-based measurements of neural network activity for acute neurotoxicity testing. Neurotoxicology. , (2016).
- Shamblott, M. J., et al. Human embryonic germ cell derivatives express a broad range of developmentally distinct markers and proliferate extensively in vitro. Proc Natl Acad Sci U S A. 98 (1), 113-118 (2001).
- Bryan, H. K., Olayanju, A., Goldring, C. E., Park, B. K. The Nrf2 cell defence pathway: Keap1-dependent and -independent mechanisms of regulation. Biochem Pharmacol. 85 (6), 705-717 (2013).
- Tufekci, K. U., Civi Bayin, E., Genc, S., Genc, K. The Nrf2/ARE Pathway: A Promising Target to Counteract Mitochondrial Dysfunction in Parkinson's Disease. Parkinsons Dis. , 314082 (2011).
- Kovac, S., et al. Nrf2 regulates ROS production by mitochondria and NADPH oxidase. Biochim Biophys Acta. 1850 (4), 794-801 (2015).
- Lee, J. M., Shih, A. Y., Murphy, T. H., Johnson, J. A. NF-E2-related factor-2 mediates neuroprotection against mitochondrial complex I inhibitors and increased concentrations of intracellular calcium in primary cortical neurons. J Biol Chem. 278 (39), 37948-37956 (2003).
- Itoh, K., et al. An Nrf2/small Maf heterodimer mediates the induction of phase II detoxifying enzyme genes through antioxidant response elements. Biochem Biophys Res Commun. 236 (2), 313-322 (1997).
- Li, L., et al. Nrf2/ARE pathway activation, HO-1 and NQO1 induction by polychlorinated biphenyl quinone is associated with reactive oxygen species and PI3K/AKT signaling. Chem Biol Interact. , 56-67 (2014).
- Cannon, J. R., et al. A highly reproducible rotenone model of Parkinson's disease. Neurobiol Dis. 34 (2), 279-290 (2009).
- Sherer, T. B., Kim, J. H., Betarbet, R., Greenamyre, J. T. Subcutaneous rotenone exposure causes highly selective dopaminergic degeneration and alpha-synuclein aggregation. Exp Neurol. 179 (1), 9-16 (2003).
- Testa, C. M., Sherer, T. B., Greenamyre, J. T. Rotenone induces oxidative stress and dopaminergic neuron damage in organotypic substantia nigra cultures. Brain Res Mol Brain Res. 134 (1), 109-118 (2005).
- Zhou, J., et al. High-efficiency induction of neural conversion in human ESCs and human induced pluripotent stem cells with a single chemical inhibitor of transforming growth factor beta superfamily receptors. Stem Cells. 28 (10), 1741-1750 (2010).
- Topol, A., Tran, N. N., Brennand, K. J. A Guide to Generating and Using hiPSC Derived NPCs for the Study of Neurological Diseases. J Vis Exp. (96), e52495 (2015).
- Jiang, Y., Zhang, M. J., Hu, B. Y. Specification of functional neurons and glia from human pluripotent stem cells. Protein Cell. 3 (11), 818-825 (2012).
- Parsons, X. H., et al. Efficient derivation of human neuronal progenitors and neurons from pluripotent human embryonic stem cells with small molecule induction. J Vis Exp. (56), e3273 (2011).
- Shi, Y., Kirwan, P., Livesey, F. J. Directed differentiation of human pluripotent stem cells to cerebral cortex neurons and neural networks. Nat Protoc. 7 (10), 1836-1846 (2012).
- Zeng, H., et al. Specification of region-specific neurons including forebrain glutamatergic neurons from human induced pluripotent stem cells. PLoS One. 5 (7), e11853 (2010).
- Zeng, X., et al. An in vitro model of human dopaminergic neurons derived from embryonic stem cells: MPP+ toxicity and GDNF neuroprotection. Neuropsychopharmacology. 31 (12), 2708-2715 (2006).
- Paşca, A. M., et al. Functional cortical neurons and astrocytes from human pluripotent stem cells in 3D culture. Nat Methods. 12 (7), 671-678 (2015).
- Almeida, S., et al. Modeling key pathological features of frontotemporal dementia with C9ORF72 repeat expansion in iPSC-derived human neurons. Acta Neuropathol. 126 (3), 385-399 (2013).
- Urbán, N., Guillemot, F. Neurogenesis in the embryonic and adult brain: same regulators, different roles. Front Cell Neurosci. 8, 396 (2014).
- Kriegstein, A., Alvarez-Buylla, A. The glial nature of embryonic and adult neural stem cells. Annu Rev Neurosci. 32, 149-184 (2009).
- Krencik, R., Weick, J. P., Liu, Y., Zhang, Z. J., Zhang, S. C. Specification of transplantable astroglial subtypes from human pluripotent stem cells. Nat Biotechnol. 29 (6), 528-534 (2011).
- Krencik, R., Zhang, S. C. Directed differentiation of functional astroglial subtypes from human pluripotent stem cells. Nat Protoc. 6 (11), 1710-1717 (2011).
- Nguyen, T., Nioi, P., Pickett, C. B. The Nrf2-antioxidant response element signaling pathway and its activation by oxidative stress. J Biol Chem. 284 (20), 13291-13295 (2009).
- Bal-Price, A., et al. Putative adverse outcome pathways relevant to neurotoxicity. Crit Rev Toxicol. 45 (1), 83-91 (2015).
- Cabezas, R., El-Bacha, R. S., Gonzalez, J., Barreto, G. E. Mitochondrial functions in astrocytes: neuroprotective implications from oxidative damage by rotenone. Neurosci Res. 74 (2), 80-90 (2012).
- Swarnkar, S., et al. Astrocyte activation: a key step in rotenone induced cytotoxicity and DNA damage. Neurochem Res. 37 (10), 2178-2189 (2012).
- Canovas-Jorda, D., Louisse, J., Pistollato, F., Zagoura, D., Bremer, S. Regenerative toxicology: the role of stem cells in the development of chronic toxicities. Expert Opin Drug Metab Toxicol. 10 (1), 39-55 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten