Method Article
Protocolo para a Diferenciação de Células-Tronco Pluripotentes Induzidas Humanas em Culturas Mistas de Neurônios e Glia para Teste de Neurotoxicidade
Neste Artigo
Resumo
As células-tronco pluripotentes induzidas por humanos (hiPSCs) são consideradas uma ferramenta poderosa para a triagem de drogas e substâncias químicas e para o desenvolvimento de novos modelos in vitro para testes de toxicidade, incluindo neurotoxicidade. Aqui, um protocolo detalhado para a diferenciação de hiPSCs em neurônios e glia é descrito.
Resumo
As células estaminais pluripotentes humanas podem se diferenciar em vários tipos de células que podem ser aplicadas a ensaios de toxicidade in vitro baseados em humanos. Uma grande vantagem é que a reprogramação de células somáticas para produzir células-tronco pluripotentes induzidas por humanos (HiPSCs) evita as questões éticas e legislativas relacionadas ao uso de células estaminais embrionárias humanas (hESCs). Os HiPSCs podem ser expandidos e diferenciados de forma eficiente em diferentes tipos de células neuronais e gliais, servindo como sistemas de teste para testes de toxicidade e, em particular, para a avaliação de diferentes caminhos envolvidos na neurotoxicidade. Este trabalho descreve um protocolo para a diferenciação de hiPSCs em culturas mistas de células neuronais e gliais. As vias de sinalização que são reguladas e / ou ativadas pela diferenciação neuronal são definidas. Esta informação é fundamental para a aplicação do modelo celular ao novo paradigma de teste de toxicidade, no qual os produtos químicos são avaliados com base na sua capacidade de peRturb caminhos biológicos. Como uma prova de conceito, a rotenona, um inibidor do complexo respiratório mitocondrial I, foi utilizada para avaliar a ativação da via de sinalização Nrf2, um regulador chave do mecanismo de defesa celular anti-oxidante-resposta-elemento- (ARE) contra o estresse oxidativo .
Introdução
O relatório do Conselho Nacional de Pesquisa dos EUA 1 prevê um novo paradigma de teste de toxicidade em que o teste de toxicidade regulatória seria deslocado de uma abordagem que dependesse de alterações fenotípicas observadas em animais para uma abordagem focada em ensaios in vitro mecanicistas usando células humanas. Os derivados de células estaminais pluripotentes (PSC) podem representar alternativas aos modelos de células cancerosas, pois as células obtidas podem se assemelhar mais às condições fisiológicas dos tecidos humanos e fornecer ferramentas mais relevantes para estudar os efeitos adversos induzidos por produtos químicos. Os dois principais tipos de culturas de PSC que são mais promissores para o teste de toxicidade são as células estaminais embrionárias humanas (hESCs) e células-tronco pluripotentes induzidas por humanos (HiPSCs), que atualmente são amplamente utilizadas nas áreas de pesquisa básica e medicina regenerativa 2 , 3 . Esta experiência agora pode ser aproveitada para o desenvolvimento de uma nova classe de toxoloTestes in vitro in vitro destinados a identificar as vias fisiológicas perturbadas envolvidas no desenvolvimento de efeitos adversos in vivo . No entanto, os métodos de teste para avaliações de segurança regulamentares baseadas em hESCs não deverão ser aceitos por todos os Estados-Membros da UE e por países em todo o mundo devido a possíveis preocupações éticas e diversas políticas legislativas nacionais que regulam o uso de células derivadas de embriões.
Os hiPSCs compartilham características semelhantes aos hESCs 4 , 5 e possuem grande potencial para métodos in vitro , tanto para identificar alvos terapêuticos como para avaliações de segurança. Além disso, a tecnologia hiPSC mitiga as restrições de um pool de doadores limitado e as preocupações éticas associadas às células derivadas de embriões. Um grande desafio para o HiPSCs é a demonstração de que essas células podem gerar de forma reprodutiva uma gama significativa de derivados de células toxicologicamente relevantes,Com características e respostas típicas dos tecidos humanos. Os níveis predefinidos dos marcadores selecionados geralmente são usados para caracterizar as populações celulares após o processo de diferenciação e fornecer informações sobre a estabilidade do processo de diferenciação.
Trabalhos anteriores avaliaram a adequação de hiPSCs para gerar culturas mistas de células neuronais e gliais e avaliar os efeitos da rotenona, um inibidor do complexo respiratório mitocondrial I, na ativação da via Nrf2, um regulador chave dos mecanismos de defesa antioxidante em Muitos tipos de células 6 , 7 .
Este trabalho descreve um protocolo usado para a diferenciação de HiPSCs em culturas mestiças neuronais e gliais, fornecendo detalhes sobre as vias de sinalização (gene e nível de proteína) que são ativadas na diferenciação neuronal / glial. Além disso, o trabalho mostra resultados representativos demonstrando como issoO modelo de células neuronais e gliais derivadas de HiPSC pode ser usado para avaliar a ativação da sinalização Nrf2 induzida por tratamento agudo (24 h) com rotenona, permitindo a avaliação da indução do estresse oxidativo.
Os fibroblastos IMR90 foram reprogramados em hiPSCs em I-Stem (França) pela transdução viral de 2 fatores de transcrição (Oct4 e Sox2) usando vetores pMIG 6 . Modelos hiPSC análogos também podem ser aplicados. Os protocolos descritos abaixo resumem todos os estágios de diferenciação de hiPSCs em células-tronco neurais (NSCs) e mais em culturas mistas de neurônios pós-mitóticos e células gliais (etapas 1 e 2, veja também o site EURL ECVAM DBALM para uma descrição detalhada de O protocolo) 8 .
Um protocolo adicional para o isolamento, expansão, criopreservação e maior diferenciação de NSCs em neurônios misturados e células gliais é detalhado nas etapas 3 e 4 (também se referem ao EURL ECVAM DBALM nósBsite para uma descrição detalhada deste protocolo) 9 . O Passo 5 descreve as análises que podem ser feitas para avaliar a identidade fenotípica das células durante os vários estágios de compromisso e diferenciação.
Protocolo
1. Expansão de células-tronco pluripotentes induzidas por humanos (hiPSC)
NOTA: os hiPSCs podem ser cultivados em um substrato de proteína adequada na presença de meio mTeSR1 contendo suplementos de mTeSR1 5x (preparado de acordo com as instruções do fabricante, placas de 100 fragmentos de colônia / placa de Petri de 60 mm). Quando as colônias hiPSC atingem um tamanho apropriado (veja um exemplo de uma colônia na Figura 2A ), passe as células conforme descrito abaixo (uma vez por semana).
- Revestir pratos com matriz de membrana basal qualificada com hESC (doravante denominada "matriz qualificada") ou qualquer outro substrato proteico adequado.
- Armazene a matriz qualificada (veja a Tabela de Materiais ) a -80 ° C em alíquotas de 200 μL em tubos de 1,5 mL frios e pipetas de 5 ou 10 mL frias.
- Antes de passar, descongelar 200 μL de matriz qualificada no gelo.
- Diluir 200 μL de matriz qualificada em 20 mL de meio DMEM / F12 (diluição 1: 100).
- Cubra placas de Petri de 60 mm com esta solução (5 mL / prato).
- Incubar os pratos revestidos a 37 ° C durante pelo menos 1 h.
- Criopreservação e descongelação de fragmentos de colônias HiPSC
- Depois de cortar as colônias de HiPSC (ver passo 1.3 para o procedimento de corte da colônia hiPSC), ressuspenda gentil e lentamente os fragmentos de colônia hiPSC em meio de congelação de células estaminais, ~ 100 fragmentos / 250 μL (veja a Tabela de Materiais).
- Alíquota dos fragmentos de colônia em frascos adequados para criopreservação (250 μL / frasco para injectáveis).
- Coloque os frascos em um recipiente cheio de 2-propanol e coloque o recipiente a -80 ° C durante um mínimo de 2 h e até 2 semanas.
- Transfira os frascos para a fase de vapor de um tanque de nitrogênio líquido.
- Para reiniciar a cultura, descongelar 1 frasco congelado em banho-maria a 37 ° C.
- Recolher delicadamente os fragmentos de colônia hiPSC em 7 mL de HiPSC completo pré-aquecidoMédio (veja a Tabela de Materiais ) em um tubo de 15 mL usando uma pipeta de 1, 2 ou 5 mL.
- Centrifugar os fragmentos de colônia hiPSC a 130 xg durante 3 min.
- Remova o sobrenadante e ressuspenda gentilmente os fragmentos de colônia hiPSC em 1 mL de meio hiPSC completo usando uma pipeta de 1, 2 ou 5 mL.
- Diluir adicionalmente a suspensão celular em 3 ou 4 mL de meio hiPSC completo.
- Placa os fragmentos de colônia hiPSC em uma placa de Petri de 60 mm com revestimento de matriz qualificado (~ 100 fragmentos / prato; prenda os pratos conforme descrito no passo 1.1).
- Incube os hiPSCs a 37 ° C e 5% de CO 2 .
- Execute uma mudança média total todos os dias.
- Aproximação das colônias HiPSC
NOTA: HiPSCs indiferenciados devem ser redondos em forma, com nucleolos grandes e sem citoplasma abundante. As colônias indiferenciadas devem ser caracterizadas por uma morfologia plana e bem embalada. Apenas colônias indiferenciadas (cerca de 1 mm iN diâmetro) deve ser cortado para uma passagem adicional.- Corte as colônias de células-tronco em quadrados de cerca de 200 μm x 200 μm usando uma seringa de 1 mL com uma agulha 30G ou qualquer outra ferramenta comercialmente disponível (veja a Tabela de Materiais). Use um microscópio estereoscópico com uma ampliação de 4x em um gabinete de fluxo laminar à temperatura ambiente.
- Destaque os fragmentos de colônia da superfície do prato usando uma pipeta de 200 μl, pipetando suavemente o meio por baixo para levantar as peças.
- Transfira os fragmentos de colônia (~ 100 peças) para uma placa qualificada com placa DMEM / F12 preenchida com 4 mL de meio hiPSC completo (ver Tabela de Materiais, revestir os pratos conforme descrito no passo 1.1).
- Incubar a (s) nova (s) placa (s) a 37 ° C e 5% de CO 2 .
- Execute uma mudança média total todos os dias e inspecione a morfologia das colônias usando um microscópio de contraste de fase com amplificações de 4X e 10X.
2. HiPSC DiffereNação em Neurônios Mistos e Glia
NOTA: O procedimento leva aproximadamente 28 dias para ser concluído, com os principais passos descritos na Figura 1 (parte superior).
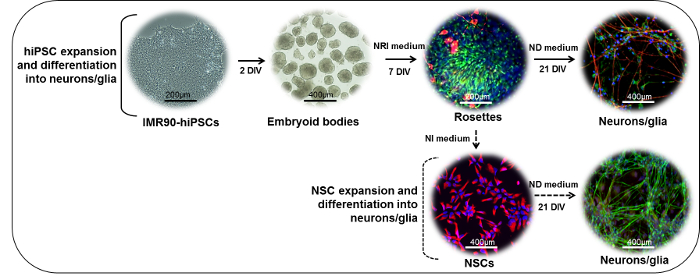
Figura 1: Representação esquemática do protocolo de diferenciação neuronal. (Parte superior) As colônias IMR90-hiPSC podem ser cortadas em fragmentos para formar corpos embrionários (EBs). Após 2 dias in vitro (DIV), os EBs podem ser plaqueados em pratos laminados ou padrão revestidos de matriz e cultivados na presença de meio de indução neuroepitelial (NRI) para gerar derivados neuroectodermais (rosetas, aqui coradas para nestin (verde) e β -III-tubulina (vermelha)). Os rosetas podem ser dissociados, coletados, reaparecidos em pratos laminados ou padrão revestidos de matriz, e diferenciados em neurônio maduro (NF200, vermelho) e glial(GFAP, verde) na presença de meio de diferenciação neuronal (ND). (Parte inferior), os NSC derivados de rosetas (nestin, vermelho) podem ser expandidos na presença de meio de indução neural (NI), criopreservados ou ainda diferenciados na presença de meio ND para formar neuronal misto (NF200, verde) e glial ( GFAP, vermelho) culturas. Clique aqui para ver uma versão maior desta figura.
- Geração de corpos embrióides (EBs) (Dias 0 → 1)
NOTA: Este procedimento requer boas habilidades e precisão manual. Os fragmentos de colônia HiPSC devem ser de tamanho igual para obter corpos embrionóneos homogêneos (EBs) nos próximos passos. As colônias morfologicamente diferenciadas (com grandes frações citoplasmáticas e pequenos nucleolos) devem ser descartadas.- Refresque o meio HiPSC (placa de Petri de 3 mL / 60 mm) antes de cortar as colônias indiferenciadas hiPSC (cerca de 1 mm de diâmetroEter, veja a Figura 2A ) sob condições estéreis (como descrito no passo 1).
- Corte as colônias indiferenciadas (como mostrado na Figura 2A e Figura 2B ) em fragmentos de aproximadamente 200 μm x 200 μm usando uma seringa de 1 mL com uma agulha 30G. Use um microscópio estereoscópico com uma ampliação de 4X em um gabinete de fluxo laminar à temperatura ambiente.
- Retire os fragmentos de colônia da superfície do prato usando uma pipeta de 200 μl, fazendo um pipeteamento suave por baixo do meio para levantar as peças.
- Transfira todos os fragmentos destacados e o meio para um tubo de 15 mL usando uma pipeta de 1, 2 ou 5 mL.
- Enxágüe o prato com 2 mL de meio hiPSC completo para recuperar todos os fragmentos.
- Centrifugar a 112 xg durante 1 min.
- Aspirar o sobrenadante e ressuspender gentilmente os fragmentos em 5 mL de meio HiPSC EB completo (ver Tabela de Materiais ).
- Placa o coFragmentos de lony em uma placa de Petri de 60 mm ultra-baixa fixação (placa de Petri de 5 mL / 60 mm).
- Incubar a placa de Petri durante a noite a 37 ° C e 5% de CO 2 .
- No dia seguinte (Dia 1), colete os EBs e seu meio em um tubo de 15 mL usando uma pipeta de 1, 2 ou 5 mL.
- Centrifugar os EBs a 112 xg por 1 min.
- Elimine cuidadosamente o sobrenadante e ressuspenda gentilmente os EBs em 5 mL de meio de HiPSC EB completo usando uma pipeta de 1, 2 ou 5 mL.
- Replaca os EBs em uma nova placa de Petri de 60 mm ultra-baixa fixação (placa de Petri de 5 mL / 60 mm).
- Incubar a placa de Petri durante a noite a 37 ° C e 5% de CO 2 .
- No dia 1, revestir os pratos com a matriz da membrana basal ( por exemplo, matrigel, a seguir designado por "matriz padrão") ou qualquer outro substrato de proteína adequado ( por exemplo , laminina).
- Armazene a matriz padrão (veja a Tabela de Materiais ) a -80 ° C emAlíquotas de 200 μL utilizando tubos de 1,5 mL frios e pipetas de 5 ou 10 mL frias.
- Destilar 200 μL de matriz padrão em gelo.
- Diluir 200 μL de matriz padrão em 20 mL de meio DMEM / F12 (diluição 1: 100).
- Cubra placas de Petri de 60 mm com esta solução (5 mL / prato).
- Incubar os pratos revestidos a 37 ° C durante a noite.
NOTA: Estes pratos serão utilizados para plaquear os EBs (cerca de 50 EBs / prato) e gerar agregados neuroepiteliais (rosetas); Veja o passo 2.3.
- Geração de agregados neuroepiteliais (rosetas) (Dias 2 → 7)
- No dia 2, remova a solução de revestimento de matriz padrão das placas de Petri de 60 mm (não precisa enxaguar as placas) e preencha-as com 5 mL / prato de meio de indução neuroepitelial completo (NRI); Veja a Tabela de Materiais .
- Transfira os EBs flutuantes (do passo 2.1.14) para pratos revestidos (~ 50 EBs / prato) usando uma pipeta de 200 μL sob um filtro estereoscópicoCroscópio com uma ampliação de 4x e colocado em um gabinete de fluxo laminar.
NOTA: É fundamental selecionar EBs de tamanho médio homogêneo (~ 200-300 μm de diâmetro). EBs muito pequenas podem não sobreviver bem durante a diferenciação neuroectodérmica, enquanto os EBs muito grandes tendem a sofrer necrose do núcleo. - Incubar os pratos a 37 ° C e 5% de CO 2 .
- No dia seguinte (Dia 3), verifique a louça no microscópio com uma ampliação de 10x para garantir que os EBs estejam conectados.
- Execute suavemente uma alteração média total com meio NRI completo.
- Altere a média de NRI todos os dias até ao dia 7, quando os agregados neuroepiteliais (rosetas) devem ser visíveis.
- No dia 7, coloque a matriz padrão (ou laminina), conforme descrito no passo 2.2, em qualquer formato de placa ou prato necessário: placas de 96 poços (100 μL / poço), placas de 24 poços (250 μL / poço), 12- Bem placas (500 μL / poço), chips MEA (para atividade elétrica, 1 mL / chip de um único poço) ou 60 mm Petri disheS (4 mL / prato).
- Incubar as placas / pratos revestidos durante pelo menos 2 h a 37 ° C e 5% de CO 2 .
- Dissociação de rosetas e diferenciação neuronal (Dias 8 → 28)
NOTA: Este procedimento requer boas habilidades e precisão manual. Para evitar a coleta de células mesodérmicas e endodérmicas, apenas as estruturas tipo ectoderme rosette devem ser dissociadas e coletadas.- No dia 8, corte as estruturas semelhantes a rosetas em fragmentos sob um microscópio estereoscópico com uma ampliação de 10X em condições estéreis. Use uma seringa de 1 mL com uma agulha 30G. Observe que as rosetas tendem a se separar facilmente do prato quando tocadas com a agulha.
- Complete o desprendimento dos fragmentos de rosetas usando uma pipeta de 200 μL.
- Transfira o prato sob o capô de fluxo laminar e colete os fragmentos de rosácea e o meio em um tubo cônico de 15 mL usando uma pipeta de 1, 2 ou 5 mL. Enxaguar o prato com 2 mL de meio NRI para recuperarTodos os fragmentos.
- Gire os fragmentos de rosetas a 112 xg durante 2 min.
- Aspirar o sobrenadante.
- Ressuspenda gentilmente o sedimento em 1 mL de 1x DPBS (sem cálcio e magnésio) e faça uma pipeta suavemente dos fragmentos de rosetas para cima e para baixo usando uma pipeta de 1000 μL para dissociá-los parcialmente.
- Adicione 4 mL de meio de NRI completo e conte as células usando azul de tripano e um contador de células automatizado (veja a Tabela de Materiais )
NOTA: Diluir 20 μL de suspensão celular em 20 μL de azul de Trypan. Este passo pode ser omitido se as células não puderem ser trazidas para uma suspensão de célula única. Se os fragmentos de rosácea não parecem completamente dissociados, os fragmentos de rosetas derivados de cerca de 50 EBs / prato de 60 mm podem ser ressuspensos em 50 mL de meio NRI completo e chapeados, conforme indicado na Tabela 1 . - Aspirar a solução de revestimento de matriz padrão (ou laminina) das placas de Petri, placas e / ou chips MEA (do passo 2.3.7). Não deixe secar.
- Placa as células em meio NRI completo de acordo com o plano de estudo (cerca de 15.000 células / cm 2 , ver Tabela 1 para indicações de volume do chapeamento).
- Incubar as placas durante a noite a 37 ° C e 5% de CO 2 .
- No dia 10, realize uma alteração de meio total usando o meio de diferenciação neuronal completo (ND); Veja a Tabela de Materiais.
- Atualize o meio completo de ND duas vezes por semana até o dia 28.
- Caracterize os derivados de células neuronais / gliais, conforme descrito no passo 5 (ver Tabela 2 para critérios gerais de aceitação).
3. Expansão e Diferenciação Neural Neurônica (NSC) derivada de HiPSC em Neurônios Mistos e Glia
NOTA: NSCs derivados de fragmentos de roseta podem ser expandidos e mantidos seguindo o procedimento descrito abaixo ( Figura 1 , parte inferior). Isso permite incrementarE número de células para diferenciação e testes químicos.
- Revestir uma placa de Petri de 60 mm (ou um balão T-25) com 5 mL de solução de revestimento de DMEM / F12 de matriz padrão e incubri-la durante pelo menos 2 h a 37 ° C e 5% de CO 2 (como descrito no passo 2.2).
- Gire os fragmentos de roseta derivados dos passos 1-2 (ver passo 2.4.) Em um tubo cônico de 15 mL a 112 xg durante 2 min.
- Ressuspenda gentilmente o sedimento em 5 mL de meio de indução neural (NI); Veja a Tabela de Materiais.
- Transfira as células para uma placa padrão de Petri de 60 mm revestida com matriz (ou balão T-25).
- Cultura de NSC derivado de rosetas na presença de NI médio, atualizando a média todos os dias até as células chegarem à confluência.
- Quando confluente, passe os NSCs como descrito nas seguintes etapas.
NOTA: Aproxime os NSCs uma vez por semana; Considere usar pratos, frascos ou pratos recém-revestidos, dependendo do plano de estudo. - Remova o NI completo e medíocreEnxágue os NSCs com DPBS (sem cálcio e magnésio).
- Adicione 1,5 mL de 0,05% de tripsina-EDTA pré-aquecida a 37 ° C para a placa de Petri de 60 mm (ou balão T-25) contendo as células e coloque-a na incubadora durante 1 min.
- Toque suavemente o prato (ou frasco) para separar as células.
- Adicione 1,5 mL de inibidor de tripsina pré-aquecido a 37 ° C e transfira as células para um tubo de 15 mL.
- Enxaguar a placa de Petri (ou balão T-25) com um volume igual de meio NI (1,5 mL) e coletar o volume no mesmo tubo de 15 mL.
- Centrifugar as células a 130 xg durante 3 min.
- Remova o sobrenadante e ressuspenda gentilmente as células em 1 mL de meio NI completo usando uma pipeta de 1.000 uL.
- Diluir adicionalmente a suspensão celular em 3 ou 4 mL de meio NI completo e contar as células usando azul de tripano e um contador de células automatizado.
- Placa os NSCs na placa de Petri de 60 mm (ou balão T-25) do passo 3.1 a uma densidade de cerca de 50 000 células / cm2.
- Execute uma alteração média total com o meio NI completo todos os dias.
- Caracterizar as células para a presença de derivados de células neuronais / gliais, como descrito no passo 5.
NOTA: Os NSCs podem ser diferenciados em culturas misturadas de neurônios e glia em meio ND completo (conforme descrito nas etapas 2.4.11-2.4.13), atualizando o meio de ND completo duas vezes por semana durante 21 dias.
4. Criopreservação e Descongelação NSC derivadas de HiPSC
NOTA: Após a passagem, os NSC podem ser congelados e descongelados seguindo este procedimento.
- NSCs passíveis com centrífuga (do passo 3.12) a 130 xg durante 3 min.
NOTA: As células devem ser contadas no passo 3.14. - Ressuspenda gentil e lentamente os NSCs a 3 x 10 6 / mL de meio de congelação (veja a Tabela de Materiais ).
- Alíquota as células em frascos adequados para criopreservação (cerca de 0,5 mL = 1,5 x 10 6 / frasco para injectáveis).
- Coloque os frascos em um recipienteLled com 2-propanol e coloque o recipiente a -80 ° C durante um mínimo de 2 h e até 2 semanas.
- Transfira os frascos para a fase de vapor de um tanque de nitrogênio líquido.
- Para reiniciar a cultura celular, descongelar 1 frasco congelado em banho-maria a 37 ° C.
- Recolher gentilmente as células em 7 mL de meio NI pré-aquecido completo em um tubo de 15 mL usando uma pipeta de 1000 μl.
- Centrifugar as células a 130 xg durante 3 min.
- Remova o sobrenadante e ressuspenda gentilmente as células em 1 mL de meio NI completo usando uma pipeta de 1000 μl.
- Diluir adicionalmente a suspensão celular em 3 ou 4 mL de meio NI completo e contar as células usando azul de tripano e um contador de células automatizado (nota: diluir 20 μL de suspensão celular em 20 μL de azul de tripano, a viabilidade após o descongelamento deve ser ≥ 80 %).
- Placa os NSCs em uma placa de Petri revestida de 60 mm (ou balão T25) a uma densidade de cerca de 50 000 células / cm2.
5. CharaCterização de células neurais e gliais derivadas de HiPSC
NOTA: Após a diferenciação, os derivados neuronais e gliais podem ser caracterizados usando diferentes técnicas, como as descritas nas seções a seguir.
- Análise quantitativa de PCR em tempo real (qPCR) 10
- Gire os fragmentos de colônias do hiPSC, EBs e / ou NSCs a 130 xg durante 3 min.
- Ressuspender o sedimento celular em 100 μL de tampão de lise de ARN frio fornecido em um kit adequado para extração de RNA.
- Alternativamente, colete derivados neuronais / gliais diretamente das placas aspirando o meio e adicionando tampão de lise de RNA frio aos poços para coletar as células.
- Isolar o RNA seguindo as instruções do fabricante.
- Reverse-transcribe 500 ng do RNA total utilizando um kit adequado para RNA para cDNA retrotranscription.
- Execute reações de qPCR em duplicado usando a mistura principal apropriada e o iniciadorConjuntos (veja a Tabela de Materiais ).
- Registre a emissão fluorescente em tempo real: 45 ciclos com recozimento de primers a 60 ° C.
- Normalize as quantidades relativas de RNA para GAPDH e β-actina como genes de referência e use hiPSCs indiferenciados ou células não tratadas para as condições de calibração (método ΔΔCt). Alternativamente, use outro método adequado.
- Imunocitoquímica e imagens de alto conteúdo (HCI) 6 , 11
- Corrigir as colónias hiPSC, NSCs e / ou derivados neuronais / gliais com paraformaldeído frio a 4% durante 15 minutos à temperatura ambiente.
- Lavar suavemente as células em 1X PBS e armazenar as placas a 4 ° C por até 1 mês.
- Quando pronto para coloração, permeabilize as células em tampão de permeabilização (1x DPBS contendo 0,1% de triton-X-100 e 3% de BSA) durante 15 min à temperatura ambiente.
- Remova a permeabilização buFfer e incubar as células em tampão de bloqueio (3% BSA / 1X DPBS) durante 15 min à temperatura ambiente para evitar a ligação não específica de anticorpos.
- Remova o tampão de bloqueio e incube as células durante a noite a 4 ° C em tampão de bloqueio contendo anticorpos primários adequados (ver Tabela de Materiais ).
- Lave as células 3 vezes com 1x PBS.
- Incubar as células durante 45 min a temperatura ambiente em tampão de bloqueio contendo anticorpos secundários conjugados com fluorochrome (ver Tabela de Materiais ), contra-coloração dos núcleos com corante DAPI.
- Quantifique a intensidade média de fluorescência e as porcentagens relativas de tipos de células usando uma plataforma de imagem de alto conteúdo adequada, se disponível (consulte a Tabela de Materiais ).
NOTAS: Para determinar o nível de intensidade do fundo fluorescente, incubar algumas células / poços com anticorpos secundários isolados. A análise citométrica de fluxo de vida (não fixa), undOs hiPSCs indiferenciados podem ser realizados para avaliar a expressão de marcadores específicos de PSC, como SSEA4 (ver Tabela de Materiais ). As colônias HiPSC indiferenciadas podem ser analisadas para a atividade da fosfatase alcalina usando kits BCIP / NBT comercialmente disponíveis, seguindo as instruções do fabricante (veja a Tabela de Materiais ). Além disso, os ensaios e análises da matriz de proteína de fase inversa (RPPA) podem ser realizados, conforme descrito na Referência 12 (para uma lista de anticorpos testados, veja a Tabela de Materiais ).
- Medições eletrofisiológicas 13
- Placa os fragmentos de roseta dissociados (após 7 DIV) ou NSCs derivados de rosetas em matrizes de multieletrodos revestidos (MEAs, veja a Tabela de Materiais ) em meio ND completo (~ 1 x 10 5 células / chip MEA de um único poço).
- Diferencie as células por 3 semanas no meio completo de ND, refrescandoE médio duas vezes por semana.
- No final da diferenciação, selar os chips MEA com uma membrana semi-permeável sob uma capa de fluxo laminar para manter as culturas estéreis para medições repetidas.
- Substitua um dos eletrodos por uma referência de terra, permitindo gravações dos eletrodos restantes.
- Registre a taxa de disparo média (MFR, número de espias / min) usando um amplificador MEA com o controle de temperatura integrado ajustado a 37 ° C e 5% de CO 2 .
- Detectar picos dos dados brutos do MEA usando o limite limite de -4.7σ (σ representa o desvio padrão do ruído basal).
- Processar os dados de pós-gravação com um software adequado.
Resultados
Caracterização de hiPSCs indiferenciados
Para avaliar o fenótipo dos hiPSCs, a análise da morfologia das colônias / células, a determinação dos marcadores específicos do PSC e o exame da expressão gênica e da atividade do fosfato alcalino devem ser realizados. HiPSCs indiferenciados devem ser redondos, com nucleolos grandes e sem citoplasma abundante. A maioria das colônias deve ser caracterizada por uma morfologia plana e bem embalada, indicativa de um fenótipo indiferenciado ( Figura 2A e Figura 2B ). Além disso, mais de 80% das colônias devem ser positivas para a coloração da atividade da fosfatase alcalina ( Figura 2C ).
Cerca de 80% das células devem ser positivas para marcadores clássicos relacionados à pluripotência, como Oct4, SSEA3, SSEA4 e Tra1-60 ( Figura 2D-H ), como mostrado pela imunocitoquímica e citometria de fluxo, enquanto que as porcentagens de células nestin + e β-III-Tubulin + devem ser significativamente baixas (cerca de 8% e 3%, respectivamente, Como mostrado na Figura 2H ). Estes resultados devem ser reprodutíveis através de passagens.
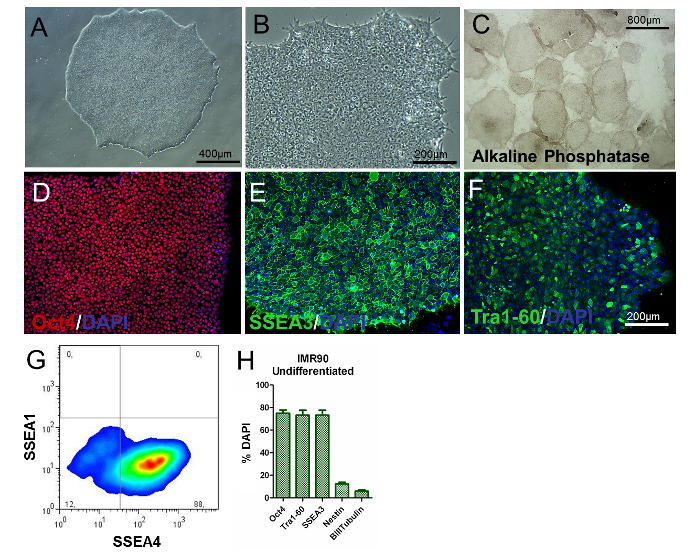
Figura 2. Caracterização de IMP90-hiPSC não diferenciados. (A e B) Imagens representativas de contraste de fase (amplificações 10X e 20X) de colônias indiferenciadas IMR90-hiPSC. (C) imagens representativas de colônias coloradas com fosfatase alcalina (ampliação de 4 x). (DF) Imagens imunocitoquímicas representativas de (D) Oct4 (vermelho), (E) SSEA3 (verde) e (F) TrA1-60 (verde). (G) Gráfico de pontos representativos da coloração SSEA1 (CD15) e SSEA4, analisada por citometria de fluxo. (H) O gráfico de barras mostra as porcentagens de Oct4 + (~ 75 - 80%), Tra1-60 + (~ 75 - 80%), SSEA3 + (~ 75 - 80%), nestin + (~ 10-15% ) E células β-III-tubulina + (~ 3 - 7%), contrastadas com DAPI e quantificadas por HCI, com uma média de 3 a 5 repetições biológicas ± o SEM (gráfico modificado da Referência 6). Clique aqui para ver uma versão maior desta figura.
Avaliação da pluripotência através da formação de EB
Os HiPSCs são pluripotentes, o que significa que eles expressam três genes relacionados à camada de germe em condições apropriadas. Para avaliar a pluripotência hiPSC, é possível aplicar umaAbordagem baseada na formação de EB espontânea, o que induz a formação das três camadas germinativas 14 . As análises dos genes específicos da camada de germe devem indicar um aumento dependente do tempo de endoderma (α-fetoproteína (AFP) e Cytokeratin 18 (KRT18)), ectoderme (Nestin, SRY-box 1 (Sox1) e caixa emparelhada 6 (Pax6) ) E mesoderma (expressão de genes relacionados com peptídeo natriurético A (NPPA) e Brachyury-T) ( Figura 3A e Figura 3B ); Veja a Tabela de Materiais.
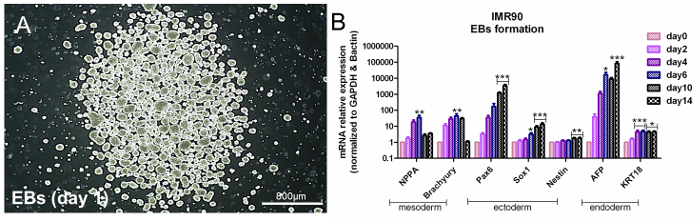
Figura 3. Avaliação da pluripotência pela formação de EB. (A) Imagem de contraste de fase representativa de EBs no dia 1. (B) O gráfico de barras mostra as análises de qPCR de mesodermal (NPPA e brachyury), ectodérmicas (Pax6, Sox1 e nestin) e endodermal (AFP e KRT18 ), Normalizados aos genes de referência, β-actina e GAPDH, e calibrados para células indiferenciadas (Dia 0). Este é o método ΔΔCt, com uma média de 5 análises independentes ± a SEM * p <0,05, ** p <0,01, *** p <0,001; Gráfico modificado da Referência 6. Clique aqui para ver uma versão maior dessa figura.
Indução de diferenciação neuronal e glial
IMR90-iPSCs podem ser diferenciados em culturas mistas de neurônios pós-mitóticos e células gliais seguindo os passos resumidos na Figura 1 e nas seções do protocolo. 5-8 dias após o chapeamento dos EBs em pratos padrão revestidos com matriz ou laminina na presença de meio NRI completo, as estruturas semelhantes a rosáceas devem começar a se tornar visíveis (= "Xfig"> Figura 4A). Os rosetas são caracterizados pela presença de células nestin + (precursores neuronais, ~ 90%), com poucas células β-III-tubulina + (células neuronais comprometidas, ~ 5-10%), estas geralmente localizadas principalmente na periferia da Rosetas ( Figura 4B , rosetas no dia 12).
Após a dissociação de rosetas e a substituição em pratos ou placas revestidos por matriz de laminina ou padrão na presença de meio ND completo, as células começam a se diferenciar em culturas misturadas de neurônios e glia, formando progressivamente grupos de corpos celulares neuronais conectados por feixes de neurites ( Figura 4C e Figura 4D ). Resultados similares devem ser obtidos quando se analisa as populações neuronais obtidas pela expansão das NSC derivadas de roseta e diferencia-las em neurônios e glia. NSCs expandidos a partir de rosetas devem ser nEstin + ( Figura 4E , inserção mostrando células nestin + ).
Após 21 dias de diferenciação, as células devem ser positivas para a β-III-tubulina; NF200; Tau; E MAP2, o marcador tardio de dendritos ( Figura 4D , 4F e Figura 4H), com pelo menos 10-15% das células positivas para a proteína ácida fibrilar glial (GFAP), um marcador astroglial ( Figura 4G e Figura 4H) . Além disso, ~ 20 - 30% das células devem reter a expressão do nestin após a diferenciação ( Figura 4H ). É importante considerar que a porcentagem de cada tipo de célula ( ou seja, neurônio, astrocito, células nestin + ) pode variar em relação a passagens, e a variabilidade dependente do usuário pode ser observada.
Ao analisar subpopulações neuronais específicas, os neurônios GABAérgicos representamEnviou ~ 15 - 20% das células totais, neurônios dopaminérgicos ~ 13 - 20% e neurônios glutamatérgicos ~ 35 - 42%, como mostrado pela imunocoloração para o ácido gama-aminobutírico (GABA), tirosina hidroxilase (TH) e glutamato vesicular Transportador 1 (VGlut1), respectivamente (ver a quantificação representativa na Figura 4H ). A indução de diferenciação também pode ser avaliada pela análise de marcadores relacionados à pluripotência ( por exemplo, Oct4, Tra1-60 e SSEA3), que deve ser significativamente downregulated em células diferenciadas versus hiPSCs indiferenciadas (não mostrado, ver Referência 6). Isso também pode ser confirmado através da análise da expressão gênica por qPCR, o que deve indicar uma diminuição de Oct4 e Nanog e a regulação positiva de genes neuronais, como a molécula de adesão de células neurais 1 (NCAM1) e a proteína 2 associada a microtúbulos (MAP2); O gene pré-sináptico, a sinaptofisina (SYP); E o gene pós-sináptico, proteína tau associada a microtúbulos (MAPT), como mostrado em < Classe forte = "xfig"> Figura 4I. Além disso, os genes neurônicos dopaminérgicos (TH e NR4A1), noradrenérgicos (PHOX2A e PHOX2B), glutamatérgicos (NARG2, GRIA1 e GAP43), GABAergic (GABRA1 e GABRA3), neurônios motores (ISL1 e LHX3) e colaterais colinérgicos (SLC5A7 e SLC18A13) Resultam em células neuronais com regulação positiva comparadas às células indiferenciadas ( Figura 4J ).
A análise da atividade elétrica espontânea, por meio do MEA, é uma leitura valiosa para avaliar a funcionalidade da rede neuronal em hiPSCs diferenciados. No final do período de diferenciação, os derivados neuronais são geralmente caracterizados por uma taxa média de disparo (MFR) de pelo menos 60 picos / min (ver o gráfico de quadriculação representativo na Figura 4K ). No entanto, as explosões não são observadas.
55702fig4.jpg "/>
Figura 4. Diferenciação de IMR90-hiPSCs em Culturas Mistas de Neurônios e Glia. (A e B) Imagens representativas de rosetas após 7 DIV (A) e após 12 DIV (B) , coradas para nestin (verde) e β-III-tubulin (vermelho)). (C e D) Imagens representativas de células diferenciadas após 22 DIV (C) e 28 DIV (D) , coradas para β-III-tubulin (vermelho) e NF200 (verde)). (E) Imagem representativa de NSCs derivada da dissociação e expansão da rosácea (inserção mostra nestin + células, vermelho). (F e G) Imagens representativas de células neuronais ( F , coradas para NF200 (vermelho) e Tau (verde)) e células gliais ( G , coradas para GFAP (vermelho)) diferenciadas de NSCs (após 21 DIV). (H) Quantificação de nestin, MAP2, GFAP, ácido gama-aminobutírico (GABA), transportador vesicular de glutamato 1 (VGlut1) e células positivas de tirosina hidroxilase (TH) por HCI, comparando derivados de IMR90-hiPSC e células diferenciadas de NSCs derivados de IMR90-hiPSC (gráfico modificado da Referência 7). (I e J) Gráficos de barras que mostram as análises qPCR de genes de pluripotência (Oct4 e Nanog) e genes neuronais (NCAM1, MAP2, SYP e MAPT) (I) e dopaminérgicos (TH e NR4A1), noradrenérgicos (PHOX2A e PHOX2B) Glutamatérgicos (NARG2, GRIA1 e GAP43), GABAergic (GABRA1 e GABRA3), neurônio motor (ISL1 e LHX3) e genes colinérgicos (SLC5A7 e SLC18A13) (J) . Todas as análises foram normalizadas para os genes de referência, β-actina e GAPDH, e calibradas para células indiferenciadas (barras verdes). Este é o método ΔΔCt, com uma média de 5 análises independentes ± a SEM * p <0,05, ** p <0,01, *** p <0,001. Os gráficos em I e J foram modificados a partir da Referência 6. (K) Gráfico de quadriculação representativa do neur derivado de IMR90-NSCOns (a gravação foi realizada por um mínimo de 600 s, as barras verticais representam espinhos únicos). Clique aqui para ver uma versão maior desta figura.
Uma assinatura específica de marcadores neuronais é regulada positivamente em IMR90-hiPSC diferenciados
No novo paradigma de teste de toxicidade, é essencial definir os eventos moleculares e celulares que ocorrem dentro de uma célula após a exposição a um determinado tóxico. Portanto, é relevante caracterizar quais caminhos de sinalização são ativados e / ou upregulados dentro do modelo celular sob investigação.
As matrizes comercialmente disponíveis para a análise da expressão do gene da proteína quinase podem ser usadas para comparar hiPSCs indiferenciadas versus células diferenciadas. DifferenOs IMPS90-hiPSCs são submetidos à regulação positiva de genes envolvidos no controle de receptores de neurotrofina, regulação de orientação axônica, modulação de crescimento de neurite, receptores de fator neurotrófico derivado de glial (GDNF), proteína de proteína morfogenética óssea (TGPB) e via plaquetária- Receptores do fator de crescimento derivado (PDGF) ( Figura 5A e Figura 5B ).
A análise do RPPA mostra a regulação positiva de uma assinatura neuronal específica em IMR90-hiPSCs diferenciados. Em particular, as vias de sinalização Erk / CREB, Akt / PDK1 / mTOR e Notch1 são ativadas após a diferenciação ( Figura 5C ).
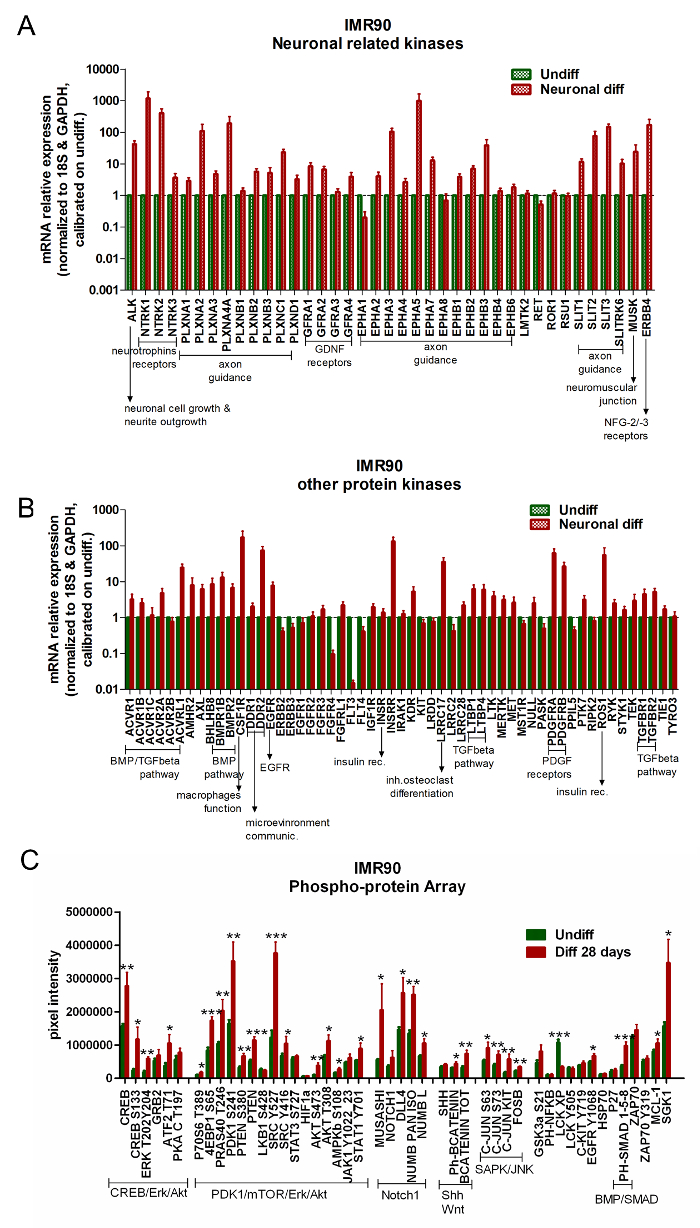
Figura 5. As células neuronais e gliais diferenciadas exibem a ativação de caminhos relacionados ao neurônio. (UMAE B) Gráficos de barras relatam as análises qPCR de quinases relacionadas com neurônios (A) e outros genes relacionados à quinase (B) . Os dados de expressão de genes foram normalizados para os genes de referência 18S e GAPDH (fornecidos na matriz) e calibrados para células indiferenciadas. Para essas análises, um gene foi considerado significativamente upregulated quando sua expressão era pelo menos 2 vezes maior do que em células indiferenciadas (2 -ΔΔtt ≥ 2); Significa 3 análises independentes ± a SEM). (C) O gráfico de barras mostra as quantificações de proteínas absolutas por meio da análise de RPPA, comparando as barras diferenciadas (barras vermelhas) e as células indiferenciadas (barras verdes). As proteínas pertencentes às mesmas cascatas da via de sinalização são agrupadas da seguinte forma: CREB / Erk / Akt, PDK1 / mTOR / Erk / Akt, Notch1, Shh e Wnt, SAPK / JNK e BMP / SMAD. Média ± a SEM de 4 análises independentes. * P <0,05, ** p <0,01, *** p <0,001; Gráfico modificado fRom Referência 6. Clique aqui para ver uma versão maior deste número.
As culturas neuronais / gliais derivadas de IMR90-hiPSC podem ser usadas para avaliar os efeitos da rotenona
Rotenone, um inibidor do complexo I da cadeia respiratória mitocondrial, é conhecido por causar estresse oxidativo, desencadeando a ativação da via Nrf2. Sob condições quiescentes, Nrf2 está ancorado no citoplasma por Keap1 (proteína Kelch-like ECH-associada 1), um repressor Nrf2, o que facilita ubiquitinação Nrf2 e proteólise 15 . Após a indução do estresse oxidativo, Nrf2 transloca para o núcleo e ativa a expressão dos genes alvo Nrf2-ARE 16 .
Neurônios derivados de IMR90-hiPSC eD células gliais podem ser usadas para avaliar os efeitos da rotenona na ativação de Nrf2 expondo as células a diferentes concentrações de rotenona ( por exemplo, 1, 10 e 100 nM) durante 24 h. Estas concentrações foram estabelecidas de acordo com estudos anteriores 17 , 18 .
Nessas concentrações e tempos de exposição, a rotenona não resultou em citotoxicidade, como mostra a quantificação de núcleos celulares DAPI + vivos ( Figura 6A ). Rotenone induziu translocação nuclear de Nrf2, especialmente depois de expor as células a rotenona 100 nM ( Figura 6B e Figura 6E ). Na mesma concentração, observou-se uma diminuição significativa no Keap1 citoplasmático ( Figura 6C ), juntamente com um aumento na NAD (P) H quinone oxidoreductase 1 (NQO1) e na Sulfiredoxina 1 (SRXN1), dois Nrf2-target enZymes 19 , 20 ( Figura 6D ).
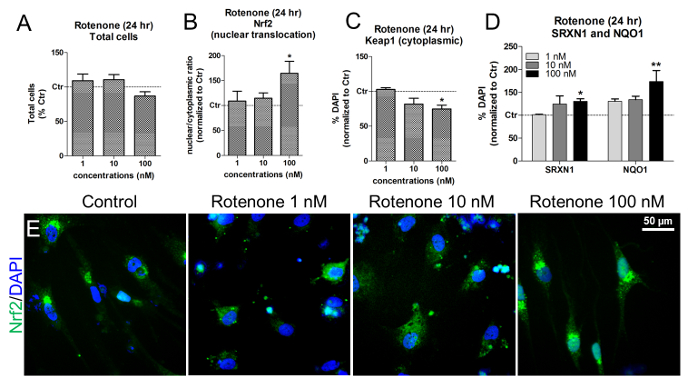
Figura 6. Efeitos da Rotenona nos níveis de proteína Nrf2 Translocação Nuclear, Keap1, SRXN1 e NQO1. (A) Quantificação de células DAPI + ao vivo ( ou seja, núcleos não-picógenos) após o tratamento com 24 h com rotenona 1, 10, 100 nM e células normalizadas para não tratadas (Control, Ctr). (B) Transferência nuclear da proteína Nrf2 ( ou seja, relações nucleares / citoplasmáticas) após 24 h de exposição à rotenona, avaliada por medidas de intensidade de fluorescência usando análise de HCI. (C) Quantificação dos níveis citoplasmáticos de proteína Keap1 após o tratamento com rotenona, avaliada pela análise de HCI. (D) Quantificação de NAD (P) H quinone oxidoreductase 1 (NQO1) e SulfiRedoxina 1 (SRXN1) por meio de imunofluorescência e HCl após 24 h de tratamento com rotenona. (E) Imagens representativas da localização da proteína Nrf2 (verde). Todos os valores são mostrados como média ± SEM de 3 repetições biológicas. * P <0,05, ** p <0,01; Figura modificada da Referência 7. Clique aqui para ver uma versão maior deste número.
Nessas concentrações e tempos de tratamento, a rotenona também provocou um aumento dependente da concentração de porcentagem de células astrogliais (GFAP + ) ( Figura 7A e Figura 7B ), sem afetar as proporções de NSCs (nestin + ) e neurônios (MAP2 + ) ( Figura 7B ). Ao analisar as proporções de subtipos neuronais específicos, o tratamento com rotenona (10 nM e 100 nM)Diminuiu significativamente o número de neurônios dopaminérgicos (TH + ) ( Figura 7C e D ), enquanto que as percentagens de neurônios GABAérgicos (GABA + ) e glutamatérgicos (VGlut1 + ) não mudaram ( Figura 7D ). Analogamente, estudos anteriores in vivo e in vitro descreveram uma morte celular neuronal dopaminérgica dependente de rotenona 21 , 22 , 23 .
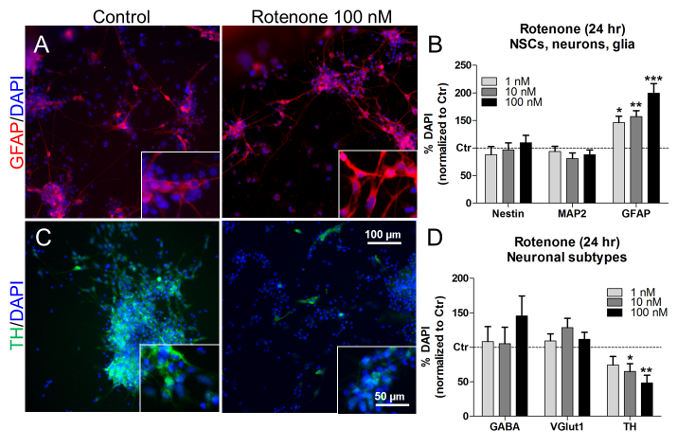
Figura 7. Efeitos da Rotenona em Células Gliais e Neurônios Dopaminérgicos. (A) Imagens representativas das células GFAP + (vermelho), com ampliação 40X nas inserções, não tratadas ou tratadas com rotenona 100 nM por 24 h. (B) Quantificação Das células nestin + , MAP2 + e GFAP + , normalizadas para células não tratadas (controle, Ctr). (C) Imagens representativas de neurônios dopaminérgicos TH + (verde), com ampliação 40X nas inserções, não tratadas ou tratadas com rotenona 100 nM durante 24 h. (D) Quantificação de células neuronais GABA + , VGlut1 + e TH + , normalizadas para células não tratadas (Ctr). Todos os valores são mostrados como média ± SEM de 3 repetições biológicas. * P <0,05, ** p <0,01, *** p <0,001; Figura modificada da Referência 7. Clique aqui para ver uma versão maior deste número.
O significado estatístico foi avaliado pela ANOVA unidirecional com o Teste de comparação múltipla da Dunnett como pós-teste (comparando todas as colunas versus a coluna de controle)Xref "> 24 ou por t-testar duas vezes ou não pareado de acordo com o tipo de análise. Todos os dados representam a média de pelo menos três repetições biológicas ± o erro padrão da média (SEM). Um asterisco sobre uma barra indica Uma diferença significativa com o grupo de controle. * P <0,05, ** p <0,01, *** p <0,001.
Tabela 1.
Nota sobre a densidade do revestimento da roseta dissociada: se os fragmentos de rosácea não parecem completamente dissociados, para atingir uma densidade de revestimento celular de cerca de 15.000 células / cm2, fragmentos de rosetas dissociadas provenientes de cerca de 50 EBs / 1 x 60mm-dish podem ser ressuspensos em 50 mL de meio de NRI completo e chapeados da seguinte maneira (dependendo do formato da placa):
| Placas Multiwell / MEA | Área de crescimento (cm 2 / bem) | Volume de suspensão celular à placa por poço (ou chip MEA) | Número máximo de placas que podem ser plaqueadas (com 50 ml de suspensão celular) |
| 96 poços | 0,3 | 100 ul | 5 |
| 48 poços | 0,7 | 220 ul | 4 |
| 24 poços | 2 | 625 ul | 3 |
| 12 poços | 4 | 1,25 ml | 3 |
| 6 poços | 10 | 3,125 ml | 2 |
| Único poço chip MEA | 3.5 | 1,1 ml | 45 |
Tabela 2: Critérios de Aceitação
| Marcador /Anticorpo | Porcentagem (em células DAPI + (ao vivo) após 28 DIV |
| B-III-tubulina (Tuj1) | 35-45% |
| MAP2 | 50-60% |
| NF200 | 45-55% |
| GFAP | 10-25% |
| Nestin | 15-25% |
Discussão
Este trabalho descreve um protocolo robusto e relativamente rápido para a diferenciação de IMR90-hiPSCs em neurônios pós-mitóticos e células gliais. Os protocolos de diferenciação neuronal previamente publicados baseados em hESCs e HiPSCs geralmente produzem altas porcentagens de precursores neurais 25 , 26 e um número significativo de células alvo neuronais 27 , 28 , 29 , 30 , 31 , 32 , 33 . Analogamente, o protocolo de diferenciação descrito aqui é adequado para gerar culturas heterogêneas de células neuronais GABAérgicas, glutamatérgicas e dopaminérgicas, juntamente com glia e uma proporção discreta de células nestin + . A presença de células neurais glutamatérgicas (~ 35-42%) e GABAérgicas (~ 15-20%) sugere queEssa cultura possui características anteriores do cérebro, características corticais e a presença de um número discreto de neurônios dopaminérgicos (~ 13-20%) também pode indicar especificidade mesencéfalo. Além disso, a permanência de uma proporção modesta de células nestin + pode ser adequada para o estudo da neurogênese e os possíveis efeitos de substâncias químicas em NSCs, que são principalmente confinados tanto ao hipocampo quanto à zona subventricular (SVZ) do prosencéfalo 34 . Outras análises imunocitoquímicas e de expressão gênica ajudariam a definir melhor a especificidade regional dos derivados de células diferenciadas.
Os dois passos mais críticos no protocolo de diferenciação descrito neste documento são: (i) o corte de colônias de hiPSC em fragmentos homogêneos (o que é crítico para a geração de EBs com tamanhos homogêneos) e (ii) o corte de estruturas neuroectodérmicas (rosetas ) Para diferenciação NSC, que requer habilidade manual significativaE precisão para evitar a coleta de células mesodérmicas e endodérmicas que podem reduzir as proporções de neurônios e células gliais obtidas após a diferenciação.
É crucial caracterizar os fenótipos das células durante a expansão (como colônias indiferenciadas ou NSCs) e durante todas as etapas de diferenciação. Em particular, os perfis de expressão de genes e proteínas dos derivados das células neuronais / gliais devem mostrar uma regulação positiva e ativação de caminhos de sinalização relacionados aos neurônios, enquanto que a expressão de marcadores de pluripotência deve ser diminuída.
A geração de EBs e derivados neuroectodermais (rosetas) podem ser desafiantes manualmente e propensos a variabilidade. Por esse motivo, desenvolvemos um protocolo para a expansão de NSC derivadas de roseta e sua maior diferenciação em células neuronais / gliais.
Possíveis limitações deste protocolo de diferenciação são principalmente (i) a porcentagem relativamente baixa de dDerivados gliais indiferenciados e (ii) Falta de funções de rede neuronal maduras (como mostra a falta de explosões). Além disso, subpopulações específicas de astrocitos podem funcionar como progenitores primários ou NSCs 35 . Embora não tenham sido observadas células níquel / GFAP duplo-positivas nesta cultura de células diferenciadas (dados não apresentados), a hipótese de que as células GFAP + nestas culturas são progenitores astrócitos e astrócitos. É plausível que, ao ampliar o tempo de diferenciação, o número de astrocitos pode aumentar, e sua morfologia pode tornar-se mais madura, como já foi indicado por trabalhos anteriores do grupo 36 , 37 de Zhang.
No novo paradigma de teste de toxicidade, o conhecimento sobre perturbações induzidas por produtos químicos das vias biológicas é de extrema importância na avaliação da adversidade química. Portanto, os sistemas de teste in vitro devem ser capazes deRelacionar efeitos adversos com os distúrbios das vias de sinalização, de acordo com o conceito de via de desfecho adverso (AOP). Como uma prova de conceito, a rotenona pode ser usada para avaliar a ativação da via Nrf2, que está envolvida na defesa celular contra o estresse oxidativo ou eletrofílico 38 e o estresse oxidativo é um evento chave importante e comum em vários AOPs relevantes para Neurotoxicidade no desenvolvimento e adulto 39 .
Rotenone deve induzir a ativação da via Nrf2, que pode ser demonstrada pela translocação nuclear da proteína Nrf2 e pelo aumento da expressão de enzimas alvo Nrf2, incluindo NQO1 e SRXN1. Verificou-se que a rotenona induz um aumento dose-dependente de níveis de proteína GFAP, indicativo de ativação de astrocitos 40 , 41 . Rotenone também diminui o número de células dopaminérgicas (TH + ), que está de acordo com previEstudos in vitro e in vivo que mostram morte celular dopaminérgica dependente de rotenona, uma vez que este tipo de neurônio é particularmente sensível ao estresse oxidativo 21 , 22 , 23 .
Em conclusão, este modelo de cultura celular neuronal e glial derivado de HiPSC é uma ferramenta valiosa para avaliar os efeitos neurotóxicos de produtos químicos que provocam estresse oxidativo, resultando na ativação da via Nrf2. Como este protocolo de diferenciação permite a geração de culturas mistas de células neuronais (neurônios GABAérgicos, dopaminérgicos e glutamatérgicos) e astrócitos, pode ser adequado para estudar a interferência entre neurônios e glia em condições fisiológicas e patológicas, como em doenças neurodegenerativas ( Por exemplo, doença de Parkinson). Além disso, a presença de uma proporção significativa de NSCs pode ajudar a avaliar os possíveis efeitos de produtos químicos no prog neuralEnors, que são conhecidos como o alvo principal de mutações ou infecções virais quimicamente induzidas 42 .
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Os autores agradecem ao Dr. Marc Peschanski (I-Stem, Évry, França), por fornecer os IMR90-hiPSCs; Dr. Giovanna Lazzari e Dr. Silvia Colleoni (Avantea srl, Cremona, Itália); Dr. Simone Haupt (Universidade de Bonn, Alemanha); Dr. Tiziana Santini (Instituto Italiano de Tecnologia, Roma), para fornecer aconselhamento sobre a avaliação da coloração por imunofluorescência; Dr. Benedetta Accordi, Dr. Elena Rampazzo e Dr. Luca Persano (Universidade de Pádua, Itália), por suas contribuições para a análise de RPPA e validação de anticorpos. Financiamento: este trabalho foi apoiado pelo projeto financiado pela UE "SCR & Tox" (Acordo de Subsídio N ° 266753).
Materiais
| Name | Company | Catalog Number | Comments |
| Complete hiPSC medium: | |||
| mTeSR1 Basal Medium | Stem Cell Technologies | 05851 | (Step 1.2.6). Complete mTeSR1 is stable when stored at 2 - 8°C for up to 2 weeks. 5X Supplements can be dispensed into working aliquots and stored at -20°C. Use frozen aliquots within 3 months. |
| mTeSR1 5X Supplements | Stem Cell Technologies | 05852 | |
| Matrigel hESC-qualified Matrix | Corning | 354277 | 1:100 (Step 1.1). Thaw Matrigel on ice, prepare 200 ul aliquots and store them in -80°C. For coating, dilute 200ul aliquot in 20 ml of DMEM/F12 medium. |
| CryoStem Freezing Medium | Stemgent | 01-0013-50 | Freeze ~ 100 fragments/250 ul/vial (Step 1.2.1) |
| Name | Company | Catalog Number | Comments |
| hiPSC EB medium: | |||
| Knockout DMEM | Thermo-Fisher | 10829-018 | (Step 2.1.7) |
| Knockout Serum Replacement (KOSR) | Thermo-Fisher | 10828-028 | 20% final concentration (Step 2.1.7) |
| Non-Essential Amino Acids | Thermo-Fisher | 11140-035 | (Step 2.1.7) |
| Penicillin/Streptomycin | Thermo-Fisher | 15140-122 | 50 U/mL final concentration (Step 2.1.7) |
| L-Glutamine 200 mM Solution | Thermo-Fisher | 25030-081 | 2 mM final concentration (Step 2.1.7) |
| β-Mercaptoethanol | Thermo-Fisher | 31350-010 | 50 µM final concentration (Step 2.1.7) |
| Name | Company | Catalog Number | Comments |
| Complete neuroepithelial induction medium (NRI): | |||
| DMEM/F12 | Thermo-Fisher | 3133-038 | (Step 2.3.1) |
| Non-Essential Amino Acids | Thermo-Fisher | 11140-035 | (Step 2.3.1) |
| N2 Supplement | Thermo-Fisher | 17502-048 | (Step 2.3.1) |
| Penicillin/Streptomycin | Thermo-Fisher | 15140-122 | 50 U/mL final concentration (Step 2.3.1) |
| Heparin Grade I-A, ≥180 USP units/mg | Sigma-Aldrich | H3149-100KU | 2 µg/ml final concentration (Step 2.3.1) |
| bFGF | Thermo-Fisher | 13256-029 | 20 ng/ml final concentration added before use (Step 2.3.1) |
| Matrigel Basement Membrane Matrix | Corning | 354234 | 1:100 (Step 2.2). Thaw Matrigel on ice, prepare 200 ul aliquots and store them in -80°C. For coating, dilute 200 ul aliquot in 20 ml of cold DMEM/F12 medium. |
| Laminin | Sigma-Aldrich | L2020 | 1:100 (Step 2.2). Dilute in PBS 1X. |
| Name | Company | Catalog Number | Comments |
| Complete Neuronal Differentiation medium (ND): | |||
| Neurobasal Medium | Thermo-Fisher | 21103049 | (Step 2.4.11) |
| B-27 Supplements (50x) | Thermo-Fisher | 17504044 | (Step 2.4.11) |
| N2 Supplement | Thermo-Fisher | 17502-048 | (Step 2.4.11) |
| Penicillin/Streptomycin | Thermo-Fisher | 15140-122 | 50 U/mL final concentration (Step 2.4.11) |
| GDNF | Thermo-Fisher | PHC7045 | 1 ng/ml final concentration. Added before use. (Step 2.4.11) |
| BDNF | Thermo-Fisher | PHC7074 | 2.5 ng/ml final concentration. Added before use. (Step 2.4.11) |
| Name | Company | Catalog Number | Comments |
| Neural induction medium (NI): | |||
| DMEM/F12 | Thermo-Fisher | 3133-038 | (Step 3.3) |
| Non-Essential Amino Acids | Thermo-Fisher | 11140-035 | (Step 3.3) |
| N2 Supplement | Thermo-Fisher | 17502-048 | (Step 3.3) |
| Penicillin/Streptomycin | Thermo-Fisher | 15140-122 | 50 U/mL final concentration (Step 3.3) |
| Heparin Grade I-A, ≥180 USP units/mg | Sigma-Aldrich | H3149-100KU | 2 µg/ml final concentration (Step 3.3) |
| B-27 Supplement (50X), minus vitamin A | Thermo-Fisher | 12587010 | (Step 3.3) |
| L-Glutamine 200 mM Solution | Thermo-Fisher | 25030-081 | 2 mM final concentration (Step 3.3) |
| bFGF | Thermo-Fisher | 13256-029 | 10 ng/ml final concentration. Added before use (Step 3.3) |
| EGF | Thermo-Fisher | PHG6045 | 10 ng/ml final concentration. Added before use (Step 3.3) |
| BDNF | Thermo-Fisher | PHC7074 | 2.5 ng/ml final concentration. Added before use (Step 3.3) |
| Defined Trypsin Inhibitor (DTI) | Thermo-Fisher | R007-100 | Pre-warm at 37°C. Add an equal amount of DTI to Trypsin-EDTA (Step 3.10) |
| Trypsin-EDTA (0.5%), no phenol red | Thermo-Fisher | 15400054 | 1:10. Dilute Trypsin-EDTA in PBS 1x (without calcium and magnesium), pre-warm the solution at 37°C (Step 3.8) |
| CryoStor cell cryopreservation medium | Sigma-Aldrich | C2874-100ML | (Step 4.2) |
| Trypan Blue (0.4%) | Sigma-Aldrich | T8154-100ML | multiple manufacturers/suppliers |
| Name | Company | Catalog Number | Comments |
| TaqMan Probesets and reagents for gene expression analysis: | |||
| RNAqueous-Micro kit | Thermo-Fisher | AM1931 | (Step 5.1.6) |
| High Capacity cDNA Reverse Transcription Kits | Thermo-Fisher | 4368814 | |
| TaqMan Gene Expression Master Mix | Thermo-Fisher | 4369016 | |
| GFAP | Thermo-Fisher | Hs00909233_m1 | |
| MAP2 | Thermo-Fisher | Hs00258900_m1 | |
| NQO1 | Thermo-Fisher | Hs02512143_s1 | |
| SRXN1 | Thermo-Fisher | Hs00607800_m1 | |
| HMOX1 | Thermo-Fisher | Hs01110250_m1 | |
| GSR | Thermo-Fisher | Hs00167317_m1 | |
| PAX6 | Thermo-Fisher | Hs01088112_m1 | |
| NES | Thermo-Fisher | Hs00707120_s1 | |
| GRIA1 | Thermo-Fisher | Hs00181348_m1 | |
| GAP43 | Thermo-Fisher | Hs00967138_m1 | |
| GABRA3 | Thermo-Fisher | Hs00968132_m1 | |
| GABRA1 | Thermo-Fisher | Hs00168058_m1 | |
| NR4A2 | Thermo-Fisher | Hs00428691_m1 | |
| TH | Thermo-Fisher | Hs00165941_m1 | |
| GAPDH | Thermo-Fisher | Hs02758991_g1 | |
| ACTB | Thermo-Fisher | Hs99999903_m1 | |
| MAPT | Thermo-Fisher | Hs00902194_m1 | |
| SYP | Thermo-Fisher | Hs00300531_m1 | |
| NANOG | Thermo-Fisher | Hs04260366_g1 | |
| POU5F1 (OCT4) | Thermo-Fisher | Hs04195369_s1 | |
| SOX1 | Thermo-Fisher | Hs01057642_s1 | |
| AFP | Thermo-Fisher | Hs00173490_m1 | |
| KRT18 | Thermo-Fisher | Hs01941416_g1 | |
| NPPA | Thermo-Fisher | Hs00383230_g1 | |
| T | Thermo-Fisher | Hs00610080_m1 | |
| NCAM1 | Thermo-Fisher | Hs00941821_m1 | |
| NR4A1 | Thermo-Fisher | Hs00374226_m1 | |
| PHOX2A | Thermo-Fisher | Hs00605931_mH | |
| PHOX2B | Thermo-Fisher | Hs00243679_m1 | |
| NARG2 | Thermo-Fisher | Hs00973298_g1 | |
| SLC18A3 | Thermo-Fisher | Hs00268179_s1 | |
| SLC5A7 | Thermo-Fisher | Hs00222367_m1 | |
| ISL1 | Thermo-Fisher | Hs00158126_m1 | |
| LHX3 | Thermo-Fisher | Hs01033412_m1 | |
| TaqMan Human Protein Kinase Array | Thermo-Fisher | 4418721 | |
| Name | Company | Catalog Number | Comments |
| Antibodies and reagents for immunostaining: | |||
| B-III-tubulin (Tuj1) | Covance | MMS-435P | 1:500 (Step 5.2.5). Other antibodies may also be used. |
| MAP2 | Sigma Aldrich | M4403 | 1:500 |
| NF200 | Sigma Aldrich | N4142 | 1:1000 |
| GFAP | Acris Antibodies GmbH | AP02002SU-N | 1:500 |
| Nestin | Sigma-Aldrich | N5413 | 1:200 |
| synaptophysin (SYN) | Abcam | AB14692 | 1:200 |
| Tau | Thermo-Fisher | MA5-12808 | 1:100 |
| Nrf2 | Abcam | AB62352 | 1:200 |
| Keap1 | Abcam | AB66620 | 1:200 |
| sulfiredoxin1 (SRXN1) | Abcam | AB92298 | 1:200 |
| NAD(P)H quinone oxidoreductase 1 (NQO1) | Abcam | AB2346 | 1:200 |
| OCT4 | Millipore | MAB4401 | 1:100 |
| SSEA3 | Millipore | MAB4303 | 1:100 |
| Tra1-60 | Millipore | MAB4360 | 1:250 |
| Tyrosine hydroxylase (TH) | Millipore | AB152 | 1:200 |
| Gamma-aminobutyric acid (GABA) | Sigma-Aldrich | A0310 | 1:100 |
| Vesicular glutamate transporter 1 (VGlut1) | Abcam | AB72311 | 1:500 |
| Paraformaldehyde | Sigma-Aldrich | P6148-500G | 4% (4% formaldehyde can also be used) |
| DPBS, no calcium, no magnesium | Thermo-Fisher | 14190144 | |
| Triton-X-100 Solution | Sigma-Aldrich | 93443-100ML | 0.1% |
| BSA 35% | Sigma-Aldrich | A7979-50ML | 3.5% |
| Donkey anti-Rabbit IgG (H+L) Cross Adsorbed Secondary Antibody, DyLight 594 conjugate | Thermo-Fisher | SA5-10040 | 1:500. (Step 5.2.7) Other fluorochrome-conjugated secondary antibodies may also be used. In this case, appropriate dilutions should be tested by the enduser. |
| Donkey anti-Mouse IgG (H+L) Cross Adsorbed Secondary Antibody, DyLight 488 conjugate | Thermo-Fisher | SA5-10166 | 1:500 |
| Donkey anti-Goat IgG (H+L) Cross Adsorbed Secondary Antibody, DyLight 488 conjugate | Thermo-Fisher | SA5-10086 | 1:500 |
| DAPI Solution (1 mg/ml) | Thermo-Fisher | 62248 | 1:1000 (Step 5.2.7) |
| Name | Company | Catalog Number | Comments |
| Antibodies for Reverse Phase Protein Array (RPPA): | |||
| 4E-BP1 (S65) | Abcam | AB81297 | 1:250 (Note after step 5.2.8) |
| Akt (T308) | Cell Signaling | 9275 | 1:100 |
| Akt (S473) | Cell Signaling | 9271 | 1:100 |
| AMPKalpha (T172) | Cell Signaling | 2531 | 1:100 |
| AMPKbeta1 (S108) | Cell Signaling | 4181 | 1:100 |
| ATF-2 (T71) | Cell Signaling | 9221 | 1:100 |
| c-Jun (S63) | Cell Signaling | 9261 | 1:200 |
| c-Jun (S73) | Cell Signaling | 9164 | 1:200 |
| c-Kit (Y719) | Cell Signaling | 3391 | 1:250 |
| CREB (S133) | Cell Signaling | 9191 | 1:100 |
| EGFR (Y1068) | Cell Signaling | 2234 | 1:50 |
| ErbB2/HER2 (Y1248) | Cell Signaling | 2247 | 1:100 |
| ERK 1/2, p44/42 (T202/Y204) | Cell Signaling | 9101 | 1:2000 |
| GSK-3alpha (S21) | Cell Signaling | 9337 | 1:50 |
| Jak1 (Y1022/1023) | Cell Signaling | 3331 | 1:100 |
| Lck (Y505) | Cell Signaling | 2751 | 1:500 |
| LKB1 (S428) | Cell Signaling | 3051 | 1:100 |
| mTOR (S2448) | Cell Signaling | 5536 | 1:100 |
| NFkB p65 (S536) | Cell Signaling | 3031 | 1:50 |
| p70 S6 Kinase (T389) | Cell Signaling | 9205 | 1:200 |
| PDK1 (S241) | Cell Signaling | 3061 | 1:100 |
| PKA C (T197) | Cell Signaling | 4781 | 1:250 |
| PRAS40 (T246) | BioSource | 44-1100 | 1:2000 |
| PTEN (S380) | Cell Signaling | 9551 | 1:500 |
| Smad1 (S463/465), Smad5 (S463/465), Smad8 (S426/428) | Cell Signaling | 9511 | 1:500 |
| Src (Y527) | Cell Signaling | 2105 | 1:500 |
| Src Family (Y416) | Cell Signaling | 2101 | 1:200 |
| Stat1 (Y701) | Cell Signaling | 9171 | 1:200 |
| Stat3 (S727) | Cell Signaling | 9134 | 1:200 |
| Zap-70 (Y319) | Enogene | E011159 | 1:100 |
| βCatenin (S33/37/T41) | Cell Signaling | 9561 | 1:250 |
| CREB | Upstate Biotechnologies | 06-863 | 1:100 |
| Fos B | Cell Signaling | 2251 | 1:200 |
| GRB2 | Cell Signaling | 3972 | 1:2000 |
| HSP70 | Stressgen | SPA-810 | 1:100 |
| c-Jun | Cell Signaling | 9165 | 1:100 |
| Kip1/p27 | BD | 610241 | 1:100 |
| Lck | Cell Signaling | 2984 | 1:250 |
| Mcl-1 | Cell Signaling | 4572 | 1:80 |
| Musashi | Cell Signaling | 2154 | 1:100 |
| NOTCH1 | Cell Signaling | 3439 | 1:100 |
| PTEN | Cell Signaling | 9552 | 1:500 |
| SGK1 | Abnova | PAB4590 | 1:250 |
| Zap-70 | Cell Signaling | 2705 | 1:250 |
| β-Catenin | Abcam | AB32572 | 1:1000 |
| Dll4 | Abcam | AB7280 | 1:500 |
| Shh | Abcam | AB53281 | 1:250 |
| HIF-1α | BD | 610958 | 1:50 |
| NUMB PAN-ISO | Upstate Biotechnologies | 07-207 | 1:400 |
| NUMB-L | Chemicon | AB15145 | 1:750 |
| Cyclin B | BD | 610220 | 1:75 |
| c-Myc | Calbiochem | OP-10 | 1:100 |
| BCIP/NBT Kit | Thermo-Fisher | 002209 | (Note after step 5.2.8). Kit used to measure alkaline phosphatase activity, similar kits can be used. |
| Name | Company | Catalog Number | Comments |
| Antibodies for Flow Cytometry: | |||
| SSEA1 Antibody, Pacific Blue conjugate | Thermo-Fisher | MHCD1528 | 1:100 (Note after step 5.2.8) |
| SSEA4 Antibody (MC813-70), Alexa Fluor 647 | Thermo-Fisher | SSEA421 | 1:100 |
| Name | Company | Catalog number | Comments |
| Specific instruments, tools and softwares: | |||
| Countess Automated Cell Counter | Thermo-Fisher | C10227 | Neubauer chamber or other suitable glass hemocytometer can be used. |
| MEA1060-Inv-BC | Multichannel Systems | MEA1060-Inv-BC | (Step 5.3) |
| MEA1060-BC control software | Multichannel Systems | MEA1060-BC | (Step 5.3) |
| NeuroExplorer | Multichannel Systems | NeuroExplorer (NE) | (Step 5.3) For post-processing of MEA data |
| Multielectrode arrays (MEA) | Multichannel Systems | 60MEA100/10iR-Ti-gr | (Step 5.3) Single-well MEA chip |
| ArrayScan XTI High Content Platform | Thermo-Fisher | ASN00002P | (Step 5.2.8) Mean fluorescence can be quantified by using specific ArrayScan algorithms (e.g., Cytotoxicity V.4 and NucTrans V.4 bioapplications). It is recommended to take minimum 20 pictures/well, and have 7-8 internal replicates per condition |
| 7900HT Fast Real-Time PCR System | Thermo-Fisher | 4351405 | (Step 5.1.6) |
| BD ULTRA-FINE Needle Insulin Syringe (with 30G needle) | BD | 328280 | (Steps 1.3.1, 2.1.2, and 2.4.1) |
| StemPro EZPassage Disposable Stem Cell Passaging Tool | ThermoFisher | 23181010 | This colony cutting tool can be used as an alternative to the use of 30G needle 1 mL syringes (Step 1.3.1) |
| Ultra-Low attachment Petri dish (60 mm) | Corning | 10010582 | (Step 2.1.8) Also other brands can be used. |
| Mr. Frosty Freezing container | Sigma-Aldrich | C1562-1EA | |
Referências
- NRC. . Toxicity Testing in the 21st Century: A Vision and a Strategy. , (2007).
- Drews, K., Jozefczuk, J., Prigione, A., Adjaye, J. Human induced pluripotent stem cells--from mechanisms to clinical applications. J Mol Med (Berl). 90 (7), 735-745 (2012).
- Ho, P. J., Yen, M. L., Yet, S. F., Yen, B. L. Current applications of human pluripotent stem cells: possibilities and challenges. Cell Transplant. 21 (5), 801-814 (2012).
- Krueger, W. H., Swanson, L. C., Tanasijevic, B., Rasmussen, T. P. Natural and artificial routes to pluripotency. Int J Dev Biol. 54 (11-12), 1545-1564 (2010).
- Pistollato, F., Bremer-Hoffmann, S., Healy, L., Young, L., Stacey, G. Standardization of pluripotent stem cell cultures for toxicity testing. Expert Opin Drug Metab Toxicol. 8 (2), 239-257 (2012).
- Pistollato, F., et al. Development of a pluripotent stem cell derived neuronal model to identify chemically induced pathway perturbations in relation to neurotoxicity: effects of CREB pathway inhibition. Toxicol Appl Pharmacol. 280 (2), 378-388 (2014).
- Zagoura, D., Canovas-Jorda, D., Pistollato, F., Bremer-Hoffmann, S., Bal-Price, A. Evaluation of the rotenone-induced activation of the Nrf2 pathway in a neuronal model derived from human induced pluripotent stem cells. Neurochem Int. , (2016).
- Standard operating procedure for differentiation of human induced pluripotent stem cells into post-mitotic neurons and glial cells. EURL ECVAM Available from: https://ecvam-dbalm.jrc.ec.europa.eu/methods-and-protocols/protocol/standard-operating-procedure-for-differentiation-of-human-induced-pluripotent-stem-cells-into-post-mitotic-neurons-and-glial-cells-%28mixed-culture%29-protocol-no.-165/key/p_1570 (2016)
- Standard operating procedure for expansion of rosette-derived neural stem cells. EURL ECVAM Available from: https://ecvam-dbalm.jrc.ec.europa.eu/methods-and-protocols/protocol/standard-operating-procedure-for-expansion-of-rosette-derived-neural-stem-cells-protocol-no.-166/key/p_1571 (2016)
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nat Protoc. 3 (6), 1101-1108 (2008).
- Brien, P. J., et al. High concordance of drug-induced human hepatotoxicity with in vitro cytotoxicity measured in a novel cell-based model using high content screening. Arch Toxicol. 80 (9), 580-604 (2006).
- Accordi, B., et al. Functional protein network activation mapping reveals new potential molecular drug targets for poor prognosis pediatric BCP-ALL. PLoS One. 5 (10), e13552 (2010).
- Vassallo, A., et al. A multi-laboratory evaluation of microelectrode array-based measurements of neural network activity for acute neurotoxicity testing. Neurotoxicology. , (2016).
- Shamblott, M. J., et al. Human embryonic germ cell derivatives express a broad range of developmentally distinct markers and proliferate extensively in vitro. Proc Natl Acad Sci U S A. 98 (1), 113-118 (2001).
- Bryan, H. K., Olayanju, A., Goldring, C. E., Park, B. K. The Nrf2 cell defence pathway: Keap1-dependent and -independent mechanisms of regulation. Biochem Pharmacol. 85 (6), 705-717 (2013).
- Tufekci, K. U., Civi Bayin, E., Genc, S., Genc, K. The Nrf2/ARE Pathway: A Promising Target to Counteract Mitochondrial Dysfunction in Parkinson's Disease. Parkinsons Dis. , 314082 (2011).
- Kovac, S., et al. Nrf2 regulates ROS production by mitochondria and NADPH oxidase. Biochim Biophys Acta. 1850 (4), 794-801 (2015).
- Lee, J. M., Shih, A. Y., Murphy, T. H., Johnson, J. A. NF-E2-related factor-2 mediates neuroprotection against mitochondrial complex I inhibitors and increased concentrations of intracellular calcium in primary cortical neurons. J Biol Chem. 278 (39), 37948-37956 (2003).
- Itoh, K., et al. An Nrf2/small Maf heterodimer mediates the induction of phase II detoxifying enzyme genes through antioxidant response elements. Biochem Biophys Res Commun. 236 (2), 313-322 (1997).
- Li, L., et al. Nrf2/ARE pathway activation, HO-1 and NQO1 induction by polychlorinated biphenyl quinone is associated with reactive oxygen species and PI3K/AKT signaling. Chem Biol Interact. , 56-67 (2014).
- Cannon, J. R., et al. A highly reproducible rotenone model of Parkinson's disease. Neurobiol Dis. 34 (2), 279-290 (2009).
- Sherer, T. B., Kim, J. H., Betarbet, R., Greenamyre, J. T. Subcutaneous rotenone exposure causes highly selective dopaminergic degeneration and alpha-synuclein aggregation. Exp Neurol. 179 (1), 9-16 (2003).
- Testa, C. M., Sherer, T. B., Greenamyre, J. T. Rotenone induces oxidative stress and dopaminergic neuron damage in organotypic substantia nigra cultures. Brain Res Mol Brain Res. 134 (1), 109-118 (2005).
- Zhou, J., et al. High-efficiency induction of neural conversion in human ESCs and human induced pluripotent stem cells with a single chemical inhibitor of transforming growth factor beta superfamily receptors. Stem Cells. 28 (10), 1741-1750 (2010).
- Topol, A., Tran, N. N., Brennand, K. J. A Guide to Generating and Using hiPSC Derived NPCs for the Study of Neurological Diseases. J Vis Exp. (96), e52495 (2015).
- Jiang, Y., Zhang, M. J., Hu, B. Y. Specification of functional neurons and glia from human pluripotent stem cells. Protein Cell. 3 (11), 818-825 (2012).
- Parsons, X. H., et al. Efficient derivation of human neuronal progenitors and neurons from pluripotent human embryonic stem cells with small molecule induction. J Vis Exp. (56), e3273 (2011).
- Shi, Y., Kirwan, P., Livesey, F. J. Directed differentiation of human pluripotent stem cells to cerebral cortex neurons and neural networks. Nat Protoc. 7 (10), 1836-1846 (2012).
- Zeng, H., et al. Specification of region-specific neurons including forebrain glutamatergic neurons from human induced pluripotent stem cells. PLoS One. 5 (7), e11853 (2010).
- Zeng, X., et al. An in vitro model of human dopaminergic neurons derived from embryonic stem cells: MPP+ toxicity and GDNF neuroprotection. Neuropsychopharmacology. 31 (12), 2708-2715 (2006).
- Paşca, A. M., et al. Functional cortical neurons and astrocytes from human pluripotent stem cells in 3D culture. Nat Methods. 12 (7), 671-678 (2015).
- Almeida, S., et al. Modeling key pathological features of frontotemporal dementia with C9ORF72 repeat expansion in iPSC-derived human neurons. Acta Neuropathol. 126 (3), 385-399 (2013).
- Urbán, N., Guillemot, F. Neurogenesis in the embryonic and adult brain: same regulators, different roles. Front Cell Neurosci. 8, 396 (2014).
- Kriegstein, A., Alvarez-Buylla, A. The glial nature of embryonic and adult neural stem cells. Annu Rev Neurosci. 32, 149-184 (2009).
- Krencik, R., Weick, J. P., Liu, Y., Zhang, Z. J., Zhang, S. C. Specification of transplantable astroglial subtypes from human pluripotent stem cells. Nat Biotechnol. 29 (6), 528-534 (2011).
- Krencik, R., Zhang, S. C. Directed differentiation of functional astroglial subtypes from human pluripotent stem cells. Nat Protoc. 6 (11), 1710-1717 (2011).
- Nguyen, T., Nioi, P., Pickett, C. B. The Nrf2-antioxidant response element signaling pathway and its activation by oxidative stress. J Biol Chem. 284 (20), 13291-13295 (2009).
- Bal-Price, A., et al. Putative adverse outcome pathways relevant to neurotoxicity. Crit Rev Toxicol. 45 (1), 83-91 (2015).
- Cabezas, R., El-Bacha, R. S., Gonzalez, J., Barreto, G. E. Mitochondrial functions in astrocytes: neuroprotective implications from oxidative damage by rotenone. Neurosci Res. 74 (2), 80-90 (2012).
- Swarnkar, S., et al. Astrocyte activation: a key step in rotenone induced cytotoxicity and DNA damage. Neurochem Res. 37 (10), 2178-2189 (2012).
- Canovas-Jorda, D., Louisse, J., Pistollato, F., Zagoura, D., Bremer, S. Regenerative toxicology: the role of stem cells in the development of chronic toxicities. Expert Opin Drug Metab Toxicol. 10 (1), 39-55 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados