Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Visualización y seguimiento de ARNm endógenos en cámaras de huevo Drosophila melanogaster en vivo
En este artículo
Resumen
Aquí, presentamos un protocolo para la visualización, detección, análisis y seguimiento del tráfico endógeno de ARNm en cámara de óvulos Drosophila melanogaster en vivo utilizando balizas moleculares, microscopía confocal de disco giratorio y análisis de código abierto Software.
Resumen
Las técnicas de diagnóstico por imágenes basadas en la fluorescencia, en combinación con los desarrollos de la microscopía ligera, han revolucionado la forma en que los biólogos celulares realizan estudios de imágenes de células vivas. Los métodos para detectar ARN se han ampliado considerablemente desde que los estudios seminales vincularon la localización de ARNm específica del sitio con la regulación de la expresión génica. Los procesos dinámicos de ARNm ahora se pueden visualizar a través de enfoques que detectan ARNm, junto con configuraciones de microscopía que son lo suficientemente rápidas como para capturar el rango dinámico de comportamiento molecular. La tecnología de baliza molecular es un enfoque basado en la hibridación capaz de detectar directamente transcripciones endógenas en células vivas. Las balizas moleculares son sondas de ácido nucleico con forma de horquilla, acondicionadas internamente y discriminantes de un solo nucleótido, que fluoran sólo tras la hibridación a una secuencia de diana única. Cuando se combina con microscopía de fluorescencia avanzada e imágenes de alta resolución, permiten realizar un seguimiento espacial y temporal del movimiento intracelular de ARNm. Aunque esta tecnología es el único método capaz de detectar transcripciones endógenas, los biólogos celulares aún no han adoptado completamente esta tecnología debido a las dificultades en el diseño de este tipo de sondas para la toma de imágenes de células vivas. Una nueva aplicación de software, PinMol,permite un diseño mejorado y rápido de las sondas más adecuadas para hibridar eficientemente a las regiones objetivo de ARNm dentro de una célula viva. Además, la adquisición de imágenes en tiempo real y el software de análisis de imágenes actual de código abierto permiten una salida de datos refinada, lo que conduce a una evaluación más fina de la complejidad subyacente a los procesos dinámicos involucrados en el ciclo de vida del ARNm.
Aquí presentamos un protocolo integral para diseñar y entregar balizas moleculares en cámaras de óvulos Drosophila melanogaster. La detección y visualización directa y muy específica de arNM maternos endógenos se realiza mediante microscopía confocal de disco giratorio. Los datos de imagen se procesan y analizan mediante la detección y seguimiento de objetos en el software Icy para obtener detalles sobre el movimiento dinámico de los ARNm, que se transportan y localizan a regiones especializadas dentro del ovocitos.
Introducción
Los estudios de biología celular que visualizan eventos dinámicos con resolución espacial y temporal han sido posibles gracias al desarrollo de técnicas de imagen de células vivas basadas en fluorescencia. En la actualidad, la visualización in vivo de ARNm se logra a través de tecnologías basadas en interacciones de aptámero-proteína de ARN, fluorescencia inducida por aptámero de ARN de tintes orgánicos y recocido de la sonda de ácido nucleico1,2, 3. Todos ellos ofrecen alta especificidad, sensibilidad y relación señal-fondo. Sin embargo, los enfoques centrados en el ARN requieren una manipulación genética extensiva, donde un transgén está diseñado para expresar un ARN con motivos estructurales artificiales que son necesarios para la unión a proteínas o tintes orgánicos. Por ejemplo, el sistema MS2/MCP requiere la coexpresión de un transgén que exprese una construcción de ARN que contenga múltiples repeticiones en tándem de la secuencia de unión para la proteína de recubrimiento del bacteriófago MS2 (MCP), y otro transgén que codifica una proteína fluorescente fusionado con MCP4,5. La adición de estos motivos estructurales secundarios al ARN, junto con una proteína voluminosa con etiqueta fluorescente, ha suscitado preocupaciones de que los procesos de ARN nativos puedan verse afectados6. Una tecnología que aborda esta preocupación y ofrece ventajas únicas adicionales es el enfoque basado en ácido nucleico, balizas moleculares (MB). Los MB permiten la detección múltiplex de ARNm endógenos, la discriminación de variaciones de nucleótidos individuales y la rápida cinética de la hibridación con ARNm7objetivo,8. Los MF son sondas de oligonucleótidos que permanecen en un pliegue de horquilla apenada antes de someterse a un cambio de conformación fluorogénica una vez que hibridan a sus objetivos (Figura1C)9. Varios grupos han tenido éxito en el uso de MBs para detectar ARN no codificantes (microRNAs y lncRNAs)10,11,12,13, retrovirus de ARN14 y proteínas de ADN dinámicas interacciones15. Se han empleado con éxito para la toma de imágenes en diversos organismos y tejidos, tales como embriones de pez cebra16, neuronas13, tejido tumoral17, cardiomiocitos diferenciadores18,y Salmonella 19.
Aquí describimos el enfoque de diseño, entrega y detección de ARNm endógenos en cámaras de huevo D. melanogaster vivas junto con una configuración de microscopía que es lo suficientemente rápida como para capturar el rango dinámico del transporte molecular activo. La cámara de óvulos D. melanogaster ha servido como un sistema modelo multicelular ideal para una amplia gama de estudios de desarrollo, desde la división temprana de células madre germinales y la expresión génica materna hasta la generación de plan corporal segmentado20, 21. Las cámaras de huevo son fácilmente aisladas, grandes y translúcidas, y son capaces de soportar horas de análisis ex vivo, lo que las hace altamente susceptibles a experimentos de imágenes. Mucho trabajo se ha centrado en la localización asimétrica de transcripciones maternas a regiones subcelulares discretas antes de ser traducidas activamente. En particular, la localización del ARNm de oskar y su posterior traducción en el polo posterior del ovocitos deben producirse de manera estrictamente regulada para evitar un fenotipo22de embrión bicaudalletal letal. el ARNm de oskar se transcribe en las 15 células germinales, llamadas células de la barulgante, y se transporta activamente a través de puentes citoplásmicos, llamados canales de anillo, en el ovocitos, la célula germinal que se convierte en el óvulo maduro y, en última instancia, se fertiliza (Figura1A ). La considerable cantidad de información ya disponible sobre el reclutamiento dinámico y el intercambio de factores proteicos hacia y desde oskar mRNP, junto con sus viajes intracelulares de largo alcance, hacen de Oskar un candidato preferido para estudiar los muchos procesos del ciclo de vida del ARNm. Los MB han sido fundamentales para revelar detalles sobre el proceso de localización de ARNm y descifrar la regulación y función de los factores proteicos que controlan el transporte de ARNm durante la oogénesis de Drosophila. En particular, mediante la microinyección de MB en células de enfermería y la realización de experimentos de imágenes de células vivas, el seguimiento de los ARNm endógenos es posible8,23.
La hoja de ruta presentada aquí ofrece los pasos de un proceso completo, desde la realización de un experimento de imágenes de células en vivo utilizando MBs, la adquisición de datos de imágenes, hasta la realización de análisis de datos para realizar un seguimiento del ARNm endógeno en su entorno celular nativo. Los pasos se pueden modificar y optimizar aún más para satisfacer las necesidades de los investigadores que trabajan con otros tejidos/tipos de células dentro de su propio entorno de laboratorio.
Protocolo
1. Diseño de MBs para imágenes de células en vivo
-
Doble la secuencia de ARN de destino para predecir la estructura secundaria del objetivo mRNA utilizando la "forma de ARN" del servidor mfold (http://unafold.rna.albany.edu/?q=mfold/RNA-Folding-Form).
- Pegue/cargue la secuencia de destino en formato FASTA, seleccione una suboptisidad del 5 o 10% (estructuras con una energía libre de plegado dentro del 5 o 10% del valor MFE, respectivamente) y ajuste el número máximo de plegados calculados en consecuencia (por ejemplo, mayor para el 10% sub-óptimo).
Nota: La inclusión de estructuras secundarias subóptimas al diseñar MBs permite la identificación de regiones dentro del ARNm objetivo que pueden ser más flexibles o más rígidas de lo previsto para la estructura de energía libre mínima (MFE) por sí sola, lo que mejora la diseño general de los MB adecuados para imágenes de células vivas. - Seleccione un "trabajo inmediato" para los objetivos de ARNm de 800 nucleótidos (nt), o un "trabajo por lotes" para longitudes de ARNm entre 801 y 8.000 nt. Guarde el archivo "ss-count" como archivo de texto simple.
- Pegue/cargue la secuencia de destino en formato FASTA, seleccione una suboptisidad del 5 o 10% (estructuras con una energía libre de plegado dentro del 5 o 10% del valor MFE, respectivamente) y ajuste el número máximo de plegados calculados en consecuencia (por ejemplo, mayor para el 10% sub-óptimo).
-
Utilice el archivo "ss-count" obtenido en el paso 1.1 como entrada para el programa PinMol (https://bratulab.wordpress.com/software/) con los parámetros deseados, para diseñar varios MB para el destino mRNA (consulte tutoriales que describen el uso del programa PinMol 24 a https://bratulab.wordpress.com/tutorial-pinmol-mac/).
- Determinar la especificidad de los MM seleccionados mediante el análisis BLAST: utilizar "blastn" con la base de datos adecuada (por ejemplo, para mBs oskar mRNA utilizan la base de datos "refseq-rna" y el organismo Drosophila melanogaster).
- Identificar cualquier expresión específica del tejido del objetivo del ARNm (por ejemplo, para oskar mRNA Flybase> High-Throughput Expression Data> FlyAtlas Anatomy Microarray o modENCODE Anatomy RNA-Seq; http://flybase.org/reports/FBgn0003015) y compártelo con cualquier blast positivo golpea. Elimine las sondas que muestran >50% de homología cruzada con otros ARNm que también se expresan en el tejido/célula de interés.
- Seleccione el par de fluoróforo y quencher adecuado para la configuración de microscopía disponible para realizar imágenes de células vivas (por ejemplo, Cy5/BHQ2)25.
2. Síntesis, purificación y caracterización mb
- Utilizar la síntesis y purificación interna como se describió anteriormente7, o servicios de proveedores comerciales, para sintetizar y purificar de uno a cinco MB (ver nota anterior), utilizando el siguiente esquema de etiquetado: [5'(Fluorophore)-(C3 o C6 linker)-(2'-O -secuencia de metílico MB)-(Quencher)3']. Purificar los MB utilizando HPLC de fase inversa, en casa o utilizando los servicios del proveedor comercial.
NOTA: Las fosfosamiditas utilizadas para la síntesis automatizada de la sonda deben tener la modificación de 2'-O-metil ribonucleótido. También se pueden utilizar quimeras de ácido nucleico bloqueado alternado (LNA) y 2'-O-metil modificaciones para aumentar la estabilidad de un híbrido entre un MB más corto y su ARNmobjetivo 26. - Sintetizar oligonucleótidos de ADN que coincidan con la secuencia de la región de ARN objetivo y, por lo tanto, son complementarios a la región de sonda de los MB, para su uso en caracterización in vitro (véanse los pasos 2.3 a 2.5; nota anterior). Maximizar la hibridación del MB con la imitación del objetivo de ADN-oligonucleótido, incluyendo en cada extremo del objetivo de ADN cuatro nucleótidos adicionales, como se encuentra en la secuencia de ARNm objetivo.
Nota: Una caracterización más rigurosa de la eficiencia del MB para detectar la secuencia dirigida se puede realizar utilizando objetivos de ARN sintetizados in vitro en lugar de oligonucleótidos de ADN complementarios8. - Realizar la desnaturalización térmica del MB solo, medir su temperatura de fusión (Tm), y confirmar que el MB asume la forma de horquilla deseada a temperatura fisiológica. Hemos observado valores Tm entre 60 y 90oC.
- Realizar la desnaturalización térmica del MB en presencia del objetivo de oligonucleótido de ADN y medir el TM del híbrido objetivo MB:DNA, como se describió anteriormente7. Se desea un Tm entre 55 y 60 oC para el híbrido MB:DNA.
- Realizar reacciones de hibridación in vitro con el objetivo de oligonucleótido de ADN correspondiente, y determinar la eficiencia de la formación híbrida MB:DNA a temperatura fisiológica, como se describió anteriormente7. Se desea una cinética de hibridación rápida con la imitación del objetivo de ADN, sin embargo, los MB que no muestran una alta eficiencia de hibridación con objetivos de ADN pueden tener un mejor rendimiento con el ARNm objetivo in vitro y/o in vivo.
3. Disección y preparación de cámaras individuales de huevo para microinyección
- Alimente las hembras recién nacidas y acopladas durante 2-3 días con pasta de levadura fresca.
- Anestetizar moscas en una almohadilla de CO2 y, usando pinzas finas (Dumont #5), transferir 1-2 hembras en una gota de aceite de Halocarbon 700 en un resbalón de la cubierta de vidrio.
- Usando un par de pinzas, oriente la mosca con el lado dorsal hacia arriba bajo un estereomicroscopio. Diseccionar el abdomen femenino haciendo una pequeña incisión en el extremo posterior y apretar suavemente el par de ovarios en el aceite.
- Explantar los ovarios en una gota de aceite en una nueva capa. Sostenga suavemente un ovario con una pinza mientras pellizca las etapas más jóvenes del ovariole con la otra pinza. el ARNm de oskar se localiza activamente a mediados de la oogénesis (etapas > 7), y las cámaras de óvulos más jóvenes (etapas < 7) son más difíciles de inyectar y no sobreviven tanto tiempo. Arrastre lentamente el deslizamiento de la cubierta (con un movimiento hacia abajo) hasta que los óvulos individuales o las cámaras de huevo estén aislados y alineados verticalmente. Separe aún más las cámaras de huevo individuales desplazando las etapas no deseadas de la cadena de óvulos de ovariole.
NOTA: Asegúrese de que las cámaras de huevo con objeto de burla individual no flotan en el aceite y que se adhieren al resbalón de la cubierta. Esto es importante tanto para la microinyección exitosa como para la adquisición de imágenes.
4. Microinyección de MBs en las Células de Enfermeras de las Cámaras de Huevo
- Preparar la solución MB, utilizando una baliza molecular (por ejemplo, osk2216Cy5), o una mezcla de dos MB que se dirigen a diferentes ARNm y que están etiquetados con fluoróforos espectralmente distintos (por ejemplo, osk2216Cy5 y drongo1111Cy3). Utilice una concentración de 200-300 ng/L cada MB en HybBuffer (50 mM Tris-HCl - pH 7,5, 1,5 mM MgCl2 y 100 mM NaCl). Para un cóctel de cuatro MM etiquetados con el mismo fluoróforo que apuntan al mismo ARNm a 200 ng/L cada uno en HybBuffer (por ejemplo, osk82, osk1236, osk2216). Gire la solución mb inmediatamente antes de cargar la aguja para microinyección.
- Seleccione el objetivo. Se recomienda un objetivo de aceite de 40x para encontrar una cámara de huevo adecuada y para realizar microinyección.
- Monte el cubreobjetos con la cámara de huevo diseccionada en la etapa del microscopio. Traiga el objetivo en la posición de enfoque e identifique una cámara de óvulos en una etapa de desarrollo de media a última, que esté adecuadamente orientada para la microinyección (es decir, con el eje perpendicular A-P a la punta de la aguja para permitir una fácil inyección dentro de una enfermera proximal al ovocitos).
- Cargue una aguja (comercial o preparada en casa27 ) con una solución de 1 ML MB (ver paso 4.1) y conéctela al microinyector. Para microinyecciones en cámaras de huevo D. melanogaster, oriente la aguja (ver Tabla de Materiales)en un ángulo <45o a la etapa del microscopio (por ejemplo, 30o) para evitar la punción de varias células de la enfermera.
- Configurar el inyector con presión de inyección de 500-1.000 hPa y presión de compensación de 100-250 hPa (ver Tabla de Materiales).
- Mueva lentamente el escenario para traer en el campo de visión un área de la gota de aceite vacío de las cámaras de huevo.
- Usando el joystick del micromanipulador, baje suavemente la aguja en la gota de aceite y ponga su punta enfocada hacia la periferia del campo de visión.
- Realice una función de "limpieza" para extraer el aire de la punta de la aguja y asegurarse de que hay flujo de la aguja.
- Lleve la aguja a la posición de inicio y concéntrese en la cámara de óvulos que se va a microinyectar, luego vuelva a enfocar la aguja y colóquela cerca del borde de la cámara de óvulos.
- Realizar un ajuste fino de la posición Z del objetivo de tal manera que la membrana que separa las células folículos de las células de la enfermera está enfocada.
- Inserte la aguja en una celda de enfermería y realice la inyección durante 2-5 s.
- Retire suavemente la aguja y retráiquela a la posición de inicio.
- Cambie el objetivo al aumento deseado para la adquisición de imágenes (60-63x o 100x), concéntrese en la cámara de óvulos y comience la adquisición.
5. Adquisición de datos mediante una configuración de microscopio confocal de disco giratorio
NOTA: Consulte la Tabla de materiales para ver nuestra configuración específica.
- Configure el protocolo de adquisición para grabar una pila XYZCt de imágenes de 8-16 bits (XYZ, volumen, c, canal, t de tiempo).
- Seleccione líneas láser para los canales deseados (por ejemplo, láser de 641 nm para Cy5 y 491 nm para GFP) y adquiera los canales secuencialmente: primero la señal de fluorescencia en cada canal y luego cambie la posición Z, para permitir un análisis de colocalización adecuado.
- Seleccione el paso Z (p. ej., 0,3 m) y los límites Z superior e inferior(p. ej. -2 a 2 m).
- Introduzca el tiempo de adquisición y la frecuencia de muestreo (por ejemplo, cada 15-30 s durante un máximo de 1 h).
- Iniciar la adquisición.
6. Procesamiento, Análisis de Datos para Obtener Información de Seguimiento y Colocalización, y Preparación de Archivos de Video
- Procesamiento de imágenes
- Descarga, desempaquete y abra Icy, una plataforma de comunidad abierta para la informática de bioimágenes (http://icy.bioimageanalysis.org/)
- Abra la pila XYZCt adquirida en el paso 5: Imagen/Secuencia >Archivo>Abrir.
- Convertir pila a ImageJ: ImageJ> Herramientas> Convertir a IJ, tienen el modo separado activado.
- Hacer un substack (una selección de un rango de pasos Z y puntos de tiempo a analizar más a fondo): ImageJ>Imagen>Pilas>Herramientas>Crear Substack...; seleccionar los canales deseados, los pasos Z y los puntos de tiempo.
- Guardar substack como archivo TIFF: ImageJ>Archivo>Guardar como>Tiff...; utilice este archivo para los pasos posteriores.
- Canales divididos: ImageJ>Imagen>Color>Dividir canales.
- Restar fondo mediante una pila de fondo: ImageJ>Process>Image Calculator..., o usando la opción Rolling ball: ImageJ>Process>Subtract Background..., seleccione el radio de bola rodante. Previsualice la imagen para el radio seleccionado antes de seleccionar "Aceptar".
Nota: La señal de fondo surgirá principalmente de un enfriamiento incorrecto del fluroroforre. La relación señal:fondo (S:B) se utiliza a menudo como indicador del "brillo" de un MB, y se mide a partir de experimentos de hibridación in vitro del MB y oligonucleótido objetivo de ADN. Por ejemplo, los MB osk1236 y osk2216 tienen un S:B de 81 y 120 euros, respectivamente. - Ajuste el brillo y el contraste de cada canal: ImageJ>Imagen>Ajustar>Brillo/Contraste, seleccione Aplicar.
- Guarde cada canal como un archivo TIFF independiente: ImageJ>Archivo>Guardar como>Tiff....
- Combinar los dos canales: ImageJ>Imagen>Color>Combinar canales...; seleccionar los canales. Guarde la nueva pila como un nuevo archivo TIFF (consulte el paso 6.1.8).
- Detección y seguimiento de puntos
- Volver a Icy: ImageJ>Herramientas>Convertir a hielo.
- Una barra de escala se superpone automáticamente en la pila al convertira a Icy, si el plugin de la barra de escala está instalado [Buscar usando Plugins>Setup>Online plugin]. Si es necesario, edite la barra de escala a través de la ventana Inspector (lado derecho de la pantalla)>ficha Capa>Nombre>Barra de escala.
- Anule la selección/inactive el icono 'ojo' de la barra De escala de la pestaña Capa>Nombre para eliminar la barra de escala de la pila original. Se puede reactivar en la pila final.
- Guarde la pila recién procesada tomando una captura de pantalla usando el icono "cámara" de la barra de menús de la ventana de imagen, "Tomar una captura de pantalla de la vista actual" y Archivo>Guardar como>Tiff....
- Determine la sensibilidad al punto, si los parámetros de sensibilidad al punto ya se han determinado, pase al paso 6.2.7.
- Detectar puntos: seleccione la ventana con la imagen o pila que se va a analizar, Detección&Seguimiento>Detección>Detector de puntos y rellene los parámetros de Configuración:
- En Input (Entrada), seleccione "currentSequenceInputDetection" (predeterminado).
- En Preprocessing, seleccione "Channel 0" (predeterminado) o el canal deseado haciendo referencia cruzada al número en la ventana Inspector>Sequence.
- En Detector, seleccione "Detectar punto brillante sobre fondo oscuro", utilice "Forzar el uso de Wavelets 2D para 3D" solo si no hay suficientes cortes Z en las pilas para realizar el análisis. Seleccione "Escala(s)" y "Sensibilidad" para cada escala (agregue más escalas para puntos más grandes). La Escala y Sensibilidad (cuanto mayor sea el número más sensible es la detección, un máximo de 140 es sugerido por Icy) son variables de prueba y error, que deben ser verificadas visualmente después y decididas.
- Para Región de interés, utilice "ROIfromSequence" (predeterminado).
- Para El filtrado, utilice "NoFiltering" (predeterminado) o seleccione "SizeFiltering" para definir el "Rango de objetos aceptados (en píxeles)".
- Salida: seleccione la configuración de salida XLS o XML (seleccione el formato XML cuando utilice 2007 MS Excel o anterior y hay >65.000 puntos). Si los resultados del detector de puntos se utilizan para el análisis de seguimiento, seleccione también "Exportar a Piscina".
- Repita la detección de manchas utilizando varios valores de escala/sensibilidad hasta que se detecten todas o la mayoría de las manchas. Registre todos los parámetros finales.
- Para el análisis de colocalización, repita la detección de puntos para el otro canal.
- Para realizar un seguimiento de los puntos, seleccione el menú desplegable Detección&Seguimiento>Seguimiento>Seguimiento de puntos>Ejecutar el detector de puntos con parámetros del paso 6.2.6., o utilice el menú desplegable "Seleccionar resultados de detección aquí" para seleccionar un conjunto de datos existente (para ello, mantenga la ventana Detector de manchas abierto desde el paso 6.2.5). Pulse el botón "Parámetros de estimación" y seleccione el movimiento de destino deseado en la ventana emergente Estimación de parámetros (por ejemplo, "es difusivo y dirigido"). Pulse el botón "Ejecutar seguimiento".
- Repita la detección de puntos y el seguimiento de otros canales al realizar el seguimiento de puntos de pilas multicanal, siguiendo los pasos 6.2.6 y 6.2.7, comenzando con la pila generada a partir del paso 6.2.7.
- Para visualizar pistas, seleccione Detección&Seguimiento>Seguimiento>Administrador de pistas: esta ventana se abre automáticamente al finalizar una ejecución de seguimiento. Para "Color Track Processor", seleccione "Habilitar" y elija la representación de color deseada para las pistas. Se puede acceder a los procesadores de pista relevantes a través del "Añadir procesador de pistas..." Menú desplegable (por ejemplo, seleccione "Track Processor Time Clip", active la ventana "Track Clipper" y elija el número deseado de detecciones que se mostrarán antes y después del punto de tiempo actual.)
- Guardar información de pistas como un archivo de pista XML: Detección&Seguimiento>Seguimiento>Administrador de pistas>Archivo>Guardar como....
- Guarde los resultados tomando una captura de pantalla usando el icono "cámara" de la barra de menús de la ventana de imagen, "Tomar una captura de pantalla de la vista actual". Las capturas de pantalla se pueden tomar con los puntos detectados y / o las pistas simplemente activando / desactivando los iconos de ojo correspondientes que se encuentran en la ventana inspector>pestaña Capa>Nombre> Envoltorio de superposición.
- Instale el plugin TimeStamp Overlay: Plugins>Setup>Online plugin>TimeStamp Overlay>Install.
- Añadir marca de tiempo: Plugins>TimeStamp Overlay (Nuevo). Siga las instrucciones en la ventana emergente (esquina inferior derecha de la pantalla) para obtener instrucciones sobre cómo colocar y formatear la marca de tiempo. El intervalo de tiempo se puede agregar/cambiar en la ventana Inspector>ficha Secuencia>Propiedades de secuencia>Editar.
- Guarde los resultados tomando otra captura de pantalla. Guardar imagen como 1) Formato Tiff y 2) como formato AVI; para el formato AVI primero convertir a renderizado RGB (Imagen/Secuencia>Renderización>imagen RGB).
- Girar la imagen a la orientación deseada: Ventana Inspector>Ficha Secuencia>Canvas>Rotación.
- Guarde la imagen rotada por "Tomar una captura de pantalla de la vista actual". Asegúrese de que el icono "ojo" de la barra de escala no esté seleccionado, ya que también rotará con la imagen.
- Elija y recorte el ROI: seleccione Región de interés>2D ROI>Elija la forma de ROI y, a continuación, cree/dibuje ROI en la imagen; Imagen/Secuencia>Plano (XY)>Recorte rápido.
- Análisis de colocación
- Preparar un protocolo de colocación; se proporcionan varios ejemplos en el sitio web de Icy (http://icy.bioimageanalysis.org/protocol/List) (ver Materiales Suplementarios).
- Cargar protocolo de colocación: Herramientas>Scripting>Protocolos>Cargar y ajustar parámetros en los bloques que interactúan (por ejemplo, en el bloque "Wavelet Spot Detecting" utilizan parámetros determinados en el paso 6.2.6.).
- Mida el tamaño de una partícula en píxeles, determine la distancia de colocación e infórmela en el bloque "Colocalizador" como "Distancia máxima".
NOTA: El tamaño de la partícula en píxeles depende del sistema de detección. Para medir el tamaño, haga zoom en una sola partícula y cuente manualmente los píxeles que abarcan el ancho de la señal. Promedio de las mediciones de al menos tres partículas. La distancia máxima que se establecerá para la colocación es el tamaño de la partícula en píxeles (esto representa la suma máxima del radio de dos partículas que se tocan). - Si lo desea, seleccione uno o más ROI para el análisis de colocalización: Región de interés>2D ROI>Elija la forma del ROI>Dibujar ROI en la imagen.
- Recortar ROI:Imagen/Secuencia>Plano (XY)>Recorte rápido.
- Realizar la colocación: Ventana del editor de protocolos>Ficha Protocolo elegido>Ejecutar. El bloque final en la ventana del editor de protocolos contendrá el porcentaje general de colocación basado en la detección de puntos, mientras que la información en cada punto de tiempo se puede encontrar en la ventana Inspector> ficha Salida.
- Realice un seguimiento de las partículas colocalizadas y únicas siguiendo el paso 6.2.7 (Puntos de seguimiento).
- Guarde como se describe en el paso 6.2.16.
Resultados
Usando PinMol, se pueden diseñar varios MM para un objetivo de ARNm (Figura1B-C). Después de la síntesis y purificación, los MB seleccionados se caracterizan y se comparan utilizando el análisis in vitro.
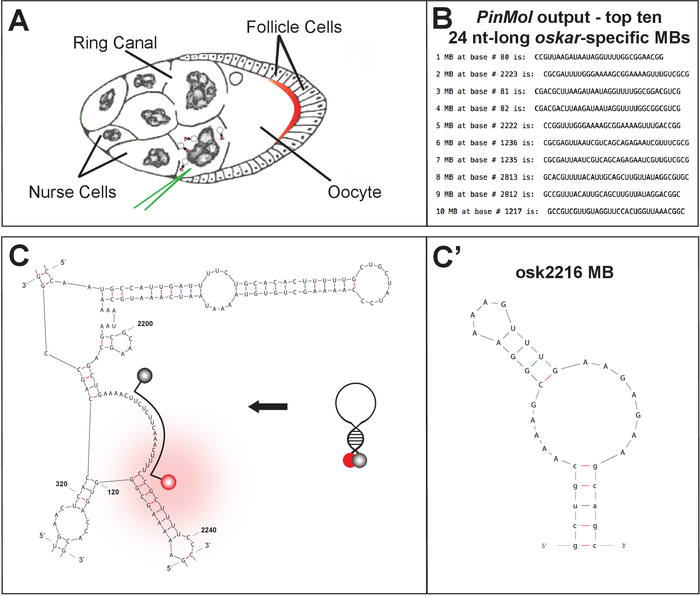
Figura 1: Técnica y descripción de tejidos para imágenes de células vi...
Discusión
La visualización en vivo del tráfico endógeno de ARNm en cámaras de huevo Drosophila se basa en el uso de mBs específicos, eficientes y resistentes a la nucleasa, que ahora se pueden diseñar fácilmente con el software PinMol. Los MB son sondas específicas diseñadas para detectar secuencias únicas dentro de un ARNm de destino (preferiblemente regiones libres de estructura secundaria), lo que hace posible la detección altamente resuelta de una transcripción. La única limitación al adoptar es...
Divulgaciones
Los autores no tienen conflicto de intereses que revelar.
Agradecimientos
Agradecemos a Salvatore A.E. Marras (Centro del Instituto de Investigación de Salud Pública de la Universidad Rutgers) por la síntesis, etiquetado y purificación de balizas moleculares, y Daniel St Johnston (Instituto Gurdon, Universidad de Cambridge) por el oskar-MS2/ Stock de mosca transgénica MCP-GFP. Este trabajo fue apoyado por un Premio CAREER 1149738 de la Fundación Nacional de Ciencias y un Premio Del Congreso del Personal Profesional-CUNY a DPB.
Materiales
| Name | Company | Catalog Number | Comments |
| Spectrofluorometer | Fluoromax-4 Horiba-Jobin Yvon | n/a | Photon counting spectrofluorometer |
| Quartz cuvette | Fireflysci (former Precision Cells Inc.) | 701MFL | |
| Dumont #5 tweezer | World Precision Instruments | 501985 | Thin tweezers are very important to separate out the individual egg chambers |
| Halocarbon oil 700 | Sigma-Aldrich | H8898 | |
| Cover slip No.1 22 x 40mm | VWR | 48393-048 | |
| Dissecting microscope | Leica MZ6 Leica Microsystems Inc. | n/a | |
| CO2 fruit fly anesthesia pad | Genesee Scienific | 59-114 | |
| Tris-HCL pH7.5 | Sigma-Aldrich | 1185-53-1 | |
| Magnesium chloride | Sigma-Aldrich | 7791-18-6 | |
| NaCl | Sigma-Aldrich | 7647-14-5 | |
| Spinning disc confocal microscope | Leica DMI-4000B inverted microscope equipped with Yokogawa CSU 10 spinning disc Leica Microsystems Inc. | n/a | |
| Hamamatsu C9100-13 ImagEM EMCCD camera | Hamamatsu | n/a | |
| PatchMan NP 2 Micromanipulator | Eppendorf Inc. | 920000037 | |
| FemtoJet Microinjector | Eppendorf Inc. | 920010504 | |
| Injection needle: Femtotips II | Eppendorf Inc. | 930000043 | |
| Loading tip: 20ul Microloader | Eppendorf Inc. | 930001007 | |
| Micro Cover glasses no. 1 or 1.5, 22x40mm | VWR | 48393-026; 48393-172 | |
| Dry yeast | Any grocery store | n/a | |
| Computer, > 20 GB RAM | Although processing can be carried out on most computers, higher capabilities will increase the speed of the processing |
Referencias
- Tyagi, S. Imaging intracellular RNA distribution and dynamics in living cells. Nature Methods. 6 (5), 331-338 (2009).
- Bao, G., Rhee, W. J., Tsourkas, A. Fluorescent probes for live-cell RNA detection. Annual Reviews of Biomedical Engineering. 11, 25-47 (2009).
- Mannack, L. V., Eising, S., Rentmeister, A. Current techniques for visualizing RNA in cells. F1000Research. 5, (2016).
- Larson, D. R., Zenklusen, D., Wu, B., Chao, J. A., Singer, R. H. Real-time observation of transcription initiation and elongation on an endogenous yeast gene. Science. 332 (6028), 475-478 (2011).
- Bertrand, E., et al. Localization of ASH1 mRNA particles in living yeast. Molecular Cell. 2 (4), 437-445 (1998).
- Garcia, J. F., Parker, R. MS2 coat proteins bound to yeast mRNAs block 5' to 3' degradation and trap mRNA decay products: implications for the localization of mRNAs by MS2-MCP system. RNA. 21 (8), 1393-1395 (2015).
- Bratu, D. P. Molecular beacons: Fluorescent probes for detection of endogenous mRNAs in living cells. Methods in Molecular Biology. 319, 1-14 (2006).
- Bratu, D. P., Cha, B. J., Mhlanga, M. M., Kramer, F. R., Tyagi, S. Visualizing the distribution and transport of mRNAs in living cells. Proceedings of the National Academy of Sciences of the Unites States of America. 100 (23), 13308-13313 (2003).
- Tyagi, S., Kramer, F. R. Molecular beacons: probes that fluoresce upon hybridization. Nature Biotechnology. 14 (3), 303-308 (1996).
- Chen, M., et al. A molecular beacon-based approach for live-cell imaging of RNA transcripts with minimal target engineering at the single-molecule level. Scientific Reports. 7 (1), 1550 (2017).
- Liu, Y., et al. Multiplex detection of microRNAs by combining molecular beacon probes with T7 exonuclease-assisted cyclic amplification reaction. Analytical and Bioanalytical Chemistry. 409 (1), 107-114 (2017).
- Baker, M. B., Bao, G., Searles, C. D. In vitro quantification of specific microRNA using molecular beacons. Nucleic Acids Research. 40 (2), e13 (2012).
- Ko, H. Y., et al. A color-tunable molecular beacon to sense miRNA-9 expression during neurogenesis. Scientific Reports. 4, 4626 (2014).
- Vet, J. A., et al. Multiplex detection of four pathogenic retroviruses using molecular beacons. Proceedings of the National Academy of Sciences of the Unites States of America. 96 (11), 6394-6399 (1999).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Methods in Molecular Biology. 429, 209-224 (2008).
- Li, W. M., Chan, C. M., Miller, A. L., Lee, C. H. Dual Functional Roles of Molecular Beacon as a MicroRNA Detector and Inhibitor. Journal of Biological Chemistry. 292 (9), 3568-3580 (2017).
- Kuang, T., Chang, L., Peng, X., Hu, X., Gallego-Perez, D. Molecular Beacon Nano-Sensors for Probing Living Cancer Cells. Trends in Biotechnology. 35 (4), 347-359 (2017).
- Ban, K., et al. Non-genetic Purification of Ventricular Cardiomyocytes from Differentiating Embryonic Stem Cells through Molecular Beacons Targeting IRX-4. Stem Cell Reports. 5 (6), 1239-1249 (2015).
- Hadjinicolaou, A. V., Demetriou, V. L., Emmanuel, M. A., Kakoyiannis, C. K., Kostrikis, L. G. Molecular beacon-based real-time PCR detection of primary isolates of Salmonella Typhimurium and Salmonella Enteritidis in environmental and clinical samples. BMC Microbiology. 9, 97 (2009).
- McLaughlin, J. M., Bratu, D. P. Drosophila melanogaster Oogenesis: An Overview. Methods in Molecular Biology. 1328, 1-20 (2015).
- Bastock, R., St Johnston, D. Drosophila oogenesis. Current Biology. 18 (23), R1082-R1087 (2008).
- Rongo, C., Gavis, E. R., Lehmann, R. Localization of oskar RNA regulates oskar translation and requires Oskar protein. Development. 121 (9), 2737-2746 (1995).
- Mhlanga, M. M., et al. In vivo colocalisation of oskar mRNA and trans-acting proteins revealed by quantitative imaging of the Drosophila oocyte. PLoS One. 4 (7), e6241 (2009).
- Bayer, L. V., Omar, O. S., Bratu, D. P., Catrina, I. E. PinMol: Python application for designing molecular beacons for live cell imaging of endogenous mRNAs. bioRxiv. , (2018).
- Marras, S. A., Kramer, F. R., Tyagi, S. Efficiencies of fluorescence resonance energy transfer and contact-mediated quenching in oligonucleotide probes. Nucleic Acids Research. 30 (21), e122 (2002).
- Bratu, D. P., Catrina, I. E., Marras, S. A. Tiny molecular beacons for in vivo mRNA detection. Methods in Molecular Biology. 714, 141-157 (2011).
- Dean, D. A. Preparation (pulling) of needles for gene delivery by microinjection. Cold Spring Harbor. 2006 (7), (2006).
- Alami, N. H., et al. Axonal transport of TDP-43 mRNA granules is impaired by ALS-causing mutations. Neuron. 81 (3), 536-543 (2014).
- Jackson, S. R., et al. Applications of Hairpin DNA-Functionalized Gold Nanoparticles for Imaging mRNA in Living Cells. Methods in Enzymology. 572, 87-103 (2016).
- Zimyanin, V. L., et al. In vivo imaging of oskar mRNA transport reveals the mechanism of posterior localization. Cell. 134 (5), 843-853 (2008).
- Catrina, I. E., Marras, S. A., Bratu, D. P. Tiny molecular beacons: LNA/2'-O-methyl RNA chimeric probes for imaging dynamic mRNA processes in living cells. ACS Chemical Biology. 7 (9), 1586-1595 (2012).
- Chen, A. K., Behlke, M. A., Tsourkas, A. Efficient cytosolic delivery of molecular beacon conjugates and flow cytometric analysis of target RNA. Nucleic Acids Research. 36 (12), e69 (2008).
- Nitin, N., Santangelo, P. J., Kim, G., Nie, S., Bao, G. Peptide-linked molecular beacons for efficient delivery and rapid mRNA detection in living cells. Nucleic Acids Research. 32 (6), e58 (2004).
- Chen, A. K., Behlke, M. A., Tsourkas, A. Avoiding false-positive signals with nuclease-vulnerable molecular beacons in single living cells. Nucleic Acids Research. 35 (16), e105 (2007).
- Bevilacqua, P. C., Ritchey, L. E., Su, Z., Assmann, S. M. Genome-Wide Analysis of RNA Secondary Structure. Annual Review of Genetics. 50, 235-266 (2016).
- Mhlanga, M. M., Vargas, D. Y., Fung, C. W., Kramer, F. R., Tyagi, S. tRNA-linked molecular beacons for imaging mRNAs in the cytoplasm of living cells. Nucleic Acids Research. 33 (6), 1902-1912 (2005).
- Eliceiri, K. W., et al. Biological imaging software tools. Nature Methods. 9 (7), 697-710 (2012).
- Bolte, S., Cordelieres, F. P. A guided tour into subcellular colocalization analysis in light microscopy. Journal of Microscopy. 224 (Pt 3), 213-232 (2006).
- Trcek, T., et al. Drosophila germ granules are structured and contain homotypic mRNA clusters. Nature Commununications. 6, 7962 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados