Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Canlı Drosophila melanogaster yumurta Chambers Içinde endojen mrnas görselleştirme ve izleme
Bu Makalede
Özet
Burada, moleküler işaretleri kullanarak canlı Drosophila melanogaster yumurta odasında endojen mRNA kaçakçılığı görselleştirme, algılama, analiz ve izleme için bir protokol sunuyoruz, iplik disk konfokka mikroskopi, ve açık kaynak Analizi Yazılım.
Özet
Floresan bazlı görüntüleme teknikleri, ışık Mikroskopisinde gelişmelerle birlikte, hücre biyologlarının canlı hücre görüntüleme çalışmaları konusunda devrim yarattı. RNAs algılama yöntemleri, seminal çalışmalarda siteye özel mRNA lokalizasyonu ile gen ifadesi yönetmeliğine bağlı olarak büyük ölçüde genişledi. Dinamik mRNA süreçleri artık mRNAs 'ı algılayan yaklaşımlar aracılığıyla, dinamik moleküler davranış aralığını yakalamak için yeterince hızlı olan mikroskopi ayarlarıyla birlikte görselleştirilebilir. Moleküler işaret teknolojisi, yaşayan hücrelerde endojen transkriptleri doğrudan tespit edebilen bir hybridizasyon tabanlı bir yaklaşımdır. Moleküler işaretler, yalnızca benzersiz bir hedef dizisine hibridizasyon üzerine floresan olan, saç iğne şeklinde, dahili olarak bastırılmış, tek nüklotid ayrımcılık nüklenik asit probları vardır. Gelişmiş Floresan mikroskopisi ve yüksek çözünürlüklü görüntüleme ile birleştiğinde, mRNAs 'ın hücre içi hareketinin uzamsal ve temporal takibini gerçekleştirmesini sağlar. Bu teknoloji, endojen transkriptleri tespit edebilen tek yöntem olmasına rağmen, hücre biyologlar canlı hücre görüntüleme için böyle problar tasarlama zorlukları nedeniyle henüz tam olarak bu teknolojiyi kucakladı değil. Yeni bir yazılım uygulaması olan pinmol, canlı hücre içindeki mRNA hedef bölgelerine verimli bir şekilde melezleşme etmek için en uygun problar gelişmiş ve hızlı tasarım sağlar. Buna ek olarak, yüksek çözünürlüklü, gerçek zamanlı görüntü edinme ve akım, açık kaynak görüntü analiz yazılımı, mRNA 'nın yaşam döngüsünde yer alan dinamik süreçlerin temelinin karmaşıklığının daha ince bir değerlendirilmesine yol açan, rafine edilmiş bir veri çıktısı için izin verir.
Burada tasarım ve Drosophila melanogaster yumurta odalarına moleküler işaretler teslim için kapsamlı bir protokol sunuyoruz. Endojen maternal mRNAs 'ın doğrudan ve son derece spesifik tespiti ve görselleştirilmesi, iplik diski konforkop mikroskobu ile gerçekleştirilir. Görüntüleme verileri, oocayt içinde özel bölgelere taşınan ve lokalize olan mRNAs 'ın dinamik hareketi hakkında ayrıntılı bilgi almak için ICG yazılımında nesne algılama ve izleme kullanılarak işlenir ve analiz edilir.
Giriş
Uzamsal ve temporal çözünürlüğe sahip dinamik olayları görselleştiren hücre biyolojisi çalışmaları, floresan bazlı canlı hücre görüntüleme tekniklerinin geliştirilmesi ile mümkün hale gelmiştir. Şu anda, In vivo mRNA görselleştirme RNA aptamer-protein etkileşimleri dayalı teknolojiler aracılığıyla elde edilir, organık boyalar RNA aptamer kaynaklı floresans ve nüklik asit prob tavlama1,2, 3. hepsi teklif yüksek özgüllük, hassasiyet ve sinyal-to-background oranı. Ancak RNA aptamer merkezli yaklaşımlar, bir transgeni protein veya organik boya bağlama için gerekli olan yapay yapısal motiflerle RNA 'ya ifade etmek üzere tasarlanan kapsamlı genetik manipülasyon gerektirir. Örneğin, MS2/MCP sistemi, bakteriyofaj MS2 kat proteini (MCP) için bağlayıcı dizinin birden fazla tandem tekrarı içeren bir RNA yapısını ifade eden bir transgeni ve bir floresan proteini kodlayan başka bir transgeni ifade etmesini gerektirir. MCP4,5' e kullanılmış. Bu tür ikincil yapısal motiflerin RNA 'ya ilavesi, hantal floresan etiketli bir proteinle birlikte, yerli RNA süreçlerinin6' nın etkilenmesi konusunda endişeler ortaya çıkmış olabilir. Bu endişeyi ele alan ve ek benzersiz avantajlar sunan bir teknoloji, nüklik asit bazlı yaklaşımdır, moleküler işaretler (MBs). MBS, endojen mrb 'lerin Multiplex tespitine, tek nüklemotid varyasyonlarının ayrımcılığını ve hedef mRNA7,8ile hibridizasyon hızlı kinetiği için izin verir. MBS onlar hedeflerine melezleşme bir kez bir flororojenik Konformasyonel değişikliği geçiren önce bir sönük saç iğne kat kalır oligonükleotit problar (Şekil 1C)9. Birkaç grup, hem kodlama olmayan Rnas 'ları (MicroRNAs ve lncrnas)10,11,12,13, RNA retrovirüsler14 ve dinamik DNA-proteini algılamak için MBS kullanmanın başarısı vardı etkileşimleri15. Onlar başarıyla çeşitli organizmalar ve dokularda, zebra balığı embriyo16, nöronlar13, tümör dokusu17, ayrım kardiyomiyosit18ve Salmonella gibi görüntüleme için istihdam edilmiştir 19 yaşında.
Burada yaşam D. melanogaster yumurta odalarında endojen mrnas için tasarım, teslimat ve algılama yaklaşımı, aktif moleküler taşıma dinamik aralığını yakalamak için yeterince hızlı bir mikroskopi set-up ile birleştiğinde açıklanmaktadır. D. melanogaster yumurta odası, erken germline kök hücre bölünmesi ve maternal gen ifadesinden bölümsel vücut planı20' ye kadar çok çeşitli gelişim çalışmaları için ideal bir çok hücreli model sistemi olarak hizmet vermiştir. 21 yaşında. Yumurta odaları kolayca izole edilir, büyük ve saydam, ve ex vivo analiz saatlerce dayanabilir, onları görüntüleme denemeleri için son derece kolay hale. Çok iş aktif tercüme edilmeden önce ayrık alt hücre bölgelere maternal transkriptleri asimetrik lokalizasyonu üzerinde duruldu. Özellikle, Oskar mRNA lokalizasyonu ve oocayt posterior Pole sonraki çeviri ölümcül bir bicaudal embriyo fenotipi22önlemek için sıkıca düzenlenmiş bir şekilde gerçekleşmesi gerekir. Oskar mRNA, Hemşire hücreleri denilen 15 germline hücrelerinde yazılmış ve aktif olarak sitoplazmik köprüler aracılığıyla taşınan, halka kanalları denilen, oocyte içine, Olgun yumurta olur ve sonuçta döllenen germline hücre (Şekil 1a ). Uzun menzilli hücre içi seyahat ile birlikte, Oskar mrnp ve gelen protein faktörleri dinamik işe alma ve değişimi ile ilgili zaten mevcut bilgi önemli miktarda, Oskar bir tercih aday yapmak çalışma mRNA yaşam döngüsünün birçok süreçleri. MBs mRNA lokalizasyonu süreci ile ilgili detayları açığa ve Drosophila oogenesis sırasında mRNA taşıma kontrol protein faktörlerinin düzenleme ve fonksiyon deşifre etmede enstrümantal olmuştur. Özellikle, MBS 'yi hemşire hücrelerine mikro injeksiyon ve canlı hücre görüntüleme deneyleri yaparak, endojen mrb 'ların takibi mümkündür8,23.
Burada sunulan yol haritası, doğal hücresel ortamında endojen mRNA 'yı izlemek için veri analizini gerçekleştirirken, MBs kullanarak canlı bir hücre görüntüleme deneyi yaparak, görüntüleme verilerini elde ederek tam bir sürecin adımlarını sunar. Adımlar değiştirilebilir ve daha fazla kendi laboratuar ayarı içinde diğer dokular/hücre türleri ile çalışan araştırmacılar ihtiyaçlarını karşılamak için optimize edilebilir.
Protokol
1. canlı hücre görüntüleme için MBs tasarımı
-
Mfold SUNUCUSUNDAN "RNA formu" kullanarak mRNA hedefin ikincil yapısını tahmin etmek IÇIN hedef RNA sırasını katlayın (http://unafold.RNA.Albany.edu/?q=mfold/RNA-Folding-form).
- Yapıştır/FASTA formatında hedef dizisini yükleyin, 5 veya% 10 alt optimalite seçin (sırasıyla MFE değerinin 5 veya% 10 ' unda katlanabilir ücretsiz enerji içeren yapılar) ve en fazla hesaplanan katlanmaların sayısını buna göre ayarlayın (örn.% 10 için daha büyük alt-optimality).
Not: MBs tasarlarken alt optimum ikincil yapıların eklenmesi, hedef mRNA içindeki bölgelerin tanımlanmasına izin verir, bu da minimum serbest enerji (MFE) yapısı için tahmin edildiği gibi daha esnek veya daha sert olabilir canlı hücre görüntüleme için uygun MBs genel tasarımı. - 801 ve 8.000 NT arasında mRNA uzunlukları için 800 nükleotidler (NT) veya "toplu iş" mRNA hedefleri için bir "acil iş" seçin. "ss-Count" dosyasını basit metin dosyası olarak kaydedin.
- Yapıştır/FASTA formatında hedef dizisini yükleyin, 5 veya% 10 alt optimalite seçin (sırasıyla MFE değerinin 5 veya% 10 ' unda katlanabilir ücretsiz enerji içeren yapılar) ve en fazla hesaplanan katlanmaların sayısını buna göre ayarlayın (örn.% 10 için daha büyük alt-optimality).
-
MRNA hedefi için birkaç MB tasarımı ( pinmol programının kullanımını açıklayan öğreticiler bakın) istenen parametrelerle pinmol programı (https://bratulab.wordpress.com/Software/) için giriş olarak adım 1,1 ' de elde edilen "ss-Count" dosyasını kullanın 24 at https://bratulab.wordpress.com/tutorial-pinmol-Mac/).
- BLAST analizi gerçekleştirerek seçilen MBs 'nin özgüllüğünü belirleyin: uygun veritabanıyla "blastn" kullanın (örneğin, Oskar mRNA 'Ya özgü MBS için "RefSeq-RNA" veritabanını ve Drosophila melanogaster organizmayı kullanın).
- MRNA hedefinin herhangi bir dokuya özgü ifadeyi tanımlayın (örn. Oskar mRNA flybase ≫ yüksek verimlilik ifade verisi ≫ FlyAtlas anatomi microarray veya modENCODE Anatomy RNA-Seq; http://flybase.org/Reports/FBgn0003015) ve ile karşılaştırın herhangi bir pozitif BLAST hits. Aynı zamanda ilgi doku/hücre içinde ifade edilen diğer MRI ile >% 50 çapraz Homoloji gösteren probları ortadan kaldırın.
- Canlı hücre görüntüleme (örn. Cy5/BHQ2)25' i gerçekleştirmek için kullanılabilen mikroskopinin ayarlanması için uygun fluorophore ve giderici çiftini seçin.
2. MB sentezi, arıtma ve karakterizasyonu
- Daha önce açıklanan7, ya da ticari sağlayıcılardan Hizmetleri, sentezlemek ve bir beş MBS arındırmak için (Yukarıdaki nota bakın), aşağıdaki etiketleme şeması kullanarak-ev sentezi ve arıtma kullanın: [5 ' (fluorophore)-(C3 veya C6 bağlayıcı)-(2 '-O -metil MB serisi)-(Quencher) 3 ']. Ters fazlı HPLC kullanarak, evde veya ticari sağlayıcının hizmetlerini kullanarak MBs 'yi arındırın.
Not: Otomatik prob sentezi için kullanılan fosforamidler 2 '-O-metil ribonükleotit modifikasyon olmalıdır. Bir de daha kısa bir MB ve hedef mRNA26arasında bir melez istikrarı artırmak için kilitli nüklik asit (nNa) ve 2 '-O-metil modifikasyonları Chimeras kullanabilirsiniz. - Hedeflenen RNA bölgesinin dizisiyle eşleşen DNA oligonükleotidler synthesizer ve böylece MBs prob bölgesine tamamlayıcı, in vitro karakterizasyon kullanmak için (bkz: 2,3 adım 2,5; Yukarıdaki Not). DNA-oligonükleotid hedef Mimic ile MB hibridizasyon maksimize, DNA hedef dört ek nükleotid her ucunda dahil ederek, hedef mRNA sırası bulunan gibi.
Not: hedeflenen sırayı algılamak için MB 'nin verimliliğinin daha titiz bir karakterizasyonu, tamamlayıcı DNA oligonükleotidler8yerine in vitro sentezlenmiş RNA hedefleri kullanılarak gerçekleştirilebilir. - Tek başına MB termal denatürasyon gerçekleştirmek, onun erime sıcaklığı ölçmek (TM), ve MB fizyolojik sıcaklıkta istenen saç iğne şeklini varsayar onaylayın. 60 ile 90 °C arasındaki TM değerlerini gözlemledik.
- DNA oligonükleotit hedef varlığında MB termal denatürasyon gerçekleştirmek ve MB ölçmek: DNA hedef melez 's TM, daha önce açıklanan7. MB: DNA hibrid için 55 ile 60 °C arasında bir TM istenidir.
- Karşılık gelen DNA oligonükleotit hedef ile in vitro hibridizasyon reaksiyonlar gerçekleştirin ve MB verimliliğini belirlemek: fizyolojik sıcaklıkta DNA hibrid oluşumu, daha önce açıklanan7. DNA hedef mimik ile hızlı hibridizasyon kinetik istenen, ancak DNA hedefleri ile yüksek hibridizasyon verimliliği göstermez MBS In vitro ve/veya in vivo hedef mRNA ile daha iyi bir performans olabilir.
3. mikroenjeksiyon için bireysel yumurta odaları diseksiyon ve hazırlanması
- Taze Maya hamur ile 2-3 gün boyunca yeni yumurtadan, evlenmiş kadın yem.
- Anesteziye bir CO2 pad üzerinde uçar ve ince cımbız (Dumont #5) kullanarak, bir cam kapak slip üzerinde 700 Halocarbon yağı bir damla içine transfer 1-2 kadın.
- Bir çift Cımbız kullanarak, bir stereomicroscope altında dorsal tarafı ile sinek yönlendirmek. Arka ucunda küçük bir kesi yaparak kadın karın incelemek ve yavaşça yağ içine yumurtalıkların çifti sıkmak.
- Yumurtalıkları yeni bir coverslip üzerinde bir yağ damlası üzerine ekidir. Diğer cımbız ile ovariole en genç aşamaları kapalı pinching ederken yavaşça bir cımbız ile bir yumurtlama tutun. Oskar mRNA aktif olarak lokalize ve Mid-oogenesis sonra (aşamaları > 7), ve Genç yumurta odaları (aşamaları < 7) enjekte etmek daha zordur ve uzun süre hayatta değilsiniz. Bireysel ovarioles veya yumurta odaları izole edilmiş ve dikey olarak hizalanıncaya kadar yavaşça kapak kayma (aşağı hareket ile) sürükleyin. Ovariole yumurta zincirinden istenmeyen aşamaları keserek daha ayrı tek yumurta odaları.
Not: Bireysel alay yumurta odaları yağ yüzen değil emin olun, ve onlar kapak kayma uygun. Bu hem başarılı mikroenjeksiyon ve görüntü edinme için önemlidir.
4. yumurta Chambers hemşire hücreleri içine MBs mikroenjeksiyon
- Bir moleküler işaret (örn. osk2216Cy5) kullanarak MB çözümünü hazırlayın veya farklı mrb 'ler hedefleyen ve ışıksal farklı fluorophores (örn. osk2216Cy5 ve drongo1111Cy3) ile etiketli iki MBS karışımı. HybBuffer 'da her MB 200-300 ng/μL konsantrasyonu kullanın (50 mM Tris-HCl-pH 7,5, 1,5 mM MgCl2 ve 100 mm NaCl). Her HybBuffer (örn. osk82, osk1236, osk2216) 200 ng/μL aynı mRNA hedefleyen aynı fluorophore ile etiketlenmiş dört MBs bir kokteyl için. Mikroenjeksiyon için iğne yüklemeden hemen önce MB çözüm aşağı spin.
- Hedefi seçin. Uygun bir yumurta odası bulmak ve mikroenjeksiyon yapmak için 40X yağ amacı önerilir.
- Ayrık yumurta odası ile lamel magazini mikroskop sahne üzerine monte edin. Odak konumda hedefi getirmek ve bir orta-geç gelişimsel aşamada bir yumurta odası tanımlamak, bu düzgün mikroenjeksiyon için yönlendirilmiş (yani, bir hemşire içinde kolay enjeksiyon için izin iğne ucu Için dik AàP ekseni ile oksit için proksimal hücre).
- ~ 1 μL MB çözeltisi (bkz . Adım 4,1) ile bir iğne (ticari veya ev27hazırlanmış) yükleyin ve microinjector bağlayın. D. melanogaster yumurta odalarında mikroenjeksiyonları için, bir açı < 45 ° mikroskop aşamaya (örneğin 30 °) birkaç hemşire hücrelerini delme önlemek için Iğne ( malzeme tablosunabakın) yönlendirmek.
- 500-1000 hPa enjeksiyon basıncı ve 100-250 hPa Dengeleme basıncı ile enjektörü ayarlayın (bkz. malzeme tablosu).
- Yavaşça yumurta odaları yağ damla geçersiz bir alan görünümü alanında getirmek için sahne hareket ettirin.
- Mikro manipülatör joystick kullanarak, hafifçe yağ damlası içine iğne düşük ve bakış alanı çevre doğru odağı içine ucu getirmek.
- İğne ucunu hava kaldırmak ve iğne akışı olduğundan emin olmak için bir ' temiz ' işlevi gerçekleştirin.
- İğne ev konumuna getirin ve mikroenjekte olmak için yumurta odasına odaklanmak, sonra odak içine iğne geri getirmek ve yumurta odasının kenarına yakın konumlandırmak.
- Folikül hücrelerini hemşire hücrelerinden ayıran membranın odak noktası olduğu gibi, hedefin Z pozisyonunun ince bir ayarını gerçekleştirin.
- Bir hemşire hücresine iğne takın ve 2-5 s için enjeksiyon gerçekleştirin.
- İğneyi yavaşça çıkarın ve ev konumuna geri çekin.
- Görüntü edinme (60-63x veya 100x) için istenen büyütme hedefini değiştirmek, yumurta odasına odaklanmak, ve edinme başlar.
5. bir Spinning disk Confocal mikroskop kurulum kullanarak veri edinme
Not: Özel Kurulumumuz için malzeme tablosunu görün.
- 8-16-bit görüntülerin XYZCt yığınını kaydetmek için edinme protokolünü ayarlayın (XYZ = hacim, C = kanal, t = zaman).
- İstenilen kanallar için lazer hatları seçin (örn. Cy5 için 641 nm lazer ve GFP için 491 Nm) ve kanalları sırayla elde etmek: ilk olarak her kanaldaki floresan sinyali ve ardından Z konumunu değiştirmek için uygun kolokalizasyon analizine izin verir.
- Z adımını (örn. 0,3 μm), üst ve alt Z sınırlarını (örn. -2 μm-2 μm) seçin.
- Edinme süresini ve örnekleme hızını girin (örn. 1 h 'ye kadar her 15-30 s).
- Satın alma işlemini başlatın.
6. işleme, Izleme ve Koyerelleştirme bilgileri almak için veri analizi ve video dosyalarının hazırlanması
- Görüntü Işleme
- Download, açmak ve açık Icy, bioımage Bilişim için açık bir topluluk platformu (http://icy.bioimageanalysis.org/)
- 5. adımda elde edilen XYZCt yığınını açın: resim/sıra > dosyası > aç.
- Stack 'i ımagej 'ye Dönüştür: ımagej > Araçları > ıJ 'ye Dönüştür, ayrılmış mod açık.
- Bir substack (Z adımlarının bir dizi ve daha da analiz edilecek zaman noktaları bir seçim) olun: ımagej > Image > yığınları > Araçları > yapmak Substack...; istediğiniz kanalları, Z-adımlarını ve zaman noktalarını seçin.
- Substack TIFF dosyası olarak kaydedin: ımagej > Dosya > > tiff olarak kaydedin...; Sonraki adımlar için bu dosyayı kullanın.
- Bölünmüş kanallar: ımagej > Image > Color > bölünmüş kanallar.
- Arka plan yığını kullanarak veya arka plan çıkarma: ımagej > Process > Image Calculator... veya Rolling Ball seçeneğini kullanarak: ımagej > Process > çıkarma Arkaplan..., Rolling Ball yarıçapı seçin. "Kabul et" seçeneğini seçmeden önce seçilen yarıçapın görüntüsünü önizleyin.
Not: arka plan sinyali ağırlıklı olarak flurorophore yanlış Quenching ortaya çıkacaktır. Sinyal: arka plan oranı (s:b) genellikle bir MB 's "parlaklık" için bir gösterge olarak kullanılır ve MB ve DNA hedef oligonükleotide in vitro hibridizasyon deneyleri ölçülür. Örneğin, MBs osk1236 ve osk2216 bir S:B var ~ 81 ve ~ 120, sırasıyla. - Her kanal için parlaklık ve kontrastı ayarlayın: ımagej > Image > > Parlaklık/kontrast ayarlayın, Uygula 'yı seçin.
- Her kanalı ayrı bir TIFF dosyası olarak kaydedin: ımagej > Dosya > > tiff olarak kaydedin....
- İki kanal birleştir: ımagej > Image > Color > birleştirme kanalları...; kanalları seçin. Yeni yığını yeni bir TIFF dosyası olarak kaydedin (bkz . Adım 6.1.8).
- Spot algılama ve Izleme
- Geri Icy Dönüştür: ımagej > Araçları > Icy dönüştürmek.
- Ölçek çubuğu eklentisi yüklüyse, bir ölçek çubuğu otomatik olarak Icy 'ye dönüşüm üzerine yığına kaplanmış [plugins > Setup > Online eklentisi kullanarak ara]. Gerekirse, Ölçek çubuğunu Denetçi penceresi (ekranın sağ tarafı) > Katman sekmesi > adı > ölçek çubuğunun üzerinden düzenleyin.
- Ölçek çubuğunu özgün yığınından kaldırmak için katman sekmesi > adı 'ndan ölçek çubuğu için ' göz ' simgesinin işaretini kaldırın/geçersiz hale getirin. Son yığınında yeniden etkinleştirilebilir.
- Yeni işlenmiş yığını görüntü penceresinin menü çubuğundan "Camera" simgesini kullanarak bir ekran görüntüsü alarak kaydedin, "geçerli görünümün bir ekran görüntüsü alın" ve Dosya > > tiff olarak kaydedin....
- Spot hassasiyet parametreleri zaten belirlendiği takdirde adım 6.2.7 üzerine hareket ederseniz nokta hassasiyetini belirleyin.
- Spotları Algıla: analiz edilecek görüntü veya yığını içeren pencereyi seçin, algılama & Izleme > algılama > Spot dedektörü ve ayarlar parametrelerini doldurun:
- Giriş için "Currentsequenceınputdetection" (varsayılan) öğesini seçin.
- Ön Işleme için, "kanal 0" (varsayılan) veya istenen kanalı Denetçi penceresi > sırası sekmesindeki numarayı çapraz başvurarak seçin.
- Dedektör için, "siyah arka planda parlak noktayı Algıla;", yalnızca analizini yapmak için yığınlarda yeterli Z-dilim yoksa "3B için 2B Dalgıcıklar kullanımını zorla" seçeneğini belirleyin. Her ölçek için "Scale (s)" ve "sensitivite" seçeneğini belirleyin (daha büyük noktalar için daha fazla ölçek ekleyin). Ölçek ve hassasiyet (büyük sayı daha hassas algılama, en fazla 140 Icy tarafından önerilen) deneme ve hata değişkenleri, bu görsel olarak daha sonra kontrol edilmelidir ve üzerine karar verilir.
- Ilgi bölgesi için "ROIfromSequence" (varsayılan) kullanın.
- Filtreleme için "NoFiltering" (varsayılan) kullanın veya "kabul edilen nesnelerin aralığı (piksel cinsinden)" tanımlamak için "SizeFiltering" seçin.
- Çıkış: seçin XLS veya XML çıkış ayarı (2007 MS Excel veya daha önce kullanırken XML biçimini seçin ve > 65000 noktalar vardır). Spot Dedektör sonuçları izleme analizi için kullanılırsa, "SwimmingPool 'a dışa aktar" seçeneğini de seçin.
- Spotlar tüm veya çoğu algılandığında kadar çeşitli Ölçek/duyarlılık değerleri kullanarak Spotlar tekrar saptama. Tüm son parametreleri kaydedin.
- Kolokalizasyon analizi için, diğer kanal için spot algılaması yineleyin.
- Spotları izlemek için algılama & Izleme > Izleme > Spot Izleme > nokta dedektörü 'ni adım 6.2.6 gelen parametrelerle çalıştırın veya mevcut bir veri kümesini seçmek için "algılama sonuçlarını burada Seç" açılır menüsünü kullanın (bunun için spot Dedektör penceresini tutun Adım 6.2.5) açın. "Tahmin parametreleri" düğmesine basın ve parametreler tahmini açılır penceresinde istediğiniz hedef hareketi seçin (örn. "hem difüzif hem de yönlendirilmiş"). "Çalıştır izleme" düğmesine basın.
- Adım 6.2.7 oluşturulan yığınla başlayan 6.2.6 ve 6.2.7 adımlarını izleyerek, çok kanallı yığınların noktalarını izlerken diğer kanallar için spot algılamayı ve izlemeyi yineleyin.
- Parçaları görselleştirmek için algılama & Izleme > Izleme > Track Manager 'ı seçin – Bu pencere izleme çalışmasını tamamladıktan sonra otomatik olarak açılır. "Renkli parça Işlemcisi" için "etkinleştir" i seçin ve parçalar için istenilen renk gösterimini seçin. İlgili parça işlemcileri "parça Işlemcisi Ekle..." ile erişilebilir. (örn. "Işlemci zaman klibini Izle" seçeneğini belirleyin, "parça kesme parçayı" penceresini etkinleştirin ve geçerli zaman noktasından önce ve sonra görüntülenecek istenen algılama sayısını seçin.)
- Parça bilgilerini bir XML Track dosyası olarak Kaydet: algılama & Izleme > Izleme > Track Manager > Dosya > Farklı Kaydet....
- Görüntü penceresinin menü çubuğundan "kamera" simgesini kullanarak bir ekran görüntüsü alarak sonuçları kaydedin, "geçerli görünümün bir ekran görüntüsünü alın". Ekran görüntüleri algılanan Spotlar ve/veya sadece Denetçi penceresi > Katman sekmesi > adı > Overlay sarıcı bulunan ilgili göz simgesini (ler) devre dışı bırakma/aktive ederek parçaları ile alınabilir.
- Zaman damgası yerleşimi eklentisi yükleyin: Eklentiler > Kurulum > Online Plugin > TimeStamp Overlay > yükleyin.
- Zaman damgası ekle: plugins > TimeStamp Overlay (yeni). Zaman damgası yerleştirme ve biçimlendirme yönergeleri için açılır pencerede (ekranın sağ alt köşesinde) yönergeleri izleyin. Zaman aralığı eklenebilir/değiştirilmiş Denetçi penceresi > sırası sekmesinde > Sequence Özellikleri > Düzenle.
- Başka bir ekran görüntüsü alarak sonuçları kaydedin. Görüntü kaydet 1) Tiff formatı ve 2) AVI biçimi olarak; AVI formatı için ilk RGB işleme (görüntü/sıra > Rendering > RGB görüntü) dönüştürün.
- Görüntüyü istediğiniz oryantasyona döndürün: Müfettiş penceresi > sıra sekmesi > Tuval > döndürme.
- Döndürülmüş görüntüyü "geçerli görünümün ekran görüntüsünü alın" olarak kaydedin. Aynı zamanda görüntü ile döndürülecek gibi ölçek çubuğunun "göz" simgesinin seçilmemiş olduğundan emin olun.
- YG 'yi seçin ve kırpın: Ilgi alanı > 2B YG 'yi seçin > YG şeklini seçin ve ardından görüntüde YG oluşturun/çizin; Görüntü/sıra > düzlem (XY) > hızlı kırpma.
- Kolokalizasyon Analizi
- Bir kolokalizasyon Protokolü hazırlayın; çeşitli örnekler Icy Web sitesinde (http://icy.bioimageanalysis.org/protocol/List) (bkz. ek malzemeler) sağlanır.
- Yükleme kolokalizasyon protokolü: Araçlar > Scripting > Protocols > yükleme ve etkileşim bloklarında parametreleri ayarlamak (örneğin, "Wavelet spot algılama" blok kullanım parametrelerinde adım 6.2.6.) belirlenir.
- Bir parçacık boyutunu piksel cinsinden ölçün, kolokalizasyon mesafeyi belirleyin ve "Colocalizer" bloğuna "maksimum mesafe" olarak girin.
Not: Parçacık piksel cinsinden boyutu algılama sistemine bağlıdır. Boyutu ölçmek için, tek bir parçacığın içine yakınlaştırın ve sinyalin genişliğini kapsayan pikselleri el ile Sayın. En az üç parçacıklardan ortalama ölçümler. Kolokalizasyon için ayarlanacak en fazla uzaklık, parçacığın piksel cinsinden boyutudur (Bu, iki parçacık dokunmadan yarıçapının maksimum toplamını temsil eder). - İstenirse, kolokalizasyon analizi için bir veya daha fazla ROI seçin: Ilgi alanı > 2B YG > YG şeklini seçin > resimde YG 'Yi çizin.
- Kırpma YG (s): resim/sıra > düzlem (XY) > hızlı kırpma.
- Koyerelleştirme gerçekleştirin: protokoller Düzenleyici penceresi > seçilen protokol sekmesi > Çalıştır. Protokoller düzenleyici penceresindeki son blok, spot algılamaya göre genel koyerelleştirme yüzdesini içerir, ancak her zaman noktasında bulunan bilgiler Inspector penceresinde > çıktı sekmesinde bulunabilir.
- Adım 6.2.7 takip ederek kolokalize ve tek parçacıkları takip edin (parça lekeler).
- 6.2.16 adımda açıklandığı şekilde kaydedin.
Sonuçlar
Pinmolkullanarak, birkaç MBS bir mRNA hedefi için tasarlanabilir (Şekil 1B-C). Sentez ve arıtma sonrasında, seçilen MBs karakterizedir ve in vitro analizini kullanarak karşılaştırılır.
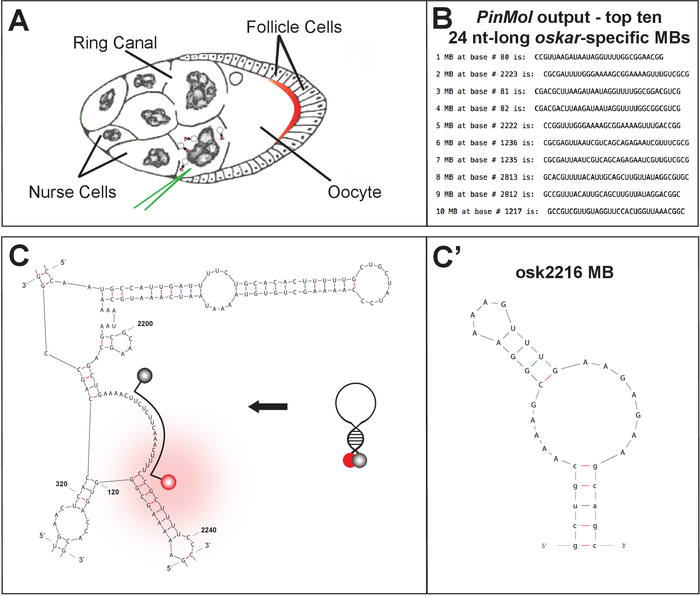
Şekil 1: endojen MRI 'ların canlı hücre görüntülemesinde teknik ve doku tanımı...
Tartışmalar
Drosophila yumurta odalarında endojen mRNA ticaretinin canlı görselleştirme artık pinmol yazılımı ile kolayca tasarlanabilen, spesifik, verimli ve çekirdeksiz dirençli MBS kullanımına dayanır. MBs bir hedef mRNA (tercihen bölgeler ikincil yapısı ücretsiz) içinde benzersiz dizileri algılamak için tasarlanmış spesifik problar, bir transkript olası son derece çözülmüş algılama yapma. Diğer dokular/hücre türleri için bu tekniği/Protokolü benimseyen tek sınırlama, faiz nu...
Açıklamalar
Yazarlar, ifşa etmek için ilgi çakışması yoktur.
Teşekkürler
Biz, sentez, etiketleme ve moleküler işaretçileri ve arıtma için Salvatore AE Marras (kamu sağlığı Araştırma Enstitüsü Merkezi, Rutgers Üniversitesi) teşekkür ve Daniel St Johnston (Gurdon Enstitüsü, Cambridge Üniversitesi) Oskar-MS2 için/ MCP-GFP transgenik sinek stok. Bu çalışma, Ulusal Bilim Vakfı karıyer Ödülü 1149738 ve DPB profesyonel personel Kongresi-CUNY Ödülü tarafından destekleniyordu.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Spectrofluorometer | Fluoromax-4 Horiba-Jobin Yvon | n/a | Photon counting spectrofluorometer |
| Quartz cuvette | Fireflysci (former Precision Cells Inc.) | 701MFL | |
| Dumont #5 tweezer | World Precision Instruments | 501985 | Thin tweezers are very important to separate out the individual egg chambers |
| Halocarbon oil 700 | Sigma-Aldrich | H8898 | |
| Cover slip No.1 22 mm x 40 mm | VWR | 48393-048 | |
| Dissecting microscope | Leica MZ6 Leica Microsystems Inc. | n/a | |
| CO2 fruit fly anesthesia pad | Genesee Scienific | 59-114 | |
| Tris-HCL pH 7.5 | Sigma-Aldrich | 1185-53-1 | |
| Magnesium chloride | Sigma-Aldrich | 7791-18-6 | |
| NaCl | Sigma-Aldrich | 7647-14-5 | |
| Spinning disc confocal microscope | Leica DMI-4000B inverted microscope equipped with Yokogawa CSU 10 spinning disc Leica Microsystems Inc. | n/a | |
| Hamamatsu C9100-13 ImagEM EMCCD camera | Hamamatsu | n/a | |
| PatchMan NP 2 Micromanipulator | Eppendorf Inc. | 920000037 | |
| FemtoJet Microinjector | Eppendorf Inc. | 920010504 | |
| Injection needle: Femtotips II | Eppendorf Inc. | 930000043 | |
| Loading tip: 20 μL Microloader | Eppendorf Inc. | 930001007 | |
| Micro Cover glasses no. 1 or 1.5, 22 mm x 40 mm | VWR | 48393-026; 48393-172 | |
| Dry yeast | Any grocery store | n/a | |
| Computer, > 20 GB RAM | Although processing can be carried out on most computers, higher capabilities will increase the speed of the processing |
Referanslar
- Tyagi, S. Imaging intracellular RNA distribution and dynamics in living cells. Nature Methods. 6 (5), 331-338 (2009).
- Bao, G., Rhee, W. J., Tsourkas, A. Fluorescent probes for live-cell RNA detection. Annual Reviews of Biomedical Engineering. 11, 25-47 (2009).
- Mannack, L. V., Eising, S., Rentmeister, A. Current techniques for visualizing RNA in cells. F1000Research. 5, (2016).
- Larson, D. R., Zenklusen, D., Wu, B., Chao, J. A., Singer, R. H. Real-time observation of transcription initiation and elongation on an endogenous yeast gene. Science. 332 (6028), 475-478 (2011).
- Bertrand, E., et al. Localization of ASH1 mRNA particles in living yeast. Molecular Cell. 2 (4), 437-445 (1998).
- Garcia, J. F., Parker, R. MS2 coat proteins bound to yeast mRNAs block 5' to 3' degradation and trap mRNA decay products: implications for the localization of mRNAs by MS2-MCP system. RNA. 21 (8), 1393-1395 (2015).
- Bratu, D. P. Molecular beacons: Fluorescent probes for detection of endogenous mRNAs in living cells. Methods in Molecular Biology. 319, 1-14 (2006).
- Bratu, D. P., Cha, B. J., Mhlanga, M. M., Kramer, F. R., Tyagi, S. Visualizing the distribution and transport of mRNAs in living cells. Proceedings of the National Academy of Sciences of the Unites States of America. 100 (23), 13308-13313 (2003).
- Tyagi, S., Kramer, F. R. Molecular beacons: probes that fluoresce upon hybridization. Nature Biotechnology. 14 (3), 303-308 (1996).
- Chen, M., et al. A molecular beacon-based approach for live-cell imaging of RNA transcripts with minimal target engineering at the single-molecule level. Scientific Reports. 7 (1), 1550 (2017).
- Liu, Y., et al. Multiplex detection of microRNAs by combining molecular beacon probes with T7 exonuclease-assisted cyclic amplification reaction. Analytical and Bioanalytical Chemistry. 409 (1), 107-114 (2017).
- Baker, M. B., Bao, G., Searles, C. D. In vitro quantification of specific microRNA using molecular beacons. Nucleic Acids Research. 40 (2), e13 (2012).
- Ko, H. Y., et al. A color-tunable molecular beacon to sense miRNA-9 expression during neurogenesis. Scientific Reports. 4, 4626 (2014).
- Vet, J. A., et al. Multiplex detection of four pathogenic retroviruses using molecular beacons. Proceedings of the National Academy of Sciences of the Unites States of America. 96 (11), 6394-6399 (1999).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Methods in Molecular Biology. 429, 209-224 (2008).
- Li, W. M., Chan, C. M., Miller, A. L., Lee, C. H. Dual Functional Roles of Molecular Beacon as a MicroRNA Detector and Inhibitor. Journal of Biological Chemistry. 292 (9), 3568-3580 (2017).
- Kuang, T., Chang, L., Peng, X., Hu, X., Gallego-Perez, D. Molecular Beacon Nano-Sensors for Probing Living Cancer Cells. Trends in Biotechnology. 35 (4), 347-359 (2017).
- Ban, K., et al. Non-genetic Purification of Ventricular Cardiomyocytes from Differentiating Embryonic Stem Cells through Molecular Beacons Targeting IRX-4. Stem Cell Reports. 5 (6), 1239-1249 (2015).
- Hadjinicolaou, A. V., Demetriou, V. L., Emmanuel, M. A., Kakoyiannis, C. K., Kostrikis, L. G. Molecular beacon-based real-time PCR detection of primary isolates of Salmonella Typhimurium and Salmonella Enteritidis in environmental and clinical samples. BMC Microbiology. 9, 97 (2009).
- McLaughlin, J. M., Bratu, D. P. Drosophila melanogaster Oogenesis: An Overview. Methods in Molecular Biology. 1328, 1-20 (2015).
- Bastock, R., St Johnston, D. Drosophila oogenesis. Current Biology. 18 (23), R1082-R1087 (2008).
- Rongo, C., Gavis, E. R., Lehmann, R. Localization of oskar RNA regulates oskar translation and requires Oskar protein. Development. 121 (9), 2737-2746 (1995).
- Mhlanga, M. M., et al. In vivo colocalisation of oskar mRNA and trans-acting proteins revealed by quantitative imaging of the Drosophila oocyte. PLoS One. 4 (7), e6241 (2009).
- Bayer, L. V., Omar, O. S., Bratu, D. P., Catrina, I. E. PinMol: Python application for designing molecular beacons for live cell imaging of endogenous mRNAs. bioRxiv. , (2018).
- Marras, S. A., Kramer, F. R., Tyagi, S. Efficiencies of fluorescence resonance energy transfer and contact-mediated quenching in oligonucleotide probes. Nucleic Acids Research. 30 (21), e122 (2002).
- Bratu, D. P., Catrina, I. E., Marras, S. A. Tiny molecular beacons for in vivo mRNA detection. Methods in Molecular Biology. 714, 141-157 (2011).
- Dean, D. A. Preparation (pulling) of needles for gene delivery by microinjection. Cold Spring Harbor. 2006 (7), (2006).
- Alami, N. H., et al. Axonal transport of TDP-43 mRNA granules is impaired by ALS-causing mutations. Neuron. 81 (3), 536-543 (2014).
- Jackson, S. R., et al. Applications of Hairpin DNA-Functionalized Gold Nanoparticles for Imaging mRNA in Living Cells. Methods in Enzymology. 572, 87-103 (2016).
- Zimyanin, V. L., et al. In vivo imaging of oskar mRNA transport reveals the mechanism of posterior localization. Cell. 134 (5), 843-853 (2008).
- Catrina, I. E., Marras, S. A., Bratu, D. P. Tiny molecular beacons: LNA/2'-O-methyl RNA chimeric probes for imaging dynamic mRNA processes in living cells. ACS Chemical Biology. 7 (9), 1586-1595 (2012).
- Chen, A. K., Behlke, M. A., Tsourkas, A. Efficient cytosolic delivery of molecular beacon conjugates and flow cytometric analysis of target RNA. Nucleic Acids Research. 36 (12), e69 (2008).
- Nitin, N., Santangelo, P. J., Kim, G., Nie, S., Bao, G. Peptide-linked molecular beacons for efficient delivery and rapid mRNA detection in living cells. Nucleic Acids Research. 32 (6), e58 (2004).
- Chen, A. K., Behlke, M. A., Tsourkas, A. Avoiding false-positive signals with nuclease-vulnerable molecular beacons in single living cells. Nucleic Acids Research. 35 (16), e105 (2007).
- Bevilacqua, P. C., Ritchey, L. E., Su, Z., Assmann, S. M. Genome-Wide Analysis of RNA Secondary Structure. Annual Review of Genetics. 50, 235-266 (2016).
- Mhlanga, M. M., Vargas, D. Y., Fung, C. W., Kramer, F. R., Tyagi, S. tRNA-linked molecular beacons for imaging mRNAs in the cytoplasm of living cells. Nucleic Acids Research. 33 (6), 1902-1912 (2005).
- Eliceiri, K. W., et al. Biological imaging software tools. Nature Methods. 9 (7), 697-710 (2012).
- Bolte, S., Cordelieres, F. P. A guided tour into subcellular colocalization analysis in light microscopy. Journal of Microscopy. 224 (Pt 3), 213-232 (2006).
- Trcek, T., et al. Drosophila germ granules are structured and contain homotypic mRNA clusters. Nature Commununications. 6, 7962 (2015).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır