Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Visualisierung und Verfolgung endogenes mRNAs in Live Drosophila melanogaster Eierkammern
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll zur Visualisierung, Detektion, Analyse und Verfolgung des endogenen mRNA-Handels in der lebenden Drosophila melanogaster Eikammer mit molekularen Baken, Spinnscheiben-Konfokalmikroskopie und Open-Source-Analyse vor. software.
Zusammenfassung
Fluoreszenzbasierte bildgebende Verfahren in Kombination mit Entwicklungen in der Lichtmikroskopie haben die Art und Weise revolutioniert, wie Zellbiologen Live-Zell-Bildgebungsstudien durchführen. Die Methoden zum Nachweis von RNAs haben sich stark ausgeweitet, seit bahnbrechende Studien die standortspezifische mRNA-Lokalisierung mit der Genexpressionsregulation verknüpften. Dynamische mRNA-Prozesse können nun über Ansätze visualisiert werden, die mRNAs erkennen, gekoppelt mit Mikroskopie-Setups, die schnell genug sind, um den dynamischen Bereich des molekularen Verhaltens zu erfassen. Die molekulare Beacon-Technologie ist ein hybridisierungsbasierter Ansatz, der in der Lage ist, endogene Transkripte in lebenden Zellen direkt zu detektieren. Molekulare Leuchttürme sind haarförmige, intern abgeschreckte, single-nukleotiddiskriminierende Nukleinsäuresonden, die erst bei Hybridisierung zu einer einzigartigen Zielsequenz fluoreszieren. In Verbindung mit fortschrittlicher Fluoreszenzmikroskopie und hochauflösender Bildgebung ermöglichen sie eine räumliche und zeitliche Verfolgung der intrazellulären Bewegung von mRNAs. Obwohl diese Technologie die einzige Methode ist, die in der Lage ist, endogene Transkripte zu erkennen, haben Zellbiologen diese Technologie aufgrund von Schwierigkeiten bei der Entwicklung solcher Sonden für die Bildgebung von lebenden Zellen noch nicht vollständig angenommen. Eine neue Softwareanwendung, PinMol, ermöglicht ein verbessertes und schnelles Design von Sonden, die am besten geeignet sind, um mRNA-Zielregionen innerhalb einer lebenden Zelle effizient zu hybridisieren. Darüber hinaus ermöglichen hochauflösende, Echtzeit-Bildaufnahme und aktuelle Open-Source-Bildanalyse-Software eine verfeinerte Datenausgabe, was zu einer feineren Bewertung der Komplexität führt, die den dynamischen Prozessen zugrunde liegt, die im Lebenszyklus der mRNA beteiligt sind.
Hier stellen wir ihnen ein umfassendes Protokoll zur Gestaltung und Lieferung molekularer Leuchttürme in Drosophila melanogaster Eierkammern vor. Die direkte und hochspezifische Detektion und Visualisierung endogener mütterlicher mRNAs erfolgt über die konfokale Mikroskopie der Spinnscheibe. Imaging-Daten werden mithilfe der Objekterkennung und -verfolgung in der eisigen Software verarbeitet und analysiert, um Details über die dynamische Bewegung von mRNAs zu erhalten, die in spezialisierte Regionen innerhalb der Eizelle transportiert und lokalisiert werden.
Einleitung
Zellbiologische Studien, die dynamische Ereignisse mit räumlicher und zeitlicher Auflösung visualisieren, wurden durch die Entwicklung fluoreszenzbasierter Live-Zell-Bildgebungstechniken ermöglicht. Derzeit wird die in vivo mRNA-Visualisierung über Technologien erreicht, die auf RNA-Aptamer-Protein-Wechselwirkungen, RNA-Aptamer-induzierter Fluoreszenz organischer Farbstoffe und Nukleinsäuresonde-Glühen1,2, 3. Sie alle bieten eine hohe Spezifität, Empfindlichkeit und Signal-Hintergrund-Verhältnis. RNA-aptamerzentrierte Ansätze erfordern jedoch eine umfassende genetische Manipulation, bei der ein Transgen entwickelt wurde, um eine RNA mit künstlichen Strukturmotiven auszudrücken, die für die Protein- oder organische Farbstoffbindung erforderlich sind. Beispielsweise erfordert das MS2/MCP-System die Koexpression eines Transgens, das ein RNA-Konstrukt exmittiert, das mehrere Tandemwiederholungen der Bindungssequenz für das Bakteriophage MS2-Beschichtungsprotein (MCP) enthält, und ein weiteres Transgen, das für ein fluoreszierendes Protein kodiert. mit MCP4,5verschmolzen. Die Zugabe solcher sekundären Strukturmotive zur RNA, zusammen mit einem sperrigen fluoreszierend getaggten Protein, hat Bedenken aufkommen lassen, dass native RNA-Prozesse betroffen sein könnten6. Eine Technologie, die diesem Anliegen begegnet und weitere einzigartige Vorteile bietet, ist der nukleinsäurebasierte Ansatz, molekulare Leuchttürme (MBs). MBs ermöglichen den Multiplex-Nachweis von endogenen mRNAs, die Diskriminierung einzelner Nukleotidvariationen und eine schnelle Kinetik der Hybridisierung mit Ziel mRNA7,8. MBs sind Oligonukleotid-Sonden, die in einer abgeschreckten Haarnadelfalte verbleiben, bevor sie sich einer fluorogenen Konformationsänderung unterziehen, sobald sie sich mit ihren Zielen hybridisieren (Abbildung 1C)9. Mehrere Gruppen hatten Erfolg bei der Verwendung von MBs, um sowohl nicht-kodierende RNAs (microRNAs und lncRNAs)10,11,12,13, RNA-Retroviren14 und dynamisches DNA-Protein zu detektieren Wechselwirkungen15. Sie wurden erfolgreich für die Bildgebung in verschiedenen Organismen und Geweben eingesetzt, wie Zebrafisch-Embryonen16, Neuronen13, Tumorgewebe17, Differenzierung Kardiomyozyten18, und Salmonellen 19.
Hier beschreiben wir den Design-, Liefer- und Detektionsansatz für endogene mRNAs in lebenden D. melanogaster Eikammern gekoppelt mit einem Mikroskopie-Setup, das schnell genug ist, um den Dynamikbereich des aktiven molekularen Transports zu erfassen. Die D. melanogaster Eikammer diente als ideales multizelluläres Modellsystem für eine breite Palette von Entwicklungsstudien, von der frühen Keimbahnstammzellteilung und mütterlichen Genexpression bis hin zur Generierung des segmentalen Körperplans20, 21. Eierkammern sind leicht zu isolieren, groß und durchscheinend und können stundenlangen Ex-vivo-Analysen standhalten, wodurch sie für bildgebende Experimente sehr zugänglich sind. Viele Arbeiten konzentrierten sich auf die asymmetrische Lokalisierung von mütterlichen Transkripten in diskrete subzelluläre Regionen, bevor sie aktiv übersetzt wurden. Insbesondere muss die Oskar mRNA-Lokalisierung und ihre anschließende Übersetzung am hinteren Pol der Eizelle streng reguliert erfolgen, um einen tödlichen bicaudalen Embryo-Phänotyp22zu vermeiden. oskar mRNA wird in den 15 Keimbahnzellen transkribiert, krankenschwestern Zellen genannt, und aktiv durch zytoplasmatische Brücken, ringal genannt, in die Oozyte transportiert, die Keimlinzelle, die das reife Ei wird und letztlich befruchtet wird (Abbildung 1A ). Die beträchtliche Menge an Informationen, die bereits über die dynamische Rekrutierung und den Austausch von Proteinfaktoren zu und von Oskar mRNP sowie über die grenzüberschreitende grenzüberschreitende Reise von Oskar mRNP zur Verfügung stehen, macht Oskar zu einem bevorzugten Studienkandidaten. die vielen Prozesse des mRNA-Lebenszyklus. MBs haben maßgeblich dazu beigetragen, Details über den Prozess der mRNA-Lokalisierung zu enthüllen und die Regulation und Funktion von Proteinfaktoren zu entschlüsseln, die den mRNA-Transport während der Drosophila-Oogenese steuern. Insbesondere durch mikroinjizierende MBs in Pflegezellen und Durchführung von Live-Zell-Bildgebungsexperimenten ist die Verfolgung endogener mRNAs möglich8,23.
Die hier vorgestellte Roadmap bietet die Schritte eines vollständigen Prozesses, von der Durchführung eines Live-Zell-Bildgebungsexperiments mit MBs über die Erfassung von Bilddaten bis hin zur Durchführung von Datenanalysen zur Verfolgung endogenes mRNA in seiner nativen zellulären Umgebung. Die Schritte können modifiziert und weiter optimiert werden, um den Anforderungen von Forschern gerecht zu werden, die mit anderen Geweben/Zelltypen innerhalb ihrer eigenen Laborumgebung arbeiten.
Protokoll
1. Design von MBs für Live Cell Imaging
-
Falten Sie die Ziel-RNA-Sequenz, um die sekundäre Struktur des mRNA-Ziels mithilfe der "RNA-Form" vom mfold-Server (http://unafold.rna.albany.edu/?q=mfold/RNA-Folding-Form) vorherzusagen.
- Fügen Sie die Zielsequenz im FASTA-Format ein, wählen Sie 5 oder 10 % Suboptimalität (Strukturen mit freier Faltenergie innerhalb von 5 bzw. 10 % des MFE-Wertes) und passen Sie die maximale Anzahl berechneter Faltungen entsprechend an (z.B. größer für 10% Suboptimalität).
Anmerkung: Die Einbeziehung suboptimaler Sekundärstrukturen bei der Entwicklung von MBs ermöglicht die Identifizierung von Regionen innerhalb der Ziel-mRNA, die flexibler oder starrer sein können als nur für die minimale freie Energiestruktur (MFE), die die Gesamtdesign von MBs, die für die Bildgebung von Lebenden Zellen geeignet sind. - Wählen Sie einen "unmittelbaren Auftrag" für mRNA-Ziele von 800 Nukleotiden (nt) oder einen "Batch-Job" für mRNA-Längen zwischen 801 und 8.000 nt. Speichern Sie die Datei "ss-count" als einfache Textdatei.
- Fügen Sie die Zielsequenz im FASTA-Format ein, wählen Sie 5 oder 10 % Suboptimalität (Strukturen mit freier Faltenergie innerhalb von 5 bzw. 10 % des MFE-Wertes) und passen Sie die maximale Anzahl berechneter Faltungen entsprechend an (z.B. größer für 10% Suboptimalität).
-
Verwenden Sie die in Schritt 1.1 erhaltene Datei "ss-count" als Eingabe für das PinMol-Programm (https://bratulab.wordpress.com/software/) mit den gewünschten Parametern, um mehrere MBs für das mRNA-Ziel zu entwerfen (siehe Tutorials zur Beschreibung der Verwendung des PinMol-Programms 24 bei https://bratulab.wordpress.com/tutorial-pinmol-mac/).
- Bestimmen Sie die Spezifität ausgewählter MBs durch BLAST-Analyse: verwenden Sie "blastn" mit der entsprechenden Datenbank (z.B. für oskar mRNA-spezifische MBs verwenden Sie die "refseq-rna"-Datenbank und den Drosophila-Melanogaster-Organismus).
- Identifizieren Sie eine gewebespezifische Expression des mRNA-Ziels (z.B. für oskar mRNA Flybase> High-Throughput Expression Data> FlyAtlas Anatomy Microarray oder modENCODE Anatomy RNA-Seq; http://flybase.org/reports/FBgn0003015) und vergleichen Sie mit positive BLAST-Treffer. Beseitigen Sie Sonden, die >50% Cross-Homologie mit anderen mRNAs zeigen, die auch im Gewebe/der Zelle von Interesse ausgedrückt werden.
- Wählen Sie das Fluorophor- und Das Quencher-Paar aus, das für die Mikroskopie-Einrichtung geeignet ist, die für die Live-Zell-Bildgebung (z. B. Cy5/BHQ2)25zur Verfügung steht.
2. MB Synthese, Reinigung und Charakterisierung
- Verwenden Sie die interne Synthese und Reinigung wie zuvor beschrieben7oder Dienstleistungen von kommerziellen Anbietern, um ein bis fünf MBs zu synthetisieren und zu reinigen (siehe oben Anm.) unter Verwendung des folgenden Kennzeichnungsschemas: [5'(Fluorophore)-(C3 oder C6 linker)-(2'-O -methyl MB Sequenz)-(Quencher)3']. Reinigen Sie MBs mit Reverse-Phase HPLC, im Haus oder mit den Dienstleistungen des kommerziellen Anbieters.
HINWEIS: Die phosphoramiditen, die für die automatisierteSondensynthese verwendet werden, müssen die 2'- O-Methylribonukleotid-Modifikation haben. Man kann auch Chimären von abwechselnden Sperrnukleinsäure (LNA)und 2'- O-Methylmodifikationen verwenden, um die Stabilität eines Hybriden zwischen einem kürzeren MB und seiner ZielmRNA26zu erhöhen. - Synthetisieren Sie DNA-Oligonukleotide, die der Sequenz der zielgerichteten RNA-Region entsprechen und somit die Sondenregion von MBs ergänzen, zur Verwendung in der In-vitro-Charakterisierung (siehe Schritte 2.3 bis 2.5; oben Anmerkung). Maximieren Sie die Hybridisierung des MB mit dem DNA-Oligonukleotid-Ziel mimik, indem Sie an jedem Ende des DNA-Ziels vier zusätzliche Nukleotide einbeziehen, wie in der Ziel-mRNA-Sequenz gefunden.
Hinweis: Eine strengere Charakterisierung der Effizienz des MB zum Nachweis der Zielsequenz kann mit in vitro synthetisierten RNA-Zielen anstelle komplementärer DNA-Oligonukleotide8durchgeführt werden. - Führen Sie die thermische Denaturierung des MB allein durch, messen Sie seine Schmelztemperatur (Tm) und bestätigen Sie, dass das MB die gewünschte Haarnadelform bei physiologischer Temperatur annimmt. Wir haben Tm-Werte zwischen 60 und 90 °C beobachtet.
- Führen Sie die thermische Denaturierung des MB in Gegenwart des DNA-Oligonukleotidziels durch und messen Sie das Tm des MB:DNA-Zielhybrids, wie zuvor beschrieben7. Für den MB:DNA-Hybrid wird ein Tm zwischen 55 und 60 °C gewünscht.
- Führen Sie In-vitro-Hybridisierungsreaktionen mit dem entsprechenden DNA-Oligonukleotid-Ziel durch und bestimmen Sie die Effizienz der MB:DNA-Hybridbildung bei physiologischer Temperatur, wie zuvor beschrieben7. Schnelle Hybridisierungskinetik mit der DNA-Zielmimik ist erwünscht, aber MBs, die keine hohe Hybridisierungseffizienz mit DNA-Targets aufweisen, können eine bessere Leistung mit der Ziel-mRNA in vitro und/oder in vivo aufweisen.
3. Zerlegung und Vorbereitung einzelner Eierkammern zur Mikroinjektion
- Füttern Sie frisch geschlüpfte, gepaarte Weibchen für 2-3 Tage mit frischer Hefepaste.
- Anästhesisieren fliegt auf einem CO 2-Pad und überträgt mit einer feinen Pinzette (Dumont #5) 1-2 Weibchen in einen Tropfen Halocarbonöl 700 auf einem Glasdeckel-
- Mit einer Pinzette orientieren Sie die Fliege mit der dorsalen Seite unter einem Stereomikroskop nach oben. Sezieren Sie den weiblichen Bauch, indem Sie einen kleinen Schnitt am hinteren Ende machen und drücken Sie das Paar Eierstöcke sanft in das Öl.
- Die Eierstöcke auf einen Öltropfen auf einem neuen Deckelabrutsche abpflanzen. Halten Sie vorsichtig einen Eierstock mit einer Pinzette, während Sie die jüngsten Stufen des Ovariole mit der anderen Pinzette abkniffen. oskar mRNA ist aktiv lokalisiert an und nach der MittlerenOogenese (Stadien > 7), und jüngere Eikammern (Stadien < 7) sind schwieriger zu injizieren und überleben nicht so lange. Ziehen Sie langsam auf den Deckelschlupf (mit einer Abwärtsbewegung), bis einzelne Eizellen oder Eikammern isoliert und vertikal ausgerichtet sind. Weitere trennen einzelne Eikammern durch Verdrängung der unerwünschten Stadien von der eiförmigen Eikette.
HINWEIS: Stellen Sie sicher, dass individuell gehänselte Eierkammern nicht im Öl schwimmen und dass sie am Deckelschlupf haften. Dies ist sowohl für eine erfolgreiche Mikroinjektion als auch für die Bildaufnahme wichtig.
4. Mikroinjektion von MBs in die Pflegezellen von Eierkammern
- Bereiten Sie die MB-Lösung mit einem molekularen Leuchtfeuer (z. B. osk2216Cy5) oder einer Mischung aus zwei MBs vor, die auf verschiedene mRNAs abzielen und mit spektral unterschiedlichen Fluorophoren (z. B. osk2216Cy5 und drongo1111Cy3) gekennzeichnet sind. Verwenden Sie eine Konzentration von 200-300 ng/l pro MB in HybBuffer (50 mM Tris-HCl - pH 7,5, 1,5 mM MgCl2 und 100 mM NaCl). Für einen Cocktail aus vier MBs, die mit demselben Fluorophor gekennzeichnet sind und die jeweils auf dieselbe mRNA bei 200 ng/l in HybBuffer abzielen (z. B. osk82, osk1236, osk2216). Drehen Sie die MB-Lösung unmittelbar vor dem Laden der Nadel für die Mikroinjektion herunter.
- Wählen Sie das Ziel aus. Ein 40-faches Ölobjektiv wird empfohlen, um eine geeignete Eikammer zu finden und mikroinziert zu werden.
- Montieren Sie den Deckelrutsch mit sezierter Eikammer auf die Mikroskopbühne. Bringen Sie das Ziel in der Fokusposition auf und identifizieren Sie eine Eikammer in einem mittleren bis späten Entwicklungsstadium, die richtig für die Mikroinjektion ausgerichtet ist (d. h. mit der A-P-Achse senkrecht zur Nadelspitze, um eine einfache Injektion innerhalb einer Krankenschwester zu ermöglichen Zelle proximal zur Oozyte).
- Laden Sie eine Nadel (kommerziell oder im Haus27 hergestellt) mit einer Lösung von 1 L MB (siehe Schritt 4.1) und schließen Sie sie an den Mikroinjektor an. Bei Mikroinjektionen in D. melanogaster Eikammern die Nadel (siehe Materialtabelle)in einem Winkel von <45° an der Mikroskopstufe (z.B. 30°) orientieren, um das Durchsatzen mehrerer Pflegezellen zu vermeiden.
- Einrichten des Injektors mit einem Einspritzdruck von 500-1.000 hPa und einem Kompensationsdruck von 100-250 hPa (siehe Materialtabelle).
- Bewegen Sie langsam die Bühne, um in das Sichtfeld einen Bereich des Öltropfens leere Eikammern zu bringen.
- Mit dem Mikromanipulator-Joystick senken Sie die Nadel vorsichtig in den Öltropfen und bringen ihre Spitze in den Fokus zur Peripherie des Sichtfeldes.
- Führen Sie eine "saubere" Funktion aus, um die Luft von der Nadelspitze zu entfernen und sicherzustellen, dass es Einen Fluss von der Nadel gibt.
- Bringen Sie die Nadel in die Heimatposition und konzentrieren Sie sich auf die zu mikroinjizierende Eikammer, dann bringen Sie die Nadel wieder in den Fokus und positionieren Sie sie in der Nähe des Rands der Eikammer.
- Führen Sie eine Feineinstellung der Z-Position des Objektivs durch, so dass die Membran, die die Follikelzellen von Pflegezellen trennt, im Fokus steht.
- Setzen Sie die Nadel in eine Ammensamenzelle ein und führen Sie eine Injektion für 2-5 s durch.
- Entfernen Sie die Nadel vorsichtig und ziehen Sie sie in die Heimatposition ein.
- Ändern Sie das Ziel in die gewünschte Vergrößerung für die Bildaufnahme (60-63x oder 100x), konzentrieren Sie sich auf die Eikammer und beginnen Sie mit der Erfassung.
5. Erfassung von Daten mit einem Spinning Disc Konfokalmikroskop Setup
HINWEIS: Siehe Tabelle der Materialien für unsere spezifische Einrichtung.
- Richten Sie das Erfassungsprotokoll ein, um einen XYZCt-Stapel mit 8-16-Bit-Bildern aufzuzeichnen (XYZ = Volume, C = Kanal, t = Zeit).
- Wählen Sie Laserlinien für die gewünschten Kanäle (z.B. 641 nm Laser für Cy5 und 491 nm für GFP) und erfassen Sie die Kanäle sequenziell: zuerst das Fluoreszenzsignal in jedem Kanal und dann die Z-Position, um eine korrekte Kolokalisierungsanalyse zu ermöglichen.
- Wählen Sie den Z-Schritt (z.B. 0,3 m) und die oberen und unteren Z-Grenzwerte(z. B. -2 bis 2 m).
- Geben Sie die Erfassungszeit und die Abtastrate ein (z.B. alle 15-30 s für bis zu 1 h).
- Initiieren Sie die Erfassung.
6. Verarbeitung, Datenanalyse zur Erfassung von Tracking- und Colokalisierungsinformationen und Vorbereitung von Videodateien
- Bildverarbeitung
- Download, Entpacken und Open Icy, eine offene Community-Plattform für Bioimage-Informatik (http://icy.bioimageanalysis.org/)
- Öffnen Sie den XYZCt-Stack, der in Schritt 5 erworben wurde: Bild/Sequenz >Datei>Öffnen.
- Konvertieren Sie Stack in ImageJ: ImageJ> Tools> Konvertieren in IJ, haben den abgetrennten Modus EIN.
- Machen Sie einen Substack (eine Auswahl von Z-Schritten und Zeitpunkten, die weiter analysiert werden sollen): ImageJ>Image>Stacks>Tools>Substack erstellen...; wählen Sie die gewünschten Kanäle, Z-Schritte und Zeitpunkte aus.
- Substack als TIFF-Datei speichern: ImageJ>Datei>Speichern unter>Tiff...; verwenden Sie diese Datei für nachfolgende Schritte.
- Geteilte Kanäle: ImageJ>Bild>Farbe>Kanäle teilen.
- Subtrahieren Sie den Hintergrund entweder mit einem Hintergrundstapel: ImageJ>Process>Image Calculator..., oder mit der Option Rolling Ball: ImageJ>Process>Hintergrund subtrahieren..., wählen Sie den Rolling Ball Radius aus. Zeigen Sie eine Vorschau des Bildes für den ausgewählten Radius an, bevor Sie "Akzeptieren" auswählen.
Hinweis: Hintergrundsignal wird hauptsächlich durch unsachgemäßes Abschrecken des Flurorophors entstehen. Das Signal:Hintergrundverhältnis (S:B) wird häufig als Indikator für die "Helligkeit" eines MB verwendet und anhand von In-vitro-Hybridisierungsexperimenten des MB- und DNA-Zieloligonukleotids gemessen. Beispielsweise verfügen die MBs osk1236 und osk2216 über ein S:B von 81 USD bzw. 120 USD. - Anpassen der Helligkeit und des Kontrasts für jeden Kanal: ImageJ>Bild>Anpassen>Helligkeit/Kontrast, wählen Sie Anwenden aus.
- Speichern Sie jeden Kanal als separate TIFF-Datei: ImageJ>Datei>Speichern als>Tiff....
- Zusammenführen der beiden Kanäle: ImageJ>Image>Farbe>Kanäle zusammenführen...; wählen Sie die Kanäle aus. Speichern Sie den neuen Stack als neue TIFF-Datei (siehe Schritt 6.1.8).
- Spot-Erkennung und -Tracking
- Konvertieren Sie zurück zu Icy: ImageJ>Tools>Konvertieren in Eis.
- Eine Maßstabsleiste wird bei der Konvertierung in Icy automatisch auf den Stapel überlagert, wenn das Scale-Bar-Plugin installiert ist [Suche mit Plugins>Setup>Online-Plugin]. Bearbeiten Sie bei Bedarf die Maßstabsleiste über das Inspektorfenster (rechte Seite des Bildschirms)>Layer-Registerkarte>Name>Scale-Leiste.
- Deaktivieren/Deaktivieren Sie das "Auge"-Symbol für die Maßstabsleiste aus der Registerkarte Layer>Name, um die Maßstabsleiste aus dem ursprünglichen Stapel zu entfernen. Es kann auf dem letzten Stapel reaktiviert werden.
- Speichern Sie den neu verarbeiteten Stapel, indem Sie einen Screenshot mit dem Symbol "Kamera" aus der Menüleiste des Bildfensters, "Screenshot der aktuellen Ansicht erstellen" und Datei>Speichern als>Tiff....
- Bestimmen Sie die Punktempfindlichkeit, wenn die Spotempfindlichkeitsparameter bereits ermittelt wurden, bewegen Sie sich auf Schritt 6.2.7.
- Flecken erkennen: Wählen Sie das Fenster mit dem zu analysierenden Bild oder Stapel, Detection&Tracking>Detection>Spot Detector aus, und füllen Sie die Einstellungen-Parameter aus:
- Wählen Sie für Eingabe "currentSequenceInputDetection" (Standard).
- Wählen Sie für die Vorverarbeitung "Kanal 0" (Standard) oder den gewünschten Kanal aus, indem Sie die Zahl im Inspektorfenster>Sequenz-Registerkarte querverweisen.
- Wählen Sie für Detector "Hellfleck über dunklem Hintergrund erkennen"; verwenden Sie "Die Verwendung von 2D-Wellenfür 3D erzwingen" nur, wenn nicht genügend Z-Slices in den Stapeln vorhanden sind, um die Analyse durchzuführen. Wählen Sie "Scale(s)" und "Sensitivity" für jede Skala (weitere Skalen für größere Flecken hinzufügen). Die Skala und Empfindlichkeit (je größer die Zahl, desto empfindlicher ist die Erkennung, maximal 140 wird von Icy vorgeschlagen) sind Versuchs- und Fehlervariablen, die anschließend visuell überprüft und entschieden werden müssen.
- Verwenden Sie für die Interessenregion "ROIfromSequence" (Standard).
- Verwenden Sie zum Filtern "NoFiltering" (Standard), oder wählen Sie "SizeFiltering", um den "Bereich der akzeptierten Objekte (in Pixel)" zu definieren.
- Ausgabe: Wählen Sie XLS- oder XML-Ausgabeeinstellung (wählen Sie xml-Format, wenn Sie 2007 MS Excel oder früher verwenden und es gibt >65.000 Spots). Wenn die Spotdetektorergebnisse für die Tracking-Analyse verwendet werden, wählen Sie auch "Export nach SwimmingPool".
- Wiederholen Sie die Erkennung von Flecken mit verschiedenen Skalierungs-/Empfindlichkeitswerten, bis alle oder die meisten Spots erkannt werden. Zeichnen Sie alle endgültigen Parameter auf.
- Wiederholen Sie für die Colokalisierungsanalyse die Spoterkennung für den anderen Kanal.
- Um Spots zu verfolgen, wählen Sie Detection&Tracking>Tracking>Spot Tracking>Spot Detector mit Parametern aus Schritt 6.2.6. ausführen, oder verwenden Sie das Pulldown-Menü "Erkennungsergebnisse hier auswählen", um ein vorhandenes Dataset auszuwählen (halten Sie dazu das Spot-Detektor-Fenster ab Schritt 6.2.5 geöffnet). Drücken Sie die Taste "Parameter schätzen" und wählen Sie die gewünschte Zielbewegung im Popup-Fenster Parameter-Schätzung aus (z. B. "ist diffusiv und gerichtet"). Drücken Sie die Taste "Tracking ausführen".
- Wiederholen Sie die Spoterkennung und -verfolgung für andere Kanäle beim Nachverfolgen von Spots von Mehrkanal-Stacks, indem Sie den Schritten 6.2.6 und 6.2.7 folgen, beginnend mit dem Stapel, der aus Schritt 6.2.7 generiert wird.
- Um Tracks zu visualisieren, wählen Sie Detection&Tracking>Tracking>Track Manager – dieses Fenster wird nach Abschluss eines Tracking-Laufs automatisch geöffnet. Wählen Sie unter "Color Track Processor" "Aktivieren" aus, und wählen Sie die gewünschte Farbdarstellung für die Spuren aus. Auf relevante Trackprozessoren kann über den "Track Processor hinzufügen..." zugegriffen werden. Pulldown-Menü (z. B. "Track Processor Time Clip" auswählen, das Fenster "Track Clipper" aktivieren und die gewünschte Anzahl von Erkennungen auswählen, die vor und nach dem aktuellen Zeitpunkt angezeigt werden sollen.)
- Speichern von Tracks-Informationen als XML-Spurdatei: Erkennung&Tracking>Tracking>Track Manager>Datei>Speichern unter....
- Speichern Sie Ergebnisse, indem Sie einen Screenshot mit dem "Kamera"-Symbol aus der Menüleiste des Bildfensters "Erstellen Sie einen Screenshot der aktuellen Ansicht" erstellen. Screenshots können mit den erkannten Spots und/oder den Spuren einfach durch Aktivieren/Deaktivieren des entsprechenden Augensymbols im Inspektorfenster>Layer-Tab>Name>Overlay-Wrapper aufgenommen werden.
- Installieren Sie das TimeStamp Overlay Plugin: Plugins>Setup>Online-Plugin>TimeStamp Overlay>Installieren.
- Timestamp hinzufügen: Plugins>TimeStamp Overlay (Neu). Befolgen Sie die Anweisungen im Popup-Fenster (untere rechte Ecke des Bildschirms) für Anweisungen zum Platzieren und Formatieren des Zeitstempels. Das Zeitintervall kann im Inspektorfenster hinzugefügt/geändert werden>Sequenz-Registerkarte>Sequenzeigenschaften>Bearbeiten.
- Speichern Sie die Ergebnisse, indem Sie einen weiteren Screenshot erstellen. Bild als 1) Tiff-Format und 2) als AVI-Format speichern; für das AVI-Format zuerst in RGB-Rendering konvertieren (Bild/Sequenz>Rendern>RGB-Bild).
- Drehen Sie das Bild in die gewünschte Ausrichtung: Inspektorfenster>Registerkarte Sequenz>Canvas>Rotation.
- Speichern Sie gedrehtes Bild mit "Erstellen Sie einen Screenshot der aktuellen Ansicht". Stellen Sie sicher, dass das "Auge"-Symbol für die Skalenleiste deaktiviert ist, da es sich auch mit dem Bild dreht.
- ROI auswählen und zuschneiden: Wählen Sie Region Of Interest>2D ROI>ROI-Form auswählen und dann ROI auf dem Image erstellen/zeichnen; Bild/Sequenz>Ebene (XY)>Schnelles Zuschneiden.
- Colokalisierungsanalyse
- Vorbereiten eines Colokalisierungsprotokolls; Auf der Icy-Website (http://icy.bioimageanalysis.org/protocol/List) (siehe Ergänzende Materialien) finden Sie mehrere Beispiele.
- Colokalisierungsprotokoll laden: Tools>Scripting>Protokolle>Laden und Anpassen von Parametern in den interagierenden Blöcken (z. B. im Block "Wavelet Spot Detecting" verwenden Parameter, die in Schritt 6.2.6. ermittelt werden).
- Messen Sie die Größe eines Partikels in Pixeln, bestimmen Sie den Abstand zur Kolokalisierung und geben Sie es als "Max-Entfernung" in den Block "Colocalizer" ein.
HINWEIS: Die Größe des Partikels in Pixelhängt hängt vom Erkennungssystem ab. Um die Größe zu messen, zoomen Sie in ein einzelnes Partikel, und zählen Sie manuell die Pixel, die die Breite des Signals überspannen. Durchschnittlich die Messungen aus mindestens drei Partikeln. Der maximale Abstand, der für die Kolokalisierung festgelegt werden soll, ist die Größe des Partikels in Pixeln (dies stellt die maximale Summe des Radius von zwei berührenden Partikeln dar). - Wählen Sie bei Bedarf einen oder mehrere ROIs für die Colokalisierungsanalyse aus: Region Of Interest>2D ROI>ROI-Form auswählen>ROI auf Image zeichnen.
- Zuschneiden ROI(s): Image/Sequence>Plane (XY)>Schnelle Ernte.
- Colokalisierung durchführen: Protokoll-Editor-Fenster>Ausgewählte Protokoll-Registerkarte>Ausführen. Der letzte Block im Editorfenster Protokolle enthält den Gesamtprozentsatz der Kolokalisierung basierend auf der Spoterkennung, während Informationen zu jedem Zeitpunkt im Inspektorfenster> Ausgabe-Registerkarte zu finden sind.
- Verfolgen Sie kolokalisierte und einzelne Partikel, indem Sie Schritt 6.2.7 (Track-Spots) folgen.
- Speichern, wie in Schritt 6.2.16 beschrieben.
Ergebnisse
Mit PinMolkönnen mehrere MBs für ein mRNA-Ziel ausgelegt werden (Abbildung 1B-C). Nach Der Synthese und Reinigung werden die ausgewählten MBs charakterisiert und mittels In-vitro-Analyse verglichen.
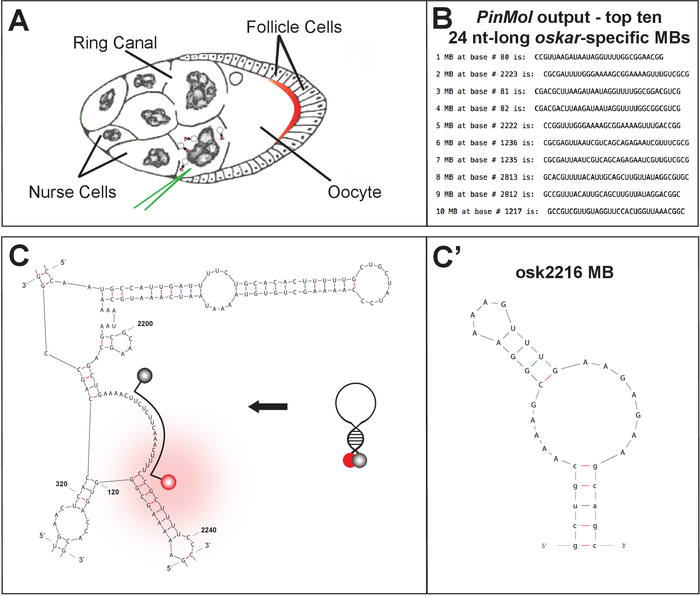
Abbildung 1: Technik und Gewebebeschreibung für die Bildgebung von endogenen mRNA...
Diskussion
Die Live-Visualisierung des endogenen mRNA-Handels in Drosophila-Eikammern beruht auf der Verwendung spezifischer, effizienter und nukleaseresistenter MBs, die nun einfach mit der PinMol-Software entworfen werden können. MBs sind spezifische Sonden, die entwickelt wurden, um eindeutige Sequenzen innerhalb einer Ziel-mRNA zu erkennen (vorzugsweise Regionen frei von sekundärer Struktur), was eine hochaufgelöste Detektion eines Transkripts ermöglicht. Die einzige Einschränkung bei der Einführung dies...
Offenlegungen
Die Autoren haben keinen Interessenkonflikt zu offenbaren.
Danksagungen
Wir danken Salvatore A.E. Marras (Public Health Research Institute Center, Rutgers University) für die Synthese, Kennzeichnung und Reinigung molekularer Leuchttürme und Daniel St Johnston (The Gurdon Institute, University of Cambridge) für die Oskar-MS2/ MCP-GFP transgene Fliegenlager. Diese Arbeit wurde durch einen National Science Foundation CAREER Award 1149738 und einen Professional Staff Congress-CUNY Award an dPB unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Spectrofluorometer | Fluoromax-4 Horiba-Jobin Yvon | n/a | Photon counting spectrofluorometer |
| Quartz cuvette | Fireflysci (former Precision Cells Inc.) | 701MFL | |
| Dumont #5 tweezer | World Precision Instruments | 501985 | Thin tweezers are very important to separate out the individual egg chambers |
| Halocarbon oil 700 | Sigma-Aldrich | H8898 | |
| Cover slip No.1 22 mm x 40 mm | VWR | 48393-048 | |
| Dissecting microscope | Leica MZ6 Leica Microsystems Inc. | n/a | |
| CO2 fruit fly anesthesia pad | Genesee Scienific | 59-114 | |
| Tris-HCL pH 7.5 | Sigma-Aldrich | 1185-53-1 | |
| Magnesium chloride | Sigma-Aldrich | 7791-18-6 | |
| NaCl | Sigma-Aldrich | 7647-14-5 | |
| Spinning disc confocal microscope | Leica DMI-4000B inverted microscope equipped with Yokogawa CSU 10 spinning disc Leica Microsystems Inc. | n/a | |
| Hamamatsu C9100-13 ImagEM EMCCD camera | Hamamatsu | n/a | |
| PatchMan NP 2 Micromanipulator | Eppendorf Inc. | 920000037 | |
| FemtoJet Microinjector | Eppendorf Inc. | 920010504 | |
| Injection needle: Femtotips II | Eppendorf Inc. | 930000043 | |
| Loading tip: 20 μL Microloader | Eppendorf Inc. | 930001007 | |
| Micro Cover glasses no. 1 or 1.5, 22 mm x 40 mm | VWR | 48393-026; 48393-172 | |
| Dry yeast | Any grocery store | n/a | |
| Computer, > 20 GB RAM | Although processing can be carried out on most computers, higher capabilities will increase the speed of the processing |
Referenzen
- Tyagi, S. Imaging intracellular RNA distribution and dynamics in living cells. Nature Methods. 6 (5), 331-338 (2009).
- Bao, G., Rhee, W. J., Tsourkas, A. Fluorescent probes for live-cell RNA detection. Annual Reviews of Biomedical Engineering. 11, 25-47 (2009).
- Mannack, L. V., Eising, S., Rentmeister, A. Current techniques for visualizing RNA in cells. F1000Research. 5, (2016).
- Larson, D. R., Zenklusen, D., Wu, B., Chao, J. A., Singer, R. H. Real-time observation of transcription initiation and elongation on an endogenous yeast gene. Science. 332 (6028), 475-478 (2011).
- Bertrand, E., et al. Localization of ASH1 mRNA particles in living yeast. Molecular Cell. 2 (4), 437-445 (1998).
- Garcia, J. F., Parker, R. MS2 coat proteins bound to yeast mRNAs block 5' to 3' degradation and trap mRNA decay products: implications for the localization of mRNAs by MS2-MCP system. RNA. 21 (8), 1393-1395 (2015).
- Bratu, D. P. Molecular beacons: Fluorescent probes for detection of endogenous mRNAs in living cells. Methods in Molecular Biology. 319, 1-14 (2006).
- Bratu, D. P., Cha, B. J., Mhlanga, M. M., Kramer, F. R., Tyagi, S. Visualizing the distribution and transport of mRNAs in living cells. Proceedings of the National Academy of Sciences of the Unites States of America. 100 (23), 13308-13313 (2003).
- Tyagi, S., Kramer, F. R. Molecular beacons: probes that fluoresce upon hybridization. Nature Biotechnology. 14 (3), 303-308 (1996).
- Chen, M., et al. A molecular beacon-based approach for live-cell imaging of RNA transcripts with minimal target engineering at the single-molecule level. Scientific Reports. 7 (1), 1550 (2017).
- Liu, Y., et al. Multiplex detection of microRNAs by combining molecular beacon probes with T7 exonuclease-assisted cyclic amplification reaction. Analytical and Bioanalytical Chemistry. 409 (1), 107-114 (2017).
- Baker, M. B., Bao, G., Searles, C. D. In vitro quantification of specific microRNA using molecular beacons. Nucleic Acids Research. 40 (2), e13 (2012).
- Ko, H. Y., et al. A color-tunable molecular beacon to sense miRNA-9 expression during neurogenesis. Scientific Reports. 4, 4626 (2014).
- Vet, J. A., et al. Multiplex detection of four pathogenic retroviruses using molecular beacons. Proceedings of the National Academy of Sciences of the Unites States of America. 96 (11), 6394-6399 (1999).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Methods in Molecular Biology. 429, 209-224 (2008).
- Li, W. M., Chan, C. M., Miller, A. L., Lee, C. H. Dual Functional Roles of Molecular Beacon as a MicroRNA Detector and Inhibitor. Journal of Biological Chemistry. 292 (9), 3568-3580 (2017).
- Kuang, T., Chang, L., Peng, X., Hu, X., Gallego-Perez, D. Molecular Beacon Nano-Sensors for Probing Living Cancer Cells. Trends in Biotechnology. 35 (4), 347-359 (2017).
- Ban, K., et al. Non-genetic Purification of Ventricular Cardiomyocytes from Differentiating Embryonic Stem Cells through Molecular Beacons Targeting IRX-4. Stem Cell Reports. 5 (6), 1239-1249 (2015).
- Hadjinicolaou, A. V., Demetriou, V. L., Emmanuel, M. A., Kakoyiannis, C. K., Kostrikis, L. G. Molecular beacon-based real-time PCR detection of primary isolates of Salmonella Typhimurium and Salmonella Enteritidis in environmental and clinical samples. BMC Microbiology. 9, 97 (2009).
- McLaughlin, J. M., Bratu, D. P. Drosophila melanogaster Oogenesis: An Overview. Methods in Molecular Biology. 1328, 1-20 (2015).
- Bastock, R., St Johnston, D. Drosophila oogenesis. Current Biology. 18 (23), R1082-R1087 (2008).
- Rongo, C., Gavis, E. R., Lehmann, R. Localization of oskar RNA regulates oskar translation and requires Oskar protein. Development. 121 (9), 2737-2746 (1995).
- Mhlanga, M. M., et al. In vivo colocalisation of oskar mRNA and trans-acting proteins revealed by quantitative imaging of the Drosophila oocyte. PLoS One. 4 (7), e6241 (2009).
- Bayer, L. V., Omar, O. S., Bratu, D. P., Catrina, I. E. PinMol: Python application for designing molecular beacons for live cell imaging of endogenous mRNAs. bioRxiv. , (2018).
- Marras, S. A., Kramer, F. R., Tyagi, S. Efficiencies of fluorescence resonance energy transfer and contact-mediated quenching in oligonucleotide probes. Nucleic Acids Research. 30 (21), e122 (2002).
- Bratu, D. P., Catrina, I. E., Marras, S. A. Tiny molecular beacons for in vivo mRNA detection. Methods in Molecular Biology. 714, 141-157 (2011).
- Dean, D. A. Preparation (pulling) of needles for gene delivery by microinjection. Cold Spring Harbor. 2006 (7), (2006).
- Alami, N. H., et al. Axonal transport of TDP-43 mRNA granules is impaired by ALS-causing mutations. Neuron. 81 (3), 536-543 (2014).
- Jackson, S. R., et al. Applications of Hairpin DNA-Functionalized Gold Nanoparticles for Imaging mRNA in Living Cells. Methods in Enzymology. 572, 87-103 (2016).
- Zimyanin, V. L., et al. In vivo imaging of oskar mRNA transport reveals the mechanism of posterior localization. Cell. 134 (5), 843-853 (2008).
- Catrina, I. E., Marras, S. A., Bratu, D. P. Tiny molecular beacons: LNA/2'-O-methyl RNA chimeric probes for imaging dynamic mRNA processes in living cells. ACS Chemical Biology. 7 (9), 1586-1595 (2012).
- Chen, A. K., Behlke, M. A., Tsourkas, A. Efficient cytosolic delivery of molecular beacon conjugates and flow cytometric analysis of target RNA. Nucleic Acids Research. 36 (12), e69 (2008).
- Nitin, N., Santangelo, P. J., Kim, G., Nie, S., Bao, G. Peptide-linked molecular beacons for efficient delivery and rapid mRNA detection in living cells. Nucleic Acids Research. 32 (6), e58 (2004).
- Chen, A. K., Behlke, M. A., Tsourkas, A. Avoiding false-positive signals with nuclease-vulnerable molecular beacons in single living cells. Nucleic Acids Research. 35 (16), e105 (2007).
- Bevilacqua, P. C., Ritchey, L. E., Su, Z., Assmann, S. M. Genome-Wide Analysis of RNA Secondary Structure. Annual Review of Genetics. 50, 235-266 (2016).
- Mhlanga, M. M., Vargas, D. Y., Fung, C. W., Kramer, F. R., Tyagi, S. tRNA-linked molecular beacons for imaging mRNAs in the cytoplasm of living cells. Nucleic Acids Research. 33 (6), 1902-1912 (2005).
- Eliceiri, K. W., et al. Biological imaging software tools. Nature Methods. 9 (7), 697-710 (2012).
- Bolte, S., Cordelieres, F. P. A guided tour into subcellular colocalization analysis in light microscopy. Journal of Microscopy. 224 (Pt 3), 213-232 (2006).
- Trcek, T., et al. Drosophila germ granules are structured and contain homotypic mRNA clusters. Nature Commununications. 6, 7962 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten