A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
חזותי ומעקב אחר mRNAs בשידור חי דרוזוסופילה מלאנוגסטר צ'יימברס
In This Article
Summary
כאן, אנו מציגים פרוטוקול עבור ויזואליזציה, זיהוי, ניתוח ומעקב של סחר האנטי-מסוגגני mRNA בחיים Drosophila ילה מלאנוסטר תא ביצה באמצעות משואות מולקולריות, מיקרוסקופיה דיסק מסתובב, וניתוח קוד פתוח תוכנה.
Abstract
שיטות הדמיה מבוססות-פלואורסצנטית, בשילוב עם התפתחויות במיקרוסקופיה קלה, יש מהפכה כיצד ביולוגים תא לנהל לימודי הדמיה של תאים חיים. שיטות זיהוי RNAs התרחבו מאוד מאז לימודי הזרע מקושרים לוקליזציה mRNA ספציפי לאתר לתקנות ביטוי גנים. תהליכי mRNA דינאמי יכול כעת להיות מדמיין באמצעות גישות המאתרות Mrna, בשילוב עם set-ups מיקרוסקופ כי הם מהירים מספיק כדי ללכוד את טווח דינמי של התנהגות מולקולרית. מסוגל לגילוי ישיר של תעתיקים. של התאים החיים משואות מולקולריות הן בצורת ראש, מנותת פנימי, יחיד-נוקלאוטיד להפלות בדיקה חומצות גרעין, אשר זרוח, רק על הכלאה לרצף יעד ייחודי. כאשר מצמידים עם מיקרוסקופ פלואורסצנטית מתקדם הדמיה ברזולוציה גבוהה, הם מאפשרים אחד לבצע מעקב מרחבי וזמני של תנועה תאיים של mRNAs. למרות טכנולוגיה זו היא השיטה היחידה המסוגלת לזהות תעתיקים אנדוגניים, ביולוגים תא עדיין לא חיבק באופן מלא בטכנולוגיה זו בשל קשיים בעיצוב בדיקות כאלה עבור הדמיה תא חי. יישום תוכנה חדש, Pinmol, מאפשר עיצוב משופר ומהיר של הגששים המתאימים ביותר לhybridize ביעילות אזורים היעד mrna בתוך תא חי. בנוסף, ברזולוציה גבוהה, רכישת תמונה בזמן אמת, הנוכחי, ניתוח תמונה קוד פתוח תוכנה לאפשר פלט נתונים מעודן, המוביל הערכה עדינה יותר של המורכבות הבסיסית של תהליכים דינמיים המעורבים במחזור החיים של mRNA.
כאן אנו מציגים פרוטוקול מקיף לעיצוב ושליחת משואות מולקולריות לתוך מלאנוזופרסטר תאי ביצה. הזיהוי וההדמיה הישירה והייחודית ביותר של מיקרו-האמדוגני מבוצעת באמצעות מיקרוסקופ מסתובב הדיסק. נתוני הדמיה מעובדים ומנותח באמצעות זיהוי אובייקטים ומעקב בתוכנה קרח כדי לקבל פרטים על התנועה הדינמית של mRNAs, אשר מועברים ומקומי לאזורים מיוחדים בתוך oocyte.
Introduction
ביולוגיה של התא מחקרים להמחיש אירועים דינמיים עם רזולוציה מרחבית וזמני התאפשרה על ידי פיתוח של טכניקות הדמיה של תאים בעלי מבוססי-זריחה חי. כיום, ב vivo mrna הדמיה מושגת באמצעות טכנולוגיות המבוססות על rna aptamer-חלבון אינטראקציות, רנ א aptamer אלף המושרה של צבעים אורגניים וחומצה גרעין בדיקה ריפוי1,2, 3. כולם מציעים שיעור גבוה, רגישות ויחס אות לרקע. עם זאת, הגישות במרכז RNA aptamer-ממורכז דורשות מניפולציה גנטית נרחבת, שם מתוכנן לבטא רנ א עם מוטיבים מבניים מלאכותיים הדרושים עבור חלבון או מחייב צבע אורגני. לדוגמה, מערכת MS2/MCP מחייבת את הביטוי המשותף של הרשת המביעה מבנה RNA המכיל מספר חזרות דו-מושביים של רצף הכריכה עבור החלבון בקטביאג MS2 מעיל (MCP), והצפנה נוספת של חלבון פלורסנט . התמזגו ל-MCP4,5 התוספת של מוטיבים מבניים כגון ל-RNA, יחד עם חלבון מתויג מגושם, העלה חששות כי תהליכי RNA הילידים עשויים להיות מושפעים6. טכנולוגיה המטפלת בדאגה זו ומציעה יתרונות ייחודיים נוספים היא הגישה המבוססת על חומצות גרעין, משואות מולקולריות (MBs). MBs לאפשר זיהוי מולטידוגני של החומרים האנדוגניים, אפליה של נוקלאוטיד יחיד וריאציות, ו קינטיקה מהירה של הכלאה עם mrnas המטרה7,8. ב-MBs משתנים בדיקה שנותרו בקיפול מכופף לפני שעברו שינוי של fluorogenic ברגע שהם hybridize ליעדים שלהם (איור 1C)9. מספר קבוצות הצליחו להשתמש ב-MBs כדי לזהות שניהם rnas שאינם קידוד (מיקרורונאס ו-lncrnas)10,11,12,13, RNA רטרווירוסים14 ו-DNA דינאמי-חלבון אינטראקציות15. הם כבר מועסק בהצלחה עבור הדמיה באורגניזמים שונים ורקמות, כגון עוברי דג העוברים16, נוירונים13, רקמות הגידול17, ההבחנה הקרדיוציטים18, סלמונלה . בן 19
כאן אנו מתארים את העיצוב, המסירה ואת הגישה לגילוי עבור mRNAs האנדוגניים בחיים D. מלאנוגסטר חדרי ביצה בשילוב עם הגדרת מיקרוסקופ מספיק מהר כדי ללכוד את טווח דינמי של הובלה מולקולרית פעיל. תא ביצת D. melanogaster שימש מערכת המודל האידיאלי מרובת תאים עבור מגוון רחב של מחקרים התפתחותיים, מ-germline מוקדם גזע התא וביטוי גנים אימהית לדור של הגוף מגזרי20, . עשריםואחד תאי ביצה מבודדים בקלות, גדול שקוף, ומסוגל לעמוד שעות של ניתוח vivo ex , מה שהופך אותם קל מאוד ניסויים הדמיה. עבודה רבה התמקדה לוקליזציה אסימטרית של תעתיקים אימהי לאזורים בדידים משנה דיסקרטית לפני שהוא מתורגם באופן פעיל. במיוחד, לוקליזציה של אוסקר mrna ואת התרגום העוקב שלה בקוטב האחורי של oocyte חייב להתרחש בצורה מוסדר היטב כדי למנוע העובר הקטלני בלאודל היום22. אוסקר mrna משועתק בתאים 15 germline, שנקרא תאים האחות, ומועברים באופן פעיל דרך גשרים cytoplasmic, קרא תעלות הטבעת, לתוך oocyte, התא germline הופך ביצה בוגרת מופרית בסופו של דבר (איור 1a ). כמות ניכרת של מידע זמין כבר לגבי גיוס דינמי וחילופי חלבונים של גורמי חלבון מ- אוסקר mrnp, יחד עם נסיעות הארוך שלה תאיים שלה, להפוך את אוסקר למועמד המועדף ללמוד תהליכים רבים של מחזור החיים mRNA. MBs כבר אינסטרומנטלי לחשוף פרטים על תהליך הלוקליזציה של mRNA ופענוח הרגולציה והתפקוד של גורמי חלבון השולטים בתחבורה mRNA במהלך Drosophila ילה oogenesis. במיוחד, על ידי מיקרו הזרקת MBs לתוך התאים האחות וביצוע ניסויים הדמיה תא חי, המעקב אחר mrnas מעקב אפשרי8,23.
מפת הדרכים המוצגת כאן מציעה את השלבים של תהליך שלם, החל בביצוע ניסוי הדמיה של תא חי באמצעות MBs, רכישת נתוני הדמיה, כדי לבצע ניתוח נתונים כדי לעקוב אחר mRNA אנדוגניים בסביבה הסלולרית הטבעית שלה. השלבים יכולים להיות שונה וממוטב נוסף כדי לענות על הצרכים של חוקרים לעבוד עם רקמות אחרות/סוגי תאים בתוך הגדרת המעבדה שלהם.
Protocol
1. עיצוב של MBs לדימות תאים חיים
-
מקפלים את היעד ברצף RNA כדי לחזות את המבנה המשני של היעד mRNA באמצעות "הטופס RNA" מהשרת mfold (http://unafold.rna.albany.edu/?q=mfold/RNA-Folding-Form).
- הדבק/העלה את רצף היעד בפורמט FASTA, בחר 5 או 10% משנה את הצורה (מבנים עם אנרגיה חופשית של קיפול בתוך 5 או 10% של ערך MFE, בהתאמה), ולהתאים את המספר המירבי של foldings מחושבים בהתאם (למשל גדול עבור 10% תת-אופטיאליות).
הערה: הכללה של מבנים משניים אופטימליים משנה בעת עיצוב MBs מאפשר זיהוי של אזורים בתוך mRNA היעד שעשוי להיות גמיש יותר או נוקשה יותר מכפי שחזוי עבור מבנה אנרגיה חופשית מינימלית (MFE) בלבד, אשר משפרת את עיצוב כולל של MBs מתאים לדימות תאים חיים. - בחר "עבודה מיידית" עבור מטרות mRNA של 800 נוקלאוטידים (nt), או "משימת אצווה" עבור אורכי mRNA בין 801 ו 8,000 nt. שמור את הקובץ "הרוזן ss" כקובץ טקסט פשוט.
- הדבק/העלה את רצף היעד בפורמט FASTA, בחר 5 או 10% משנה את הצורה (מבנים עם אנרגיה חופשית של קיפול בתוך 5 או 10% של ערך MFE, בהתאמה), ולהתאים את המספר המירבי של foldings מחושבים בהתאם (למשל גדול עבור 10% תת-אופטיאליות).
-
השתמש בקובץ "ss-count" שהתקבל בשלב 1.1 כקלט לתוכנית הפינמול (https://bratulab.wordpress.com/software/) עם הפרמטרים הרצויים, כדי לעצב מספר MBs עבור יעד mrna (ראה מדריכים המתארים שימוש בתוכנית הפינמול 24 ב https://bratulab.wordpress.com/tutorial-pinmol-mac/).
- לקבוע את הספציפיות של MBs שנבחרו על ידי ביצוע ניתוח הפיצוץ: השתמש "blastn" עם מסד הנתונים המתאים (למשל עבור אוסקר Mrna-MBs ספציפי להשתמש במסד הנתונים "מרפזה-rna" ואת Drosophila ילה melanogaster האורגניזם).
- זיהוי כל ביטוי ספציפי לרקמה של היעד mRNA (למשל עבור אוסקר Mrna flybase ≫ ביטוי תפוקה גבוהה נתונים ≫ flybase אנטומיה מיקרו מערך או מודדנקוד אנטומיה RNA-Seq; http://flybase.org/reports/FBgn0003015) ולהשוות עם כל כניסות חיוביות הפיצוץ. סלק בדיקה שתציג > 50% בעלי הומולוגיה עם mRNAs אחרים אשר מבוטאים גם ברקמה/תא הריבית.
- בחר את ה-פלואורואופפור וזוג הקוכר המתאימים לקבוצת המיקרוסקופיה הזמינה לביצוע הדמיה של תאים חיים (לדוגמה Cy5/BHQ2)25.
2. סינתזה של MB, טיהור ואפיון
- השתמש בסינתזה בתוך הבית ובטיהור כמתואר בעבר7, או שירותים מספקים מסחריים, כדי לסנתז ולטהר אחד עד חמש MBs (ראה לעיל הערה), באמצעות סכימת התיוג הבאה: [5] (פלואורואופפור)-(2 '-O) -מתיל מגה-בתים)-(קונצ'ר) 3 ']. לטהר את השימוש בשיטת הייעוץ בשלב ההפוך, בבית או בשימוש בשירותי הספק המסחרי.
הערה: הזרחנות המשמשות לסינתזה בדיקה אוטומטית חייבת להיות השינוי של 2 '-O-מתיל. ניתן גם להשתמש בצ של מתחלפים של חומצה בגרעין נעול לסירוגין (LNA) ו 2 '-O-מתיל שינויים כדי להגדיל את היציבות של היברידית בין MB קצר יותר mrna היעד שלה26. - לסנתז דנ א של מחלת ה-DNA התואמים את הרצף של אזור ה-RNA המיועד ולכן משלימים את אזור הבדיקה של MBs, לשימוש באפיון חוץ גופית (ראה שלבים 2.3 עד 2.5; מעל הערה). למקסם את הכלאה של המגה-בתים עם ה-DNA-olig, מטרה היעד לחקות, על ידי כלילת בכל קצה של מטרה ה-DNA ארבעה נוקלאוטידים נוספים, כפי שנמצא ברצף mRNA היעד.
הערה: אפיון קפדני יותר של יעילות המגה-בתים לזהות את הרצף המיועד ניתן לבצע באמצעות מטרות RNA מסונתז על-ידי מבחנה במקום של DNA משלימה olig, משלימים8. - בצע הדציה תרמית של ה-MB בלבד, למדוד את טמפרטורת ההיתוך שלה (Tm), ולאשר כי ה-MB מניח את הצורה הרצויה מאוד בטמפרטורה פיזיולוגית. הבחנו בערכי Tm בין 60 ל-90 ° c.
- בצע דנטורציה תרמית של ה-MB בנוכחות ה-DNA olig, היעד ולמדוד את MB: DNA היעד של היברידית Tm, כפי שתוארה בעבר7. Tm בין 55 ו 60 ° c רצוי עבור MB: DNA היברידית.
- לבצע תגובות היברידיות מחוץ לבית עם היעד המתאים DNA olig, ולקבוע את היעילות של MB: היווצרות DNA היברידית בטמפרטורה פיזיולוגית, כפי שתוארה בעבר7. הכלאה מהירה קינטיקה עם היעד DNA לחקות רצוי, אולם MBs כי לא להראות יעילות הכלאה גבוהה עם מטרות DNA יכול להיות ביצועים טובים יותר עם mRNA היעד ב מבחנה ו/או vivo.
3. חיתוך והכנת תאי ביצה בודדים עבור מיקרוהזרקה
- להאכיל החדש בקעו, נשים מזדווג עבור 2-3 ימים עם להדביק שמרים טריים.
- באמצעות מלקחיים משובחים (דומונט5), העבירו 1-2 נקבות לתוך טיפת שמן הלוקרבון 700 על כיסוי זכוכית.
- באמצעות זוג מלקחיים, כוון את הזבוב עם הצד האחד למעלה מתחת לstereomicroscope. לנתח את הבטן הנשית על ידי יצירת חתך קטן בקצה האחורי ולסחוט בעדינות את זוג השחלות לתוך השמן.
- לחקור את השחלות על טיפת שמן על שמיכות חדש. בעדינות להחזיק שחלה אחת עם פינצטה אחד תוך צובט את השלבים הצעירים של ovariole עם הטוויצר השני. אוסקר mrna מקומי באופן פעיל לאחר באמצע oogenesis (שלבים > 7), ותאי ביצה צעירים (שלבים < 7) קשה יותר להזריק ולא לשרוד זמן רב. גררו באיטיות על שובר הכיסוי (עם תנועה כלפי מטה) עד שיחידות האויונות או תאי הביצה מבודדים ומיושרים אנכית. לתאי ביצה נפרדים נוספים על ידי הפרדת שלבים בלתי רצויים משרשרת הביצים של הovariole.
הערה: הקפידו שתאי ביצה שיציקו באופן אינדיבידואלי אינם צפים בשמן, ושהם נצמדים לכריכה המכסה. הדבר חשוב הן עבור מיקרו-הזרקה מוצלחת ורכישת תמונה.
4. הזרקה של MBs לתאי האחות של צ'יימברס ביצים
- להכין את הפתרון MB, באמצעות משואת מולקולרי אחד (g. osk2216Cy5), או שילוב של שתי MBs היעד mRNAs שונים ואשר מסומנים באמצעות fluorophores נפרדות (למשל osk2216Cy5 ו drongo1111Cy3). השתמש בריכוז של 200-300 ng/μL כל MB ב HybBuffer (50 mM טריס-HCl-pH 7.5, 1.5 mM MgCl2 ו-100 מ"מ הנאל). עבור קוקטייל של ארבע MBs עם תווית של אותו fluorophore כי הם מיקוד אותו mRNA ב 200 ng/μL כל אחד ב HybBuffer (למשל, osk82, osk1236, osk2216). ספין למטה את הפתרון MB מיד לפני טעינת המחט למיקרו הזרקה.
- בחר את המטרה. מטרת שמן 40x מומלצת למציאת חדר ביצים מתאים ולביצוע מיקרוהזרקה.
- הר את הסרבל עם חדר ביצה גזור על המיקרוסקופ הבמה. העלה את המטרה במיקום המיקוד ולזהות תא ביצה בשלב התפתחותי אמצע עד מאוחר, כי הוא מכוון כראוי למיקרו הזרקה (כלומר, עם ציר AàP בניצב לקצה המחט כדי לאפשר זריקה קלה בתוך אחות התא האבוציט של התאים.
- טען מחט (מסחרי או מוכן בבית27) עם פתרון ~ 1 ΜL MB (ראה שלב 4.1) ולחבר אותו למיקרו מזרק. לזריקות מיקרו בתאי ביצה D. melanogaster , האוריינט המחט (ראה לוח חומרים) בזווית < 45 ° לשלב המיקרוסקופ (למשל 30 °) כדי למנוע מספר תאים אחות.
- הגדרת מזרק עם לחץ ההזרקה של 500-1000 hPa והלחץ פיצוי של 100-250 hPa (ראה טבלת חומרים).
- הזיזו לאט את הבמה כדי להביא את שדה הראייה לאזור של השמן הריק של תאי הביצה.
- באמצעות ג'ויסטיק המיקרומניפולציה, הנמך בעדינות את המחט לתוך טיפת השמן והבא את קצהו לפוקוס לעבר הפריפריה של שדה הראייה.
- לבצע ' נקי ' פונקציה כדי להסיר את האוויר מקצה המחט כדי להבטיח כי יש זרימה מן המחט.
- הביאו את המחט לתנוחת הבית והתרכזו בחדר הביצה כדי להיות מוזרקים, ואז להביא את המחט בחזרה לפוקוס ולמקם אותו ליד הקצה של תא ביצה.
- לבצע התאמה קנס של המטרה של Z-מיקום כזה הממברנה המפרידים את התאים זקיק מתאי האחות הוא בפוקוס.
- הכנס את המחט לתא אחות ולבצע הזרקה עבור 2-5 s.
- להסיר בעדינות את המחט ולמשוך אותו לתנוחת הבית.
- שנה את המטרה להגדלה הרצויה של רכישת התמונה (60-63 x או 100x), התמקד בחדר הביצה והתחל ברכישה.
5. רכישת נתונים באמצעות דיסק מסתובב ההתקנה מיקרוסקופ מיקוד
הערה: ראה טבלת חומרים עבור הכיוונון הספציפי שלנו.
- הגדר פרוטוקול רכישה כדי להקליט מחסנית XYZCt של 8-16-bit תמונות (XYZ = נפח, C = ערוץ, t = time).
- בחר קווי לייזר עבור הערוצים הרצויים (למשל 641 ננומטר לייזר עבור Cy5 ו 491 nm עבור GFP) ולרכוש את הערוצים ברצף: תחילה את האות הפלואורסצנטית בכל ערוץ ולאחר מכן לשנות את המיקום Z, כדי לאפשר ניתוח לוקליזציה הנכון.
- בחר בשלב z (למשל 0.3 יקרומטר) ובמגבלות Z העליונות והתחתונות (לדוגמה-2 יקרומטר עד 2 יקרומטר).
- להזין את זמן הרכישה ואת קצב הדגימה (למשל כל 15-30 s עד 1 h).
- . התחל ברכישה
6. עיבוד, ניתוח נתונים לקבלת מידע מעקב ולוקליזציה, והכנת קבצי וידאו
- עיבוד תמונה
- הורדה, אריזה ופתיחה של אייסי, פלטפורמה קהילתית פתוחה לאינפורמטיקה ביודמית (http://icy.bioimageanalysis.org/)
- פתח את מחסנית XYZCt שנרכשה בשלב 5: תמונה/רצף > קובץ > פתוח.
- המר מחסנית ל-ImageJ: ImageJ > כלים > המרה ל-IJ, יש מצב מנותק ON.
- ליצור מחסנית משנה (מבחר של מגוון של שלבים Z ונקודות זמן להיות מנותח עוד): ImageJ > תמונה > ערימות > כלים > להפוך מחסנית משנה...; בחר את הערוצים הרצויים, Z-שלבים ונקודות זמן.
- שמור מחסנית משנה כקובץ TIFF: ImageJ > קובץ > שמור כ> Tiff...; השתמש בקובץ זה עבור השלבים הבאים.
- ערוצי פיצול: ImageJ > תמונה > צבע > ערוצים מפוצלים.
- הפחת רקע באמצעות מחסנית רקע: ImageJ > תהליך > מחשבון תמונה..., או באמצעות האפשרות ' כדור מתגלגל ': ה> הליך > הפחת רקע..., בחר ברדיוס הכדור המתגלגל. הצג בתצוגה מקדימה את התמונה עבור הרדיוס שנבחר לפני בחירת "קבל".
הערה: אות הרקע עולה בעיקר מקוצ'ינג מגונה של הפלוראוריפור. האות: יחס הרקע (S:B) משמש לעתים קרובות כאינדיקטור עבור "בהירות" של מגה-בתים, והוא נמדד מתוך ניסויים היברידיזציה של מטרה במגה-בתים ושגיאות DNA. לדוגמה, MBs osk1236 ו-osk2216 יש S:B של ~ 81 ו-~ 120, בהתאמה. - כוונן את הבהירות והניגודיות עבור כל ערוץ: ImageJ > תמונה > כוונן את > בהירות/ניגודיות, בחר באפשרות החל.
- שמור כל ערוץ כקובץ TIFF נפרד: ImageJ > קובץ > שמור כ> Tiff....
- מיזוג שני הערוצים: ImageJ > תמונה > צבע > ערוצי מיזוג...; בחור את הערוצים. שמור את המחסנית החדשה כקובץ TIFF חדש (ראה step 6.1.8).
- איתור ומעקב ספוט
- להמיר בחזרה אייסי: ImageJ > כלים > להמיר קרח.
- סרגל בקנה מידה מצופה באופן אוטומטי על המחסנית בעת המרה לקרח, אם התוסף בר קנה המידה מותקן [חיפוש באמצעות תוספים > תוכנית ההתקנה > מקוונת). במקרה הצורך, ערכו את סרגל הסרגל באמצעות חלון המפקח (הצד הימני של המסך) > הכרטיסיה ' שכבה ' > שם > סרגל סרגל.
- בטלו את הסימון בסמל ' עין ' בסרגל הכרטיסיות ' שכבה > שם ' כדי להסיר את סרגל קנה המידה מהמחסנית המקורית. ניתן להפעיל אותו מופעל בערימה הסופית.
- שמור את המחסנית החדשה שעובדו על-ידי צילום מסך באמצעות הסמל "מצלמה" מתוך שורת התפריטים של חלון התמונה, צלם צילום מסך של התצוגה הנוכחית והקובץ > שמור בתור > Tiff....
- קביעת רגישות לספוט, אם פרמטרי הרגישות של הספוט כבר נקבעו למעבר אל שלב 6.2.7.
- זיהוי מקומות: בחר את החלון עם התמונה או המחסנית שיש לנתח, זיהוי & מעקב > זיהוי > גלאי ספוט ומלא את פרמטרי ההגדרות:
- כדי להזין קלט, בחר באפשרות "האיתור הנוכחי של גילוי קלט" (ברירת המחדל).
- עבור עיבוד מקדים, בחר באפשרות "ערוץ 0" (ברירת מחדל) או בערוץ הרצוי על-ידי הפניה מקושרת למספר בכרטיסיה ' חלון מפקח > רצף '.
- עבור גלאי, בחר "לזהות נקודה בהירה על רקע כהה;" להשתמש "כוח השימוש של 2D אדוות עבור 3D" רק אם אין מספיק Z-פרוסות בערימות כדי לבצע את הניתוח. בחר באפשרות "קנה מידה" ו-"רגישות" לכל קנה מידה (הוסף סולמות נוספים לנקודות גדולות יותר). קנה המידה והרגישות (ככל שהמספר יותר רגיש הוא הזיהוי, מקסימום של 140 מוצע על ידי אייסי) הם המשפט משתני שגיאה, כי יש לבדוק ויזואלית לאחר מכן החליטו על.
- עבור ' אזור העניינים ', השתמש באפשרות ' שימוש ברצף ' (ברירת המחדל).
- לסינון, השתמש באפשרות "NoFiltering" (ברירת מחדל), או בחר באפשרות "סינון שינוי גודל" כדי להגדיר את "טווח האובייקטים המקובלים (בפיקסלים)".
- פלט: בחירת XLS או הגדרת פלט XML (בחר פורמט XML בעת שימוש 2007 MS Excel או מוקדם יותר ויש > נקודות 65000). אם התוצאות של גלאי הספוט משמשות לניתוח המעקב, בחרו גם ' ייצוא לבריכת שחייה '.
- חזור על זיהוי של נקודות באמצעות ערכי קנה מידה/רגישות שונים עד לזיהוי כל הנקודות או רוב הכתמים. הקלט את כל הפרמטרים הסופיים.
- לקבלת ניתוח לוקליזציה לשפות אחרות, זיהוי נקודה חוזרת עבור הערוץ האחר.
- כדי לעקוב אחר כתמים, בחר באפשרות זיהוי & מעקב > מעקב > ספוט > הפעל את גלאי הספוט באמצעות הפרמטרים משלב 6.2.6., או השתמש בתפריט הנפתח "בחר תוצאות זיהוי כאן" כדי לבחור ערכת נתונים קיימת (לשם כך, השאר את חלון גלאי הספוט פתוח משלב 6.2.5). לחץ על כפתור "הערכת פרמטרים" ובחר את תנועת היעד הרצויה בחלון שערוך פרמטרים (למשל "הוא גם מפזרים ומכוון"). לחץ על כפתור "הפעל מעקב".
- חזור על איתור ומעקב אחר נקודות עבור ערוצים אחרים בעת מעקב אחר מקומות של ערימות רב-ערוצי, לאחר השלבים 6.2.6 ו-6.2.7, החל במחסנית שנוצרת משלב 6.2.7.
- כדי להמחיש מסלולים, בחר באפשרות זיהוי & מעקב > מעקב אחר > מנהל מעקב-חלון זה נפתח באופן אוטומטי עם סיום הפעלת המעקב. עבור "מעבד מעקב אחר צבעים", בחר "אפשר" ובחר את הייצוג הרצוי של צבע עבור הרצועות. ניתן לגשת אל מעבדי הרצועה הרלוונטיים באמצעות "הוספת מעבד מעקב..." תפריט משיכה (לדוגמה, בחר באפשרות "עקוב אחר קליפ זמן מעבד", הפעל את החלון "מעקב אחר מעבדים", ובחר את מספר הזיהוי הרצוי שיוצג לפני ואחרי נקודת הזמן הנוכחית.)
- שמור שירים מידע כקובץ מעקב XML: זיהוי & מעקב אחר > מעקב אחר > מנהל מעקב אחר > קובץ > שמירה בשם...
- להציל את התוצאות על ידי לקיחת צילום מסך באמצעות סמל "מצלמה" מתוך שורת התפריטים של חלון תמונה, "לקחת צילום מסך של השקפה הנוכחי". צילומי מסך ניתן לקחת עם מקומות שזוהו ו/או את הרצועות פשוט על ידי הפעלת/הפעלה של סמל העין המקבילה (עם) שנמצאו המפקח חלון > שכבה הכרטיסיה > שם > עטיפה כיסוי.
- התקן את התוסף לכיסוי חותמת הזמן: תוספים > תוכנית ההתקנה > מקוונת plugin > כיסוי חותמת זמן > התקן.
- הוספת חותמת זמן: תוספים > כיסוי חותמת זמן (חדש). בצע את ההוראות על החלון המוקפץ (בפינה הימנית התחתונה של המסך) לקבלת הנחיות לגבי הצבת ועיצוב חותמת הזמן. ניתן להוסיף/לשנות את מרווח הזמן בכרטיסיה רצף > > מאפייני רצף > עריכה.
- שמור תוצאות על ידי לקיחת מסך נוסף. שמור תמונה כ 1) תבנית Tiff ו-2) כתבנית AVI; לתבנית AVI המרה ראשונה לעיבוד RGB (תמונה/רצף > רינדור > תמונת RGB).
- סובב תמונה לכיוון הרצוי: חלון מפקח > כרטיסיה רצף > > סיבוב.
- שמור תמונה שסובבה על-ידי "צלם צילום מסך של התצוגה הנוכחית". הקפידו לבטל את הבחירה בסמל ' עין ' בסרגל הקנה מידה, כפי שהוא יסתובב גם עם התמונה.
- לבחור ולחתוך ROI: בחר אזור של עניין > 2D ROI > בחר הצורה ROI ולאחר מכן ליצור/לצייר ROI על התמונה; תמונה/רצף > מישור (XY) > יבול מהיר.
- ניתוח לוקליזציה
- הכן פרוטוקול לוקליזציה; מספר דוגמאות מסופקות באתר הקרח (http://icy.bioimageanalysis.org/protocol/List) (ראה חומרים משלימים).
- טען פרוטוקול התאמה לשפות אחרות: כלים > Scripting > פרוטוקולים > לטעון ולהתאים פרמטרים בבלוקי האינטראקציה (לדוגמה, ב-"זיהוי אדוה Spot" בלוק השתמש בפרמטרים שנקבעו בשלב 6.2.6.).
- למדוד את הגודל של חלקיק בפיקסלים, לקבוע את המרחק לוקליזציה ולהכניס אותו לתוך "לוקליזציה" לחסום כמו "מרחק מקסימום".
הערה: גודל החלקיק בפיקסלים תלוי במערכת האיתור. למדידת גודל, התקרבות לחלקיק בודד וספירה ידנית של הפיקסלים המתפרסים על רוחב האות. ממוצע המידות של שלושה חלקיקים לפחות. המרחק המירבי שיש להגדיר עבור התאמה לשפות אחרות הוא גודל החלקיק בפיקסלים (הדבר מייצג את הסכום המירבי של רדיוס של שני חלקיקים הנוגעים). - אם רצונך בכך, בחר ROIs אחת או יותר עבור ניתוח לוקליזציה: אזור של עניין > 2D ROI > לבחור את הצורה ROI > צייר ROI על התמונה.
- המשך חיתוך (ים): תמונה/רצף > מישור (XY) > יבול מהיר.
- ביצוע התאמה לשפות אחרות: > הכרטיסיה פרוטוקול נבחר > הפעלה. הבלוק הסופי בחלון של עורך פרוטוקולים יכיל את אחוז הלוקליזציה הכולל בהתאם לזיהוי ספוט, בעוד שמידע בכל נקודת זמן נמצא בחלון המפקח > הכרטיסיה פלט.
- מעקב אחר חלקיקים מקומיים ובודדים באמצעות שלב 6.2.7 (מעקב אחר נקודות).
- שמור כמתואר בשלב 6.2.16.
תוצאות
באמצעות Pinmol, ניתן לתכנן מספר MBs עבור יעד mrna אחד (איור 1b-C). לאחר הסינתזה והטיהור, השימוש ב-MBs הנבחר מאופיין בניתוח חוץ-גופית.
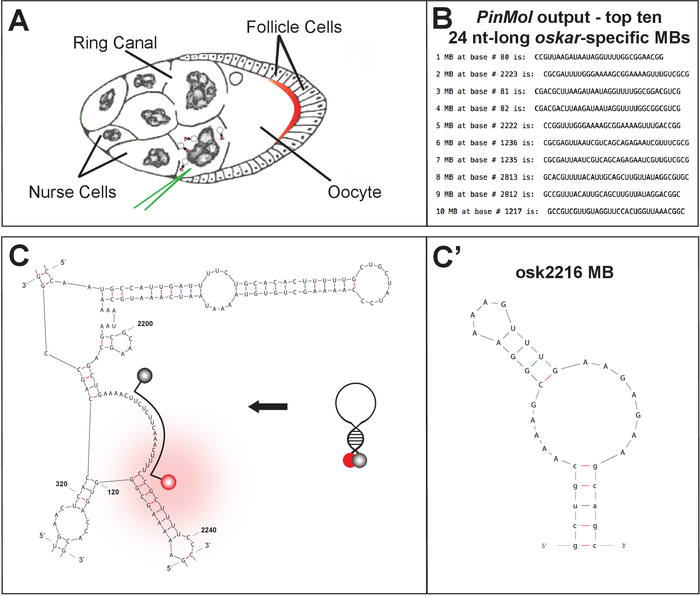
איור 1: טכניקה ותיאור הרקמה עבור הדמ?...
Discussion
ויזואליזציה בשידור חי של סחר הנשים האנסוגני ב- Drosophila ילה תאי ביצה מסתמך על שימוש ספציפי, יעיל, ו nuclease עמידים לשימוש, אשר כעת ניתן לתכנן בקלות עם תוכנת pinmol . MBs הם רגשים ספציפיים שנועדו לזהות רצפים ייחודיים בתוך mRNA היעד (רצוי אזורים ללא מבנה משני), מה שמאפשר זיהוי מאוד מוחלט של תעתיק. ?...
Disclosures
למחברים אין ניגוד אינטרסים לגלות.
Acknowledgements
אנו מודים סלווטורה A.E. Marras (מרכז מחקר בריאות הציבור, באוניברסיטת רטגרס) לסינתזה, תיוג וטיהור של משואות מולקולרית, דניאל St ג'ונסטון (מכון גורדון, אוניברסיטת קיימברידג ') עבור אוסקר-MS2/ מניות לטיסה. עבודה זו היתה נתמכת על ידי פרס לאומי של קרן המדע בשנת 1149738 ופרס צוות מקצועי לקונגרס-CUNY ל DPB.
Materials
| Name | Company | Catalog Number | Comments |
| Spectrofluorometer | Fluoromax-4 Horiba-Jobin Yvon | n/a | Photon counting spectrofluorometer |
| Quartz cuvette | Fireflysci (former Precision Cells Inc.) | 701MFL | |
| Dumont #5 tweezer | World Precision Instruments | 501985 | Thin tweezers are very important to separate out the individual egg chambers |
| Halocarbon oil 700 | Sigma-Aldrich | H8898 | |
| Cover slip No.1 22 x 40mm | VWR | 48393-048 | |
| Dissecting microscope | Leica MZ6 Leica Microsystems Inc. | n/a | |
| CO2 fruit fly anesthesia pad | Genesee Scienific | 59-114 | |
| Tris-HCL pH7.5 | Sigma-Aldrich | 1185-53-1 | |
| Magnesium chloride | Sigma-Aldrich | 7791-18-6 | |
| NaCl | Sigma-Aldrich | 7647-14-5 | |
| Spinning disc confocal microscope | Leica DMI-4000B inverted microscope equipped with Yokogawa CSU 10 spinning disc Leica Microsystems Inc. | n/a | |
| Hamamatsu C9100-13 ImagEM EMCCD camera | Hamamatsu | n/a | |
| PatchMan NP 2 Micromanipulator | Eppendorf Inc. | 920000037 | |
| FemtoJet Microinjector | Eppendorf Inc. | 920010504 | |
| Injection needle: Femtotips II | Eppendorf Inc. | 930000043 | |
| Loading tip: 20ul Microloader | Eppendorf Inc. | 930001007 | |
| Micro Cover glasses no. 1 or 1.5, 22x40mm | VWR | 48393-026; 48393-172 | |
| Dry yeast | Any grocery store | n/a | |
| Computer, > 20 GB RAM | Although processing can be carried out on most computers, higher capabilities will increase the speed of the processing |
References
- Tyagi, S. Imaging intracellular RNA distribution and dynamics in living cells. Nature Methods. 6 (5), 331-338 (2009).
- Bao, G., Rhee, W. J., Tsourkas, A. Fluorescent probes for live-cell RNA detection. Annual Reviews of Biomedical Engineering. 11, 25-47 (2009).
- Mannack, L. V., Eising, S., Rentmeister, A. Current techniques for visualizing RNA in cells. F1000Research. 5, (2016).
- Larson, D. R., Zenklusen, D., Wu, B., Chao, J. A., Singer, R. H. Real-time observation of transcription initiation and elongation on an endogenous yeast gene. Science. 332 (6028), 475-478 (2011).
- Bertrand, E., et al. Localization of ASH1 mRNA particles in living yeast. Molecular Cell. 2 (4), 437-445 (1998).
- Garcia, J. F., Parker, R. MS2 coat proteins bound to yeast mRNAs block 5' to 3' degradation and trap mRNA decay products: implications for the localization of mRNAs by MS2-MCP system. RNA. 21 (8), 1393-1395 (2015).
- Bratu, D. P. Molecular beacons: Fluorescent probes for detection of endogenous mRNAs in living cells. Methods in Molecular Biology. 319, 1-14 (2006).
- Bratu, D. P., Cha, B. J., Mhlanga, M. M., Kramer, F. R., Tyagi, S. Visualizing the distribution and transport of mRNAs in living cells. Proceedings of the National Academy of Sciences of the Unites States of America. 100 (23), 13308-13313 (2003).
- Tyagi, S., Kramer, F. R. Molecular beacons: probes that fluoresce upon hybridization. Nature Biotechnology. 14 (3), 303-308 (1996).
- Chen, M., et al. A molecular beacon-based approach for live-cell imaging of RNA transcripts with minimal target engineering at the single-molecule level. Scientific Reports. 7 (1), 1550 (2017).
- Liu, Y., et al. Multiplex detection of microRNAs by combining molecular beacon probes with T7 exonuclease-assisted cyclic amplification reaction. Analytical and Bioanalytical Chemistry. 409 (1), 107-114 (2017).
- Baker, M. B., Bao, G., Searles, C. D. In vitro quantification of specific microRNA using molecular beacons. Nucleic Acids Research. 40 (2), e13 (2012).
- Ko, H. Y., et al. A color-tunable molecular beacon to sense miRNA-9 expression during neurogenesis. Scientific Reports. 4, 4626 (2014).
- Vet, J. A., et al. Multiplex detection of four pathogenic retroviruses using molecular beacons. Proceedings of the National Academy of Sciences of the Unites States of America. 96 (11), 6394-6399 (1999).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Methods in Molecular Biology. 429, 209-224 (2008).
- Li, W. M., Chan, C. M., Miller, A. L., Lee, C. H. Dual Functional Roles of Molecular Beacon as a MicroRNA Detector and Inhibitor. Journal of Biological Chemistry. 292 (9), 3568-3580 (2017).
- Kuang, T., Chang, L., Peng, X., Hu, X., Gallego-Perez, D. Molecular Beacon Nano-Sensors for Probing Living Cancer Cells. Trends in Biotechnology. 35 (4), 347-359 (2017).
- Ban, K., et al. Non-genetic Purification of Ventricular Cardiomyocytes from Differentiating Embryonic Stem Cells through Molecular Beacons Targeting IRX-4. Stem Cell Reports. 5 (6), 1239-1249 (2015).
- Hadjinicolaou, A. V., Demetriou, V. L., Emmanuel, M. A., Kakoyiannis, C. K., Kostrikis, L. G. Molecular beacon-based real-time PCR detection of primary isolates of Salmonella Typhimurium and Salmonella Enteritidis in environmental and clinical samples. BMC Microbiology. 9, 97 (2009).
- McLaughlin, J. M., Bratu, D. P. Drosophila melanogaster Oogenesis: An Overview. Methods in Molecular Biology. 1328, 1-20 (2015).
- Bastock, R., St Johnston, D. Drosophila oogenesis. Current Biology. 18 (23), R1082-R1087 (2008).
- Rongo, C., Gavis, E. R., Lehmann, R. Localization of oskar RNA regulates oskar translation and requires Oskar protein. Development. 121 (9), 2737-2746 (1995).
- Mhlanga, M. M., et al. In vivo colocalisation of oskar mRNA and trans-acting proteins revealed by quantitative imaging of the Drosophila oocyte. PLoS One. 4 (7), e6241 (2009).
- Bayer, L. V., Omar, O. S., Bratu, D. P., Catrina, I. E. PinMol: Python application for designing molecular beacons for live cell imaging of endogenous mRNAs. bioRxiv. , (2018).
- Marras, S. A., Kramer, F. R., Tyagi, S. Efficiencies of fluorescence resonance energy transfer and contact-mediated quenching in oligonucleotide probes. Nucleic Acids Research. 30 (21), e122 (2002).
- Bratu, D. P., Catrina, I. E., Marras, S. A. Tiny molecular beacons for in vivo mRNA detection. Methods in Molecular Biology. 714, 141-157 (2011).
- Dean, D. A. Preparation (pulling) of needles for gene delivery by microinjection. Cold Spring Harbor. 2006 (7), (2006).
- Alami, N. H., et al. Axonal transport of TDP-43 mRNA granules is impaired by ALS-causing mutations. Neuron. 81 (3), 536-543 (2014).
- Jackson, S. R., et al. Applications of Hairpin DNA-Functionalized Gold Nanoparticles for Imaging mRNA in Living Cells. Methods in Enzymology. 572, 87-103 (2016).
- Zimyanin, V. L., et al. In vivo imaging of oskar mRNA transport reveals the mechanism of posterior localization. Cell. 134 (5), 843-853 (2008).
- Catrina, I. E., Marras, S. A., Bratu, D. P. Tiny molecular beacons: LNA/2'-O-methyl RNA chimeric probes for imaging dynamic mRNA processes in living cells. ACS Chemical Biology. 7 (9), 1586-1595 (2012).
- Chen, A. K., Behlke, M. A., Tsourkas, A. Efficient cytosolic delivery of molecular beacon conjugates and flow cytometric analysis of target RNA. Nucleic Acids Research. 36 (12), e69 (2008).
- Nitin, N., Santangelo, P. J., Kim, G., Nie, S., Bao, G. Peptide-linked molecular beacons for efficient delivery and rapid mRNA detection in living cells. Nucleic Acids Research. 32 (6), e58 (2004).
- Chen, A. K., Behlke, M. A., Tsourkas, A. Avoiding false-positive signals with nuclease-vulnerable molecular beacons in single living cells. Nucleic Acids Research. 35 (16), e105 (2007).
- Bevilacqua, P. C., Ritchey, L. E., Su, Z., Assmann, S. M. Genome-Wide Analysis of RNA Secondary Structure. Annual Review of Genetics. 50, 235-266 (2016).
- Mhlanga, M. M., Vargas, D. Y., Fung, C. W., Kramer, F. R., Tyagi, S. tRNA-linked molecular beacons for imaging mRNAs in the cytoplasm of living cells. Nucleic Acids Research. 33 (6), 1902-1912 (2005).
- Eliceiri, K. W., et al. Biological imaging software tools. Nature Methods. 9 (7), 697-710 (2012).
- Bolte, S., Cordelieres, F. P. A guided tour into subcellular colocalization analysis in light microscopy. Journal of Microscopy. 224 (Pt 3), 213-232 (2006).
- Trcek, T., et al. Drosophila germ granules are structured and contain homotypic mRNA clusters. Nature Commununications. 6, 7962 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved