需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
可视化和跟踪活果蝇黑色素细胞卵室的内源性 mRNA
摘要
在这里,我们提出了一个协议,用于可视化,检测,分析和跟踪内源性mRNA贩运在活的果蝇黑色素细胞卵室使用分子信标,旋转盘共聚焦显微镜,和开源分析软件。
摘要
基于荧光的成像技术,结合光显微镜的发展,彻底改变了细胞生物学家进行活细胞成像研究的方式。自从开创性研究将位点特异性mRNA定位与基因表达调控联系起来以来,检测RNA的方法已经大大扩展。动态 mRNA 过程现在可以通过检测 mRNA 的方法进行可视化,再加上足够快的显微镜设置,可以捕获分子行为的动态范围。分子信标技术是一种基于杂交的方法,能够直接检测活细胞中的内源性转录。分子信标是发夹状、内部淬火、单核苷酸鉴别核酸探针,只有在杂交到独特的靶序列时才会产生荧光。当与先进的荧光显微镜和高分辨率成像相结合时,它们使人们能够对mRNA的细胞内运动进行空间和时间跟踪。虽然这项技术是能够检测内源转录本的唯一方法,但由于在设计活细胞成像的此类探针方面有困难,细胞生物学家尚未完全接受这项技术。一种新的软件应用程序PinMol,允许增强和快速设计的探头最适合有效地杂交到活细胞内的mRNA目标区域。此外,高分辨率、实时图像采集和当前开源图像分析软件可实现精细的数据输出,从而更精细地评估 mRNA 生命周期中涉及的动态过程的复杂性。
在这里,我们提出了一个全面的协议,用于设计和交付分子信标到果蝇黑色素细胞的卵子室。通过旋转盘共聚焦显微镜对内源性母体mRNA进行直接和高度特异性的检测和可视化。成像数据使用冰面软件中的物体检测和跟踪进行处理和分析,以获取有关 mRNA 动态移动的详细信息,mRNA 被传输并本地化到卵母细胞内的专门区域。
引言
通过开发基于荧光的活细胞成像技术,能够用空间和时间分辨率来可视化动态事件。目前,在体内mRNA可视化是通过基于RNAaptamer-蛋白相互作用、RNAaptamer诱导的有机染料荧光和核酸探退火1、2、3.均提供高特异性、灵敏度和信背景比。然而,以RNA贴合剂为中心的方法需要大量的基因操作,其中转基因被设计为表达具有蛋白质或有机染料结合所需的人工结构图案的RNA。例如,MS2/MCP 系统需要共同表达一个转基因表达的RNA结构,该结构包含噬菌体 MS2 涂层蛋白 (MCP) 的多重串联重复,另一个转基因编码荧光蛋白融合到MCP4,5。在RNA中加入这种二次结构图案,以及大量荧光标记的蛋白质,引起了人们的担忧,即原生RNA过程可能受到影响6。一项解决了这一问题并提供额外独特优势的技术是核酸方法,分子信标(MB)。MB允许多向地源性mRNA检测,区分单核苷酸变异,以及快速动力学与目标mRNA7,8杂交。MB是寡核苷酸探针,一旦杂交到目标(图1C)9,在经历氟性形成变化之前,它们仍停留在淬火发夹褶皱中。几个组已经成功地使用MB检测非编码RNA(微RNA和lncRNA)10,11,12,13,RNA逆转录病毒14和动态DNA蛋白交互15.它们已成功应用于各种生物体和组织成像,如斑马鱼胚胎16、神经元13、肿瘤组织17、区分心肌细胞18和沙门氏菌19.
在这里,我们描述了活D.melanogaster卵室内源性mRNA的设计、交付和检测方法,以及足够快的显微镜设置,能够捕获活性分子传输的动态范围。D. melanogaster卵子室是一个理想的多细胞模型系统,用于广泛的发育研究,从早期生殖系干细胞分裂和母体基因表达到分段体计划20的生成, 21.蛋室易于隔离,大而半透明,能够承受数小时的体外分析,使其高度易于成像实验。在积极翻译之前,许多工作都集中在母体抄本到离散亚细胞区域的不对称本地化上。特别是,oskar mRNA定位及其随后在卵母细胞后极的翻译必须以严格调节的方式发生,以避免致命的双体胚胎表型22。oskar mRNA被转录在15个生殖系细胞中,称为护士细胞,并通过细胞质桥(称为环管)积极输送到卵母细胞,即成为成熟卵子并最终受精的生殖细胞(图1A)).现有大量关于蛋白质因子动态招募和交换的信息,以及其远程细胞内旅行,使oskar成为学习的首选mRNA生命周期的许多过程。MB有助于揭示mRNA定位过程的细节,并破译控制果蝇细胞生成过程中控制mRNA传输的蛋白质因子的调控和功能。特别是,通过将MB微注射到护士细胞中并进行活细胞成像实验,可以追踪内源性mRNA8,23。
此处提供的路线图提供了完整流程的步骤,从使用 MB 进行活细胞成像实验、获取成像数据,到执行数据分析以跟踪其原生细胞环境中的内源性 mRNA。可以修改和进一步优化这些步骤,以满足在他们自己的实验室环境中处理其他组织/细胞类型的研究人员的需求。
Access restricted. Please log in or start a trial to view this content.
研究方案
1. 活细胞成像MB的设计
-
折叠目标RNA序列,使用mfold服务器(http://unafold.rna.albany.edu/?q=mfold/RNA-Folding-Form)中的"RNA形式"预测mRNA目标的二次结构。
- 以 FASTA 格式粘贴/上传目标序列,选择 5 或 10% 次优极性(自由能量在 MFE 值的 5 或 10% 以内折叠的结构),并相应地调整计算折叠的最大数量(例如,较大的 10%次优度)。
注意:在设计MB时,包括次优二级结构,可以识别目标mRNA内可能比仅预测的最低自由能(MFE)结构更灵活或更刚性的区域,从而改进适合活细胞成像的MB的整体设计。 - 为 800 个核苷酸 (nt) 的 mRNA 目标选择"立即作业",或为 mRNA 长度介于 801 到 8,000 nt 之间的"批量作业"选择"立即作业"。将"ss-count"文件保存为简单文本文件。
- 以 FASTA 格式粘贴/上传目标序列,选择 5 或 10% 次优极性(自由能量在 MFE 值的 5 或 10% 以内折叠的结构),并相应地调整计算折叠的最大数量(例如,较大的 10%次优度)。
-
使用步骤 1.1 中获取的"s-count"文件作为PinMol程序 (https://bratulab.wordpress.com/software/) 的输入,为 mRNA 目标设计多个 MB(请参阅描述PinMol程序使用情况的教程24 https://bratulab.wordpress.com/tutorial-pinmol-mac/)。
- 通过执行 BLAST 分析确定所选 MB 的特异性:将"blastn"与适当的数据库一起使用(例如,对于oskar mRNA 特异性 MB,使用"refseq-rna"数据库和果蝇黑色素体生物体)。
- 识别 mRNA 靶的任何组织特定表达(例如,oskar mRNA Flybase> 高通量表达数据 > FlyAtlas 解剖微阵列或 modENCODE 解剖RNA-Seq;http://flybase.org/reports/FBgn0003015)并与任何积极的 BLAST 命中。消除显示 >50% 与其他 mRNA 交叉同源的探针,这些探针也表达在感兴趣的组织/细胞中。
- 选择适合用于进行活细胞成像的显微镜设置的荧光对和淬火对(例如 Cy5/BHQ2)25。
2. MB 合成、纯化和特性化
- 使用上述7或商业提供商的服务使用内部合成和纯化,使用以下标记方案合成和纯化 1 到 5 MB(见上文注):[5'(荧光素)-(C3 或 C6 链接器)-(2'-O)-甲基 MB 序列)-(昆切)3'*。使用逆相 HPLC、内部或使用商业提供商的服务净化 MB。
注:用于自动探针合成的磷酰胺石必须具有2'-O-甲基核苷酸修饰。也可以使用交替锁定核酸(LNA)和2'-O-甲基修饰的嵌合体,以提高更短的MB与其目标mRNA26之间的杂交稳定性。 - 合成DNA寡核苷酸,与靶向RNA区域的序列相匹配,因此与MB的探针区域互补,用于体外表征(见步骤2.3至2.5;上注)。通过DNA-寡核苷酸靶点模拟,通过在DNA靶点的每一端包括四个额外的核苷酸,最大化甲基溴的杂交,如目标mRNA序列中所发现的那样。
注意:使用体外合成RNA靶点而不是互补DNA寡核苷酸8进行更严格地描述MB检测靶点序列的效率。 - 单独对 MB 进行热变性,测量其熔化温度 (Tm),并确认 MB 在生理温度下假定所需的发夹形状。我们观察到60至90°C之间的Tm值。
- 在DNA寡核苷酸靶场下对MB进行热变性,并测量MB:DNA靶混合体Tm,如前所述7。MB:DNA杂交需要55至60°C的Tm。
- 使用相应的DNA寡核苷酸靶点进行体外杂交反应,并确定MB:DNA杂交形成在生理温度下的效率,如前文所述。需要使用DNA靶点模拟的快速杂交动力学,但是,在体外和/或体内,如果MB没有显示高杂交效率,则具有更好的性能。
3. 微注射单个卵子室的解剖和制备
- 用新鲜酵母膏喂养新孵化的交配雌性2-3天。
- 麻醉苍蝇在CO2垫上,并使用精细钳子(杜蒙特#5),将1-2只雌性转移到玻璃盖滑道上的卤碳油700滴中。
- 使用一对钳子,在立体显微镜下,将苍蝇的背侧与背侧定向。通过在后端做一个小切口,将一对卵巢轻轻挤压到油中,切除女性腹部。
- 将卵巢放在新盖玻片上的油滴上。用一个钳子轻轻握住一个卵巢,同时用另一个钳子捏掉卵巢的最年轻阶段。oskar mRNA 在中生(阶段 > 7)和年轻的卵室(阶段 < 7)是主动局部的,并且不会存活太久。缓慢拖动盖滑(向下移动),直到单个卵子或蛋室被隔离并垂直对齐。通过从卵子链中取代不需要的阶段,进一步分离单个蛋室。
注:确保单独取笑的蛋室不会漂浮在油中,并且它们粘附在盖滑上。这对成功的显微注射和图像采集都非常重要。
4. 将MB微注射到卵室的护士室
- 使用一个分子信标(例如 osk2216Cy5)或两个针对不同 mRNA 的 MB 组合,并用光谱上不同的荧光团(例如 osk2216Cy5 和 drongo111Cy3)进行制备 MB 解决方案。在 HybBuffer 中每 MB 的浓度为 200-300 纳克/μL(50 mM Tris-HCl - pH 7.5,1.5 mM MgCl2和 100 mM NaCl)。对于在 HybBuffer 中,以相同的 mRNA 为目标相同的 mRNA 的四个 MB 的鸡尾酒,每个 mRNA 以 200 纳克/μL(例如 osk82、osk1236、osk2216)。在装针进行显微注射之前,立即将 MB 溶液旋转。
- 选择目标。建议采用 40x 油镜,用于寻找合适的蛋室和执行显微注射。
- 将带有解剖蛋室的盖玻片安装到显微镜上。在焦点位置提出目标,并在中后期发育阶段确定一个卵室,该卵子室适合显微注射(即,与垂直于针尖的 A+P 轴,以便轻松在护士体内注射细胞接近卵母细胞)。
- 用 +1 μL MB 溶液加载针头(商业或准备在27室内),并将其连接到显微注射器。对于D.melanogaster卵室的微注射,将针头(见材料表)以<45°的角度定向到显微镜阶段(例如30°),以避免刺穿多个护士细胞。
- 设置喷射压力为 500-1,000 hPa 和补偿压力为 100-250 hPa 的喷油器(参见材料表)。
- 慢慢移动舞台,把视野中的油滴空隙的蛋室带进视野。
- 使用微操作器操纵杆,轻轻地将针头降至油滴中,使其尖端聚焦到视野外围。
- 执行"清洁"功能,从针尖上清除空气,并确保针头流出。
- 将针头带到主位置,并聚焦于要微注射的卵室,然后将针头重新聚焦并放置在蛋室边缘附近。
- 对目标的Z位置进行微调,使卵泡细胞与护士细胞分离的膜处于焦点。
- 将针头插入护士室,进行2-5s的注射。
- 轻轻取下针头并将其缩回主位置。
- 将目标更改为图像采集所需的放大倍率(60-63x 或 100x),聚焦蛋室,然后开始采集。
5. 使用旋转盘共聚焦显微镜设置采集数据
注:有关我们特定设置,请参阅材料表。
- 设置采集协议以记录由 8-16 位图像组成的 XYZCt 堆栈(XYZ = 音量、C = 通道、t = 时间)。
- 为所需通道选择激光线(例如,Cy5 的 641 nm 激光和 GFP 的 491 nm 激光),并按顺序获取通道:首先在每个通道中发出荧光信号,然后更改 Z 位置,以便进行适当的共定位分析。
- 选择 Z 步长(例如 0.3 μm)和顶部和底部 Z 限制(例如-2 μm 到 2 μm)。
- 输入采集时间和采样速率(例如,每 15-30 s 一次,长达 1 小时)。
- 启动采集。
6. 处理、数据分析以获取跟踪和共定位信息,以及准备视频文件
- 图像处理
- 下载、解包和打开 Icy,一个用于生物图像信息学的开放社区平台(http://icy.bioimageanalysis.org/)
- 打开步骤 5 中获取的 XYZCt 堆栈:图像/序列 >文件 >打开。
- 将堆栈转换为 ImageJ:ImageJ> 工具> 转换为 IJ,具有分离模式打开。
- 制作子堆栈(需要进一步分析的 Z 步长和时间点范围的选择):ImageJ>Image>Stacks>工具>使子堆栈...选择所需的通道、Z 步长和时间点。
- 将子堆栈另存为 TIFF 文件:ImageJ>文件>另存为>Tiff...;使用此文件执行后续步骤。
- 拆分通道:ImageJ>Image>颜色>拆分通道。
- 使用背景堆栈减去背景:ImageJ>Process>图像计算器...,或使用滚动球选项:ImageJ>Process>减去背景...,选择滚动球半径。在选择"接受"之前,预览所选半径的图像。
注意:背景信号将主要产生于氟荧光团的淬火不当。信号:背景比(S:B)常用作MB"亮度"的指标,通过MB和DNA靶位寡核苷酸的体外杂交实验进行测量。例如,MB osk1236 和 osk2216 的 S:B 分别为 +81 和 +120。 - 调整每个通道的亮度和对比度:ImageJ>图像>调整>亮度/对比度,选择"应用"。
- 将每个通道另存为单独的 TIFF 文件:ImageJ>文件>另存为>Tiff....
- 合并两个通道:ImageJ>Image>颜色>合并通道...选择通道。将新堆栈另存为新 TIFF 文件(请参阅步骤 6.1.8)。
- 点检测和跟踪
- 转换回冰:图像J>工具>转换为冰。
- 如果安装了比例尺插件[使用插件搜索>安装程序>在线插件],则比例栏在转换为 Icy 时会自动叠加到堆栈上。如果需要,通过"检查器"窗口(屏幕右侧)编辑比例尺>图层选项卡>Name>缩放栏。
- 从"图层"选项卡取消选择/停用比例栏的"眼睛"图标>Name 以从原始堆栈中删除比例尺。它可以在最后堆栈上重新激活。
- 使用图像窗口菜单栏中的"相机"图标、"拍摄当前视图的屏幕截图"和"文件>另存为>Tiff"进行屏幕截图,从而保存新处理的堆栈。
- 确定点灵敏度(如果已确定点灵敏度参数)移动到步骤 6.2.7。
- 检测点:选择带有要分析的图像或堆栈的窗口,检测和跟踪>检测>点检测器,并填写"设置"参数:
- 对于输入,选择"当前序列输入检测"(默认值)。
- 对于预处理,通过交叉引用"检查器"窗口中的数字,选择"通道 0"(默认)或所需通道。
- 对于探测器,选择"在暗背景上检测亮点;"仅当堆栈中没有足够的 Z 切片执行分析时,才使用"强制使用 2D 小波进行 3D"。为每个比例选择"缩放"和"灵敏度"(为较大的斑点添加更多比例)。比例和灵敏度(数字越大,检测越敏感,Icy 建议最多 140)是试验变量和误差变量,必须在事后目视进行检查并做出决定。
- 对于感兴趣的区域,请使用"ROIfrom 序列"(默认)。
- 对于筛选,使用"无筛选"(默认值)或选择"大小筛选"来定义"接受对象的范围(以像素为单位)"。
- 输出:选择 XLS 或 XML 输出设置(使用 2007 MS Excel 或更早版本时选择 XML 格式,并且有 >65,000 个点)。如果检测点结果用于跟踪分析,则还选择"导出到"游泳池"。
- 使用各种比例/灵敏度值重复检测斑点,直到检测到所有或大部分斑点。记录所有最终参数。
- 对于共定位分析,对其他通道重复点检测。
- 要跟踪点,请选择"检测和跟踪">跟踪>污点跟踪>使用步骤 6.2.6 中的参数运行污点检测器,或使用"在此处选择检测结果"下拉菜单选择现有数据集(为此,保留污点检测器窗口从步骤 6.2.5 打开)。按"估计参数"按钮,在"参数估计"弹出窗口中选择所需的目标运动(例如,"既是扩散的,也是定向的")。按"运行跟踪"按钮。
- 在跟踪多通道堆栈的点时,按照步骤 6.2.6 和 6.2.7 对其他通道重复点检测和跟踪,从步骤 6.2.7 生成的堆栈开始。
- 要可视化轨道,请选择"检测和跟踪">跟踪>跟踪管理器 - 此窗口在完成跟踪运行后自动打开。对于"颜色轨道处理器",选择"启用"并选择轨道所需的颜色表示形式。相关轨道处理器可通过"添加轨道处理器..."进行访问。下拉菜单(例如,选择"跟踪处理器时间剪辑",启用"跟踪剪辑"窗口,并选择在当前时间点前后显示的所需检测数。
- 将跟踪信息另存为 XML 跟踪文件:检测和跟踪>跟踪>跟踪管理器>文件>另存为......
- 使用图像窗口菜单栏中的"相机"图标"拍摄屏幕截图,以"拍摄当前视图的屏幕截图"来保存结果。只需激活/停用检查器窗口中的相应眼睛图标(s),即可使用检测到的斑点和/或轨道进行屏幕截图。"层"选项卡>Name>叠加包装器。
- 安装时间戳叠加插件:插件>安装程序>在线插件>时间戳叠加>安装。
- 添加时间戳:插件>时间戳叠加(新建)。按照弹出窗口(屏幕右下角)上的说明操作,了解放置和设置时间戳格式的说明。时间间隔可以在"检查器"窗口中添加/更改>序列选项卡>序列属性>编辑。
- 通过拍摄另一个屏幕截图来保存结果。将图像另存为 1) Tiff 格式,2) 保存为 AVI 格式;对于 AVI 格式,首先转换为 RGB 渲染(图像/序列>渲染>RGB 图像)。
- 将图像旋转到所需方向:检查器窗口>序列选项卡>Canvas>旋转。
- 通过"拍摄当前视图的屏幕截图"保存旋转的图像。确保取消选择比例尺的"眼睛"图标,因为它也会随图像旋转。
- 选择并裁剪 ROI:选择感兴趣的区域>2D ROI>选择 ROI 形状,然后在图像上创建/绘制 ROI;图像/序列>平面(XY)>快速裁剪。
- 共定位分析
- 准备一个共定位协议;Icy 网站 (http://icy.bioimageanalysis.org/protocol/List) 提供了几个示例(参见补充材料)。
- 加载共定位协议:工具>脚本>协议>加载,并调整交互块中的参数(例如,在步骤6.2.6中确定的"小波点检测"模块中使用参数中)。
- 以像素为单位测量粒子的大小,确定共定位距离并将其输入到"Colocalizer"块中,将其输入为"最大距离"。
注:以像素为单位的粒子大小取决于检测系统。要测量大小,可以放大到单个粒子并手动计算跨越信号宽度的像素。从至少三个粒子中测量值平均。为共定位设置的最大距离是粒子的大小(表示两个粒子接触的半径的最大总和)。 - 如果需要,请选择一个或多个 ROI 进行共定位分析:感兴趣区域>2D ROI>选择 ROI 形状>在图像上绘制 ROI。
- 作物投资回报率:图像/序列>平面(XY)>快速裁剪。
- 执行共定位:协议编辑器窗口>选择协议选项卡>运行。协议编辑器窗口中的最后一个块将包含基于点检测的整体共定位百分比,而每个时间点的信息可以在"检查器"窗口> 输出选项卡中找到。
- 按照步骤 6.2.7(跟踪点)跟踪共位和单个粒子。
- 如步骤 6.2.16 所述保存。
Access restricted. Please log in or start a trial to view this content.
结果
使用PinMol,可以为一个 mRNA 目标设计多个 MB(图 1B-C)。经过合成和纯化,使用体外分析对选定的MB进行特征描述和比较。
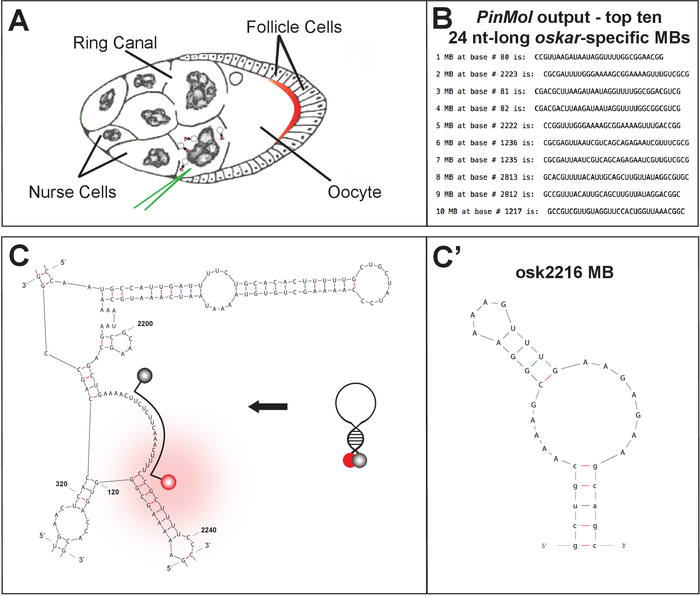
图1:内源性mRNA活细胞成像技术和组织描述。(A) 用于显微注射的中期果蝇?...
Access restricted. Please log in or start a trial to view this content.
讨论
果蝇卵室内源性mRNA贩运的实时可视化依赖于使用特定、高效且耐核酸酶的MB,现在可以使用PinMol软件轻松设计。MB 是特定探针,旨在检测目标 mRNA 内的独特序列(最好是无二次结构的区域),从而能够对转录记录进行高解析检测。在其他组织/细胞类型采用此技术/协议时,唯一的限制是感兴趣的样本的 MB 交付效率。虽然其他方法要求对组织进行基因操作,以表达贴合剂和RNA结合蛋白,并带有荧?...
Access restricted. Please log in or start a trial to view this content.
披露声明
提交人没有利益冲突可披露。
致谢
我们感谢Salvatore A.E.Marras(罗格斯大学公共卫生研究所中心)对分子信标的合成、标记和纯化,并感谢丹尼尔·圣约翰斯顿(剑桥大学古登研究所)的奥斯卡-MS2/ MCP-GFP转基因苍蝇库存。这项工作得到了国家科学基金会CAREER奖1149738和专业员工大会-CUNY奖的支持。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| Spectrofluorometer | Fluoromax-4 Horiba-Jobin Yvon | n/a | Photon counting spectrofluorometer |
| Quartz cuvette | Fireflysci (former Precision Cells Inc.) | 701MFL | |
| Dumont #5 tweezer | World Precision Instruments | 501985 | Thin tweezers are very important to separate out the individual egg chambers |
| Halocarbon oil 700 | Sigma-Aldrich | H8898 | |
| Cover slip No.1 22 mm x 40 mm | VWR | 48393-048 | |
| Dissecting microscope | Leica MZ6 Leica Microsystems Inc. | n/a | |
| CO2 fruit fly anesthesia pad | Genesee Scienific | 59-114 | |
| Tris-HCL pH 7.5 | Sigma-Aldrich | 1185-53-1 | |
| Magnesium chloride | Sigma-Aldrich | 7791-18-6 | |
| NaCl | Sigma-Aldrich | 7647-14-5 | |
| Spinning disc confocal microscope | Leica DMI-4000B inverted microscope equipped with Yokogawa CSU 10 spinning disc Leica Microsystems Inc. | n/a | |
| Hamamatsu C9100-13 ImagEM EMCCD camera | Hamamatsu | n/a | |
| PatchMan NP 2 Micromanipulator | Eppendorf Inc. | 920000037 | |
| FemtoJet Microinjector | Eppendorf Inc. | 920010504 | |
| Injection needle: Femtotips II | Eppendorf Inc. | 930000043 | |
| Loading tip: 20 μL Microloader | Eppendorf Inc. | 930001007 | |
| Micro Cover glasses no. 1 or 1.5, 22 mm x 40 mm | VWR | 48393-026; 48393-172 | |
| Dry yeast | Any grocery store | n/a | |
| Computer, > 20 GB RAM | Although processing can be carried out on most computers, higher capabilities will increase the speed of the processing |
参考文献
- Tyagi, S. Imaging intracellular RNA distribution and dynamics in living cells. Nature Methods. 6 (5), 331-338 (2009).
- Bao, G., Rhee, W. J., Tsourkas, A. Fluorescent probes for live-cell RNA detection. Annual Reviews of Biomedical Engineering. 11, 25-47 (2009).
- Mannack, L. V., Eising, S., Rentmeister, A. Current techniques for visualizing RNA in cells. F1000Research. 5, (2016).
- Larson, D. R., Zenklusen, D., Wu, B., Chao, J. A., Singer, R. H. Real-time observation of transcription initiation and elongation on an endogenous yeast gene. Science. 332 (6028), 475-478 (2011).
- Bertrand, E., et al. Localization of ASH1 mRNA particles in living yeast. Molecular Cell. 2 (4), 437-445 (1998).
- Garcia, J. F., Parker, R. MS2 coat proteins bound to yeast mRNAs block 5' to 3' degradation and trap mRNA decay products: implications for the localization of mRNAs by MS2-MCP system. RNA. 21 (8), 1393-1395 (2015).
- Bratu, D. P. Molecular beacons: Fluorescent probes for detection of endogenous mRNAs in living cells. Methods in Molecular Biology. 319, 1-14 (2006).
- Bratu, D. P., Cha, B. J., Mhlanga, M. M., Kramer, F. R., Tyagi, S. Visualizing the distribution and transport of mRNAs in living cells. Proceedings of the National Academy of Sciences of the Unites States of America. 100 (23), 13308-13313 (2003).
- Tyagi, S., Kramer, F. R. Molecular beacons: probes that fluoresce upon hybridization. Nature Biotechnology. 14 (3), 303-308 (1996).
- Chen, M., et al. A molecular beacon-based approach for live-cell imaging of RNA transcripts with minimal target engineering at the single-molecule level. Scientific Reports. 7 (1), 1550(2017).
- Liu, Y., et al. Multiplex detection of microRNAs by combining molecular beacon probes with T7 exonuclease-assisted cyclic amplification reaction. Analytical and Bioanalytical Chemistry. 409 (1), 107-114 (2017).
- Baker, M. B., Bao, G., Searles, C. D. In vitro quantification of specific microRNA using molecular beacons. Nucleic Acids Research. 40 (2), e13(2012).
- Ko, H. Y., et al. A color-tunable molecular beacon to sense miRNA-9 expression during neurogenesis. Scientific Reports. 4, 4626(2014).
- Vet, J. A., et al. Multiplex detection of four pathogenic retroviruses using molecular beacons. Proceedings of the National Academy of Sciences of the Unites States of America. 96 (11), 6394-6399 (1999).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Methods in Molecular Biology. 429, 209-224 (2008).
- Li, W. M., Chan, C. M., Miller, A. L., Lee, C. H. Dual Functional Roles of Molecular Beacon as a MicroRNA Detector and Inhibitor. Journal of Biological Chemistry. 292 (9), 3568-3580 (2017).
- Kuang, T., Chang, L., Peng, X., Hu, X., Gallego-Perez, D. Molecular Beacon Nano-Sensors for Probing Living Cancer Cells. Trends in Biotechnology. 35 (4), 347-359 (2017).
- Ban, K., et al. Non-genetic Purification of Ventricular Cardiomyocytes from Differentiating Embryonic Stem Cells through Molecular Beacons Targeting IRX-4. Stem Cell Reports. 5 (6), 1239-1249 (2015).
- Hadjinicolaou, A. V., Demetriou, V. L., Emmanuel, M. A., Kakoyiannis, C. K., Kostrikis, L. G. Molecular beacon-based real-time PCR detection of primary isolates of Salmonella Typhimurium and Salmonella Enteritidis in environmental and clinical samples. BMC Microbiology. 9, 97(2009).
- McLaughlin, J. M., Bratu, D. P. Drosophila melanogaster Oogenesis: An Overview. Methods in Molecular Biology. 1328, 1-20 (2015).
- Bastock, R., St Johnston, D. Drosophila oogenesis. Current Biology. 18 (23), R1082-R1087 (2008).
- Rongo, C., Gavis, E. R., Lehmann, R. Localization of oskar RNA regulates oskar translation and requires Oskar protein. Development. 121 (9), 2737-2746 (1995).
- Mhlanga, M. M., et al. In vivo colocalisation of oskar mRNA and trans-acting proteins revealed by quantitative imaging of the Drosophila oocyte. PLoS One. 4 (7), e6241(2009).
- Bayer, L. V., Omar, O. S., Bratu, D. P., Catrina, I. E. PinMol: Python application for designing molecular beacons for live cell imaging of endogenous mRNAs. bioRxiv. , (2018).
- Marras, S. A., Kramer, F. R., Tyagi, S. Efficiencies of fluorescence resonance energy transfer and contact-mediated quenching in oligonucleotide probes. Nucleic Acids Research. 30 (21), e122(2002).
- Bratu, D. P., Catrina, I. E., Marras, S. A. Tiny molecular beacons for in vivo mRNA detection. Methods in Molecular Biology. 714, 141-157 (2011).
- Dean, D. A. Preparation (pulling) of needles for gene delivery by microinjection. Cold Spring Harbor. 2006 (7), (2006).
- Alami, N. H., et al. Axonal transport of TDP-43 mRNA granules is impaired by ALS-causing mutations. Neuron. 81 (3), 536-543 (2014).
- Jackson, S. R., et al. Applications of Hairpin DNA-Functionalized Gold Nanoparticles for Imaging mRNA in Living Cells. Methods in Enzymology. 572, 87-103 (2016).
- Zimyanin, V. L., et al. In vivo imaging of oskar mRNA transport reveals the mechanism of posterior localization. Cell. 134 (5), 843-853 (2008).
- Catrina, I. E., Marras, S. A., Bratu, D. P. Tiny molecular beacons: LNA/2'-O-methyl RNA chimeric probes for imaging dynamic mRNA processes in living cells. ACS Chemical Biology. 7 (9), 1586-1595 (2012).
- Chen, A. K., Behlke, M. A., Tsourkas, A. Efficient cytosolic delivery of molecular beacon conjugates and flow cytometric analysis of target RNA. Nucleic Acids Research. 36 (12), e69(2008).
- Nitin, N., Santangelo, P. J., Kim, G., Nie, S., Bao, G. Peptide-linked molecular beacons for efficient delivery and rapid mRNA detection in living cells. Nucleic Acids Research. 32 (6), e58(2004).
- Chen, A. K., Behlke, M. A., Tsourkas, A. Avoiding false-positive signals with nuclease-vulnerable molecular beacons in single living cells. Nucleic Acids Research. 35 (16), e105(2007).
- Bevilacqua, P. C., Ritchey, L. E., Su, Z., Assmann, S. M. Genome-Wide Analysis of RNA Secondary Structure. Annual Review of Genetics. 50, 235-266 (2016).
- Mhlanga, M. M., Vargas, D. Y., Fung, C. W., Kramer, F. R., Tyagi, S. tRNA-linked molecular beacons for imaging mRNAs in the cytoplasm of living cells. Nucleic Acids Research. 33 (6), 1902-1912 (2005).
- Eliceiri, K. W., et al. Biological imaging software tools. Nature Methods. 9 (7), 697-710 (2012).
- Bolte, S., Cordelieres, F. P. A guided tour into subcellular colocalization analysis in light microscopy. Journal of Microscopy. 224 (Pt 3), 213-232 (2006).
- Trcek, T., et al. Drosophila germ granules are structured and contain homotypic mRNA clusters. Nature Commununications. 6, 7962(2015).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。