È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Visualizzazione e monitoraggio di mRNA endogeni nelle camere uovo di Drosophila melanogaster dal vivo
In questo articolo
Riepilogo
Qui, presentiamo un protocollo per la visualizzazione, il rilevamento, l'analisi e il monitoraggio del traffico di mRNA endogeno nel vivo Drosophila melanogaster camera d'uovo utilizzando fari molecolari, microscopia confocale del disco rotante, e analisi open-source software.
Abstract
Le tecniche di imaging basate sulla fluorescenza, in combinazione con gli sviluppi della microscopia luminosa, hanno rivoluzionato il modo in cui i biologi cellulari conducono studi di imaging a cellule vive. I metodi per rilevare gli RNA si sono notevolmente espansi a partire dagli studi seminali collegati alla localizzazione dell'mRNA specifico del sito alla regolazione dell'espressione genica. I processi dinamici dell'mRNA possono ora essere visualizzati tramite approcci che rilevano gli mRNA, associati a set-up di microscopia abbastanza veloci da catturare la gamma dinamica del comportamento molecolare. La tecnologia dei fari molecolari è un approccio basato sull'ibridazione in grado di rilevare direttamente le trascrizioni endogene nelle cellule viventi. I fari molecolari sono sonde di acido nucleico a forma di tornante, accoltellato internamente, mononucleotide, che fluoresce solo dall'ibridazione in una sequenza bersaglio unica. Quando accoppiati con la microscopia a fluorescenza avanzata e l'imaging ad alta risoluzione, consentono di eseguire il monitoraggio spaziale e temporale del movimento intracellulare degli mRNA. Anche se questa tecnologia è l'unico metodo in grado di rilevare le trascrizioni endogene, i biologi cellulari non hanno ancora pienamente abbracciato questa tecnologia a causa delle difficoltà nella progettazione di tali sonde per l'imaging delle cellule vive. Una nuova applicazione software, PinMol, consente una progettazione avanzata e rapida delle sonde più adatte per ibridarsi in modo efficiente alle regioni bersaglio mRNA all'interno di una cellula vivente. Inoltre, l'acquisizione di immagini ad alta risoluzione e in tempo reale e l'attuale software di analisi delle immagini open source consentono un output di dati raffinato, portando a una valutazione più fine della complessità alla base dei processi dinamici coinvolti nel ciclo di vita del mRNA.
Qui presentiamo un protocollo completo per la progettazione e la consegna di fari molecolari nelle camere d'uovo della Drosophila melanogaster. Il rilevamento e la visualizzazione diretta e altamente specifica di mRNA materni endogeni avviene tramite la microscopia confocale del disco rotante. I dati di imaging vengono elaborati e analizzati utilizzando il rilevamento e il rilevamento degli oggetti nel software Icy per ottenere dettagli sul movimento dinamico degli mRNA, che vengono trasportati e localizzati in regioni specializzate all'interno dell'oocito.
Introduzione
Gli studi di biologia cellulare che visualizzano eventi dinamici con risoluzione spaziale e temporale sono stati resi possibili dallo sviluppo di tecniche di imaging delle cellule vive basate sulla fluorescenza. Attualmente, la visualizzazione mRNA in vivo si ottiene tramite tecnologie che si basano su interazioni RNA aptamer-proteina, fluorescenza indotta dall'RNA di coloranti organici e sonda dell'acido nucleico annealing1,2, 3. Tutti offrono un'elevata specificità, sensibilità e rapporto segnale-sfondo. Tuttavia, gli approcci centrati sull'RNA aptamer richiedono un'ampia manipolazione genetica, in cui un transgene è progettato per esprimere un RNA con motivi strutturali artificiali necessari per il legame di proteine o coloranti organici. Ad esempio, il sistema MS2/MCP richiede la co-espressione di un transgene che esprime un costrutto di RNA contenente più ripetizioni tandem della sequenza di legame per la proteina del pelo di batterifafo maMS2 (MCP) e un altro transgene che codifica una proteina fluorescente fusa in MCP4,5. L'aggiunta di tali motivi strutturali secondari all'RNA, insieme a una ingombrante proteina fluorescente, ha sollevato preoccupazioni sul fatto che i processi dell'RNA nativo possano essere influenzati6. Una tecnologia che affronta questa preoccupazione e offre ulteriori vantaggi unici è l'approccio basato sull'acido nucleico, i fari molecolari (MB). Gli MB consentono il rilevamento multiplex di mRNA endogeni, la discriminazione delle variazioni dei singoli nucleotidi e la cinetica veloce dell'ibridazione con mRNA bersaglio7,8. Gli MB sono sonde oligonucleotidi che rimangono in una prua spenta prima di subire un cambiamento conformazionale fluorogenico una volta ibridati ai loro obiettivi (Figura 1C)9. Diversi gruppi hanno avuto successo nell'utilizzo di MB per rilevare sia RNA non codificanti (microRNA e lncRNA)10,11,12,13, retrovirus RNA14 e DNA-protein dinamico interazioni15. Sono stati impiegati con successo per l'imaging in vari organismi e tessuti, come gli embrioni di pesce zebra16, i neuroni13, il tessuto tumorale17, differenziando i cardiomiociti18e la Salmonella 19.
Qui descriviamo l'approccio di progettazione, consegna e rilevamento per mRNA endogeni nelle camere d'uovo D. melanogaster viventi accoppiate con un set-up di microscopia che è abbastanza veloce per catturare la gamma dinamica del trasporto molecolare attivo. La camera d. melanogaster è servita come un sistema modello multicellulare ideale per una vasta gamma di studi sullo sviluppo, dalla divisione precoce delle cellule staminali germinali e l'espressione genica materna alla generazione del piano settoriale segmentale20, 21. Le camere delle uova sono facilmente isolate, grandi e traslucide e sono in grado di sopportare ore di analisi ex vivo, rendendole altamente suscettibili agli esperimenti di imaging. Molto lavoro si è concentrato sulla localizzazione asimmetrica delle trascrizioni materne in regioni subcellulari discrete prima di essere tradotte attivamente. In particolare, la localizzazione dell'oskar mRNA e la sua successiva traduzione al polo posteriore degli ovociti devono avvenire in modo strettamente regolamentato per evitare un fenotipo22di embrione biacaudale letale. oskar mRNA è trascritto nelle 15 cellule germinali, chiamate cellule infermiere, e trasportato attivamente attraverso ponti citoplasmici, chiamati ponti ad anello, nell'ovocita, la cellula germinale che diventa l'uovo maturo e viene infine fecondata (Figura 1A ). La notevole quantità di informazioni già disponibili per quanto riguarda il reclutamento dinamico e lo scambio di fattori proteici da e verso oskar mRNP, insieme ai suoi viaggi intracellulari a lungo raggio, rendono oskar un candidato preferito per studiare i numerosi processi del ciclo di vita dell'mRNA. Gli MB sono stati fondamentali per rivelare dettagli sul processo di localizzazione dell'mRNA e decifrare la regolazione e la funzione dei fattori proteici che controllano il trasporto dell'mRNA durante l'oogenesi della Drosophila. In particolare, microiniettando MB nelle cellule dell'infermiera ed eseguendo esperimenti di imaging in cellule vive, è possibile il monitoraggio degli mRNA endogeni8,23.
La tabella di marcia qui presentata offre le fasi di un processo completo, dall'esecuzione di un esperimento di imaging a cellule vive con manzi, acquisizione di dati di imaging, all'esecuzione di analisi dei dati per monitorare l'mRNA endogeno nel suo ambiente cellulare nativo. I passaggi possono essere modificati e ulteriormente ottimizzati per soddisfare le esigenze dei ricercatori che lavorano con altri tessuti / tipi di cellule all'interno del proprio ambiente di laboratorio.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Progettazione di MB per Live Cell Imaging
-
Piegare la sequenza di RNA bersaglio per prevedere la struttura secondaria del bersaglio mRNA utilizzando la "forma RNA" dal server mfold (http://unafold.rna.albany.edu/?q=mfold/RNA-Folding-Form).
- Incollare/caricare la sequenza di destinazione in formato FASTA, selezionare 5 o 10% di sub-optimality (strutture con un'energia libera di ripiegamento entro il 5 o 10% del valore MFE, rispettivamente) e regolare il numero massimo di pieghe calcolate di conseguenza (ad es. più grande per il 10% sub-ottimalità).
Nota: l'inclusione di strutture secondarie non ottimali durante la progettazione di MB consente l'identificazione di regioni all'interno dell'mRNA di destinazione che possono essere più flessibili o più rigide rispetto alle previsioni della sola struttura di energia libera minima (MFE), progettazione complessiva di MB adatti per l'imaging di cellule vive. - Selezionare un "lavoro immediato" per le destinazioni mRNA di 800 nucleotidi (nt) o un "processo batch" per lunghezze di mRNA comprese tra 801 e 8.000 nt. Salva il file "ss-count" come semplice file di testo.
- Incollare/caricare la sequenza di destinazione in formato FASTA, selezionare 5 o 10% di sub-optimality (strutture con un'energia libera di ripiegamento entro il 5 o 10% del valore MFE, rispettivamente) e regolare il numero massimo di pieghe calcolate di conseguenza (ad es. più grande per il 10% sub-ottimalità).
-
Utilizzare il file "ss-count" ottenuto nel passaggio 1.1 come input per il programma PinMol (https://bratulab.wordpress.com/software/) con i parametri desiderati, per progettare diversi MB per il target mRNA (vedere tutorial che descrivono l'utilizzo del programma PinMol 24 allhttps://bratulab.wordpress.com/tutorial-pinmol-mac/).
- Determinare la specificità dei MB selezionati eseguendo l'analisi BLAST: utilizzare "blastn" con il database appropriato (ad esempio per gli MB specifici di oskar mRNA utilizzare il database "refseq-rna" e l'organismo melanogaster Drosophila).
- Identificare qualsiasi espressione specifica del bersaglio mRNA (ad esempio per oskar mRNA Flybase> High-Throughput Expression Data> FlyAtlas Anatomy Microarray o modENCODE Anatomy RNA-Seq; http://flybase.org/reports/FBgn0003015) e confrontare con eventuali colpi BLAST positivi. Eliminare le sonde che mostrano >50% cross-homology con altri mRNA che sono espressi anche nel tessuto / cellula di interesse.
- Selezionare la coppia di fluorofori e quencher appropriata per il set-up di microscopia disponibile per eseguire l'imaging delle cellule vive (ad esempio Cy5/BHQ2)25.
2. MB Sintesi, Purificazione e Caratterizzazione
- Utilizzare la sintesi e la purificazione in-house come descritto in precedenza7, o servizi da fornitori commerciali, per sintetizzare e purificare da uno a cinque MB (vedi nota sopra), utilizzando il seguente schema di etichettatura: [5'(Fluorophore)-(C3 o C6 linker)-(2'-O -sequenza MB metilica)-(Quencher)3']. Purificare gli MB utilizzando HPLC in fase inversa, in casa o utilizzando i servizi del fornitore commerciale.
NOT: I fosforamiditi utilizzati per la sintesi automaticadella sonda devono avere la modifica del ribonucleotide 2'- O-metile. Si possono anche usare chimere di acido solare-nucleico alternato(LNA) e modifiche 2'- O-metile per aumentare la stabilità di un ibrido tra un MB più corto e il suo mRNA bersaglio26. - Sintetizzare gli oligonucleotidi del DNA che corrispondono alla sequenza della regione RNA mirata e quindi sono complementari alla regione della sonda degli MB, per l'uso nella caratterizzazione in vitro (vedi i passaggi da 2.3 a 2.5; nota sopra). Massimizzare l'ibridazione del MB con il mimio bersaglio DNA-oligonucleotide, includendo su ogni estremità del DNA bersaglio quattro nucleotidi aggiuntivi, come si trova nella sequenza di mRNA bersaglio.
Nota: una caratterizzazione più rigorosa dell'efficienza dell'MB per rilevare la sequenza mirata può essere eseguita utilizzando bersagli in vitro sintetizzati RNA invece di oligonucleotidi di DNA complementari8. - Eseguire la denaturazione termica del MB da solo, misurarne la temperatura di fusione (Tm) e verificare che l'MB assuma la forma desiderata a temperatura fisiologica. Abbiamo osservato valori di Tm tra 60 e 90 gradi centigradi.
- Eseguire la denaturazione termica del MB in presenza del bersaglio oligonucleotide del DNA e misurare la Tm dell'ibrido di destinazione MB:DNA, come descritto in precedenza7. Per l'ibrido MB:DNA si desidera un Tm compreso tra 55 e 60 gradi centigradi.
- Eseguire reazioni di ibridazione in vitro con il corrispondente obiettivo di oligonucleotide del DNA e determinare l'efficienza della formazione ibrida MB:DNA a temperatura fisiologica, come descritto in precedenza7. La cinetica di ibridazione rapida con l'imitazione del bersaglio del DNA è desiderata, tuttavia gli MB che non mostrano un'elevata efficienza di ibridazione con bersagli del DNA possono avere prestazioni migliori con l'mRNA bersaglio in vitro e/o in vivo.
3. Dissezione e preparazione di camere uovo individuali per microiniezione
- Nutrire le femmine appena schiuse e accoppiate per 2-3 giorni con pasta di lievito fresco.
- Anestesizza vola su un pad di CO2 e, usando una pinzetta fine (Dumont #5), trasferisci 1-2 femmine in una goccia di olio di alocarbonio 700 su un coperchio di copertura di vetro.
- Utilizzando un paio di pinzette, orientare la mosca con il lato dorsale sotto uno stereomicroscopio. Dissezionare l'addome femminile facendo una piccola incisione all'estremità posteriore e spremere delicatamente la coppia di ovaie nell'olio.
- Espiantare le ovaie su una goccia d'olio su una nuova vela. Tenere delicatamente un'ovaia con una pinzetta mentre si pizzicano le fasi più giovani dell'ovariole con l'altra pinzetta. oskar mRNA è attivamente localizzato a metà e dopo l'oogenesi (fasi > 7), e le camere d'uovo più giovani (stadi < 7) sono più difficili da iniettare e non sopravvivono più a lungo. Trascinare lentamente sullo scivolo di copertura (con un movimento verso il basso) fino a quando le singole ovarioos o camere d'uovo sono isolate e allineate verticalmente. Separare ulteriormente le camere a uovo singolo spostando le fasi indesiderate dalla catena delle uova di ovario.
NOT: Assicurarsi che le camere d'uovo tisued individualmente non galleggiano nell'olio, e che aderiscono alla ricevuta di copertura. Questo è importante sia per la microiniezione di successo che per l'acquisizione di immagini.
4. Microiniezione di MB nelle celle infermiere delle camere d'uovo
- Preparare la soluzione MB, utilizzando un faro molecolare (ad esempio osk2216Cy5), o una miscela di due MB che si rivolgono a mRNA diversi e che sono etichettati con fluorofori distinti spettrali (ad esempio osk2216Cy5 e drongo1111Cy3). Utilizzare una concentrazione di 200-300 ng/L ogni MB in HybBuffer (50 mM MM Tris-HCl - pH 7.5, 1,5 mM MgCl2 e 100 mM NaCl). Per un cocktail di quattro MB etichettati con lo stesso fluoroforo che prendono di mira lo stesso mRNA a 200 ng/L ciascuno in HybBuffer (ad esempio osk82, osk1236, osk2216). Abbassare la soluzione MB immediatamente prima di caricare l'ago per la microiniezione.
- Selezionare l'obiettivo. Per trovare una camera d'uovo appropriata e per eseguire la microiniezione si raccomanda un obiettivo di olio di 40x.
- Montare la coverslip con camera d'uovo sezionata sullo stadio del microscopio. Portare l'obiettivo nella posizione di messa a fuoco e identificare una camera d'uovo in una fase di sviluppo medio-tarda, che sia opportunamente orientata per la microiniezione (ad esempio, con l'asse AàP perpendicolare alla punta dell'ago per consentire una facile iniezione all'interno di un infermiere cellula prossimamica all'ovocita).
- Caricare un ago (commerciale o preparato in casa27) con una soluzione da 1 MB (vedere il punto 4.1) e collegarlo al microiniettore. Per le microiniezioni nelle camere d'uovo D. melanogaster, orientare l'ago (vedi Tabella deimateriali) con un angolo <45% rispetto allo stadio del microscopio (ad es. 30o) per evitare di forare diverse cellule infermieristiche.
- Impostare l'iniettore con pressione di iniezione di 500-1,000 hPa e pressione di compensazione di 100-250 hPa (vedi Tabella dei materiali).
- Spostare lentamente lo stadio per portare nel campo visivo un'area della goccia d'olio priva di camere d'uovo.
- Utilizzando il joystick micromanipolatore, abbassare delicatamente l'ago nella goccia d'olio e portare la sua punta a fuoco verso la periferia del campo visivo.
- Eseguire una funzione "pulita" per rimuovere l'aria dalla punta dell'ago e per garantire che ci sia flusso dall'ago.
- Portare l'ago in posizione iniziale e concentrarsi sulla camera dell'uovo per essere microiniettato, quindi riportare l'ago a fuoco e posizionarlo vicino al bordo della camera d'uovo.
- Eseguire una regolazione fine della posizione z dell'obiettivo in modo che la membrana che separa le cellule follicoli dalle cellule dell'infermiera sia a fuoco.
- Inserire l'ago in una cella dell'infermiera ed eseguire l'iniezione per 2-5 s.
- Rimuovere delicatamente l'ago e ritrarlo alla posizione di casa.
- Modificare l'obiettivo per l'ingrandimento desiderato per l'acquisizione di immagini (60-63x o 100x), concentrarsi sulla camera delle uova e iniziare l'acquisizione.
5. Acquisizione dei dati mediante la configurazione di un microscopio confocale del disco rotante
NOT: Vedere Tabella dei materiali per la configurazione specifica.
- Impostare il protocollo di acquisizione per registrare uno stack di immagini a 8-16 bit (XY , volume, C , canale, t e ora).
- Selezionare le linee laser per i canali desiderati (ad esempio laser da 641 nm per Cy5 e 491 nm per GFP) e acquisire i canali in sequenza: prima il segnale di fluorescenza in ogni canale e quindi cambiare la posizione di , per consentire una corretta analisi di co-localizzazione.
- Selezionare il passo di , ovvero 0,3 m, e i limiti superiore e inferiore (ades. da -2 m a 2 m).
- Immettere il tempo di acquisizione e la frequenza di campionamento (ad es. ogni 15-30 s per un massimo di 1 h).
- Avviare l'acquisizione.
6. Elaborazione, analisi dei dati per ottenere informazioni di tracciamento e co-localizzazione e preparazione dei file video
- Elaborazione delle immagini
- Scarica, disfare e aprire Icy, una piattaforma comunitaria aperta per l'informatica bioimage (http://icy.bioimageanalysis.org/)
- Aprire lo stack XY-Ct acquisito nel passaggio 5: Immagine/Sequenza >File>Apri.
- Converti pila in ImageJ: ImageJ> Strumenti> Converti in IJ, hanno Modalità scollegata attivata.
- Effettuare un sottostack (una selezione di un intervallo di passaggi e punti temporali da analizzare ulteriormente): ImageJ>Immagine>Stacks>Strumenti>Make Substack...; selezionare i canali, i passi z e i punti di tempo desiderati.
- Salva sottopila come file TIFF: ImageJ>File>Salva con nome>Tiff...; utilizzare questo file per i passaggi successivi.
- Dividi canali: ImageJ>Immagine>Colore>Canali dividi.
- Sottrarre lo sfondo utilizzando una pila di sfondo: ImageJ>Process>Image Calculator..., oppure utilizzando l'opzione Palla a rotazione: ImageJ>Processo>Sottrai sfondo..., selezionare il raggio della palla a rotazione. Visualizzare l'anteprima dell'immagine per il raggio selezionato prima di selezionare "Accetta".
Nota: Il segnale di fondo deriva principalmente da uno spegnimento improprio del fluroroforo. Il rapporto signal:background (S:B) è spesso usato come indicatore per la "luminosità" di un MB, e viene misurato da esperimenti di ibridazione in vitro dell'oligonucleotide MB e del bersaglio del DNA. Ad esempio, gli MB osk1236 e osk2216 hanno rispettivamente un valore S:B pari a 81 e 120 USD. - Regolare la luminosità e il contrasto per ogni canale: ImageJ>Image>Adjust>Brightness/Contrast (Regola la luminosità e il contrasto) per ciascun canale: ImageJ>Image>Adjust>Regola luminosità/contrasto (Regola )& ris).
- Salvare ogni canale come file TIFF separato: ImageJ>File>Salva con nome>Tiff....
- Unire i due canali: ImageJ>Immagine>Colore>Unisci canali...; selezionare i canali. Salvare il nuovo stack come nuovo file TIFF (vedere il passaggio 6.1.8).
- Rilevamento e tracciamento degli spot
- Convertire di nuovo in icy: ImageJ>Strumenti>Converti in icy.
- Una barra della scala viene automaticamente sovrapposta allo stack al momento della conversione in Icy, se è installato il plug-in della barra della scala [Ricerca tramite plugin>Setup>Plugin online]. Se necessario, modificare la barra della scala tramite la finestra Impostazioni (lato destro dello schermo)>Scheda Livello>Nome>Barra della scala.
- Deselezionate/disattivate l'icona "occhio" per la barra Scala dalla scheda Livello>Nome per rimuovere la barra della scala dalla pila originale. Può essere riattivato nello stack finale.
- Salvare la pila appena elaborata prendendo uno screenshot utilizzando l'icona "camera" dalla barra dei menu della finestra immagine, "Cattura uno screenshot della vista corrente" e File>Salva con nome>Tiff....
- Determinare la sensibilità spot, se i parametri di sensibilità spot sono già stati determinati spostarsi al passaggio 6.2.7.
- Rileva punti: seleziona la finestra con l'immagine o la pila da analizzare, Rilevamento&Tracciamento>Rilevamento>Rilevatore spot e compila i parametri Impostazioni:
- Per Input, selezionare "currentSequenceInputDetection" (impostazione predefinita).
- Per Pre-elaborazione, selezionare "Canale 0" (predefinito) o il canale desiderato facendo riferimento incrociato al numero nella scheda Finestra di controllo>Sequenza.
- Per Rilevatore, selezionare "Rileva punto luminoso su sfondo scuro;" utilizzare "Forza l'uso di Wavelets 2D per 3D" solo se non ci sono abbastanza fette di z nelle pile per eseguire l'analisi. Selezionare "Scale(s)" e "Sensitivity" per ogni scala (aggiungere più scale per punti più grandi). La Scala e la Sensibilità (maggiore è il numero più sensibile è il rilevamento, un massimo di 140 è suggerito da Icy) sono variabili di tentativi ed errori, che devono essere controllate visivamente in seguito e decise.
- Per Regione di interesse, utilizzare "ROIfromSequence" (impostazione predefinita).
- Per Filtro, utilizzare "NoFiltering" (impostazione predefinita) o selezionare "SizeFiltering" per definire l'intervallo di oggetti accettati (in pixel)".
- Output: selezionare l'impostazione di output XLS o XML (selezionare Formato XML quando si utilizza 2007 MS Excel o versioni precedenti e sono presenti >65.000 punti). Se i risultati del rilevatore di spot vengono utilizzati per l'analisi di tracciamento, selezionare anche "Esporta in SwimmingPool".
- Ripetere il rilevamento dei punti utilizzando vari valori di scala/sensibilità fino a quando non vengono rilevati tutti o la maggior parte dei punti. Registrare tutti i parametri finali.
- Per l'analisi della colocalizzazione, ripetere il rilevamento spot per l'altro canale.
- Per tenere traccia dei punti, selezionare Rilevamento&Tracciamento>Tracking>Spot Tracking>Esegui il rilevatore di punti con i parametri del passaggio 6.2.6, oppure utilizzare il menu a discesa "Seleziona risultati rilevamento qui" per selezionare un set di dati esistente (per questo, mantenere la finestra Spot Detector aperto dal passaggio 6.2.5). Premere il pulsante "Parametri stima" e selezionare il movimento di destinazione desiderato nella finestra a comparsa Stima parametri (ad esempio "è sia diffusa che diretta"). Premere il pulsante "Run tracking".
- Ripeti il rilevamento e il tracciamento dei punti per altri canali durante il monitoraggio dei punti di pile multicanale, seguendo i passaggi 6.2.6 e 6.2.7, a partire dalla pila generata dal passaggio 6.2.7.
- Per visualizzare le tracce, selezionare Rilevamento&Tracciamento>Tracking>Track Manager – questa finestra si apre automaticamente al termine di un'esecuzione di rilevamento. Per "Color Track Processor", selezionare "Abilita" e scegliere la rappresentazione di colore desiderata per le tracce. I processori di traccia rilevanti sono accessibili tramite il "aggiungi processore traccia..." menu a discesa (ad esempio, selezionare "Traccia clip tempo processore", attivare la finestra "Track Clipper" e scegliere il numero desiderato di rilevamenti da visualizzare prima e dopo il punto temporale corrente.)
- Salva tiene traccia delle informazioni come file di traccia XML: Rilevamento&Rilevamento>Tracciamento>Gestione tracce>File>Salva con nome....
- Salvare i risultati catturando uno screenshot utilizzando l'icona "fotocamera" dalla barra dei menu della finestra immagine, "Acquisire uno screenshot della vista corrente". Le schermate possono essere scattate con i punti e/o le tracce rilevate semplicemente attivando/disattivando le icone a forma di occhio corrispondenti presenti nella finestra di ispezione>scheda Livello>Nome>Wrapper overlay.
- Installare il plug-in TimeStamp Overlay: Plugins>Setup>Online plugin>TimeStamp Overlay>Install.
- Aggiungi timestamp: Plugins>TimeStamp Overlay (New). Seguire le istruzioni nella finestra pop-up (angolo inferiore destro dello schermo) per istruzioni sul posizionamento e la formattazione dell'indicatore di data e ora. L'intervallo di tempo può essere aggiunto/modificato nella finestra di ispezione>scheda Sequenza>Proprietà sequenza>Modifica.
- Salvare i risultati catturando un'altra schermata. Salvare l'immagine come 1) formato Tiff e 2) in formato AVI; per il formato AVI prima convertire in rendering RGB (Immagine/Sequenza>Rendering>RGB image).
- Ruotare l'immagine all'orientamento desiderato: Finestra di controllo>scheda Sequenza>Canvas>Rotazione.
- Salvare l'immagine ruotata con "Cattura uno screenshot della vista corrente". Assicurarsi che l'icona "occhio" per la barra della scala sia deselezionata, in quanto ruoterà anche con l'immagine.
- Scegliere e ritagliare il ROI: selezionare Regione di interesse>ROI 2D>Scegli forma ROI e quindi creare/disegnare IL ROI sull'immagine; Immagine/Sequenza>Piano (XY)>Ritaglio rapido.
- Analisi della co-localizzazione
- Preparare un protocollo di co-localizzazione; sul sito Web icy (http://icy.bioimageanalysis.org/protocol/List) sono disponibili diversi esempi (vedere Materialisupplementari ).
- Caricare il protocollo di colocalizzazione: Tools>Scripting>Protocols>Load e regolare i parametri nei blocchi che interagiscono (ad esempio nel blocco "Wavelet Spot Detecting" utilizzano i parametri determinati nel passaggio 6.2.6.).
- Misurare le dimensioni di una particella in pixel, determinare la distanza di co-localizzazione e inserirla nel blocco "Colocalizzare" come "Distanza massima".
NOT: La dimensione della particella in pixel dipende dal sistema di rilevamento. Per misurare le dimensioni, ingrandisci una singola particella e conta manualmente i pixel che coprono la larghezza del segnale. Calcolare la media delle misurazioni da almeno tre particelle. La distanza massima da impostare per la colocalizzazione è la dimensione della particella in pixel (questa rappresenta la somma massima del raggio di due particelle che si toccano). - Se lo si desidera, selezionare uno o più ROI per l'analisi della co-localizzazione: Regione di interesse>ROI 2D>Scegli forma ROI>Disegna ROI sull'immagine.
- ROI di ritaglio: Immagine/Sequenza>Piano (XY)>Ritaglio rapido.
- Esegui colocalizzazione: finestra dell'editor dei protocolli>Scheda Protocollo scelto>Esegui. Il blocco finale nella finestra dell'editor Protocolli conterrà la percentuale di co-localizzazione complessiva basata sul rilevamento spot, mentre le informazioni in ogni punto temporale sono disponibili nella finestra di ispezione> scheda Output.
- Traccia colocalizzate e particelle singole seguendo il passo 6.2.7 (tracce).
- Salvare come descritto nel passaggio 6.2.16.Save as described in step 6.2.16.
Access restricted. Please log in or start a trial to view this content.
Risultati
Utilizzando PinMol, diversi MB possono essere progettati per un bersaglio mRNA (Figura 1B-C). Dopo la sintesi e la purificazione, i MB selezionati vengono caratterizzati e confrontati mediante l'analisi in vitro.
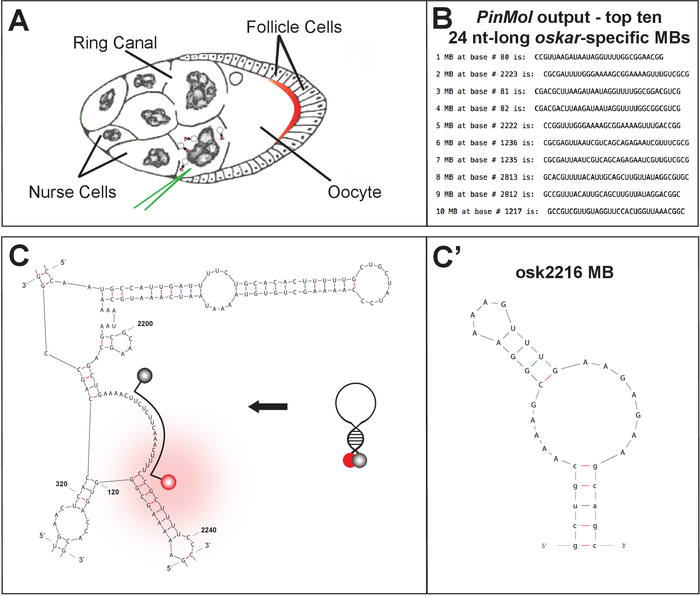
Figura 1: Tecnica e descrizione dei tessuti per l'imaging di cellule vi...
Access restricted. Please log in or start a trial to view this content.
Discussione
La visualizzazione dal vivo del traffico endogeno di mRNA nelle camere delle uova della Drosophila si basa sull'uso di MB specifici, efficienti e resistenti alla nucasiera, che ora possono essere facilmente progettati con il software PinMol. Gli MB sono sonde specifiche progettate per rilevare sequenze univoche all'interno di un mRNA bersaglio (preferibilmente regioni prive di struttura secondaria), rendendo possibile il rilevamento altamente risolto di una trascrizione. L'unica limitazione quando si ad...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno alcun conflitto di interessi da divulgare.
Riconoscimenti
Ringraziamo Salvatore A.E. Marras (Public Health Research Institute Center, Rutgers University) per la sintesi, l'etichettatura e la purificazione dei fari molecolari, e Daniel St Johnston (The Gurdon Institute, Università di Cambridge) per l'oskar-MS2/ Stock di mosca transgenico MCP-GFP. Questo lavoro è stato sostenuto da un National Science Foundation CAREER Award 1149738 e da un Professional Staff Congress-CUNY Award al DPB.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Spectrofluorometer | Fluoromax-4 Horiba-Jobin Yvon | n/a | Photon counting spectrofluorometer |
| Quartz cuvette | Fireflysci (former Precision Cells Inc.) | 701MFL | |
| Dumont #5 tweezer | World Precision Instruments | 501985 | Thin tweezers are very important to separate out the individual egg chambers |
| Halocarbon oil 700 | Sigma-Aldrich | H8898 | |
| Cover slip No.1 22 mm x 40 mm | VWR | 48393-048 | |
| Dissecting microscope | Leica MZ6 Leica Microsystems Inc. | n/a | |
| CO2 fruit fly anesthesia pad | Genesee Scienific | 59-114 | |
| Tris-HCL pH 7.5 | Sigma-Aldrich | 1185-53-1 | |
| Magnesium chloride | Sigma-Aldrich | 7791-18-6 | |
| NaCl | Sigma-Aldrich | 7647-14-5 | |
| Spinning disc confocal microscope | Leica DMI-4000B inverted microscope equipped with Yokogawa CSU 10 spinning disc Leica Microsystems Inc. | n/a | |
| Hamamatsu C9100-13 ImagEM EMCCD camera | Hamamatsu | n/a | |
| PatchMan NP 2 Micromanipulator | Eppendorf Inc. | 920000037 | |
| FemtoJet Microinjector | Eppendorf Inc. | 920010504 | |
| Injection needle: Femtotips II | Eppendorf Inc. | 930000043 | |
| Loading tip: 20 μL Microloader | Eppendorf Inc. | 930001007 | |
| Micro Cover glasses no. 1 or 1.5, 22 mm x 40 mm | VWR | 48393-026; 48393-172 | |
| Dry yeast | Any grocery store | n/a | |
| Computer, > 20 GB RAM | Although processing can be carried out on most computers, higher capabilities will increase the speed of the processing |
Riferimenti
- Tyagi, S. Imaging intracellular RNA distribution and dynamics in living cells. Nature Methods. 6 (5), 331-338 (2009).
- Bao, G., Rhee, W. J., Tsourkas, A. Fluorescent probes for live-cell RNA detection. Annual Reviews of Biomedical Engineering. 11, 25-47 (2009).
- Mannack, L. V., Eising, S., Rentmeister, A. Current techniques for visualizing RNA in cells. F1000Research. 5, (2016).
- Larson, D. R., Zenklusen, D., Wu, B., Chao, J. A., Singer, R. H. Real-time observation of transcription initiation and elongation on an endogenous yeast gene. Science. 332 (6028), 475-478 (2011).
- Bertrand, E., et al. Localization of ASH1 mRNA particles in living yeast. Molecular Cell. 2 (4), 437-445 (1998).
- Garcia, J. F., Parker, R. MS2 coat proteins bound to yeast mRNAs block 5' to 3' degradation and trap mRNA decay products: implications for the localization of mRNAs by MS2-MCP system. RNA. 21 (8), 1393-1395 (2015).
- Bratu, D. P. Molecular beacons: Fluorescent probes for detection of endogenous mRNAs in living cells. Methods in Molecular Biology. 319, 1-14 (2006).
- Bratu, D. P., Cha, B. J., Mhlanga, M. M., Kramer, F. R., Tyagi, S. Visualizing the distribution and transport of mRNAs in living cells. Proceedings of the National Academy of Sciences of the Unites States of America. 100 (23), 13308-13313 (2003).
- Tyagi, S., Kramer, F. R. Molecular beacons: probes that fluoresce upon hybridization. Nature Biotechnology. 14 (3), 303-308 (1996).
- Chen, M., et al. A molecular beacon-based approach for live-cell imaging of RNA transcripts with minimal target engineering at the single-molecule level. Scientific Reports. 7 (1), 1550(2017).
- Liu, Y., et al. Multiplex detection of microRNAs by combining molecular beacon probes with T7 exonuclease-assisted cyclic amplification reaction. Analytical and Bioanalytical Chemistry. 409 (1), 107-114 (2017).
- Baker, M. B., Bao, G., Searles, C. D. In vitro quantification of specific microRNA using molecular beacons. Nucleic Acids Research. 40 (2), e13(2012).
- Ko, H. Y., et al. A color-tunable molecular beacon to sense miRNA-9 expression during neurogenesis. Scientific Reports. 4, 4626(2014).
- Vet, J. A., et al. Multiplex detection of four pathogenic retroviruses using molecular beacons. Proceedings of the National Academy of Sciences of the Unites States of America. 96 (11), 6394-6399 (1999).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Methods in Molecular Biology. 429, 209-224 (2008).
- Li, W. M., Chan, C. M., Miller, A. L., Lee, C. H. Dual Functional Roles of Molecular Beacon as a MicroRNA Detector and Inhibitor. Journal of Biological Chemistry. 292 (9), 3568-3580 (2017).
- Kuang, T., Chang, L., Peng, X., Hu, X., Gallego-Perez, D. Molecular Beacon Nano-Sensors for Probing Living Cancer Cells. Trends in Biotechnology. 35 (4), 347-359 (2017).
- Ban, K., et al. Non-genetic Purification of Ventricular Cardiomyocytes from Differentiating Embryonic Stem Cells through Molecular Beacons Targeting IRX-4. Stem Cell Reports. 5 (6), 1239-1249 (2015).
- Hadjinicolaou, A. V., Demetriou, V. L., Emmanuel, M. A., Kakoyiannis, C. K., Kostrikis, L. G. Molecular beacon-based real-time PCR detection of primary isolates of Salmonella Typhimurium and Salmonella Enteritidis in environmental and clinical samples. BMC Microbiology. 9, 97(2009).
- McLaughlin, J. M., Bratu, D. P. Drosophila melanogaster Oogenesis: An Overview. Methods in Molecular Biology. 1328, 1-20 (2015).
- Bastock, R., St Johnston, D. Drosophila oogenesis. Current Biology. 18 (23), R1082-R1087 (2008).
- Rongo, C., Gavis, E. R., Lehmann, R. Localization of oskar RNA regulates oskar translation and requires Oskar protein. Development. 121 (9), 2737-2746 (1995).
- Mhlanga, M. M., et al. In vivo colocalisation of oskar mRNA and trans-acting proteins revealed by quantitative imaging of the Drosophila oocyte. PLoS One. 4 (7), e6241(2009).
- Bayer, L. V., Omar, O. S., Bratu, D. P., Catrina, I. E. PinMol: Python application for designing molecular beacons for live cell imaging of endogenous mRNAs. bioRxiv. , (2018).
- Marras, S. A., Kramer, F. R., Tyagi, S. Efficiencies of fluorescence resonance energy transfer and contact-mediated quenching in oligonucleotide probes. Nucleic Acids Research. 30 (21), e122(2002).
- Bratu, D. P., Catrina, I. E., Marras, S. A. Tiny molecular beacons for in vivo mRNA detection. Methods in Molecular Biology. 714, 141-157 (2011).
- Dean, D. A. Preparation (pulling) of needles for gene delivery by microinjection. Cold Spring Harbor. 2006 (7), (2006).
- Alami, N. H., et al. Axonal transport of TDP-43 mRNA granules is impaired by ALS-causing mutations. Neuron. 81 (3), 536-543 (2014).
- Jackson, S. R., et al. Applications of Hairpin DNA-Functionalized Gold Nanoparticles for Imaging mRNA in Living Cells. Methods in Enzymology. 572, 87-103 (2016).
- Zimyanin, V. L., et al. In vivo imaging of oskar mRNA transport reveals the mechanism of posterior localization. Cell. 134 (5), 843-853 (2008).
- Catrina, I. E., Marras, S. A., Bratu, D. P. Tiny molecular beacons: LNA/2'-O-methyl RNA chimeric probes for imaging dynamic mRNA processes in living cells. ACS Chemical Biology. 7 (9), 1586-1595 (2012).
- Chen, A. K., Behlke, M. A., Tsourkas, A. Efficient cytosolic delivery of molecular beacon conjugates and flow cytometric analysis of target RNA. Nucleic Acids Research. 36 (12), e69(2008).
- Nitin, N., Santangelo, P. J., Kim, G., Nie, S., Bao, G. Peptide-linked molecular beacons for efficient delivery and rapid mRNA detection in living cells. Nucleic Acids Research. 32 (6), e58(2004).
- Chen, A. K., Behlke, M. A., Tsourkas, A. Avoiding false-positive signals with nuclease-vulnerable molecular beacons in single living cells. Nucleic Acids Research. 35 (16), e105(2007).
- Bevilacqua, P. C., Ritchey, L. E., Su, Z., Assmann, S. M. Genome-Wide Analysis of RNA Secondary Structure. Annual Review of Genetics. 50, 235-266 (2016).
- Mhlanga, M. M., Vargas, D. Y., Fung, C. W., Kramer, F. R., Tyagi, S. tRNA-linked molecular beacons for imaging mRNAs in the cytoplasm of living cells. Nucleic Acids Research. 33 (6), 1902-1912 (2005).
- Eliceiri, K. W., et al. Biological imaging software tools. Nature Methods. 9 (7), 697-710 (2012).
- Bolte, S., Cordelieres, F. P. A guided tour into subcellular colocalization analysis in light microscopy. Journal of Microscopy. 224 (Pt 3), 213-232 (2006).
- Trcek, T., et al. Drosophila germ granules are structured and contain homotypic mRNA clusters. Nature Commununications. 6, 7962(2015).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon