このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ライブショウジョウバエメラノガスター卵室における内因性mRNAの可視化と追跡
要約
ここでは、分子ビーコン、スピニングディスク共焦点顕微鏡、オープンソース分析を用いたライブショウジョウバエメラノガスター卵室における内因性mRNA人身売買の可視化、検出、分析、追跡のためのプロトコルを提示する。ソフトウェア。
要約
蛍光ベースのイメージング技術は、光顕微鏡の開発と組み合わせることで、細胞生物学者が生細胞イメージング研究を行う方法に革命を起こしました。RNAを検出する方法は、セミナル研究が遺伝子発現調節に部位特異的mRNA局在化を結合して以来、大幅に拡大してきました。動的mRNAプロセスは、mRNAを検出するアプローチを介して可視化することができ、分子挙動の動的範囲を捕捉するのに十分な速さである顕微鏡検査のセットアップと組み合わされます。分子ビーコン技術は、生細胞内の内因性転写物を直接検出できるハイブリダイゼーションベースのアプローチです。分子ビーコンは、ヘアピン状、内部的にクエンチされた、単一ヌクレオチド識別核酸プローブであり、これは、ユニークな標的配列へのハイブリダイゼーション時にのみ蛍光する。高度な蛍光顕微鏡と高解像度イメージングと組み合わせることで、mRNAの細胞内移動の空間的および時間的な追跡を行うことが可能になります。この技術は内因性転写物を検出できる唯一の方法ですが、細胞生物学者は、生細胞イメージングのためのそのようなプローブの設計が困難なため、この技術をまだ完全に受け入れていません。新しいソフトウェアアプリケーションPinMolは、生きている細胞内のmRNA標的領域に効率的にハイブリダイズするのに最適なプローブの強化され、迅速な設計を可能にします。さらに、高解像度のリアルタイム画像取得と現在のオープンソース画像解析ソフトウェアにより、洗練されたデータ出力が可能になり、mRNAのライフサイクルに関与する動的プロセスの基礎となる複雑さの詳細な評価が可能になります。
ここでは、ショウジョウバエメラメラノガスター卵室に分子ビーコンを設計し、提供するための包括的なプロトコルを提示します。内因性mRNAの直接的かつ非常に特異的な検出および可視化は、紡糸ディスク共焦点顕微鏡を介して行われる。イメージングデータは、氷のソフトウェアで物体検出とトラッキングを使用して処理および分析され、卵母細胞内の特殊な領域に輸送およびローカライズされるmRNAの動的な動きに関する詳細を取得します。
概要
空間的および時間的分解能で動的事象を可視化する細胞生物学研究は、蛍光ベースのライブ細胞イメージング技術の開発によって可能になった。現在、生体内mRNAビジュアライゼーションは、RNAアプタマータンパク質相互作用、有機色素および核酸プローブのRNAアプタマー誘導蛍光1、2に基づく技術を介して達成される。3.それらはすべて高い特異性、感度および信号対バックグラウンド比を提供する。しかし、RNAアプタマー中心のアプローチは広範な遺伝子操作を必要とし、トランスジーンはタンパク質または有機色素結合に必要な人工構造モチーフを持つRNAを発現するように設計される。例えば、MS2/MCPシステムは、バクテリオファージMS2コートタンパク質(MCP)に対する結合配列の複数のタンデム反復を含むRNA構築物を発現するトランスジーンの同時発現と、蛍光タンパク質をコードする別のトランスジーンを必要とする。MCP 4に融合,5.このような二次構造モチーフをRNAに添加することは、かさばる蛍光タンパク質と共に、天然RNAプロセスが6に影響を与えるかもしれないという懸念を提起している。この懸念に対処し、追加のユニークな利点を提供する技術は、核酸ベースのアプローチ、分子ビーコン(MB)です。MBは、内因性mRNAの多重検出、単一ヌクレオチド変動の判分、および標的mRNA7,8とのハイブリダイゼーションの高速運動学を可能にする。MBは、標的にハイブリダイズした後に蛍動体形成的な立体構造変化を受ける前に、クエンチングヘアピンフォールドに残るオリゴヌクレオチドプローブである(図1C)9。いくつかのグループは、非コードRNA(マイクロRNAおよびlncRNA)10、11、12、13、RNAレトロウイルス14および動的DNAタンパク質の両方を検出するためにMBを使用することに成功しました。インタラクション15.ゼブラフィッシュ胚16、ニューロン13、腫瘍組織17、心筋細胞18、サルモネラ菌など、様々な生物や組織でのイメージングに成功しています。19.
ここでは、生きているD.メラノガスター卵室における内因性mRNAの設計、送達および検出アプローチについて、活性分子輸送の動的範囲を捕捉するのに十分な速さである顕微鏡検査のセットアップと相まって説明する。D.メラノガスター卵室は、初期の生殖細胞分裂や母体遺伝子発現からセグメントボディプラン20の生成まで、幅広い発達研究のための理想的な多細胞モデルシステムとして機能してきました。 21.卵室は、簡単に単離され、大きく半透明であり、外生分析の時間に耐えることができるので、イメージング実験に非常に適しています。多くの研究は、積極的に翻訳される前に離散的な細胞領域への母体転写の非対称的な局在化に焦点を当ててきた。特に、オスカーmRNA局在化およびその後の卵母細胞の後極でのその後の翻訳は、致命的な二頭上胚表現型22を避けるために厳密に規制された方法で起こらなければならない。oskar mRNAは、ナース細胞と呼ばれる15個の生殖細胞に転写され、環管と呼ばれる細胞質の橋を通って積極的に卵母細胞に輸送され、成熟卵となり最終的に受精される生殖細胞(図1A)).オスカーmRNPとの間のタンパク質因子の動的な募集と交換に関して既に入手可能なかなりの量の情報は、その長距離細胞内旅行と共に、オスカーを研究する好ましい候補にするmRNAライフサイクルの多くのプロセス。MBは、mRNA局在化のプロセスに関する詳細を明らかにし、ショウジョウバエの発生時にmRNA輸送を制御するタンパク質因子の調節と機能を解読するのに役立ってきました。特に、看護師細胞にMBをマイクロ注入し、生細胞イメージング実験を行うことにより、内因性mRNAの追跡が可能である8,23。
ここで示すロードマップは、MBを用いてライブ細胞イメージング実験を行うことから、イメージングデータを取得すること、そのネイティブ細胞環境で内因性mRNAを追跡するためのデータ分析を実行することまで、完全なプロセスのステップを提供します。ステップは、独自のラボ設定内で他の組織/細胞タイプを扱う研究者のニーズを満たすために変更し、さらに最適化することができます。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. 生細胞イメージング用MBの設計
-
ターゲットRNA配列を折りたたみ、mfoldサーバからの「RNA形態」を用いてmRNAターゲットの二次構造を予測する(http://unafold.rna.albany.edu/?q=mfold/RNA-Folding-Form)。
- FASTA形式でターゲットシーケンスを貼り付け/アップロードし、5%または10%のサブ最適性(MFE値の5%または10%以内の自由エネルギーを持つ構造)を選択し、それに応じて計算された折りたたみの最大数を調整します(例えば、10%の場合は大きい)。サブオプティマイティ)。
注: MB を設計する際に最適でない二次構造を含めることで、最小自由エネルギー(MFE)構造のみの予測よりも柔軟または剛性が高いターゲットmRNA内の領域の同定が可能となり、生細胞イメージングに適したMBの全体的な設計。 - 800ヌクレオチド(nt)のmRNAターゲットの「即時ジョブ」を選択するか、801~8,000 ntのmRNA長さの「バッチ・ジョブ」を選択します。
- FASTA形式でターゲットシーケンスを貼り付け/アップロードし、5%または10%のサブ最適性(MFE値の5%または10%以内の自由エネルギーを持つ構造)を選択し、それに応じて計算された折りたたみの最大数を調整します(例えば、10%の場合は大きい)。サブオプティマイティ)。
-
ステップ1.1で得られた「ssカウント」ファイルを、目的のパラメータを持つPinMolプログラム(https://bratulab.wordpress.com/software/)の入力として使用し、mRNAターゲット用のいくつかのMBを設計する(PinMolプログラムの使用法を説明するチュートリアルを参照)。24 https://bratulab.wordpress.com/tutorial-pinmol-mac/)で)
- BLAST分析を実行して選択されたMBの特異性を決定する:適切なデータベースで「ブラストン」を使用する(例えば、oskar mRNA特異的MBは「refseq-rna」データベースおよびショウジョウバエメラノガスター生物を使用する)。
- mRNA標的の組織特異的発現(例えば、oskar mRNA Flybase> ハイスループット発現データ> FlyAtlas解剖学マイクロアレイまたはmodENCODE解剖学RNA-Seq;http://flybase.org/reports/FBgn0003015)を同定し、任意の肯定的なBLASTヒット。目的の組織/細胞でも発現される他のmRNAとの>50%クロス相同を示すプローブを排除します。
- ライブセルイメージングを実行するために利用可能な顕微鏡検査のセットアップに適した蛍光素とクエンチャーのペアを選択します(例えばCy5/BHQ2)25。
2. MB合成、精製、特性分類
- 前述の7、または商用プロバイダからのサービスとして社内合成と精製を使用して、1~5MB(上記注を参照)を合成および精製し、次のラベリングスキームを使用して[5'(C3またはC6リンカー)-(2'-O)-メチルMB配列)-(クエンチャー)3']。リバースフェーズHPLCを使用して、社内で、または商用プロバイダのサービスを使用してMBを浄化します。
注:自動プローブ合成に使用されるリンアミドは、2'-O-メチルリボヌクレオチド修飾を持っている必要があります。また、連結核酸(LNA)と2'-O-メチル修飾のキメラを使用して、より短いMBとその標的mRNA26との間のハイブリッドの安定性を高めることができる。 - 標的RNA領域の配列に一致し、従ってMBのプローブ領域に相補的であるDNAオリゴヌクレオチドを合成し、インビトロキャラクタリゼーションで使用する(上記の手順2.3から2.5;上記の注記を参照)。DNA-オリゴヌクレオチド標的模倣を伴うMBのハイブリダイゼーションを最大化し、標的mRNA配列に見られるように4つの追加ヌクレオチドをDNA標的の各端に含む。
注:標的配列を検出するMBの効率のより厳密な特性化は、相補的なDNAオリゴヌクレオチド8の代わりにインビトロ合成RNA標的を用いて行うことができる。 - MB単独の熱変性を行い、その融解温度(Tm)を測定し、MBが生理的温度で所望のヘアピン形状を想定していることを確認する。我々は60と90 °Cの間のTm値を観察した。
- DNAオリゴヌクレオチド標的の存在下でMBの熱変変化を行い、前述の7としてMB:DNA標的ハイブリッドのTmを測定する。MB:DNAハイブリッドには55~60°CのTmが望まれています。
- 対応するDNAオリゴヌクレオチド標的とのインビトロハイブリダイゼーション反応を行い、生理学的温度でのMB:DNAハイブリッド形成の効率を決定し、前述の7.DNA標的模倣による高速ハイブリダイゼーション動態が望まれるが、DNA標的との高いハイブリダイゼーション効率を示さないMBは、インビトロおよび/またはインビボにおける標的mRNAとのより良い性能を持つ可能性がある。
3. マイクロインジェクションのための個々の卵室の解剖と準備
- 新鮮な酵母ペーストで2〜3日間、新しく孵化した雌を飼育します。
- CO2パッド上のハエを麻酔し、細かいピンセット(dumont#5)を使用して、ガラスカバースリップ上のハロカーボンオイル700の滴に1-2メスを転送します。
- ピンセットのペアを使用して、ステレオ顕微鏡の下で後ろ側を上にハエを向けます。後端に小さな切開を行い、女性の腹部を解剖し、卵巣のペアをオイルに静かに絞ります。
- 卵巣を新しいカバースリップのオイルドロップに置き植えます。オバリオレの最も若いステージを他のピンザーでつまみながら、1つのピンザーで1つの卵巣をそっと保持します。oskar mRNAは、中年発生後および中間発生後に積極的に局在化し(ステージ>7)、若い卵室(ステージ<7)は注入がより困難であり、長く生き残らない。個々の卵巣または卵室が分離され、垂直に整列されるまで、カバースリップ(下向きの動きで)をゆっくりとドラッグします。さらに、卵巣卵鎖から不要な段階を置き換えることによって、単一の卵室を分離する。
注:個別にからかわれた卵室が油に浮かばず、カバースリップに付着していることを確認します。これは、マイクロインジェクションと画像集録の両方で重要です。
4. 卵室のナース細胞へのMBのマイクロインジェクション
- MB溶液を、1つの分子ビーコン(例えばosk2216Cy5)を用いて、または異なるmRNAを標的とし、スペクトル的に異なる蛍光体で標識された2つのMBの混合物(例えばosk2216Cy5およびdrongo111Cy3)を用いて調記する。HybBuffer(50 mMトリス-HCl - pH 7.5、1.5 mM MgCl2および100 mM NaCl)で各MBの濃度200-300 ng/μLを使用してください。HybBufferでそれぞれ200 ng/μLで同じmRNAを標的とする同じ蛍次眼素で標識された4つのMBのカクテル(例えばosk82、osk1236、osk2216)。マイクロインジェクション用の針をロードする直前にMB溶液をスピンダウンします。
- 目的を選択します。40倍のオイルの目的は、適切な卵室を見つけ、マイクロインジェクションを行うための推奨されます。
- 解剖卵室でカバースリップを顕微鏡ステージに取り付けます。フォーカス位置で目的を持ち上げ、マイクロインジェクションのために適切に配向された中間から後期の発達段階で卵室を特定する(すなわち、AáP軸が針先に垂直で、看護師内で容易な注入を可能にする)卵母細胞に近位の細胞)。
- 約1 μL MB溶液(ステップ4.1参照)で針(市販またはハウス27で調製)をロードし、マイクロインジェクターに接続します。D.メラノガスター卵室のマイクロインジェクションの場合は、複数の看護師細胞に穿刺されないように、針(材料の表を参照)を顕微鏡段階(例えば30°)に向けます。
- 500-1,000 hPaの注入圧力および100-250 hPaの補償圧力が付いている注入器を組み立てなさい(材料のテーブルを参照)。
- ゆっくりとステージを移動して、卵室のオイルドロップボイドの領域を視野に入れます。
- マイクロマニピュレータジョイスティックを使用して、オイルドロップに針を静かに下げ、視野の周辺に向かってその先端に焦点を当てます。
- 針の先端から空気を取り除き、針からの流れがあることを確認するために「クリーン」機能を実行します。
- 家の位置に針を持参し、マイクロ注入される卵室に焦点を当て、その後、焦点に戻って、卵室の端の近くに配置します。
- 看護師細胞から卵胞細胞を分離する膜が焦点を合わせ始めるような目的のZ位置の微調整を行う。
- 看護師の細胞に針を挿入し、2-5 sの注射を行う。
- 針をそっと取り外し、ホームポジションに引き込みます。
- 画像取得(60-63xまたは100x)の目的倍率に目標を変更し、卵室に焦点を当て、取得を開始します。
5. スピニングディスク共焦点顕微鏡設定によるデータの取得
注:特定の設定については、資料の表を参照してください。
- 8-16 ビット・イメージの XYZCt スタックを記録する集録プロトコルを設定します (XYZ = ボリューム、C = チャネル、t = 時間)。
- 目的のチャネル(例えば、Cy5の場合は641nmレーザー、GFPの場合は491 nm)のレーザーラインを選択し、チャンネルを順番に取得します:最初に各チャンネルの蛍光信号を取得し、次にZ位置を変更して、適切なコローカリゼーション解析を可能にします。
- Z ステップ (0.3 μm など) を選択し、上下の Z限界 (-2 μm から 2 μm など) を選択します。
- 集録時間とサンプリングレートを入力します(例えば、1時間まで15〜30sごとに)。
- 取得を開始します。
6. トラッキングおよびコローカリゼーション情報の取得のためのデータ分析、およびビデオファイルの準備
- 画像処理
- バイオイメージ情報学のためのオープンコミュニティプラットフォームであるIcyをダウンロード、アンパック、オープン(http://icy.bioimageanalysis.org/)
- ステップ 5: イメージ/シーケンス >ファイル >開く XYZCt スタックを開きます。
- スタックを ImageJ に変換する: ImageJ> ツール > IJ に変換し、デタッチモードをオンにしました。
- サブスタックを作成する (さらに分析する Z ステップとタイム ポイントの範囲の選択): ImageJ>イメージ>スタック>ツール>サブスタックを作成...;目的のチャンネル、Zステップ、タイムポイントを選択します。
- サブスタックを TIFF ファイルとして保存する: ImageJ>ファイル>保存 As>Tiff...;このファイルを後続の手順に使用します。
- スプリットチャンネル: ImageJ>イメージ>カラー>スプリットチャンネル。
- 背景スタックを使用して背景を減算する: ImageJ>プロセス>画像計算機...、またはローリングボールオプションを使用して:ImageJ>プロセス>減算背景...、ローリングボール半径を選択します。選択した半径の画像をプレビューしてから、「同意する」を選択します。
注: バックグラウンド信号は、主にフルロフォアの不適切な消光によって発生します。信号:背景比(S:B)はMBの「明るさ」の指標としてよく使用され、MBおよびDNA標的オリゴヌクレオチドのインビトロハイブリダイゼーション実験から測定される。たとえば、MB osk1236 と osk2216 はそれぞれ S:B が ~81 と ~120 です。 - 各チャンネルの明るさとコントラストを調整する: ImageJ>Image>調整>明るさ/コントラスト、[適用]を選択します。
- 各チャネルを別々の TIFF ファイルとして保存する: ImageJ>ファイル>保存 As>Tiff....
- 2 つのチャネルをマージする: ImageJ>イメージ>カラー>チャンネルのマージ...チャンネルを選択します。新しいスタックを新しい TIFF ファイルとして保存します (手順 6.1.8を参照)。
- スポット検出とトラッキング
- 氷に戻す: ImageJ>ツール>氷に変換します。
- スケールバーは、スケールバープラグインがインストールされている場合、Icyへの変換時に自動的にスタック上にオーバーレイされます[プラグイン>セットアップ>オンラインプラグインを使用して検索]。必要に応じて、[インスペクタ]ウィンドウ(画面の右側)でスケールバーを編集します>レイヤータブ>[名前]>スケールバーを編集します。
- [レイヤー] タブ>[名前] から [スケール] バーの 「目」アイコンの選択を解除/無効にすると、元のスタックからスケール バーが削除されます。最終的なスタックで再アクティブ化できます。
- イメージ ウィンドウのメニュー バーから "カメラ" アイコンを使用してスクリーンショットを撮り、"現在のビューのスクリーンショットを撮る"、および File>Tiff....
- スポット感度パラメータが既に決定されている場合は、ステップ 6.2.7 に移動します。
- スポットの検出: 分析する画像またはスタックを持つウィンドウを選択し、検出&トラッキング>検出>スポットディテクタを選択し、設定パラメータを入力します。
- 入力の場合は、「現在のシーケンス入力検出」(デフォルト)を選択します。
- 前処理の場合は、[インスペクタ]ウィンドウ>シーケンスタブで番号を相互参照して「チャンネル0」(デフォルト)または目的のチャンネルを選択します。
- 検出器の場合は、「暗い背景の上に明るいスポットを検出する」を選択し、解析を実行するのに十分な Z スライスがスタックにない場合にのみ、「3D 用 2D ウェーブレットの強制使用」を使用します。スケールごとに「スケール」と「感度」を選択します(大きなスポットにスケールを追加します)。スケールと感度(検出の感度が高いほど、最大140は氷で示唆される)は試行錯誤変数であり、その後視覚的にチェックして決定する必要があります。
- 対象地域の場合は、「ROIfromSequence」(デフォルト)を使用します。
- フィルタリングの場合は、「フィルタリングなし」(デフォルト)を使用するか、「サイズフィルタリング」を選択して「受け入れられたオブジェクトの範囲(ピクセル単位)」を定義します。
- 出力: XLS または XML 出力設定を選択します (2007 MS Excel 以前を使用している場合は XML 形式を選択し、>65,000 スポットがあります)。スポット検出器の結果をトラッキング解析に使用する場合は、「スイミングプールにエクスポート」も選択します。
- スポットのすべてまたは大部分が検出されるまで、さまざまなスケール/感度値を使用してスポットを繰り返し検出します。すべての最後のパラメータを記録します。
- コローカリゼーション分析では、他のチャネルのスポット検出を繰り返します。
- スポットを追跡するには、[検出&トラッキング>スポットトラッキング>スポットトラッキング>ステップ6.2.6.からパラメータを使用してスポット検出器を実行する]を選択するか、プルダウンメニューを使用して既存のデータセットを選択します(このため、スポット検出器ウィンドウを保持します)。ステップ6.2.5から開きます)。「パラメータの推定」ボタンを押し、パラメータ推定ポップアップウィンドウで目的のターゲットモーションを選択します(例えば、「拡散と指向の両方」)。「トラッキングを実行」ボタンを押します。
- マルチチャネル スタックのスポットを追跡する際に、ステップ 6.2.6 および 6.2.7 の手順に従って、ステップ 6.2.7 から生成されたスタックから始めて、他のチャネルのスポット検出と追跡を繰り返します。
- トラックを視覚化するには、[検出&トラッキング>トラッキングマネージャ] - トラッキング実行の完了時に自動的に開きます。「カラートラックプロセッサ」で「有効にする」を選択し、トラックの色の表現を選択します。関連するトラックプロセッサは、「トラックプロセッサを追加...」を介してアクセスすることができます。プルダウンメニュー(例:「トラックプロセッサタイムクリップ」を選択し、「トラッククリッパー」ウィンドウを有効にし、現在の時点の前後に表示される検出の必要な数を選択します)。
- トラック情報を XML トラック ファイルとして保存する: 検出&トラッキング >トラッキング マネージャ >ファイルとして保存する...
- 画像ウィンドウのメニューバー「現在のビューのスクリーンショットを撮る」から「カメラ」アイコンを使用してスクリーンショットを撮って結果を保存します。スクリーンショットは、検出されたスポットやトラックと一緒に、インスペクタウィンドウで見つかった対応する目のアイコンをアクティブ/非アクティブ化するだけで撮影できます。>レイヤータブ>オーバーレイラッパー。
- タイムスタンプオーバーレイプラグインをインストールする: プラグイン>セットアップ>オンラインプラグイン>タイムスタンプオーバーレイ>インストール。
- タイムスタンプを追加する: プラグイン>タイムスタンプオーバーレイ (新規)タイムスタンプの配置と書式設定の手順については、ポップアップ ウィンドウ (画面の右下隅) の指示に従います。時間間隔は、インスペクタ ウィンドウ >シーケンス タブ>シーケンスプロパティ>編集で追加または変更できます。
- 別のスクリーンショットを撮って結果を保存します。画像を 1) Tiff 形式、2) AVI 形式として保存します。AVI 形式の場合、最初に RGB レンダリング (イメージ/シーケンス>レンダリング>RGB イメージ) に変換します。
- 画像を目的の向きに回転させる: インスペクタ ウィンドウ>シーケンスタブ>キャンバス>回転。
- 「現在のビューのスクリーンショットを撮る」によって回転画像を保存します。スケールバーの「目」アイコンが選択解除されていることを確認します。
- ROI を選択してトリミングする: [関心のある領域]を選択します>2D ROI>ROI シェイプを選択し、画像上に ROI を作成/描画します。画像/シーケンス>平面(XY)>高速トリミング。
- コローカリゼーション分析
- コローカライズ プロトコルを準備します。いくつかの例は、氷のウェブサイト(http://icy.bioimageanalysis.org/protocol/List)で提供されています(補足資料を参照)。
- ロードコローカリゼーションプロトコル: ツール>Scripting>プロトコル>ロード、および相互作用ブロック内のパラメータを調整します(例:「ウェーブレットスポット検出」ブロックの使用パラメータはステップ6.2.6で決定されます)。
- パーティクルのサイズをピクセル単位で測定し、コローカライズ距離を決定し、「コローカライザ」ブロックに「最大距離」として入力します。
注:ピクセル単位の粒子のサイズは、検出システムによって異なります。サイズを測定するには、単一のパーティクルにズームインし、信号の幅にまたがるピクセルを手動でカウントします。少なくとも3つの粒子からの測定値を平均化します。コローカライズに設定する最大距離は、パーティクルのサイズをピクセル単位で表します(これは、接触する 2 つのパーティクルの半径の最大合計を表します)。 - 必要に応じて、コローカライズ分析用に 1 つ以上の ROI を選択します: 関心のある領域>2D ROI>ROI シェイプ>イメージに ROI を描画します。
- トリミング ROI(上):イメージ/シーケンス>平面 (XY)>高速トリミング。
- コローカライズの実行: プロトコル エディタ ウィンドウ >選択したプロトコル タブ>実行。プロトコル エディタ ウィンドウの最後のブロックには、スポット検出に基づく全体的なコローカライズの割合が含まれますが、各時点の情報は [インスペクター] ウィンドウ > 出力タブで確認できます。
- ステップ 6.2.7(スポットを追跡)に従って、コローカライズされたパーティクルと単一のパーティクルを追跡します。
- 手順 6.2.16 で説明したように保存します。
Access restricted. Please log in or start a trial to view this content.
結果
PinMolを使用して、複数の MB を 1 つの mRNA ターゲットに対して設計できます (図 1B-C)。合成および精製後、選択されたMBはインビトロ分析を用いて特徴付け、比較される。
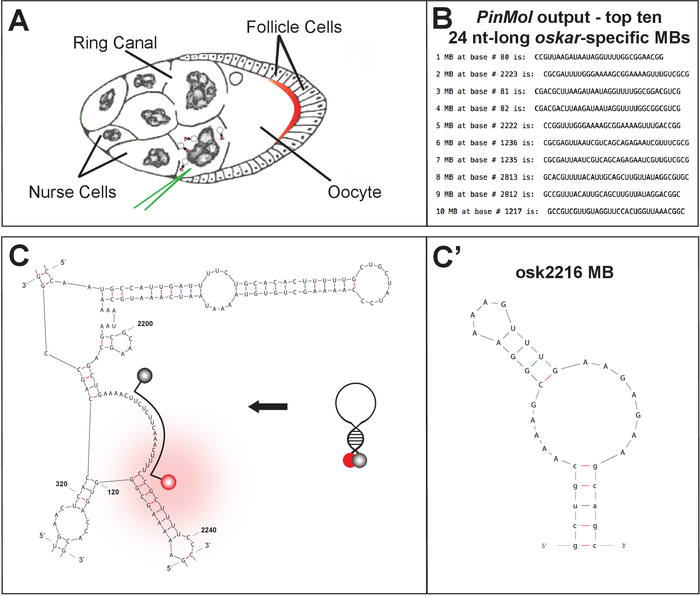
図1:内因性mRNAの生細胞イメージングのた?...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
ショウジョウバエの卵室における内因性mRNAの密有性のライブ可視化は、PinMolソフトウェアを使用して簡単に設計できる特定の、効率的な、ヌクレアーゼ耐性MBの使用に依存しています。MBは、標的mRNA内の一意の配列(好ましくは二次構造のない領域)を検出するように設計された特定のプローブであり、転写物の高度に解決された検出を可能にする。他の組織/細胞タイプにこの技?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者は開示する利益相反を持っていない。
謝辞
サルヴァトーレA.E.マラス(ラトガース大学公衆衛生研究所センター)が分子ビーコンの合成、標識、精製を行い、ダニエル・セント・ジョンストン(ケンブリッジ大学ガードン研究所)がオスカー-MS2/ MCP-GFPトランスジェニックフライストック。この作品は、国立科学財団キャリア賞1149738とDPBに専門スタッフ会議-CUNY賞によってサポートされました。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Spectrofluorometer | Fluoromax-4 Horiba-Jobin Yvon | n/a | Photon counting spectrofluorometer |
| Quartz cuvette | Fireflysci (former Precision Cells Inc.) | 701MFL | |
| Dumont #5 tweezer | World Precision Instruments | 501985 | Thin tweezers are very important to separate out the individual egg chambers |
| Halocarbon oil 700 | Sigma-Aldrich | H8898 | |
| Cover slip No.1 22 mm x 40 mm | VWR | 48393-048 | |
| Dissecting microscope | Leica MZ6 Leica Microsystems Inc. | n/a | |
| CO2 fruit fly anesthesia pad | Genesee Scienific | 59-114 | |
| Tris-HCL pH 7.5 | Sigma-Aldrich | 1185-53-1 | |
| Magnesium chloride | Sigma-Aldrich | 7791-18-6 | |
| NaCl | Sigma-Aldrich | 7647-14-5 | |
| Spinning disc confocal microscope | Leica DMI-4000B inverted microscope equipped with Yokogawa CSU 10 spinning disc Leica Microsystems Inc. | n/a | |
| Hamamatsu C9100-13 ImagEM EMCCD camera | Hamamatsu | n/a | |
| PatchMan NP 2 Micromanipulator | Eppendorf Inc. | 920000037 | |
| FemtoJet Microinjector | Eppendorf Inc. | 920010504 | |
| Injection needle: Femtotips II | Eppendorf Inc. | 930000043 | |
| Loading tip: 20 μL Microloader | Eppendorf Inc. | 930001007 | |
| Micro Cover glasses no. 1 or 1.5, 22 mm x 40 mm | VWR | 48393-026; 48393-172 | |
| Dry yeast | Any grocery store | n/a | |
| Computer, > 20 GB RAM | Although processing can be carried out on most computers, higher capabilities will increase the speed of the processing |
参考文献
- Tyagi, S. Imaging intracellular RNA distribution and dynamics in living cells. Nature Methods. 6 (5), 331-338 (2009).
- Bao, G., Rhee, W. J., Tsourkas, A. Fluorescent probes for live-cell RNA detection. Annual Reviews of Biomedical Engineering. 11, 25-47 (2009).
- Mannack, L. V., Eising, S., Rentmeister, A. Current techniques for visualizing RNA in cells. F1000Research. 5, (2016).
- Larson, D. R., Zenklusen, D., Wu, B., Chao, J. A., Singer, R. H. Real-time observation of transcription initiation and elongation on an endogenous yeast gene. Science. 332 (6028), 475-478 (2011).
- Bertrand, E., et al. Localization of ASH1 mRNA particles in living yeast. Molecular Cell. 2 (4), 437-445 (1998).
- Garcia, J. F., Parker, R. MS2 coat proteins bound to yeast mRNAs block 5' to 3' degradation and trap mRNA decay products: implications for the localization of mRNAs by MS2-MCP system. RNA. 21 (8), 1393-1395 (2015).
- Bratu, D. P. Molecular beacons: Fluorescent probes for detection of endogenous mRNAs in living cells. Methods in Molecular Biology. 319, 1-14 (2006).
- Bratu, D. P., Cha, B. J., Mhlanga, M. M., Kramer, F. R., Tyagi, S. Visualizing the distribution and transport of mRNAs in living cells. Proceedings of the National Academy of Sciences of the Unites States of America. 100 (23), 13308-13313 (2003).
- Tyagi, S., Kramer, F. R. Molecular beacons: probes that fluoresce upon hybridization. Nature Biotechnology. 14 (3), 303-308 (1996).
- Chen, M., et al. A molecular beacon-based approach for live-cell imaging of RNA transcripts with minimal target engineering at the single-molecule level. Scientific Reports. 7 (1), 1550(2017).
- Liu, Y., et al. Multiplex detection of microRNAs by combining molecular beacon probes with T7 exonuclease-assisted cyclic amplification reaction. Analytical and Bioanalytical Chemistry. 409 (1), 107-114 (2017).
- Baker, M. B., Bao, G., Searles, C. D. In vitro quantification of specific microRNA using molecular beacons. Nucleic Acids Research. 40 (2), e13(2012).
- Ko, H. Y., et al. A color-tunable molecular beacon to sense miRNA-9 expression during neurogenesis. Scientific Reports. 4, 4626(2014).
- Vet, J. A., et al. Multiplex detection of four pathogenic retroviruses using molecular beacons. Proceedings of the National Academy of Sciences of the Unites States of America. 96 (11), 6394-6399 (1999).
- Li, J., Cao, Z. C., Tang, Z., Wang, K., Tan, W. Molecular beacons for protein-DNA interaction studies. Methods in Molecular Biology. 429, 209-224 (2008).
- Li, W. M., Chan, C. M., Miller, A. L., Lee, C. H. Dual Functional Roles of Molecular Beacon as a MicroRNA Detector and Inhibitor. Journal of Biological Chemistry. 292 (9), 3568-3580 (2017).
- Kuang, T., Chang, L., Peng, X., Hu, X., Gallego-Perez, D. Molecular Beacon Nano-Sensors for Probing Living Cancer Cells. Trends in Biotechnology. 35 (4), 347-359 (2017).
- Ban, K., et al. Non-genetic Purification of Ventricular Cardiomyocytes from Differentiating Embryonic Stem Cells through Molecular Beacons Targeting IRX-4. Stem Cell Reports. 5 (6), 1239-1249 (2015).
- Hadjinicolaou, A. V., Demetriou, V. L., Emmanuel, M. A., Kakoyiannis, C. K., Kostrikis, L. G. Molecular beacon-based real-time PCR detection of primary isolates of Salmonella Typhimurium and Salmonella Enteritidis in environmental and clinical samples. BMC Microbiology. 9, 97(2009).
- McLaughlin, J. M., Bratu, D. P. Drosophila melanogaster Oogenesis: An Overview. Methods in Molecular Biology. 1328, 1-20 (2015).
- Bastock, R., St Johnston, D. Drosophila oogenesis. Current Biology. 18 (23), R1082-R1087 (2008).
- Rongo, C., Gavis, E. R., Lehmann, R. Localization of oskar RNA regulates oskar translation and requires Oskar protein. Development. 121 (9), 2737-2746 (1995).
- Mhlanga, M. M., et al. In vivo colocalisation of oskar mRNA and trans-acting proteins revealed by quantitative imaging of the Drosophila oocyte. PLoS One. 4 (7), e6241(2009).
- Bayer, L. V., Omar, O. S., Bratu, D. P., Catrina, I. E. PinMol: Python application for designing molecular beacons for live cell imaging of endogenous mRNAs. bioRxiv. , (2018).
- Marras, S. A., Kramer, F. R., Tyagi, S. Efficiencies of fluorescence resonance energy transfer and contact-mediated quenching in oligonucleotide probes. Nucleic Acids Research. 30 (21), e122(2002).
- Bratu, D. P., Catrina, I. E., Marras, S. A. Tiny molecular beacons for in vivo mRNA detection. Methods in Molecular Biology. 714, 141-157 (2011).
- Dean, D. A. Preparation (pulling) of needles for gene delivery by microinjection. Cold Spring Harbor. 2006 (7), (2006).
- Alami, N. H., et al. Axonal transport of TDP-43 mRNA granules is impaired by ALS-causing mutations. Neuron. 81 (3), 536-543 (2014).
- Jackson, S. R., et al. Applications of Hairpin DNA-Functionalized Gold Nanoparticles for Imaging mRNA in Living Cells. Methods in Enzymology. 572, 87-103 (2016).
- Zimyanin, V. L., et al. In vivo imaging of oskar mRNA transport reveals the mechanism of posterior localization. Cell. 134 (5), 843-853 (2008).
- Catrina, I. E., Marras, S. A., Bratu, D. P. Tiny molecular beacons: LNA/2'-O-methyl RNA chimeric probes for imaging dynamic mRNA processes in living cells. ACS Chemical Biology. 7 (9), 1586-1595 (2012).
- Chen, A. K., Behlke, M. A., Tsourkas, A. Efficient cytosolic delivery of molecular beacon conjugates and flow cytometric analysis of target RNA. Nucleic Acids Research. 36 (12), e69(2008).
- Nitin, N., Santangelo, P. J., Kim, G., Nie, S., Bao, G. Peptide-linked molecular beacons for efficient delivery and rapid mRNA detection in living cells. Nucleic Acids Research. 32 (6), e58(2004).
- Chen, A. K., Behlke, M. A., Tsourkas, A. Avoiding false-positive signals with nuclease-vulnerable molecular beacons in single living cells. Nucleic Acids Research. 35 (16), e105(2007).
- Bevilacqua, P. C., Ritchey, L. E., Su, Z., Assmann, S. M. Genome-Wide Analysis of RNA Secondary Structure. Annual Review of Genetics. 50, 235-266 (2016).
- Mhlanga, M. M., Vargas, D. Y., Fung, C. W., Kramer, F. R., Tyagi, S. tRNA-linked molecular beacons for imaging mRNAs in the cytoplasm of living cells. Nucleic Acids Research. 33 (6), 1902-1912 (2005).
- Eliceiri, K. W., et al. Biological imaging software tools. Nature Methods. 9 (7), 697-710 (2012).
- Bolte, S., Cordelieres, F. P. A guided tour into subcellular colocalization analysis in light microscopy. Journal of Microscopy. 224 (Pt 3), 213-232 (2006).
- Trcek, T., et al. Drosophila germ granules are structured and contain homotypic mRNA clusters. Nature Commununications. 6, 7962(2015).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved