Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Fotometría multifibra para registrar la actividad neuronal en animales en movimiento libre
En este artículo
Resumen
Este protocolo detalla cómo implementar y realizar grabaciones de fotometría multifibra, cómo corregir artefactos independientes del calcio y consideraciones importantes para la toma de imágenes de fotometría de doble color.
Resumen
Registrar la actividad de un grupo de neuronas en un animal que se mueve libremente es una tarea difícil. Además, a medida que el cerebro se disecciona en subgrupos funcionales más pequeños y más pequeños, se vuelve primordial registrar a partir de proyecciones y/o subpoblaciones genéticamente definidas de las neuronas. La fotometría de fibra es un enfoque accesible y potente que puede superar estos desafíos. Mediante la combinación de metodologías ópticas y genéticas, la actividad neuronal se puede medir en estructuras cerebrales profundas mediante la expresión de indicadores de calcio codificados genéticamente, que traducen la actividad neuronal en una señal óptica que se puede medir fácilmente. El protocolo actual detalla los componentes de un sistema de fotometría multifibra, cómo acceder a estructuras cerebrales profundas para entregar y recoger luz, un método para tener en cuenta los artefactos de movimiento y cómo procesar y analizar señales fluorescentes. El protocolo detalla consideraciones experimentales al realizar imágenes de color simple y doble, ya sea de fibras ópticas de implante simple o múltiple.
Introducción
La capacidad de correlacionar las respuestas neuronales con aspectos específicos del comportamiento de un animal es fundamental para entender el papel que desempeña un grupo particular de neuronas en la dirección o respuesta a una acción o estímulo. Dada la complejidad del comportamiento animal, con la infinidad de estados internos y estímulos externos que pueden afectar incluso a las acciones más simples, el registro de una señal con resolución de un solo ensayo dota a los investigadores de las herramientas necesarias para superarlas Limitaciones.
La fotometría de fibra se ha convertido en la técnica de elección para muchos investigadores en el campo de la neurociencia de sistemas debido a su relativa simplicidad en comparación con otras técnicas de grabación in vivo, su alta relación señal-ruido, y la capacidad de grabar en una variedad de paradigmas conductuales1,2,3,4,5,6,7,8. A diferencia de los métodos electrofisiológicos tradicionales, la fotometría es el enfoque óptico más comúnmente utilizado junto con indicadores de calcio codificados genéticamente (GECI, la serie GCaMP)9. Las GECI cambian su capacidad de fluorescencia en función de si están o no vinculadas al calcio. Debido a que la concentración interna de calcio en las neuronas está muy estrictamente regulada y los canales de calcio cerrados por voltaje se abren cuando una neurona dispara un potencial de acción, aumentos transitorios en la concentración interna de calcio, que resultan en aumentos transitorios en el capacidad de un GECI a la fluorescencia, puede ser un buen proxy para el disparo neuronal9.
Con la fotometría de fibra, la luz de excitación se dirige hacia abajo una fibra óptica delgada y multimodo en el cerebro, y una señal de emisión se recoge de nuevo a través de la misma fibra. Debido a que estas fibras ópticas son ligeras y flexibles, un animal puede moverse en gran medida sin obstáculos, haciendo que esta técnica sea compatible con una amplia gama de pruebas y condiciones conductuales. Algunas condiciones, como los movimientos rápidos o la flexión del cable de conexión de fibra óptica más allá del radio en el que puede mantener la reflexión interna total, pueden introducir artefactos de señal. Para desambiguar la señal del ruido, podemos explotar una propiedad de GCaMP conocida como el "punto isosbestico". Brevemente, con GCaMP, a medida que la longitud de onda de la luz de excitación se desplaza hacia la izquierda, su emisión en el estado ligado al calcio disminuye y la emisión en el estado de calcio no unido aumenta marginalmente. El punto en el que la intensidad relativa de estas dos emisiones es igual se denomina punto isosbestico. Cuando GCaMP se excita en este punto, su emisión no se ve afectada por los cambios en las concentraciones internas de calcio, y la varianza en la señal se debe más a menudo a la atenuación de la señal de sobredoblamiento del cordón de parche de fibra óptica o movimiento del tejido neural en relación con la fibra implantada.
La electrofisiología de una sola unidad sigue siendo el estándar de oro para las grabaciones in vivo que se mueven libremente debido a su resolución de nivel de una sola célula y de un solo pico. Sin embargo, puede ser difícil identificar la identidad molecular de las células que se registran, y el análisis post-hoc puede ser bastante laborioso. Si bien la fotometría de fibra no tiene resolución de una sola célula, sí permite a los investigadores hacer preguntas imposibles de abordar con técnicas tradicionales. Combinando estrategias virales con animales transgénicos, la expresión de GECI se puede dirigir a tipos neuronales definidos genéticamente para registrar la actividad neuronal definida por la población o la proyección, que se puede realizar mediante el seguimiento de la señal de calcio directamente en el axón terminales10,11. Además, mediante el implante de múltiples cánulas de fibra óptica, es posible monitorear simultáneamente la actividad neuronal de varias regiones cerebrales y vías en el mismo animal12,13.
En este manuscrito, describimos una técnica para fotometría de fibra única y multifibra, cómo corregir artefactos independientes del calcio y detallar cómo realizar grabaciones mono y doble color. También proporcionamos ejemplos de los tipos de preguntas que se pueden hacer y sus crecientes niveles de complejidad (véase la figura 1). La configuración de fotometría de fibra para grabaciones multifibra detalladas en este protocolo se puede construir utilizando una lista de materiales que se encuentran en https://sites.google.com/view/multifp/hardware(Figura 2).
Es esencial que el sistema esté equipado para longitudes de onda de excitación de 410 nm y 470 nm para la emisión de fluorescencia independiente del calcio y dependiente del calcio de GCaMP6 o sus variantes. Para configuraciones personalizadas o si no hay ningún software disponible para ejecutar el sistema, se puede utilizar el programa libre y de código abierto Bonsai (http://www.open-ephys.org/bonsai/). Alternativamente, la fotometría de fibra se puede ejecutar a través de MATLAB (por ejemplo, https://github.com/deisseroth-lab/multifiber)12 u otro lenguaje de programación14. El software y el hardware del sistema deben permitir la manipulación de los LED de 410 nm y 470 nm y de la cámara, la extracción de imágenes(Figura 2),y el cálculo de la intensidad fluorescente media en las regiones de interés (ROI) dibujadas alrededor de las fibras en las imágenes. La salida debe ser una tabla de valores de intensidad media registrados con los LED de 470 nm y 410 nm de cada fibra en el cable de conexión. Cuando se realizan experimentos multifibra, las fibras agrupadas de 400 m pueden limitar el movimiento de los ratones. En tales casos, se recomienda el uso de cables de parche de 200 m, que proporcionan más flexibilidad. También puede ser posible utilizar cables ficticios más pequeños durante el entrenamiento de ratones.
Es crucial poder extraer puntos de tiempo para eventos de interés durante la adquisición de fotometría de fibra. Si el sistema no proporciona fácilmente un sistema integrado para integrar TTLs para eventos específicos, una estrategia alternativa es asignar una marca de tiempo a los puntos de tiempo individuales registrados para alinearse con tiempos y eventos específicos durante el experimento. La marca de tiempo se puede realizar utilizando el reloj del ordenador.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Todos los experimentos se realizaron de acuerdo con los Comités Institucionales de Cuidado y Uso de Animales de la Universidad de California, San Diego, y la Guía Canadiense para el Cuidado y Uso de Animales de Laboratorio y fueron aprobados por la Universidad Laval Animal Protection Comité.
1. Alineación de la trayectoria óptica entre la cámara CMOS (semiconductor de óxido metálico complementario) y el cable de conexión individual o ramificado
- Afloje todos los tornillos del traductor de 5 ejes (11, Figura 2B).
- Tornillo en el cable de conexión (12, Figura 2B)al adaptador [SMA (subminiatura A) o FC (conector de fibra óptica)] que se fija al traductor de 5 ejes.
- Encienda la luz de excitación de 470 nm (1, Figura 2B)a baja potencia (100 w) y coloque la punta del cordón de conexión que apunta a una corredera de plástico autofluorescente. Esto no tiene ninguna relación con las grabaciones futuras, pero es únicamente para visualizar el proceso de alineación.
- Grabe desde la cámara CMOS (13, Figura 2B)en modo en vivo. Aumente la ganancia o ajuste la tabla de búsqueda (LUT) hasta que la imagen no sea completamente negra. El punto es poder ver una imagen en el punto focal del objetivo (10, Figura 2B).
- Avance el traductor de 5 ejes hacia el objetivo, asegurando que la luz de 470 nm esté centrada en la fibra en el extremo SMA o FC del cable de conexión, hasta que una imagen pueda ser resuelta en la cámara.
- Ajuste los ejes X e Y hasta que la imagen esté centrada y bien resuelta.
- Visualice la luz emitida desde el extremo de la férula del cable de conexión. Debe aparecer como un círculo isotrópico. Si se utiliza un cable de conexión ramificado, la cantidad de luz emitida en los extremos de la férula de cada cable de conexión debe ser similar. Si el círculo no es isotrópico o la luz emitida es desigual, ajuste el traductor de 5 ejes en el eje X-Y.
2. Configuración de ROI alrededor de fibras para la medición de la intensidad fluorescente media
- Encienda todas las luces de excitación para visualizar mejor las fibras. Ajuste la ganancia de la cámara de modo que no haya píxeles saturados y una imagen clara de las fibras esté presente.
- Grabe en vivo o tome una imagen preliminar.
- Dibuje los ROI alrededor de las fibras y guárdelos para la medición de los valores medios de intensidad durante las grabaciones (Figura 2A).
- Para múltiples grabaciones de fibra, pruebe la independencia en las señales.
- Grabación en vivo de todas las fibras.
- Apunte una fibra hacia una fuente de luz y toque con un dedo. Las fluctuaciones muy grandes deben ocurrir únicamente en ese canal (fuga aceptable 1:1000).
- Si las señales no son independientes, vuelva a dibujar los ROC más conservadores y repita la prueba de independencia.
- Para etiquetar y realizar un seguimiento de qué ROI corresponde a qué fibra, cinta de color o esmalte de uñas se puede aplicar al extremo de las fibras. Tome una foto antes del inicio de cualquier experimento como un recordatorio secundario.
3. Configuración de la arena de grabación
- Cuelgue el cable de conexión por encima de la arena con soportes, abrazaderas o soportes.
- Asegúrese de que el animal pueda moverse libremente por toda la arena, desinhibido por la longitud de la fibra.
- Si se utiliza una caja de operandos o un campo abierto, asegúrese de que el cordón de parche será capaz de llegar al animal con una flexión mínima. Si esto requiere un pinchazo en la nariz, asegúrese de que hay suficiente espacio en la parte superior para evitar la flexión de la fibra. Evite cualquier flexión o torsión excesiva del cable de conexión.
4. Grabaciones in vivo
NOTA: El procedimiento de implantación de cánula de fibra óptica para experimentos de fotometría de fibra es idéntico al procedimiento de optogenética descrito en Sparta et al15. Recomendamos el uso de cemento dental (ver Tabla de Materiales),que proporciona un fuerte anclaje de la tapa principal al hueso del cráneo. El cemento dental será particularmente útil en los casos en que no se puedan utilizar tornillos de anclaje.
- Inspeccione visualmente el extremo distal de las fibras del cordón de parche por ojo y con un microscopio de minifibra. Si la superficie de las fibras está rayada, vuelva a pulir las fibras con película de pulido/lapeado de fibra con grano fino (1 m y 0,3 m).
- Limpie los extremos distales del cable de conexión con un 70% de etanol y un aplicador de punta de algodón.
- Limpie las cánulas de fibra óptica con un 70% de etanol y un aplicador de punta de algodón.
- Conecte el extremo de la férula del cable de conexión a la fibra implantada utilizando una funda de cerámica dividida cubierta con un tubo retráctil negro. Durante la conexión, asegúrese de que el manguito esté apretado, de lo contrario utilice un manguito nuevo.
NOTA: Habrá una gran cantidad de pérdida de señal si hay espacio entre la férula del cable de conexión y el implante, y las grabaciones no funcionarán. - Permita que el animal se recupere durante unos minutos antes del inicio de las pruebas de comportamiento.
- Comience a grabar la señal óptica y ejecute el experimento.
- Durante la grabación, mantenga un ojo cuidadoso en el seguimiento en vivo para garantizar grabaciones de calidad. Se espera que la señal disminuya rápidamente en función del tiempo en los primeros 2 minutos de grabación. Este efecto es causado por la caída del LED mediada por calor, por lo que el aumento en el calor aumenta la resistencia del elemento óptico.
- Si se produce un salto en la señal que excede la cinética de encendido/apagado de GCaMP, esto es a menudo una indicación de que el manguito no está lo suficientemente apretado y el espacio entre el cable de conexión y el implante está cambiando. En este caso, detenga el experimento y vuelva a conectar el animal con una nueva manga.
5. Análisis de datos de fotometría de fibra
NOTA: Este es un método para el análisis de datos que funciona bien para la mayoría de las grabaciones. Sin embargo, se pueden implementar enfoques alternativos. El código de ejemplo para el análisis de datos se puede encontrar aquí: https://github.com/katemartian/Photometry_data_processing.
- Extraer valores medios de intensidad de fluorescencia registrados a partir de 470 nm (Int470) y 410 nm (Int410) LED, correspondientes a cada fibra individual.
- Suaviza cada señal usando un algoritmo de media móvil(Figura 3A).
- Realizar la corrección de línea base de cada señal(Figura 3A y 3B) utilizando el algoritmo de mínimos cuadrados penalizados (https://github.com/zmzhang/airPLS AirPLS) de rectificación iterativa adaptable (https://github.com/zmzhang/airPLS) para eliminar la pendiente y la frecuencia baja fluctuaciones en las señales.
- Estandarizar cada señal utilizando el valor medio y la desviación estándar(Figura 3C):
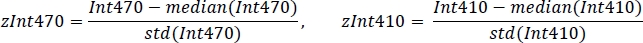
- Utilizando la regresión lineal robusta no negativa, ajuste las señales estándares zInt410 a zInt470 (Figura 3D) a la función de regresión:
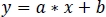
- Utilice los parámetros de la regresión lineal(a , b) para encontrar nuevos valores de zInt410 ajustados a zInt470 (fitInt410, Figura 3D,E):
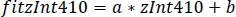
- Utilice los parámetros de la regresión lineal(a , b) para encontrar nuevos valores de zInt410 ajustados a zInt470 (fitInt410, Figura 3D,E):
- Calcular el dF/F normalizado (z dF/F) (Figura 3F):
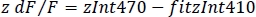
6. Grabaciones simultáneas de doble color
- Añadir al sistema de fotometría un LED de 560 nm para excitar el sensor de calcio fluorescente rojo y los espejos y filtros dicroicos apropiados (véase Kim et al., 2016 para una descripción detallada)12.
- Agregue un divisor de imagen entre el objetivo y la cámara CMOS para separar las longitudes de onda de emisión verde y roja (consulte la figura 5). El divisor de imagen formará dos imágenes reflejadas en el sensor de la cámara, correspondientes a las señales rojas y verdes (por ejemplo, un cable de conexión con 3 ramas creará una imagen con 6 fibras).
- Dibuja los ROI alrededor de todas las fibras en ambos colores como se detalló anteriormente. Asegúrese de identificar claramente cada ROI con la fibra y el canal correspondientes (verde y rojo)(Figura 4A).
- Desencadenar excitación simultánea con LEDs de 470 nm y 560 nm y alternarlos con LED de 410 nm(Figura 5A).
7. Análisis de datos de doble color
- Siga los pasos de la Sección 5 para encontrar fitInt410 para la señal Int470 y calcular z dF/F.
- Debido a que el punto isosbestico para los GECI desplazados en rojo es generalmente desconocido, la señal registrada con 410 nm LED en el canal verde se puede utilizar para la corrección del movimiento a través de ambos canales. Siga los pasos de la Sección 5 para encontrar fitInt410 para la señal Int560 y calcular z dF/F.
Access restricted. Please log in or start a trial to view this content.
Resultados
Los correlatos neuronales de las respuestas conductuales pueden variar dependiendo de una variedad de factores. En este ejemplo, utilizamos fotometría de fibra in vivo para medir la actividad de los terminales de axón desde el área hipotalámica lateral (LHA) que terminan en la habenula lateral (LHb). Los ratones de tipo salvaje fueron inyectados con un virus asociado a adeno (AAV) que codificaba GCaMP6s (AAV-hSyn-GCaMP6s) en la LHA y se implantó una fibra óptica con la punta inmediatamente por encima del LHb
Access restricted. Please log in or start a trial to view this content.
Discusión
La fotometría de fibra es un enfoque accesible que permite a los investigadores registrar la dinámica de calcio a granel de poblaciones neuronales definidas en animales que se mueven libremente. Este método se puede combinar con una amplia gama de pruebas de comportamiento, incluyendo tareas "pesadas de movimiento" tales como pruebas de natación forzada2, miedo-acondicionamiento18, interacciones sociales1,4, y o...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Sage Aronson es el CEO y fundador de Neurophotometrics Ltd., que vende sistemas de fotometría multifibra.
Agradecimientos
Este trabajo fue apoyado por una subvención del Consejo de Investigación de Ciencias Naturales e Ingeniería de Canadá (NSERC: RGPIN-2017-06131) a C.P. C. P. es un FRSQ Chercheur-Boursier. También agradecemos al Plateforme d'Outils Moléculaires (https://www.neurophotonics.ca/fr/pom) la producción de los vectores virales utilizados en este estudio.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 1/4"-20 Stainless Steel Cap Screw, 1" Long | Thorlabs | SH25S100 | |
| 1/4"-20 Stainless Steel Cap Screw, 1/2" Long | Thorlabs | SH25S050 | |
| 1/4"-20 Stainless Steel Cap Screw, 3/8" Long | Thorlabs | SH25S038 | |
| 1000 µm, 0.50 NA, SMA-SMA Fiber Patch Cable | Thorlabs | M59L01 | |
| 12.7 mm Optical Post | Thorlabs | TR30/M | |
| 12.7 mm Pedestal Post Holder | Thorlabs | PH20EM | |
| 15 V, 2.4 A Power Supply Unit with 3.5 mm Jack Connector for T-Cube | Thorlabs | KPS101 | |
| 20x objective | Thorlabs | RMS20X | #10 in Figure 2, #11 in Figure 5 |
| 30 mm Cage Cube with Dichroic Filter Mount | Thorlabs | CM1-DCH/M | #8-9 in Figure 2, #8-10 in Figure 5 |
| 405 nm LED | Doric Lenses | CLED_405 | #2 in Figure 2 |
| 410 nm bandpass filter | Thorlabs | FB410-10 | #5 in Figure 2; #7 in Figure 5 |
| 465 nm. LED | Doric Lenses | CLED_465 | #1 in Figure 2 |
| 470 nm bandpass filter | Thorlabs | FB470-10 | #4 in Figure 2; #6 in Figure 5 |
| 560 nm bandpass filter | Semrock | FF01-560/14-25 | #5 in Figure 5 |
| 560 nm LED | Doric Lenses | CLED_560 | #1 in Figure 3 |
| 5-axis kinematic Mount | Thorlabs | K5X1 | #11 in Figure 2, #12 in Figure 5 |
| Achromatic Doublet | Thorlabs | AC254-035-A-ML | #7 in Figure 2 |
| Adaptor for 405 collimator | Thorlabs | AD11F | #3 in Figure 2; #4 in Figure 5 |
| Adaptor for ajustable collimator | Thorlabs | AD127-F | #3 in Figure 2; #4 in Figure 5 |
| Aluminum Breadboard | Thorlabs | MB1824 | |
| Clamping Fork | Thorlabs | CF125 | |
| Cube connector | Thorlabs | CM1-CC | |
| Dual 493/574 dichroic | Semrock | FF493/574-Di01-25x36 | #10 in Figure 5 |
| Emission filter for GCaMP6 | Semrock | FF01-535/22-25 | #6 in Figure 2 |
| Enclosure with Black Hardboard Panels | Thorlabs | XE25C9 | |
| Externally SM1-Threaded End Cap for Machining | Thorlabs | SM1CP2M | |
| Fast-change SM1 Lens Tube Filter Holder | Thorlabs | SM1QP | #4-6 in Figure 2, #5-7 in Figure 5 |
| Fixed Collimator for 405 nm light | Thorlabs | F671SMA-405 | #3 in Figure 2; #4 in Figure 5 |
| Fixed collimator for 470 and 560 nm light | Thorlabs | F240SMA-532 | #3 in Figure 2; #4 in Figure 5 |
| Green emission filter | Semrock | FF01-520/35-25 | In light beam splitter |
| High-Resolution USB 3.0 CMOS Camera | Thorlabs | DCC3260M | #13 in Figure 2, #15 in Figure 5 |
| Light beam splitter | Neurophotometrics | SPLIT | #14 in Figure 5 |
| Longpass Dichroic Mirror, 425 nm Cutoff | Thorlabs | DMLP425R | #8 in Figure 2, #9 in Figure 5 |
| Longpass Dichroic Mirror, 495 nm Cutoff | Semrock | FF495-Di03 | #9 in Figure 2, #8 in Figure 5 |
| Metabond dental cement | C&B | ||
| M8 - M8 cable | Doric Lenses | Cable_M8-M8 | |
| Optic fiber cannulas | Doric Lenses | Need to specify that these will be used to photometry experiments requiring low autofluorescence | |
| Optic fiber Patchcords | Doric Lenses | Need to specify that these will be used to photometry experiments requiring low autofluorescence | |
| Red emission filter | Semrock | FF01-600/37-25 | In light beam splitter |
| T7 LabJack | LabJack | ||
| T-cube LED Driver | Thorlabs | LEDD1B | |
| USB 3.0 I/O Cable, Hirose 25, for DCC3240 | Thorlabs | CAB-DCU-T3 |
Referencias
- Gunaydin, L. A., et al. Natural Neural Projection Dynamics Underlying Social Behavior. Cell. 157 (7), 1535-1551 (2014).
- Proulx, C. D., et al. A neural pathway controlling motivation to exert effort. Proceedings of the National Academy of Sciences of the United States of America. 115 (22), 5792-5797 (2018).
- Muir, J., et al. In Vivo Fiber Photometry Reveals Signature of Future Stress Susceptibility in Nucleus Accumbens. Neuropsychopharmacology. 43 (2), 255-263 (2017).
- Wang, D., et al. Learning shapes the aversion and reward responses of lateral habenula neurons. eLife. 6, (2017).
- de Jong, J. W., et al. A Neural Circuit Mechanism for Encoding Aversive Stimuli in the Mesolimbic Dopamine System. Neuron. 101 (1), 133-151 (2018).
- Lerner, T. N., et al. Intact-Brain Analyses Reveal Distinct Information Carried by SNc Dopamine Subcircuits. Cell. 162 (3), 635-647 (2015).
- Calipari, E. S., et al. In vivo imaging identifies temporal signature of D1 and D2 medium spiny neurons in cocaine reward. Proceedings of the National Academy of Sciences of the United States of America. 113 (10), 2726-2731 (2016).
- González, A. J., et al. Inhibitory Interplay between Orexin Neurons and Eating. Current Biology. 26 (18), 2486-2491 (2016).
- Chen, T. -W., et al. Ultrasensitive fluorescent proteins for imaging neuronal activity. Nature. 499 (7458), 295-300 (2013).
- Barker, D. J., et al. Lateral Preoptic Control of the Lateral Habenula through Convergent Glutamate and GABA Transmission. Cell Reports. 21 (7), 1757-1769 (2017).
- Siciliano, C. A., Tye, K. M. Leveraging calcium imaging to illuminate circuit dysfunction in addiction. Alcohol. 74, 47-63 (2018).
- Kim, C. K., et al. Simultaneous fast measurement of circuit dynamics at multiple sites across the mammalian brain. Nature Methods. 13 (4), 325-328 (2016).
- Sych, Y., Chernysheva, M., Sumanovski, L. T., Helmchen, F. High-density multi-fiber photometry for studying large-scale brain circuit dynamics. Nature Methods. 16 (6), 553-560 (2019).
- Akam, T., Walton, M. E. pyPhotometry: Open source Python based hardware and software for fiber photometry data acquisition. Scientific Reports. 9 (1), 3521(2019).
- Sparta, D. R., et al. Construction of implantable optical fibers for long-term optogenetic manipulation of neural circuits. Nature Protocol. 7 (1), 12-23 (2011).
- Stamatakis, A. M., et al. Lateral Hypothalamic Area Glutamatergic Neurons and Their Projections to the Lateral Habenula Regulate Feeding and Reward. The Journal of Neuroscience. 36 (2), 302-311 (2016).
- Tervo, G. D., et al. A Designer AAV Variant Permits Efficient Retrograde Access to Projection Neurons. Neuron. 92 (2), 372-382 (2016).
- Yu, K., da Silva, P., Albeanu, D. F., Li, B. Central Amygdala Somatostatin Neurons Gate Passive and Active Defensive Behaviors. The Journal of Neuroscience. 36 (24), 6488-6496 (2016).
- Falkner, A. L., Grosenick, L., Davidson, T. J., Deisseroth, K., Lin, D. Hypothalamic control of male aggression-seeking behavior. Nature Neuroscience. 19 (4), 596-604 (2016).
- Ren, J., et al. Anatomically Defined and Functionally Distinct Dorsal Raphe Serotonin Sub-systems. Cell. 175 (2), 472-487 (2018).
- Barnett, L. M., Hughes, T. E., Drobizhev, M. Deciphering the molecular mechanism responsible for GCaMP6m’s Ca2+-dependent change in fluorescence. PLOS ONE. 12 (2), 0170934(2017).
- Sun, F., et al. A Genetically Encoded Fluorescent Sensor Enables Rapid and Specific Detection of Dopamine in Flies, Fish, and Mice. Cell. 174 (2), 481-496 (2018).
- Patriarchi, T., et al. Ultrafast neuronal imaging of dopamine dynamics with designed genetically encoded sensors. Science. 360 (6396), (2018).
- Feng, J., et al. A Genetically Encoded Fluorescent Sensor for Rapid and Specific In Detection of Norepinephrine. Neuron. 102 (4), 745-761 (2019).
- Akerboom, J., et al. Genetically encoded calcium indicators for multi-color neural activity imaging and combination with optogenetics. Frontiers in Molecular Neuroscience. 6, 1-29 (2013).
- Dana, H., et al. Sensitive red protein calcium indicators for imaging neural activity. eLife. 5, (2016).
- Wang, H., Jing, M., Li, Y. Lighting up the brain: genetically encoded fluorescent sensors for imaging neurotransmitters and neuromodulators. Current Opinion in Neurobiology. 50, 171-178 (2018).
- Lu, L., et al. Wireless optoelectronic photometers for monitoring neuronal dynamics in the deep brain. Proceedings of the National Academy of Sciences. 115 (7), 1374-1383 (2018).
- Jennings, J. H., et al. Visualizing Hypothalamic Network Dynamics for Appetitive and Consummatory Behaviors. Cell. 160 (3), 516-527 (2014).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados