Method Article
Caracterización funcional de las ligasinas de ubiquitina RING-Tipo E3 In Vitro y In Planta
En este artículo
Resumen
El objetivo de este manuscrito es presentar un esquema para los estudios bioquímicos y funcionales completos de las ligasinas de ubiquitina tipo RING E3. Esta canalización de varios pasos, con protocolos detallados, valida una actividad enzimática de la proteína probada y demuestra cómo vincular la actividad a la función.
Resumen
La ubiquitinación, como modificación posttranslacional de las proteínas, desempeña un importante papel regulador en la homeostasis de las células eucariotas. La unión covalente de 76 modificadores de ubiquitina de aminoácidos a una proteína diana, dependiendo de la longitud y topología de la cadena de poliubiquitina, puede resultar en diferentes resultados que van desde la degradación de las proteínas hasta los cambios en la localización y/o actividad de la proteína modificada. Tres enzimas catalizan secuencialmente el proceso de ubiquitinación: enzima activadora de ubiquitina E1, enzima conjugada de ubiquitina E2 y ligasa de ubiquitina E3. La ubiquitina ligaa E3 determina la especificidad del sustrato y, por lo tanto, representa un tema de estudio muy interesante. Aquí presentamos un enfoque integral para estudiar la relación entre la actividad enzimática y la función de la ligasa de ubiquitina tipo RING E3. Este protocolo de cuatro pasos describe 1) cómo generar un mutante deficiente de la ligasa E3 a través de mutagénesis dirigida por el sitio dirigida al dominio RING conservado; 2–3) cómo examinar la actividad de ubiquitinación tanto in vitro como en planta; 4) cómo vincular esos análisis bioquímicos a la importancia biológica de la proteína probada. La generación de un mutante con deficiencia de ligasa E3 que todavía interactúa con su sustrato pero que ya no lo ubiquitiniza para la degradación facilita la prueba de interacciones enzima-sustrato in vivo. Además, la mutación en el dominio RING conservado a menudo confiere un fenotipo negativo dominante que se puede utilizar en estudios funcionales de eliminación como un enfoque alternativo a un enfoque de interferencia de ARN. Nuestros métodos fueron optimizados para investigar el papel biológico del efector de nematodos parásitos de la planta RHA1B, que secuestra el sistema de ubiquitinación del huésped en las células vegetales para promover el parasitismo. Con ligera sin modificaciones del sistema de expresión in vivo, este protocolo se puede aplicar al análisis de cualquier ligadura E3 tipo RING independientemente de sus orígenes.
Introducción
La gran mayoría de las ligasas de ubiquitina E3 pertenecen a proteínas de tipo RING (Really Interesting New Gene). El dominio RING-finger fue identificado originalmente por Freemont et al. 1 y funcionalmente descrito como un dominio que media la interacción proteína-proteína2. El dedo canónico RING es un tipo especial de dominio de coordinación de zinc definido como una secuencia de consenso de ocho Cys (C) conservados y su (H) espaciados específicamente por otros residuos de aminoácidos (X), C-X2-C-X9–39-C-X1–3-H-X2–3-C/H-X2-C-X4–48-C-X2-C. Dos iones Zn2+ se estabilizan mediante residuos de núcleo C y H a través de una topología única de "cross-brace" con C1/C2 y C/H5/C6 coordinando el primer ion Zn2+, mientras que C3/H4 y C7/C8 enlazan el segundo(Figura 1A)3,4. Dependiendo de la presencia de C o H en el quinto sitio de coordinación Zn2+,se definieron dos subclases canónicas de proteínas RING-finger: C3HC4 y C3H2C3 (RING-HC y RING-H2, respectivamente). Debido a que el dominio RING de la ligasa de ubiquitina E3 media la interacción entre las enzimas y sustratos conjugantes E2, se ha demostrado que la mutación de estos residuos esenciales de C y H interrumpe la actividad de la ligasa5. Se han descrito otras cinco subclases menos comunes de ligasligas RING E3 (RING-v, RING-C2, RING-D, RING-S/T y RING-G)6. Las ligasas de ubiquitina tipo RING E3 se pueden subdividir aún más en enzimas E3 simples y complejas. La sencilla subunidad única RING E3 ligases contiene tanto el sitio de reconocimiento de sustrato sy el dominio RING de unión E2. Por el contrario, el complejo multisubunidad RING tipo E3 o bien recluta el sustrato o media la unión del e2-ubiquitina intermedia al complejo E3. El dominio RING de residuos de Lys que sirve como un sitio o sitios de unión a la ubiquitina primaria para la autoubiquitinación también podrían ser importantes para la actividad de la ligasa E3.
No todas las proteínas que contienen RING funcionan como ligases E3. Por lo tanto, la predicción bioinformática del dominio RING-finger y la capacidad de ubiquitinación de proteínas dependientes de E2 deben ser validadas bioquímicamente y vinculadas al papel biológico de la proteína probada. Aquí, describimos un protocolo paso a paso que describe cómo detectar y caracterizar funcionalmente la actividad enzimática de las ligasinas ubiquitinas tipo RING E3, tanto in vitro como en planta, a través de un enfoque de mutagénesis dirigida por el sitio. Los resultados representativos de esta canalización se muestran para la ligada de tipo RING E3 RHA1B. RHA1B es una proteína efectora producida por el nematodo parasitario de la planta Globodera pallida para suprimir la inmunidad de la planta y manipular la morfología de las células de la raíz de la planta. Para protegerse de la invasión de patógenos/parásitos, las plantas han evolucionado el dominio de unión a nucleótidos y los receptores inmunes de tipo de repetición rica en leucina (NB-LRR) que detectan la presencia de un patógeno o parásito y, como consecuencia, desarrollan la respuesta hipersensible (HR), que es una forma de muerte celular rápida y localizada que ocurre en el sitio de infección para detener la colonización de patógenos. Uno de estos receptores inmunes es la proteína gpa2 de patata que confiere resistencia a algunos aislados de G. pallida (poblaciones de campo D383 y D372)7.
Utilizando los protocolos presentados, se ha encontrado recientemente que RHA1B interfiere con la señalización inmune de la planta de una manera dependiente de E3 al apuntar a la planta Gpa2 inmunoreceptor para la ubiquitinación y degradación8.
Protocolo
1. Mutagénesis dirigida por el sitio(Figura 1)
- Identificar las Cys conservadas y Sus aminoácidos en el dominio RING(Figura 1A) y las imprimaciones de diseño que llevan el codón de sustitución de interés flanqueado por 15 pares base a ambos lados del sitio de mutación(Figura 1B).
- Introducir la mutación deseada por la amplificación basada en PCR del plásmido que alberga el gen de interés utilizando imprimaciones mutagénicas y polimerasa de ADN de alta fidelidad que contiene Pfu en 50 s de volumen total de reacción de PCR, como se muestra en la Tabla 1 y la Tabla 2 según el protocolo del fabricante.
- Digerir el ADN parental metilado y semimetilado derivado de Escherichia coliañadiendo 3 l de enzima de restricción DpnI directamente a la reacción de PCR (paso 1.2) e incubando a 37oC durante 2 h.
NOTA: La metilación es una modificación de la proteína posttranscripcional que se añade al plásmido producido y aislado de las bacterias. Nuevas copias del plásmido generado por PCR carecen de metilación, por lo tanto, las nuevas copias permanecerán intactas durante el tratamiento con DpnI. - Purificar los plásmidos mutagenizados utilizando un kit comercial de extracción de ADN basado en la tecnología de columna de espín y eluir el ADN con 50 ml de agua.
- Transformar las células químicamente competentes de DH5 E. coli con 0,5 ml del ADN de plásmido mutageneizado recuperado de acuerdo con el protocolo del fabricante. En resumen, incubar células competentes con ADN en hielo durante 30 minutos, luego escupirlas térmicamente durante 20 s a 42 oC, y colocar los tubos de nuevo en hielo durante 2 minutos.
- Verifique la mutación deseada por Sanger secuenciando los plásmidos de ADN aislados de E. coli.
2. Purificación de proteínas recombinante y ensayo de ubiquitinación in vitro
- Clonar el tipo salvaje RING y mutar genes RING de interés en el vector pMAL-c2 (siga el protocolo del fabricante; Cuadro 3) para fusionar estos genes con la etiqueta de epítopo MBP que permite la purificación de un solo paso utilizando resina de amilosa. Introducir las construcciones resultantes en la cepa E. coli BL21 como se describe en el paso 1.5.
- Cultivar la cepa de E. coli BL21 que alberga la construcción deseada en medio líquido LB de 50 ml a 37 oC durante 2-3 h hasta que llegue a la fase logarítmica (OD600 de 0.4–0.6).
- Añadir IPTG a una concentración final de 0,1–1 mM para inducir la expresión de proteína recombinante etiquetada con MBP de interés e incubar el cultivo de E. coli durante 2-3 h a 28 oC. Coloque la cultura sobre hielo después de la incubación.
NOTA: Realice los pasos 2.4–2.13 en hielo para proteger las proteínas de la degradación. - Para comprobar la eficiencia de inducción, recoja 1,5 ml de células inducidas, Girarlas a 13.000 x g durante 2 min, quitar el sobrenadante y resuspender las células en 20 l de 2x tampón de carga SDS-PAGE (24 mM Tris-HCl pH 6.8, 0.8% SDS, 10% (v/v) glicerol, 4 mM DTT, 0.04% (w/v) bromofenol azul).
- Hervir las muestras durante 5 minutos y ejecutarlas con un gel 10% SDS-PAGE. Para evaluar visualmente la acumulación de proteína de fusión MBP (peso molecular de proteína de interés + 42,5 kDa MBP), manchar el gel durante 20 min agitando con tampón de tinción coomassie (50% metanol, 10% ácido acético, 0,1% azul Coomassie) y destainando durante la noche con el tampón de destaining (20% metanol, 10% ácido acético).
- Cosecha las células restantes de E. coli por centrifugación a 1.350 x g durante 6 min, desecha el sobrenadante y resuspende el pellet celular con 5 ml de tampón de columna (20 mM Tris HCl, 200 mM de NaCl, 1 mM EDTA, inhibidor de la proteasa bacteriana).
NOTA: Este es un buen lugar para detener el protocolo durante la noche. Las células congeladas se pueden almacenar hasta 1 semana a -20 oC. - Descomponer las células de E. coli colocando el tubo que contiene las bacterias en un baño de agua helada y aplicando 10 ciclos de sonicación: 10 s sonicación a 30% de amperios seguidos de 20 s descansos.
- Muestra de centrífuga a 13.000 x g a 4oC durante 10 min y guarda el sobrenadante (extracto crudo).
- Preparar 500 ml de resina de amilosa en un tubo de 15 ml. Lavar la resina añadiendo 10 ml de tampón de columna fría y centrifugación a 1.800 x g,4 oC durante 5 min. Haz esto 2x.
- Añadir 5 ml de extracto crudo al tubo con la resina de amilosa e incubar durante la noche a 4oC.
- Centrifugar a 1.800 x g a 4oC durante 5 min y desechar el sobrenadante.
- Añadir 10 ml de tampón de columna al pellet de resina e incubar durante 20 min. A continuación, centrífuga a 1.800 x g a 4 oC durante 5 min. Repita este paso 2x.
- Eluir la proteína de fusión con 0,5 ml de tampón de columna que contenga 10 mM de maltosa incubando una muestra durante 2 h a 4 oC. Centrifugar a 1.800 x g a 4oC durante 5 min y recoger la proteína eluted. Repita este paso 2x.
- Dializar 1 ml de la fracción proteica contra el PBS frío. Proteína aliquot en tubos de un solo uso (10-20 l) para evitar la congelación y almacenar a -80 oC hasta que sea necesario.
- Mida la concentración de proteínas utilizando el ensayo Bradford9.
- Configurar la reacción de ubiquitinación in vitro en un volumen total de 30 l mezclando 40 ng de E1 (por ejemplo, AtUBA1), 100 ng de E2 (por ejemplo, AtUBC8, SlUBC1/4/6/7/12/13/17/20/22/27/32), 1 g de proteína tipo MBP- y 2 g de FLAG-Ub (o HA-Ub) en el tampón de ubiquitinación (50 mM Tris-HCl pH 7.5, 2 mM ATP, 5 mM MgCl2, 30 mM de fosfato de creatina, 50 g/ml de creatina fosfoquinasa). Incubar la mezcla a 30oC durante 2 h.
NOTA: Premake 20x tampón de ubiquitinación y guárdalo hasta 6 meses a -20 oC en pequeñas alícuotas para un solo uso. La creatina fosfoquinasa pierde fácilmente su actividad enzimática cuando el tampón se descongela y se congela repetidamente. - Terminar la reacción mezclando las muestras de 30 l con 7,5 l de búfer de carga de 5 x SDS-PAGE (60 mM Tris-HCl pH 6,8, 2% SDS, 25% (v/v) glicerol, 10 mM de TDT, 0,1% (p/v) de bromofenol azul) y hervir durante 5 min.
- Separe las proteínas con una electroforesis de gel de poliacrilamida del 7,5% SDS (SDS-PAGE), luego transfiera a la membrana PDVF y detecte la ubiquitinación por hinchazón occidental utilizando el anti-FLAG (o anti-HA).
- Mancha la membrana PVDF con Azul Coomassie para verificar la carga igual de proteína de tipo MBP-RING probada.
3. Expresión de proteína transitoria mediada por Agrobacteriumen hojas de Nicotiana benthamiana y en el ensayo de ubiquitinación de plantaquitinación
- Las cepas apropiadas de la racha Agrobacterium tumefaciens que llevan el gen de interés etiquetado con epítopos (por ejemplo, HA-RHA1B, HA-RHA1BC135S, HA-RHA1BK146R, HA-Ub) y vector vacío como control en el medio LB que contiene los antibióticos de selección adecuados.
- Después de 2 días de crecimiento a 28oC, recoge colonias individuales y hazlas crecer en medio líquido LB con los antibióticos adecuados a 28oC/250 rpm durante otras 24 h.
- Transfiera 100 ml de cultivo agrobacteriano a 3 ml de LB fresco con los antibióticos apropiados e incubar el cultivo durante 4-6 h adicionales a 28 oC con rotación (250 rpm) a la fase de crecimiento exponencial tardío.
- Espíe las células agrobacterianas a 1.800 x g durante 6 min, deseche el sobrenadante y resuspenda las células con 3 ml de tampón de lavado (50 mM MES pH 5.6, 28 mM de glucosa, 2 mM NaH2PO4). Repita este paso 2x.
- Después del segundo lavado, resuspenda las células en el tampón de inducción (50 mM MES pH 5.6, 28 mM de glucosa, 2 mM NaH2PO4, 200 m acetosyringone, 37 mM NH4Cl, 5 mM MgSO4.7H2O, 4 mM KCl, 18 M FeSO4.7H2O, 2 mM CaCl2). Incubar las células con tampón de inducción durante 10–12 h adicionales a 28 oC.
NOTA: Acetosyringona induce la transferencia de ADN-T. - Centrifugar las células a 1.800 x g durante 6 min, desechar el sobrenadante y resuspender las células con 2 ml de tampón de infiltración (10 mM MES pH 5.5, 200 m de acetosyringona).
NOTA: Si Agrobacterias se agrega después de la incubación con tampón de inducción, deje que las células agregadas se hundan en la parte inferior del tubo dejándolo en el banco durante unos minutos, y transfiera la suspensión clara de Agrobacterium a un nuevo tubo antes de continuar con el paso 3.6. - Mida la concentración de bacterias utilizando el valor OD600 (la densidad óptica a una absorbancia de 600 nm). Ajuste los valores de OD600 a los deseados.
NOTA: Por lo general, un valor de600 OD entre 0,2 y 0,4 funciona mejor para una sola expresión de tinción agrobacteriana. Si se aplica una combinación de diferentes cepas agrobacterianas, los valores totales de OD600 de las cepas agrobacterianas no deben exceder de 1. - Agroinfiltrate de 4 semanas de edad N. bethamiana se va pinchando suavemente con una aguja, seguido de Agrobacterium inyectando a mano con una jeringa sin la aguja. Círculo de la zona de la hoja infiltrada con el marcador (generalmente 1-2 cm de diámetro).
- Recoger los tejidos de las hojas infiltradas 36 h después de la infiltración. Moler el tejido en un polvo fino con nitrógeno líquido.
- Polvo de tejido resuspendido con 300 l de tampón de extracción de proteínas (50 mM Tris-HCl pH 7.5, 150 mM NaCl, 5 mM EDTA, 2 mM TDT, 10% glicerol, 1% polivinilpolipirrolidona, 1 mM PMSF, cóctel inhibidor de la proteasa vegetal) y centrífuga a 15.000 x g durante 15 min a 4oC.
- Transfiera el sobrenadante a un tubo nuevo. Añadir 5x búfer de carga SDS-PAGE a una concentración final de 1x y hervir durante 5 min.
- Separe las proteínas brutas en geles 10% SDS-PAGE, transfiera a membranas PVDF y sondas con anti-HA para detectar en la ubiquitinación de la planta.
4. Establecer el vínculo entre la actividad enzimática y la función en planta
NOTA: Por ejemplo, RHA1B promueve la degradación de la proteína resistente Gpa2 para suprimir la muerte celular de HR. Este paso muestra cómo verificar que esas actividades virulentas de RHA1B son dependientes de E3.
- Las cepas de Agrobacterium tumefaciens apropiadas de racha que llevan genes etiquetados de interés (en este ejemplo HA-RHA1B, HA-RHA1BC135S,HA-RHA1BK146R, myc-Gpa2, RBP1) y vector vacío como control. Siga los pasos 3.1–3.8 para la preparación e inyección de Agrobacterium en hojas de N. bethamiana.
- Para la degradación de la proteína de sustrato dependiente de E3, siga los pasos 3.9–3.12 y realice la hincha occidental utilizando anticuerpos apropiados para detectar la acumulación de proteínas en las células vegetales (por ejemplo, anti-HA y anti-MYC).
- Para la inhibición de la muerte celular mediada por respuesta hipersensible dependiente de E3 (HR), monitoree las hojas agroinlinlinminficadas para los síntomas de muerte celular de HR de 2 a 4 días después de la infiltración.
Resultados
En esta sección, se proporcionan resultados representativos para el protocolo utilizado para el examen de una sola subunidad E3 ubiquitina ligasa RHA1B que tiene un dominio de tipo RING-H2 predicho por PROSITE (132–176 aminoácidos)10. Como se muestra en la Figura 1, para obtener una proteína mutante deficiente en E3, al menos una de las ocho Cos o Hs conservadas en el dominio RING(Figura 1A) necesita ser mutada(Figura 1B). Así, como primer paso, se generaron dos versiones mutantes de RHA1B, RHA1BC135S (una sustitución de Cys por Ser en el C3 conservado del dominio RING) y RHA1BK146R (una sustitución de Lys por Arg en el único Lys presente en RHA1B). Aunque una sola subunidad E3 ligases media la transferencia de ubiquitina de ubiquitina que alberga E2 al sustrato en lugar de interactuar directamente con la ubiquitina, la autoubiquitinación de E3 en Lys podría ser necesaria para su actividad enzimática máxima.
Los resultados de la hincha occidental en la Figura 2A muestran un resultado típico positivo del ensayo de ubiquitinación in vitro, con un frotis multibanda que comienza en el peso molecular de la proteína probada (por ejemplo, RHA1B con fusión mpB a 100 kDa) y progresa hacia arriba. El anticuerpo anti-HA reconocido HA-etiquetado UB incorporado en la cadena de poli-ubiquitinación de diferentes longitudes, creando este típico frotis de escalera asociado a la ubiquitina. Para validar los resultados positivos, la Figura 2A también presenta todos los controles negativos importantes que faltan en componentes individuales (E1, E2, Ub o MBP-RHA1B) o utilizando MBP como control y falta de la señal de ubiquitinación manchada. Además, la tinción azul Coomassie de la membrana PVDF mostró la misma carga de MBP-RHA1B o MBP en todos los controles.
La Figura 2B muestra cómo variaron los resultados de la ubiquitinación in vitro dependiendo de la combinación específica E2/E3. En este ejemplo, se probaron 11 E2 diferentes que representaban 10 familias E2 diferentes. La actividad de ubiquitinación detectada varió de ninguna señal (sin frotis) a un frotis multibanda a partir de diferentes pesos moleculares, lo que indica diferentes patrones de ubiquitinación.
La Figura 3 muestra los resultados del ensayo de ubiquitinación para las versiones RING y K-mutantes de la proteína probada. La falta de actividad enzimática para RHA1BC135S está respaldada por su incapacidad para generar un frotis multibanda in vitro(Figura 3A)o promover la señal de poliubiquitinación en planta(Figura 3B). Es notable que la sobreexpresión de UB con etiqueta HA en planta por sí sola dio ubiquitinación de nivel basal en todas las muestras probadas, incluido el control vectorial, en contraste con la fuerte señal de ubiquitinación conferida por la actividad enzimática de tipo salvaje RHA1B. Además, el análisis del mutante RHA1BK146R sugiere que el residuo K146 también es esencial para la actividad E3 de RHA1B. Aunque se detectó una señal marginal de autoubiquitinación in vitro(Figura 3A),el ensayo in planta determinó que el mutante es deficiente en E3(Figura 3B,sólo se detectó la señal de ubiquitinación de fondo).
Después de generar y validar bioquímicamente el mutante con deficiencia de E3, se pueden diseñar estudios funcionales para determinar el papel biológico asociado a E3 de la ligarosa de ubiquitina RING E3 probada. En el caso de RHA1B, este efector de nematodos suprime la señalización inmune de la planta, como se manifiesta por la supresión de la muerte de la célula HR desencadenada por Gpa2. Como se presenta en la Figura 4A, a diferencia del tipo salvaje RHA1B, el mutante RHA1BC135S que carecía de actividad de ligaa E3 no interfirió con la muerte de células HR. Dado que el resultado más común de la ubiquitinación proteica es su degradación mediada por proteasoma, las mutaciones que residen en el dominio RING también se pueden utilizar para verificar una capacidad dependiente de E3 para desencadenar la degradación de sus sustratos directos y/o indirectos. Por lo tanto, significativamente, los resultados de la hincha occidental en la Figura 4B confirman que Gpa2 no se acumuló en presencia de tipo salvaje RHA1B pero RHA1BC135S no tuvo ningún impacto en la estabilidad de la proteína Gpa2.
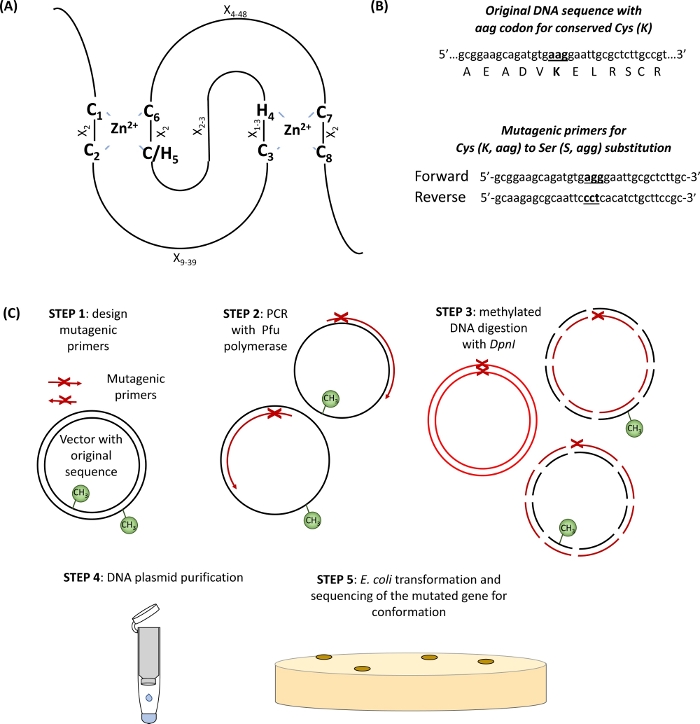
Figura 1: Representación esquemática del principio y los pasos implicados en la mutagénesis dirigida por el sitio. (A) dominio RING-CH/H2 con Cys conservado y Sus aminoácidos resaltados. (B) Un ejemplo de diseño de imprimaciones mutagénicas. (C) Pasos de mutagénesis dirigida por el sitio. Haga clic aquí para ver una versión más grande de esta figura.
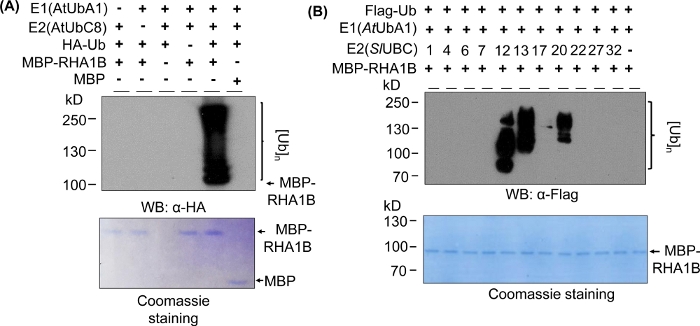
Figura 2: Ensayo representativo de ubiquitinación in vitro. (A) El gel superior muestra el ensayo de ubiquitinación incluyendo todos los controles negativos, y el gel inferior muestra la carga igual. (B) El rango de resultados esperados dependiendo de las enzimas E2. Esta cifra ha sido modificada de Kud et al8. Haga clic aquí para ver una versión más grande de esta figura.
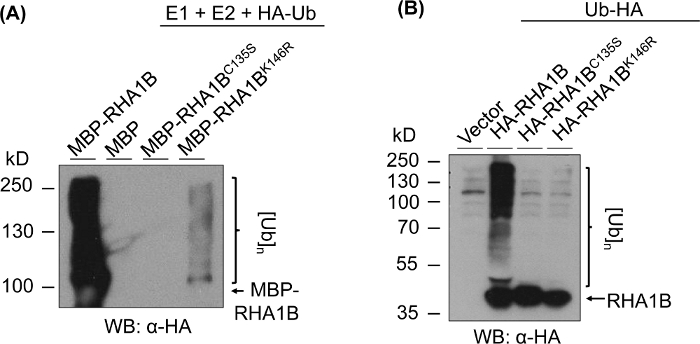
Figura 3: Resultados del ensayo de ubiquitinación para los mutantes RING y K (RHA1BC135S y RHA1BK146R). (A) Resultados de ubicuidación in vitro para RHA1BC135S y RHA1BK146R. (B) En los resultados del ensayo de ubiquitinación de planta para RHA1BC135S y RHA1BK146R. Esta cifra ha sido modificada de Kud et al8. Haga clic aquí para ver una versión más grande de esta figura.
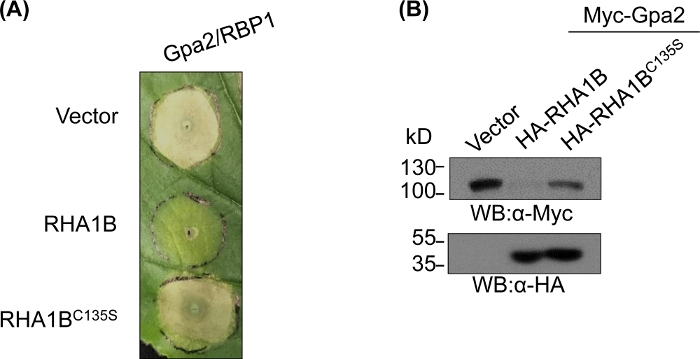
Figura 4: Estudio funcional representativo para las funciones biológicas dependientes de E3. Un ejemplo de estudios funcionales que muestran la función biológica dependiente de E3. (A) Supresión de la muerte de células HR dependientes de E3 y (B) degradación de un inmunorreceptor vegetal Gpa2. Esta cifra ha sido modificada de Kud et al8. Haga clic aquí para ver una versión más grande de esta figura.
| Configuración de PCR | |
| 1 L | plásmido (100 ng) |
| 1.5 L | Imprimación mutagénica F (10 m) |
| 1.5 L | Imprimación mutagénica R (10 m) |
| 1 L | dNWP (10 mM) |
| 5 l | buffer (10x) |
| 1 L | Ultra Pfu polimerasa (2,5 U/l) |
| 39 l | ddH2O |
| 50 l | VOLUMEN TOTAL |
Tabla 1: Configuración de la reacción de PCR
| programa termociclador | |||
| 1 | 95 oC | 30 s | |
| 2 | 95 oC | 30 s | |
| 3 | 60 oC | 30 s | |
| 4 | 72 oC | 5 min | repetir 2-4 30 veces |
| 5 | 72 oC | 5 min | |
Tabla 2: Programa termociclador PCR
| reacción de ligadura configurada para el ejemplo RHA1B | ||
| 1.5 L | pMAL-c2::MBP vector linealizado por digestión con BamHI y SalI (60 ng) | |
| 7 L | Inserto RHA1B/RHA1BC135S o RHA1BK146R digerido con BamHI y SalI (25 ng) | |
| 1 L | T4 tampón de ligasa (10x) | |
| 0,5 l | Ligasa T4 (400 U/L) | |
| 10 l | VOLUMEN TOTAL | |
Tabla 3: Reacción de ligadura configurada para el ejemplo RHA1B.
Discusión
Elaclarar la base bioquímica y mecanicista de las ligasas de ubiquitina TIPO RING E3 puede contribuir en gran medida a nuestra comprensión de su importancia biológica en el desarrollo, la señalización del estrés y el mantenimiento de la homeostasis. El protocolo descrito aquí combina un enfoque de mutagénesis con estudios funcionales in vitro y en planta. Mediante la introducción de una única sustitución de aminoácidos en los residuos conservados del dominio RING a través de mutagénesis directa al sitio, el mutante con deficiencia de E3 resultante puede ser probado en paralelo con proteína de tipo silvestre para vincular la actividad enzimática con la funcionalidad.
Es fundamental identificar adecuadamente el dominio RING, en particular sus Cys conservados y Sus residuos. Las herramientas en línea como PROSITE se pueden utilizar para hacerlo10. Para desestabilizar el dominio RING responsable de reclutar la enzima E2, Cys normalmente se sustituye por Ser, que es su reemplazo estructural más cercano que carece de la capacidad de crear un enlace de disulfuro utilizado para la coordinación de zinc. Lorick et al. mostraron que la mutación en cualquiera de esos residuos críticos de Cys aboliría la actividad de ubiquitinación de la subunidad única tipo RING E3 ligases5. Aunque algunos residuos de Cys también son importantes para los complejos de ligasas E3 multiunidad que contienen proteínas de tipo RING, debido a la estructura tridimensional multifacética y dinámica de esos complejos de ubiquitinación y a su diferente papel de las proteínas tipo RING, las sustituciones únicas de residuos conservados en el dominio RING en la ligasa E3 multiunit no ha tenido éxito en la generación de un fenotipo11con deficiencia de ligasa.
Para la mutagénesis dirigida por el sitio, encontramos que el uso de vectores plásmidos más pequeños y ciclos de amplificación más bajos generalmente produjo una mayor eficiencia para la mutagénesis. La enzima Pfu se puede sustituir por cualquier otra polimerasa de ADN de alta fidelidad y alta procesatividad. Además, si el gen de interés contiene codones raros, otra mancha de E. coli, Rosetta, se puede utilizar para lograr un mayor rendimiento de la proteína recombinante. Además, tanto el tiempo de incubación como la temperatura para la inducción IPTG se pueden optimizar aún más. Las temperaturas más bajas reducen la tasa de división de E. coli, lo que podría ser favorable para la expresión de ciertas proteínas. Aunque una mayor concentración de IPTG podría mejorar la expresión de proteínas, también inhibe los procesos de división de E. coli y no se recomienda.
Las ligasas RING de subunidad única tipo E3 no sólo funcionan como un andamio molecular que posiciona el intermedio E2-Ub cerca del sustrato, sino que también estimula la actividad de transferencia de ubiquitina de sus E2 cognados. Además, dado que una combinación E2/E3 es importante para la longitud y los enlaces de la cadena de poliubiquitina que determina el destino de un sustrato modificado, cualquier consideración de los E3 de tipo RING debe incluir a sus socios enzimáticos, E2s12. Como se muestra en la Figura 3B,no todos los E2 probados son compatibles con la ligada RHA1B. Por lo tanto, los ensayos de ubiquitinación in vitro deben llevarse en paralelo con múltiples enzimas E2 que representan diferentes clases E2 para evitar resultados falsos negativos.
Aquí se presenta el ensayo enzimático in vitro que detecta la capacidad de autoubiquitinación de las proteínas de tipo RING probadas. Sin embargo, con pequeñas modificaciones, este protocolo se puede adaptar fácilmente para detectar la ubiquitinación in vitro de sustratos. Para ello, la mezcla de ubiquitinación in vitro del paso 2.15 debe complementarse con la proteína recombinante del potencial sustrato de ligasa E3 (500 ng). Después de una incubación de 2 h a 30 oC, la proteína ubiquitinizada debe capturarse utilizando 15 oL de matriz de afinidad anti-HA (si se utiliza HA-Ub, o matriz de afinidad anti-FLAG si se utiliza FLAG-Ub) por agitación durante 2 h a 4 oC. Después de lavar las perlas 4 veces con el tampón de lavado Ub frío (20 mM Tris pH 7.5, 100 mM NaCl, 0.1 mM EDTA, 0.05% Tween 20, 1x PMSF), deseche todo menos 40 l del tampón y pase al paso 2.16. La señal de ubiquitinación, detectada por anticuerpos específicos del ub etiquetado con epítopos y sustrato, respectivamente, que surge del peso molecular de la proteína del sustrato, confirma la especificidad sustrato/enzima.
Además, la identificación in vivo de sustratos de ligasa E3 se asocia con múltiples desafíos debido a la interacción transitoria enzima-sustrato y la rápida degradación de la proteína diana ubiquitinada. El uso de un mutante deficiente de ligasa E3, que todavía interactúa con su objetivo pero ya no lo ubiquitina13, es una alternativa muy útil a la adición de inhibidor proteasomal MG132, que no siempre interfiere lo suficiente con la función de proteasoma 26S.
Una característica común de las ligasas tipo RING E3 es una tendencia a formar y funcionar como homo- y/o heterodímeros. Curiosamente, la sustitución en los residuos conservados del dominio RING suele estar asociada a un fenotipo negativo dominante en el que la ligasa mutada tipo RING E3 bloquea la actividad enzimática de una proteína nativa de tipo silvestre13. Por lo tanto, la sobreexpresión de los mutantes RING en planta puede ser un enfoque alternativo para eliminar el gen de la ligasa E3.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Nuestro trabajo fue posible gracias al apoyo financiero de la subvención competitiva de la Iniciativa de Investigación Agrícola y Alimentaria (2017-67014-26197; 2017-67014-26591) del USDA National Institute of Food and Agriculture, USDA-NIFA Farm Bill, Northwest Potato Potato Consorcio, y ISDA Specialty Crop.
Materiales
| Name | Company | Catalog Number | Comments |
| Acetic acid | Sigma-Aldrich | A6283 | |
| Acetosyringone | Sigma-Aldrich | D134406 | |
| Amylose resin | NEB | E8021S | |
| ATP | Sigma-Aldrich | A1852 | |
| Bacterial protease inhibitor | Sigma-Aldrich | P8465 | |
| Bromphenol Blue | VWR | 97061-690 | |
| CaCl2 | Sigma-Aldrich | C1016 | |
| Centrifuge | Beckman Coulter | model: Avanti J-25 | |
| Commassie Blue | VWR | 97061-738 | |
| Creatine phosphate | Sigma-Aldrich | P7936 | |
| Creatine phosphokinase | Sigma-Aldrich | C3755 | |
| DNA clean & concentrator Kit | ZYMO RESEARCH | D4029 | |
| DpnI | NEB | R0176S | |
| DTT | Sigma-Aldrich | D0632 | |
| E. coli BL21 | Thermo Fisher Scientific | C600003 | |
| E. coli DH5α competent cells | Thermo Fisher Scientific | 18265017 | |
| EDTA | Sigma-Aldrich | 324504 | |
| FeSO4 7H2O | Sigma-Aldrich | F7002 | |
| FLAG-Ub | BostonBiochem | U-120 | |
| Glucose | VWR | 188 | |
| Glycerol | Sigma-Aldrich | G5516 | |
| HA-Ub | BostonBiochem | U-110 | |
| Heat block | VWR | model: 10153-318 | |
| Incubator | VWR | model: 1525 Digital Incubator | |
| Incubator shaker | Thermo Fisher Scientific | model: MaxQ 4000 | |
| IPTG | Roche | 10724815001 | |
| KCl | Sigma-Aldrich | P9333 | |
| LB Broth | Sigma-Aldrich | L3022 | |
| Liquide nitrogen | university chemistore | ||
| Maltose | Sigma-Aldrich | 63418 | |
| MES | Sigma-Aldrich | M3671 | |
| Methanol | Sigma-Aldrich | 34860 | |
| MgCl2 | Sigma-Aldrich | 63138 | |
| MgSO4 7H2O | Sigma-Aldrich | 63138 | |
| Microcentrifuge | Eppendorf | model: 5424 | |
| Miniprep plasmid purification kit | ZYMO RESEARCH | D4015 | |
| monoclonal anti-FLAG antibody | Sigma-Aldrich | F3165 | |
| monoclonal anti-HA antibody | Sigma-Aldrich | H9658 | |
| monoclonal anti-MYC antibody | Sigma-Aldrich | WH0004609M2 | |
| Mortar | VWR | 89038-144 | |
| NaCl | Sigma-Aldrich | S7653 | |
| NaH2PO4 | Sigma-Aldrich | S8282 | |
| NanoDrop | Thermo Fisher Scientific | model: 2000 Spectrophotometer | |
| Needle | Thermo Fisher Scientific | 14-826-5C | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| PCR machine | Bio-Rad | model: C1000 | |
| Pestle | VWR | 89038-160 | |
| Pfu Ultra | Agilent Technologies | 600380 | |
| Plant protease inhibitor coctail | Sigma-Aldrich | P9599 | |
| pMAL-c2 | NEB | N8076S | |
| PMSF | Sigma-Aldrich | P7626 | |
| Polyvinylpolypyrrolidone | Sigma-Aldrich | P6755 | |
| SDS | Sigma-Aldrich | 1614363 | |
| Sonicator | Qsonica Sonicators | model: Q125 | |
| Syringe | Thermo Fisher Scientific | 22-253-260 | |
| Tris | Sigma-Aldrich | T1503 | |
| T4 ligase | NEB | M0202S |
Referencias
- Freemont, P. S., Hanson, I. M., Trowsdale, J. A novel gysteine-rich sequence motif. Cell. 64, 483-484 (1991).
- Borden, K. L. B. RING fingers and B-boxes: Zinc-binding protein-protein interaction domains. Biochemistry and Cell Biology. 76, 351-358 (1998).
- Barlow, P. N., Luisi, B., Milner, A., Elliott, M., Everett, R. Structure of the C3HC4 Domain by 1H-nuclear Magnetic Resonance Spectroscopy: A New Structural Class of Zinc-finger. Journal of Molecular Biology. 237, 201-211 (1994).
- Borden, K. L. B., et al. The solution structure of the RING finger domain from the acute promyelocytic leukaemia proto-oncoprotein PML. The EMBO Journal. 14, 1532-1541 (1995).
- Lorick, K. L., et al. RING fingers mediate ubiquitin-conjugating enzyme (E2)-dependent ubiquitination. Proceedings of the National Academy of Sciences. 96, 11364-11369 (1999).
- Jiménez-López, D., Muñóz-Belman, F., González-Prieto, J. M., Aguilar-Hernández, V., Guzmán, P. Repertoire of plant RING E3 ubiquitin ligases revisited: New groups counting gene families and single genes. PLoS ONE. 13, 1-28 (2018).
- Sacco, M. A., et al. The Cyst Nematode SPRYSEC Protein RBP-1 Elicits Gpa2- and RanGAP2-Dependent Plant Cell Death. PLoS Pathogens. 5, 1-14 (2009).
- Kud, J., et al. The potato cyst nematode effector RHA1B is a ubiquitin ligase and uses two distinct mechanisms to suppress plant immune signaling. PLoS Pathogens. 15, 1007720 (2019).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72, 248-254 (1976).
- Sigrist, C. J. A., et al. New and continuing developments at PROSITE. Nucleic Acids Research. 41, 344-347 (2013).
- Dove, K. K., Stieglitz, B., Duncan, E. D., Rittinger, K., Klevit, R. E. Molecular insights into RBR E3 ligase ubiquitin transfer mechanisms. EMBO Reports. 17, 1221-1235 (2016).
- Metzger, M. B., Pruneda, J. N., Klevit, R. E., Weissman, A. M. RING-type E3 ligases: Master manipulators of E2 ubiquitin-conjugating enzymes and ubiquitination. Biochimica et Biophysica Acta - Molecular Cell Research. 1843, 47-60 (2014).
- Xie, Q., et al. SINAT5 promotes ubiquitin-related degradation of NAC1 to attenuate auxin signals. Nature. 419, 167-170 (2002).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados