Method Article
Функциональная характеристика РИНГ-типа E3 Ubiquitin Ligases in Vitro и В Планте
В этой статье
Резюме
Целью данной рукописи является представление набросков для комплексных биохимических и функциональных исследований посвяжецких лигазах типа RING E3. Этот многоступенчатый конвейер с подробными протоколами проверяет ферментативную активность проверенного белка и демонстрирует, как связать эту активность с функционированием.
Аннотация
Убиквитинация, как постпереводная модификация белков, играет важную регулятивную роль в гомеостаза хукартотических клеток. Ковалентное присоединение 76 аминокислот ный убиквитин модификаторов к целевому белку, в зависимости от длины и топологии полиубиквитина цепи, может привести к различным результатам, начиная от деградации белка изменения в локализации и / или деятельности модифицированного белка. Три фермента последовательно катализируют процесс убиквитина: e1 убиквитин-активирующего фермента, E2 убиквитин-конъюгирующего фермента, и E3 убиквитин лигазы. E3 убиквитин лигаза определяет специфику субстрата и, следовательно, представляет собой очень интересный предмет исследования. Здесь мы представляем комплексный подход к изучению взаимосвязи между ферментативной активностью и функцией убиквитиновой лиги цигазы типа RING. Этот четырехступенчатый протокол описывает 1) как создать E3 лигаза дефицит мутант через сайт-направленный мутагенез, ориентированный на сохраненный домен RING; 2-3) как изучить активность вездесущности как в пробирке, так и в плану; 4) как связать эти биохимический анализ с биологической значимостью проверенного белка. Поколение мутанта E3, дефицитного лигазе, который все еще взаимодействует со своим субстратом, но больше не уведомляет его для деградации, облегчает тестирование взаимодействий фермент-субстрата in vivo. Кроме того, мутация в сохраненной области RING часто предоставляет доминирующий отрицательный фенотип, который может быть использован в функциональных нокаут-исследований в качестве альтернативного подхода к РНК-интерференции. Наши методы были оптимизированы для исследования биологической роли растения паразитарных нематод эффектор RHA1B, который захватывает принимающей системы убликвиции в клетках растений для содействия тунеядства. При незначительной модификации системы экспрессии in vivo этот протокол может быть применен к анализу любой лиги e3 типа RING независимо от ее происхождения.
Введение
Подавляющее большинство E3 убиквитин лигаза принадлежат к RING(Really Interesting New Gene) типа белков. Домен RING-finger был первоначально идентифицирован Freemont et al. 1 и функционально описывается как домен, опосредовающий белково-белковое взаимодействие2. Канонический палец RING представляет собой особый тип цинка координации домена определяется как консенсус последовательность из восьми сохраненных Cys (C) и его (H) специально расположены другие аминокислотные остатки (X), C-X2-C-X9-39-C-X1-3-H-X2-3-C/H-X2-C-X 4-C-X.C.2-C-X.C.2-C-X.C.2-C-X.C.2-C-X.C.2-C-X.C.2-C-X-C-X.C-X.C.2-C-X.C.2-C-X-C-X.C.2-C-X.C.2-C-X-C-X-C-X.C.2-C-X-C-X-C-X-C-X-C-X-C-X-C-X-C-X-C-X-C-X-C-X-C-X-C-2-C-X-C-X-C-X-C-X-C-X-C-X-C-X-C-X-C-2-C-X.C.2-C-X-C-X-C Два иона «n2» стабилизируются по остаткам ядра C и H через уникальную топологию «кросс-скобки» с C1/C2 и C/H5/C6, координирующими первый ион «n2» в то время как C3/H4 и C7/C8 связывают второй(рисунок 1A)3,4. В зависимости от наличия C или H в пятом месте координации «n2»были определены два канонических подкласса белков RING-finger: C3HC4 и C3H2C3 (RING-HC и RING-H2, соответственно). Поскольку область RING E3 ubiquitin ligase опосредует взаимодействие между ферментами E2 и субстратами, мутация этих основных остатков C и H, как было показано, нарушает активность лигазы5. Описаны еще пять менее распространенных подклассов лигазаring E3 (RING-v, RING-C2, RING-D, RING-S/T и RING-G)6. Убиквитин-лигаза типа РИНГ можно дополнительно разделить на простые и сложные ферменты E3. Простое одноподразделение RING E3 ligases содержит как сайт распознавания субстрата, так и домен E2-связывания RING. В отличие от этого, мультисубунит типа RING E3 комплекс либо набирать субстрата или опосредует связывание E2-убиквитин промежуточных к E3 комплекса. Остаток домен RING Lys (ы), который служит основным сайтом вложений убиквитина (ы) для самоубиквитина, также может быть важен для деятельности E3 ligase.
Не все содержащие RING белки функционируют как лигаза E3. Таким образом, биоинформатическое предсказание домена RING-finger и способность к убиквитивании белка, зависящему от Е2, должно быть биохимически проверено и связано с биологической ролью проверенного белка. Здесь мы описываем пошаговый протокол с изложением того, как обнаружить и функционально охарактеризовать ферментативную активность убиквитинов типа RING E3, как in vitro, так и в плане, с помощью подхода мутагенеза, направленного на сайт. Репрезентативные результаты этого трубопровода отображаются для РИНГ типа E3 ligase RHA1B. RHA1B является эффектор белка, вырабатываемого растений паразитической кисты nematode Globodera паллида для подавления иммунитета растений и манипулировать морфологией корневых клеток растений. Чтобы защитить себя от патогенного/ паразитного вторжения, растения развили нуклеотид-связывающей области и лейцина богатых повторить (NB-LRR) типа иммунных рецепторов, которые обнаруживают наличие патогена или паразита и, как следствие, развивать гиперчувствительный ответ (HR), который является одной из форм быстрой и локализованной смерти клеток, происходящих на месте инфекции, чтобы остановить колонизации патогенов. Одним из таких иммунных рецепторов является картофельный белок Gpa2, который придает устойчивость некоторым изолятам G. pallida (полевые популяции D383 и D372)7.
Используя представленные протоколы, было недавно установлено, что RHA1B мешает иммунной сигнализации растений в E3-зависимым образом, ориентируясь на иммунорецептор завода Gpa2 для убликвитиния и деградации8.
протокол
1. Сайт-направленный мутагенез(Рисунок 1)
- Определите сохраненные Cys и его аминокислоты в домене RING(Рисунок 1A) и дизайн грунтовки проведения замены кодон интереса в окружении 15 базовых пар по обе стороны от места мутации (Рисунок 1B).
- Ввести желаемую мутацию на основе ПЦР усиления плазмида укрывательство гена интереса с использованием мутагенных грунтовки и высокой точности ДНК-полимеразы, содержащие Pfu в 50 Зл общего объема реакции ПЦР, как показано в таблице 1 и таблице 2 в соответствии с протоколом производителя.
- Дайджест Escherichiacoli-производной родительской метилированных и полуметилированных ДНК, добавив 3 Зл фермента ограничения DpnI непосредственно к реакции ПЦР (шаг 1.2) и инкубации при 37 кс для 2 ч.
ПРИМЕЧАНИЕ: Метилирование является посттранскриптовая модификация белка, которая добавляется в плазмид производства и выделены из бактерий. Новые копии ПЦР-генерируемых плазмид отсутствие метилирования, поэтому, новые копии останутся нетронутыми во время лечения DpnI. - Очистите мутагенизированные плазмиды с помощью коммерческого комплекта для извлечения ДНК на основе технологии спин-колонки и увядните ДНК с 50 зл и водой.
- Преобразуйте ХИМИЧЕСКИ компетентные клетки DH5'e. coli с 0,5 л восстановленной мутагенизированной плазмидной ДНК в соответствии с протоколом производителя. Вкратце, инкубировать компетентные клетки с дна на льду на 30 min, после этого жар-шокирует их для 20 s на 42 c, и устанавливает пробки снова на льду на 2 min. Инкубировать клетки с 500 qL средств LB на 37 c для 1 h на 250 rpm и после этого поместите их на селективных паштетах.
- Проверить желаемую мутацию Сэнгер секвенирования ДНК плазмиды изолированы от кишечной палочки.
2. Рекомбинантная очистка белка и анализ убиквитинации в пробирке
- Клонировать дикий тип RING и мутировавших генов RING, представляющих интерес в вектор pMAL-c2 (следуйте протоколу производителя; Таблица 3) чтобы сплавить эти гены с эпитопом MBP тегом, который позволяет одношагнюю очистку с использованием амилозы. Введите полученные конструкции в штамм E. coli BL21, описанный в шаге 1.5.
- Выращивайте штамм E. coli BL21, укрывающий нужную конструкцию в жидкой среде 50 мл ЛБ при 37 градусах По Цельсия в течение 2-3 ч, пока не достигнет логарифмической фазы (OD600 0,4-0.6).
- Добавьте IPTG к конечной концентрации 0,1-1 мм, чтобы вызвать выражение интересуемого интересуемого для MBP рекомбинантного белка MBP и инкубировать культуру кишечной палочки на 2-3 ч при 28 градусах Цельсия. Поместите культуру на лед после инкубации.
ПРИМЕЧАНИЕ: Выполните шаги 2.4-2.13 на льду для защиты белков от деградации. - Чтобы проверить эффективность индукции, соберите 1,5 мл индуцированных клеток, спина их вниз на 13000 х г в течение 2 мин, удалить супернатант, и resuspend клетки в 20 злител 2x SDS-PAGE загрузки буфера (24 mM Tris-HCl рН 6,8, 0,8% SDS, 10% (v/v) глицерол, 4 мМ DTT, 0,04% (ww/blue brom).
- Отварить образцы в течение 5 минут и запустить их на 10% SDS-PAGE гель. Чтобы визуально оценить накопление белка MBP-фьюжн (молекулярный вес белка интерес42,5 kDa MBP), пятно гель в течение 20 минут, агитируя с Coomassie окрашивания буфера (50% метанола, 10% уксусной кислоты, 0,1% Coomassie синий) и дестанагирование ночь с буфер дестабилизации (20% метанола, 10% уксусной кислоты).
- Урожай оставшихся клеток кишечной палочки центрифугирование на 1350 х г в течение 6 мин, отбросить супернатант, и resuspend клетки гранулы с 5 мл столбцов (20 мм Tris HCl, 200 мм NaCl, 1 мМ EDTA, бактериальный ингибитор протеазы).
ПРИМЕЧАНИЕ: Это хорошее место, чтобы остановить протокол на ночь. Замороженные ячейки могут храниться до 1 недели при -20 градусах Цельсия. - Разбейте клетки кишечной палочки, поместив трубку, содержащую бактерии, в ванну с ледяной водой и применяя 10 циклов звукоизоляции: 10 s sonication на 30% усилителе, а затем 20 с перерывами.
- Centrifuge образец на 13000 х г при 4 кв кв в течение 10 минут и сохранить супернатант (сырой экстракт).
- Приготовьте 500 л амилозы в трубке длиной 15 мл. Вымойте сена, добавив 10 мл холодного буфера столбца и центрифуги при 1800 х г,4 кв. м в течение 5 мин. Сделай это в 2 x.
- Добавьте 5 мл сырого экстракта в трубку с мичтовой мизины и инкубировать на ночь при 4 градусах Цельсия.
- Центрифуга при 1800 х г при 4 градусах по Цельсию в течение 5 мин и отбросьте супернатант.
- Добавьте 10 мл буфера столбца в гранулы из шалотки из минины и насищите в течение 20 минут. Затем центрифуга на 1800 х г при 4 c в течение 5 мин. Повторите этот шаг 2x.
- Выясните синтез белка с 0,5 мл столбцового буфера, содержащего 10 мМ мальтоза путем инкубации образца для 2 ч при 4 C. Центрифуге при 1800 х г при 4 градусах по Цельсию в течение 5 мин и соберите элетированный белок. Повторите этот шаг 2x.
- Диализ 1 мл белковой фракции против холодного PBS. Аликвотный белок в одноразовые трубки (10-20 л), чтобы избежать замораживания оттаивания и хранить при -80 градусов по Цельсию до тех пор, пока это необходимо.
- Измерьте концентрацию белка с помощью Брэдфорда анализ9.
- Настройка реакции убиквитинации in vitro в общем объеме 30 qL путем смешивания 40 нг E1 (например, AtUBA1), 100 нг E2 (например, AtUBC8, SlUBC1/4/6/7/12/13/17/20/22/27/32), 1BP и 2 мкг FLAG-Ub (или HA-Ub) в буфере вездесущности (50 мм Tris-HCl pH 7.5, 2 mM ATP, 5 мМ MgCl2, 30 мм креатина фосфата, 50 мкг/мл креатина фосфокиназы). Инкубировать смесь при 30 градусах по Цельсию в течение 2 ч.
ПРИМЕЧАНИЕ: Premake 20x убиквитинации буфера и хранить его до 6 месяцев при -20 градусов по Цельсию в небольших aliquots для одного использования. Креатин фосфокиназа легко теряет свою ферментативную активность, когда буфер размораживаются и замораживаются неоднократно. - Прекратите реакцию, смешивая 30 образцов СЛ с 7,5 л 5x SDS-PAGE погрузочный буфер (60 мм Tris-HCl pH 6.8, 2% SDS, 25% (v/v) глицерол, 10 мМ DTT, 0,1% (w/v) бромов синий) и кипения в течение 5 минут.
- Отделите белки с 7,5% SDS-полиакриламид гель электрофорез (SDS-PAGE), а затем передать на мембрану PDVF, и обнаружить убиквитинации с помощью анти-FLAG (или анти-HA).
- Пятно PVDF мембраны с Coomassie синий, чтобы проверить равную нагрузку испытания Белка типа MBP-RING.
3. Агробактерии-опосредованного переходного выражения белка в листьях Nicotiana benthamiana и в анализе убликвиции растений
- Полоса соответствующих Agrobacterium tumefaciens штаммов, перевозящих эпитоп-тегами гена интереса (например, HA-RHA1B, HA-RHA1BC135S, HA-RHA1BK146R, HA-Ub) и пустой вектор в качестве контроля на среде LB, содержащий соответствующие антибиотики выбора.
- После 2 дней роста при 28 градусах Цельсия, подобрать одиночных колоний и выращивать их в ЖИДКОй среде LB с соответствующими антибиотиками на 28 кв/250 об/мин еще 24 ч.
- Перенесите 100 л агробактериальной культуры на 3 мл свежего LB с соответствующими антибиотиками и инкубировать культуру в течение дополнительных 4-6 ч при 28 градусах Цельсия с вращением (250 об/мин) до поздней фазы экспоненциального роста.
- Спин вниз агробактериальных клеток на 1800 х г в течение 6 мин, отбросить супернатант, и resuspend клетки с 3 мл мыть буфера (50 мм MES pH 5,6, 28 мм глюкозы, 2 мМ2PO4). Повторите этот шаг 2x.
- После второй стирки, resuspend клетки в индукционном буфере (50 мМ MES pH 5.6, 28 mM глюкозы, 2 мМ2 PO4, 200 мкм acetosyringone, 37 мМ NH4Cl, 5 мМ MgSO4.7H2O, 4 мМ KCl, 18 mM MM4.7H2. Инкубировать клетки индукционным буфером для дополнительного 10-12 ч при 28 градусах Цельсия.
ПРИМЕЧАНИЕ: Ацетосирингон индуцирует передачу Т-ДНК. - Центрифуги клетки на 1800 х г в течение 6 мин, отбросить супернатант, и resuspend клетки с 2 мл инфильтрационного буфера (10 мм ММ MES pH 5.5, 200 мкм ацетосирирингон).
ПРИМЕЧАНИЕ: Если Agrobacteria агрегат после инкубации с индукционным буфером, пусть агрегированные клетки опускаются на дно трубки, оставляя его на скамейке в течение нескольких минут, и передать четкую подвеску Agrobacterium в новую трубку, прежде чем приступить к шагу 3.6. - Измерьте концентрацию бактерий, используя значение OD600 (оптическая плотность при абсорбции 600 нм). Отрегулируйте значения OD600 на желаемые.
ПРИМЕЧАНИЕ: Обычно значение OD600 между 0.2-0.4 работает наиболее наилучшим образомнаилучшим образом для одиночного агробактериального выражения пятна. При применении сочетания различных агробактериальных штаммов, то общие значенияOD 600 агробактериальных штаммов не должны превышать 1. - Agroinfiltrate 4-недельный Н. bethamiana листья, нежно колоть их с иглой, а затем ручной инъекции Агробактерии со шприцем без иглы. Обведите проинфильтртированную область листа маркером (обычно диаметром 1-2 см).
- Соберите проникнутые ткани листьев 36 h после инфильтрации. Измельчить ткань до мелкого порошка с жидким азотом.
- Переутомляемый тканевой порошок с 300 йл буфера экстракции белка (50 мм Tris-HCl pH 7.5, 150 мМ NaCl, 5 мМ EDTA, 2 мМ DTT, 10% глицерола, 1% поливинилполипирролидон, 1 мМ PMSF, коктейль-ингибитор протеазы растений) и центрифуга при 15 000 мг на 15 мин при 4 градусах Цельсия.
- Перенесите супернатант на новую трубку. Добавьте 5x sDS-PAGE погрузочный буфер к конечной концентрации 1x и кипятите в течение 5 мин.
- Отдельные сырые белки на 10% SDS-PAGE гели, передача на PVDF мембраны, и зонд с анти-HA для обнаружения в убиквитинации растений.
4. Установление связи между ферментативной деятельностью и функцией в плант
ПРИМЕЧАНИЕ: Например, RHA1B способствует деградации резистентного белка Gpa2 для подавления смерти HR клеток. Этот шаг показывает, как проверить, что эти вирулентные действия RHA1B являются E3-зависимыми.
- Полоса соответствующих Agrobacterium tumefaciens штаммов проведения помечены гены интереса (в этом примере HA-RHA1B, HA-RHA1BC135S, HA-RHA1BK146R, myc-Gpa2, RBP1) и пустой вектор в качестве контроля. Выполните шаги 3.1-3.8 для приготовления агробактерии и инъекции на листьях N. bethamiana.
- Для E3-зависимой деградации белка субстрата, следуйте шагам 3.9-3.12 и выполняйте западное blotting используя соотвествующие антитела для того чтобы обнаружить накопление протеина в клетках завода (например, anti-HA и anti-MYC).
- Для E3-зависимых гиперчувствительных ответ (HR) опосредованное ингибирование смерти клетки, контролировать агроинфильтранных листьев для HR-клеточных симптомов смерти 2-4 дней после инфильтрации.
Результаты
В этом разделе, репрезентативные результаты предоставляются для протокола, используемого для изучения одного подразделения E3 убиквитин лигаза RHA1B, который имеет PROSITE-предсказал RING-H2 типа домена (132-176 аминокислот)10. Как показано на рисунке 1, для того, чтобы получить E3-дефицитный мутант белка, по крайней мере один из восьми консервированных Cs или Hs в домене RING (Рисунок 1А) должен быть мутагенизирован(Рисунок 1B). Таким образом, в качестве первого шага были созданы две мутантные версии RHA1B, RHA1BC135S (замена Cys Ser в сохраненном C3 домена RING) и RHA1BK146R (замена Lys by Arg в единственном Lys, присутствующем в RHA1B). Хотя одноподразделение E3 ligases посредничает убиквитин передачи от убиквитин укрывательство E2 в субстрате, а не непосредственно взаимодействующих с убиквитин, самоубиквиции E3 в Лисе может потребоваться для его максимальной ферментативной деятельности.
Западные результаты blotting на рисунке 2a показывают типичный положительный результат убликвитисации in vitro, с multibanding мазком starting at молекулярный вес испытанного протеина (например, MPB-сросенный RHA1B 100 kDa) и прогрессируя вверх. Анти-HA антитела признали HA-тегами Ub включены в поли-убиквитинации цепи различной длины, создавая этот типичный убиквитин связанных лестницы, как мазок. Для подтверждения положительных результатов на рисунке 2А также представлены все важные отрицательные элементы управления, не достижимые отдельными компонентами (E1, E2, Ub или MBP-RHA1B) или использующие MBP в качестве контроля и отсутствие смазанный сигнал вездесущности. Кроме того, синее окрашивание мембраны PVDF показало равную нагрузку MBP-RHA1B или MBP во всех элементах управления.
На рисунке 2B показано, как результаты повсеместного убликвитирования in vitro менялись в зависимости от конкретной комбинации E2/E3. В этом примере было протестировано 11 различных E2, представляющих 10 различных семейств E2. Обнаруженная активность убиквитинации варьировалась от отсутствие сигнала (без мазка) до многополосного мазка, начинающегося с разных молекулярных весов, что указывает на различные модели убиквитинации.
На рисунке 3 показаны результаты анализов вездесущности для версий РИНГ- и K-мутантов проверенного белка. Отсутствие ферментативной активности для RHA1BC135S поддерживается его неспособностью либо генерировать многополосный мазок in vitro(рисунок 3A),либо способствовать поли-убиквитизации сигнала в растения(Рисунок 3B). Примечательно, что переэкспрессия HA-tagged Ub в плану сама по себе дала базальный убиквитинация во всех проверенных образцах, включая контроль переносчиков, в отличие от сильного сигнала убиквитизации, присваиваемого ферментативной активностью дикого типа RHA1B. Кроме того, анализ мутанта RHA1BK146R позволяет предположить, что остаток K146 также имеет важное значение для деятельности E3 RHA1B. Хотя маргинальный сигнал самоубиквитирования был обнаружен в пробирке(рисунок 3A), в анализе планты определяется мутант E3-дефицит(Рисунок 3B, только фон убиквитинации сигнал обнаружен).
После генерации и биохимически йапоза мутанта, дефиците е3, функциональные исследования могут быть разработаны для определения ассоциированной Е3 биологической роли испытанной убиквитинной лигиции РИНГ E3. В случае RHA1B, этот эффектнера нематода подавляет иммунную сигнализацию растений, как проявляется подавлением смерти HR-клеток, вызванных Gpa2. Как представлено на рисунке 4A, в отличие от дикого типа RHA1B, RHA1BC135S мутант отсутствует E3 ligase деятельности не вмешиваться в смерти HR ячейки. Учитывая, что наиболее распространенным результатом убиквитина белка является его протеасомо-опосредованное деградация, мутации, проживающие в домене RING, также могут быть использованы для проверки способности, зависящей от Е3, чтобы вызвать деградацию их прямых и/или косвенных субстратов. Таким образом, значительно, западные результаты blotting в Рисунке 4B подтверждают что Gpa2 не аккумулировало в присутсвии одичалого типа RHA1B но RHA1BC135S не имело никакое влияние на стабилности протеина Gpa2.
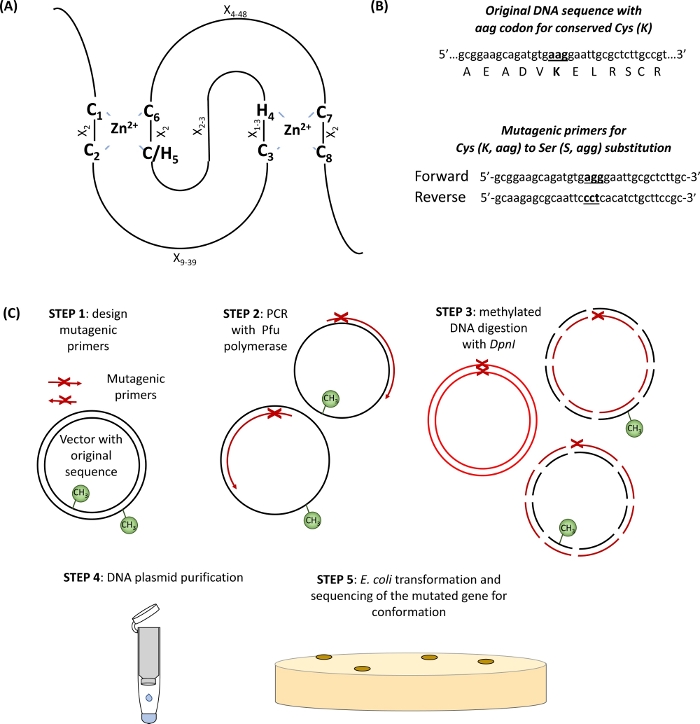
Рисунок 1: Схематическое представление принципа и шагов, связанных с мутагенезом, направленным на сайт. (A) RING-CH/H2 домен с сохраненными Ки с и его аминокислоты выделены. (B) Пример мутагенных грунтовки дизайн. (C) Шаги сайта направлены мутагенез. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
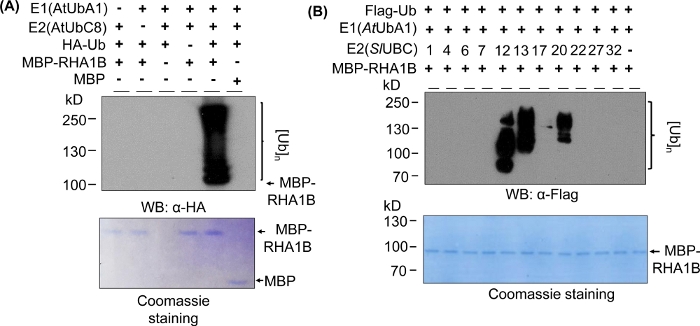
Рисунок 2: Представитель in vitro увселиления анализа. (A) Верхний гель показывает убиквиционную проверку, включая все негативные элементы управления, а нижний гель показывает равную нагрузку. (B) Диапазон ожидаемых результатов в зависимости от ферментов E2. Эта цифра была изменена с Kud et al8. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
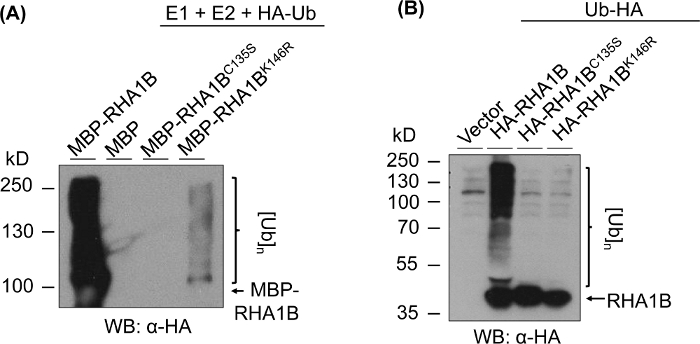
Рисунок 3: Результаты опроса по повсеместному осваивания для RING- и K-мутантов (RHA1BC135S и RHA1BK146R). (A) Результаты повсеместного убиквитирования RHA1BC135S и RHA1BK146R. (B) В плану повсеместного исследования результатов для RHA1BC135S и RHA1BK146R. Эта цифра была изменена с Kud et al8. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
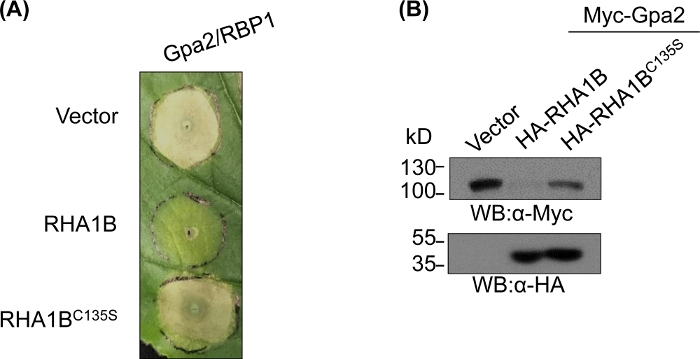
Рисунок 4: Представительфункциональное исследование для Е3 зависимых биологических функций. Пример функциональных исследований, показывающих е3-зависимую биологическую функцию. (A) E3-зависимых ПОдавления смерти клетки HR и (B) деградации иммунорецептора растений Gpa2. Эта цифра была изменена с Kud et al8. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
| Настройка ПЦР | |
| 1 зл | плазмид (100 нг) |
| 1,5 л | F мутагенная грунтовка (10 мкм) |
| 1,5 л | R мутагенная грунтовка (10 мкм) |
| 1 зл | dNTPs (10 mM) |
| 5 зл | буфер (10x) |
| 1 зл | Ультра-пу полимераза (2,5 U/л) |
| 39 зл | ddH2O |
| 50 зл | ОБЩИЙ ОБЪЕМ |
Таблица 1: Настройка реакции ПЦР
| термоциклоза | |||
| 1 | 95 кв. c | 30 с | |
| 2 | 95 кв. c | 30 с | |
| 3 | 60 кк | 30 с | |
| 4 | 72 кк с | 5 мин. | повторить 2-4 30 раз |
| 5 | 72 кк с | 5 мин. | |
Таблица 2: Программа термоциклора ПЦР
| реакции перевязки, созданной для примера RHA1B | ||
| 1,5 л | pMAL-c2::MBP линейный вектор путем пищеварения с BamHI и SalI (60 нг) | |
| 7 л | RHA1B/RHA1BC135S или RHA1BK146R вставка переваривается с BamHI и SalI (25 нг) | |
| 1 зл | Буфер лиги T4 (10x) | |
| 0,5 л л | Лигаза Т4 (400 U/L) | |
| 10 зл | ОБЩИЙ ОБЪЕМ | |
Таблица 3: Реакция лигации, созданная для примера RHA1B.
Обсуждение
Прояснения биохимической и механистической основы РИНГ типа E3 убиквитин лигаза может внести большой вклад в наше понимание их биологического значения в развитии, стресс сигнализации, и поддержание гомеостаза. Протокол описано здесь пары mutagenesis подход с in vitro и в планку функциональных исследований. Вводя одну замещение аминокислот в сохраненных остатках домена RING через прямой мутагенез, полученный E3-дефицитный мутант может быть протестирован параллельно с диким белком типа, чтобы связать ферментативную активность с функциональностью.
Очень важно правильно определить домен RING, в частности его сохраненные Ки и его остатки. Онлайн-инструменты, такие как PROSITE могут быть использованы для этого10. Для дестабилизации домена RING, ответственного за набор фермента E2, Cys обычно заменяется Ser, который является его ближайшей структурной заменой, не способной создать дисульфидную связь, используемую для координации цинка. Lorick et al. показали, что мутация в любом из этих критических остатков Cys отменит убиквитивационную активность одного подразделения ринг-типа E3 ligases5. Хотя некоторые остатки Cys также важны для многоузловых e3 лигазических комплексов, содержащих белки типа RING, из-за многогранной и динамической трехмерной структуры этих комплексов убиквитина и различной роли белков типа РИНГ, единичные замены сэкономленных остатковв домене RING в многоузловой Лиге E3 ligase не были успешными в генерации a ligase.
Для сайта направлены мутагенез, мы обнаружили, что использование небольших переносчиков плазмида и более низких циклов усиления обычно дает более высокую эффективность для мутагенеза. Фермент Pfu можно заменить любой другой высокой точностью и высокой процессивативностью ДНК-полимеразой. Кроме того, если интересующий ген содержит редкие кодоны, другое пятно кишечной палочки, Rosetta, может быть использовано для достижения более высокой урожайности рекомбинантного белка. Кроме того, можно дополнительно оптимизировать как время инкубации, так и температуру индукции IPTG. Более низкие температуры снижают скорость деления кишечной палочки, что может быть благоприятным для выражения определенных белков. Хотя более высокая концентрация IPTG может улучшить экспрессию белка, она также подавляет процессы деления кишечной палочки и не рекомендуется.
Одноразовая субединица типа E3 ligases типа E3 не только функционирует как молекулярная эшафот, которая позиционирует промежуточный E2-Ub в непосредственной близости от субстрата, но и стимулирует активность переноса убиквитина их cognate E2s. Кроме того, учитывая, что комбинация E2/E3 важна для длины и связей поликубиквина цепи, которая определяет судьбу модифицированного субстрата, любое рассмотрение ТИПА RING E3s должно включать в себя их ферментативные партнеры, E2s12. Как показано на рисунке 3B, не все проверенные E2s совместимы с LIgase RHA1B. Таким образом, in vitro убиквитинация анализы должны осуществляться параллельно с несколькими ферментами E2, представляющих различные классы E2, чтобы избежать ложных отрицательных результатов.
Здесь представлена ферментативная проверка in vitro, которая определяет способность самоубиквитинации проверенных белков типа РИНГ. Однако, с небольшими изменениями, этот протокол может быть легко адаптирован для обнаружения in vitro повсеместного субстрата. С этой целью смесь убиквитивания in vitro со ступени 2.15 должна быть дополнена рекомбинантным белком потенциального субстрата E3 ligase (500 нг). После 2 ч инкубации при 30 градусах Цельсия, убиквитинат белок должен быть захвачен с помощью 15 зЛ анти-HA сродства матрицы (если HA-Ub используется, или анти-FLAG сродство матрицы, если FLAG-Ub используется) агитации в течение 2 ч при 4 градусах по Цельсию. После мытья бисера 4x раз с холодным буфером мытья Ub (20 mM Tris pH 7.5, 100 mM NaCl, 0.1 mM EDTA, 0.05% Tween 20, 1x PMSF), сбросите все, кроме 40 л буфера и перейдите к шагу 2.16. Сигнал убиквитинации, обнаруженный антителами, специфичными для эпитопа-тегами Ub и субстрата, соответственно, выходящими из молекулярного веса белка субстрата, подтверждает специфичность субстрата/фермента.
Кроме того, идентификация субстратов E3 ligase in vivo обычно связана с многочисленными проблемами, связанными с переходным взаимодействием ферментосубстрата и быстрой деградацией убиквитинаированного целевого белка. Использование E3 лигазы дефицит мутант, который по-прежнему взаимодействует со своей целью, но больше не убиквитинат его13, является очень полезной альтернативой добавлению протеасомального ингибитора MG132, который не всегда достаточно вмешиваться в 26S протеасомы функции.
Общей характеристикой лигаза хавки типа РИНГ а это тенденция к формированию и функционированию в качестве гомо-и/или гетеродимеров. Интересно, что замена в сохраненных остатках домена RING обычно ассоциируется с доминирующим отрицательным фенотипом, где мутировавшая лигаза типа РИНГ блокирует ферментативную активность местного дикого белка13. Таким образом, переэкспрессия ингинговых мутантов в плане может быть альтернативным подходом к выбиванию гена Лигаза E3.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Наша работа стала возможной благодаря финансовой поддержке конкурсного гранта Инициативы по сельскохозяйственным и продовольственным исследованиям (2017-67014-26197; 2017-67014-26591) Национального института продовольствия и сельского хозяйства МИНИСТЕРСТВА сельского хозяйства США, USDA-NIFA Farm Bill, Northwest Potato консорциум, и ISDA Специальность урожая.
Материалы
| Name | Company | Catalog Number | Comments |
| Acetic acid | Sigma-Aldrich | A6283 | |
| Acetosyringone | Sigma-Aldrich | D134406 | |
| Amylose resin | NEB | E8021S | |
| ATP | Sigma-Aldrich | A1852 | |
| Bacterial protease inhibitor | Sigma-Aldrich | P8465 | |
| Bromphenol Blue | VWR | 97061-690 | |
| CaCl2 | Sigma-Aldrich | C1016 | |
| Centrifuge | Beckman Coulter | model: Avanti J-25 | |
| Commassie Blue | VWR | 97061-738 | |
| Creatine phosphate | Sigma-Aldrich | P7936 | |
| Creatine phosphokinase | Sigma-Aldrich | C3755 | |
| DNA clean & concentrator Kit | ZYMO RESEARCH | D4029 | |
| DpnI | NEB | R0176S | |
| DTT | Sigma-Aldrich | D0632 | |
| E. coli BL21 | Thermo Fisher Scientific | C600003 | |
| E. coli DH5α competent cells | Thermo Fisher Scientific | 18265017 | |
| EDTA | Sigma-Aldrich | 324504 | |
| FeSO4 7H2O | Sigma-Aldrich | F7002 | |
| FLAG-Ub | BostonBiochem | U-120 | |
| Glucose | VWR | 188 | |
| Glycerol | Sigma-Aldrich | G5516 | |
| HA-Ub | BostonBiochem | U-110 | |
| Heat block | VWR | model: 10153-318 | |
| Incubator | VWR | model: 1525 Digital Incubator | |
| Incubator shaker | Thermo Fisher Scientific | model: MaxQ 4000 | |
| IPTG | Roche | 10724815001 | |
| KCl | Sigma-Aldrich | P9333 | |
| LB Broth | Sigma-Aldrich | L3022 | |
| Liquide nitrogen | university chemistore | ||
| Maltose | Sigma-Aldrich | 63418 | |
| MES | Sigma-Aldrich | M3671 | |
| Methanol | Sigma-Aldrich | 34860 | |
| MgCl2 | Sigma-Aldrich | 63138 | |
| MgSO4 7H2O | Sigma-Aldrich | 63138 | |
| Microcentrifuge | Eppendorf | model: 5424 | |
| Miniprep plasmid purification kit | ZYMO RESEARCH | D4015 | |
| monoclonal anti-FLAG antibody | Sigma-Aldrich | F3165 | |
| monoclonal anti-HA antibody | Sigma-Aldrich | H9658 | |
| monoclonal anti-MYC antibody | Sigma-Aldrich | WH0004609M2 | |
| Mortar | VWR | 89038-144 | |
| NaCl | Sigma-Aldrich | S7653 | |
| NaH2PO4 | Sigma-Aldrich | S8282 | |
| NanoDrop | Thermo Fisher Scientific | model: 2000 Spectrophotometer | |
| Needle | Thermo Fisher Scientific | 14-826-5C | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| PCR machine | Bio-Rad | model: C1000 | |
| Pestle | VWR | 89038-160 | |
| Pfu Ultra | Agilent Technologies | 600380 | |
| Plant protease inhibitor coctail | Sigma-Aldrich | P9599 | |
| pMAL-c2 | NEB | N8076S | |
| PMSF | Sigma-Aldrich | P7626 | |
| Polyvinylpolypyrrolidone | Sigma-Aldrich | P6755 | |
| SDS | Sigma-Aldrich | 1614363 | |
| Sonicator | Qsonica Sonicators | model: Q125 | |
| Syringe | Thermo Fisher Scientific | 22-253-260 | |
| Tris | Sigma-Aldrich | T1503 | |
| T4 ligase | NEB | M0202S |
Ссылки
- Freemont, P. S., Hanson, I. M., Trowsdale, J. A novel gysteine-rich sequence motif. Cell. 64, 483-484 (1991).
- Borden, K. L. B. RING fingers and B-boxes: Zinc-binding protein-protein interaction domains. Biochemistry and Cell Biology. 76, 351-358 (1998).
- Barlow, P. N., Luisi, B., Milner, A., Elliott, M., Everett, R. Structure of the C3HC4 Domain by 1H-nuclear Magnetic Resonance Spectroscopy: A New Structural Class of Zinc-finger. Journal of Molecular Biology. 237, 201-211 (1994).
- Borden, K. L. B., et al. The solution structure of the RING finger domain from the acute promyelocytic leukaemia proto-oncoprotein PML. The EMBO Journal. 14, 1532-1541 (1995).
- Lorick, K. L., et al. RING fingers mediate ubiquitin-conjugating enzyme (E2)-dependent ubiquitination. Proceedings of the National Academy of Sciences. 96, 11364-11369 (1999).
- Jiménez-López, D., Muñóz-Belman, F., González-Prieto, J. M., Aguilar-Hernández, V., Guzmán, P. Repertoire of plant RING E3 ubiquitin ligases revisited: New groups counting gene families and single genes. PLoS ONE. 13, 1-28 (2018).
- Sacco, M. A., et al. The Cyst Nematode SPRYSEC Protein RBP-1 Elicits Gpa2- and RanGAP2-Dependent Plant Cell Death. PLoS Pathogens. 5, 1-14 (2009).
- Kud, J., et al. The potato cyst nematode effector RHA1B is a ubiquitin ligase and uses two distinct mechanisms to suppress plant immune signaling. PLoS Pathogens. 15, 1007720 (2019).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72, 248-254 (1976).
- Sigrist, C. J. A., et al. New and continuing developments at PROSITE. Nucleic Acids Research. 41, 344-347 (2013).
- Dove, K. K., Stieglitz, B., Duncan, E. D., Rittinger, K., Klevit, R. E. Molecular insights into RBR E3 ligase ubiquitin transfer mechanisms. EMBO Reports. 17, 1221-1235 (2016).
- Metzger, M. B., Pruneda, J. N., Klevit, R. E., Weissman, A. M. RING-type E3 ligases: Master manipulators of E2 ubiquitin-conjugating enzymes and ubiquitination. Biochimica et Biophysica Acta - Molecular Cell Research. 1843, 47-60 (2014).
- Xie, Q., et al. SINAT5 promotes ubiquitin-related degradation of NAC1 to attenuate auxin signals. Nature. 419, 167-170 (2002).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены