Method Article
インビトロとプランタにおけるRING型E3ユビキチンリガーゼの機能特性評価
要約
この原稿の目的は、RING型E3ユビキチンリガーゼの包括的な生化学的および機能的研究の概要を提示することです。このマルチステップパイプラインは、詳細なプロトコルを用いて、試験されたタンパク質の酵素活性を検証し、活性を機能にリンクする方法を示す。
要約
ユビキチン化は、タンパク質の翻訳後修飾として、真核細胞の恒常性において重要な調節的役割を果たす。標的タンパク質への76アミノ酸ユビキチン修飾剤の共有結合は、ポリウビキチン鎖の長さおよびトポロジーに応じて、タンパク質分解から修飾タンパク質の局在化および/または活性の変化に至るまで、異なる結果をもたらす可能性がある。3つの酵素がユビキチン化プロセスを順次触媒する:E1ユビキチン活性化酵素、E2ユビキチン共役酵素、およびE3ユビキチンリガーゼ。E3ユビキチンリガーゼは、基質特異性を決定し、したがって、非常に興味深い研究対象を表す。ここでは、RING型E3ユビキチンリガーゼの酵素活性と機能との関係を研究するための包括的なアプローチを提示する。この4段階のプロトコルは、保存されたRINGドメインを標的とした部位特異的突然変異誘発を介してE3リガーゼ欠損変異体を生成する方法を説明する。2-3)インビトロとプランタの両方のユビキチン化活性を調べる方法;4)これらの生化学的分析を試験タンパク質の生物学的意義に結びつける方法。E3リガーゼ欠損変異体の生成は、まだその基質と相互作用するが、もはや分解のためにそれをユビキチン化し、生体内での酵素基質相互作用の試験を容易にする。さらに、保存されたRINGドメインにおける突然変異は、RNA干渉アプローチの代替アプローチとして機能的ノックアウト研究で利用できる支配的な陰性表現型をしばしば付与する。我々の方法は、植物細胞内の宿主ユビキチン化システムをハイジャックして寄生を促進する植物寄生線虫エフェクターRHA1Bの生物学的役割を調べるために最適化された。in vivo発現システムのわずかな変更によって、このプロトコルは起源に関係なくあらゆるRING型E3リガーゼの分析に適用することができる。
概要
E3ユビキチンリガーゼの大部分は、RING(Really Interesting New Gene)型タンパク質に属する。RINGフィンガードメインは、もともとフリーモントらによって識別されました.1および機能的にタンパク質間相互作用を媒介するドメインとして記載される2.正規の輪ばれる指は、他のアミノ酸残基(X)によって特異的に間隔を空けた8つの保存されたCys(C)およびHis(H)のコンセンサス配列として定義される特殊なタイプの亜鉛調整ドメインであり、C-X2-C-X1-3-H-X2-3-C/C-X 2 -C-X 4-48 -C-X-C-X 2 -C-X2-C-X4-48-C-X 2 -C-X2つのZn2+イオンは、C1/C2およびC/H5 /C6を有するユニークな「クロスブレース」トポロジーを介してコアCおよびH残基によって安定化され、C3/H4およびC7/C8は第2(図1A)3、4を結合する最初のZn 2+イオンを調整する。第5Zn2+配位部位におけるCまたはHの存在に応じて、リングフィンガータンパク質の2つの正規サブクラスが定義された:C3HC4およびC3H2C3(RING-HCおよびRING-H2、それぞれ)。E3ユビキチンリガーゼのRINGドメインはE2共役酵素と基質との相互作用を媒介するので、これらの必須CおよびH残基の突然変異はリガーゼ活性5を破壊することが示されている。RING E3 リガーゼのさらに 5 つのあまり一般的でないサブクラス (RING-v、RING-C2、RING-D、RING-S/T、および RING-G)6が説明されています。RING型E3ユビキチンリガーゼは、さらに単純で複雑なE3酵素に細分化することができる。単純な単一サブユニットRINGE3リガーゼは、基板認識部位およびE2結合リングドメインの両方を含む。対照的に、マルチサブユニットRING型E3複合体は、E2-ユビキチン中間体のE3複合体へのリクルート基板または媒仲介する。自己ユビキチン化のための一次ユビキチン結合部位として機能するRINGドメインLys残基は、E3リガーゼ活性にとっても重要である可能性がある。
すべてのRING含有タンパク質がE3リガーゼとして機能するわけではありません。したがって、RING-fingerドメインのバイオインフォマティクス予測とE2依存性タンパク質ユビキチン化の能力は、生化学的に検証され、試験されたタンパク質の生物学的役割にリンクする必要があります。ここでは、サイト指向の突然変異誘発アプローチを通じて、in vitroとプランタの両方でRING型E3ユビキチンリガーゼの酵素活性を検出し、機能的に特徴付ける方法を概説するステップバイステッププロトコルについて説明する。このパイプラインからの代表的な結果は、RING型E3リガーゼRHA1Bについて示されています。RHA1Bは、植物の免疫を抑制し、植物根細胞の形態を操作するために植物寄生性嚢胞線腫性グロボデラ・パリダによって産生されるエフェクタータンパク質である。病原体/寄生虫の侵入から身を守るために、植物は病原体または寄生虫の存在を検出するヌクレオチド結合ドメインおよびロイシンリッチ反復(NB-LRR)型免疫受容体を進化させ、その結果、感染部位で起こる急速で局所的な細胞死の一形態である過敏性応答(HR)を発症し、病原体のコロニー形成を阻止する。そのような免疫受容体の1つは、G.パリダのいくつかの単離物(フィールド集団D383およびD372)7に対する耐性を付与するジャガイモGpa2タンパク質である。
提示されたプロトコルを用いて、RHA1Bが植物Gpa2免疫受容体をユビキチン化および分解8に標的化することによりE3依存的に植物免疫シグナル伝達を妨害することが最近判明した。
プロトコル
1. 部位特異的突然変異誘発 (図 1)
- RINGドメイン内の保存されたCysおよびHisアミノ酸を同定し(図1A)、および変異部位の両側に15塩基対で横たわって目的の置換コドンを運ぶ設計プライマー(図1B)。
- 製造業者のプロトコルに従って、全PCR反応量の50μLでPfuを含む変異原プライマーおよび高忠実度DNAポリメラーゼを用いて目的の遺伝子を収容するプラスミドのPCRベースの増幅による所望の変異を導入する。
- 大腸菌由来の親メチル化および半メチル化DNAを消化し、DpnI制限酵素の3μLをPCR反応に直接加え(ステップ1.2)、37°Cで2時間インキュベートします。
注:メチル化は、産生され、細菌から単離されたプラスミドに添加される転写後タンパク質修飾である。PCR生成プラスミドの新しいコピーはメチル化を欠いているため、DpnI治療中に新しいコピーはそのまま残ります。 - スピンカラム技術に基づく市販のDNA抽出キットを用いて変異型プラスミドを精製し、50μLの水でDNAを溶出します。
- DH5α大腸菌は、製造業者のプロトコルに従って、回収された変異型プラスミドDNAの0.5μLで化学的に有能な細胞を変換します。簡単に言えば、氷上のDNAを含む有能な細胞を30分間インキュベートし、42°Cで20時間加熱衝撃し、2分間氷上にチューブを置き、500μLのLBメディアを37°Cで250rpmで1時間インキュベートし、選択的なパテに広げます。
- 大腸菌から単離されたDNAプラスミドをシーケンシングするサンガーによって所望の突然変異を検証する。
2. 組換えタンパク質精製とインビトロユビキチン化アッセイ
- 目的の野生型RINGと変異したRING遺伝子をpMAL-c2ベクターに複製します(メーカーのプロトコルに従ってください。表 3)これらの遺伝子を、アミロース樹脂を用いたワンステップ精製を可能にするMBPエピトープタグと融合させる。ステップ1.5で説明したように、得られた構成要素を大腸菌BL21株に導入する。
- 大腸菌株BL21を成長させ、対数相(0.4~0.6のOD600)に達するまで、37°Cで50mL LB液体培地中の所望の構造を2〜3時間持ち込む。
- IPTGを0.1~1mMの最終濃度に加えて、目的とするMBPタグ組換えタンパク質の発現を誘導し、28°Cで2~3時間大腸菌培養物をインキュベートします。インキュベーション後に氷の上に培養物を置きます。
メモ:氷上でステップ2.4~2.13を実行して、タンパク質を劣化から保護します。 - 誘導効率を確認するには、誘導細胞の1.5 mLを収集し、 2分間13,000 x gでスピンダウンし、上清を取り除き、2x SDS-PAGEローディングバッファ(24 mM Tris-HCl pH 6.8、0.8%SDS、10%(v/v)グリセロール、4 mM DTT、0.04%(w/0/d
- サンプルを5分間沸騰させ、10%SDS-PAGEゲルで実行します。MBP融合タンパク質(目的のタンパク質の分子量+42.5 kDa MBP)の蓄積を視覚的に評価するには、クマシー染色バッファー(メタノール50%、酢酸10%、クマシーブルー0.1%)で攪拌し、一晩で脱色することにより、ゲルを20分間染色します。脱染み緩衝液(20%メタノール、10%酢酸)。
- 残りの大腸菌細胞を1,350xgで6分間遠心分離して採取し、上清を廃棄し、5mLのカラムバッファー(20mM Tris HCl、200 mM NaCl、1 mM EDTA、細菌プロテアーゼ阻害剤)で細胞ペレットを再懸濁させる。
注: これは、プロトコルを一晩停止するのに適した場所です。凍結細胞は-20°Cで1週間まで保存することができる。 - 氷水浴に細菌を含むチューブを配置し、10の超音波処理サイクルを適用することにより、大腸菌細胞を分解する:30%アンプで10 s超音波処理の後に20 sの休憩が続きます。
- 4°Cで13,000 x gの遠心分離試料を10分間保存し、上清(粗抽出物)を保存した。
- 15 mLチューブに500μLのアミロース樹脂を調製します。冷たいカラムバッファーを10mL加えて樹脂を洗浄し、1,800 x gで遠心分離し、4°Cで5分間洗浄します。これを2x行います。
- アミロース樹脂で5mLの粗抽出物をチューブに加え、4°Cで一晩インキュベートします。
- 4°Cで1,800×gで5分間遠心分離し、上清を廃棄する。
- 樹脂ペレットに10mLのカラムバッファーを加え、20分間インキュベートします。次に、4 分間 4 °C で 1,800 x gの遠心分離を行います。
- 10 mMマルトースを含むカラムバッファーを0.5mLで融合タンパク質に溶出し、4°Cで2時間のサンプルをインキュベートします。4°Cで1,800 x gで遠心分離し、溶出タンパク質を回収する。この手順 2x を繰り返します。
- 冷たいPBSに対するタンパク質画分の透析量1mL。アリコタンパク質を単独使用チューブ(10~20°L)に入れ、凍結解凍を回避し、必要になるまで-80°Cで保存します。
- ブラッドフォードアッセイ9を用いてタンパク質濃度を測定する。
- E1の40 ng(例えば、AtUBA1)、E2の100ng(例えば、AtUBC8、SlUBC8、SlUBC1/4/6/7/12/13/17/20/22/27/32)、1つの量を混合することにより、総体積30μLでインビトロユビキチン反応を設定し、1つのタイプ ユビキチン化バッファー内の 2 μg FLAG-Ub (または HA-Ub) (50 mM Tris-HCl pH 7.5, 2 mM ATP, 5 mM MgCl2, 30 mM クレアチンリン酸, 50 gg/mL クレアチンホスホキナーゼ).混合物を30°Cで2時間インキュベートする。
注:プレメイク20xユビキチンレーションバッファと1回の使用のための小さなアリコートで-20°で6ヶ月まで保存します。クレアチンホスホキナーゼは、緩衝液を解凍し、繰り返し凍結すると、酵素活性を容易に失う。 - 30 μL サンプルを 5x SDS-PAGE ローディング バッファー (60 mM Tris-HCl pH 6.8, 2% SDS, 25% (v/v) グリセロール、10 mM DTT、0.1% (w/v) ブロモフェノールブルー) と沸騰させて、5 分間沸騰させて反応を終了します。
- 7.5%SDS-ポリアクリルアミドゲル電気泳動(SDS-PAGE)でタンパク質を分離し、PDVF膜に転写し、抗FLAG(または抗HA)を用いてウェスタンブロッティングによるユビキチン化を検出します。
- PVDF膜をクマシーブルーで染色し、テスト済みのMBP-RING型タンパク質の均等な負荷を検証します。
3.アグロバクテリウム-ニコチアナ・ベンタミアナ葉およびプランタユビキチン化アッセイにおける媒介性一過性タンパク質発現
- 目的のエピトープタグ付き遺伝子を担持するスクト適切なアグロバクテリウム・トゥメファシエンス株(例えば、HA-RHA1B、HA-RHA1BC135S、HA-RHA1BK146R、HA-Ub)および空ベクターを適切な抗生物質を含むLB培地上の対照として
- 28°Cで成長の2日後、単一のコロニーをピックアップし、別の24時間のために28°C /250 rpmで適切な抗生物質でLB液体培地中でそれらを成長させます。
- 100μLのアグロバクテリア培養物を適切な抗生物質で3mLの新鮮なLBに移し、後期指数成長期に回転(250rpm)で28°Cでさらに4〜6時間培養をインキュベートします。
- 1,800 x gでアグロバクテリア細胞を6分間スピンダウンし、上清を廃棄し、3mLの洗浄緩衝液(50mM MES pH 5.6、28 mMグルコース、2 mM NaH2PO4)で細胞を再懸濁する。この手順 2x を繰り返します。
- 2回目の洗浄後、誘導緩衝液中の細胞を再サスペンド(50mM MES pH 5.6、28 mMグルコース、2mM NaH2PO4、200 μMアセトシリンギン、37 mM NH4Cl、5 mM MgSO4.7H2O、4 mM KCl、18 μM FeSO 4.7H2O、2mMCaCl2誘導緩衝液で細胞をインキュベートし、さらに10~12時間、28°Cでインキュベートします。
注:アセトシリンゾンはT-DNA転移を誘発する。 - 細胞を1,800 x gで6分間遠心分離し、上清を廃棄し、2mLの浸潤緩衝液(10mM MES pH 5.5、200μMアセトシンギントン)で細胞を再休止する。
注:誘導緩衝液でインキュベーションした後に凝集するアグロバクテリアの場合は、凝集した細胞をベンチに数分間放置してチューブの底部に沈み、クリアアグロバクテリウム懸濁液を新しいチューブに移してから、ステップ3.6に進みます。 - OD600値(600nmの吸光度での光学密度)を用いて細菌の濃度を測定する。OD600の値を目的の値に調整します。
注: 通常、0.2 ~ 0.4 の OD600値は、単一のアグロバクテリア染色発現に最適です。異なる農薬株の組み合わせが適用される場合、アグロバクテリア株の合計OD600値は1を超えてはなりません。 - アグロインフィルトは、4週齢のN.ベタミアナが針でそっと刺し、続いて針のない注射器でアグロバクテリウムを手で注射することによって葉をする。浸潤した葉の領域をマーカーで囲みます(通常直径は1~2cm)。
- 浸潤した葉組織36時間浸潤を収集する。液体窒素で微細粉末に組織を粉砕します。
- タンパク質抽出バッファーの300 μLで再懸濁された組織粉末(50 mM Tris-HCl pH 7.5、150 mM NaCl、5 mM EDTA、2 mM DTT、10%グリセロール、1%ポリビニルポリピロリドン、1mM PMSF、植物プロテアーゼ阻害剤カクテル)および遠心分離剤で15,000 x 1500°Cで15,000 x1500°Cで15,000 x 1500°cで
- 新しいチューブに上清を移します。5x SDS-PAGEローディングバッファを1xの最終濃度に加え、5分間沸騰させます。
- 10%SDS-PAGEゲル上の粗タンパク質を分離し、PVDF膜上に転写し、抗HAでプローブしてプランタユビキチン化で検出します。
4. 植物の酵素活性と機能との関連の確立
注:例えば、RHA1Bは、HR細胞死を抑制するために耐性タンパク質Gpa2の分解を促進する。このステップでは、RHA1B のこれらの悪質なアクティビティが E3 に依存していることを確認する方法を示します。
- ストリーク適切なアグロバクテリウム・トゥメファシエンス株は、タグ付けされた目的遺伝子を運ぶ株(この例ではHA-RHA1B、HA-RHA1B C135S、HA-RHA1BK146R、myc-Gpa2、RBP1)および対照として空のベクターである。N.ベタミアナの葉にアグロバクテリウムの調製と注射のためのステップ3.1-3.8に従ってください。
- E3依存性基質タンパク質分解については、ステップ3.9~3.12に従い、適切な抗体を用いてウェスタンブロッティングを行い、植物細胞内のタンパク質蓄積を検出します(例えば、抗HAおよび抗MYC)。
- E3依存性過敏性応答(HR)媒介細胞死抑制については、HR細胞死症状のアグロインフィルト葉を2~4日後にモニタリングします。
結果
このセクションでは、PROSITE予測RING-H2型ドメイン(132〜176アミノ酸)10を有する単一サブユニットE3ユビキチンリガーゼRHA1Bの検査に使用されるプロトコルに代表する結果が提供される。図1に示すように、E3欠損変異タンパク質を得るためには、RINGドメイン内の保存された8つのCsまたはHsのうちの少なくとも1つ(図1A)を変異化する必要がある(図1B)。したがって、第1段階として、RHA1Bの2つの変異型バージョン、RHA1BC135S(RINGドメインの保存されたC3におけるSerによるサイの置換)およびRHA1BK146R(RHA1Bに存在する唯一のLysにおけるArgによるリスの置換)が生成された。単一サブユニットE3リガセは、ユビキチンを収容するE2から基板へのユビキチン移動を仲介するが、LysにおけるE3の自己ユビキチン化は、その最大の酵素活性のために必要とされるかもしれない。
図2Aにおけるウェスタンブロッティング結果は、試験されたタンパク質の分子量(例えば、MPB融合RHA1B〜100kDa)から始まり、上方に進行するマルチバンドスミアを有する典型的なインビトロユビキチン化アッセイ結果を示す。抗HA抗体は、異なる長さのポリユビキチン化鎖に組み込まれたHAタグ付きUbを認識し、この典型的なユビキチン関連ラダー様スミアを作成した。正の結果を検証するために、図 2Aは、個々のコンポーネント (E1、E2、Ub、または MBP-RHA1B) を欠いているか、MBP を制御として使用し、塗りつぶされたユビキチン化信号を欠いているすべての重要な負のコントロールも示しています。さらに、PVDF膜のクマシーブルー染色は、すべてのコントロールにおいてMBP-RHA1BまたはMBPの等しい負荷を示した。
図2Bは、特定のE2/E3の組み合わせに応じてインビトロユビキチン化結果がどのように変化するかを示す。この例では、10個の異なるE2ファミリーを表す11の異なるE2がテストされました。検出されたユビキチン化活性は、シグナルなし(スミアなし)から異なる分子量から始まるマルチバンドスミアまで、異なるユビキチン化パターンを示す範囲を示した。
図3は、試験されたタンパク質のRING-およびK変異型バージョンのユビキチンアッセイ結果を示す。RHA1BC135Sの酵素活性の欠如は、インビトロ(図3A)でマルチバンドスミアを生成したり、プランタで多ユビキチン化シグナルを促進したりすることができないことで支持されている(図3B)。それ自体でプランタのHAタグ付きUbの過剰発現は、野生型RHA1Bの酵素活性によって与えられる強いユビキチン化シグナルとは対照的に、ベクター制御を含むすべての試験されたサンプルにおいて基礎レベルのユビキチン化を与えたことは注目に値する。さらに、RHA1BK146R変異体に関する分析は、K146残基がRHA1BのE3活性にも不可欠であることを示唆している。限界自己ユビキチン化シグナルはインビトロ(図3A)で検出されたが、プランタアッセイでは変異体がE3欠損であると判定した(図3B、バックグラウンドユビキチン化シグナルのみが検出された)。
E3欠損変異体を生成および生化学的に検証した後、機能的研究は、試験されたRING E3ユビキチンリガーゼのE3関連生物学的役割を決定するように設計することができる。RHA1Bの場合、この線虫エフェクターは、Gpa2トリガHR細胞死の抑制によって現れるように、植物の免疫シグナル伝達を抑制する。図4Aに示すように、野生型RHA1Bとは異なり、E3リガーゼ活性を欠くRHA1BC135S変異体はHR細胞死を妨げなかった。タンパク質ユビキチン化の最も一般的な結果は、そのプロテアソーム媒介性分解であることを考えると、RINGドメインに存在する突然変異は、直接的および/または間接的な基質の分解をトリガするE3依存性の能力を検証するためにも使用することができる。したがって、有意に、図4Bにおけるウェスタンブロッティング結果は、Gpa2が野生型RHA1Bの存在下で蓄積しなかったことを確認したが、RHA1BC135SはGpa2タンパク質安定性に影響を及ぼさなかった。
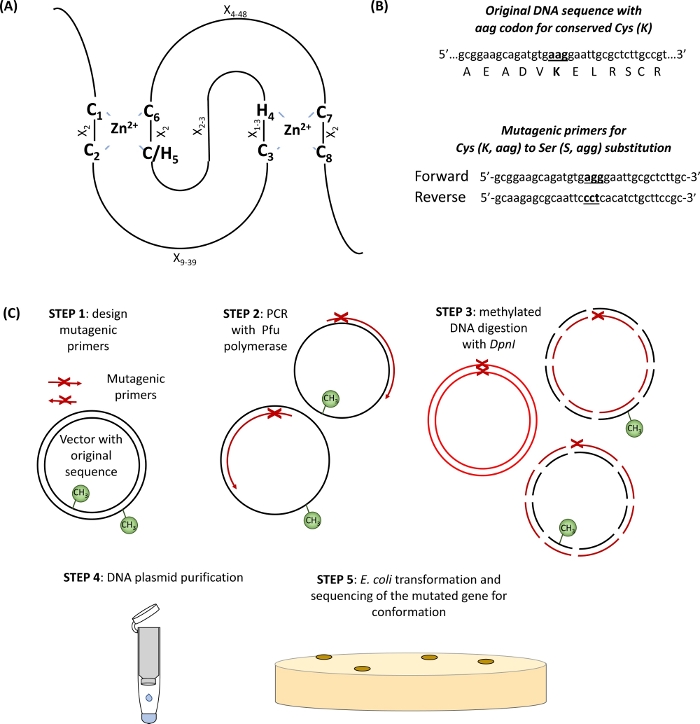
図1:部位特異的突然変異誘発に関与する原理とステップの概略表現(A)保存されたCysとHisアミノ酸が強調されたRING-CH/H2ドメイン。(B) 変異原性プライマー設計の一例。(C)部位特異的突然変異誘発のステップこの図の大きなバージョンを表示するには、ここをクリックしてください。
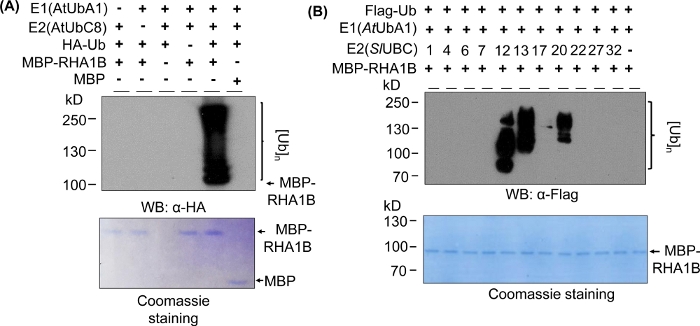
図2:インビトロユビキチンアッセイの代表者。(A)トップゲルは、すべての負のコントロールを含むユビキチンアッセイを示し、底ゲルは等しい負荷を示す。(B) E2酵素に応じた期待される結果の範囲。この図は Kud ら8から変更されています。この図の大きなバージョンを表示するには、ここをクリックしてください。
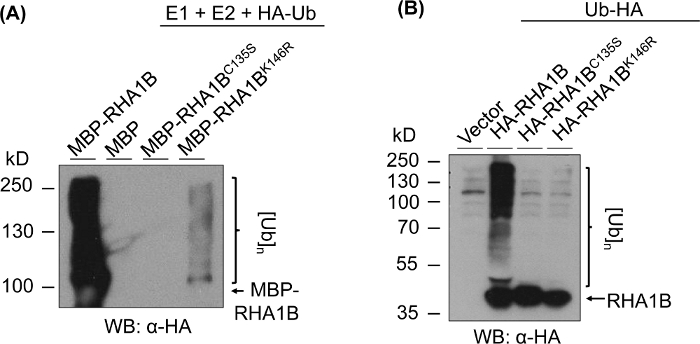
図3:RING-およびK-変異体(RHA1BC135SおよびRHA1BK146R)に対するユビキチン化アッセイ結果。(A) RHA1BC135SおよびRHA1BK146Rのインビトロユビキチン化結果(B)RHA1BC135SおよびRHA1BK146Rに対するプランタユビキチン化アッセイ結果この図は Kud ら8から変更されています。この図の大きなバージョンを表示するには、ここをクリックしてください。
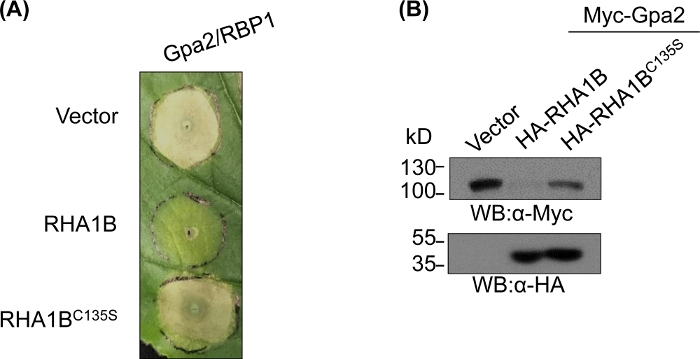
図4:E3依存性生物学的機能に関する代表的な機能的研究E3依存性生物学的機能を示す機能的研究の一例。(A)E3依存性HR細胞死抑制および(B)植物免疫受容体Gpa2の分解。この図は Kud ら8から変更されています。この図の大きなバージョンを表示するには、ここをクリックしてください。
| PCR セットアップ | |
| 1°L | プラスミド(〜100ng) |
| 1.5°L | F変異原プライマー(10μM) |
| 1.5°L | R変異原プライマー(10μM) |
| 1°L | dNTP (10 mM) |
| 5°L | バッファ (10x) |
| 1°L | ウルトラPfuポリメラーゼ (2.5 U/μl) |
| 39°L | ddH2O |
| 50°L | 総容積 |
表1:PCR反応の設定
| サーモサイカープログラム | |||
| 1 | 95 °C | 30 s | |
| 2 | 95 °C | 30 s | |
| 3 | 60 °C | 30 s | |
| 4 | 72 °C | 5 分 | 2-4 30回繰り返す |
| 5 | 72 °C | 5 分 | |
表 2: PCR サーモサイカー プログラム
| RHA1Bの例に対して設定されたライゲーション反応 | ||
| 1.5°L | pMAL-c2::バンヒとSalIによる消化によるMBP線形化ベクトル(60ng) | |
| 7°L | RHA1B/RHA1BC135S または RHA1BK146R 挿入物は、BamHI および SalI (25 ng) で消化されます。 | |
| 1°L | T4リガーゼバッファー(10倍) | |
| 0.5°L | T4リガーゼ(400 U/μL) | |
| 10°L | 総容積 | |
表3:RHA1B例に対してライゲーション反応を設定した。
ディスカッション
RING型E3ユビキチンリガーゼの生化学的・機械的基礎を解明することは、ホメオスタシスの発達、ストレスシグナル伝達、維持における生物学的意義の理解に大きく貢献することができます。ここで説明するプロトコルは、インビトロおよびプランタ機能研究と突然変異誘発アプローチを組み合わします。サイト直接突然変異誘発を介してRINGドメインの保存された残基に単一のアミノ酸置換を導入することにより、得られたE3欠損変異体を野生型タンパク質と並行して試験し、酵素活性を機能性と結び付けることができる。
RINGドメイン、特に保存されたCysとHis残基を適切に識別することが重要です。PROSITEなどのオンラインツールを使用すると、10を行うことができます。E2酵素の採用を担うRINGドメインを不安定化させるために、Cysは通常、亜鉛配位に使用されるジスルフィド結合を作成する能力を欠く最も近い構造置換であるSerで置換される。Lorick et al. は、これらの重要な Cys 残基のいずれかに対する突然変異が、単一サブユニット RING 型 E3 リガセス5のユビキチン活性を廃止することを示した。一部のCys残基は、RING型タンパク質を含むマルチユニットE3リガーゼ複合体においても重要であるが、それらのユビキチン化複合体の多面的かつ動的な三次元構造とRING型タンパク質の異なる役割のために、多単位E3リガーゼにおけるRINGドメインにおける保存残基の単一置換は、リガーゼ欠乏表現型11の生成に成功していない。
部位特異的突然変異誘発では、小さいプラスミドベクターと低い増幅サイクルを用いることは、通常、突然変異誘発に対してより高い効率をもたらすことを発見した。Pfu酵素は、他の高忠実度および高いプロセス性DNAポリメラーゼで置換することができる。さらに、目的の遺伝子が希少なコドンを含む場合、別の大腸菌染色、ロゼッタ、組換えタンパク質のより高い収率を達成するために使用することができる。さらに、IPTG誘導のためのインキュベーション時間および温度の両方をさらに最大限に活用することができる。温度が低いと大腸菌分裂率が低下し、特定のタンパク質の発現に有利になる可能性があります。IPTGの濃度が高いほどタンパク質発現が改善される可能性がありますが、大腸菌分裂プロセスも阻害し、お勧めできません。
単一サブユニットRING型E3リガーゼは、E2-Ub中間体を基質に近接して位置づける分子足場として機能するだけでなく、そのコグネイトE2sのユビキチン移動活性を刺激する。さらに、E2/E3の組み合わせが、改変基板の運命を決定するポリウビキチン鎖の長さと結合にとって重要であることを考えると、RING型E3sの考慮には、その酵素パートナーE2s12を含める必要がある。図3Bに示すように、テスト済みのE2はすべてRHA1Bリガーゼと互換性があるわけではありません。したがって、インビトロユビキチン化アッセイは、誤った陰性の結果を避けるために、異なるE2クラスを表す複数のE2酵素と並行して行われるべきである。
ここで提示するin vitro酵素アッセイは、試験されたRING型タンパク質の自己ユビキチン化能力を検出する。しかし、小さな改変により、このプロトコルは、基板のインビトロユビキチン化を検出するために容易に適応することができる。このことから、ステップ2.15からのインビトロユビキチン化混合物は、潜在的なE3リガーゼ基質(500ng)の組換えタンパク質を添加する必要があります。30°Cで2時間のインキュベーションの後、ユビキチン化タンパク質は、抗HA親和性マトリックスの15μL(HA-Ubを使用する場合、またはFLAG-Ubを使用する場合は抗FLAG親和性マトリックス)を使用して、4°Cで2時間攪拌することにより捕捉する必要があります。冷たいUb洗浄バッファー(20 mM Tris pH 7.5、100 mM NaCl、0.1 mM EDTA、0.05%Tween 20、1x PMSF)でビーズを4倍洗浄した後、バッファの40μLを除くすべてを破棄し、ステップ2.16に移動します。ユビキチン化シグナルは、エピトープタグ付きUbおよび基質に特異的な抗体によって検出され、それぞれ、基質タンパク質の分子量から出現し、基質/酵素特異性を確認する。
さらに、生体内のE3リガーゼ基質の同定は、通常、一過性酵素基質相互作用およびユビキチン化標的タンパク質の急速な分解に起因する複数の課題に関連している。E3リガーゼ欠損変異体を使用すると、まだ標的と相互作用するが、13をユビキチン化しなくなったが、プロテアソーム阻害剤MG132を添加する非常に有用な代替手段であり、26Sプロテアソーム機能を常に十分に妨害するとは限らない。
RING型E3リガーゼの共通の特徴は、ホモおよび/またはヘテロダイマーとして形成および機能する傾向がある。興味深いことに、RINGドメインの保存残基における置換は、通常、変異したRING型E3リガーゼが天然の野生型タンパク質13の酵素活性を遮断する優性陰性表現型と関連している。したがって、プランタにおけるRING変異体の過剰発現は、E3リガーゼ遺伝子をノックアウトするための代替アプローチであり得る。
開示事項
著者たちは何も開示する必要はない。
謝辞
私たちの仕事は、USDA国立食品農業研究所、USDA-NIFAファームビル、ノースウエストポテトビルの農業・食品研究イニシアチブ競争助成金(2017-67014-26197;2017-67014-26591)からの資金援助によって可能になりました。コンソーシアム、ISDAスペシャルティクロップ
資料
| Name | Company | Catalog Number | Comments |
| Acetic acid | Sigma-Aldrich | A6283 | |
| Acetosyringone | Sigma-Aldrich | D134406 | |
| Amylose resin | NEB | E8021S | |
| ATP | Sigma-Aldrich | A1852 | |
| Bacterial protease inhibitor | Sigma-Aldrich | P8465 | |
| Bromphenol Blue | VWR | 97061-690 | |
| CaCl2 | Sigma-Aldrich | C1016 | |
| Centrifuge | Beckman Coulter | model: Avanti J-25 | |
| Commassie Blue | VWR | 97061-738 | |
| Creatine phosphate | Sigma-Aldrich | P7936 | |
| Creatine phosphokinase | Sigma-Aldrich | C3755 | |
| DNA clean & concentrator Kit | ZYMO RESEARCH | D4029 | |
| DpnI | NEB | R0176S | |
| DTT | Sigma-Aldrich | D0632 | |
| E. coli BL21 | Thermo Fisher Scientific | C600003 | |
| E. coli DH5α competent cells | Thermo Fisher Scientific | 18265017 | |
| EDTA | Sigma-Aldrich | 324504 | |
| FeSO4 7H2O | Sigma-Aldrich | F7002 | |
| FLAG-Ub | BostonBiochem | U-120 | |
| Glucose | VWR | 188 | |
| Glycerol | Sigma-Aldrich | G5516 | |
| HA-Ub | BostonBiochem | U-110 | |
| Heat block | VWR | model: 10153-318 | |
| Incubator | VWR | model: 1525 Digital Incubator | |
| Incubator shaker | Thermo Fisher Scientific | model: MaxQ 4000 | |
| IPTG | Roche | 10724815001 | |
| KCl | Sigma-Aldrich | P9333 | |
| LB Broth | Sigma-Aldrich | L3022 | |
| Liquide nitrogen | university chemistore | ||
| Maltose | Sigma-Aldrich | 63418 | |
| MES | Sigma-Aldrich | M3671 | |
| Methanol | Sigma-Aldrich | 34860 | |
| MgCl2 | Sigma-Aldrich | 63138 | |
| MgSO4 7H2O | Sigma-Aldrich | 63138 | |
| Microcentrifuge | Eppendorf | model: 5424 | |
| Miniprep plasmid purification kit | ZYMO RESEARCH | D4015 | |
| monoclonal anti-FLAG antibody | Sigma-Aldrich | F3165 | |
| monoclonal anti-HA antibody | Sigma-Aldrich | H9658 | |
| monoclonal anti-MYC antibody | Sigma-Aldrich | WH0004609M2 | |
| Mortar | VWR | 89038-144 | |
| NaCl | Sigma-Aldrich | S7653 | |
| NaH2PO4 | Sigma-Aldrich | S8282 | |
| NanoDrop | Thermo Fisher Scientific | model: 2000 Spectrophotometer | |
| Needle | Thermo Fisher Scientific | 14-826-5C | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| PCR machine | Bio-Rad | model: C1000 | |
| Pestle | VWR | 89038-160 | |
| Pfu Ultra | Agilent Technologies | 600380 | |
| Plant protease inhibitor coctail | Sigma-Aldrich | P9599 | |
| pMAL-c2 | NEB | N8076S | |
| PMSF | Sigma-Aldrich | P7626 | |
| Polyvinylpolypyrrolidone | Sigma-Aldrich | P6755 | |
| SDS | Sigma-Aldrich | 1614363 | |
| Sonicator | Qsonica Sonicators | model: Q125 | |
| Syringe | Thermo Fisher Scientific | 22-253-260 | |
| Tris | Sigma-Aldrich | T1503 | |
| T4 ligase | NEB | M0202S |
参考文献
- Freemont, P. S., Hanson, I. M., Trowsdale, J. A novel gysteine-rich sequence motif. Cell. 64, 483-484 (1991).
- Borden, K. L. B. RING fingers and B-boxes: Zinc-binding protein-protein interaction domains. Biochemistry and Cell Biology. 76, 351-358 (1998).
- Barlow, P. N., Luisi, B., Milner, A., Elliott, M., Everett, R. Structure of the C3HC4 Domain by 1H-nuclear Magnetic Resonance Spectroscopy: A New Structural Class of Zinc-finger. Journal of Molecular Biology. 237, 201-211 (1994).
- Borden, K. L. B., et al. The solution structure of the RING finger domain from the acute promyelocytic leukaemia proto-oncoprotein PML. The EMBO Journal. 14, 1532-1541 (1995).
- Lorick, K. L., et al. RING fingers mediate ubiquitin-conjugating enzyme (E2)-dependent ubiquitination. Proceedings of the National Academy of Sciences. 96, 11364-11369 (1999).
- Jiménez-López, D., Muñóz-Belman, F., González-Prieto, J. M., Aguilar-Hernández, V., Guzmán, P. Repertoire of plant RING E3 ubiquitin ligases revisited: New groups counting gene families and single genes. PLoS ONE. 13, 1-28 (2018).
- Sacco, M. A., et al. The Cyst Nematode SPRYSEC Protein RBP-1 Elicits Gpa2- and RanGAP2-Dependent Plant Cell Death. PLoS Pathogens. 5, 1-14 (2009).
- Kud, J., et al. The potato cyst nematode effector RHA1B is a ubiquitin ligase and uses two distinct mechanisms to suppress plant immune signaling. PLoS Pathogens. 15, 1007720 (2019).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72, 248-254 (1976).
- Sigrist, C. J. A., et al. New and continuing developments at PROSITE. Nucleic Acids Research. 41, 344-347 (2013).
- Dove, K. K., Stieglitz, B., Duncan, E. D., Rittinger, K., Klevit, R. E. Molecular insights into RBR E3 ligase ubiquitin transfer mechanisms. EMBO Reports. 17, 1221-1235 (2016).
- Metzger, M. B., Pruneda, J. N., Klevit, R. E., Weissman, A. M. RING-type E3 ligases: Master manipulators of E2 ubiquitin-conjugating enzymes and ubiquitination. Biochimica et Biophysica Acta - Molecular Cell Research. 1843, 47-60 (2014).
- Xie, Q., et al. SINAT5 promotes ubiquitin-related degradation of NAC1 to attenuate auxin signals. Nature. 419, 167-170 (2002).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved