Method Article
Imágenes fluorescentes de calcio y posterior hibridación in situ para la caracterización de precursores neuronales en Xenopus laevis
En este artículo
Resumen
Presentamos un protocolo de dos partes que combina imágenes fluorescentes de calcio con hibridación in situ, permitiendo al experimentador correlacionar patrones de actividad de calcio con perfiles de expresión génica a nivel de una sola célula.
Resumen
La actividad espontánea del calcio intracelular se puede observar en una variedad de tipos de células y se propone desempeñar un papel crítico en una variedad de procesos fisiológicos. En particular, la regulación adecuada de los patrones de actividad del calcio durante la embriogénesis es necesaria para muchos aspectos del desarrollo neuronal de vertebrados, incluyendo el cierre adecuado del tubo neural, la sinaptogénesis y la especificación del fenotipo del neurotransmisor. Si bien la observación de que los patrones de actividad del calcio pueden diferir tanto en frecuencia como en amplitud sugiere un mecanismo convincente por el cual estos flujos podrían transmitir señales codificadas a los efectores aguas abajo y regular la expresión génica, enfoques a nivel de la población han carecido de la precisión necesaria para seguir explorando esta posibilidad. Además, estos enfoques limitan los estudios del papel de las interacciones células-células al excluir la capacidad de determinar el estado de la determinación neuronal en ausencia de contacto entre células. Por lo tanto, hemos establecido un flujo de trabajo experimental que combina imágenes de calcio de lapso de tiempo de explantes neuronales disociadas con un ensayo de hibridación in situ de fluorescencia, permitiendo la correlación inequívoca del patrón de actividad del calcio con el patrón molecular fenotipo en un nivel de una sola célula. Pudimos utilizar con éxito este enfoque para distinguir y caracterizar patrones específicos de actividad de calcio asociados con la diferenciación de células neuronales y células progenitoras neuronales, respectivamente; más allá de esto, sin embargo, el marco experimental descrito en este artículo podría adaptarse fácilmente para investigar correlaciones entre cualquier perfil de actividad de serie temporal y la expresión de un gen o genes de interés.
Introducción
El calcio citosólico libre es fundamental para una variedad de procesos biológicos, que van desde la proliferación celular y la migración a la apoptosis y la autofagia1,2,3. Dentro de estas vías, el calcio puede ejercer efectos aguas abajo sobre la expresión génica interactuando con dominios de unión al calcio para inducir cambios conformacionales que modulan la actividad proteica y las interacciones. Por ejemplo, un sensor de calcio neuronal conocido como el modulador antagonista del elemento regulador descendente (DREAM) se mantiene en una conformación intermedia desdoblada cuando está unido por calcio, evitando que interactúe con sus objetivos de proteína y ADN4. Sin embargo, más allá de servir como una molécula de señalización simple, la naturaleza dinámica de los transitorios de calcio intracelular permite que estos patrones de actividad codifican señales más complejas basadas en amplitud o frecuencia5,6. La translocación nuclear del factor de transcripción nuclear de las células T activadas (NFAT) se ve reforzada por oscilaciones de calcio de alta frecuencia, pero inhibida por oscilaciones de baja frecuencia7. Con tende de ello, un trabajo reciente ha sugerido que NFAT puede responder a la exposición acumulativa al calcio8. Tanto la calcineurinacomola proteína quinasa II dependiente de La calmodulina /calmodulina II (CaMKII) también presentan respuestas distintas a los transitorios de calcio de una frecuencia, duración o amplitud9específicas. Para añadir un nivel adicional de complejidad regulatoria, los modelos computacionales sugieren que muchas proteínas de unión al calcio aguas abajo se vuelven más o menos dependientes de la frecuencia en respuesta a la presencia o ausencia de competidores de unión10,11.
Dentro del sistema nervioso en desarrollo, dos clases principales de comportamientos de actividad del calcio se han definido y asociado con procesos biológicos específicos. Las afluencias de calcio se clasifican como "picos" si se producen dentro de las células individuales, alcanzan una intensidad máxima del 400% de la línea de base en cinco segundos y presentan una doble decaimiento exponencial12. Este tipo de señal se asocia principalmente con neurotransmisor fenotipo especificación13. Por el contrario, las "ondas" se definen como transitorios de calcio más lentos y menos extremos en los que la concentración de calcio intracelular de una célula se eleva a 200% de la línea de base durante un período de treinta segundos o más, y luego se descompone durante varios minutos12. Estas señales a menudo se propagan a través de múltiples células vecinas, y su presencia se ha asociado con el crecimiento de neurita y la proliferación celular14,15. Sin embargo, aunque estas dos clases se han definido en función de perfiles cinéticos característicos, sigue sin estar claro exactamente qué características de estos patrones están siendo detectadas por las células y traducidas por los efectores descendentes.
Comprender la relación entre las oscilaciones de calcio intracelular y la expresión génica proporcionaría una visión crucial de uno de los mecanismos reguladores que garantiza el desarrollo y el patrón adecuados del sistema nervioso. Con este fin, los estudios de la médula espinal embrionaria han demostrado que el aumento de la actividad del pico de calcio durante el desarrollo se asocia con niveles más altos de neuronas inhibitorias, mientras que la disminución de la actividad del pico de calcio se asocia con niveles más altos de neuronas excitatorias13. Sin embargo, estos ensayos a nivel de población no se han utilizado para asociar la actividad del calcio con la expresión génica a nivel de una sola célula.
Abordar estas preguntas en el nivel de la celda única ofrece varias ventajas distintas sobre el trabajo anterior. Por un parte, la capacidad de evaluar la actividad del calcio y la expresión génica en muchas células individualmente permite observar el repertorio completo de patrones de actividad distintos sin ser ofuscado por una medición a granel. Además, el estudio de estas relaciones en el cultivo primario de una sola célula significa que se mantendrán los vínculos autónomos celulares entre la actividad del calcio y la expresión génica, mientras que las interacciones que requieran comunicación de células celulares serán abrogadas. Por lo tanto, este enfoque permite que estos mecanismos autónomos celulares se estudien de forma aislada. Sin embargo, también permite que el papel de la actividad de calcio no autónoma de las células sea esclarecido e interrogado. Por ejemplo, las células se pueden diseccionar de un embrión en la etapa de la placa neural, cultivadas hasta que los controles del hermano alcanzan la etapa del tubo neural y luego se comparan con las células que se han diseccionado recientemente desde un embrión en etapa del tubo neural. Esto permite la comparación directa de las células que retuvieron la comunicación de células celulares a través de un período clave de desarrollo con aquellas en las que se abolió la comunicación de células celulares.
Al tratar de abordar las limitaciones de los enfoques experimentales anteriores, desarrollamos un protocolo que permitiría evaluar tanto la actividad del calcio como la expresión génica en células progenitoras neuronales individuales, facilitando la correlación de patrones de actividad específicos con programas de diferenciación posteriores. El tejido neural se diseccionó de Xenopus laevis en varias etapas del desarrollo neuronal, se disociaba en células individuales e se imageó mediante microscopía confocal en presencia de un indicador de calcio fluorescente. Después de las imágenes de células vivas, las muestras se fijaron y analizaron mediante hibridación in situ por fluorescencia (FISH) para detectar la expresión de un gen o isoforma de interés. Es importante destacar que las células individuales se pueden realizar un seguimiento de ambos experimentos de diagnóstico por imágenes, lo que significa que el perfil de actividad de calcio de una célula y su nivel de expresión génica se pueden asociar entre sí(Figura 1). El protocolo aquí indicado está destinado a sondear las relaciones entre los patrones de actividad del calcio y la expresión génica a través del neurodesarrollo embrionario en Xenopus laevis. Sin embargo, el marco experimental más amplio (imágenes de curso de tiempo de una sola célula seguidas de FISH y el registro de imágenes) se puede modificar y aplicar a prácticamente cualquier tipo de célula, reportero fluorescente y gen de interés.
Protocolo
Todo el trabajo relacionado con animales se realizó de acuerdo con los protocolos aprobados por el Comité Institucional de Cuidado y Uso de Animales (IACUC) en el Colegio de William y Mary.
1. Cuidado de animales y manejo de embriones
- Inducir el apareamiento natural mediante la administración de una inyección subcutánea de gonadotropina coriónica humana (HCG) en el saco linfático dorsal de Xenopus laevis adulto a una dosis de 600 U para las mujeres y 400 U para los hombres.
- Después de la inyección, coloque al menos un macho y una rana hembra en un tanque de retención a temperatura ambiente durante la noche. La puesta de óvulos normalmente comienza 9-12 h después de la administración de HCG.
- Recoger embriones. Dejelly lavándose suavemente con 2% de cisteína (pH 8.0) durante 2-4 min.
- Enjuague los embriones 3x en 0.1x Solución de Timbre Modificado (MMR) (100 mM NaCl, 2 mM KCl, 1 mM MgSO4, 2 mM CaCl2, 5 mM HEPES, pH ajustado a 7.4–7.6).
- Transfiera los embriones a platos Petri de vidrio de 100 mm que contengan 0,1x MMR y 50 g/mL de gentamicina. Una densidad de 50-100 embriones por placa es apropiada.
- Incubar los platos a 14 oC a 22 oC y permitir que los embriones se desarrollen hasta que lleguen a la(s) etapa(s) de desarrollo deseada(s). Retire periódicamente las células no fertilizadas y los embriones necróticos o en desarrollo anormal con una pipeta de transferencia plástica.
NOTA: Para garantizar la coherencia, la puesta en escena del desarrollo se realiza de acuerdo con criterios morfológicos definidos por Nieuwkoop y Faber16. Las etapas de interés variarán en función del enfoque experimental. Por ejemplo, los puntos de referencia clave del neurodesarrollo están asociados con la etapa 14 (inicio de la neurulación), la etapa 18 (inicio del cierre del tubo neural) y la etapa 22 (inicio del alargamiento del brote de cola).
2. Disección de embriones y preparación de muestras
- Preparación de la solución
- Prepare 2 mM Ca2+ solución que contenga 116 mM DeNaCl, 0,67 mM KCl, 2 mM De CaCl2x 2H2O, 1,31 mM MgSO4y Tris de 4,6 mM. Por cada solución de 100 ml, añadir 1 ml de penicilina/estreptomicina (10.000 U/ml de penicilina; 10.000 g/ml de estreptomicina). Ajuste el pH a 7,8 y filtre.
- Preparar la solución libre de calcio y magnesio (CMF) combinando 116 mM NaCl, 0,67 mM KCl, 4,6 mM Tris y 0,4 mM EDTA. Ajuste el pH a 7.8 y el autoclave para esterilizar. Por cada solución de 100 ml, añadir 1 ml de penicilina/estreptomicina (10.000 U/ml de penicilina; 10.000 g/ml de estreptomicina). Ajuste el pH a 7,8 y filtre.
- Preparación de placas para disección e imágenes
- Esterilizar a los rayos UV dos platos de plástico Petri de 35 mm y un plato de cultivo celular de 35 mm (ver Tabla de Materiales).
- Mientras trabaja en una campana de flujo laminar, prepare dos tubos cónicos de plástico de 50 ml que contengan 10 ml de solución de 2 mM Ca2+ cada uno.
- Permanezca trabajando en la campana de flujo laminar y agregue 2 ml de solución de 2 mM Ca2+ a una placa Petri de plástico de 35 mm, 2 ml de solución de 2 mM Ca2+ a una solución de cultivo celular de 35 mm y 2 ml de solución CMF a una placa de plástico Petri de 35 mm.
- Fuera de la campana de flujo laminar, llene dos platos de plástico Petri de 100 mm y una placa de plástico Petri de 35 mm con 0,1x MMR con gentamicina (50 g/ml). Llene una placa Petri de plástico de 60 mm con un 70% de etanol.
- Inmediatamente antes de la disección, añadir 0,01 g de colagenasa B a uno de los tubos de 50 ml que contienen la solución de Ca2+. Mezcle bien y transfiera la solución a un plato Petri de plástico fresco de 60 mm.
- Con la ayuda de un microscopio de disección, identifique los embriones de la etapa de desarrollo deseada. Utilice una pipeta de transferencia estéril para transferir al menos seis embriones apropiados a una de las placas de 100 mm que contengan 0,1 mMR + gentamicina preparada en el paso 2.2.4. Esto servirá como la placa de retención.
- Utilice una pipeta de transferencia estéril para transferir un embrión a la segunda placa de 100 mm que contenga 0,1x MMR + gentamicina. Esto servirá como placa de disección.
- Si diseccionan embriones lo suficientemente viejos como para moverse (aproximadamente etapa 23 o más), anestetizar cada embrión antes de la disección transcrigiéndolo a un plato que contenga un éster etílico de ácido 3,1% 3% diluido en 0,1x MMR con gentamicina (50 g/ml). Una vez que el embrión esté inmovilizado, transfieralo de nuevo al plato que contiene 0,1x MMR con gentamicina (50 g/ml) y continúe con la disección.
- Retire cuidadosamente la membrana vitelina que rodea el embrión. Esto se puede hacer más fácilmente mediante el uso de un par de fórceps contundentes para estabilizar el embrión mientras se utiliza un par de fórceps finos para agarrar la membrana. Tire con cuidado con los fórceps finos para despegar la membrana vitelina.
- Utilice fórceps finos para separar las regiones dorsal y ventrales del embrión. Esto se puede hacer usando los fórceps para 'pinchar' el embrión a lo largo del eje anterior-posterior, cortándolo por la mitad. Con una pipeta de transferencia estéril, transfiera la parte dorsal a la placa de 60 mm con solución de colagenasa preparada en el paso 2.2.5. Deseche la porción ventral.
- Deje que la explanta dorsal se incuba en la solución de colagenasa durante 1-2 minutos a temperatura ambiente. Transfiera suavemente de nuevo a la placa de disección.
- Complete la disección eliminando cuidadosamente toda la contaminación endodérmica y mesodeérmica residual del tejido neural presuntivo del ectodermo. En el caso de los embriones en la etapa 22 o anterior, también se debe extraer y desechar el tubo neural.
NOTA: Si es necesario durante la disección, el plato de etanol 70% preparado en el paso 2.2.4 se puede utilizar para limpiar o volver a esterilizar los fórceps. - Una vez completada la disección, transfiera suavemente la explanta ción a la placa de 35 mm de la solución Ca2+ preparada en el paso 2.2.3.
- Repita los pasos 2.4-2.9 hasta que se hayan recogido cuatro explantas.
- Utilice un micropipeta P1000 para transferir los cuatro explantes a la placa de 35 mm que contiene la solución CMF, teniendo cuidado de evitar cualquier contacto entre los explantes y la interfaz aire-agua. Gire suavemente el plato para que todos los explantes se agrupen en el centro de la placa.
- Incubar 1 h a temperatura ambiente para permitir que los explantes se disocien.
NOTA: Para ayudar en la disociación, se puede agregar 0.025%–0.01% de trippsina a la solución CMF. Esto puede ser necesario para la disociación eficiente de embriones mayores (etapa 22 y mayores). - En este punto, al menos dos embriones adecuadamente escenificados deben permanecer en la placa de retención. Transfiera estos embriones a un plato fresco lleno de 0,1x MMR con gentamicina preparada en el paso 2.2.4 y permita que se desarrollen sin perturbaciones con el plato cubierto para que coincida con el plato explanta. Estos embriones servirán como controles de hermanos.
- Utilice el superpegamento para fijar un cubreobjetos micro-reglado (ver Tabla de Materiales)a la parte inferior del plato de cultivo celular de 35 mm preparado en el paso 2.2.3.
NOTA: Coloque pequeños trozos de superpegamento alrededor de los bordes de la cubierta, luego presiónelo firmemente contra la parte inferior del plato de cultivo celular. Las marcas posicionales se oscurecerán en cualquier lugar en el que el pegamento entre en contacto con la rejilla, por lo que es importante mantener la parte central cuadriculada del cubreobjetos libre de adhesivo. - Después de que los explantes se hayan disociado durante 1 h, utilice una micropipeta P100 para transferirlos al plato de cultivo celular. Para placar tantas células como sea posible en la porción cuadriscada del plato, sostenga la pipeta en un ángulo poco profundo cerca de la superficie del plato, coloque la punta de la pipeta en la esquina de la rejilla mirando hacia adentro, y expulse firmemente la suspensión celular a través de la rejilla ar idealmente, las células se asentarán en un racimo denso apretado.
- Incubar durante 1 h a temperatura ambiente para permitir que las células se adhieran a la placa. Determinar y registrar la etapa de desarrollo de los embriones de control de hermanos cuando comienza esta incubación.
- Combinar 5 ml 1 mM de Fluo-4 AM (ver Tabla de Materiales)con 2 l de ácido Pluronic F-127.
NOTA: Fluo-4 AM es sensible a la luz y debe mantenerse en un tubo a prueba de luz o cubierto de papel de aluminio en todo momento. - Una vez completada la incubación, mueva el plato de muestra a un cuarto oscuro u otro lugar protegido contra la luz. Utilice un micropipeta para extraer 100 ml de solución del borde del plato. Agregue esta solución a la alícuota del ácido Fluo-4 AM/Pluronic F-127, la pipeta hacia arriba y hacia abajo para mezclar, y devuelva el volumen completo al plato de muestra. Gire suavemente para mezclar.
- Cubra la placa con papel de aluminio y deje incubar durante 1 h a temperatura ambiente. Determinar y registrar la etapa de desarrollo de los embriones de control de hermanos cuando comienza esta incubación.
- Al final de la incubación, utilizar el tubo cónico restante de 2 mM Ca2+ para realizar tres lavados de medios de la siguiente manera: 1) quitar 1 ml de solución del plato, añadir 3 ml de solución fresca, 2) quitar 3 ml de solución del plato, añadir 3 ml de solución fresca, 3) quitar 3 ml de solución del plato, añadir 3 ml de solución fresca.
3. Imágenes de calcio
NOTA: Las imágenes de calcio se realizaron utilizando un microscopio confocal invertido(Tabla de materiales).
- Coloque la placa de muestra en la etapa del microscopio, teniendo cuidado de protegerla de la exposición a la luz ambiental. Una vez que la placa esté asegurada, utilice un marcador para etiquetar el punto frontal de la placa de modo que se pueda encontrar el mismo campo de visión en las imágenes posteriores.
- Localice la muestra bajo el microscopio , primero a 10 veces y luego a un aumento 20veces, y seleccione un campo de visión adecuado para la toma de imágenes. Un campo de visión ideal es denso de células, pero no tan denso que las celdas se agrupan o son difíciles de distinguir individualmente.
- Ajuste el enfoque del microscopio para que la cubierta reglada por la rejilla sea visible. Los números marcados en el coverslip sirven como identificadores únicos para un locus de rejilla particular y se pueden utilizar para localizar el mismo campo de visión para imágenes adicionales. Si el campo de visión seleccionado originalmente no se superpone con ningún número, reajuste hasta que un número identificable esté en el marco.
- Tome una imagen de campo brillante del campo de visión seleccionado con el cubreobjetos reglado por la cuadrícula en el foco.
- Ajuste la configuración de enfoque y tome una imagen de campo brillante del campo de visión seleccionado con las celdas enfocadas.
- Con la capa celular enfocada, ilumine las muestras con un láser de 488 nm. Los valores HV y Offset se pueden optimizar para cada experimento para garantizar que se detecte un rango dinámico de fluorescencia en el canal FITC.
- Para una imagen de dos horas, modifique la configuración de imágenes para grabar 901 fotogramas con un tiempo de escaneo de 3,93 s y un intervalo de 8 s. Ejecute la configuración para adquirir la imagen.
- Una vez completada la toma de imágenes, retire la placa de la etapa del microscopio. Retire 1 ml de solución de la placa y reemplácela con 1 ml de 2meFA (200 mM MOPS, 2 mM EGTA y 2 mM MgSO4 en 7,4% de formaldehído).
- Incubar la placa durante 2 h a temperatura ambiente o durante la noche a 4oC. Determinar y registrar la etapa de desarrollo de los embriones de control de hermanos cuando comienza esta incubación.
- Una vez completada la fijación, retire toda la solución de la placa y reemplácela por 2 ml de 1x PBS. Almacene las placas a 4 oC para su posterior procesamiento.
4. Análisis de expresión génica: Síntesis de sonda
- Como se describe a continuación, genere una sonda de ARN antisentido para la hibridación in situ. Además, genere una sonda de detección para el mismo gen para su uso como un control negativo.
- Para purificar el ADN plásmido que contiene la secuencia de la plantilla de sonda, inocular 150 ml de caldo LB con reservas de glicerol bacteriano que contengan el plásmido de la plantilla. Incubar a 37oC con agitación durante la noche o hasta que el cultivo se turbia.
- Purifica el ADN plásmido del cultivo bacteriano usando tu método de elección.
NOTA: Utilizamos el kit de preparación midi McNary-Nagel para obtener altos rendimientos de ADN plásmido. - Para confirmar que el plásmido contiene la plaquita esperada, realice un resumen de restricción y analice los productos en un gel de agarosa. El plásmido sin cortar también se puede analizar en un gel de agarosa para comprobar la contaminación genómica del ADN.
- Para linealizar el ADN de la plantilla, configure una reacción de resumen de restricción de 100 l que contenga 20 g de ADN de plásmido, 2 ml de enzima de restricción adecuada y 1 pampón apropiado. Incubar a 37oC durante al menos 2 h.
- Extraer el ADN linealizado mediante la realización de una extracción de fenol/cloroformo seguida de una extracción de cloroformo.
- Precipita el ADN con 100% de etanol. Esto se puede hacer rápidamente añadiendo dos volúmenes de etanol frío a la muestra e incubando a -80 oC hasta que se solidifique (15-30 min).
- Utilice una centrífuga refrigerada para peletizar el ADN girando durante 20 minutos a 12.000 x g/4 oC.
- Retire el sobrenadante y lave el pellet con 200 ml de etanol al 70%. Girar durante 5 min a 12.000 x g/4 oC.
- Retirar el sobrenadante y el pellet seco al aire durante aproximadamente 5 min. Resuspender en 20 oL de 1x TE y conservar a 4 oC hasta su uso posterior.
- Para sintetizar y purificar la sonda de ARN antisentido, cree una mezcla rNTP de 2,5 mM combinando 15 ml de rCTP de 10 mM, 15 ml de rGTP de 10 mM, 15 ml de rATP de 10 mM, 9,75 ml de rUTP de 10 mM y 5,25 ml de 10 mM de excavación-11 UTP (ver tabla de materiales).
- Configurar una reacción de transcripción in vitro de 50 ml que contenga 4 g de ADN de plantilla linealizada a partir del paso 4.2-4.10, 15 l de mezcla de rNTP de 2,5 ml a partir del paso 4.11, 10 l de tampón de transcripción de 5x, 5 ml de TDT de 0,1 M, 0,5 ml de inhibidor de la ANR (20 U/L) y 1,5 ml de polimerasa de ARN adecuada (T3, T7 o SP6). Incubar 1 h a 37oC.
- Añadir 1,5 ml de ARN polimerasa adicional a la reacción y volver a 37 oC durante una hora adicional.
- Añadir 1 l de RQ1 DNAse a la reacción e incubar a 37 oC durante 10 minutos para degradar la plantilla de ADN.
- Añadir 30 l de solución de LiCl de 7,5 M a la muestra. Pipetear para mezclar e incubar a -20oC durante al menos 1 h.
- Con una centrífuga refrigerada, gire la muestra 25 min a 14.000 x g/4 oC.
- Retire el sobrenadante y enjuague el pellet con 500 ml de etanol al 70%. Girar durante 5 min a 14.000 x g/4 oC.
- Retire el sobrenadante y el pellet seco al aire durante aproximadamente 5 min. Resuspenden en 20 ml de agua libre de nucleasas.
- Crear en 10x stock de sonda diluyendo la muestra a una concentración de 10 ng/-L en zona de influencia de hibridación (50% formamida, 5x SSC (citrato salino-sódico; 750 mM NaCl, 75 mM de citrato de sodio, pH 7,0), ARN torula de 1 mg/ml, 0,1 % de Tween-20, 1 x solución de Denhardt, 0,1% CHAPS, 10 mM de EDTA y 100 g/ml de heparina). Conservar a -20oC hasta su uso posterior.
5. Análisis de la expresión génica: Hibridación de Fluorescencia In Situ
NOTA: Todos los lavados deben realizarse con aproximadamente 1 ml de solución utilizando una pipeta de transferencia estéril y envuelta individualmente. La pipeta debe colocarse en el borde de la placa al extraer o añadir la solución, y los lavados deben realizarse lo más suavemente posible para garantizar que las células no se desalojen de la superficie de la placa y se pierdan.
- Retire 1x PBS de la placa (del paso 3.10). Reemplazar con 1x PBS fresco e incubar 5 min a temperatura ambiente.
- Combinar 25 ml de trietanolamina de 0,1 M (pH 8,0) con 62,5 ml de anhídrido acético. Mezcla bien. Lave el plato con esta solución durante 10 min.
- Lavar la placa con 1 sSC durante 5 min.
- Lave la placa con 0.02 M HCl durante 10 min para permear las células.
- Lavar 2x con 1x PBS durante 5 min cada uno.
- Retire la solución y agregue 1 ml de búfer de hibridación (50% de formamida, 5x SSC (750 mM NaCl, 75 mM de citrato de sodio, pH 7.0), 1 mg/ml de ARN torula, 0,1% Tween-20, 1x Solución de Denhardt, 0,1% CHAPS, EDTA de 10 mM y 100 g/ml de heparina) a placa. Incubar con agitación durante al menos 6 h a 60oC.
- Retire el búfer de hibridación y sustitúyalo por una solución de sonda de ARN de 750 ml (diluida forma ndo el material de 10x fabricado en el paso 4.19. Las sondas de ARN sense se pueden utilizar como un control negativo.
- Incubar con agitación durante 8-14 h a 60oC.
- Retire la sonda y guárdela a -20 oC.
NOTA: La dilución de 1x sonda se puede reutilizar hasta tres veces antes de ser descartada. - Enjuague la placa con 0,2x SSC.
- Lavar con SSC fresco 0,2x durante 1 h a 60oC.
- Mueva las placas a temperatura ambiente y equilibre durante 5 min.
- Lave la placa con 0,2x SSC durante 5 min.
- Lave el plato con 1PBT durante 15 min.
- Lavar la placa con 2% H2O2 en 1x PBT (0,1% Triton-x-100) durante 1 h.
NOTA: Esta solución es sensible a la luz, por lo que debe ser fresca para cada experimento y protegida de la luz. Las placas también deben estar protegidas de la luz o láminas durante esta incubación. - Lavar la placa con 1 x TBST (150 mM NaCl, 50 mM Tris-HCl, pH 7,5, 0,1%Tween-20) durante 15 min.
- Diluir el reactivo de bloqueo al 2% en el tampón de ácido maleico (100 mM de ácido maleico, 150 mM de NaCl, pH 7,5). Bloquear las células de esta mezcla durante al menos 1 h a temperatura ambiente.
- Reemplace la solución de bloqueo con anticuerpos anti-digoxigeno-POD diluidos 1:1.000 en 2% Reactivo de bloqueo en el tampón de ácido maléico. Incubar durante la noche a 4oC.
- Enjuague la placa 3x con 1 x TBST.
- Lavar 4 veces con 2 ml de 1 x TBST durante al menos 15 minutos por lavado con balanceo continuo.
- Lavar 2x con 1pbT durante al menos 10 minutos por lavado con balanceo continuo.
- Diluir la tiramida conjugada con Cy3 1:25 en 1x PBT. Lavar la placa con 750 ml de esta dilución durante 5 min.
NOTA: Esta solución es extremadamente sensible a la luz, y las placas deben mantenerse frustradas o protegidas de la luz durante el resto del experimento para evitar el deterioro de la señal. - Añadir 2,5 sL de 0,3% H2O2 a esta solución e incubar con balanceo continuo durante 40 minutos adicionales a temperatura ambiente.
- Lavar 4 veces con 1 tbST durante al menos 15 minutos por lavado con balanceo continuo.
- Enjuague con 1pbT.
- Fijar las células incubando durante 1 h a temperatura ambiente en 1x MEMFA (100 mM MOPS, 1 mM EGTA y 1 mM MgSO4 en 3.7% formaldehído).
- Retire la solución y sustitúyala por 1pbS. Conservar las placas en un recipiente con papel de aluminio a 4oC hasta su posterior posterior procesamiento.
6. Células de imagen
NOTA: Las imágenes se realizaron con un microscopio confocal invertido.
- Coloque la placa de muestra en la etapa del microscopio, alineando la marca realizada en el paso 3.1 al frente del escenario.
- Enfoque la imagen de modo que la cubierta revestida de cuadrícula sea visible y, utilizando la imagen de cuadrícula tomada en el paso 3.4 como referencia, ajuste el campo de visión para que coincida con el campo de visión capturado en la imagen de calcio (Sección 3).
- Adquiera imágenes de campo brillante tanto de la cuadrícula como de las celdas.
- Ilumina las muestras con un láser TRITC de 595 nm. Ajuste los valores de ganancia para distinguir adecuadamente la señal del fondo utilizando las imágenes de las celdas de control negativas y adquirir una imagen fija.
NOTA: Idealmente, los niveles de fluorescencia de fondo se determinan sobre la base de una placa de control negativa procesada en paralelo con una sonda de ARN de detección no objetivo. Los ajustes de ganancia se ajustan de modo que esta placa aparece completamente negra (correspondiente a los niveles de fondo), y luego se mantienen constantes para otras placas que se crean imágenes de ese lote experimental.
7. Procesamiento de datos
NOTA: El procesamiento de datos se realizó utilizando el software Nikon Elements.
- Abra la imagen de calcio de 2 h del paso 3.7. Identifique los píxeles correspondientes a cada celda individual seleccionando Binario > Detección de puntos > Puntos brillantes. Asegúrese de que el canal FITC esté seleccionado.
- Los círculos de color aparecerán sobre celdas individuales después de que se haya generado esta capa. Ajuste los parámetros de distribución y tamaño de celda para que se reconozcan y asocien tantas celdas como sea posible a un identificador único.
- Realice un seguimiento de las celdas en todos los fotogramas de la imagen navegando a Ver > Controles de análisis > Opciones de seguimiento. Establezca 5 fotogramas como el espacio máximo entre pistas, elimine objetos asociados con menos de 600 fotogramas y seleccione la opción Cerrar huecos. Seleccione Realizar un seguimiento de los archivos binarios para aplicar el seguimiento de celdas a la imagen.
- Después de realizar un seguimiento de las celdas, elimine manualmente cualquier objeto que no corresponda a una celda individual (por ejemplo, un grupo de celdas). Sin embargo, los puntos de datos no deben excluirse de un análisis adicional basado en la morfología del seguimiento de la actividad cálcica.
- En el panel Imagen, seleccione Ver superposición > Mostrar ID de objeto binario. Desplácese hasta el final de la imagen (Marco 901) y seleccione Edición > Crear instantánea de vista (RGB de 8 bits)> Fotograma actual > Aceptar. Esto creará una instantánea del último fotograma de la imagen con el IDENTIFICADOR binario asociado de cada celda visible. Guarde esta imagen.
- Exportar datos de series temporales seleccionando todos los objetos seguidos de Exportar datos a Excel. Guarde la salida como un archivo CSV.
- Abra la imagen FISH. Optimice la detección de puntos como en los pasos 7.1–7.2, utilizando el canal TRITC en lugar del canal FITC. Elimine manualmente los binarios asignados incorrectamente y, a continuación, seleccione Resultados de medición automatizados > Actualizar medición para calcular la intensidad de la señal de cada celda. Exporte una instantánea de imagen y una tabla de datos repitiendo los pasos 7.5 y 7.6.
- Cree una hoja de cálculo donde la columna A se etiqueta COMO ID binario FISH y la columna B se etiqueta como ID binario de calcio. Abra las imágenes exportadas en los pasos 7.5 y 7.7. Para cada objeto identificado en la imagen FISH (paso 7.7), registre el ID binario en la columna A. A continuación, localice la celda correspondiente en la imagen de calcio (paso 7.5) y registre ese ID binario en la columna B. Las celdas que no se pueden identificar con confianza en ambas imágenes no deben agregarse a la hoja de cálculo.
NOTA: Puede ser útil utilizar un programa de edición de fotos como Adobe Photoshop o el editor de imágenes GIMP para abrir ambas imágenes, hacer una semitransparente y superponerla en su imagen de socio para identificar y vincular más fácilmente los dos ID binarios asociados a cada celda. - Para cada célula identificada, cotejar (ya sea manualmente o con un script) sus datos de calcio de serie temporal (asociados con el ID binario de calcio y exportados en el paso 7.6) y sus datos de expresión génica (asociados con el ID binario FISH y exportados en el paso 7.7).
NOTA: El procesamiento y análisis de datos posteriores puede implicar la recopilación de estos datos en una sola tabla de datos y la aplicación de una serie de técnicas analíticas, incluyendo el recuento de picos, el análisis fractal y la entropía de Markovian que permiten al investigador discernir patrones novedosos de actividad de calcio 17,18.
Resultados
En la Figura 2Ase puede ver un ejemplo exitoso de células disociadas preparadas para la toma de imágenes de calcio. Las celdas están densamente chapadas, lo que permite recopilar la máxima cantidad de información de cada imagen, pero no tan densamente chapadas que las celdas individuales no se pueden distinguir con confianza entre sí. La fluorescencia se detecta para cada célula definida durante el período de imágenes de 2 h. La visualización de una gráfica compuesta que contiene las trazas de todas las celdas registradas en un experimento revela el grado en que las mediciones a granel o de población pueden oscurecer patrones más matizados de comportamiento de pico(Figura 2B). Cuando se aíslan los perfiles registrados de células individuales, se pueden identificar claramente ejemplos de la actividad irregular de picoteo característica de las células progenitoras neuronales. A diferencia de las neuronas maduras, las células neuronales embrionarias presentan naturaleza irregular, altamente variable y compleja de la actividad del calcio(Figura 2B). Para cuantificar esta complejidad, se ha aplicado la aplicación de diferentes métodos de análisis de datos17,18, incluyendo diversos parámetros para definir un pico(Figura 2C).
La hibridación in situ por fluorescencia exitosa, incluido el diseño y la síntesis exitosos de una sonda de ARNm antisentido, se puede evaluar comparando la placa experimental con un control de fondo incubado con un control de ARN de detección no vinculante(Figura 3A,B). Un control de sonda positivo también se puede realizar procesando un tipo de celda conocido por expresar el ARNm de destino en niveles detectables.
La identificación de la misma célula a través de imágenes de calcio y FISH requiere que las células conserven aproximadamente la misma posición durante la hibridación y el procesamiento de la sonda. Si las placas se manipulan de forma aproximada o los lavados se realizan con demasiada fuerza, las células se pueden desalojar de la superficie de la placa y se pueden perder cuando la solución se desecha o se deposita en una ubicación diferente en la placa, lo que hace imposible que se hagan coincidir a través de las imágenes(Figura 4A). Si esta interrupción afecta solamente algunas de las celdas en el campo de visión, todavía puede ser posible detectar y asignar algunas celdas dentro de la imagen(Figura 4B). Sin embargo, la cantidad máxima de datos se obtiene de un experimento en el que FISH se realiza cuidadosamente y pocas celdas se pierden o se reposicionan entre imágenes(Figura 4C).
Una vez que se han recopilado datos para describir tanto la actividad del calcio como la expresión génica de un número razonable de células en la etapa de desarrollo de interés, se pueden realizar análisis adicionales para evaluar las correlaciones entre estas dos características(Figura 1). Se han aplicado una serie de métricas para cuantificar los patrones de actividad del calcio, incluyendo el recuento de picos/frecuencia, la potencia media, la estimación del exponente de Hurst y la medición de la entropía de Markovian17,18. La expresión génica puede definirse cuantitativamente por nivel absoluto de fluorescencia o calificarse en una escala binaria (sí/no), dependiendo de las preguntas experimentales que se aborden.
Los resultados de experimentos que cotejaron la actividad del calcio con la expresión de genes marcadores progenitores neuronales revelaron numerosas asociaciones entre patrones específicos de actividad del calcio y fenotipos de neurotransmisores. En la etapa de la placa neural (etapa 14), las células GABAérgicas que expresan el marcador de neurona inhibitoria gad1.1 exhiben actividad de calcio que es más regular y de mayor amplitud que la de las células que carecen de expresión gad1.1 (Figura 5A). Además, mientras que estas células gad1.1-expressing se asocian con niveles más altos de pico de gran amplitud, el pico de baja amplitud es más frecuente en las células glutamatérgicas que expresan el marcador de neurona excitatoria slc17a7.
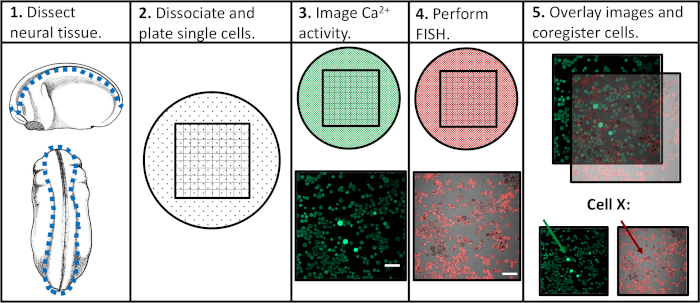
Figura 1: Esquema del flujo de trabajo experimental. Barra de escala a 100 m. Las imágenes en los paneles 3-5 fueron tomadas de Paudel et al. (2019)17. Haga clic aquí para ver una versión más grande de esta figura.
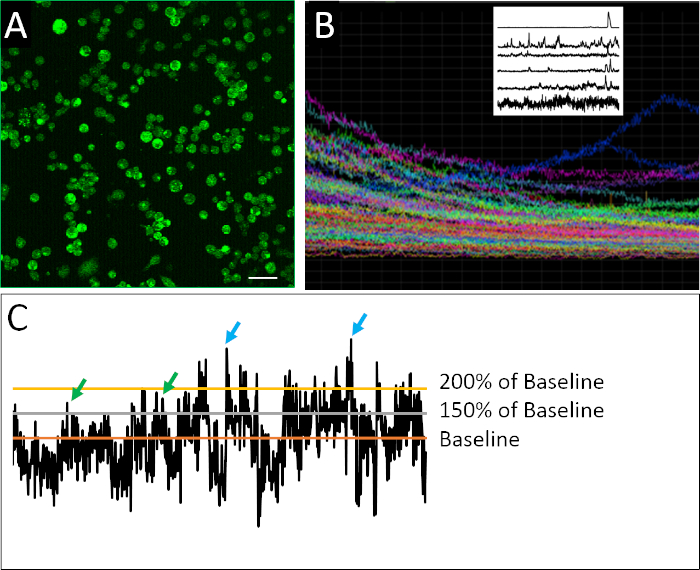
Figura 2: Imágenes de calcio y perfiles de actividad de ejemplo. (A) Actividad de calcio intracelular según lo informado por Fluo4-AM. Cada imagen de 2 h se compone de 901 marcos, con un marco representativo mostrar aquí. (B) Gráfica compuesta de intensidad de fluorescencia a lo largo del tiempo en todas las células dentro del campo de visión de imagen. Los rastros indican claramente fotoblanqueo de las horas extras del tinte indicador (Fluo4). La gráfica ráster en la parte superior izquierda muestra rastros representativos de la actividad del calcio después de la aplicación de un algoritmo de destendencia desarrollado por Eilers y Boelen19,donde las células que se muestran aquí exhiben diverso patrón de comportamiento de pico. (C) Aplicación de diferentes umbrales (150% y 200% de la línea de base, donde la línea de base es el promedio de intensidad fluorescente destendencia) para definir un pico (flechas verdes y azules). Barra de escala a 100 m. Haga clic aquí para ver una versión más grande de esta figura.
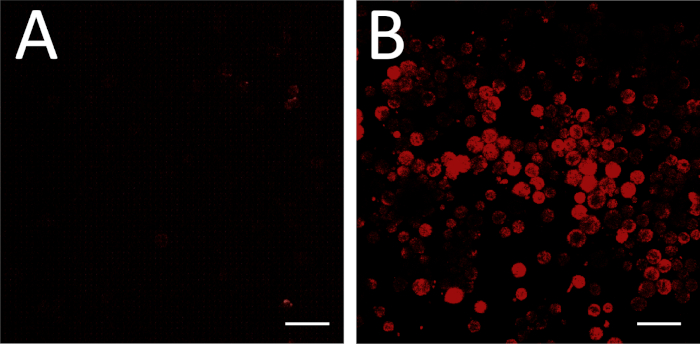
Figura 3: Imagen FISH. (A) FISH realizado con una sonda de ARN de detección no vinculante como un control negativo. Los ajustes de imagen se han ajustado para que ninguna célula parezca fluorescente. Algunos campos de visión pueden incluir desechos no celulares con cierta fluorescencia, como se ve en las esquinas superior derecha e inferior derecha de (A); estos pueden ser ignorados con el propósito de la configuración de fondo. (B) Los mismos ajustes de imagen se utilizan para crear una imagen de una placa experimental (sonda de ARN antisentido). La fluorescencia en estas condiciones corresponde a la expresión génica por encima del fondo. Barra de escala a 100 m. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Superposición de imágenes y registro de registro. Representaciones esquemáticas de una muestra de actividad de calcio (células representadas por círculos verdes rellenos) y después de FISH (células representadas por círculos rojos sombreados). (A) Las celdas que se han movido significativamente durante el manejo y procesamiento de muestras no se pueden identificar de forma fiable en las dos imágenes. (B) La interrupción de la celda puede afectar solo a algunas celdas en el campo de visión. Algunas células se pueden identificar claramente en ambas imágenes, mientras que otras no se pueden emparejar con confianza. (C) Si las muestras se manejan cuidadosamente, la mayoría de las células permanecerán intactas y se pueden identificar en ambas imágenes. Haga clic aquí para ver una versión más grande de esta figura.
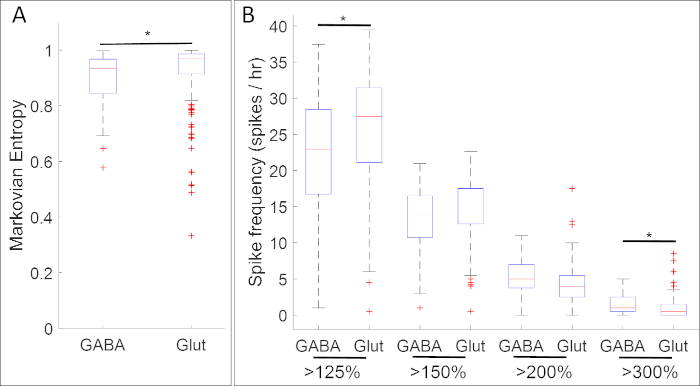
Figura 5: Un ejemplo de aplicación de este método, diagramas de caja que muestran asociaciones entre la actividad del calcio y la expresión génica (GABA y Glut para genes gad1.1 y slc17a7 respectivamente) en la etapa de placa neural Xenopus laevis. En la etapa 14, las células postivas (GABA) gad1.1 presentan una actividad de calcio más alta y más regular según lo definido por (A) Entropía de Markovian18 y (B) recuentos de picos utilizando umbrales 125%, 150%, 200% y 300% del promedio de la intensidad fluorescente destendencia (línea de base)17 que las células positivas slc17a7 (Glut). Las estrellas indican diferencias estadísticamente significativas de acuerdo con la prueba Kolmogorov-Smirnov de dos muestras corregida por Bonferroni (p < 0,05) y las estadísticas d de Cohen para el tamaño del efecto (n a 5 cultivos y >100 células; * 0,2 a á d a < 0,5). La cifra fue redibujada y adaptada del conjunto de datos obtenido de Paudel et al.17. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Se han observado patrones característicos de actividad cálcica en las células que componen el sistema nervioso en desarrollo, con tipos específicos de actividad asociados con procesos de neurodesarrollo distintos. Sin embargo, una mayor comprensión de los mecanismos por los cuales estos patrones de actividad de gran densidad de información se traducen en respuestas transcripcionales requiere que la información sobre la actividad del calcio y la expresión génica se recopile con resolución de una sola célula. Mientras que los sistemas que exhiben más actividad estereotipada del calcio, como las neuronas maduras, se pueden ensayo razonablemente a granel, los patrones irregulares que caracterizan el sistema nervioso embrionario se enmascaran fácilmente con grabaciones menos precisas.
El marco experimental establecido en este protocolo es fácilmente adaptable a una amplia variedad de tipos de células y reporteros fluorescentes. El tejido que contiene prácticamente cualquier tipo de célula o combinación de tipos de células se puede diseccionar de un organismo modelo de interés y chapado para imágenes de una sola célula. Además de permitir la identificación celular y aislar el efecto de los procesos autónomos de células, un enfoque de cultivo celular primario permite al experimentador definir los componentes de medios como desee. Por ejemplo, se han realizado experimentos que comparan la actividad de precursores neuronales en la solución de 2 mM Ca2+ para investigar si las relaciones entre la frecuencia de los picos y el fenotipo de neurotransmisores en la médula espinal embrionaria pueden recapitularse sin la influencia de las interacciones célula-célula13,20.
Si bien este protocolo aprovecha el marcador fluorescente Fluo4-AM para detectar la actividad de calcio intracelular, dependiendo de los criterios de selección, los usuarios pueden seleccionar otros marcadores disponibles comercialmente21,incluyendo indicadores de calcio codificados genéticamente. Del mismo modo, se podrían utilizar marcadores alternativos para monitorear los cambios dinámicos en la concentración de un ion de interés (incluyendo K+, Na+, y Zn2+),potencial de membrana o pH celular. La configuración de imágenes y la duración de la imagen se pueden modificar según sea necesario.
Aunque correlacionamos la actividad del calcio y el fenotipo neuronal como una aplicación específica, este método también es aplicable para una variedad de otras propiedades celulares. Por ejemplo, la hibridación in situ por fluorescencia se puede realizar con sondas contra cualquier gen de interés, incluyendo el marcador neuronal ChAT o el factor de transcripción Engrailed, permitiendo la detección sensible de un panel personalizable de especies de ARNm. Estas sondas se pueden diseñar para ser específicas de isoformas, soportando la especificidad de destino adicional si se desea. Double FISH se puede realizar utilizando sondas conjugadas con varios dos fluoróforos diferentes, permitiendo la evaluación simultánea de la expresión de múltiples genes. Sin embargo, los lavados adicionales requeridos por este tipo de experimento se asocian con una mayor probabilidad de pérdida o movimiento celular y requieren experiencia y delicadeza para realizarse con éxito.
Independientemente de las modificaciones específicas del experimento realizadas en este protocolo, hay varios pasos clave que requieren una atención cuidadosa. Las disecciones deben realizarse con cuidado para eliminar todos los tejidos contaminantes o las poblaciones celulares; debido a que el patrón espacial se pierde cuando los explantes se disocian, las células restantes de los tejidos vecinos se intercalarán e indistinguibles de las células de interés. Después de que las células estén chapadas, las muestras deben manipularse con la la forma más suave posible para evitar que las células se desalojen. Lo más importante es que esto significa que todos los cambios en la solución deben realizarse lenta y cuidadosamente, con la pipeta colocada en el borde de la placa cuando la solución se está retirando y agregando. Esto asegurará que las células se puedan identificar con confianza en imágenes de calcio y FISH. Si las celdas se interrumpen durante el procesamiento, puede ser imposible identificar algunas o todas las celdas correspondientes entre las dos imágenes. Aconsejamos erring en el lado de la precaución con estas asignaciones, de tal manera que sólo inequívocamente las células correspondientes se utilizan para el análisis posterior.
Dependiendo de la cuestión biológica que se trate, una variedad de enfoques de análisis pueden ser apropiados. La actividad de calcio en serie temporal se puede procesar y cuantificar de diversas maneras, con flexibilidad del experimentador a la hora de elegir parámetros de destendencia, métricas de análisis y parámetros de análisis (por ejemplo, el % del umbral de línea base utilizado para definir un pico de calcio). Las correlaciones entre la actividad del calcio y el nivel de expresión génica se pueden dibujar analizando la expresión génica como un valor de fluorescencia absoluto o relativo extraído de la imagen FISH. Alternativamente, las correlaciones entre la actividad del calcio y la expresión génica (presencia/ausencia) se pueden extraer definiendo un umbral de fluorescencia para la señal de expresión génica positiva y asignando identificadores de "sí" o "no" a células individuales. En su conjunto, este esquema experimental proporciona una canalización increíblemente flexible para la recopilación y el análisis preliminar de datos de series temporales junto con datos de expresión génica coincidentes con células. Tales experimentos serán críticos para comprender mejor las complejas relaciones entre la dinámica celular y los cambios transcripcionales, como lo ejemplifica la identificación de patrones de actividad de calcio característicos de los precursores neuronales con engorde inhibidor y excitatorio en Xenopus laevisembrionario.
Divulgaciones
No se declaran conflictos de intereses.
Agradecimientos
Agradecemos a Wendy Herbst y Lindsay Schleifer por sus contribuciones al desarrollo de estos protocolos. Este trabajo fue apoyado por subvenciones de los Institutos Nacionales de Salud (1R15NS067566-01, 1R15HD077624-01 y 1R15HD096415-01) a MSS.
Materiales
| Name | Company | Catalog Number | Comments |
| For Animal Husbandry & Cell Culture | |||
| CHORULON (chorionic gonodotropin) | Merck Animal Health | ||
| Gentamycin sulfate salt | Millipore Sigma | G1264 | |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fisher Scientific | 15140122 | |
| Pyrex petri dishes, 100 mm x 20 mm | Millipore Sigma | CLS3160102 | |
| Corning Falcon Easy-Grip Tissue Culture Dishes, 35mm | Fisher Scientific | 08-772A | |
| Corning Falcon Easy-Grip Tissue Culture Dishes, 60mm | Fisher Scientific | 08-772F | |
| Falcon Standard Tissue Culture Dishes | Fisher Scientific | 08-772E | |
| Thermo Scientifc Nunc Cell Culture / Petri Dishes, 35x10mm Dish, Nunclon Delta | Fisher Scientific | 12-565-90 | |
| Fisherbrand Standard Disposable Transfer Pipettes, Nongraduated; Length: 5.875 in.; Capacity: 7.7 mL | Fisher Scientific | 13-711-7M | |
| Ethyl 3-aminobenzoate methanesulfonate | Millipore Sigma | E10521 | |
| Collagenase B | Millipore Sigma | 11088807001 | |
| Dumont #55 Forceps, Dumostar | Fine Science Tools | 11295-51 | |
| Dumont #5 Forceps, Dumostar | Fine Science Tools | 11295-00 | |
| Cellattice Micro-Ruled Cell Culture Surface | Nexcelom Bioscience | CLS5-25D-050 | |
| For Calcium Imaging | |||
| Fluo-4, AM, cell permeant | Thermo Fisher Scientific | F14201 | |
| Pluronic F-127, 0.2 µm filtered (10% Solution in Water) | Thermo Fisher Scientific | P6866 | |
| For RNA Probe Generation | |||
| PureYield Plasmid Miniprep System | Promega | A1222 | |
| rATP | Promega | P1132 | |
| rCTP | Promega | P1142 | |
| rGTP | Promega | P1152 | |
| rUTP | Promega | P1162 | |
| Digoxigenin-11-UTP | Millipore Sigma | 3359247910 | |
| Rnase Inhibitor | Thermo Fisher Scientific | N8080119 | |
| T3 RNA Polymerase | Promega | P2083 | |
| T7 RNA Polymerase | Promega | P2075 | |
| SP6 RNA Polymerase | Promega | P1085 | |
| RQ1 Rnase-Free Dnase | Promega | M6101 | |
| LiCl Precipitation Solution (7.5 M) | Thermo Fisher Scientific | AM9480 | |
| For Fluorescence In Situ Hybridization | |||
| Acetic Anhydride | Thermo Fisher Scientific | 320102 | |
| Blocking Reagent | Millipore Sigma | 11096176001 | |
| Anti-Digoxigenin-POD, Fab fragments | Millipore Sigma | 11207733910 | |
| Cy3 Mono-Reactive NHS Ester | Millipore Sigma | GEPA13105 | |
| Solution Components | |||
| Calcium chloride, 96% extra pure, powder, anhydrous, ACROS Organixs | Fisher Scientific | AC349610 | |
| Calcium chloride dihydrate | Millipore Sigma | C3306 | |
| CHAPS hydrate | Millipore Sigma | C3023 | |
| Denhardt's Solution (50X) | Thermo Fisher Scientific | 750018 | |
| DTT, Molecular Grade (DL-Dithiothreitol) | Promega | P1171 | |
| Ethylenediaminetetraacetic Acid, Disodium Salt Dihydrate | Fisher Scientific | S311 | |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N',N'-tetraacetic acid | Millipore Sigma | E3889 | |
| Formamide (Deionized) | Thermo Fisher Scientific | AM9342 | |
| Herparin sodium salt from porcine intestinal mucosa | Millipore Sigma | H3393 | |
| HEPES (Ultra Pure) | Thermo Fisher Scientific | 11344041 | |
| Hydrogen peroxide solution | Millipore Sigma | H1109 | |
| L-Cysteine | Millipore Sigma | 168149 | |
| Magnesium chloride, pure, ACROS Organics | Fisher Scientific | AC223211000 | |
| Magnesium sulfate, 97% pure, ACROS Organixs, anhydrous | Fisher Scientific | AC413480050 | |
| Maleic Acid, 99%, ACROS Organics | Fisher Scientific | ACS125231000 | |
| MOPS (Fine White Crystals/Molecular Biology), Fisher BioReagents | Fisher Scientific | BP308 | |
| Potassium chloride | Millipore Sigma | P9541 | |
| Ribonucleic acid from torula yeast, Type IX | Millipore Sigma | R3629 | |
| Sodium chloride | Millipore Sigma | S7653 | |
| Triethanolamine | Millipore Sigma | 90279 | |
| Tris | Millipore Sigma | GE17-1321-01 | |
| TWEEN 20 | Millipore Sigma | P9416 | |
| Equipment | |||
| Laminar Flow Hood | model of choice | ||
| Dissecting Microscope | model of choice | ||
| Inverted Fluorescence Microscope | Nikon | TE200 | |
| NIS-Elements Imaging Software | Nikon | ||
| Shaking Incubator | model of choice | ||
| Refrigerated Centrifuge | model of choice | ||
| Miscellaneous | |||
| Corning bottle-top vaccum filter system, 0.22 μm pore, 500 mL bottle capacity | Millipore Sigma | CLS430769 | |
| Falcon 50mL Conical Centrifuge Tubes | Fisher Scientific | 14-432-22 |
Referencias
- Humeau, J., et al. Calcium signaling and cell cycle: Progression or death. Cell Calcium. 70, 3-15 (2017).
- Kim, J. M., Lee, M., Kim, N., Heo, W. D. Optogenetic toolkit reveals the role of Ca2+ sparklets in coordinated cell migration. PNAS. 112 (21), 5951-5957 (2016).
- Orrenius, S., Zhivotovsky, B., Nicotera, P. Regulation of cell death: the calcium-apoptosis link. Nature Reviews Molecular Cell Biology. 4 (7), 552-565 (2003).
- Pham, K., et al. Ca2+ and Mg2+ module conformational dynamics and stability of downstream regulatory element antagonist modulator. Protein Science. 24 (5), 741-751 (2015).
- Smedler, E., Uhlén, P. Frequency decoding of calcium oscillations. Biochimica et Biophysica Acta. 1840 (3), 964-969 (2014).
- Moreau, M., Néant, I., Webb, S. E., Miller, A. L., Riou, J. F., Leclerc, C. Ca(2+) coding and decoding strategies for the specification of neural and renal precursor cells during development. Cell Calcium. 59 (2-3), 75-83 (2016).
- Tomida, T., Hirose, K., Takizawa, A., Shibasaki, F., Iino, M. NFAT functions as a working memory of Ca2+ signals in decoding Ca2+ oscillation. EMBO. 22 (15), 3825-3832 (2003).
- Hannanta-Anan, P., Chow, B. Y. Optogenetic Control of Calcium Oscillation Waveform Defines NFAT as an Integrator of Calcium Load. Cell Systems. 2 (4), 283-288 (2016).
- Li, L., Stefan, M. I. Le Novère N. Calcium input frequency, duration and amplitude differentially module the relative activation of calcineurin and CaMKII. PLoS One. 7 (9), 43810(2012).
- Romano, D. R., Pharris, M. C., Patel, N. M., Kinzer-Ursem, T. L. Competitive tuning: Competition's role in setting the frequency-dependence of Ca2+-dependent proteins. PLoS Computational Biology. 13 (11), 1005820(2017).
- Pharris, M. C., Patel, N. M., Kinzer-Ursen, T. L. Competitive Tuning Among Ca2+/Calmodulin-Dependent Proteins: Analysis of in silico Model Robustness and Parameter Variability. Cellular and Molecular Bioengineering. 11 (5), 353-365 (2018).
- Gu, X., Olson, E. C., Spitzer, N. C. Spontaneous neuronal calcium spikes during early differentiation. Journal of Neuroscience. 14 (11), 6325-6335 (1994).
- Borodinsky, L. N., Root, C. M., Cronin, J. A., Sann, S. B., Gu, X., Spitzer, N. C. Activity-dependent homeostatic specification of transmitter expression in embryonic neurons. Nature. 429 (6991), 523-530 (2004).
- Ciccolini, F., Collins, T. J., Sudhoelter, J., Lipp, P., Berridge, M. J., Bootman, M. D. Local and Global Spontaneous Calcium Events Regulate Neurite Outgrowth and Onset of GABAergic Phenotype during Neural Precursor Differentiation. Journal of Neuroscience. 23 (1), 103-111 (2003).
- Weissman, T. A., Riquelme, P. A., Ivic, L., Flint, A. C., Kriegstein, A. R. Calcium Waves Propagate through Radial Glial Cells and Modulate Proliferation in the Developing Neocortex. Neuron. 43 (5), 647-661 (2004).
- Nieuewkoop, P. D., Faber, J. The stages of Xenopus embryonic development. Normal Table of Xenopus laevis. , North Holland Publishing Company. (1994).
- Paudel, S., et al. Calcium Activity Dynamics Correlate with Neuronal Phenotype at a Single Cell Level and in a Threshold-Dependent Manner. International Journal of Molecular Science. 20 (8), 1880(2019).
- Marken, J. P., et al. A Markovian Entropy Measure for the Analysis of Calcium Activity Time Series. PLoS One. 11 (12), 0168342(2016).
- Eilers, P. H. C., Boelens, H. F. M. Baseline Correction with Asymmetric Least Squares Smoothing. Leiden University Medical Centre Report. , (2005).
- Guemez-Gamboa, A., et al. Non-cell-autonomous mechanism of activity-dependent neurotransmitter switching. Neuron. 82 (5), 1004-1016 (2014).
- Paredes, R., Madelaine,, et al. Chemical calcium indicators. Methods. 46 (3), 143-151 (2008).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados