Method Article
Fluoreszierende Calcium-Bildgebung und anschließende In-Situ-Hybridisierung zur neuronalen Vorläufercharakterisierung bei Xenopus laevis
In diesem Artikel
Zusammenfassung
Wir präsentieren ein zweiteiliges Protokoll, das fluoreszierende Calcium-Bildgebung mit In-situ-Hybridisierung kombiniert, so dass der Experimentator Muster der Calciumaktivität mit Genexpressionsprofilen auf einzelzelligem Niveau korrelieren kann.
Zusammenfassung
Spontane intrazelluläre Kalziumaktivität kann in einer Vielzahl von Zelltypen beobachtet werden und wird vorgeschlagen, kritische Rollen in einer Vielzahl von physiologischen Prozessen zu spielen. Insbesondere ist eine angemessene Regulierung der Kalziumaktivitätsmuster während der Embryogenese für viele Aspekte der neuronalen Entwicklung von Wirbeltieren erforderlich, einschließlich des richtigen Neuralrohrverschlusses, der Synaptogenese und der Phänotypspezifikation des Neurotransmitters. Während die Beobachtung, dass sich Kalziumaktivitätsmuster sowohl in Derfrequenz als auch in Amplitude unterscheiden können, einen zwingenden Mechanismus nahelegt, mit dem diese Flussmittel kodierte Signale an nachgeschaltete Effektoren übertragen und die Genexpression regulieren könnten, Ansätze auf Bevölkerungsebene haben nicht die nötige Präzision, um diese Möglichkeit weiter zu erkunden. Darüber hinaus begrenzen diese Ansätze Studien über die Rolle von Zell-Zell-Wechselwirkungen, indem sie die Fähigkeit ausschließt, den Zustand der neuronalen Bestimmung in Abwesenheit von Zell-Zell-Kontakt zu untersuchen. Daher haben wir einen experimentellen Workflow etabliert, der die Zeitraffer-Calcium-Bildgebung dissoziierter neuronaler Explanten mit einem Fluoreszenz-In-situ-Hybridisierungstest kombiniert, der die eindeutige Korrelation von Calciumaktivitätsmuster mit molekularen Phänotyp auf einzelzelligem Niveau. Wir konnten diesen Ansatz erfolgreich nutzen, um spezifische Calciumaktivitätsmuster zu unterscheiden und zu charakterisieren, die mit der Differenzierung von Nervenzellen bzw. neuronalen Vorläuferzellen verbunden sind; Darüber hinaus könnte jedoch der in diesem Artikel beschriebene experimentelle Rahmen leicht angepasst werden, um Korrelationen zwischen jedem Zeitreihenaktivitätsprofil und der Expression eines Gens oder Gens von Interesse zu untersuchen.
Einleitung
Freies zytosolisches Kalzium ist entscheidend für eine Vielzahl von biologischen Prozessen, von Zellproliferation und Migration bis hin zu Apoptose und Autophagie1,2,3. Innerhalb dieser Bahnen kann Kalzium nachgelagerte Auswirkungen auf die Genexpression ausüben, indem es mit Calcium-bindenden Domänen interagiert, um konformationale Veränderungen zu induzieren, die die Proteinaktivität und -wechselwirkungen modulieren. Zum Beispiel wird ein neuronaler Kalziumsensor, der als Downstream Regulatory Element Antagonist Modulator (DREAM) bekannt ist, in einer entfalteten Zwischenkonformation gehalten, wenn er durch Kalzium gebunden wird, wodurch er daran gehindert wird, mit seinem Protein und seinenDNA-Zielen4 zu interagieren. Abgesehen davon, dass sie als einfaches Signalmolekül dienen, ermöglicht die dynamische Natur intrazellulärer Calciumtransienten diesen Aktivitätsmustern jedoch, komplexere Amplituden- oder frequenzbasierte Signale5,6zu kodieren. Die kernnukleare Translokation des Transkriptionsfaktors Kernfaktor aktivierter T-Zellen (NFAT) wird durch hochfrequente Calciumschwingungen verstärkt, aber durch niederfrequente Schwingungen gehemmt7. Die jüngsten Arbeiten deuten darauf hin, dass NFAT tatsächlich auf die kumulative Kalziumexposition reagieren könnte8. Sowohl Calcineurin als auch Ca2+/calmodulin-abhängige Proteinkinase II (CaMKII) weisen auch unterschiedliche Reaktionen auf Kalziumtransienten einer bestimmten Häufigkeit, Dauer oder Amplitude9auf. Um eine zusätzliche Ebene der regulatorischen Komplexität zu erhöhen, deuten Rechenmodelle darauf hin, dass viele nachgeschaltete Calcium-bindende Proteine mehr oder weniger frequenzabhängig werden, als Reaktion auf das Vorhandensein oder Fehlen verbindlicher Wettbewerber10,11.
Innerhalb des sich entwickelnden Nervensystems wurden zwei Hauptklassen von Calciumaktivitätsverhalten definiert und mit spezifischen biologischen Prozessen in Verbindung gebracht. Kalziumzuflüsse werden als "Spikes" klassifiziert, wenn sie innerhalb einzelner Zellen auftreten, innerhalb von fünf Sekunden eine Spitzenintensität von 400 % der Ausgangswerte erreichen und einen doppelten exponentiellen Zerfall aufweisen12. Diese Art von Signal ist in erster Linie mit Neurotransmitter Phänotyp Spezifikation13verbunden. Im Gegensatz dazu werden "Wellen" als langsamere, weniger extreme Kalziumtransienten definiert, bei denen die intrazelluläre Kalziumkonzentration einer Zelle über einen Zeitraum von dreißig Sekunden oder mehr auf 200 % der Ausgangsbasis ansteigt und dann über mehrere Minuten12zerfällt. Diese Signale verbreiten sich oft über mehrere benachbarte Zellen, und ihre Anwesenheit wurde mit Neuritenwachstum und Zellproliferation14,15in Verbindung gebracht. Obwohl diese beiden Klassen auf der Grundlage charakteristischer kinetischer Profile definiert wurden, bleibt unklar, welche Merkmale dieser Muster tatsächlich von Zellen erkannt und von nachgeschalteten Effektoren übersetzt werden.
Das Verständnis des Zusammenhangs zwischen intrazellulären Kalziumoszillationen und Genexpression würde einen entscheidenden Einblick in einen der Regulierungsmechanismen geben, der eine angemessene Entwicklung und Musterung des Nervensystems sicherstellt. Zu diesem Zweck haben Studien des embryonalen Rückenmarks gezeigt, dass erhöhte Calcium-Spike-Aktivität während der Entwicklung mit höheren Konzentrationen von hemmenden Neuronen verbunden ist, während verminderte Calcium-Spike-Aktivität mit höheren Konzentrationen von erregenden Neuronen verbunden ist13. Diese Assays auf Populationsebene wurden jedoch nicht verwendet, um Kalziumaktivität mit Genexpression auf einer Einzelzellebene zu assoziieren.
Die Annäherung an diese Fragen auf der Ebene der Einzelzelle bietet mehrere deutliche Vorteile gegenüber früheren Arbeiten. Zum einen ermöglicht die Fähigkeit, die Kalziumaktivität und genexpression in vielen Zellen einzeln zu beurteilen, das gesamte Repertoire unterschiedlicher Aktivitätsmuster zu beobachten, ohne durch eine Massenmessung verschleiert zu werden. Darüber hinaus bedeutet das Studium dieser Beziehungen in der einzelzelligen Primärkultur, dass zellautonome Verbindungen zwischen Kalziumaktivität und Genexpression beibehalten werden, während Wechselwirkungen, die eine Zell-Zell-Kommunikation erfordern, aufgehoben werden. Daher ermöglicht dieser Ansatz, diese zellautonomen Mechanismen isoliert zu untersuchen. Es ermöglicht jedoch auch, die Rolle der nicht-zellautonomen Kalziumaktivität aufzuklären und zu verhören. Beispielsweise können Zellen im Stadium der neuralen Platte von einem Embryo seziert, kultiviert werden, bis die Geschwistersteuerung das Neuronröhrenstadium erreicht, und dann mit Zellen verglichen werden, die frisch aus einem Neuronröhren-Stadium-Embryo seziert wurden. Dies ermöglicht einen direkten Vergleich von Zellen, die die Zell-Zell-Kommunikation über einen wichtigen Entwicklungszeitraum hinweg beibehalten, mit denen die Zell-Zell-Kommunikation abgeschafft wurde.
Um die Grenzen früherer experimenteller Ansätze anzugehen, entwickelten wir ein Protokoll, das die Beurteilung sowohl der Kalziumaktivität als auch der Genexpression in einzelnen neuronalen Vorläuferzellen ermöglicht und die Korrelation spezifischer Aktivitätsmuster mit nachfolgenden Differenzierungsprogrammen erleichtert. Neuralgewebe wurde von Xenopus laevis in verschiedenen Stadien der neuronalen Entwicklung seziert, in einzelne Zellen dissoziiert und mittels konfokaler Mikroskopie in Gegenwart eines fluoreszierenden Kalziumindikators abgebildet. Nach der Live-Zell-Bildgebung wurden Proben fixiert und mittels Fluoreszenz-in-situ-Hybridisierung (FISH) untersucht, um die Expression eines Gens oder einer Isoform von Interesse zu erkennen. Wichtig ist, dass einzelne Zellen in beiden bildgebenden Experimenten nachverfolgt werden können, was bedeutet, dass das Calciumaktivitätsprofil einer Zelle und ihr Genexpressionsniveau miteinander assoziiert werden können (Abbildung 1). Das hier vorgestellte Protokoll soll Die Beziehungen zwischen Kalziumaktivitätsmustern und Genexpression über die embryonale Neuroentwicklung in Xenopus laevisuntersuchen. Der breitere experimentelle Rahmen (Einzelzell-Zeit-Kurs-Bildgebung, gefolgt von FISH und Bild-Coregistration) kann jedoch modifiziert und auf praktisch jeden Zelltyp, fluoreszierenden Reporter und Gen von Interesse angewendet werden.
Protokoll
Alle Arbeiten mit Tieren wurden in Übereinstimmung mit Protokollen durchgeführt, die vom Institutional Animal Care and Use Committee (IACUC) am College of William and Mary genehmigt wurden.
1. Tierpflege und Embryo-Handhabung
- Induzieren Sie die natürliche Paarung durch Verabreichung einer subkutanen Injektion von humanem Choriongonadotropin (HCG) in den dorsalen Lymphsack des erwachsenen Xenopus laevis in einer Dosis von 600 U für Frauen und 400 U für Männer.
- Nach der Injektion mindestens einen männlichen und einen weiblichen Frosch über Nacht in einen Raumtemperatur-Haltetank geben. Die Eiablage beginnt in der Regel 9-12 h nach der HCG-Verabreichung.
- Sammeln Sie Embryonen. Entjelly durch sanftes Waschen mit 2% Cystein (pH 8,0) für 2-4 min.
- Spülen Sie die Embryonen 3x in 0.1x Mark es Modified Ringer es Solution (MMR) (100 mM NaCl, 2 mM KCl, 1 mM MgSO4, 2 mM CaCl2, 5 mM HEPES, pH angepasst auf 7.4–7.6).
- Übertragen Sie die Embryonen auf 100 mm Glas Petrischalen mit 0,1x MMR und 50 g/ml Gentamycin. Eine Dichte von 50-100 Embryonen pro Platte ist angemessen.
- Die Schalen bei 14 °C–22°C bebrüten und die Embryonen entwickeln lassen, bis sie das gewünschte Entwicklungsstadium erreichen. Entfernen Sie regelmäßig unbefruchtete Zellen und nekrotische oder sich abnorm entwickelnde Embryonen mit einer Kunststoff-Transferpipette.
ANMERKUNG: Um Konsistenz zu gewährleisten, wird die Entwicklungsinszenierung nach morphologischen Kriterien durchgeführt, die von Nieuwkoop und Faber16definiert wurden. Die Stadien des Interesses variieren je nach experimentellem Fokus. Beispielsweise sind wichtige neuroentwicklungstechnische Landmarken mit Stufe 14 (Beginn der Neurulation), Stufe 18 (Beginn des Neuralrohrverschlusses) und Stufe 22 (Beginn der Schwanzknospendehnung) verbunden.
2. Embryo-Dissektion und Probenvorbereitung
- Lösungsvorbereitung
- Bereiten Sie 2 mM Ca2+ Lösung mit 116 mM NaCl, 0,67 mM KCl, 2 mM CaCl2,2H2O, 1,31 mM MgSO4und 4,6 mM Tris vor. Geben Sie für jede 100 ml-Lösung 1 ml Penicillin/Streptomycin (10.000 U/ml Penicillin; 10.000 g/ml Streptomycin) hinzu. PH auf 7,8 einstellen und filtersterilisieren.
- Bereiten Sie kalzium- und magnesiumfreie (CMF) Lösung durch Kombination von 116 mM NaCl, 0,67 mM KCl, 4,6 mM Tris und 0,4 mM EDTA. Stellen Sie den pH-Wert auf 7,8 und den Autoklaven ein, um zu sterilisieren. Geben Sie für jede 100 ml-Lösung 1 ml Penicillin/Streptomycin (10.000 U/ml Penicillin; 10.000 g/ml Streptomycin) hinzu. PH auf 7,8 einstellen und filtersterilisieren.
- Herstellung von Platten für Diesektion und Bildgebung
- UV-sterilisieren sie zwei 35 mm Kunststoff-Petrischalen und eine 35 mm Zellkulturschale (siehe Materialtabelle).
- Bereiten Sie bei der Arbeit in einer laminaren Durchflusshaube zwei 50 ml Kunststoffkonusrohre vor, die jeweils 10 ml 2 mM Ca2+ Lösung enthalten.
- Bleiben Sie in der laminaren Durchflusshaube und fügen Sie 2 ml 2 mM Ca2+ Lösung zu einer 35 mm Kunststoff Petrischale, 2 ml 2 mM Ca2+ Lösung zu einer 35 mm Zellkulturschale und 2 ml CMF-Lösung zu einer 35 mm Kunststoff Petrischale hinzu.
- Außerhalb der laminaren Durchflusshaube zwei 100 mm Kunststoff Petrischalen und eine 35 mm Kunststoff Petrischale mit 0,1x MMR mit Gentamycin (50 g/ml) füllen. Füllen Sie eine 60 mm Kunststoff Petrischale mit 70% Ethanol.
- Unmittelbar vor der Zerlegung 0,01 g Collagenase B in eines der 50 ml-Rohre, die Ca2+ Lösung enthalten. Gut mischen und die Lösung auf eine frische 60 mm Kunststoff Petrischale übertragen.
- Identifizieren Sie mit Hilfe eines Sezierensmikroskops Embryonen des gewünschten Entwicklungsstadiums. Verwenden Sie eine sterile Transferpipette, um mindestens sechs geeignete Embryonen auf eine der 100-mm-Platten zu übertragen, die 0,1x MMR + Gentamycin enthalten, die in Schritt 2.2.4 hergestellt werden. Dies dient als Halteplatte.
- Verwenden Sie eine sterile Transferpipette, um einen Embryo auf die zweite 100-mm-Platte mit 0,1x MMR + Gentamycin zu übertragen. Dies dient als Sezierplatte.
- Wenn Embryonen seziert werden, die alt genug sind, um sich zu bewegen (ungefähr Stadium 23 oder älter), befeuchten Sie jeden Embryo vor der Zerlegung, indem Sie ihn in eine Schale übertragen, die 0,1% 3-Aminobenzoesäure-Ethylester enthält, die in 0,1x MMR mit Gentamycin (50 g/ml) verdünnt wird. Sobald der Embryo immobilisiert ist, übertragen Sie ihn zurück in die Schale, die 0,1x MMR mit Gentamycin (50 g/ml) enthält, und fahren Sie mit der Zerlegung fort.
- Entfernen Sie vorsichtig die Vitelline-Membran, die den Embryo umgibt. Dies kann am einfachsten durch die Verwendung eines Paares von stumpfen Zangen, um den Embryo zu stabilisieren, während mit einem Paar feine Zange, um die Membran zu greifen. Ziehen Sie vorsichtig mit den feinen Zangen, um die Vitelline-Membran auseinander zu schälen.
- Verwenden Sie feine Zangen, um die dorsalen und ventralen Bereiche des Embryos zu trennen. Dies kann durch die Verwendung der Zange erfolgen, um den Embryo entlang der vorderen hinteren Achse zu "kneifen" und ihn zu halbieren. Mit einer sterilen Transferpipette den dorsalen Teil mit Kollagenaselösung in Schritt 2.2.5 auf die 60 mm Platte übertragen. Entsorgen Sie den ventralen Teil.
- Lassen Sie die dorsale Explantation in der Kollagenaselösung für 1–2 min bei Raumtemperatur inkubieren. Übertragen Sie es vorsichtig zurück auf die Sezierplatte.
- Vervollständigen Sie die Dissektion, indem Sie sorgfältig alle restlichen endodermalen und mesodermalen Verunreinigungen aus dem mutmaßlichen Neuronalgewebe des Ektoderms entfernen. Bei Embryonen ab Stufe 22 sollte das Neuralrohr ebenfalls entfernt und entsorgt werden.
HINWEIS: Bei Bedarf kann die in Schritt 2.2.4 zubereitete Schale aus 70 % Ethanol verwendet werden, um die Zange zu löschen oder erneut zu sterilisieren. - Nach Abschluss der Sezierung die Explantation vorsichtig auf die in Schritt 2.2.3 hergestellte 35-mm-Platte der Ca2+-Lösung übertragen.
- Wiederholen Sie die Schritte 2.4-2.9, bis vier Ex-Pflanzen gesammelt wurden.
- Verwenden Sie eine P1000-Mikropipette, um alle vier Explants auf die 35-mm-Platte mit CMF-Lösung zu übertragen, wobei darauf geachtet wird, dass kein Kontakt zwischen den Explants und der Luft-Wasser-Schnittstelle besteht. Wirbeln Sie die Schale vorsichtig, so dass sich alle Explants in der Mitte der Platte häufen.
- 1 h bei Raumtemperatur inkubieren, damit sich Die Explantationen dissoziieren können.
HINWEIS: Zur Unterstützung der Dissoziation können der CMF-Lösung 0,025% – 0,01% Trypsin hinzugefügt werden. Dies kann für eine effiziente Dissoziation älterer Embryonen erforderlich sein (Stufe 22 und älter). - An dieser Stelle sollten mindestens zwei entsprechend inszenierte Embryonen auf der Halteplatte verbleiben. Übertragen Sie diese Embryonen auf eine frische Schale, die mit 0,1x MMR gefüllt ist, mit Gentamycin, das in Schritt 2.2.4 zubereitet wird, und lassen Sie sie sich ungestört mit der abgedeckten Schale entwickeln, die der Explantschale entspricht. Diese Embryonen dienen als Geschwisterkontrollen.
- Verwenden Sie Superkleber, um einen mikrogeregelten Deckelschlupf (siehe Materialtabelle)an der Unterseite der in Schritt 2.2.3 hergestellten 35 mm Zellkulturschale zu befestigen.
HINWEIS: Legen Sie kleine Tupfer Superkleber um die Ränder des Deckels, dann drücken Sie es fest gegen die Unterseite der Cell Culture Dish. Die Positionsmarkierungen werden überall dort verdeckt, wo der Kleber mit dem Gitter in Berührung kommt, daher ist es wichtig, den zentralen Gitterteil des Deckelrutsches frei von Klebstoff zu halten. - Nachdem sich die Expflanzen 1 h getrennt haben, verwenden Sie eine P100-Mikropipette, um sie zum Zellkulturgericht zu übertragen. Um so viele Zellen wie möglich auf den gerasterten Teil der Schale zu verkleben, halten Sie die Pipette in einem flachen Winkel nahe der Oberfläche der Schale, positionieren Sie die Pipettenspitze in der Ecke des Gitters nach innen gerichtet, und vertreiben Sie die Zellsuspension fest über die gerasterte ar ea. Idealerweise setzen sich Zellen in einem dichten dichten Cluster ab.
- 1 h bei Raumtemperatur inkubieren, damit die Zellen an der Platte haften können. Bestimmen und erfassen Sie die Entwicklungsphase der Geschwisterkontrollembryonen, wenn diese Inkubation beginnt.
- Kombinieren Sie 5 l 1 mM Fluo-4 AM (siehe Materialtabelle) mit 2 l mit 10% Pluronischer F-127-Säure.
HINWEIS: Fluo-4 AM ist lichtempfindlich und sollte jederzeit in einem lichtsicheren oder folienbedeckten Rohr aufbewahrt werden. - Nachdem die Inkubation abgeschlossen ist, verschieben Sie die Probenschale in eine Dunkelkammer oder an einen anderen lichtgeschützten Ort. Verwenden Sie eine Mikropipette, um 100 l Lösung vom Rand der Schale zu entfernen. Fügen Sie diese Lösung zum Aliquot von Fluo-4 AM/Pluronic F-127 Säure, Pipette nach oben und unten, um zu mischen, und geben Sie das volle Volumen auf die Probenschale zurück. Sanft wirbeln, um es zu mischen.
- Bedecken Sie die Platte mit Aluminiumfolie und lassen Sie für 1 h bei Raumtemperatur inkubieren. Bestimmen und erfassen Sie die Entwicklungsphase der Geschwisterkontrollembryonen, wenn diese Inkubation beginnt.
- Am Ende der Inkubation verwenden Sie die verbleibende konische Röhre von 2 mM Ca2+, um drei Medienwäschen in folgender Weise durchzuführen: 1) entfernen Sie 1 ml Lösung aus der Schale, fügen Sie 3 ml frische Lösung, 2) entfernen 3 ml Lösung aus der Schale, fügen Sie 3 ml frische Lösung, 3) entfernen 3 ml Lösung aus der Schale, fügen Sie 3 ml frische Lösung.
3. Calcium Imaging
HINWEIS: Die Calcium-Bildgebung wurde mit einem invertierten konfokalen Mikroskop (Materialtabelle )durchgeführt.
- Legen Sie die Probenplatte auf die Mikroskopstufe und achten Sie darauf, sie vor Umgebungslichteinwirkung zu schützen. Sobald die Platte gesichert ist, verwenden Sie einen Marker, um den vorderen Punkt der Platte zu beschriften, so dass das gleiche Sichtfeld in der nachfolgenden Abbildung gefunden werden kann.
- Suchen Sie die Probe unter dem Mikroskop – zuerst bei 10X und dann bei der 20-fachen Vergrößerung – und wählen Sie ein geeignetes Sichtfeld für die Bildgebung aus. Ein ideales Sichtfeld ist zelldicht, aber nicht so dicht, dass Zellen verklumpt oder schwer einzeln zu unterscheiden sind.
- Stellen Sie den Mikroskopfokus so ein, dass der gittergeregelte Abdeckungsslip sichtbar ist. Die auf dem Deckblatt markierten Zahlen dienen als eindeutige Kennungen für einen bestimmten Raster-Lokus und können verwendet werden, um dasselbe Sichtfeld für zusätzliche Bildgebung zu lokalisieren. Wenn sich das ursprünglich ausgewählte Sichtfeld nicht mit Zahlen überlappt, müssen Sie nachjustieren, bis eine identifizierbare Zahl im Rahmen ist.
- Nehmen Sie ein helles Feldbild des ausgewählten Sichtfeldes mit dem gitterregelbaren Deckslip im Fokus.
- Passen Sie die Fokuseinstellungen an, und nehmen Sie ein helles Feldbild des ausgewählten Sichtfelds mit den Zellen im Fokus.
- Mit der Zellschicht im Fokus, beleuchten Sie die Proben mit einem 488 nm Laser. HV- und Offset-Werte können für jedes Experiment optimiert werden, um sicherzustellen, dass ein dynamischer Fluoreszenzbereich auf dem FITC-Kanal erkannt wird.
- Ändern Sie für ein zweistündiges Bild die Bildkonfiguration, um 901 Frames mit einer Scanzeit von 3,93 s und einem Intervall von 8 s aufzuzeichnen.
- Sobald die Bildgebung abgeschlossen ist, entfernen Sie die Platte von der Mikroskopstufe. Entfernen Sie 1 ml Lösung von der Platte und ersetzen Sie sie durch 1 ml 2x MEMFA (200 mM MOPS, 2 mM EGTA und 2 mM MgSO4 in 7,4% Formaldehyd).
- Inkubieren Sie die Platte für 2 h bei Raumtemperatur oder über Nacht bei 4 °C. Bestimmen und erfassen Sie die Entwicklungsphase der Geschwisterkontrollembryonen, wenn diese Inkubation beginnt.
- Nachdem die Fixierung abgeschlossen ist, entfernen Sie alle Lösung von der Platte und ersetzen Sie sie durch 2 ml 1x PBS. Platten bei 4 °C zur Weiterverarbeitung lagern.
4. Genexpressionsanalyse: Sondensynthese
- Wie unten beschrieben, erzeugen Sie eine Antisense-RNA-Sonde für die In-situ-Hybridisierung. Darüber hinaus generieren Sie eine Sinnsonde für das gleiche Gen für die Verwendung als Negative Kontrolle.
- Um Plasmid-DNA zu reinigen, die die Sondenschablonensequenz enthält, impfen Sie 150 ml LB-Brühe mit bakteriellen Glycerinvorräten, die das Template-Plasmid enthalten. Bei 37 °C mit Schütteln über Nacht oder bis die Kultur trüb ist.
- Reinigen Sie Plasmid-DNA aus der Bakterienkultur mit Ihrer Methode Ihrer Wahl.
HINWEIS: Wir verwenden das McNary-Nagel Midi-Prep-Kit, um hohe Erträge an Plasmid-DNA zu erhalten. - Um zu bestätigen, dass das Plasmid den erwarteten Einsatz enthält, führen Sie einen Restriktions-Digest durch und analysieren Sie die Produkte auf einem Agarose-Gel. Ungeschnittenes Plasmid kann auch auf einem Agarose-Gel analysiert werden, um auf genomische DNA-Kontamination zu überprüfen.
- Um die Schablonen-DNA linearisieren, richten Sie eine 100-L-Restriktions-Digest-Reaktion ein, die 20 g Plasmid-DNA, 2 l geeignetes Restriktionsenzym und 1x entsprechenden Puffer enthält. Bei 37 °C für mindestens 2 h inkubieren.
- Extrahieren Sie linearisierte DNA durch Durchführung einer Phenol-/Chlorformextraktion, gefolgt von einer Chloroformextraktion.
- Niederschlagen Sie DNA mit 100% Ethanol. Dies kann schnell geschehen, indem man der Probe zwei Mengen kaltes Ethanol hinzufügt und sie bei -80 °C inkubiert, bis sie sich erstarrt (15-30 min).
- Verwenden Sie eine gekühlte Zentrifuge, um DNA durch Spinnen für 20 min bei 12.000 x g/4 °C zu pellet.
- Entfernen Sie den Überstand und waschen Sie das Pellet mit 200 l 70% Ethanol. Drehen Sie 5 min bei 12.000 x g/4 °C.
- Das überstande und lufttrockene Pellet ca. 5 min entfernen. In 20 l 1x TE aufbewahren und bis zur weiteren Verwendung bei 4 °C lagern.
- Um die Antisense-RNA-Sonde zu synthetisieren und zu reinigen, erstellen Sie einen 2,5 mM rNTP-Mix, indem Sie 15 l mit 10 mM rCTP, 15 l mit 10 ml rGTP, 15 l von 10 mM rATP, 9,75 l mit 10 mM rUTP und 5,25 l mit 10 mM Dig-11 UTP (siehe Tabelle der Materialien)kombinieren.
- Richten Sie eine 50 l In-vitro-Transkriptionsreaktion ein, die 4 g linearisierte Vorlagen-DNA aus Schritt 4.2-4.10 enthält, 15 l mit 2,5 mM rNTP-Mischung aus Schritt 4,11, 10 l 5x Transkriptionspuffer, 5 l 0,1 M DTT, 0,5 l RNAse-Inhibitor (20 U/L) und 1,5 l geeigneter RNA-Polymerase (T3, T7 oder SP6). 1 h bei 37 °C inkubieren.
- Fügen Sie der Reaktion eine zusätzliche RNA-Polymerase von 1,5 l hinzu und kehren Sie für eine weitere Stunde auf 37 °C zurück.
- Fügen Sie 1 l RQ1 DNAse zur Reaktion hinzu und inkubieren Sie bei 37 °C für 10 min, um die DNA-Vorlage zu verschlechtern.
- Fügen Sie 30 L 7,5 M LiCl-Lösung zu probe. Pipette bei -20 °C mindestens 1 h mischen und inkubieren.
- Mit einer gekühlten Zentrifuge die Probe 25 min bei 14.000 x g/4 °C drehen.
- Entfernen Sie den Überstand und spülen Sie das Pellet mit 500 l 70% Ethanol. Drehen Sie 5 min bei 14.000 x g/4 °C.
- Entfernen Sie das überstande und lufttrockene Pellet für ca. 5 min. Resuspend in 20 l nukleasefreies Wasser.
- Erstellen Sie bei einem 10-fachen Sondenbestand, indem Sie die Probe im Hybridisierungspuffer auf eine Konzentration von 10 ng/L verdünnen (50% Formamid, 5x SSC (Saline-Natriumcitrat; 750 mM NaCl, 75 mM Natriumcitrat, pH 7,0), 1 mg/ml Torul-RNA, 0,1 % Tween-20, 1x Denhardt-Lösung, 0,1% CHAPS, 10 mM EDTA und 100 g/ml Heparin). Bei -20 °C bis zur weiteren Verwendung lagern.
5. Genexpressionsanalyse: Fluoreszenz In Situ Hybridisierung
HINWEIS: Alle Waschungen sollten mit ca. 1 ml Lösung mit einer sterilen, individuell umwickelten Transferpipette durchgeführt werden. Die Pipette sollte beim Entfernen oder Hinzufügen von Lösung sanieren, und Waschungen sollten so schonend wie möglich durchgeführt werden, um sicherzustellen, dass Zellen nicht von der Plattenoberfläche entfernt werden und verloren gehen.
- 1x PBS von der Platte entfernen (ab Schritt 3.10). Ersetzen Sie es durch frische 1x PBS und brüten Sie 5 min bei Raumtemperatur.
- Kombinieren Sie 25 ml 0,1 M Triethanolamin (pH 8,0) mit 62,5 l Essigsäureanhydrid. Gut mischen. Waschen Sie die Platte mit dieser Lösung für 10 min.
- Waschen Sie die Platte mit 1x SSC für 5 min.
- Waschen Sie die Platte mit 0,02 M HCl für 10 min, um Zellen zu permeabilisieren.
- 2x mit 1x PBS für je 5 min waschen.
- Entfernen Sie die Lösung und fügen Sie 1 ml Hybridisationspuffer (50% Formamid, 5x SSC (750 mM NaCl, 75 mM Natriumcitrat, pH 7,0), 1 mg/ml Torul-RNA, 0,1% Tween-20, 1x Denhardt-Lösung, 0,1% CHAPS, 10 mM EDTA und 100 g/ml Heparin) zur Platte hinzu. Mit Schütteln mindestens 6 h bei 60 °C bebrüten.
- Entfernen Sie den Hybridisierungspuffer und ersetzen Sie die in Schritt 4.19 hergestellte 10-fache Mischung durch eine 1x RNA-Sondenlösung (verdünnt. Sense RNA Sonden können als Negativkontrolle verwendet werden.
- Mit Schütteln für 8-14 h bei 60 °C bebrüten.
- Entfernen Sie die Sonde und lagern Sie bei -20 °C.
HINWEIS: 1x Sondenverdünnung kann bis zu dreimal wiederverwendet werden, bevor sie verworfen wird. - Spülen Sie die Platte mit 0,2x SSC.
- Mit frischem 0.2x SSC für 1 h bei 60 °C waschen.
- Bewegen Sie die Platten auf Raumtemperatur und gleichnden Sie 5 min.
- Waschen Sie die Platte mit 0,2x SSC für 5 min.
- Waschen Sie die Platte mit 1x PBT für 15 min.
- Waschen Sie die Platte mit 2%H2O2 in 1x PBT (0,1% Triton-x-100) für 1 h.
HINWEIS: Diese Lösung ist lichtempfindlich, daher sollte sie für jedes Experiment frisch gemacht und vor Licht abgeschirmt werden. Die Platten sollten während dieser Inkubation auch vor Licht geschützt oder vereitelt werden. - Waschen Sie die Platte mit 1x TBST (150 mM NaCl, 50 mM Tris-HCl, pH 7.5, 0.1%Tween-20) für 15 min.
- Verdünnen Sie das blockblockierende Reagenz auf 2% im Maleinsäurepuffer (100 mM Maleinsäure, 150 mM NaCl, pH 7,5). Blockieren Sie die Zellen in dieser Mischung für mindestens 1 h bei Raumtemperatur.
- Ersetzen Sie die Blockierlösung durch Anti-Digoxygenin-POD-Antikörper, der 1:1.000 in 2% Blocking Reagenz im Maleinsäurepuffer verdünnt wird. Über Nacht bei 4 °C inkubieren.
- Spülen Sie die Platte 3x mit 1x TBST.
- 4x mit 2 ml 1x TBST für mindestens 15 min pro Wäsche mit kontinuierlichem Schaukeln waschen.
- 2x mit 1x PBT mindestens 10 min pro Wäsche mit kontinuierlichem Schaukeln waschen.
- Cy3-konjugiertes Tyramid 1:25 in 1x PBT verdünnen. Waschen Sie die Platte mit 750 l dieser Verdünnung für 5 min.
HINWEIS: Diese Lösung ist extrem lichtempfindlich, und Platten sollten für den Rest des Experiments vor Licht geesteht oder abgeschirmt werden, um eine Verschlechterung des Signals zu vermeiden. - Fügen Sie dieser Lösung 2,5 l von 0,3 %H2O2 hinzu und inkubieren Sie mit kontinuierlicher Schaukelung für weitere 40 min bei Raumtemperatur.
- 4x mit 1x TBST für mindestens 15 min pro Wäsche mit kontinuierlichem Schaukeln waschen.
- Spülen Sie mit 1x PBT.
- Fixieren Sie die Zellen durch Inkubation für 1 h bei Raumtemperatur in 1x MEMFA (100 mM MOPS, 1 mM EGTA und 1 mM MgSO4 in 3,7% Formaldehyd).
- Entfernen Sie die Lösung und ersetzen Sie sie durch 1x PBS. Bewahren Sie die Platten bis zur Weiterverarbeitung in einem vereitelten Behälter bei 4 °C auf.
6. Bildgebungszellen
HINWEIS: Die Bildgebung wurde mit einem invertierten konfokalen Mikroskop durchgeführt.
- Platzieren Sie die Probenplatte auf der Mikroskopstufe, und richten Sie die in Schritt 3.1 vorgenommene Markierung an der Vorderseite der Bühne aus.
- Fokussieren Sie das Bild so, dass der gittergefütterte Deckblatt sichtbar ist, und passen Sie mithilfe des Rasterbildes in Schritt 3.4 als Referenz das Sichtfeld an das im Kalziumbild erfasste Sichtfeld an (Abschnitt 3).
- Erfassen Sie Hellfeldbilder sowohl des Rastergitters als auch der Zellen.
- Beleuchten Sie die Proben mit einem 595 nm TRITC Laser. Passen Sie die Verstärkungswerte an, um das Signal mit den Bildern von den negativen Kontrollzellen entsprechend vom Hintergrund zu unterscheiden, und erfassen Sie ein Standbild.
HINWEIS: Idealerweise werden die Fluoreszenzspiegel im Hintergrund auf der Grundlage einer negativen Kontrollplatte bestimmt, die parallel zu einer nicht zielorientierten Sinn-RNA-Sonde verarbeitet wird. Die Gain-Einstellungen werden so eingestellt, dass diese Platte vollständig schwarz erscheint (entsprechend den Hintergrundebenen), und dann für andere Platten, die aus dieser experimentellen Charge abgebildet sind, konstant gehalten wird.
7. Datenverarbeitung
HINWEIS: Die Datenverarbeitung wurde mit der Nikon Elements-Software durchgeführt.
- Öffnen Sie das 2 h Calciumbild aus Schritt 3.7. Identifizieren Sie die Pixel, die jeder einzelnen Zelle entsprechen, indem Sie Binär > Spot-Erkennung > Helle Fleckenauswählen. Stellen Sie sicher, dass der FITC-Kanal ausgewählt ist.
- Farbige Kreise werden über einzelnen Zellen angezeigt, nachdem diese Ebene generiert wurde. Passen Sie die Zellenverteilungs- und Größenparameter so an, dass so viele Zellen wie möglich erkannt und einem eindeutigen Bezeichner zugeordnet werden.
- Verfolgen Sie die Zellen über alle Frames des Bildes, indem Sie zu Ansicht > Analysesteuerelemente > Nachverfolgungsoptionennavigieren. Legen Sie 5 Frames als maximale Lücke zwischen Denspuren fest, löschen Sie Objekte, die weniger als 600 Frames zugeordnet sind, und wählen Sie die Option Lücken schließen aus. Wählen Sie Binärdateien nachverfolgen aus, um die Zellverfolgung auf das Bild anzuwenden.
- Nachdem die Zellen nachverfolgt wurden, löschen Sie manuell jedes Objekt, das nicht einer einzelnen Zelle entspricht (z. B. ein Zellklumpen). Datenpunkte sollten jedoch nicht von einer weiteren Analyse auf der Grundlage der Morphologie der Kalziumaktivitätsspur ausgeschlossen werden.
- Wählen Sie im Bildbereich Overlay anzeigen > Binäre Objekt-ID anzeigenaus. Scrollen Sie zum Ende des Bildes (Frame 901) und wählen Sie Bearbeiten > Ansichtsmomentaufnahme erstellen (8Bit RGB)> Aktueller Frame > OK. Dadurch wird eine Momentaufnahme des letzten Bildrahmens erstellt, wobei die zugehörige Binär-ID jeder Zelle sichtbar ist. Speichern Sie dieses Bild.
- Exportieren Von Zeitreihendaten, indem Sie alle Objekte auswählen, gefolgt von Daten nach Excel exportieren. Speichern Sie die Ausgabe als CSV-Datei.
- Öffnen Sie das FISH-Bild. Optimieren Sie die Spoterkennung wie in den Schritten 7.1–7.2, indem Sie den TRITC-Kanal anstelle des FITC-Kanals verwenden. Löschen Sie manuell alle falsch zugewiesenen Binärdateien, und wählen Sie dann Automatisierte Messergebnisse > Messung aktualisieren aus, um die Signalintensität jeder Zelle zu berechnen. Exportieren Sie einen Bildmomentaufnahme und eine Datentabelle, indem Sie die Schritte 7.5 und 7.6 wiederholen.
- Erstellen Sie eine Kalkulationstabelle, in der Spalte A mit FISH Binary ID und Spalte B mit DerCalcium Binary IDbeschriftet ist. Öffnen Sie die in den Schritten 7.5 und 7.7 exportierten Bilder. Zeichnen Sie für jedes im FISH-Bild (Schritt 7.7) identifizierte Objekt die Binär-ID in Spalte A auf. Suchen Sie dann die entsprechende Zelle im Kalziumbild (Schritt 7.5) und notieren Sie diese binäre ID in Spalte B. Zellen, die in beiden Bildern nicht sicher identifiziert werden können, sollten der Kalkulationstabelle nicht hinzugefügt werden.
HINWEIS: Es kann hilfreich sein, ein Fotobearbeitungsprogramm wie Adobe Photoshop oder GIMP-Bildeditor zu verwenden, um beide Bilder zu öffnen, ein halbtransparentes Bild zu erstellen und es auf sein Partnerbild zu überlagern, um die beiden binären IDs, die jeder Zelle zugeordnet sind, leichter zu identifizieren und zu verknüpfen. - Für jede identifizierte Zelle sammeln (entweder manuell oder mit einem Skript) ihre Zeitreihen-Calciumdaten (mit der Calcium Binary ID verknüpft und in Schritt 7.6 exportiert) und seine Genexpressionsdaten (mit der FISH Binary ID verknüpft und in Schritt 7.7 exportiert).
ANMERKUNG: Nachgeschaltete Datenverarbeitung und -analyse können die Zusammenstellung dieser Daten in einer einzigen Datentabelle und die Anwendung einer Reihe von Analysetechniken umfassen, einschließlich Spike-Zählung, Fraktalanalyse und Markovian-Entropie, die es dem Prüfer ermöglichen, neuartige Muster der Calciumaktivität zu erkennen 17,18.
Ergebnisse
Ein erfolgreiches Beispiel für dissoziierte Zellen, die für die Kalzium-Bildgebung vorbereitet wurden, ist in Abbildung 2A zusehen. Die Zellen sind dicht plattiert, so dass die maximale Menge an Informationen aus jedem Bild gesammelt werden kann, aber nicht so dicht plattiert ist, dass einzelne Zellen nicht sicher voneinander unterschieden werden können. Fluoreszenz wird für jede definierte Zelle während des 2-h-Bildgebungszeitraums nachgewiesen. Die Visualisierung eines zusammengesetzten Diagramms, das die Spuren für alle in einem Experiment aufgezeichneten Zellen enthält, zeigt, inwieweit Massen- oder Populationsmessungen differenziertere Muster des Spaltverhaltens verschleiern können (Abbildung 2B). Wenn die aufgezeichneten Profile einzelner Zellen isoliert werden, können Beispiele für die unregelmäßige Spiking-Aktivität, die für neuronale Vorläuferzellen charakteristisch ist, eindeutig identifiziert werden. Im Gegensatz zu reifen Neuronen weisen embryonale neuronale Zellen eine unregelmäßige, hochvariable und komplexe Natur der Kalziumaktivität auf (Abbildung 2B). Um diese Komplexität zu quantifizieren, wurden die Anwendung verschiedener Datenanalysemethodenangewendet 17,18, einschließlich verschiedener Parameter, um eine Spitze zu definieren ( Abbildung2C).
Eine erfolgreiche Fluoreszenz-in-situ-Hybridisierung, einschließlich erfolgreicher Konstruktion und Synthese einer Antisense-mRNA-Sonde, kann durch den Vergleich der Versuchsplatte mit einer Hintergrundkontrolle, die mit einer unverbindlichen Sinn-RNA-Kontrolle inkubiert wird, bewertet werden (Abbildung 3A,B). Eine positive Sondenkontrolle kann auch durch Verarbeitung eines Zelltyps durchgeführt werden, von dem bekannt ist, dass er die Ziel-mRNA auf nachweisbaren Ebenen ausdrückt.
Die Identifizierung derselben Zelle über die Kalzium- und FISH-Bildgebung erfordert, dass die Zellen während der Sondenhybridisierung und -verarbeitung in etwa die gleiche Position behalten. Wenn Platten grob behandelt werden oder Washes zu kräftig ausgeführt werden, können Zellen von der Plattenoberfläche entfernt werden und entweder verloren gehen, wenn die Lösung verworfen oder an einer anderen Stelle auf der Platte abgelagert wird, so dass es unmöglich ist, sie bildübergreifend abzugleichen (Abbildung 4A). Wenn diese Unterbrechung nur einige der Zellen im Sichtfeld betrifft, kann es dennoch möglich sein, einige Zellen innerhalb des Bildes zu erkennen und zuzuweisen (Abbildung 4B). Die maximale Datenmenge wird jedoch aus einem Experiment gewonnen, bei dem FISH sorgfältig durchgeführt wird und nur wenige Zellen zwischen Bildern verloren gehen oder neu positioniert werden (Abbildung 4C).
Sobald Daten gesammelt wurden, um sowohl die Kalziumaktivität als auch die Genexpression einer angemessenen Anzahl von Zellen in der Entwicklungsphase(n) von Interesse zu beschreiben, können weitere Analysen durchgeführt werden, um Korrelationen zwischen diesen beiden Merkmalen zu bewerten (Abbildung 1). Eine Reihe von Metriken wurden angewendet, um Kalziumaktivitätsmuster zu quantifizieren, einschließlich Spike-Zählung/Frequenz, Durchschnittsleistung, Hurst-Exponentenschätzung und Markovian-Entropiemessung17,18. Die Genexpression kann quantitativ durch absolute Fluoreszenz definiert oder auf einer binären (Ja/Nein) Skala abgestuft werden, abhängig von den angesprochenen experimentellen Fragen.
Die Ergebnisse von Experimenten, die die Kalziumaktivität mit der Expression neuronaler Vorläufermarkergene zusammenfassten, zeigten zahlreiche Assoziationen zwischen spezifischen Mustern der Kalziumaktivität und Neurotransmitter-Phänotypen. Im Stadium der neuralen Platte (Stufe 14) weisen GABAerge Zellen, die den hemmenden Neuronenmarker gad1.1 exemittieren, eine regelmäßigere und höhere Amplitude auf als Zellen, die keine Gad1.1-Expression aufweisen (Abbildung 5A). Während diese gad1.1-exemittierendenZellen mit höheren Konzentrationen von Hochamplituden-Spiking assoziiert sind, ist das Spiking mit niedriger Amplitude häufiger in glutamatergen Zellen, die den exzitatorischen Neuronenmarker slc17a7exeklaten.
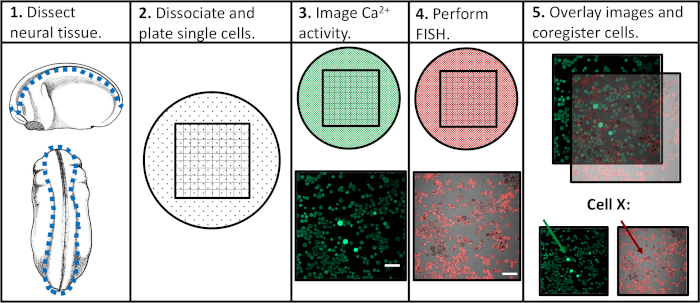
Abbildung 1: Schematic des experimentellen Workflows. Skalenbalken = 100 m. Die Bilder in den Tafeln 3-5 stammen von Paudel et al. (2019)17. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
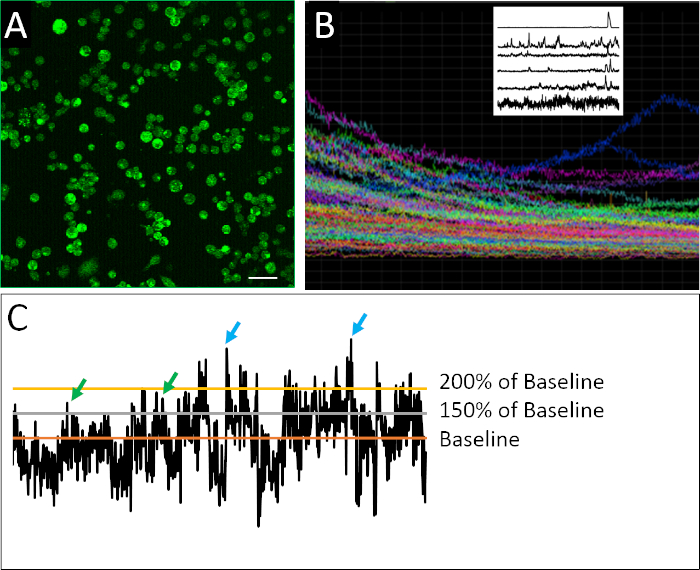
Abbildung 2: Calcium-Bildgebung und Beispielaktivitätsprofile. (A) Intrazelluläre Calciumaktivität, wie von Fluo4-AM berichtet. Jedes 2-H-Bild besteht aus 901 Bildern, mit einer repräsentativen Rahmenshow hier. (B) Zusammengesetztes Diagramm der Fluoreszenzintensität im Laufe der Zeit in allen Zellen innerhalb des abgebildeten Sichtfeldes. Die Spuren deuten deutlich auf photobleaching von Indikatorfarbstoff (Fluo4) Überstunden. Raster-Plot oben links zeigt repräsentative Spuren der Kalziumaktivität nach Anwendung eines De-Trending-Algorithmus von Eilers und Boelen19entwickelt , wo Zellen hier gezeigt zeigen unterschiedliche Muster der Spiking-Verhalten. (C) Anwendung verschiedener Schwellenwerte (150 % und 200 % des Ausgangswerts, wobei die Basislinie der Durchschnitt der de-trendierten Fluoreszenzintensität ist), um eine Spitze (grüne und blaue Pfeile) zu definieren. Skalenbalken = 100 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
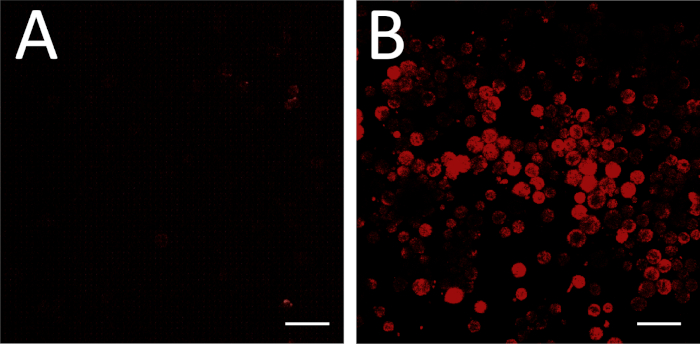
Abbildung 3: FISH-Bildgebung. (A) FISH wurde mit einer unverbindlichen Sense-RNA-Sonde als Negativkontrolle durchgeführt. Die Bildeinstellungen wurden so angepasst, dass keine Zellen fluoreszierend erscheinen. Einige Sichtfelder können nichtzellige Trümmer mit einer bestimmten Fluoreszenz enthalten, wie sie in der oberen rechten und unteren rechten Ecke von (A) zu sehen sind. Diese können aus Gründen der Hintergrundeinstellung ignoriert werden. (B) Die gleichen bildgebenden Einstellungen werden dann verwendet, um eine experimentelle Platte (Antisense-RNA-Sonde) abzubilden. Fluoreszenz unter diesen Bedingungen entspricht der Genexpression über dem Hintergrund. Skalenbalken = 100 m. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Bild-Overlay und Co-Registrierung. Schematische Darstellungen einer Probe, die für die Kalziumaktivität (Zellen dargestellt durch gefüllte grüne Kreise) und nach FISH (Zellen, die durch schattierte rote Kreise dargestellt werden) abgebildet sind. (A) Zellen, die sich während der Probenhandhabung und -verarbeitung erheblich bewegt haben, können in den beiden Bildern nicht zuverlässig identifiziert werden. (B) Zellunterbrechungen können nur einige Zellen im Sichtfeld betreffen. Einige Zellen können in beiden Bildern eindeutig identifiziert werden, während andere nicht sicher abgeglichen werden können. (C) Wenn Proben sorgfältig behandelt werden, bleiben die meisten Zellen ungestört und können in beiden Bildern identifiziert werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
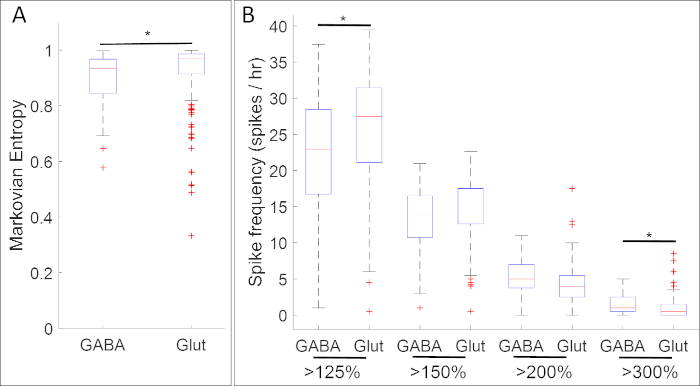
Abbildung 5: Ein Anwendungsbeispiel für diese Methode, Boxplots, die Assoziationen zwischen Kalziumaktivität und Genexpression (GABA und Glut für Gene gad1.1 bzw. slc17a7) im neuralen Plattenstadium Xenopus laevis zeigen. In Stufe 14 weisen gad1.1-postive Zellen (GABA) eine höhere Amplitude und eine regelmäßigere Kalziumaktivität auf, wie sie durch (A) Markovian entropy18 und (B) bezeichnet wird, mit den Schwellenwerten 125%, 150%, 200% und 300% des Durchschnitts der de-trendierten fluoreszierenden Intensität (Baseline)17 als slc17a7 positive Zellen (Glut). Sterne zeigen statistisch signifikante Unterschiede nach Bonferroni-korrigierten Zwei-Stichproben-Kolmogorov-Smirnov-Test (p < 0,05) und Cohens d-Statistiken zur Effektgröße (n = 5 Kulturen und >100 Zellen; * 0,2 x |d| < 0,5). Die Figur wurde neu gezeichnet und an angepasst aus dem Datensatz von Paudel et al.17. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Charakteristische Muster der Kalziumaktivität wurden in den Zellen beobachtet, aus denen das sich entwickelnde Nervensystem besteht, wobei bestimmte Arten von Aktivität mit unterschiedlichen neuroentwicklungsbedingten Prozessen verbunden sind. Ein besseres Verständnis der Mechanismen, mit denen diese informationsdichten Aktivitätsmuster in transkriptionelle Reaktionen übersetzt werden, erfordert jedoch, dass Informationen über die Kalziumaktivität und die Genexpression mit einzelliger Auflösung gesammelt werden. Während Systeme, die eine stereotypere Kalziumaktivität aufweisen, wie reife Neuronen, auf einer Massenebene vernünftigerweise untersucht werden können, werden die unregelmäßigen Muster, die das embryonale Nervensystem charakterisieren, leicht durch weniger präzise Aufnahmen maskiert.
Der in diesem Protokoll festgelegte experimentelle Rahmen kann leicht an eine Vielzahl von Zelltypen und fluoreszierenden Reportern angepasst werden. Gewebe, das praktisch jeden Zelltyp oder jede Kombination von Zelltypen enthält, kann aus einem Modellorganismus von Interesse seziert und für die einzellige Bildgebung plattiert werden. Neben der Zellidentifikation und der Isolierung der Wirkung zellautonomer Prozesse ermöglicht ein Primärzellkulturansatz dem Experimentator, Medienkomponenten nach Belieben zu definieren. Zum Beispiel wurden Experimente durchgeführt, die die Aktivität neuronaler Vorläufer in einer 2 mM Ca2+-Lösung verglichen, um zu untersuchen, ob die Beziehungen zwischen Spikefrequenz und Neurotransmitter-Phänotyp im embryonalen Rückenmark ohne den Einfluss von Zell-Zell-Wechselwirkungen rekapituliert werden können13,20.
Während dieses Protokoll den Fluoreszenzmarker Fluo4-AM nutzt, um intrazelluläre Kalziumaktivität zu erkennen, können Benutzer je nach Selektionskriterien andere kommerziell erhältliche Marker21wählen, einschließlich genetisch kodierter Calciumindikatoren. In ähnlicher Weise könnten alternative Marker verwendet werden, um dynamische Veränderungen der Konzentration eines Ionen von Interesse (einschließlich K+, Na+und Zn2+),Membranpotenzial oder zellulären pH-Wert zu überwachen. Bildeinstellungen und Bilddauer können bei Bedarf geändert werden.
Obwohl wir Kalziumaktivität und neuronalen Phänotyp als spezifische Anwendung korrelierten, ist diese Methode auch für eine Vielzahl anderer zellulärer Eigenschaften anwendbar. Beispielsweise kann die Fluoreszenz-In-situ-Hybridisierung mit Sonden gegen jedes Gen von Interesse durchgeführt werden, einschließlich des neuronalen Markers ChAT oder des Transkriptionsfaktors Engrailed, was den empfindlichen Nachweis eines anpassbaren Panels von mRNA-Arten ermöglicht. Diese Sonden können isoformspezifisch konzipiert werden und unterstützen auf Wunsch zusätzliche Zielspezifität. Double FISH kann mit Sonden durchgeführt werden, die zu mehreren zwei verschiedenen Fluorophoren konjugiert sind, was die gleichzeitige Beurteilung der Expression mehrerer Gene ermöglicht. Die zusätzlichen Washes, die für diese Art von Experiment erforderlich sind, sind jedoch mit einer erhöhten Wahrscheinlichkeit von Zellverlust oder -bewegung verbunden und erfordern Erfahrung und Delikatesse, um erfolgreich durchgeführt zu werden.
Ungeachtet aller experimentierfreudigen Änderungen an diesem Protokoll gibt es mehrere wichtige Schritte, die sorgfältige Aufmerksamkeit erfordern. Die Zerlegungen sollten mit Vorsicht durchgeführt werden, um alle kontaminierenden Gewebe oder Zellpopulationen zu entfernen; Da die räumliche Musterung verloren geht, wenn die Explanten getrennt werden, werden alle verbleibenden Zellen aus benachbarten Geweben mit den von Interesse interessierten Zellen durchsetzt und nicht von ihnen unterschieden. Nach der Plattiertwerden der Zellen sollten Proben so schonend wie möglich behandelt werden, um zu verhindern, dass Zellen sich entfernen. Am wichtigsten ist, dass dies bedeutet, dass alle Lösungsänderungen langsam und sorgfältig durchgeführt werden sollten, wobei die Pipette am Rand der Platte platziert wird, wenn die Lösung entfernt und hinzugefügt wird. Dadurch wird sichergestellt, dass Zellen sowohl in Kalzium- als auch in FISH-Bildern zuverlässig identifiziert werden können. Wenn Zellen während der Verarbeitung gestört werden, kann es unmöglich sein, einige oder alle der entsprechenden Zellen zwischen den beiden Bildern zu identifizieren. Wir raten bei diesen Zuordnungen zu einer Vorsicht, so dass für die weitere Analyse nur eindeutig entsprechende Zellen verwendet werden.
Je nach der angesprochenen biologischen Frage können verschiedene Analyseansätze angebracht sein. Die Calciumaktivität von Zeitreihen kann auf verschiedene Weise verarbeitet und quantifiziert werden, wobei die Experimentierflexibilität bei der Auswahl von De-Trending-Parametern, Analysemetriken und Analyseparametern (z. B. der % des Basisschwellenwerts, der zur Definition einer Kalziumspitze verwendet wird) verwendet wird. Korrelationen zwischen Dercalciumaktivität und dem Grad der Genexpression können durch die Analyse der Genexpression als absoluter oder relativer Fluoreszenzwert aus dem FISH-Bild gezogen werden. Alternativ können Korrelationen zwischen Kalziumaktivität und Genexpression (Präsenz/Abwesenheit) gezogen werden, indem ein Fluoreszenzschwellenwert für das positive Genexpressionssignal definiert und einzelnen Zellen "Ja"- oder "Nein"-Identifikatoren zugewiesen werden. Insgesamt bietet dieses experimentelle Schema eine unglaublich flexible Pipeline für die Erfassung und vorläufige Analyse von Zeitreihendaten in Verbindung mit zellübereinstimmenden Genexpressionsdaten. Solche Experimente werden entscheidend für ein besseres Verständnis der komplexen Beziehungen zwischen zellulärer Dynamik und transkriptionellen Veränderungen sein, wie die Identifizierung von Kalziumaktivitätsmustern zeigt, die für hemmend-fette und exzitatorisch fette neuronale Vorläufer in embryonalen Xenopus laevischarakteristisch sind.
Offenlegungen
Es wurden keine Interessenkonflikte angemeldet.
Danksagungen
Wir danken Wendy Herbst und Lindsay Schleifer für ihren Beitrag zur Entwicklung dieser Protokolle. Diese Arbeit wurde durch Zuschüsse der National Institutes of Health (1R15NS067566-01, 1R15HD077624-01 und 1R15HD096415-01) an MSS unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| For Animal Husbandry & Cell Culture | |||
| CHORULON (chorionic gonodotropin) | Merck Animal Health | ||
| Gentamycin sulfate salt | Millipore Sigma | G1264 | |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fisher Scientific | 15140122 | |
| Pyrex petri dishes, 100 mm x 20 mm | Millipore Sigma | CLS3160102 | |
| Corning Falcon Easy-Grip Tissue Culture Dishes, 35mm | Fisher Scientific | 08-772A | |
| Corning Falcon Easy-Grip Tissue Culture Dishes, 60mm | Fisher Scientific | 08-772F | |
| Falcon Standard Tissue Culture Dishes | Fisher Scientific | 08-772E | |
| Thermo Scientifc Nunc Cell Culture / Petri Dishes, 35x10mm Dish, Nunclon Delta | Fisher Scientific | 12-565-90 | |
| Fisherbrand Standard Disposable Transfer Pipettes, Nongraduated; Length: 5.875 in.; Capacity: 7.7 mL | Fisher Scientific | 13-711-7M | |
| Ethyl 3-aminobenzoate methanesulfonate | Millipore Sigma | E10521 | |
| Collagenase B | Millipore Sigma | 11088807001 | |
| Dumont #55 Forceps, Dumostar | Fine Science Tools | 11295-51 | |
| Dumont #5 Forceps, Dumostar | Fine Science Tools | 11295-00 | |
| Cellattice Micro-Ruled Cell Culture Surface | Nexcelom Bioscience | CLS5-25D-050 | |
| For Calcium Imaging | |||
| Fluo-4, AM, cell permeant | Thermo Fisher Scientific | F14201 | |
| Pluronic F-127, 0.2 µm filtered (10% Solution in Water) | Thermo Fisher Scientific | P6866 | |
| For RNA Probe Generation | |||
| PureYield Plasmid Miniprep System | Promega | A1222 | |
| rATP | Promega | P1132 | |
| rCTP | Promega | P1142 | |
| rGTP | Promega | P1152 | |
| rUTP | Promega | P1162 | |
| Digoxigenin-11-UTP | Millipore Sigma | 3359247910 | |
| Rnase Inhibitor | Thermo Fisher Scientific | N8080119 | |
| T3 RNA Polymerase | Promega | P2083 | |
| T7 RNA Polymerase | Promega | P2075 | |
| SP6 RNA Polymerase | Promega | P1085 | |
| RQ1 Rnase-Free Dnase | Promega | M6101 | |
| LiCl Precipitation Solution (7.5 M) | Thermo Fisher Scientific | AM9480 | |
| For Fluorescence In Situ Hybridization | |||
| Acetic Anhydride | Thermo Fisher Scientific | 320102 | |
| Blocking Reagent | Millipore Sigma | 11096176001 | |
| Anti-Digoxigenin-POD, Fab fragments | Millipore Sigma | 11207733910 | |
| Cy3 Mono-Reactive NHS Ester | Millipore Sigma | GEPA13105 | |
| Solution Components | |||
| Calcium chloride, 96% extra pure, powder, anhydrous, ACROS Organixs | Fisher Scientific | AC349610 | |
| Calcium chloride dihydrate | Millipore Sigma | C3306 | |
| CHAPS hydrate | Millipore Sigma | C3023 | |
| Denhardt's Solution (50X) | Thermo Fisher Scientific | 750018 | |
| DTT, Molecular Grade (DL-Dithiothreitol) | Promega | P1171 | |
| Ethylenediaminetetraacetic Acid, Disodium Salt Dihydrate | Fisher Scientific | S311 | |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N',N'-tetraacetic acid | Millipore Sigma | E3889 | |
| Formamide (Deionized) | Thermo Fisher Scientific | AM9342 | |
| Herparin sodium salt from porcine intestinal mucosa | Millipore Sigma | H3393 | |
| HEPES (Ultra Pure) | Thermo Fisher Scientific | 11344041 | |
| Hydrogen peroxide solution | Millipore Sigma | H1109 | |
| L-Cysteine | Millipore Sigma | 168149 | |
| Magnesium chloride, pure, ACROS Organics | Fisher Scientific | AC223211000 | |
| Magnesium sulfate, 97% pure, ACROS Organixs, anhydrous | Fisher Scientific | AC413480050 | |
| Maleic Acid, 99%, ACROS Organics | Fisher Scientific | ACS125231000 | |
| MOPS (Fine White Crystals/Molecular Biology), Fisher BioReagents | Fisher Scientific | BP308 | |
| Potassium chloride | Millipore Sigma | P9541 | |
| Ribonucleic acid from torula yeast, Type IX | Millipore Sigma | R3629 | |
| Sodium chloride | Millipore Sigma | S7653 | |
| Triethanolamine | Millipore Sigma | 90279 | |
| Tris | Millipore Sigma | GE17-1321-01 | |
| TWEEN 20 | Millipore Sigma | P9416 | |
| Equipment | |||
| Laminar Flow Hood | model of choice | ||
| Dissecting Microscope | model of choice | ||
| Inverted Fluorescence Microscope | Nikon | TE200 | |
| NIS-Elements Imaging Software | Nikon | ||
| Shaking Incubator | model of choice | ||
| Refrigerated Centrifuge | model of choice | ||
| Miscellaneous | |||
| Corning bottle-top vaccum filter system, 0.22 μm pore, 500 mL bottle capacity | Millipore Sigma | CLS430769 | |
| Falcon 50mL Conical Centrifuge Tubes | Fisher Scientific | 14-432-22 |
Referenzen
- Humeau, J., et al. Calcium signaling and cell cycle: Progression or death. Cell Calcium. 70, 3-15 (2017).
- Kim, J. M., Lee, M., Kim, N., Heo, W. D. Optogenetic toolkit reveals the role of Ca2+ sparklets in coordinated cell migration. PNAS. 112 (21), 5951-5957 (2016).
- Orrenius, S., Zhivotovsky, B., Nicotera, P. Regulation of cell death: the calcium-apoptosis link. Nature Reviews Molecular Cell Biology. 4 (7), 552-565 (2003).
- Pham, K., et al. Ca2+ and Mg2+ module conformational dynamics and stability of downstream regulatory element antagonist modulator. Protein Science. 24 (5), 741-751 (2015).
- Smedler, E., Uhlén, P. Frequency decoding of calcium oscillations. Biochimica et Biophysica Acta. 1840 (3), 964-969 (2014).
- Moreau, M., Néant, I., Webb, S. E., Miller, A. L., Riou, J. F., Leclerc, C. Ca(2+) coding and decoding strategies for the specification of neural and renal precursor cells during development. Cell Calcium. 59 (2-3), 75-83 (2016).
- Tomida, T., Hirose, K., Takizawa, A., Shibasaki, F., Iino, M. NFAT functions as a working memory of Ca2+ signals in decoding Ca2+ oscillation. EMBO. 22 (15), 3825-3832 (2003).
- Hannanta-Anan, P., Chow, B. Y. Optogenetic Control of Calcium Oscillation Waveform Defines NFAT as an Integrator of Calcium Load. Cell Systems. 2 (4), 283-288 (2016).
- Li, L., Stefan, M. I. Le Novère N. Calcium input frequency, duration and amplitude differentially module the relative activation of calcineurin and CaMKII. PLoS One. 7 (9), 43810(2012).
- Romano, D. R., Pharris, M. C., Patel, N. M., Kinzer-Ursem, T. L. Competitive tuning: Competition's role in setting the frequency-dependence of Ca2+-dependent proteins. PLoS Computational Biology. 13 (11), 1005820(2017).
- Pharris, M. C., Patel, N. M., Kinzer-Ursen, T. L. Competitive Tuning Among Ca2+/Calmodulin-Dependent Proteins: Analysis of in silico Model Robustness and Parameter Variability. Cellular and Molecular Bioengineering. 11 (5), 353-365 (2018).
- Gu, X., Olson, E. C., Spitzer, N. C. Spontaneous neuronal calcium spikes during early differentiation. Journal of Neuroscience. 14 (11), 6325-6335 (1994).
- Borodinsky, L. N., Root, C. M., Cronin, J. A., Sann, S. B., Gu, X., Spitzer, N. C. Activity-dependent homeostatic specification of transmitter expression in embryonic neurons. Nature. 429 (6991), 523-530 (2004).
- Ciccolini, F., Collins, T. J., Sudhoelter, J., Lipp, P., Berridge, M. J., Bootman, M. D. Local and Global Spontaneous Calcium Events Regulate Neurite Outgrowth and Onset of GABAergic Phenotype during Neural Precursor Differentiation. Journal of Neuroscience. 23 (1), 103-111 (2003).
- Weissman, T. A., Riquelme, P. A., Ivic, L., Flint, A. C., Kriegstein, A. R. Calcium Waves Propagate through Radial Glial Cells and Modulate Proliferation in the Developing Neocortex. Neuron. 43 (5), 647-661 (2004).
- Nieuewkoop, P. D., Faber, J. The stages of Xenopus embryonic development. Normal Table of Xenopus laevis. , North Holland Publishing Company. (1994).
- Paudel, S., et al. Calcium Activity Dynamics Correlate with Neuronal Phenotype at a Single Cell Level and in a Threshold-Dependent Manner. International Journal of Molecular Science. 20 (8), 1880(2019).
- Marken, J. P., et al. A Markovian Entropy Measure for the Analysis of Calcium Activity Time Series. PLoS One. 11 (12), 0168342(2016).
- Eilers, P. H. C., Boelens, H. F. M. Baseline Correction with Asymmetric Least Squares Smoothing. Leiden University Medical Centre Report. , (2005).
- Guemez-Gamboa, A., et al. Non-cell-autonomous mechanism of activity-dependent neurotransmitter switching. Neuron. 82 (5), 1004-1016 (2014).
- Paredes, R., Madelaine,, et al. Chemical calcium indicators. Methods. 46 (3), 143-151 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten