Method Article
ゼノプス・ラエビスにおける神経前駆体特性評価のための蛍光カルシウムイメージングとその後のその後の位置ハイブリダイゼーション
要約
蛍光カルシウムイメージングとin situハイブリダイゼーションを組み合わせた2部構成のプロトコルを提示し、実験者がカルシウム活性のパターンを単一細胞レベルの遺伝子発現プロファイルと相関させることが可能です。
要約
自然細胞内カルシウム活性は、様々な細胞型で観察でき、様々な生理学的過程において重要な役割を果たすと提案されている。特に、胚発生時のカルシウム活性パターンの適切な調節は、適切な神経管閉鎖、シナプト形成、神経伝達物質の表現型の仕様を含む脊椎動物の神経発達の多くの側面に必要である。カルシウム活性パターンが周波数と振幅の両方で異なる可能性があるという観察は、これらのフラックスが下流のエフェクターにコードされた信号を伝達し、遺伝子発現を調節する説得力のあるメカニズムを示唆しているが、既存の人口レベルのアプローチは、この可能性をさらに探求するために必要な精度を欠いている。さらに、これらのアプローチは、細胞間接触の非存在下における神経細胞決定の状態をアッセイする能力を排除することによって、細胞間相互作用の役割の研究を制限する。そこで、解解神経系外在物の経時・経過カルシウムイメージングと、その場でのハイブリダイゼーションにおける蛍光を組み合わせ、カルシウム活性パターンと分子の明確な相関を可能にする実験ワークフローを確立しました。単一細胞レベルの表現型。我々は、神経細胞と神経前駆細胞の分化に関連する特定のカルシウム活性パターンをそれぞれ区別し、特徴付けるために、このアプローチを使用することができました。しかし、この記事で説明した実験フレームワークは、時系列の活動プロファイルと、目的の遺伝子または遺伝子の発現との間の相関関係を調査するために容易に適応することができる。
概要
遊離細胞質カルシウムは、細胞増殖および移動からアポトーシスおよびオートファジー1、2、3に至るまで、様々な生物学的プロセスに重要である。これらの経路内では、カルシウムは、カルシウム結合ドメインと相互作用してタンパク質の活性および相互作用を調節する立体構造変化を誘導することによって、遺伝子発現に下流の効果を及ぼし得る。例えば、下流制御要素アンタゴニスト変調器(DREAM)として知られる神経カルタカルシウムセンサーは、カルシウムに結合すると展開された中間立体構造に保持され、そのタンパク質およびDNA標的4と相互作用するのを防ぐ。しかし、単純なシグナル伝達分子として機能する以外に、細胞内カルシウム過渡の動的性質により、これらの活性パターンは、より複雑な振幅または周波数ベースの信号5、6をコードすることを可能にする。活性化T細胞の核因子の転写因子の核転位は、高周波カルシウム振動によって増強されるが、低周波振動によって阻害される7。説得力のある, 最近の研究は、NFATが実際に累積カルシウム暴露に応答するかもしれないことを示唆しています 8 .カルシニューリンおよびCa2+/カルモジュリン依存性プロテインキナーゼII(CaMKII)は、また、特定の周波数、持続時間、または振幅9のカルシウム過渡に対して明確な応答を示す。調節の複雑さの追加レベルを追加するために、計算モデルは、多くの下流のカルシウム結合タンパク質が、結合競合者10,11の存在または不在に応じて多かれ少なかれ周波数依存性になることを示唆している。
発達中の神経系の中で、カルシウム活性行動の2つの主要なクラスが定義され、特定の生物学的プロセスに関連付けられている。カルシウム流入は、個々の細胞内で発生した場合に「スパイク」に分類され、5秒以内にベースラインの400%近いピーク強度に達し、二重指数比12を示す。このタイプのシグナルは、主に神経伝達物質の表現型仕様13と関連している。これに対して、「波」は、細胞の細胞内カルシウム濃度が30秒以上の期間にわたってベースラインの〜200%に上昇し、その後、数分間12にわたって減衰する、より遅く、より極端でないカルシウム過渡気として定義される。これらのシグナルは、多くの場合、複数の隣接する細胞に伝播し、その存在は、神経突起の成長と細胞増殖に関連付けられている14,15.しかし、これら2つのクラスは特徴的な運動プロファイルに基づいて定義されていますが、これらのパターンのどの特性が実際に細胞によって検出され、下流のエフェクターによって翻訳されているかは正確には不明です。
細胞内カルシウム振動と遺伝子発現の関係を理解することは、神経系の適切な発達とパターン化を保証する調節メカニズムの1つに重要な洞察を提供するであろう。この終わりのために、胚性脊髄の研究は、発達中のカルシウムスパイク活性の増加がより高いレベルの抑制性ニューロンに関連していることを実証しているが、カルシウムスパイク活性の低下は興奮性ニューロンのより高いレベルに関連している13。しかし、これらの集団レベルのアッセイは、単細胞レベルでの遺伝子発現とカルシウム活性を関連付けるために使用されていません。
単一セルのレベルでこれらの質問に近づくと、前の作業よりもいくつかの明確な利点があります。一つには、多くの細胞におけるカルシウム活性および遺伝子発現を個別に評価する能力により、バルクレベル測定によって難読化されることなく、明確な活性パターンの完全なレパートリーを観察することができます。さらに、単一細胞の初次培養におけるこれらの関係を研究することは、カルシウム活性と遺伝子発現との間の細胞自律的なリンクが維持され、細胞間通信を必要とする相互作用が失われることを意味する。したがって、このアプローチは、これらの細胞自律的なメカニズムを単独で研究することを可能にする。しかし、それはまた、非細胞自律性カルシウム活性の役割を解明し、尋問することを可能にする。例えば、細胞は神経プレート段階で胚から解剖され、兄弟コントロールが神経管段階に達するまで培養され、次に神経管段階の胚から新たに解剖された細胞と比較することができる。これにより、重要な発生期間にわたって細胞間通信を保持していた細胞と、細胞間通信が廃止された細胞とを直接比較することができます。
これまでの実験手法の限界に取り組むため、個々の神経前駆細胞におけるカルシウム活性と遺伝子発現の評価を可能にするプロトコルを開発し、その後の分化プログラムとの特定の活性パターンの相関を促進する。神経組織は、神経発生の様々な段階でゼノプス・レービスから解剖され、単一細胞に解離され、蛍光カルシウム指標の存在下で共焦点顕微鏡を介して画像化された。生細胞イメージングに続いて、サンプルを固定し、蛍光でハイブリダイゼーション(FISH)を介して、目的の遺伝子またはアイソフォームの発現を検出した。重要なことに、個々の細胞は、両方のイメージング実験にわたって追跡することができ、細胞のカルシウム活性プロファイルとその遺伝子発現レベルは互いに関連付けることができることを意味する(図1)。ここで報告されるプロトコルは、Xenopus laevisの胚性神経発達全体におけるカルシウム活性パターンと遺伝子発現の関係を調査することを目的としています。しかし、より広範な実験フレームワーク(単一細胞時間経過イメージングとFISHと画像の共登録)は、事実上任意の細胞タイプ、蛍光レポーター、および目的の遺伝子に改変および適用することができます。
プロトコル
動物に関するすべての作業は、ウィリアム・アンド・メアリー大学の機関動物管理使用委員会(IACUC)によって承認されたプロトコルに従って行われました。
1. 動物のケアと胚の取り扱い
- ヒト絨毛性ゴナドトロピン(HCG)の皮下注射を成人ゼノプス・レービスの背側リンパ嚢に投与することによって、女性は600U、男性は400Uの用量で自然交配を誘発する。
- 注射後、少なくとも1匹の雄と1匹の雌のカエルを室温の保持タンクに一晩入れる。産卵は、通常、HCG投与後9〜12時間開始する。
- 胚を収集します。2~4分の2%システイン(pH 8.0)でやさしく洗ってデゼリー。
- 0.1xマークの修飾リンガー溶液(MMR)(100mM NaCl、2 mM KCl、1mM MgSO4、2mM CaCl2、5mM HEPES、pHを7.4〜7.6に調整)で胚3倍をすすすります。
- 胚を0.1x MMRと50 μg/mLゲンタマイシンを含む100mmガラスペトリ皿に移します。プレートあたり50〜100個の胚の密度が適切である。
- 14 °C ~ 22°C で料理をインキュベートし、胚が目的の発達段階に達するまで発生できるようにします。定期的に未受精細胞や壊死またはプラスチックトランスファーピペットを持つ異常に発達している胚を除去します。
注: 一貫性を確保するために、開発ステージングは、Nieuwkoop および Faber16によって定義された形態学的基準に従って実行されます。対象となる段階は、実験フォーカスによって異なります。例えば、主要な神経発達のランドマークは、ステージ14(神経節の発症)、ステージ18(神経管閉鎖の発症)、およびステージ22(テールバッド伸びの発症)に関連しています。
2. 胚解剖と試料調製
- 溶液調製
- 116 mM NaCl、0.67 mM KCl、2 mM CaCl2·2H2O、1.31 mM MgSO4、および 4.6 mM トリスを含む 2 mM Ca2 +溶液を準備します。100 mL溶液ごとに、ペニシリン/ストレプトマイシン1mL(10,000 U/mLペニシリン、10,000 μg/mLストレプトマイシン)を加えます。pHを7.8に調整し、フィルター滅菌します。
- 116 mM NaCl、0.67 mM KCl、4.6 mM トリス、0.4 mM EDTA を組み合わせて、カルシウムとマグネシウムフリー(CMF)溶液を調製します。pHを7.8に調整し、オートクレーブを調整して滅菌します。100 mL溶液ごとに、ペニシリン/ストレプトマイシン1mL(10,000 U/mLペニシリン、10,000 μg/mLストレプトマイシン)を加えます。pHを7.8に調整し、フィルター滅菌します。
- 解剖・撮像用プレートの調製
- UV滅菌2つの35ミリメートルプラスチックペトリ皿と1つの35ミリメートルの細胞培養皿(材料のテーブルを参照)。
- 層流フードで作業しながら、2 mM Ca2+溶液の10 mLを含む2つの50 mLプラスチック円錐形チューブを準備します。
- 層流フードで作業を続け、2 mL の 2 mL の Ca2+溶液を 1 つの 35 mm プラスチック ペトリ皿に、2 mL の 2 mL の Ca2 +溶液を 1 つの 35 mm 細胞培養皿に、2 mL の CMF 溶液を 1 つの 35 mm プラスチック ペトリ皿に追加します。
- 層流フードの外側では、2つの100ミリメートルプラスチックペトリ皿と1つの35ミリメートルプラスチックペトリ皿をゲンタマイシン(50 μg / mL)で0.1x MMRで満たします。60mmのプラスチックペトリ皿に70%エタノールを充填します。
- 解解の直前に、Ca2+溶液を含む50mLチューブの1つに0.01gのコラゲーゼBを加えます。よく混ぜ、新鮮な60ミリメートルプラスチックペトリ皿に溶液を転送します。
- 解剖顕微鏡の助けを借りて、所望の発生段階の胚を同定する。滅菌移動ピペットを使用して、ステップ2.2.4で調製した0.1x MMR+ゲンタマイシンを含む100mmプレートの1つに少なくとも6つの適切な胚を移送する。これは、保持プレートとして機能します。
- 滅菌トランスファーピペットを使用して、1つの胚を0.1x MMR +ゲンタマイシンを含む第2の100mmプレートに移します。これは、解剖プレートとして機能します。
- 移動できる年齢の胚(およそ段階23以上)を解剖する場合は、0.1%3-アミノ安息香酸エチルエステルを含む皿にヘタマイシン(50μg/mL)で希釈して、解剖前に各胚を麻酔します。胚が固定化されたら、ゲンタマイシン(50 μg/mL)で0.1x MMRを含む皿に戻し、解剖を続けます。
- 慎重に胚を囲むビテリン膜を取り除きます。これは、膜を把握するために一対の微細な鉗子を使用しながら、胚を安定させるために鈍い鉗子のペアを使用することによって最も容易に行うことができる。細かい鉗子で慎重に引っ張って、ビテリン膜を剥離します。
- 微細な鉗子を使用して、胚の後側と腹側の領域を分離する。これは、鉗子を使用して胚を前後軸に沿って「ピンチ」し、半分に切断することによって行うことができます。滅菌トランスファーピペットを用いて、ステップ2.2.5で調製したコラゲターゼ溶液を用いて、60mmプレートに転写する。腹側部分を捨てる。
- 後方の植物をコラゲターゼ溶液中で室温で1~2分間インキュベートします。解剖プレートにそっと移します。
- 外胚葉の推定神経組織から残留した内皮および中皮汚染をすべて慎重に除去することによって、解剖を完了する。ステージ22以上の胚の場合、神経管も取り除いて廃棄する必要があります。
注:解剖中に必要な場合は、ステップ2.2.4で調製した70%エタノールの皿を使用して鉗子をクリアまたは再殺菌することができます。 - 解剖が完了したら、ステップ2.2.3で調製したCa2+溶液の35mmプレートに外植物を穏やかに移します。
- 4 つの外植が収集されるまで、手順 2.4~ 2.9 を繰り返します。
- P1000マイクロピペットを使用して、4つの外植植物すべてをCMF溶液を含む35mmプレートに移し、外植植物と空気水界面との接触を避けるように注意してください。すべての外植物がプレートの中央に集まるように皿をそっと渦巻きます。
- 室温で1時間インキュベートし、外植物が解き合えるようにする。
注: 解離を支援するために、0.025% ~ 0.01% のトリプシンを CMF ソリューションに追加できます。これは、古い胚の効率的な解離に必要である可能性があります (ステージ 22 以上). - この時点で、少なくとも2つの適切にステージングされた胚は、保持プレート上に残るべきである。ステップ2.2.4で調製したゲンタマイシンで0.1x MMRで満たされた新鮮な皿にこれらの胚を移し、彼らは外植皿に合わせて覆われた料理で邪魔されずに開発することができます。これらの胚は兄弟コントロールとして機能します。
- superglue を使用して、ステップ 2.2.3 で準備した 35 mm セル培養ディッシュの底部に、マイクロルール付きカバースリップ (材料の表を参照) を取り付けます。
注:カバースリップの端にスーパーグルーの小さなダブを置き、細胞培養皿の下側にしっかりと押し付けます。位置マーキングは、接着剤がグリッドに接触する場所で隠されるため、カバースリップの中央グリッド部分を接着剤から解放することが重要です。 - 外植植物が1時間解絶した後、P100マイクロピペットを使用して細胞培養皿に移します。皿の格子状部分にできるだけ多くの細胞をプレートするために、皿の表面に近い浅い角度でピペットを保持し、内側に向かってグリッドの隅にピペット先端を配置し、グリッドされたarを横切ってしっかりと細胞懸濁液を排出します理想的には、細胞は密な密なクラスターに沈着します。
- 細胞がプレートに付着できるように室温で1時間インキュベートする。このインキュベーションが始まると、兄弟コントロール胚の発達段階を決定し、記録する。
- 5 μL 1 mM Fluo-4 AM (材料表を参照) を 2 μL の 10% プルロン酸 F-127 酸と組み合わせます。
注:Fluo-4 AMは光に敏感で、常に光安全またはホイルで覆われたチューブに保管する必要があります。 - インキュベーションが完了したら、サンプルディッシュを暗室または他の光で保護された場所に移動します。マイクロピペットを使用して、皿の端から100μLの溶液を取り除きます。Fluo-4 AM/Pluronic F-127酸のアリコートにこの溶液を加え、ピペットを上下に混ぜ、フルボリュームをサンプル皿に戻します。そっと渦巻き混ぜる。
- プレートをアルミホイルで覆い、室温で1時間インキュベートします。このインキュベーションが始まると、兄弟コントロール胚の発達段階を決定し、記録する。
- インキュベーションの最後に、2 mM Ca2+の残りの円錐形チューブを使用して、皿から1 mLの溶液を取り除き、2)3 mLの新鮮な溶液を加え、3 mLの新鮮な溶液を加え、3mLの新鮮な溶液を加え、新鮮な溶液を3mL加えます。
3. カルシウムイメージング
注:カルシウムイメージングは、反転共焦点顕微鏡(材料表)を用いて行った。
- サンプルプレートを顕微鏡ステージに置き、周囲光の露出から保護するように注意してください。プレートが固定されたら、マーカーを使用してプレートの前面の点にラベルを付け、その後のイメージングで同じ視野を見つけることができます。
- 顕微鏡でサンプルを見つけ出し(最初に10Xで、次に20倍の倍率で)、画像撮影に適した視野を選択します。理想的な視野はセル密度が高いが、セルが束れているか、個別に区別しにくいほど密度が高くない。
- グリッドに定めのカバースリップが見えるように顕微鏡の焦点を調整します。カバースリップに記されている数字は、特定のグリッド軌跡の一意の識別子として機能し、追加のイメージングのために同じ視野を見つけるために使用できます。最初に選択したビューフィールドが数字と重ならない場合は、識別可能な数値がフレーム内に入るまで、変更を取り入れ換えます。
- グリッドに準拠したカバースリップに焦点を当てて、選択した視野の明視野画像を撮ります。
- フォーカスの設定を調整し、セルにフォーカスを合わせて選択した視野の明視野画像を取ります。
- 細胞層に焦点を当てて、488 nmレーザーでサンプルを照らす。HV とオフセット値は、各実験に対して最適化して、FITC チャネルで蛍光のダイナミック レンジが検出されるようにすることができます。
- 2 時間のイメージの場合は、スキャン時間が 3.93 s、間隔が 8 秒の 901 フレームを記録するようにイメージング構成を変更して、イメージを取得します。
- イメージングが完了したら、顕微鏡段階からプレートを取り出します。プレートから1 mLの溶液を取り出し、2mLのMEMFA(200mM MOPS、2 mM EGTA、2 mM MgSO4の7.4%ホルムアルデヒド)に交換します。
- プレートを室温で2時間、または4°Cで一晩インキュベートする。このインキュベーションが始まると、兄弟コントロール胚の発達段階を決定し、記録する。
- 固定が完了したら、プレートからすべての溶液を取り出し、1x PBSの2 mLに交換します。さらに処理するために、プレートを4°Cに保管してください。
4. 遺伝子発現解析:プローブ合成
- 後述のとおり、in situハイブリダイゼーション用のアンチセンスRNAプローブを生成する。さらに、陰性制御として使用する同一遺伝子に対するセンスプローブを生成する。
- プローブテンプレート配列を含むプラスミドDNAを精製するために、鋳型プラスミドを含む細菌グリセロールストックを用いてLBブロスの150mLを接種する。37°Cで一晩または培養が濁るまでインキュベートする。
- お好みの方法を用いて細菌培養からプラスミドDNAを精製します。
注:我々はプラスミドDNAの高い収率を得るためにマクナリー・ナーゲルミディプレップキットを使用しています。 - プラスミドに期待されるインサートが含まれていることを確認するには、制限消化を行い、アガロースゲル上の製品を分析します。未切断のプラスミドは、アガロースゲル上で分析してゲノムDNA汚染をチェックすることもできます。
- テンプレートDNAを線形化するには、20 μgのプラスミドDNA、2μLの適切な制限酵素、および1xの適切なバッファーを含む100 μL制限消化反応を設定します。37°Cで少なくとも2時間インキュベートする。
- フェノール/クロロホルム抽出を行い、クロロホルム抽出を行うことで、リニアライズDNAを抽出します。
- 100%エタノールでDNAを沈殿させる。これは、2巻の冷エタノールをサンプルに加え、固化するまで-80°Cでインキュベートすることで迅速に行うことができます(15〜30分)。
- 冷蔵遠心分離機を使用して、12,000 x g/4 °Cで20分間回転させてDNAをペレット化します。
- 上清を取り出し、200μLの70%エタノールでペレットを洗います。12,000 x g/4 °Cで5分間スピンします。
- 上清とエアドライペレットを約5分間取り除き、20μLの1x TEで再サスペンドし、さらに使用するまで4°Cで保管します。
- アンチセンスRNAプローブを合成して精製するには、10 mM rCTPの15 μL、10 mM rGTPの15 μL、10 mM rATPの15 μL、10mM rATPの9.75 μL、10mMの1UTM-11材料の5.25μLを組み合わせて、2.5mM rNTPミックスを作成します(UTPの5.25μL)。
- ステップ 4.2-4.10 から 4 μg の線形化されたテンプレート DNA を含む 50 μL in vitro 転写反応をセットアップします。 ステップ4.11から2.5 mM rNTPミックスの15μL、5x転写バッファーの10μL、0.1M DTTの5μL、RNAse阻害剤(20 U/μL)の0.5μL、および1.5μLの適切なRNAポリメラーゼ(T3、T7、またはSP6)の。37 °Cで1時間インキュベート。
- 反応に1.5 μlのRNAポリメラーゼを加え、さらに1時間37°Cに戻します。
- 反応にRQ1 DNAAseを1μL加え、37°Cで10分間インキュベートしてDNAテンプレートを分解します。
- サンプルに30μLの7.5 M LiCl溶液を加えます。ピペットを-20°Cで少なくとも1時間混合してインキュベートする。
- 冷蔵遠心分離機を使用して、サンプルを14,000 x g/4 °Cで25分回転させます。
- 上清を取り出し、500 μLの70%エタノールでペレットをすすいでください。14,000 x g/4 °Cで5分間スピンします。
- 約5分間の上清とエアドライペレットを取り除き、ヌクレアーゼを含まない水を20μLで再栓します。
- ハイブリダイゼーションバッファーでサンプルを10 ng/μLの濃度に希釈して、10倍のプローブストックで作成します(50%ホルムアミド、 5x SSC(生理塩水ナトリウムクエン酸;750mM NaCl、75mMクエン酸ナトリウム、pH 7.0)、1mg/mLトルラRNA、0.1%トウィーン20、1xデンハルト溶液、0.1%CHAPS、10mM EDTA、および100μg/mLヘリン。さらに使用するまで-20°Cで保管してください。
5. 遺伝子発現解析:蛍光インシチューハイブリダイゼーション
注:すべてのワッシュは、滅菌、個別にラップされたトランスファーピペットを使用して約1 mLの溶液で行う必要があります。溶液を取り外したり添加したりする際には、ピペットをプレートの端に配置し、細胞がプレート表面から外れ、失われないように、できるだけ穏やかに行う必要があります。
- プレートから1x PBSを取り除きます(ステップ3.10から)。新鮮な1x PBSに置き換え、室温で5分間インキュベートします。
- 25 mL 0.1 メタトリエタノールアミン(pH 8.0)と62.5 μLの無水酢酸を組み合わせます。よく混ぜます。この溶液で10分間皿を洗います。
- プレートを1x SSCで5分間洗います。
- プレートを0.02 M HClで10分間洗浄し、細胞を透過させます。
- 1x PBSで2xをそれぞれ5分間洗います。
- 溶液を取り除き、ハイブリダイゼーションバッファーの1 mLを追加します(50%ホルムアミド、 5x SSC(750 mM NaCl、75 mMクエン酸ナトリウム、pH 7.0)、1 mg/mLトルラRNA、0.1%トゥイーン-20、1xデンハルト溶液、0.1%CHAPS、10 mM EDTA、および100 μg/mLヘパリン)60°Cで少なくとも6時間振とうたってインキュベートする。
- ハイブリダイゼーションバッファーを取り出し、750 μLの1x RNAプローブ溶液(ステップ4.19で作られた10xストックを希釈した形)に置き換えます。センスRNAプローブは陰性制御として使用することができる。
- 60 °Cで8-14時間振と共にインキュベート。
- プローブを取り外し、-20°Cで保管します。
メモ:1xプローブ希釈は廃棄される前に3回まで再利用することができます。 - プレートを0.2x SSCですすいでください。
- 新鮮な0.2x SSCで60°Cで1時間洗浄します。
- プレートを室温に移動し、5分間平衡させます。
- プレートを0.2x SSCで5分間洗います。
- プレートを1x PBTで15分間洗います。
- プレートを1x PBT(0.1%トリトン-x-100)で2%H2O2で1時間洗います。
注:このソリューションは光に敏感であるため、実験ごとに新たに作り、光から遮蔽する必要があります。プレートはまた、光からシールドするか、このインキュベーション中に箔付けする必要があります。 - プレートを1x TBST(150 mM NaCl、50 mMトリス-HCl、pH 7.5、0.1%トゥイーン-20)で15分間洗います。
- マレイン酸バッファーで2%に希釈ブロッキング試薬(100 mMマレイン酸、150 mM NaCl、pH 7.5)。この混合物の細胞を室温で少なくとも1時間ブロックする。
- ブロッキング溶液を、マレイン酸バッファー中の2%ブロッキング試薬中1:1,000希釈した抗ジゲオキシンジン-POD抗体に置き換えます。4°Cで一晩インキュベートする。
- プレートを3xで1x TBSTですすいでください。
- 連続ロッキングで1回15分以上の2mLで4倍の洗浄を行います。
- 連続ロッキングで1回の洗浄につき少なくとも10分間、1x PBTで2xを洗浄します。
- 薄いCy3-共役チラミド 1:25 1x PBT.この希釈液を750μLで5分間洗います。
注:このソリューションは非常に光に敏感であり、プレートは、信号の劣化を避けるために、実験の残りの部分のために、箔付けまたは光からシールドしておく必要があります。 - この溶液に0.3%H2O2の2.5μLを加え、連続ロッキングで室温でさらに40分間インキュベートします。
- 連続ロッキングで1回の15分以上の15分間、1x TBSTで4倍の洗浄を行います。
- 1x PBTですすい。
- 1x MEMFA(100 mM MOPS、1 mM EGTA、1 mM MgSO4の 3.7% ホルムアルデヒド)で 1 時間、室温で 1 時間インキュベートして細胞を固定します。
- ソリューションを取り外し、1x PBS に置き換えます。プレートを4°Cのホイル容器に入れ、さらに処理するまで保管します。
6. 細胞のイメージング
注:画像化は、逆焦点顕微鏡を用いて行った。
- サンプルプレートを顕微鏡ステージに置き、ステップ3.1で作ったマークをステージの前に揃えます。
- グリッドに並んだカバースリップが表示されるように画像をフォーカスし、手順 3.4 で撮影したグリッド イメージを基準として、カルシウム画像でキャプチャされた視野に合わせて視野を調整します(セクション 3)。
- グリッドとセルの両方の明視野画像を取得します。
- 595 nm TRITC レーザーでサンプルを照らします。ゲイン値を調整して、負の制御セルからの画像を使用して、背景から信号を適切に区別し、静止画を取得します。
注:理想的には、バックグラウンド蛍光レベルは、非標的性センスRNAプローブと並行して処理された陰性制御プレートに基づいて決定されます。ゲイン設定は、このプレートが完全に黒く表示されるように調整され(背景レベルに対応)、その実験バッチからイメージされた他のプレートに対して一定に保たれます。
7. データ処理
注:データ処理はニコンエレメントソフトウェアを使用して行われました。
- ステップ3.7から2hカルシウム画像を開きます。[バイナリ] > [スポット検出] > [明るいスポット] を選択して、個々のセルに対応するピクセルを識別します。FITC チャンネルが選択されていることを確認します。
- このレイヤーが生成された後、色付きの円が個々のセルに表示されます。セルの分布とサイズのパラメータを調整して、できるだけ多くのセルが認識され、一意の識別子に関連付けられます。
- [表示] →[分析コントロール] > [トラッキング オプション] に移動して、画像のすべてのフレームのセルを追跡します。トラック間の最大ギャップとして 5 フレームを設定し、600 フレーム未満のオブジェクトを削除して、[ギャップを閉じる]オプションを選択します。[バイナリを追跡] を選択して、セルトラッキングを画像に適用します。
- セルを追跡した後、個々のセルに対応していないオブジェクト (セルの束など) を手動で削除します。しかし、データ点は、カルシウム活性トレースの形態に基づくさらなる分析から除外されるべきではありません。
- [イメージ] ペインで、[オーバーレイの表示|バイナリ オブジェクト ID の表示] を選択します。イメージの最後 (フレーム 901) までスクロールし、[編集] → [ビュー スナップショットの作成 ](8 ビット RGB)> [現在のフレーム] > [OK]を選択します。これにより、各セルに関連付けられたバイナリ ID が表示されたイメージの最後のフレームのスナップショットが作成されます。このイメージを保存します。
- すべてのオブジェクトを選択し、[データを Excel にエクスポート] をクリックして時系列データをエクスポートします。出力を CSV ファイルとして保存します。
- FISH 画像を開きます。FITC チャネルの代わりに TRITC チャネルを使用して、ステップ 7.1 ~ 7.2 のようにスポット検出を最適化します。間違って割り当てられたバイナリを手動で削除し、[自動計測結果] > [計測の更新] を選択して、各セルの信号強度を計算します。手順 7.5 と 7.6 を繰り返して、イメージ スナップショットとデータ テーブルをエクスポートします。
- 列 A がFISH バイナリ ID、列 B が[カルシウム バイナリ ID]というラベルが付いたスプレッドシートを作成します。ステップ 7.5 および 7.7 でエクスポートされたイメージを開きます。FISH イメージで識別される各オブジェクト (ステップ 7.7) について、列 A にバイナリID を記録します。次に、カルシウム画像 (手順 7.5) で対応するセルを見つけ、列 B にそのバイナリ ID を記録します。
メモ:Adobe PhotoshopやGIMP画像エディタなどの写真編集プログラムを使用して、両方の画像を開き、1つの半透明にし、パートナー画像にオーバーレイして、各セルに関連付けられた2つのバイナリIDをより簡単に識別してリンクすると便利です。 - 同定された細胞ごとに、時系列カルシウムデータ(カルシウムバイナリIDに関連付け、ステップ7.6でエクスポート)とその遺伝子発現データ(FISH Binary IDに関連付けられており、ステップ7.7でエクスポート)を照合します。
注: 下流のデータ処理と分析では、これらのデータを単一のデータテーブルに照合し、スパイクカウント、フラクタル分析、およびマルコフンエントロピーを含む一連の分析技術を適用して、研究者がカルシウム活性17、18の新しいパターンを識別することを可能にする。
結果
カルシウムイメージングのために調製された解化細胞の成功例を図2Aに示すことができる。細胞は密にメッキされ、各画像から収集される情報の最大量を可能にするが、個々の細胞が互いに自信を持って区別できないほど密にメッキされていない。2hイメージング期間にわたって定義された各細胞について蛍光が検出される。実験で記録されたすべての細胞の痕跡を含む複合プロットの視覚化は、バルクまたは集団の測定がスパイク動作のより微妙なパターンをあいまいにすることができる程度を明らかにします(図2B)。個々の細胞の記録されたプロファイルが単離されると、神経前駆細胞の不規則なスパイク活動特性の例が明確に同定され得る。成熟したニューロンとは異なり、胚性ニューロン細胞は、不規則で、非常に可変的で、カルシウム活性の複雑な性質を示す(図2B)。この複雑さを定量化するために、異なるデータ解析方法の適用は、スパイクを定義する多様なパラメータを含む17,18を適用して適用された(図2C)。
アンチセンスmRNAプローブの設計と合成に成功することを含む、situハイブリダイゼーションにおける蛍光の成功は、非結合感知体RNA制御でインキュベートされたバックグラウンドコントロールに対して実験プレートを比較することによって評価することができる(図3A、B)。陽性プローブ制御は、検出可能なレベルで標的mRNAを発現することが知られている細胞型を処理することによっても行うことができる。
カルシウムとFISHイメージングで同じ細胞を同定するには、プローブのハイブリダイゼーションと処理中に細胞がほぼ同じ位置を保持する必要があります。プレートが大まかに処理されたり、ワッシュが強引に行われたりすると、細胞はプレート表面から外れ、溶液が廃棄されたりプレート上の別の場所に堆積したりすると失われる可能性があり、画像間で一致することが不可能になります(図4A)。この中断が視野の一部のセルにのみ影響する場合でも、画像内の一部のセルを検出して割り当てることはできるかもしれません (図 4B)。しかし、FISHを慎重に行い、画像間で失われたり、位置を変える細胞が少ない実験から得られるデータの最大量(図4C)。
目的の発生段階で妥当な数の細胞のカルシウム活性と遺伝子発現の両方を記述するデータが収集されたら、これらの2つの特徴の相関を評価するためにさらなる分析を行うことができます(図1)。スパイクのカウンティング/周波数、平均パワー、ハースト指数推定、マルコフエントロピー測定17、18など、カルシウム活性パターンを定量化するために、多くの指標が適用されています。遺伝子発現は、絶対蛍光レベルによって定量的に定義するか、または対処されている実験の質問に応じて、バイナリ(はい/いいえ)スケールで等級分けすることができます。
神経前駆マーカー遺伝子の発現とカルシウム活性を照合する実験の結果、カルシウム活性の特定のパターンと神経伝達物質の表現型との間に多数の関連が明らかになった。神経板ステージ(ステージ14)では、阻害性ニューロンマーカー gad1.1を発現するGABAergic細胞は、gad1.1発現を欠いている細胞よりも規則的かつ高振幅のカルシウム活性を示す(図5A)。さらに、これらのgad1.1-発現細胞がより高いレベルの高振幅スパイクに関連しているのに対し、興奮性ニューロンマーカー lclclclclclclclclclc17a7を発現するグルタミン酸細胞では低振幅スパイクがより頻繁に起こる。
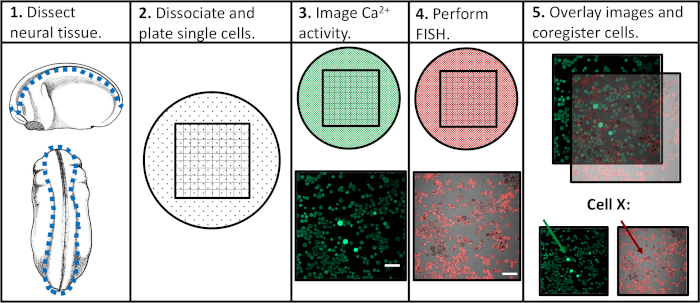
図1:実験ワークフローの概略図スケールバー = 100 μmパネル3-5の画像は、パウデルら(2019)17から撮影されました。この図の大きなバージョンを表示するには、ここをクリックしてください。
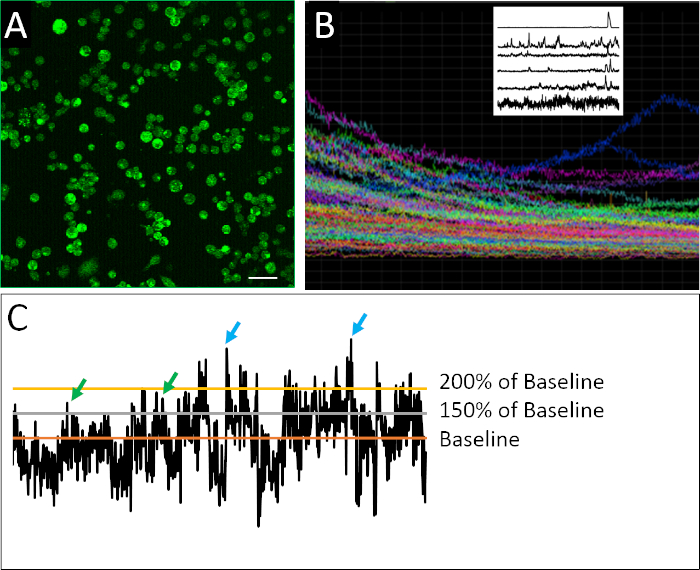
図2:カルシウムイメージングとアクティビティプロファイルの例(A) Fluo4-AMによって報告された細胞内カルシウム活性。各2時間の画像は901フレームで構成され、1つの代表的なフレームがここに示されています。(B) 画像化視野内のすべての細胞における経時の蛍光強度の複合プロット。痕跡は明らかにインジケータ染料(Fluo4)残業のフォトブリーチを示しています。左上のラスタープロットは、エイラーズとボーレン19によって開発された脱トレンドアルゴリズムの適用後のカルシウム活性の代表的な痕跡を示し、ここで示された細胞は多様なスパイク行動パターンを示す。(C) 異なる閾値(ベースラインの150%および200%、ベースラインはトレンド解除蛍光強度の平均)の適用をスパイク(緑と青の矢印)に定義する。スケールバー = 100 μmこの図の大きなバージョンを表示するには、ここをクリックしてください。
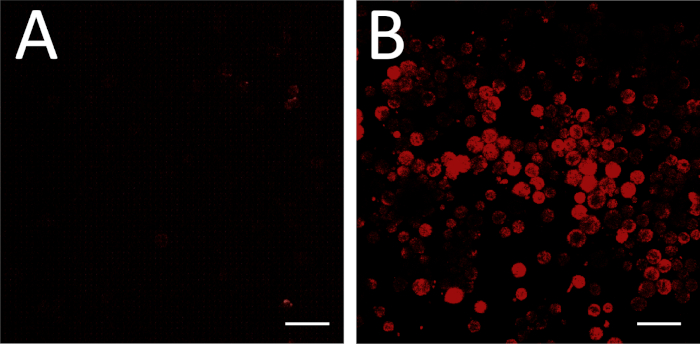
図3:魚撮像(A) 負制御として非結合感知体RNAプローブを用いて行ったFISH。蛍光を見ない細胞がないようにイメージング設定が調整されています。一部の視野には、(A)の右上および右下隅に見られるような、ある種の蛍光を伴う非細胞破片が含まれる場合があります。これらは、背景設定のために無視することができます。(B) 実験プレート(アンチセンスRNAプローブ)の画像化には、同じイメージング設定が使用されます。これらの条件下での蛍光は、上記の遺伝子発現に対応するバックグラウンドである。スケールバー = 100 μmこの図の大きなバージョンを表示するには、ここをクリックしてください。

図4:画像のオーバーレイと一連の登録カルシウム活性(緑色の丸で満たされたセル)とFISH(赤い赤い円で表されるセル)の後に画像化されたサンプルの概略図。(A) サンプルの処理と処理中に大幅に移動したセルは、2 つの画像で確実に識別できません。(B) 細胞の破壊は、視野の一部のセルにのみ影響を及ぼす可能性があります。両方の画像で明確に識別できるセルもあれば、自信を持って一致させることができないセルもあります。(C) サンプルを慎重に扱う場合、ほとんどのセルは邪魔されず、両方の画像で識別できます。この図の大きなバージョンを表示するには、ここをクリックしてください。
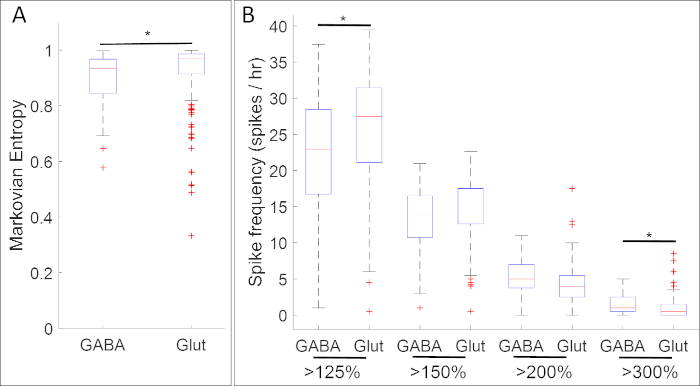
図5:この方法の適用例として、神経板段階ゼノプス・レービスにおけるカルシウム活性と遺伝子発現との関連を示す箱ひげ図(それぞれ遺伝子gad1.1およびslc17a7に対するGABAおよびGlut)ステージ14では、gad1.1-ポスチング細胞(GABA)は、(A)マルコフエントロピー18および(B)スパイク数閾値125%、150%、200%および300%の閾値を使用して定義されるより高い振幅およびより規則的なカルシウム活性を示す(ベースライン177a(slc17a)よりも大きい。星は、ボンフェローニ補正2サンプルコルモゴロフスミルノフ検定(p < 0.05)と効果サイズのコーエンのd統計(n = 5培養および>100セル;* 0.2 ≤ |d| < 0.5)の両方に従って統計的に有意な差を示します。この図は、パウデルら17から得られたデータセットから再描画され、適応された。この図の大きなバージョンを表示するには、ここをクリックしてください。
ディスカッション
カルシウム活性の特徴的なパターンは、発達中の神経系を構成する細胞において観察されており、特定のタイプの活動が明確な神経発達過程に関連している。しかし、これらの情報密度の高い活性パターンが転写応答に変換されるメカニズムをさらに理解するには、カルシウム活性と遺伝子発現に関する情報を単一細胞分解能で収集する必要があります。成熟したニューロンのようなよりステレオタイプなカルシウム活性を示すシステムは、バルクレベルで合理的にアッセイすることができるが、胚性神経系を特徴付ける不規則なパターンは、より正確でない記録によって容易にマスクされる。
このプロトコルで確立された実験フレームワークは、多種多様な細胞タイプおよび蛍光レポーターに容易に適応可能である。事実上あらゆる細胞タイプまたは細胞タイプの組み合わせを含む組織は、目的のモデル生物から解剖され、単一細胞イメージングのためにメッキされ得る。細胞の同定と細胞自律プロセスの効果の分離を可能にすることに加えて、一次細胞培養アプローチは、実験者が望ましい媒体成分を定義することを可能にする。例えば、2mMのCa2+溶液におけるニューロン前駆体の活性を比較した実験は、胚性脊髄におけるスパイク頻度と神経伝達物質表現型との関係が細胞間相互作用13、20の影響を受けずに再現できるかどうかを調べるために行われた。
このプロトコルは蛍光マーカーFluo4-AMを利用して細胞内カルシウム活性を検出するが、選択基準に応じて、ユーザーは、遺伝的にコードされたカルシウム指標を含む他の市販のマーカー21を選択することができる。同様に、代替マーカーは、目的のイオン(K+、Na+、およびZn2+を含む)、膜電位、または細胞pHの濃度に対する動的変化を監視するために使用することができる。画像の設定と画像の継続時間は必要に応じて変更できます。
具体的な用途としてカルシウム活性と神経表現型を相関させたが、この方法は他の様々な細胞特性にも適用可能である。例えば、situハイブリダイゼーション中の蛍光は、ニューロンマーカーChATまたは転写因子Engrailedを含む任意の目的の遺伝子に対するプローブを用いて行うことができるので、mRNA種のカスタマイズ可能なパネルの感度検出を可能にする。これらのプローブは、アイソフォーム特異的になるように設計することができ、必要に応じて追加のターゲット特異性をサポートします。Double FISHは、複数の異なる蛍光異管に共役したプローブを使用して行うことができるので、複数の遺伝子の発現を同時に評価することができます。しかし、この種の実験で必要とされる追加のワッシュは、細胞の喪失や動きの可能性の増加に関連しており、経験と繊細さが正常に実行される必要があります。
このプロトコルに対して行われた実験固有の変更に関係なく、注意が必要ないくつかの重要な手順があります。すべての汚染組織または細胞集団を除去するために注意して、分節を行う必要があります。外植が解離されると空間パターンは失われるため、隣接する組織の残りの細胞は、関心のある細胞と散在し、区別がつかないようになります。細胞がメッキされた後、サンプルは細胞が外れないようにできるだけ穏やかに扱われるべきである。最も重要なことは、これは、溶液が除去され、追加されるときにピペットをプレートの端に置いて、すべての溶液の変更がゆっくりと慎重に行われるべきであることを意味します。これにより、カルシウム画像とFISH画像の両方で細胞が確実に同定できるようになります。処理中に細胞が破壊された場合、2つの画像間の対応する細胞の一部または全部を特定することができない可能性があります。我々は、明確に対応する細胞だけがさらなる分析に使用されるように、これらの割り当てに注意する側に誤りをお勧めします。
対処される生物学的問題に応じて、様々な分析アプローチが適切な場合があります。時系列カルシウム活性は、さまざまな方法で処理および定量することができ、実験者はトレンド除去パラメータ、分析メトリック、分析パラメータ(例えば、カルシウムスパイクを定義するために使用されるベースライン閾値の%)を柔軟に選択できます。カルシウム活性と遺伝子発現レベルとの相関は、FISH画像から抽出された絶対蛍光値または相対蛍光値として遺伝子発現を解析することによって導き出すことができる。また、カルシウム活性と遺伝子発現(存在/不在)との相関は、陽性遺伝子発現シグナルの蛍光閾値を定義し、個々の細胞に「はい」または「いいえ」の識別子を割り当てることによって引き出すことができます。全体として、この実験的なスキーマは、細胞に一致した遺伝子発現データと組み合わせて時系列データの収集と予備分析のための非常に柔軟なパイプラインを提供します。このような実験は、胚性ゼノプス・レエビスにおける抑制性脂肪および興奮性脂肪神経前駆体のカルシウム活性パターンの同定によって例示されるように、細胞動態と転写変化との複雑な関係をよりよく理解するために重要であろう。
開示事項
利益相反は宣言されていません。
謝辞
ウェンディ・ハーブストとリンジー・シュライファーがこれらのプロトコルの開発に貢献してくれたことに感謝します。この作業は、国立衛生研究所(1R15NS067566-01、1R15HD077624-01、および1R15HD096415-01)からMSSへの助成金によってサポートされました。
資料
| Name | Company | Catalog Number | Comments |
| For Animal Husbandry & Cell Culture | |||
| CHORULON (chorionic gonodotropin) | Merck Animal Health | ||
| Gentamycin sulfate salt | Millipore Sigma | G1264 | |
| Penicillin-Streptomycin (10,000 U/mL) | Thermo Fisher Scientific | 15140122 | |
| Pyrex petri dishes, 100 mm x 20 mm | Millipore Sigma | CLS3160102 | |
| Corning Falcon Easy-Grip Tissue Culture Dishes, 35mm | Fisher Scientific | 08-772A | |
| Corning Falcon Easy-Grip Tissue Culture Dishes, 60mm | Fisher Scientific | 08-772F | |
| Falcon Standard Tissue Culture Dishes | Fisher Scientific | 08-772E | |
| Thermo Scientifc Nunc Cell Culture / Petri Dishes, 35x10mm Dish, Nunclon Delta | Fisher Scientific | 12-565-90 | |
| Fisherbrand Standard Disposable Transfer Pipettes, Nongraduated; Length: 5.875 in.; Capacity: 7.7 mL | Fisher Scientific | 13-711-7M | |
| Ethyl 3-aminobenzoate methanesulfonate | Millipore Sigma | E10521 | |
| Collagenase B | Millipore Sigma | 11088807001 | |
| Dumont #55 Forceps, Dumostar | Fine Science Tools | 11295-51 | |
| Dumont #5 Forceps, Dumostar | Fine Science Tools | 11295-00 | |
| Cellattice Micro-Ruled Cell Culture Surface | Nexcelom Bioscience | CLS5-25D-050 | |
| For Calcium Imaging | |||
| Fluo-4, AM, cell permeant | Thermo Fisher Scientific | F14201 | |
| Pluronic F-127, 0.2 µm filtered (10% Solution in Water) | Thermo Fisher Scientific | P6866 | |
| For RNA Probe Generation | |||
| PureYield Plasmid Miniprep System | Promega | A1222 | |
| rATP | Promega | P1132 | |
| rCTP | Promega | P1142 | |
| rGTP | Promega | P1152 | |
| rUTP | Promega | P1162 | |
| Digoxigenin-11-UTP | Millipore Sigma | 3359247910 | |
| Rnase Inhibitor | Thermo Fisher Scientific | N8080119 | |
| T3 RNA Polymerase | Promega | P2083 | |
| T7 RNA Polymerase | Promega | P2075 | |
| SP6 RNA Polymerase | Promega | P1085 | |
| RQ1 Rnase-Free Dnase | Promega | M6101 | |
| LiCl Precipitation Solution (7.5 M) | Thermo Fisher Scientific | AM9480 | |
| For Fluorescence In Situ Hybridization | |||
| Acetic Anhydride | Thermo Fisher Scientific | 320102 | |
| Blocking Reagent | Millipore Sigma | 11096176001 | |
| Anti-Digoxigenin-POD, Fab fragments | Millipore Sigma | 11207733910 | |
| Cy3 Mono-Reactive NHS Ester | Millipore Sigma | GEPA13105 | |
| Solution Components | |||
| Calcium chloride, 96% extra pure, powder, anhydrous, ACROS Organixs | Fisher Scientific | AC349610 | |
| Calcium chloride dihydrate | Millipore Sigma | C3306 | |
| CHAPS hydrate | Millipore Sigma | C3023 | |
| Denhardt's Solution (50X) | Thermo Fisher Scientific | 750018 | |
| DTT, Molecular Grade (DL-Dithiothreitol) | Promega | P1171 | |
| Ethylenediaminetetraacetic Acid, Disodium Salt Dihydrate | Fisher Scientific | S311 | |
| Ethylene glycol-bis(2-aminoethylether)-N,N,N',N'-tetraacetic acid | Millipore Sigma | E3889 | |
| Formamide (Deionized) | Thermo Fisher Scientific | AM9342 | |
| Herparin sodium salt from porcine intestinal mucosa | Millipore Sigma | H3393 | |
| HEPES (Ultra Pure) | Thermo Fisher Scientific | 11344041 | |
| Hydrogen peroxide solution | Millipore Sigma | H1109 | |
| L-Cysteine | Millipore Sigma | 168149 | |
| Magnesium chloride, pure, ACROS Organics | Fisher Scientific | AC223211000 | |
| Magnesium sulfate, 97% pure, ACROS Organixs, anhydrous | Fisher Scientific | AC413480050 | |
| Maleic Acid, 99%, ACROS Organics | Fisher Scientific | ACS125231000 | |
| MOPS (Fine White Crystals/Molecular Biology), Fisher BioReagents | Fisher Scientific | BP308 | |
| Potassium chloride | Millipore Sigma | P9541 | |
| Ribonucleic acid from torula yeast, Type IX | Millipore Sigma | R3629 | |
| Sodium chloride | Millipore Sigma | S7653 | |
| Triethanolamine | Millipore Sigma | 90279 | |
| Tris | Millipore Sigma | GE17-1321-01 | |
| TWEEN 20 | Millipore Sigma | P9416 | |
| Equipment | |||
| Laminar Flow Hood | model of choice | ||
| Dissecting Microscope | model of choice | ||
| Inverted Fluorescence Microscope | Nikon | TE200 | |
| NIS-Elements Imaging Software | Nikon | ||
| Shaking Incubator | model of choice | ||
| Refrigerated Centrifuge | model of choice | ||
| Miscellaneous | |||
| Corning bottle-top vaccum filter system, 0.22 μm pore, 500 mL bottle capacity | Millipore Sigma | CLS430769 | |
| Falcon 50mL Conical Centrifuge Tubes | Fisher Scientific | 14-432-22 |
参考文献
- Humeau, J., et al. Calcium signaling and cell cycle: Progression or death. Cell Calcium. 70, 3-15 (2017).
- Kim, J. M., Lee, M., Kim, N., Heo, W. D. Optogenetic toolkit reveals the role of Ca2+ sparklets in coordinated cell migration. PNAS. 112 (21), 5951-5957 (2016).
- Orrenius, S., Zhivotovsky, B., Nicotera, P. Regulation of cell death: the calcium-apoptosis link. Nature Reviews Molecular Cell Biology. 4 (7), 552-565 (2003).
- Pham, K., et al. Ca2+ and Mg2+ module conformational dynamics and stability of downstream regulatory element antagonist modulator. Protein Science. 24 (5), 741-751 (2015).
- Smedler, E., Uhlén, P. Frequency decoding of calcium oscillations. Biochimica et Biophysica Acta. 1840 (3), 964-969 (2014).
- Moreau, M., Néant, I., Webb, S. E., Miller, A. L., Riou, J. F., Leclerc, C. Ca(2+) coding and decoding strategies for the specification of neural and renal precursor cells during development. Cell Calcium. 59 (2-3), 75-83 (2016).
- Tomida, T., Hirose, K., Takizawa, A., Shibasaki, F., Iino, M. NFAT functions as a working memory of Ca2+ signals in decoding Ca2+ oscillation. EMBO. 22 (15), 3825-3832 (2003).
- Hannanta-Anan, P., Chow, B. Y. Optogenetic Control of Calcium Oscillation Waveform Defines NFAT as an Integrator of Calcium Load. Cell Systems. 2 (4), 283-288 (2016).
- Li, L., Stefan, M. I. Le Novère N. Calcium input frequency, duration and amplitude differentially module the relative activation of calcineurin and CaMKII. PLoS One. 7 (9), 43810(2012).
- Romano, D. R., Pharris, M. C., Patel, N. M., Kinzer-Ursem, T. L. Competitive tuning: Competition's role in setting the frequency-dependence of Ca2+-dependent proteins. PLoS Computational Biology. 13 (11), 1005820(2017).
- Pharris, M. C., Patel, N. M., Kinzer-Ursen, T. L. Competitive Tuning Among Ca2+/Calmodulin-Dependent Proteins: Analysis of in silico Model Robustness and Parameter Variability. Cellular and Molecular Bioengineering. 11 (5), 353-365 (2018).
- Gu, X., Olson, E. C., Spitzer, N. C. Spontaneous neuronal calcium spikes during early differentiation. Journal of Neuroscience. 14 (11), 6325-6335 (1994).
- Borodinsky, L. N., Root, C. M., Cronin, J. A., Sann, S. B., Gu, X., Spitzer, N. C. Activity-dependent homeostatic specification of transmitter expression in embryonic neurons. Nature. 429 (6991), 523-530 (2004).
- Ciccolini, F., Collins, T. J., Sudhoelter, J., Lipp, P., Berridge, M. J., Bootman, M. D. Local and Global Spontaneous Calcium Events Regulate Neurite Outgrowth and Onset of GABAergic Phenotype during Neural Precursor Differentiation. Journal of Neuroscience. 23 (1), 103-111 (2003).
- Weissman, T. A., Riquelme, P. A., Ivic, L., Flint, A. C., Kriegstein, A. R. Calcium Waves Propagate through Radial Glial Cells and Modulate Proliferation in the Developing Neocortex. Neuron. 43 (5), 647-661 (2004).
- Nieuewkoop, P. D., Faber, J. The stages of Xenopus embryonic development. Normal Table of Xenopus laevis. , North Holland Publishing Company. (1994).
- Paudel, S., et al. Calcium Activity Dynamics Correlate with Neuronal Phenotype at a Single Cell Level and in a Threshold-Dependent Manner. International Journal of Molecular Science. 20 (8), 1880(2019).
- Marken, J. P., et al. A Markovian Entropy Measure for the Analysis of Calcium Activity Time Series. PLoS One. 11 (12), 0168342(2016).
- Eilers, P. H. C., Boelens, H. F. M. Baseline Correction with Asymmetric Least Squares Smoothing. Leiden University Medical Centre Report. , (2005).
- Guemez-Gamboa, A., et al. Non-cell-autonomous mechanism of activity-dependent neurotransmitter switching. Neuron. 82 (5), 1004-1016 (2014).
- Paredes, R., Madelaine,, et al. Chemical calcium indicators. Methods. 46 (3), 143-151 (2008).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved