Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Fabricación de redes de nanotubos lipídicos multicomponente utilizando el ensayo de motilidad de quinesina deslizante
En este artículo
Resumen
Este protocolo describe un proceso para fabricar redes de nanotubos lipídicos utilizando la motilidad de la quinesina deslizante junto con vesículas lipídicas unilamelares gigantes.
Resumen
Las redes de nanotubos lipídicos (LNT) representan un sistema modelo in vitro para estudiar el transporte molecular y la biofísica lipídica con relevancia para los túbulos lipídicos ubicuos que se encuentran en las células eucariotas. Sin embargo, los LNT in vivo son estructuras altamente no equilibradas que requieren energía química y motores moleculares para ser ensamblados, mantenidos y reorganizados. Además, la composición de los LNT in vivo es compleja, compuesta por múltiples especies lipídicas diferentes. Los métodos típicos para extruir LNT requieren mucho tiempo y mano de obra, y requieren pinzas ópticas, microperlas y micropipetas para extraer por la fuerza nanotubos de vesículas lipídicas gigantes. Aquí se presenta un protocolo para el ensayo de motilidad de deslizamiento (GMA), en el que las redes LNT a gran escala se generan rápidamente a partir de vesículas unilamelares gigantes (GUV) utilizando la motilidad de microtúbulos alimentada por kinesina. Utilizando este método, las redes de LNT se forman a partir de una amplia gama de formulaciones lipídicas que imitan la complejidad de los LNT biológicos, lo que los hace cada vez más útiles para estudios in vitro de biofísica lipídica y transporte asociado a la membrana. Además, este método es capaz de producir de manera confiable redes LNT en poco tiempo (<30 min) utilizando equipos de laboratorio de uso común. Las características de la red LNT, como la longitud, el ancho y la partición de lípidos, también se pueden ajustar al alterar la composición lipídica de los GUV utilizados para fabricar las redes.
Introducción
La fabricación de redes de nanotubos lipídicos (LNT) es de creciente interés para el examen in vitro de estructuras lipídicas no equilibradas 1,2,3. Las células utilizan túbulos lipídicos para el transporte difusivo de proteínas4 y ácidos nucleicos5, así como para la comunicación de célula a célula 6,7. El retículo endoplásmico y el aparato de Golgi son particularmente interesantes, ya que estos orgánulos unidos a la membrana son los lugares principales para la síntesis de lípidos y proteínas, así como el transporte de estas biomoléculas integrales dentro del citoplasma de una célula 8,9. Las membranas de estos orgánulos están compuestas por múltiples especies de lípidos, incluidos esfingolípidos, colesterol y fosfolípidos10 que en última instancia ayudan a definir su funcionalidad. Por lo tanto, para replicar y estudiar más de cerca estos orgánulos, los LNT in vitro deben fabricarse a partir de vesículas con formulaciones lipídicas cada vez más complejas11.
Las vesículas unilamelares gigantes (GUV) se utilizan de manera generalizada para estudiar el comportamiento de la membrana lipídica porque se pueden sintetizar de manera confiable con formulaciones complejas que incluyen colesterol, fosfatidilcolina (PC), fosfatidiletanolamina (PE), fosfatidilserina (PS) y fosfatidilinositol (PI)12,13. Aquí se describe un método para fabricar LNT a partir de GUV con diferentes formulaciones lipídicas utilizando el ensayo de motilidad deslizante (GMA), en el que los LNT se extruyen en función del trabajo realizado por los motores de quinesina y los filamentos de microtúbulos que actúan sobre los GUV. En este sistema, las proteínas motoras de quinesina adsorbidas a una superficie impulsan microtúbulos biotinilados, convirtiendo la energía química de la hidrólisis del ATP en trabajo útil (específicamente, la extrusión de LNT a partir de vesículas biotiniladas)11. La red LNT resultante proporciona una plataforma modelo para estudiar los efectos de las diferencias en las fases lipídicas sobre los cambios en la morfología LNT.
Brevemente, las proteínas motoras de quinesina se introducen en una cámara de flujo en una solución que contiene caseína, lo que permite la adsorción de los motores en la superficie de vidrio de la cámara. A continuación, los microtúbulos biotinilados en una solución que contiene ATP fluyen a través de la cámara y se les permite unirse a los motores de quinesina y comenzar la motilidad. Luego se introduce una solución de estreptavidina en la cámara y se deja que se una de forma no covalente a los microtúbulos. Finalmente, los GUV que contienen un lípido biotinilado se introducen en la cámara y se unen a los microtúbulos recubiertos de estreptavidina, luego extruyen LNT para formar redes a gran escala en el transcurso de 15-30 min. Este método produce redes LNT grandes y ramificadas utilizando equipos de laboratorio estándar y reactivos a un bajo costo11.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Preparación de soluciones de microtúbulos de stock
PRECAUCIÓN: Siempre se deben usar gafas de seguridad, guantes y una bata de laboratorio durante todo el protocolo.
- Prepare 5x tampón BRB80: agregue 24.19 g de PIPES (piperazina-N,N′-bis[ácido 2-etanosulfónico]) y 0.38 g de EGTA (etilenglicol-bis[éter β-aminoetilo]-N,N,N′,N′,N′-ácido tetraacético) a una botella de vidrio de 1 L. Agregue 1 ml de 1 M MgCl2 y ajuste el pH a 6.9 con KOH. Agregue agua desionizada para llevar la solución a un volumen final de 500 ml.
- Preparar 100 mM de solución GTP: pesar 52 mg de GTP y suspender en 1 mL de agua destilada. Divida la solución de 100 mM en alícuotas de 20 μL y guárdela a -20 °C.
- Preparar solución GPEM: mezclar 200 μL de 5x BRB80, 10 μL de 100 mM de solución GTP, 100 μL de 100% de glicerol y 600 μL de agua desionizada. Divida la solución GPEM en alícuotas de 100 μL y guárdela a -20 °C.
- Preparar la solución de microtúbulos reconstituyendo viales de tubulina liofilizada disponible comercialmente (un vial cada uno de biotinilado, marcado fluorescentemente y sin etiquetar) en solución de GPEM fría (4 °C) a una concentración de stock de 5 mg/ml.
- Realice la polimerización de microtúbulos mezclando 4 μL de tubulina biotinilada, 4 μL de tubulina marcada fluorescentemente y 24 μL de tubulina sin etiquetar (todo a concentraciones de 5 mg / ml) para crear una proporción de 1: 1: 6 a un volumen final de 32 ml. Manténgalo en hielo. Divida la mezcla de tubulina en alícuotas de 2 μL y guárdela a -80 °C hasta que sea necesario.
NOTA: La polimerización eficiente requiere que la concentración de tubulina sea igual o superior a la concentración crítica (5 mg/ml)14. Aquí, la selección de la relación de tubulina se optimiza para una concentración suficiente de tubulina biotinilada para unir eficientemente la estreptavidina y los GUV, así como una concentración suficiente de tubulina fluorescente para la caracterización microscópica.
2. Preparación de vesículas unilamelares gigantes (GUV)
- Preparación de la película de agarosa
NOTA: Este protocolo está adaptado de Greene et al.15.- Prepare una solución al 1% p/v mezclando 1 g de agarosa en 100 ml de agua desionizada en matraz Erlenmeyer de 250 ml. Use un microondas estándar para calentar la solución de agarosa durante 1-2 min.
NOTA: La solución se volverá translúcida una vez que la agarosa se disuelva por completo. Deje que la solución se enfríe a 65–75 °C antes de usarla. - Utilice una punta de pipeta cortada de 1.000 μL para pipetear 300–400 μL de solución de agarosa en una cubierta de vidrio de 25 mm x 25 mm. Mientras sostiene el borde de la funda con los dedos enguantados, use otra punta de pipeta de 1,000 μL para extender la agarosa derretida uniformemente a través de la cubierta.
NOTA: Mantener la agarosa a 65–75 °C permitirá una propagación eficiente en la superficie de la cubierta. - Seque las cubiertas recubiertas de agarosa en una incubadora de 37 °C durante al menos 2 h, momento en el que la agarosa se volverá transparente. Guarde las cubiertas colocando la superficie recubierta de agarosa hacia arriba sobre una superficie limpia, como papel sin pelusa o película a base de cera a temperatura ambiente (RT).
- Prepare una solución al 1% p/v mezclando 1 g de agarosa en 100 ml de agua desionizada en matraz Erlenmeyer de 250 ml. Use un microondas estándar para calentar la solución de agarosa durante 1-2 min.
- Formulación lipídica
- Recupere los lípidos disueltos en cloroformo de un congelador de -20 °C y colóquelos en una campana de humos químicos hasta que alcancen RT.
- Calcular el volumen de stock lipídico de cada componente lipídico necesario para la formulación utilizando la siguiente fórmula:
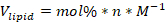
Donde: Vlípido es el volumen de lípido a utilizar, mol % es el porcentaje molar del componente lipídico, n es el número total de moles de lípido utilizado en la formulación, y M es la concentración de lípido en unidades molares.
NOTA: Por ejemplo, si se utiliza una formulación que contiene 45 mol% 1,2-dioleoil-sn-glicero-3-fosfocolina (DOPC) con una concentración de solución madre de 12,72 mM, y 1 micromol de lípidos totales en la formulación, el volumen de material DOPC utilizado en la formulación sería: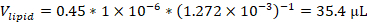
- Mezcle los lípidos juntos en la proporción calculada en un vial de vidrio en la campana de humos químicos.
- Pipetear 30 μL de solución lipídica sobre las hojas de cubierta recubiertas de agarosa en una placa caliente precalentada que se coloca por encima del punto de fusión del componente lipídico saturado de la formulación (por ejemplo, placa caliente de 50 °C para un lípido con Tm de 40 °C).
- Extienda la solución a través de la película de agarosa en un movimiento circular utilizando el borde largo de una aguja de 18 G hasta que el cloroformo se haya evaporado y se haya formado una capa uniforme de lípidos. Sostenga el borde de la funda con los dedos enguantados mientras realiza este paso.
NOTA: Se debe tener cuidado de no dañar la capa de agarosa con la aguja. - Coloque el cubrehojas con capa de agarosa y película lipídica en una placa de Petri cubierta de papel de aluminio, con el lado de la película hacia arriba, y coloque la placa de Petri en un desecador al vacío durante al menos 2 h para eliminar el disolvente residual.
- Mientras tanto, prepare una solución de sacarosa de 560 mM mezclando 1,92 g de sacarosa con 10 ml de agua desionizada.
NOTA: La concentración de la solución de sacarosa depende de la osmolaridad de los GUV tampón en los que se diluyen los GUV. Por lo general, la osmolaridad de la solución de sacarosa no debe ser más de un 10% mayor que el tampón en el que se diluirán los GUV, específicamente el tampón de motilidad (ver paso 3.11). - Formación GUV
- Adhiera una cámara adhesiva a la cubierta recubierta con la película lipídica presionando suavemente la cámara adhesiva sobre la cubierta recubierta de lípidos con la película lipídica hacia arriba, asegurando que se forme un sello hermético.
- Añadir 400 μL de solución de sacarosa de 560 mM (preparada en la etapa 2.3) a la cámara.
- Coloque la funda en la cámara de humedad y cierre la tapa.
- Coloque la cámara de humedad en una placa caliente precalentada colocada por encima del punto de fusión del componente lipídico saturado de la formulación.
- Permita que se formen vesículas durante ≥1 h antes de la recuperación.
NOTA: La formación de vesículas se puede verificar con microscopía de fluorescencia con una lente de objetivo de aire 40x.
3. Preparación de existencias y reactivos de ensayo de motilidad
- Preparación de stock de caseína
- Agregue 3 g de caseína seca a un tubo de centrífuga cónica de 50 ml, luego agregue 30 ml de 1x BRB80 y gire hasta que la solución se vuelva viscosa. Centrifugar el tubo a 15.000 x g durante 30 min.
- Transfiera el sobrenadante a otro tubo de centrífuga cónica de 50 ml y deseche el pellet. Filtre la solución a través de un filtro de jeringa de 1 μm, recogiendo la solución en un vial cónico de 50 ml. Filtrar la solución a través de un filtro de 0,2 μm, recogiendo la solución en un vial cónico de 50 ml.
- Determine la concentración de proteínas midiendo la absorbancia a 280 nm utilizando un espectrofotómetro UV-Vis y una cubeta de cuarzo.
- Calcule la concentración de caseína en mg/ml utilizando la siguiente fórmula:
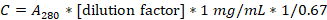
- Diluir la solución a 20 mg/ml en 1x BRB80, dividir en alícuotas de 100 μL y almacenar a -20 °C.
- Prepare la solución madre de glucosa oxidasa (2 mg/ml) mezclando 2 mg de glucosa oxidasa con 1 ml de 1x BRB80. Dividir en alícuotas de 100 μL y conservar a -20 °C.
- Preparar la solución madre de catalasa (0,8 mg/ml) mezclando 0,8 mg de catalasa con 1 ml de 1x BRB80. Dividir en alícuotas de 100 μL y conservar a -20 °C.
- Preparar 2 M de solución de glucosa suspendiendo 0,3 g de D-glucosa en 1 mL de agua desionizada. Dividir en alícuotas de 100 μL y conservar a -20 °C.
- Preparar 100 mM de TDT suspendiendo 0,015 g de TDT en 1 mL de agua desionizada. Dividir en alícuotas de 100 μL y conservar a -20 °C.
- Preparar 100 mM mg-ATP en suspensión de 0,055 g de ATP disódico en una solución de 1 ml de 100 mM de MgCl2. Dividir en alícuotas de 100 μL y conservar a -20 °C.
- Preparar una solución de Mg-AMP-PNP de 100 mM suspendiendo 0,055 g de AMP-PNP en una solución de 1 ml de 100 mM de MgCl2, luego dividir en alícuotas de 100 μL y almacenar a -20 °C.
- Preparar 100 mM de solución de Trolox añadiendo 25,03 mg de Trolox a 1 mL de metanol y almacenar a -20 °C.
- Prepare 10 mg/ml de solución de estreptavidina añadiendo 1 mg de estreptavidina a 100 μL de BRB80, luego divida en alícuotas de 2 μL y guárdela a -80ۛ °C.
- Prepare BRB90CAT mezclando 200 μL de 5x BRB80, 20 μL de solución de caseína, 10 μL de solución de MgATP, 10 μL de Trolox, 5 μL de solución de paclitaxel y 765 μL de agua DI. Conservar a 4 °C.
- Prepare la solución de motilidad mezclando 192 μL de BRB80CAT, 2 μL de solución de D-glucosa, 2 μL de solución de glucosa oxidasa, 2 μL de solución de DTT y 2 μL de solución de catalasa. Conservar a 4 °C.
- Preparar una solución de quinesina de 1 μM diluyendo la solución de quinesina madre en BRB80CAT (por ejemplo, 2 μL de solución de quinesina de 50 mM en 98 μL de BRB80CAT) y conservar a 4 °C.
- Preparar una solución de microtúbulos de 10 μg/mL diluyendo 10 μL de microtúbulos estabilizados en 90 μL de BRB80CAT a temperatura ambiente. Almacenar en RT.
- Preparar una solución de estreptavidina de 10 μg/ml añadiendo 0,1 μL de solución de estreptavidina de 10 mg/ml en 99,9 μL de solución de motilidad. Conservar a 4 °C.
- Prepare una solución de 12x GUV diluyendo 5 μL de caldo de GUV en 55 μL de solución de motilidad. Conservar a 4 °C.
- Polimerización de la tubulina en microtúbulos
- Recoger una alícuota de 2 μL de tubulina previamente preparada del congelador de -80 °C (preparada en la etapa 1.5) y colocarla en un baño de agua de 37 °C durante 30 min.
- Prepare una solución de paclitaxel de 2 mM agregando 1,71 mg de paclitaxel en 1 ml de DMSO anhidro, divida en alícuotas de 10 μL y almacene a -20 °C.
- Prepare recién BRB80T mezclando 99.5 μL de 1x BRB80 con 0.5 μL de paclitaxel de 2 mM. Caliente 100 μL de BRB80T a 37 °C en el baño de agua.
- Después de 30 min, agregue los 100 μL de BRB80T a la alícuota de tubulina para estabilizar los microtúbulos. Almacenar en RT.
4. Ensayo de motilidad deslizante (GMA)
- Prepare una cámara de flujo colocando dos tiras de cinta adhesiva de doble cara separadas por 5 mm en un portaobjetos de vidrio. Repita este proceso hasta que tres capas de cinta comprendan cada tira.
- Coloque una funda en la parte superior de la cinta, luego presione suavemente hacia abajo en la interfaz de la cubierta / cinta con una pinza o bolígrafo para garantizar una adherencia suficiente.
NOTA: El canal debe tener 5 mm de ancho por 25 mm de largo por 300 mm de alto. - Pipetear 30 μL de solución de quinesina de 1 mm (preparada en la etapa 3.12) en la celda de flujo y dejar incubar durante 5 min.
NOTA: La caseína forma una bicapa en la superficie de la cubierta / corredera de vidrio y facilita la fijación de la cola de kinesina a la superficie. - Pipetear 30 μL de solución de microtúbulos de 10 μg/ml (preparada en la etapa 3.13) en la celda de flujo, utilizando una toallita de laboratorio presionada suavemente contra el extremo opuesto del canal de flujo para facilitar el intercambio de la solución. Incubar durante 5 min.
- Lave la celda de flujo 1x–3x con 1x solución de motilidad en RT (preparado en el paso 3.11).
NOTA: La microscopía de fluorescencia que utiliza un objetivo de aire 40x se puede usar en este punto para confirmar la unión y la motilidad de los microtúbulos. Los microtúbulos aparecen como filamentos fluorescentes (decenas de micras de longitud) que se mueven (se deslizan) a través de la superficie a ~ 0.5 μm / s (Figura 1). - Pipetear 30 μL de solución de estreptavidina de 10 μg/ml (preparada en la etapa 3.14) en la celda de flujo utilizando una toallita de laboratorio presionada suavemente contra el extremo opuesto del canal de flujo para facilitar el intercambio de la solución. Incubar durante 10 min.
- Flujo de 30 μL de solución 12x GUV (preparada en el paso 3.15) en el flujo utilizando una toallita de laboratorio presionada suavemente contra el extremo opuesto del canal de flujo para facilitar el intercambio de soluciones. Incubar durante 30 min.
- Agregue 2 μL de solución AMP-PNP de 100 mM (preparada en la etapa 3.7) para detener la motilidad, luego selle la cámara con sellador.
5. Caracterización de la red LNT
- Transfiera la cámara de flujo a un microscopio invertido para obtener imágenes.
- Elija el conjunto de filtros apropiado en función de las longitudes de onda de los lípidos fluorescentes o la tubulina utilizados. Por ejemplo, cuando use lípidos marcados con Texas Red, use un filtro de excitación de 560 nm / 25 nm y un filtro de emisión de 607 nm / 36 nm.
- Utilice un objetivo de aceite 100x para enfocarse en la superficie de la cubierta.
- Imagen de las redes LNT utilizando microscopía de fluorescencia.
NOTA: Los LNT son estructuras lineales que se extruyen de las vesículas más grandes. LNT mucho más pequeños que los GUV y tienen señales de fluorescencia más débiles. Por lo tanto, la exposición y el contraste deben ajustarse en consecuencia a los LNT de imagen. Estos ajustes también conducen a la sobreexposición de los GUV, y por lo tanto, se recomienda que la lamelaridad y la separación de fases en los GUV se caractericen de forma independiente. - Enfoque el microscopio en una red de interés y tome una imagen estándar o en mosaico.
- Ajuste el tiempo de exposición y los filtros de densidad neutra para obtener imágenes de los LNT y minimizar la exposición saturada de los GUV. Adquiere imágenes tanto en canales rojos como verdes.
NOTA: Aquí, el canal rojo permite la visualización de los lípidos rojos de Texas, mientras que el canal verde permite la visualización de los microtúbulos (por ejemplo, lípidos verdes de Oregón y colorantes HiLyte488). - Cree una imagen compuesta superponiendo los canales rojo y verde (Figura 1).
- Caracterización de redes LNT mediante la medición de la longitud LNT
- Abra las imágenes adquiridas utilizando un software de análisis de imágenes como ImageJ.
- Calibre la escala para el microscopio mediante la función de escala establecida, rellene los píxeles hasta el factor de conversión mm y haga clic en Aceptar.
NOTA: El factor de conversión depende del microscopio, la lente del objetivo y la cámara, y se puede obtener utilizando una diapositiva de calibración del microscopio. Generalmente se expresa en píxeles/mm. - Utilice la herramienta Línea multipunto para medir la longitud de los nanotubos a partir del GUV principal. Mantenga pulsada la tecla Ctrl +M para medir la longitud.
- Continúe midiendo las longitudes de los tubos individuales siguiendo los pasos anteriores. La herramienta de procesamiento de imágenes guardará cada nueva medición en la ventana de resultados.
- Mantenga presionada la tecla Ctrl + D después de dibujar cada línea para realizar un seguimiento de los tubos que se han medido.
- Medición del espesor del LNT (Figura 2)
- Abra la imagen en ImageJ y seleccione la función Umbral en la pestaña Imagen .
- Haga clic en Aplicar para aplicar el umbral.
- Dibuje un rectángulo de longitud conocida sobre el tubo deseado (los píxeles negros tienen un valor de 0 y los píxeles rojos tienen un valor de 255).
- Medir la densidad integrada del área.
- Divida la densidad por la longitud (en píxeles) del LNT para obtener el grosor (en píxeles).
NOTA: Las mediciones de espesor solo se pueden comparar entre imágenes cuando la configuración de imagen y el umbral se establecen de manera idéntica.
- Determinación de la partición lipídica en nodos de LNT (Figura 3)
- Abra la imagen en ImageJ.
- Utilice la herramienta Línea para dibujar una línea sobre el nodo deseado.
- Mida la intensidad del nodo en los canales Oregon Green y Texas Red.
- Mueva la línea al LNT y mida la intensidad del LNT en los canales Oregon Green y Texas Red.
Access restricted. Please log in or start a trial to view this content.
Resultados
Las redes LNT (Figura 4) se fabricaron utilizando el protocolo descrito, que utiliza el trabajo realizado por el transporte de quinesina de microtúbulos para extruir LNT de GUV. Brevemente, los GUV se prepararon utilizando rehidratación en gel de agarosa utilizando solución de sacarosa, y los microtúbulos se polimerizaron en solución GPEM y se estabilizaron en BRB80T. A continuación, los motores de quinesina se introdujeron en una celda de flujo formando una capa activa de motores en l...
Access restricted. Please log in or start a trial to view this content.
Discusión
Las redes LNT son una herramienta útil para estudios in vitro de las propiedades de la membrana y el transporte de biomoléculas como las proteínas transmembrana. Además, el uso de formulaciones lipídicas complejas para fabricar redes LNT permite estudios biológicamente más relevantes. Otros estudios de fabricación han utilizado 1) formulaciones lipídicas simples y vesículas multilamelares o 2) técnicas de motilidad más engorrosas para fabricar redes a partir de GUV compuestas de formulaciones lipídicas compl...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Sandia National Laboratories es un laboratorio multimisión administrado y operado por National Technology & Engineering Solutions of Sandia, LLC., una subsidiaria de propiedad total de Honeywell International, Inc., para la Administración Nacional de Seguridad Nuclear del DEPARTAMENTO de Energía de los Estados Unidos bajo el contrato DE-NA-0003525. Este documento describe los resultados técnicos objetivos y el análisis. Cualquier punto de vista u opinión subjetiva que pueda expresarse en el documento no representa necesariamente los puntos de vista del Departamento de Energía de los Estados Unidos o del Gobierno de los Estados Unidos.
Agradecimientos
Este trabajo fue apoyado por el Departamento de Energía de los Estados Unidos, Oficina de Ciencias Básicas de la Energía, División de Ciencias e Ingeniería de Materiales (BES-MSE). La síntesis de kinesina y la microscopía de fluorescencia se realizaron a través de un proyecto de usuario (ZIM) en el Centro de Nanotecnologías Integradas, una instalación de usuario de la Oficina de Ciencia operada para la Oficina de Ciencia del Departamento de Energía de los Estados Unidos (DOE).
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 100x/1.4 Numerical Aperture Oil Immersion Objective | Olympus | 1-U2B836 | Olympus UPlanSApo 100x/1.40 Oil Objective Infinity Corrected, RMS Thread Working Distance 0.12mm |
| 3.0 ND Filter | Olympus | Neutral Density Filter | |
| AMP-PNP | Sigma-Aldrich | A2647 | (β,γ-imidoadenosine 5′-triphosphate) |
| ATP | Sigma-Aldrich | A7699 | Adenosine 5'-triphosphate disodium salt hydrate BioXtra |
| Brightline Pinkel DA/FI/TR/Cy5/Cy7-5X-A000 filter set | Semrock | LED-DA/FI/TR/Cy5/Cy7-5X-A-000 | BrightLine Pinkel filter set, optimized for DAPI, FITC, TRITC, Cy5 & Cy7 and other like fluorophores, illuminated with LED-based light sources |
| Casein | Sigma-Aldrich | 22090 | Casein hydrolysate for microbiology |
| Catalase | Sigma-Aldrich | C9322 | Catalase from Bovine Liver |
| Chloroform | Sigma-Aldrich | 288306 | Chloroform anhydrous contains 0.5-1.0% ethanol as stabilizer |
| Cholesterol | Avanti | 700000P | cholesterol (ovine wool, >98%) (powder) |
| D-Glucose | Sigma-Aldrich | G7021 | D-(+)-Glucose powder, BioReagent, suitable for cell culture, suitable for insect cell culture, suitable for plant cell culture, ≥99.5% |
| DOPC | Avanti | 850375C | 1,2-Dioleoyl-sn-glycero-3-phosphocholine (in chloroform) |
| DOPE-Biotin | Avanti | 870282C | 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine-N-(biotinyl) (sodium salt) |
| DPPC | Avanti | 850355P | 1,2-dipalmitoyl-sn-glycero-3-phosphocholine (powder) |
| DPPE-Biotin | Avanti | 870285P | 1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(biotinyl) (sodium salt) |
| DTT | Sigma-Aldrich | 43816 | DL-Dithiothreitol solution 1 M |
| EGTA | Sigma-Aldrich | E4378 | EGTA, Egtazic acid, Ethylene-bis(oxyethylenenitrilo)tetraacetic acid, Glycol ether diamine tetraacetic acid |
| Glucose Oxidase | Sigma-Aldrich | G6125 | Glucose Oxidase from Aspergillus niger Type II, ≥10,000 units/g solid (without added oxygen) |
| Glycerol | Fisher | G33 | Glycerol (Certified ACS), Fisher Chemical |
| GTP | Sigma-Aldrich | G8877 | Guanosine 5′-triphosphate sodium salt hydrate |
| IX-81 Olympus Microscope | Olympus | N/A | IX81 Inverted Microscope from Olympus |
| KOH | Sigma-Aldrich | 1050121000 | Potassium Hydroxide |
| Magnesium Chloride | Sigma-Aldrich | M1028 | 1.00 M magnesium chloride solution |
| Orca Flash 4.0 Digital Camera | Hamamatsu | C13440-20CU | ORCA-Flash 4.0 V3 Digital CMOS camera |
| Oregon Green-DHPE | Invitrogen | O12650 | Oregon Green 488 1,2-Dihexadecanoyl-sn-Glycero-3-Phosphoethanolamine |
| Paclitaxel | ThermoFisher | P3456 | Paclitaxel (Taxol Equivalent) - for use in research only |
| PIPES | Sigma-Aldrich | P6757 | 1,4-Piperazinediethanesulfonic acid, Piperazine-1,4-bis(2-ethanesulfonic acid), Piperazine-N,N′-bis(2-ethanesulfonic acid) |
| Texas Red-DHPE | Invitrogen | T1395MP | Texas Red 1,2-Dihexadecanoyl-sn-Glycero-3-Phosphoethanolamine, Triethylammonium Salt |
| Trolox | Sigma-Aldrich | 238813 | (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid |
| Tubulin, Biotin | Cytoskeleton | T333P | Tubulin protein (biotin) porcine brain |
| Tubulin, Hy-Lite 488 | Cytoskeleton | TL488M | Tubulin protein (fluorescent HiLyte 488) porcine brain |
| Tubulin, Unlabeled | Cytoskeleton | T240 | Tubulin protein porcine brain |
Referencias
- Bouxsein, N. F., Carroll-Portillo, A., Bachand, M., Sasaki, D. Y., Bachand, G. D. A continuous network of lipid nanotubes fabricated from the gliding motility of kinesin powered microtubule filaments. Langmuir. 29 (9), 2992-2999 (2013).
- Paxton, W. F., Bouxsein, N. F., Henderson, I. M., Gomez, A., Bachand, G. D. Dynamic assembly of polymer nanotube networks via kinesin powered microtubule filaments. Nanoscale. 7 (25), 10998-11004 (2015).
- Leduc, C., et al. Cooperative extraction of membrane nanotubes by molecular motors. Proceedings of the National Academy of Sciences of the United States of America. 101 (49), 17096-17101 (2004).
- Lippincott-Schwartz, J., Roberts, T. H., Hirschberg, K. Secretory protein trafficking and organelle dynamics in living cells. Annual Review of Cell and Developmental Biology. 16, 557-589 (2000).
- Belting, M., Wittrup, A. Nanotubes, exosomes, and nucleic acid-binding peptides provide novel mechanisms of intercellular communication in eukaryotic cells: implications in health and disease. Journal of Cell Biology. 183 (7), 1187-1191 (2008).
- Rustom, A., Saffrich, R., Markovic, I., Walther, P., Gerdes, H. H. Nanotubular highways for intercellular organelle transport. Science. 303 (5660), 1007-1010 (2004).
- Onfelt, B., Nedvetzki, S., Yanagi, K., Davis, D. M. Cutting edge: Membrane nanotubes connect immune cells. Journal of Immunology. 173 (3), 1511-1513 (2004).
- Sciaky, N., et al. Golgi tubule traffic and the effects of brefeldin A visualized in living cells. J Cell Biol. 139 (5), 1137-1155 (1997).
- Sprong, H., van der Sluijs, P., van Meer, G. How proteins move lipids and lipids move proteins. Nature Reviews Molecular Cell Biology. 2 (7), 504-513 (2001).
- Keenan, T. W., Morre, D. J. Phospholipid class and fatty acid composition of golgi apparatus isolated from rat liver and comparison with other cell fractions. Biochemistry. 9 (1), 19-25 (1970).
- Imam, Z. I., Bachand, G. D. Multicomponent and Multiphase Lipid Nanotubes Formed by Gliding Microtubule-Kinesin Motility and Phase-Separated Giant Unilamellar Vesicles. Langmuir. 35 (49), 16281-16289 (2019).
- Wesolowska, O., Michalak, K., Maniewska, J., Hendrich, A. B. Giant unilamellar vesicles - a perfect tool to visualize phase separation and lipid rafts in model systems. Acta Biochimica Polonica. 56 (1), 33-39 (2009).
- Momin, N., et al. Designing lipids for selective partitioning into liquid ordered membrane domains. Soft Matter. 11 (16), 3241-3250 (2015).
- Fygenson, D. K., Braun, E., Libchaber, A. Phase diagram of microtubules. Physical Review E. 50, 1579(1994).
- Greene, A. C., Sasaki, D. Y., Bachand, G. D. Forming Giant-sized Polymersomes Using Gel-assisted Rehydration. Journal of Visualized Experiments. (111), (2016).
- Bachand, M., et al. Directed self-assembly of 1D microtubule nano-arrays. Royal Society of Chemistry Advances. 4 (97), 54641-54649 (2014).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados