È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Fabbricazione di reti di nanotubi lipidici multicomponenti utilizzando il test di motilità di Kinesin gliding
In questo articolo
Riepilogo
Questo protocollo descrive un processo per fabbricare reti di nanotubi lipidici utilizzando la motilità della kinesina planante in combinazione con vescicole lipidiche unilamellari giganti.
Abstract
Le reti di nanotubi lipidici (LNT) rappresentano un sistema modello in vitro per lo studio del trasporto molecolare e della biofisica lipidica con rilevanza per i tubuli lipidici ubiquitari presenti nelle cellule eucariotiche. Tuttavia, gli LNT in vivo sono strutture altamente in equilibrio che richiedono energia chimica e motori molecolari per essere assemblati, mantenuti e riorganizzati. Inoltre, la composizione degli LNT in vivo è complessa, comprendente più specie lipidiche diverse. I metodi tipici per estrudere gli LNT sono sia dispendiosi in termini di tempo che di lavoro e richiedono pinzette ottiche, microsfere e micropipette per estrarre forzatamente nanotubi da vescicole lipidiche giganti. Qui viene presentato un protocollo per il test di motilità planante (GMA), in cui le reti LNT su larga scala vengono rapidamente generate da vescicole unilamellari giganti (GUV) utilizzando la motilità dei microtubuli alimentati da chinesina. Utilizzando questo metodo, le reti LNT sono formate da una vasta gamma di formulazioni lipidiche che imitano la complessità degli LNT biologici, rendendoli sempre più utili per studi in vitro di biofisica lipidica e trasporto associato alla membrana. Inoltre, questo metodo è in grado di produrre in modo affidabile reti LNT in breve tempo (<30 min) utilizzando apparecchiature di laboratorio comunemente utilizzate. Anche le caratteristiche della rete LNT come lunghezza, larghezza e partizionamento lipidico sono sintonizzabili alterando la composizione lipidica dei GUV utilizzati per fabbricare le reti.
Introduzione
La fabbricazione di reti di nanotubi lipidici (LNT) è di crescente interesse per l'esame in vitro delle strutture lipidiche non equivalenti 1,2,3. Le cellule utilizzano tubuli lipidici per il trasporto diffusivo delle proteine4 e degli acidi nucleici5 e per la comunicazione cellula-cellula 6,7. Il reticolo endoplasmatico e l'apparato di Golgi sono particolarmente interessanti, in quanto questi organelli legati alla membrana sono le posizioni primarie per la sintesi lipidica e proteica, nonché per il trasporto di queste biomolecole integrali all'interno del citoplasma di una cellula 8,9. Le membrane di questi organelli sono composte da più specie lipidiche tra cui sfingolipidi, colesterolo e fosfolipidi10 che alla fine aiutano a definire la loro funzionalità. Pertanto, per replicare e studiare più da vicino questi organelli, gli LNT in vitro devono essere fabbricati da vescicole con formulazioni lipidiche sempre più complesse11.
Le vescicole unilamellari giganti (GUV) sono utilizzate in modo pervasivo per studiare il comportamento della membrana lipidica perché possono essere sintetizzate in modo affidabile con formulazioni complesse che includono colesterolo, fosfatidilcolina (PC), fosfatidiletanolammina (PE), fosfatidilserina (PS) e fosfatidilinositolo (PI) 12,13. Qui è descritto un metodo per fabbricare LNT da GUV con diverse formulazioni lipidiche utilizzando il test di motilità planante (GMA), in cui gli LNT vengono estrusi in base al lavoro svolto dai motori kinesin e dai filamenti di microtubuli che agiscono sui VAV. In questo sistema, le proteine motorie della chinesina adsorbite in superficie spingono microtubuli biotinilati, convertendo l'energia chimica dall'idrolisi dell'ATP in lavoro utile (in particolare, l'estrusione di LNT da vescicole biotinilate)11. La rete LNT risultante fornisce una piattaforma modello per studiare gli effetti delle differenze nelle fasi lipidiche sui cambiamenti nella morfologia LNT.
In breve, le proteine motorie della chinesina vengono introdotte in una camera di flusso in una soluzione contenente caseina, che consente l'adsorbimento dei motori sulla superficie di vetro della camera. Successivamente, i microtubuli biotinilati in una soluzione contenente ATP fluiscono attraverso la camera e vengono lasciati legare ai motori della kinesina e iniziare la motilità. Una soluzione di streptavidina viene quindi introdotta nella camera e lasciata legare in modo non covalente ai microtubuli. Infine, i GUV contenenti un lipide biotinilato vengono introdotti nella camera e si legano ai microtubuli rivestiti di streptavidina, quindi estrudono gli LNT per formare reti su larga scala nel corso di 15-30 minuti. Questo metodo produce grandi reti LNT ramificate utilizzando apparecchiature di laboratorio standard e reagenti a basso costo11.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Preparazione di soluzioni di microtubuli di riserva
ATTENZIONE: occhiali di sicurezza, guanti e un camice da laboratorio devono essere sempre indossati durante tutto il protocollo.
- Preparare 5x tampone BRB80: aggiungere 24,19 g di PIPES (piperazina-N,N′-bis[acido 2-etansolfonico]) e 0,38 g di EGTA (glicole etilenico-bis[β-aminoetiletere]-N,N,N′,N′-acido tetraacetico) in un flacone di vetro da 1 L. Aggiungere 1 mL di 1 M MgCl2 e regolare il pH a 6,9 con KOH. Aggiungere acqua deionizzata per portare la soluzione a un volume finale di 500 ml.
- Preparare 100 mM di stock di soluzione GTP: pesare 52 mg di GTP e sospendere in 1 mL di acqua distillata. Dividere la soluzione da 100 mM in aliquote da 20 μL e conservare a -20 °C.
- Preparare la soluzione GPEM: mescolare 200 μL di 5x BRB80, 10 μL di soluzione GTP da 100 mM, 100 μL di glicerolo al 100% e 600 μL di acqua deionizzata. Dividere la soluzione GPEM in aliquote da 100 μL e conservare a -20 °C.
- Preparare la soluzione di microtubuli ricostituendo flaconcini di tubulina liofilizzata disponibile in commercio (un flaconcino ciascuno di biotinilato, marcato fluorescentemente e non etichettato) in soluzione GPEM fredda (4 °C) ad una concentrazione di stock di 5 mg/ml.
- Eseguire la polimerizzazione dei microtubuli mescolando 4 μL di tubulina biotinilata, 4 μL di tubulina marcata fluorescentemente e 24 μL di tubulina non etichettata (tutti a concentrazioni di 5 mg / mL) per creare un rapporto di 1: 1: 6 a un volume finale di 32 mL. Tienilo sul ghiaccio. Dividere la miscela di tubuline in aliquote da 2 μL e conservare a -80 °C fino a quando necessario.
NOTA: una polimerizzazione efficiente richiede che la concentrazione di tubulina sia uguale o superiore alla concentrazione critica (5 mg/mL)14. Qui, la selezione del rapporto di tubulina è ottimizzata per una concentrazione sufficiente di tubulina biotinilata per legare in modo efficiente streptavidina e VAV, nonché una concentrazione sufficiente di tubulina fluorescente per la caratterizzazione microscopica.
2. Preparazione di vescicole unilamellari giganti (GUV)
- Preparazione del film di agarosio
NOTA: Questo protocollo è adattato da Greene et al.15.- Preparare una soluzione all'1% p/v mescolando 1 g di agarosio in 100 mL di acqua deionizzata in un matraccio Erlenmeyer da 250 ml. Utilizzare un forno a microonde standard per riscaldare la soluzione di agarosio per 1-2 minuti.
NOTA: La soluzione diventerà traslucida una volta che l'agarosio sarà completamente dissolto. Lasciare raffreddare la soluzione a 65-75 °C prima dell'uso. - Utilizzare una punta di pipetta da 1.000 μL tagliata per pipettare 300-400 μL di soluzione di agarosio su una slitta di vetro da 25 mm x 25 mm. Mentre si tiene il bordo del coperchio con le dita guantate, utilizzare un'altra punta della pipetta da 1.000 μL per distribuire uniformemente l'agarosio fuso attraverso il coperchio.
NOTA: il mantenimento dell'agarosio a 65-75 °C consentirà una diffusione efficiente sulla superficie del coperchio. - Asciugare le coperture rivestite di agarosio in un incubatore a 37 °C per almeno 2 ore, a quel punto l'agarosio diventerà trasparente. Conservare le coperture posizionando la superficie rivestita di agarosio rivolta verso l'alto su una superficie pulita come carta priva di lanugine o pellicola a base di cera a temperatura ambiente (RT).
- Preparare una soluzione all'1% p/v mescolando 1 g di agarosio in 100 mL di acqua deionizzata in un matraccio Erlenmeyer da 250 ml. Utilizzare un forno a microonde standard per riscaldare la soluzione di agarosio per 1-2 minuti.
- Formulazione lipidica
- Recuperare i lipidi disciolti in cloroformio da un congelatore a -20 °C e metterli in una cappa chimica fino a raggiungere RT.
- Calcolare il volume di riserva lipidica di ciascun lipide componente necessario per la formulazione utilizzando la seguente formula:
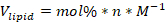
Dove: Vlipid è il volume di lipidi da utilizzare, mol % è la percentuale molare della componente lipidica, n è il numero totale di moli di lipidi utilizzati nella formulazione e M è la concentrazione di lipidi in unità molari.
NOTA: Ad esempio, se si utilizza una formulazione contenente 45 mol% 1,2-dioleoil-sn-glicero-3-fosfocolina (DOPC) con una concentrazione di soluzione madre di 12,72 mM e 1 micromolo di lipidi totali nella formulazione, il volume di stock DOPC utilizzato nella formulazione sarebbe: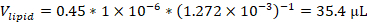
- Mescolare i lipidi insieme al rapporto calcolato in una fiala di vetro nella cappa chimica dei fumi.
- Pipettare 30 μL di soluzione lipidica sulle coperture rivestite di agarosio su una piastra calda preriscaldata posta al di sopra del punto di fusione del componente lipidico saturo della formulazione (ad esempio, piastra calda a 50 °C per un lipide con Tm di 40 °C).
- Distribuire la soluzione attraverso il film di agarosio con un movimento circolare usando il bordo lungo di un ago da 18 G fino a quando il cloroformio è evaporato e si è formato uno strato uniforme di lipidi. Tenere premuto il bordo del coperchio con le dita guantate durante l'esecuzione di questo passaggio.
NOTA: Bisogna fare attenzione a non danneggiare lo strato di agarosio con l'ago. - Posizionare la copertura con strato di agarosio e film lipidico in una capsula di Petri ricoperta di foglio di alluminio, lato del film rivolto verso l'alto e posizionare la capsula di Petri in un essiccatore sottovuoto per almeno 2 ore per rimuovere il solvente residuo.
- Nel frattempo, preparare una soluzione di saccarosio da 560 mM mescolando 1,92 g di saccarosio con 10 ml di acqua deionizzata.
NOTA: La concentrazione della soluzione di saccarosio dipende dall'osmolarità del tampone in cui i CAV sono diluiti. Tipicamente, l'osmolarità della soluzione di saccarosio non deve essere superiore del 10% rispetto al tampone in cui verranno diluiti i GUV, in particolare il tampone di motilità (vedere il punto 3.11). - Formazione di GUV
- Far aderire una camera adesiva al coperchio rivestito con il film lipidico premendo delicatamente la camera adesiva sul coperchio rivestito di lipidi con il film lipidico rivolto verso l'alto, assicurandosi che si formi una tenuta ermetica.
- Aggiungere 400 μL di soluzione di saccarosio da 560 mM (preparata al punto 2.3) alla camera.
- Posizionare il coperchio nella camera di umidità e chiudere il coperchio.
- Posizionare la camera di umidità su una piastra calda preriscaldata posta sopra il punto di fusione del componente lipidico saturo della formulazione.
- Lasciare formare le vescicole per ≥1 ora prima del recupero.
NOTA: La formazione di vescicole può essere controllata con microscopia a fluorescenza con una lente ad aria ottica 40x.
3. Preparazione delle scorte e dei reagenti per il saggio di motilità
- Preparazione dello stock di caseina
- Aggiungere 3 g di caseina secca in un tubo conico da 50 mL per centrifuga, quindi aggiungere 30 mL di 1x BRB80 e ruotare fino a quando la soluzione diventa viscosa. Centrifugare il tubo a 15.000 x g per 30 min.
- Trasferire il surnatante in un altro tubo centrifugo conico da 50 ml ed eliminare il pellet. Filtrare la soluzione attraverso un filtro a siringa da 1 μm, raccogliendo la soluzione in un flaconcino conico da 50 mL. Filtrare la soluzione attraverso un filtro da 0,2 μm, raccogliendo la soluzione in un flaconcino conico da 50 ml.
- Determinare la concentrazione proteica misurando l'assorbanza a 280 nm utilizzando uno spettrofotometro UV-Vis e una cuvetta di quarzo.
- Calcolare la concentrazione di caseina in mg/mL utilizzando la seguente formula:
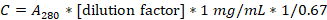
- Diluire la soluzione a 20 mg/mL in 1x BRB80, dividere in aliquote da 100 μL e conservare a -20 °C.
- Preparare la soluzione madre di glucosio ossidasi (2 mg/mL) mescolando 2 mg di glucosio ossidasi con 1 mL di 1x BRB80. Dividere in aliquote da 100 μL e conservare a -20 °C.
- Preparare la soluzione madre di catalasi (0,8 mg/ml) mescolando 0,8 mg di catalasi con 1 mL di 1x BRB80. Dividere in aliquote da 100 μL e conservare a -20 °C.
- Preparare la soluzione di glucosio 2 M sospendendo 0,3 g di D-glucosio in 1 mL di acqua deionizzata. Dividere in aliquote da 100 μL e conservare a -20 °C.
- Preparare 100 mM di stock DTT sospendendo 0,015 g di DTT in 1 mL di acqua deionizzata. Dividere in aliquote da 100 μL e conservare a -20 °C.
- Preparare 100 mM di Mg-ATP di stock sospendendo 0,055 g di ATP disodico in 1 mL di soluzione da 100 mM MgCl2. Dividere in aliquote da 100 μL e conservare a -20 °C.
- Preparare la soluzione mg-AMP-PNP da 100 mM sospendendo 0,055 g di AMP-PNP in una soluzione da 1 mL da 100 mM MgCl2, quindi dividere in aliquote da 100 μL e conservare a -20 °C.
- Preparare la soluzione di Trolox da 100 mM aggiungendo 25,03 mg di Trolox a 1 mL di metanolo e conservare a -20 °C.
- Preparare 10 mg/mL di soluzione di streptavidina aggiungendo 1 mg di streptavidina a 100 μL di BRB80, quindi dividere in aliquote da 2 μL e conservare a -80ۛ °C.
- Preparare BRB90CAT mescolando 200 μL di 5x BRB80, 20 μL di soluzione di caseina, 10 μL di soluzione di MgATP, 10 μL di Trolox, 5 μL di soluzione di paclitaxel e 765 μL di acqua DI. Conservare a 4 °C.
- Preparare la soluzione di motilità mescolando 192 μL di BRB80CAT, 2 μL di soluzione di D-glucosio, 2 μL di soluzione di glucosio ossidasi, 2 μL di soluzione di DTT e 2 μL di soluzione di catalasi. Conservare a 4 °C.
- Preparare una soluzione di kinesina da 1 μM diluendo la soluzione di kinesina madre in BRB80CAT (ad esempio, 2 μL di 50 mM di soluzione di kinesina in 98 μL di BRB80CAT) e conservare a 4 °C.
- Preparare una soluzione di microtubuli da 10 μg/mL diluendo 10 μL di microtubuli stabilizzati in 90 μL di BRB80CAT a temperatura ambiente. Negozio presso RT.
- Preparare una soluzione di streptavidina da 10 μg/mL aggiungendo 0,1 μL di 10 mg/mL di soluzione di streptavidina in 99,9 μL di soluzione di motilità. Conservare a 4 °C.
- Preparare una soluzione di GUV 12x diluendo 5 μL di stock di GUV in 55 μL di soluzione di motilità. Conservare a 4 °C.
- Polimerizzazione della tubulina in microtubuli
- Raccogliere un'aliquota di tubulina di 2 μL precedentemente preparata dal congelatore a -80 °C (preparato al punto 1.5) e metterla in un bagno d'acqua a 37 °C per 30 minuti.
- Preparare una soluzione di paclitaxel da 2 mM aggiungendo 1,71 mg di paclitaxel in 1 mL di DMSO anidro, dividere in aliquote da 10 μL e conservare a -20 °C.
- Preparare al momento BRB80T mescolando 99,5 μL di 1x BRB80 con 0,5 μL di 2 mM paclitaxel. Riscaldare da 100 μL di BRB80T a 37 °C a bagnomaria.
- Dopo 30 minuti, aggiungere i 100 μL di BRB80T all'aliquota di tubulina per stabilizzare i microtubuli. Negozio presso RT.
4. Saggio di motilità planante (GMA)
- Preparare una camera di flusso apponendo due strisce di nastro biadesivo separate da 5 mm su una diapositiva di vetro. Ripetere questo processo fino a quando tre strati di nastro comprendono ogni striscia.
- Posizionare una coverslip sulla parte superiore del nastro, quindi premere delicatamente sull'interfaccia coverslip/nastro con una pinzetta o una penna per garantire un'adesione sufficiente.
NOTA: il canale deve avere una larghezza di 5 mm per 25 mm di lunghezza per 300 mm di altezza. - Pipettare 30 μL di soluzione di kinesina da 1 mm (preparata nel passaggio 3.12) nella cella di flusso e lasciarla incubare per 5 minuti.
NOTA: La caseina forma un doppio strato sulla superficie del coperchio/vetrino e facilita l'aggancio della coda di kinesina alla superficie. - Pipettare 30 μL di soluzione di microtubulo da 10 μg/mL (preparata nella fase 3.13) nella cella di flusso, utilizzando una salvietta da laboratorio premuta delicatamente contro l'estremità opposta del canale di flusso per facilitare lo scambio di soluzione. Incubare per 5 min.
- Lavare la cella di flusso 1x–3x con 1x soluzione di motilità a RT (preparata al punto 3.11).
NOTA: La microscopia a fluorescenza che utilizza un obiettivo aereo 40x può essere utilizzata a questo punto per confermare l'attaccamento e la motilità dei microtubuli. I microtubuli appaiono come filamenti fluorescenti (decine di micron di lunghezza) che si muovono (scivolano) attraverso la superficie a ~ 0,5 μm / s (Figura 1). - Pipettare 30 μL di soluzione di streptavidina da 10 μg/mL (preparata nella fase 3.14) nella cella di flusso utilizzando una salvietta da laboratorio premuta delicatamente contro l'estremità opposta del canale di flusso per facilitare lo scambio della soluzione. Incubare per 10 min.
- Flusso 30 μL di soluzione 12x GUV (preparata nella fase 3.15) nel flusso utilizzando una salvietta da laboratorio premuta delicatamente contro l'estremità opposta del canale di flusso per facilitare lo scambio di soluzioni. Incubare per 30 min.
- Aggiungere 2 μL di soluzione AMP-PNP da 100 mM (preparata al punto 3.7) per arrestare la motilità, quindi sigillare la camera con sigillante.
5. Caratterizzazione della rete LNT
- Trasferire la camera di flusso su un microscopio invertito per l'imaging.
- Scegli il set di filtri appropriato in base alle lunghezze d'onda dei lipidi fluorescenti o della tubulina utilizzati. Ad esempio, quando si utilizzano lipidi marcati in rosso del Texas, utilizzare un filtro di eccitazione a 560 nm / 25 nm e un filtro di emissione a 607 nm / 36 nm.
- Utilizzare un obiettivo olio 100x per concentrarsi sulla superficie del coverslip.
- Immagine delle reti LNT utilizzando la microscopia a fluorescenza.
NOTA: gli LNT sono strutture lineari che estrudono dalle vescicole più grandi. Gli LNT sono molto più piccoli dei GUV e hanno segnali di fluorescenza più deboli. Pertanto, l'esposizione e il contrasto devono essere regolati di conseguenza agli LNT dell'immagine. Queste regolazioni portano anche alla sovraesposizione dei VAV e, pertanto, si raccomanda che la lamellarità e la separazione di fase nei VVV siano caratterizzate in modo indipendente. - Focalizzare il microscopio su una rete di interesse e prendere un'immagine standard o piastrellata.
- Regolare il tempo di esposizione e i filtri a densità neutra per visualizzare l'immagine degli LNT e ridurre al minimo l'esposizione satura dei GUV. Acquisisci immagini sia nei canali rosso che verde.
NOTA: Qui, il canale rosso consente la visualizzazione dei lipidi Texas-Red, mentre il canale verde consente la visualizzazione dei microtubuli (ad esempio, lipidi Oregon Green e coloranti HiLyte488). - Creare un'immagine composita sovrapponendo i canali rosso e verde (Figura 1).
- Caratterizzazione di reti LNT mediante la misurazione della lunghezza LNT
- Aprire le immagini acquisite utilizzando un software di analisi delle immagini come ImageJ.
- Calibrate la scala per il microscopio utilizzando la funzione di scala impostata, riempite i pixel fino al fattore di conversione mm e fate clic su OK.
NOTA: il fattore di conversione dipende dal microscopio, dall'obiettivo e dalla fotocamera e può essere ottenuto utilizzando un vetrino di calibrazione del microscopio. È generalmente espresso in pixel/mm. - Utilizzate lo strumento Linea multipunto per misurare la lunghezza dei nanotubi a partire dal GUV padre. Tenere premuto Ctrl +M per misurare la lunghezza.
- Continuare a misurare le lunghezze dei singoli tubi seguendo i passaggi precedenti. Lo strumento di elaborazione delle immagini salverà ogni nuova misurazione nella finestra dei risultati.
- Tenere premuto Ctrl + D dopo aver disegnato ogni linea per tenere traccia di quali tubi sono stati misurati.
- Misurazione dello spessore LNT (Figura 2)
- Aprire l'immagine in ImageJ e selezionare la funzione Soglia nella scheda Immagine .
- Fare clic su Applica per applicare la soglia.
- Disegna un rettangolo di lunghezza nota sul tubo desiderato (i pixel neri hanno un valore di 0 e i pixel rossi hanno un valore di 255).
- Misurare la densità integrata dell'area.
- Dividere la densità per la lunghezza (in pixel) dell'LNT per ottenere lo spessore (in pixel).
NOTA: le misurazioni dello spessore possono essere confrontate tra le immagini solo quando le impostazioni di imaging e la soglia sono impostate in modo identico.
- Determinazione del partizionamento lipidico nei nodi degli LNT (Figura 3)
- Aprire l'immagine in ImageJ.
- Utilizzate lo strumento linea per disegnare una linea sul nodo desiderato.
- Misurare l'intensità del nodo in entrambi i canali Oregon Green e Texas Red.
- Spostare la linea sul LNT e misurare l'intensità LNT in entrambi i canali Oregon Green e Texas Red.
Access restricted. Please log in or start a trial to view this content.
Risultati
Le reti LNT (Figura 4) sono state fabbricate utilizzando il protocollo descritto, che utilizza il lavoro svolto dal trasporto kinesin di microtubuli per estrudere LNT dai GUV. In breve, i VAV sono stati preparati utilizzando la reidratazione del gel di agarosio utilizzando soluzione di saccarosio e i microtubuli sono stati polimerizzati in soluzione GPEM e stabilizzati in BRB80T. Successivamente, i motori kinesin sono stati introdotti in una cella di flusso formando uno strato attivo di moto...
Access restricted. Please log in or start a trial to view this content.
Discussione
Le reti LNT sono uno strumento utile per studi in vitro sulle proprietà della membrana e sul trasporto di biomolecole come le proteine transmembrana. Inoltre, l'utilizzo di formulazioni lipidiche complesse per fabbricare reti LNT consente studi più rilevanti dal punto di vista biologico. Altri studi di fabbricazione hanno utilizzato 1) semplici formulazioni lipidiche e vescicole multilamellari o 2) tecniche di motilità più ingombranti per fabbricare reti da GUV composte da formulazioni lipidiche complesse. Il metodo ...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Sandia National Laboratories è un laboratorio multi-missione gestito e operato da National Technology & Engineering Solutions di Sandia, LLC., una consociata interamente controllata da Honeywell International, Inc., per la National Nuclear Security Administration del DOE degli Stati Uniti sotto contratto DE-NA-0003525. Questo documento descrive risultati tecnici oggettivi e analisi. Eventuali opinioni o opinioni soggettive che potrebbero essere espresse nel documento non rappresentano necessariamente le opinioni del Dipartimento dell'Energia degli Stati Uniti o del Governo degli Stati Uniti.
Riconoscimenti
Questo lavoro è stato supportato dal Dipartimento dell'Energia degli Stati Uniti, Office of Basic Energy Sciences, Division of Materials Sciences and Engineering (BES-MSE). La sintesi della kinesina e la microscopia a fluorescenza sono state eseguite attraverso un progetto utente (ZIM) presso il Center for Integrated Nanotechnologies, una struttura per utenti dell'Office of Science gestita per l'Office of Science del Dipartimento dell'Energia degli Stati Uniti (DOE).
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 100x/1.4 Numerical Aperture Oil Immersion Objective | Olympus | 1-U2B836 | Olympus UPlanSApo 100x/1.40 Oil Objective Infinity Corrected, RMS Thread Working Distance 0.12mm |
| 3.0 ND Filter | Olympus | Neutral Density Filter | |
| AMP-PNP | Sigma-Aldrich | A2647 | (β,γ-imidoadenosine 5′-triphosphate) |
| ATP | Sigma-Aldrich | A7699 | Adenosine 5'-triphosphate disodium salt hydrate BioXtra |
| Brightline Pinkel DA/FI/TR/Cy5/Cy7-5X-A000 filter set | Semrock | LED-DA/FI/TR/Cy5/Cy7-5X-A-000 | BrightLine Pinkel filter set, optimized for DAPI, FITC, TRITC, Cy5 & Cy7 and other like fluorophores, illuminated with LED-based light sources |
| Casein | Sigma-Aldrich | 22090 | Casein hydrolysate for microbiology |
| Catalase | Sigma-Aldrich | C9322 | Catalase from Bovine Liver |
| Chloroform | Sigma-Aldrich | 288306 | Chloroform anhydrous contains 0.5-1.0% ethanol as stabilizer |
| Cholesterol | Avanti | 700000P | cholesterol (ovine wool, >98%) (powder) |
| D-Glucose | Sigma-Aldrich | G7021 | D-(+)-Glucose powder, BioReagent, suitable for cell culture, suitable for insect cell culture, suitable for plant cell culture, ≥99.5% |
| DOPC | Avanti | 850375C | 1,2-Dioleoyl-sn-glycero-3-phosphocholine (in chloroform) |
| DOPE-Biotin | Avanti | 870282C | 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine-N-(biotinyl) (sodium salt) |
| DPPC | Avanti | 850355P | 1,2-dipalmitoyl-sn-glycero-3-phosphocholine (powder) |
| DPPE-Biotin | Avanti | 870285P | 1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(biotinyl) (sodium salt) |
| DTT | Sigma-Aldrich | 43816 | DL-Dithiothreitol solution 1 M |
| EGTA | Sigma-Aldrich | E4378 | EGTA, Egtazic acid, Ethylene-bis(oxyethylenenitrilo)tetraacetic acid, Glycol ether diamine tetraacetic acid |
| Glucose Oxidase | Sigma-Aldrich | G6125 | Glucose Oxidase from Aspergillus niger Type II, ≥10,000 units/g solid (without added oxygen) |
| Glycerol | Fisher | G33 | Glycerol (Certified ACS), Fisher Chemical |
| GTP | Sigma-Aldrich | G8877 | Guanosine 5′-triphosphate sodium salt hydrate |
| IX-81 Olympus Microscope | Olympus | N/A | IX81 Inverted Microscope from Olympus |
| KOH | Sigma-Aldrich | 1050121000 | Potassium Hydroxide |
| Magnesium Chloride | Sigma-Aldrich | M1028 | 1.00 M magnesium chloride solution |
| Orca Flash 4.0 Digital Camera | Hamamatsu | C13440-20CU | ORCA-Flash 4.0 V3 Digital CMOS camera |
| Oregon Green-DHPE | Invitrogen | O12650 | Oregon Green 488 1,2-Dihexadecanoyl-sn-Glycero-3-Phosphoethanolamine |
| Paclitaxel | ThermoFisher | P3456 | Paclitaxel (Taxol Equivalent) - for use in research only |
| PIPES | Sigma-Aldrich | P6757 | 1,4-Piperazinediethanesulfonic acid, Piperazine-1,4-bis(2-ethanesulfonic acid), Piperazine-N,N′-bis(2-ethanesulfonic acid) |
| Texas Red-DHPE | Invitrogen | T1395MP | Texas Red 1,2-Dihexadecanoyl-sn-Glycero-3-Phosphoethanolamine, Triethylammonium Salt |
| Trolox | Sigma-Aldrich | 238813 | (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid |
| Tubulin, Biotin | Cytoskeleton | T333P | Tubulin protein (biotin) porcine brain |
| Tubulin, Hy-Lite 488 | Cytoskeleton | TL488M | Tubulin protein (fluorescent HiLyte 488) porcine brain |
| Tubulin, Unlabeled | Cytoskeleton | T240 | Tubulin protein porcine brain |
Riferimenti
- Bouxsein, N. F., Carroll-Portillo, A., Bachand, M., Sasaki, D. Y., Bachand, G. D. A continuous network of lipid nanotubes fabricated from the gliding motility of kinesin powered microtubule filaments. Langmuir. 29 (9), 2992-2999 (2013).
- Paxton, W. F., Bouxsein, N. F., Henderson, I. M., Gomez, A., Bachand, G. D. Dynamic assembly of polymer nanotube networks via kinesin powered microtubule filaments. Nanoscale. 7 (25), 10998-11004 (2015).
- Leduc, C., et al. Cooperative extraction of membrane nanotubes by molecular motors. Proceedings of the National Academy of Sciences of the United States of America. 101 (49), 17096-17101 (2004).
- Lippincott-Schwartz, J., Roberts, T. H., Hirschberg, K. Secretory protein trafficking and organelle dynamics in living cells. Annual Review of Cell and Developmental Biology. 16, 557-589 (2000).
- Belting, M., Wittrup, A. Nanotubes, exosomes, and nucleic acid-binding peptides provide novel mechanisms of intercellular communication in eukaryotic cells: implications in health and disease. Journal of Cell Biology. 183 (7), 1187-1191 (2008).
- Rustom, A., Saffrich, R., Markovic, I., Walther, P., Gerdes, H. H. Nanotubular highways for intercellular organelle transport. Science. 303 (5660), 1007-1010 (2004).
- Onfelt, B., Nedvetzki, S., Yanagi, K., Davis, D. M. Cutting edge: Membrane nanotubes connect immune cells. Journal of Immunology. 173 (3), 1511-1513 (2004).
- Sciaky, N., et al. Golgi tubule traffic and the effects of brefeldin A visualized in living cells. J Cell Biol. 139 (5), 1137-1155 (1997).
- Sprong, H., van der Sluijs, P., van Meer, G. How proteins move lipids and lipids move proteins. Nature Reviews Molecular Cell Biology. 2 (7), 504-513 (2001).
- Keenan, T. W., Morre, D. J. Phospholipid class and fatty acid composition of golgi apparatus isolated from rat liver and comparison with other cell fractions. Biochemistry. 9 (1), 19-25 (1970).
- Imam, Z. I., Bachand, G. D. Multicomponent and Multiphase Lipid Nanotubes Formed by Gliding Microtubule-Kinesin Motility and Phase-Separated Giant Unilamellar Vesicles. Langmuir. 35 (49), 16281-16289 (2019).
- Wesolowska, O., Michalak, K., Maniewska, J., Hendrich, A. B. Giant unilamellar vesicles - a perfect tool to visualize phase separation and lipid rafts in model systems. Acta Biochimica Polonica. 56 (1), 33-39 (2009).
- Momin, N., et al. Designing lipids for selective partitioning into liquid ordered membrane domains. Soft Matter. 11 (16), 3241-3250 (2015).
- Fygenson, D. K., Braun, E., Libchaber, A. Phase diagram of microtubules. Physical Review E. 50, 1579(1994).
- Greene, A. C., Sasaki, D. Y., Bachand, G. D. Forming Giant-sized Polymersomes Using Gel-assisted Rehydration. Journal of Visualized Experiments. (111), (2016).
- Bachand, M., et al. Directed self-assembly of 1D microtubule nano-arrays. Royal Society of Chemistry Advances. 4 (97), 54641-54649 (2014).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon