É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Fabricando redes nanotubos lipídes de lipídicas multi-componentes usando o ensaio de motilidade de mobilidade de cinévola gliding
Neste Artigo
Resumo
Este protocolo descreve um processo para a fabricação de redes lipídicas de nanotubos usando motilidade de quinase deslizante em conjunto com vesículas lipídicas unilamellar gigantes.
Resumo
As redes lipídicas de nanotubos (LNT) representam um sistema de modelo in vitro para estudar o transporte molecular e a biofísica lipídica com relevância para os túbulos lipídicos onipresentes encontrados em células eucarióticas. No entanto, os LNTs in vivo são estruturas altamente não-equilibradas que requerem energia química e motores moleculares para serem montados, mantidos e reorganizados. Além disso, a composição dos In vivo LNTs é complexa, composta por várias espécies lipídicas diferentes. Métodos típicos para extrusão de LNTs são demorados e intensivos em mão-de-obra, e requerem pinças ópticas, microesferas e micropipettos para puxar à força nanotubos de vesículas lipídicas gigantes. Apresentado aqui é um protocolo para o ensaio de motilidade deslizante (GMA), no qual redes LNT em larga escala são rapidamente geradas a partir de vesículas unilamellar gigantes (GUVs) usando motilidade de microtúbula alimentada por cinesina. Usando este método, as redes LNT são formadas a partir de uma ampla gama de formulações lipídicas que imitam a complexidade dos LNTs biológicos, tornando-as cada vez mais úteis para estudos in vitro de biofísica lipídica e transporte associado à membrana. Além disso, este método é capaz de produzir redes LNT de forma confiável em um curto espaço de tempo (<30 min) utilizando equipamentos de laboratório comumente usados. Características da rede LNT, como comprimento, largura e particionamento lipíduo também são incapazes de alterar a composição lipídica dos GUVs usados para a fabricação das redes.
Introdução
A fabricação de redes lipídicas de nanotubos (LNT) é de crescente interesse pelo exame in vitro de estruturas lipídicas de noquilibrium 1,2,3. As células usam túbulos lipídios para o transporte difuso de proteínas4 e ácidos nucleicos5, bem como comunicação célula-celular 6,7. O ritúlume endoplasmático e o aparelho golgi são particularmente interessantes, pois essas organelas ligadas à membrana são os locais principais para síntese lipídica e proteica, bem como o transporte dessas biomoléculas integrais dentro do citoplasma de uma célula 8,9. As membranas dessas organelas são compostas de múltiplas espécies lipídicas, incluindo sphingolipids, colesterol e fosfolipídios10 que, em última análise, ajudam a definir sua funcionalidade. Assim, para replicar mais de perto e estudar essas organelas, os LNTs in vitro devem ser fabricados a partir de vesículas com formulações lipídicas cada vez mais complexas11.
Vesículas unilamellar gigantes (GUVs) são usadas de forma generalizada para estudar o comportamento da membrana lipídica porque podem ser sintetizadas de forma confiável com formulações complexas que incluem colesterol, fosfatidylcolina (PC), fosfatidyletanolamina (PE), fosfatidylserina (PS) e fosfatidylinositol (PI)12,13. Descrito aqui é um método para fabricar LNTs de GUVs com formulações lipídicas variadas usando o ensaio de motilidade deslizante (GMA), no qual os LNTs são extrudados com base no trabalho realizado por motores de cinesina e filamentos de microtúbulos que atuam em GUVs. Neste sistema, as proteínas motoras de cinesina adsorvidas a uma superfície propel microtúbulos biotinínos, convertendo energia química da hidrólise de ATP em trabalho útil (especificamente, a extrusão de LNTs de vesículas biotinína)11. A rede LNT resultante fornece uma plataforma modelo para estudar os efeitos das diferenças nas fases lipídicas sobre as alterações na morfologia LNT.
Resumidamente, as proteínas motoras da cinesina são introduzidas em uma câmara de fluxo em uma solução contendo caseína, que permite a adsorção dos motores na superfície de vidro da câmara. Em seguida, microtúbulos biotinínos em uma solução contendo fluxo ATP através da câmara e são autorizados a ligar-se aos motores de cinesina e começar a motilidade. Uma solução streptavidin é então introduzida na câmara e permitida a ligação não covalente aos microtúbulos. Finalmente, os GUVs contendo um lipídio biotinilatado são introduzidos na câmara e se ligam aos microtúbulos revestidos de streptavidin, em seguida, extrude LNTs para formar redes de grande escala ao longo de 15-30 min. Este método produz grandes redes LNT ramificadas utilizando equipamentos de laboratório padrão e reagentes a um baixo custo11.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Preparação de soluções de microtúbulos de estoque
ATENÇÃO: Óculos de segurança, luvas e um jaleco devem ser sempre usados em todo o protocolo.
- Prepare 5x tampão BRB80: adicione 24,19 g de PIPES (piperazine-N,N'-bis[2-ácido elfofônico]) e 0,38 g de EGTA (etileno glycol-bis[β-aminoetil éter]-N,N,N′,N′-tetraacético ácido) a uma garrafa de vidro 1 L. Adicione 1 mL de 1 M MgCl2 e ajuste o pH para 6,9 com KOH. Adicione água deionizada para levar a solução a um volume final de 500 mL.
- Prepare 100 mM de estoque de solução GTP: pese 52 mg de GTP e suspenda em 1 mL de água destilada. Divida a solução de 100 mM em alíquotas de 20 μL e armazene a -20 °C.
- Prepare a solução GPEM: misture 200 μL de 5x BRB80, 10 μL de solução GTP de 100 mM, 100 μL de 100% glicerol e 600 μL de água desionizada. Divida a solução GPEM em alíquotas de 100 μL e armazene em -20 °C.
- Prepare a solução de microtúbulo reconstituindo frascos de tubulina liofilizada comercialmente disponível (um frasco cada de biotinilato, rotulado fluorescente e sem rótulo) em solução GPEM fria (4 °C) para uma concentração de estoque de 5 mg/mL.
- Realize a polimerização de microtúbulos misturando 4 μL de tubulina biotinilada, 4 μL de tubulina fluorescente e 24 μL de tubulina sem rótulo (todas em concentrações de 5 mg/mL) para criar uma razão de 1:1:6 em um volume final de 32 mL. Mantenha-o no gelo. Divida a mistura de tubulina em 2 alíquotas de μL e armazene a -80 °C até que seja necessário.
NOTA: A polimerização eficiente requer que a concentração de tubulina seja igual ou superior à concentração crítica (5 mg/mL)14. Aqui, a seleção da razão tubulina é otimizada para uma tubulina biotinilada de concentração suficiente para ligar eficientemente streptavidina e GUVs, bem como uma concentração suficiente de tubulina fluorescente para caracterização microscópica.
2. Preparação de vesículas unilamellar gigantes (GUVs)
- Preparação do filme de Agarose
NOTA: Este protocolo é adaptado de Greene et al.15.- Prepare uma solução de 1% c/v misturando 1 g de agarose em 100 mL de água deionizada em frascos erlenmeyer de 250 mL. Use um micro-ondas padrão para aquecer a solução agarose por 1-2 min.
NOTA: A solução se tornará translúcida assim que a agarose estiver completamente dissolvida. Deixe a solução esfriar a 65-75 °C antes de usar. - Use uma ponta de pipeta de 1.000 μL cortada para pipeta de 300 a 400 μL de solução agarose em uma tampa de vidro de 25 mm x 25 mm. Enquanto segura a borda do deslizamento com os dedos enluvados, use outra ponta de pipeta de 1.000 μL para espalhar a agarose derretida uniformemente através da mancha de cobertura.
NOTA: Manter a agarose a 65-75 °C permitirá uma disseminação eficiente na superfície do deslizamento de tampas. - Seque as tampas revestidas de agarose em uma incubadora de 37 °C por pelo menos 2 h, momento em que a agarose se tornará transparente. Armazene as tampas colocando a superfície revestida de agarose voltada para cima em uma superfície limpa, como papel sem fiapos ou filme à base de cera à temperatura ambiente (RT).
- Prepare uma solução de 1% c/v misturando 1 g de agarose em 100 mL de água deionizada em frascos erlenmeyer de 250 mL. Use um micro-ondas padrão para aquecer a solução agarose por 1-2 min.
- Formulação lipídica
- Recupere lipídios dissolvidos em clorofórmio a partir de um congelador de -20 °C e coloque-os em uma capa de fumaça química até chegar ao RT.
- Calcule o volume de estoque lipídeto de cada lipídio componente necessário para a formulação usando a seguinte fórmula:
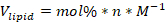
Onde: Vlipídio é o volume de lipídio para usar, mol % é a porcentagem molar do componente lipídulo, n é o número total de mols de lipídio usado na formulação, e M é a concentração de lipídio em unidades molares.
NOTA: Por exemplo, se usar uma formulação contendo 45 mol% 1,2-dioleoyl-sn-gliceo-3-fosfocholina (DOPC) com uma concentração de solução de estoque de 12,72 mM, e 1 micromole de lipídios totais na formulação, o volume de estoque DOPC usado na formulação seria: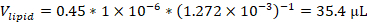
- Misture os lipídios na proporção calculada em um frasco de vidro na capa de fumaça química.
- Pipeta 30 μL de solução lipídica sobre as tampas revestidas de agarose em uma placa quente pré-aqueda que é definida acima do ponto de fusão do componente lipídudo saturado da formulação (por exemplo, placa quente de 50 °C para um lipídio com Tm de 40 °C).
- Espalhe a solução através do filme agarose em um movimento circular usando a longa borda de uma agulha de 18 G até que o clorofórmio tenha evaporado e uma camada uniforme de lipídio tenha se formado. Segure a borda da mancha com os dedos enluvados enquanto realiza esta etapa.
NOTA: Deve-se tomar cuidado para não danificar a camada de agarose com a agulha. - Coloque o deslizamento com camada de agarose e filme lipídico em uma placa de Petri coberta de papel alumínio, lado do filme voltado para cima, e coloque a placa de Petri em um desiccator de vácuo por pelo menos 2 h para remover solvente residual.
- Enquanto isso, prepare a solução de sacarose de 560 mM misturando 1,92 g de sacarose com 10 mL de água desionizada.
NOTA: A concentração da solução de sacarose depende da osmolaridade dos GUVs tampão serem diluídos. Normalmente, a osmolaridade da solução de sacarose não deve ser mais do que 10% maior do que o buffer em que os GUVs serão diluídos, especificamente o tampão de motilidade (ver passo 3.11). - Formação do GUV
- Adere a uma câmara adesiva ao deslizamento revestido com o filme lipíduo pressionando suavemente a câmara adesiva no deslizamento revestido de lipídio com o filme lipídudo voltado para cima, garantindo que uma vedação apertada seja formada.
- Adicione 400 μL de solução de sacarose de 560 mM (preparada na etapa 2.3) à câmara.
- Coloque a mancha na câmara de umidade e feche a tampa.
- Coloque a câmara de umidade em uma placa quente pré-aquelizada acima do ponto de fusão do componente lipídudo saturado da formulação.
- Permita que as vesículas se formem por ≥1 h antes da recuperação.
NOTA: A formação da vesícula pode ser verificada com microscopia de fluorescência com lente objetiva de ar de 40x.
3. Preparação de estoques de ensaios de motilidade e reagentes
- Preparação de estoque casein
- Adicione 3 g de caixa seca a um tubo de centrífuga cônica de 50 mL, depois adicione 30 mL de 1x BRB80 e gire até que a solução se torne viscosa. Centrifugar o tubo a 15.000 x g por 30 min.
- Transfira o supernatante para outro tubo de centrífuga cônica de 50 mL e descarte a pelota. Filtre a solução através de um filtro de seringa de 1 μm, coletando a solução em um frasco cônico de 50 mL. Filtre a solução através de um filtro de 0,2 μm, coletando a solução em um frasco cônico de 50 mL.
- Determine a concentração de proteína medindo a absorvância a 280 nm usando um espectrofotômetro UV-Vis e cuvette de quartzo.
- Calcule a concentração de caseína em mg/mL usando a seguinte fórmula:
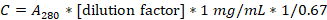
- Diluir a solução para 20 mg/mL em 1x BRB80, dividir em 100 alíquotas de μL e armazenar a -20 °C.
- Prepare a solução de estoque de glicose oxidase (2 mg/mL) misturando 2 mg de glicose oxidase com 1 mL de 1x BRB80. Divida em 100 alíquotas de μL e armazene a -20 °C.
- Prepare a solução de estoque catalase (0,8 mg/mL) misturando 0,8 mg de catalase com 1 mL de 1x BRB80. Divida em 100 alíquotas de μL e armazene a -20 °C.
- Prepare a solução de glicose de 2 M suspendendo 0,3 g de D-glicose em 1 mL de água deionizada. Divida em 100 alíquotas de μL e armazene a -20 °C.
- Prepare 100 mM DTT estoque suspendendo 0,015 g de DTT em 1 mL de água deionizada. Divida em 100 alíquotas de μL e armazene a -20 °C.
- Prepare 100 mM Mg-ATP ações suspendendo 0,055 g de disodium ATP em solução de 1 mL de 100 mM MgCl2. Divida em 100 alíquotas de μL e armazene a -20 °C.
- Prepare a solução Mg-AMP-PNP de 100 mM suspendendo 0,055 g de AMP-PNP em uma solução de 1 mL de 100 mM MgCl2, depois divida em alíquotas de 100 μL e armazene a -20 °C.
- Prepare a solução trolox de 100 mM adicionando 25,03 mg de Trolox a 1 mL de metanol e armazene a -20 °C.
- Prepare a solução streptavidin de 10 mg/mL adicionando 1 mg de streptavidina a 100 μL de BRB80, em seguida, divida em 2 alíquotas de μL e armazene a -80ۛ °C.
- Prepare o BRB90CAT misturando 200 μL de 5x BRB80, 20 μL de solução de caseína, 10 μL de solução MgATP, 10 μL de Trolox, 5 μL de solução paclitaxel e 765 μL de água DI. Armazenar a 4 °C.
- Prepare a solução de motilidade misturando 192 μL de BRB80CAT, 2 μL de solução D-glicose, 2 μL de solução de glicose oxidase, 2 μL de solução DTT e 2 μL de solução de catalase. Armazenar a 4 °C.
- Prepare uma solução de quinase de 1 μM diluindo a solução de quinase de estoque em BRB80CAT (por exemplo, 2 μL de solução de quinase de 50 mM em 98 μL de BRB80CAT) e armazene a 4 °C.
- Prepare uma solução de microtúbulo de 10 μg/mL diluindo 10 μL de microtúbulos estabilizados em 90 μL de temperatura ambiente BRB80CAT. Loja na RT.
- Prepare uma solução streptavidin de 10 μg/mL adicionando 0,1 μL de solução streptavidin de 10 mg/mL em 99,9 μL de solução de motilidade. Armazenar a 4 °C.
- Prepare uma solução DE 12x GUV diluindo 5 μL de estoque guv em 55 μL de solução de motilidade. Armazenar a 4 °C.
- Polimerização da tubulina em microtúbulos
- Colete uma alíquota de 2 μL previamente preparada de tubulina a partir do congelador -80 °C (preparado na etapa 1.5) e coloque em um banho de água de 37 °C por 30 min.
- Prepare uma solução de paclitaxel de 2 mM adicionando 1,71 mg de paclitaxel em 1 mL de DMSO anidro, divida em alíquotas de 10 μL e armazene a -20 °C.
- Prepare-se recentemente BRB80T misturando 99,5 μL de 1x BRB80 com 0,5 μL de paclitaxel de 2 mM. Aqueça 100 μL de BRB80T a 37 °C no banho de água.
- Após 30 min, adicione os 100 μL de BRB80T à alíquota da tubulina para estabilizar os microtúbulos. Loja na RT.
4. Ensaio de motilidade deslizante (GMA)
- Prepare uma câmara de fluxo afixando duas tiras de fita dupla face separadas por 5 mm em uma lâmina de vidro. Repita este processo até que três camadas de fita compõem cada tira.
- Coloque uma mancha de cobertura na parte superior da fita e pressione suavemente na interface de tinta/fita com uma pinça ou caneta para garantir a adesão suficiente.
NOTA: O canal deve ter 5 mm de largura por 25 mm de comprimento por 300 mm de altura. - Pipeta 30 μL de solução de quinase de 1 mm (preparada na etapa 3.12) para a célula de fluxo e deixá-la incubar por 5 min.
NOTA: A caixa forma uma bicamada na superfície do deslizamento de tampa/deslizamento de vidro e facilita a fixação da cauda da quinase à superfície. - Pipeta 30 μL de solução de microtúbulo de 10 μg/mL (preparada na etapa 3.13) para a célula de fluxo, utilizando uma limpeza de laboratório pressionada suavemente contra a extremidade oposta do canal de fluxo para facilitar a troca de soluções. Incubar por 5 minutos.
- Lave a célula de fluxo 1x-3x com solução de 1x motilidade em RT (preparada na etapa 3.11).
NOTA: A microscopia de fluorescência usando um objetivo de ar de 40x pode ser usada neste momento para confirmar o apego e a motilidade do microtúbulo. Microtúbulos aparecem como filamentos fluorescentes (dezenas de mícrons de comprimento) movendo-se (deslizando) pela superfície a ~0,5 μm/s (Figura 1). - Pipeta 30 μL de 10 μg/mL streptavidin solução (preparada na etapa 3.14) para a célula de fluxo usando um lenço de laboratório pressionado suavemente contra a extremidade oposta do canal de fluxo para facilitar a troca de soluções. Incubar por 10 minutos.
- Fluxo 30 μL de solução GUV de 12x (preparado na etapa 3.15) para o fluxo usando uma limpeza de laboratório pressionada suavemente contra a extremidade oposta do canal de fluxo para facilitar a troca de soluções. Incubar por 30 min.
- Adicione 2 μL de solução AMP-PNP de 100 mM (preparada na etapa 3.7) para parar a motilidade e, em seguida, selar a câmara com selante.
5. Caracterização da rede LNT
- Transfira a câmara de fluxo para um microscópio invertido para imagem.
- Escolha o conjunto de filtros apropriado com base nos comprimentos de onda dos lipídios fluorescentes ou tubulina utilizados. Por exemplo, ao usar lipídios com etiquetas vermelhas do Texas, use um filtro de excitação de 560 nm/25 nm e filtro de emissão de 607 nm/36 nm.
- Use um objetivo de óleo de 100x para focar na superfície da mancha de cobertura.
- Imagem das redes LNT usando microscopia de fluorescência.
NOTA: LNTs são estruturas lineares extrudando das vesículas maiores. LNTs muito menores que guvs e têm sinais de fluorescência mais fracos. Assim, a exposição e o contraste devem ser ajustados de acordo com os LNTs de imagem. Esses ajustes também levam à superexposição dos GUVs e, portanto, recomenda-se que a lamelaridade e a separação de fases nos GUVs sejam caracterizadas independentemente. - Concentre o microscópio em uma rede de interesse e tire uma imagem padrão ou de ladrilhos.
- Ajuste o tempo de exposição e os filtros de densidade neutra para a imagem dos LNTs e minimize a exposição saturada dos GUVs. Adquira imagens em canais vermelhos e verdes.
NOTA: Aqui, o canal vermelho permite a visualização dos lipídios Texas-Red, enquanto o canal verde permite a visualização dos microtúbulos (por exemplo, lipídios do Oregon Green e corantes HiLyte488). - Crie uma imagem composta sobrepondo os canais vermelho e verde (Figura 1).
- Caracterização das redes LNT medindo o comprimento LNT
- Abra as imagens adquiridas usando um software de análise de imagens como o ImageJ.
- Calibrar a escala para o microscópio usando o recurso de escala de conjunto, preencher os pixels para o fator de conversão mm e clicar em OK.
NOTA: O fator de conversão depende do microscópio, lente objetiva e câmera, e pode ser obtido usando um slide de calibração do microscópio. É geralmente expresso em pixels/mm. - Use a ferramenta de linha Multipoint para medir o comprimento dos nanotubos a partir do GUV pai. Segure Ctrl +M para medir o comprimento.
- Continue medindo os comprimentos dos tubos individuais seguindo os passos acima. A ferramenta de processamento de imagens salvará cada nova medição na janela de resultados.
- Segure Ctrl + D após desenhar cada linha para acompanhar quais tubos foram medidos.
- Medindo a espessura LNT (Figura 2)
- Abra a imagem no ImageJ e selecione o recurso Limiar na guia Imagem .
- Clique em Aplicar para aplicar o limiar.
- Desenhe um retângulo de comprimento conhecido sobre o tubo desejado (pixels pretos têm um valor de 0, e pixels vermelhos têm um valor de 255).
- Meça a densidade integrada da área.
- Divida a densidade pelo comprimento (em pixels) do LNT para obter a espessura (em pixels).
NOTA: As medidas de espessura só podem ser comparadas entre as imagens quando as configurações de imagem e limiar são definidas de forma idêntica.
- Determinando o particionamento lipídeca em nódulos de LNTs (Figura 3)
- Abra a imagem no ImageJ.
- Use a ferramenta Linha para desenhar uma linha sobre o nó desejado.
- Meça a intensidade do nó nos canais Oregon Green e Texas Red.
- Mova a linha para o LNT e meça a intensidade LNT nos canais Oregon Green e Texas Red.
Access restricted. Please log in or start a trial to view this content.
Resultados
As redes LNT (Figura 4) foram fabricadas utilizando o protocolo descrito, que utiliza o trabalho realizado pelo transporte de cinesina de microtúbulos para extrusão de LNTs de GUVs. Resumidamente, os GUVs foram preparados utilizando a reidratação do gel de agarose utilizando solução de sacarose, e os microtúbulos foram polimerizados em solução GPEM e estabilizados em BRB80T. Em seguida, motores de cinesina foram introduzidos em uma célula de fluxo formando uma camada ativa de motor...
Access restricted. Please log in or start a trial to view this content.
Discussão
As redes LNT são uma ferramenta útil para estudos in vitro para propriedades de membrana e o transporte de biomoléculas, como proteínas transmembranas. Além disso, o uso de formulações lipídicas complexas para fabricar redes LNT permite estudos mais biologicamente relevantes. Outros estudos de fabricação têm usado ou 1) formulações lipídicas simples e vesículos multilamellar ou 2) técnicas de motilidade mais complicadas para fabricar redes de GUVs compostas por formulações lipídicas complexas. O métod...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Sandia National Laboratories é um laboratório multi-missão gerenciado e operado pela National Technology & Engineering Solutions of Sandia, LLC., uma subsidiária integral da Honeywell International, Inc., para a Administração Nacional de Segurança Nuclear do DOE dos EUA sob contrato DE-NA-0003525. Este artigo descreve resultados técnicos e análises objetivas. Quaisquer opiniões ou opiniões subjetivas que possam ser expressas no artigo não representam necessariamente as opiniões do Departamento de Energia dos EUA ou do Governo dos Estados Unidos.
Agradecimentos
Este trabalho foi apoiado pelo Departamento de Energia dos EUA, Escritório de Ciências Básicas de Energia, Divisão de Ciências e Engenharia de Materiais (BES-MSE). A síntese de cinesina e a microscopia de fluorescência foram realizadas por meio de um projeto de usuário (ZIM) no Center for Integrated Nanotechnologies, um Office of Science User Facility operado para o Escritório de Ciência do Departamento de Energia dos EUA (DOE).
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| 100x/1.4 Numerical Aperture Oil Immersion Objective | Olympus | 1-U2B836 | Olympus UPlanSApo 100x/1.40 Oil Objective Infinity Corrected, RMS Thread Working Distance 0.12mm |
| 3.0 ND Filter | Olympus | Neutral Density Filter | |
| AMP-PNP | Sigma-Aldrich | A2647 | (β,γ-imidoadenosine 5′-triphosphate) |
| ATP | Sigma-Aldrich | A7699 | Adenosine 5'-triphosphate disodium salt hydrate BioXtra |
| Brightline Pinkel DA/FI/TR/Cy5/Cy7-5X-A000 filter set | Semrock | LED-DA/FI/TR/Cy5/Cy7-5X-A-000 | BrightLine Pinkel filter set, optimized for DAPI, FITC, TRITC, Cy5 & Cy7 and other like fluorophores, illuminated with LED-based light sources |
| Casein | Sigma-Aldrich | 22090 | Casein hydrolysate for microbiology |
| Catalase | Sigma-Aldrich | C9322 | Catalase from Bovine Liver |
| Chloroform | Sigma-Aldrich | 288306 | Chloroform anhydrous contains 0.5-1.0% ethanol as stabilizer |
| Cholesterol | Avanti | 700000P | cholesterol (ovine wool, >98%) (powder) |
| D-Glucose | Sigma-Aldrich | G7021 | D-(+)-Glucose powder, BioReagent, suitable for cell culture, suitable for insect cell culture, suitable for plant cell culture, ≥99.5% |
| DOPC | Avanti | 850375C | 1,2-Dioleoyl-sn-glycero-3-phosphocholine (in chloroform) |
| DOPE-Biotin | Avanti | 870282C | 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine-N-(biotinyl) (sodium salt) |
| DPPC | Avanti | 850355P | 1,2-dipalmitoyl-sn-glycero-3-phosphocholine (powder) |
| DPPE-Biotin | Avanti | 870285P | 1,2-dipalmitoyl-sn-glycero-3-phosphoethanolamine-N-(biotinyl) (sodium salt) |
| DTT | Sigma-Aldrich | 43816 | DL-Dithiothreitol solution 1 M |
| EGTA | Sigma-Aldrich | E4378 | EGTA, Egtazic acid, Ethylene-bis(oxyethylenenitrilo)tetraacetic acid, Glycol ether diamine tetraacetic acid |
| Glucose Oxidase | Sigma-Aldrich | G6125 | Glucose Oxidase from Aspergillus niger Type II, ≥10,000 units/g solid (without added oxygen) |
| Glycerol | Fisher | G33 | Glycerol (Certified ACS), Fisher Chemical |
| GTP | Sigma-Aldrich | G8877 | Guanosine 5′-triphosphate sodium salt hydrate |
| IX-81 Olympus Microscope | Olympus | N/A | IX81 Inverted Microscope from Olympus |
| KOH | Sigma-Aldrich | 1050121000 | Potassium Hydroxide |
| Magnesium Chloride | Sigma-Aldrich | M1028 | 1.00 M magnesium chloride solution |
| Orca Flash 4.0 Digital Camera | Hamamatsu | C13440-20CU | ORCA-Flash 4.0 V3 Digital CMOS camera |
| Oregon Green-DHPE | Invitrogen | O12650 | Oregon Green 488 1,2-Dihexadecanoyl-sn-Glycero-3-Phosphoethanolamine |
| Paclitaxel | ThermoFisher | P3456 | Paclitaxel (Taxol Equivalent) - for use in research only |
| PIPES | Sigma-Aldrich | P6757 | 1,4-Piperazinediethanesulfonic acid, Piperazine-1,4-bis(2-ethanesulfonic acid), Piperazine-N,N′-bis(2-ethanesulfonic acid) |
| Texas Red-DHPE | Invitrogen | T1395MP | Texas Red 1,2-Dihexadecanoyl-sn-Glycero-3-Phosphoethanolamine, Triethylammonium Salt |
| Trolox | Sigma-Aldrich | 238813 | (±)-6-Hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid |
| Tubulin, Biotin | Cytoskeleton | T333P | Tubulin protein (biotin) porcine brain |
| Tubulin, Hy-Lite 488 | Cytoskeleton | TL488M | Tubulin protein (fluorescent HiLyte 488) porcine brain |
| Tubulin, Unlabeled | Cytoskeleton | T240 | Tubulin protein porcine brain |
Referências
- Bouxsein, N. F., Carroll-Portillo, A., Bachand, M., Sasaki, D. Y., Bachand, G. D. A continuous network of lipid nanotubes fabricated from the gliding motility of kinesin powered microtubule filaments. Langmuir. 29 (9), 2992-2999 (2013).
- Paxton, W. F., Bouxsein, N. F., Henderson, I. M., Gomez, A., Bachand, G. D. Dynamic assembly of polymer nanotube networks via kinesin powered microtubule filaments. Nanoscale. 7 (25), 10998-11004 (2015).
- Leduc, C., et al. Cooperative extraction of membrane nanotubes by molecular motors. Proceedings of the National Academy of Sciences of the United States of America. 101 (49), 17096-17101 (2004).
- Lippincott-Schwartz, J., Roberts, T. H., Hirschberg, K. Secretory protein trafficking and organelle dynamics in living cells. Annual Review of Cell and Developmental Biology. 16, 557-589 (2000).
- Belting, M., Wittrup, A. Nanotubes, exosomes, and nucleic acid-binding peptides provide novel mechanisms of intercellular communication in eukaryotic cells: implications in health and disease. Journal of Cell Biology. 183 (7), 1187-1191 (2008).
- Rustom, A., Saffrich, R., Markovic, I., Walther, P., Gerdes, H. H. Nanotubular highways for intercellular organelle transport. Science. 303 (5660), 1007-1010 (2004).
- Onfelt, B., Nedvetzki, S., Yanagi, K., Davis, D. M. Cutting edge: Membrane nanotubes connect immune cells. Journal of Immunology. 173 (3), 1511-1513 (2004).
- Sciaky, N., et al. Golgi tubule traffic and the effects of brefeldin A visualized in living cells. J Cell Biol. 139 (5), 1137-1155 (1997).
- Sprong, H., van der Sluijs, P., van Meer, G. How proteins move lipids and lipids move proteins. Nature Reviews Molecular Cell Biology. 2 (7), 504-513 (2001).
- Keenan, T. W., Morre, D. J. Phospholipid class and fatty acid composition of golgi apparatus isolated from rat liver and comparison with other cell fractions. Biochemistry. 9 (1), 19-25 (1970).
- Imam, Z. I., Bachand, G. D. Multicomponent and Multiphase Lipid Nanotubes Formed by Gliding Microtubule-Kinesin Motility and Phase-Separated Giant Unilamellar Vesicles. Langmuir. 35 (49), 16281-16289 (2019).
- Wesolowska, O., Michalak, K., Maniewska, J., Hendrich, A. B. Giant unilamellar vesicles - a perfect tool to visualize phase separation and lipid rafts in model systems. Acta Biochimica Polonica. 56 (1), 33-39 (2009).
- Momin, N., et al. Designing lipids for selective partitioning into liquid ordered membrane domains. Soft Matter. 11 (16), 3241-3250 (2015).
- Fygenson, D. K., Braun, E., Libchaber, A. Phase diagram of microtubules. Physical Review E. 50, 1579(1994).
- Greene, A. C., Sasaki, D. Y., Bachand, G. D. Forming Giant-sized Polymersomes Using Gel-assisted Rehydration. Journal of Visualized Experiments. (111), (2016).
- Bachand, M., et al. Directed self-assembly of 1D microtubule nano-arrays. Royal Society of Chemistry Advances. 4 (97), 54641-54649 (2014).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados