Method Article
Entrega del complejo de ribonucleoproteína Cas9/sgRNA en células inmortalizadas y primarias a través de partículas similares a virus ("nanocuchillas")
En este artículo
Resumen
Hemos desarrollado un protocolo simple y económico para cargar complejos de ribonucleoproteína Cas9 / ARN de guía única (sgRNA) dentro de partículas similares a virus. Estas partículas, llamadas "Nanoblades", permiten la entrega eficiente del complejo Cas9/sgRNA en células inmortalizadas y primarias, así como in vivo.
Resumen
El sistema agrupado de repeticiones palindrómicas cortas (CRISPR)-Cas agrupadas e interespaciadas ha democratizado la edición del genoma en células eucariotas y ha llevado al desarrollo de numerosas aplicaciones innovadoras. Sin embargo, la entrega de la proteína Cas9 y el ARN de guía única (sgRNA) en las células diana puede ser técnicamente un desafío. Los vectores virales clásicos, como los derivados de lentivirus (LV) o virus adenoasociados (AAV), permiten la entrega eficiente de transgenes que codifican para la proteína Cas9 y su sgRNA asociado en muchas células primarias e in vivo. Sin embargo, estos vectores pueden sufrir inconvenientes como la integración del transgén en el genoma de la célula objetivo, una capacidad de carga limitada y la expresión a largo plazo de la proteína Cas9 y el ARN guía en las células objetivo.
Para superar algunos de estos problemas, se desarrolló un vector de entrega basado en el virus de la leucemia murina (MLV) para empaquetar la proteína Cas9 y su ARN guía asociado en ausencia de cualquier transgén codificante. Al fusionar la proteína Cas9 con el extremo C de la proteína estructural Gag de MLV, se formaron partículas similares a virus (VLP) cargadas con la proteína Cas9 y sgRNA (llamadas "Nanoblades"). Las nanocuchillas se pueden recolectar del medio de cultivo de las células productoras, purificarse, cuantificarse y usarse para transducir células diana y entregar el complejo activo Cas9 / sgRNA. Las nanocuchillas entregan su carga de ribonucleoproteína (RNP) de forma transitoria y rápida en una amplia gama de células primarias e inmortalizadas y pueden programarse para otras aplicaciones, como la activación transcripcional transitoria de genes dirigidos, utilizando proteínas Cas9 modificadas. Las nanocuchillas son capaces de editar el genoma in vivo en el hígado de ratones adultos inyectados y en ovocitos para generar animales transgénicos. Finalmente, se pueden complejar con ADN del donante para una reparación dirigida por homología "libre de transfección". La preparación de Nanoblade es simple, relativamente de bajo costo y se puede llevar a cabo fácilmente en cualquier laboratorio de biología celular.
Introducción
En comparación con otras nucleasas programables, el sistema CRISPR-Cas simplificó y democratizó drásticamente el procedimiento de orientación y escisión del genoma específico de la secuencia en las células eucariotas. A través de la simple expresión de un sgRNA, los usuarios pueden programar la proteína Cas9 (o variantes optimizadas) para casi cualquier locus celular1. En este escenario, la entrega de la proteína Cas9 y sgRNA se convierte en la principal limitación al realizar la mutagénesis dirigida al sitio. En las células inmortalizadas, el sgRNA y la proteína Cas se pueden expresar fácilmente a partir de plásmidos transfectados para lograr una orientación genómica eficiente en la mayoría de las células. Sin embargo, la expresión constitutiva del complejo Cas9/sgRNA puede aumentar la actividad fuera del objetivo de la proteína Cas9 e introducir cambios no deseados en loci2 no específicos. En las células primarias, la transfección del ADN puede ser técnicamente difícil de lograr y conducir a una mala expresión o a un pequeño porcentaje de células transfectadas. Las alternativas a la transfección clásica de ADN comprenden el uso de vectores virales que entregan un transgén que codifica para cas9 y sgRNA o la electroporación de la proteína Cas9 recombinante acoplada a un sgRNA sintético. Sin embargo, estos enfoques pueden conducir a la integración transgénica dentro del genoma del huésped celular (como es el caso de los vectores clásicos de expresión retroviral y lentiviral), la restricción por factores celulares y conducir a la expresión constitutiva de la proteína Cas9 y sgRNA.
La electroporación del complejo Cas9/sgRNA RNP puede superar la mayoría de estos problemas y conducir a una administración eficiente y transitoria en células primarias e in vivo, así como permitir una respuesta dependiente de la dosis. Sin embargo, generalmente se basa en equipos y reactivos costosos y también es difícil de mejorar si se tiene que tratar una gran cantidad de células. Como alternativa a las técnicas mencionadas anteriormente, estos autores han desarrollado "Nanoblades", un vector de administración retroviral para la proteína Cas9 y sgRNA3 que es conceptualmente similar a otros sistemas de entrega de proteínas de cápside derivadas de virus4,5,6,7,8. Las nanocuchillas explotan la capacidad natural de la poliproteína Gag de los retrovirus para producir, cuando se expresan solas en células cultivadas, VLP que se liberan en el medio extracelular9. Al fusionar la proteína Cas9 con el extremo C-terminal de la poliproteína Gag del virus de la leucemia murina (MLV) y coexpresar el sgRNA y las glicoproteínas de la envoltura viral, la proteína Cas9 puede encapsidarse dentro de las VLP o Nanoblades liberadas. Tras la purificación, las Nanoblades pueden incubarse con células diana o inyectarse in vivo para mediar en la administración rápida, transitoria y dependiente de la dosis del complejo Cas9/sgRNA RNP3.
Las nanoblades se pueden programar con múltiples sgRNAs para la edición simultánea en diferentes loci o con variantes Cas9 para realizar otras aplicaciones como la activación transcripcional específica del objetivo o la represión3. A diferencia de la electroporación de proteínas, que se basa en la expresión recombinante, las variantes cas recientemente descritas de la literatura se pueden clonar fácilmente en el vector de expresión de fusión Gag, lo que lo convierte en una plataforma versátil. Las nanocuchillas se pueden complejar o cargar con oligodesoxinucleótidos simples y de doble cadena (ssODN) para realizar la reparación dirigida a la homología3. La producción de nanoblade es relativamente simple y barata. Además, las Nanoblades se pueden almacenar a 4 °C durante muchos días o a -80 °C para su almacenamiento a largo plazo. Por lo general, las nanocuchillas median la edición eficiente del genoma libre de transgenes en la mayoría de las células inmortalizadas y cultivadas primariamente. Sin embargo, algunas células primarias pueden ser sensibles a la presencia de partículas virales, lo que resulta en un aumento de la mortalidad. Las células del sistema inmune innato también pueden reaccionar a la presencia de Nanoblades (debido a su origen viral) y activarse. En estos casos, el protocolo de transducción debe optimizarse para limitar el tiempo de exposición a Nanoblades y minimizar los efectos inespecíficos. Las nanoblades representan una alternativa viable y fácil de implementar a otros métodos de entrega CRISPR disponibles.
Protocolo
1. Diseño y clonación de sgRNA
NOTA: Las directrices para el diseño de sgRNAs se pueden obtener de múltiples fuentes como https://blog.addgene.org/how-to-design-your-grna-for-crispr-genome-editing o de Hanna y Doench10.
- Una vez que se hayan diseñado las secuencias de sgRNA de 20 nucleótidos, ordene los siguientes oligonucleótidos de ADN monocatenario:
- Adelante: 5' caccgNNNNNNNNNNNNNNNNNNNNNNNNN 3' (N corresponde al locus objetivo sin la secuencia del motivo protoespaciador-adyacente (PAM))
- Inversa: 5' aaacNNNNNNNNNNNNNNNNNNNNNNNNN 3' (N corresponde al complemento inverso del locus objetivo sin la secuencia PAM)
NOTA: No se requieren modificaciones especiales al ordenar los oligonucleótidos (no se requiere fosfato de 5').
- Hibridar los dos oligonucleótidos de ADN en un tubo de reacción en cadena de la polimerasa (PCR) de 0,2 ml mezclando 5 μL de tampón de recocido (500 mM NaCl; 100 mM Tris-HCl; 100 mM MgCl2; 10 mM TDT; pH 7,9 a 25 °C), 1 μL de cada oligonucleótido de ADN (solución madre de 100 μM en agua) y 42 μL de agua.
- En un bloque de PCR, incubar las muestras a 95 °C durante 15 s y luego disminuir la temperatura a 20 °C con una rampa de 0,5 °C/s. Conservar a temperatura ambiente o conservar a -20 °C.
NOTA: El protocolo se puede pausar aquí. - Digerir 10 μg de los plásmidos de expresión de sgRNA BLADE o SUPERBLADE con 10 unidades de enzima de restricción BsmBI-v2 durante 3 h a 55 °C en un volumen total de reacción de 50 μL.
NOTA: El vector digerido debe liberar un inserto de ADN de ~1.9 kb y un segundo fragmento de ADN de ~3.3 kb. - Cargue la reacción de restricción en un gel de agarosa al 1% teñido con 5 μg/ml de bromuro de etidio (o una tinción de gel de ADN alternativa más segura).
NOTA: Use el equipo de protección adecuado cuando manipule bromuro de etidio, que se sospecha que causa defectos genéticos.- En una mesa ultravioleta (UV) establecida en una longitud de onda de 312 nm (para evitar dañar el ADN), corte el fragmento de ADN de 3,3 kb del gel y colóquelo en un tubo de microcentrífuga de 1,5 ml.
NOTA: Use el equipo de protección adecuado (guantes y gafas de protección UV) cuando manipule bromuro de etidio y trabaje en la mesa UV. - Extraiga adn del gel en rodajas que contiene el fragmento de ADN de 3,3 kb utilizando un kit de extracción de gel de ADN dedicado (consulte la Tabla de materiales). Cuantificar la cantidad de ADN purificado usando un espectrofotómetro.
NOTA: El protocolo se puede pausar aquí.
- En una mesa ultravioleta (UV) establecida en una longitud de onda de 312 nm (para evitar dañar el ADN), corte el fragmento de ADN de 3,3 kb del gel y colóquelo en un tubo de microcentrífuga de 1,5 ml.
- Ligar los oligonucleótidos de ADN hibridados hacia adelante y hacia atrás desde el paso 1.2 hasta el vector BLADES o SUPERBLADE digerido por BsmB1 y purificado en gel desde el paso 1.5.2. Para esto, agregue 2 μL de tampón de ADN ligasa T4, 50 ng del vector purificado en gel (del paso 1.5.2), 1 μL de los oligonucleótidos de ADN hibridados (del paso 1.2), agua para formar el volumen a 19 μL y 1 μL de ADN ligasa T4. Incubar la reacción a 25 °C durante 10 min.
- Transformar el producto de ligadura en bacterias competentes (ver la Tabla de Materiales) como se describe en11. Coloque las bacterias transformadas en una placa de agar de ampicilina Luria Bertani e incube durante la noche a 37 ° C.
- Seleccione varias colonias aisladas en la placa de agar para realizar la minipreparación de ADN11 (consulte la Tabla de Materiales), y realice la secuenciación de Sanger utilizando un cebador delantero U6 (5' GACTATCATATGCTTACCGT 3') para verificar la correcta ligadura de la secuencia variable sgRNA.
NOTA: Se pueden usar otros plásmidos de expresión de sgRNA si no codifican para la proteína Cas9, lo que podría interferir con la producción de Nanoblade.
2. Preparación de plásmidos
- Realizar la maxipreparación (ver la Tabla de Materiales) de todos los plásmidos requeridos, y preparar 10 μg alícuotas a 1 μg/mL para almacenar a -20 °C. Evite los ciclos repetidos de congelación/descongelación de los plásmidos; use alícuotas dos veces antes de desecharlas.
3. Preparación de Nanoblade
- En el día 1, sembró entre 3,5 y 4 × 106 células HEK293T (ver la Tabla de Materiales) en 10 ml del medio Eagle modificado (DMEM) de Dulbecco que contiene glucosa alta, piruvato de sodio, L-glutamina, suero bovino fetal al 10% (FBS) y penicilina/estreptomicina en un plato de cultivo celular de 10 cm. Mueva la placa de 10 cm suavemente hacia atrás y hacia adelante, luego de derecha a izquierda (repita esta secuencia 5x) para distribuir las células de manera homogénea sobre la placa de cultivo. Incubar células a 37 °C en un incubador celular con 5% de CO2.
NOTA: Todos los procedimientos relacionados con el manejo de células cultivadas y Nanoblades deben realizarse bajo una campana de flujo laminar de cultivo celular para evitar su contaminación. - Día 2: Transfección por plásmidos
- Las células deben ser 70-80% confluentes 24 h después del recubrimiento (Figura 1A). Reemplace el medio con 10 ml de DMEM fresco que contenga glucosa alta, piruvato de sodio, L-glutamina, 10% fbS (se puede omitir la penicilina y la estreptomicina aunque no es obligatorio) antes de la transfección.
NOTA: En este paso, es importante que las células no sean confluentes. De lo contrario, la eficiencia de la transfección, así como la producción de partículas, podrían reducirse. - Para cada placa de 10 cm, prepare las siguientes cantidades de plásmidos en un tubo de 1,5 ml: 0,3 μg pCMV-VSV-G, 0,7 μg pBaEVRless, 2,7 μg MLV Gag/Pol, 1,7 μg BIC-Gag-Cas9, 4,4 μg de BLADES o PLÁsmido SUPERBLADES que codifique el sgRNA clonado (o 2,2 μg cada uno si se utilizan dos sgRNA).
- Agregue 500 μL de tampón de transfección (consulte la Tabla de materiales), vórtice durante 10 s y luego centrífuga durante 1 s. Agregue 20 μL del reactivo de transfección (consulte la Tabla de materiales), vórtice el tubo durante 1 s y luego centrífuga durante 1 s.
- Incubar durante 10 min a temperatura ambiente y añadir toda la solución gota a gota a las células en medio DMEM utilizando un pipettor P1000. Mueva la placa de 10 cm suavemente hacia atrás y hacia adelante, luego de derecha a izquierda (repita esta secuencia 5x) para distribuir uniformemente el reactivo de transfección sobre las células. Incubar células a 37 °C durante al menos 40 h en un incubador de células con un 5% de CO2.
NOTA: Si se desea, el medio se puede cambiar 4 h después de la transfección.
- Las células deben ser 70-80% confluentes 24 h después del recubrimiento (Figura 1A). Reemplace el medio con 10 ml de DMEM fresco que contenga glucosa alta, piruvato de sodio, L-glutamina, 10% fbS (se puede omitir la penicilina y la estreptomicina aunque no es obligatorio) antes de la transfección.
- El día 3, verifique la morfología de las células transfectadas bajo el microscopio.
NOTA: Las células productoras comenzarán a fusionarse. Esta es una ocurrencia normal debido a la expresión de envolturas virales fusogénicas (Figura 1B, C). - Día 4: Cosecha de Nanoblades
NOTA: Al menos 40 h después de la transfección, las células se habrían fusionado debido a la expresión de las envolturas virales fusogénicas y, a veces, las células están completamente separadas del soporte de la placa (Figura 1D).- Recolectar 9 ml del sobrenadante del medio de cultivo utilizando una pipeta de 10 ml.
NOTA: Las nanocuchillas son VLP capaces de entregar la proteína Cas9 y su sgRNA asociado en las células primarias e in vivo. Aunque no se consideran organismos modificados genéticamente ya que carecen de material genético, pueden inducir cambios genéticos. Por lo tanto, deben manipularse con precaución para evitar cualquier contacto con los usuarios (especialmente si están programados para dirigirse a los genes supresores de tumores). Se aconseja a los usuarios que sigan sus pautas de seguridad locales para la manipulación de vectores retrovirales y trabajen en un laboratorio de nivel BSL-2 al preparar VLP y realizar experimentos de transducción. Las nanocuchillas se pueden inactivar con 70% de etanol o 0,5% de hipoclorito de sodio. También es recomendable tratar todos los residuos plásticos (puntas de pipeta, placas de cultivo de tejidos, tubos de centrifugación) con hipoclorito de sodio al 0,5% durante al menos 10 min para inactivar las Nanoblades. - Centrifugar el sobrenadante recolectado a 500 × g durante 5 min para eliminar los desechos celulares y recuperar el sobrenadante sin perturbar el pellet celular.
NOTA: Si las Nanoblades están destinadas a ser utilizadas en células primarias, filtre el sobrenadante usando un filtro de 0.45 μm o 0.8 μm. Tenga en cuenta que este paso reduce drásticamente el título de Nanoblade, ya que una fracción significativa se bloqueará en la membrana del filtro. - Peletizar las Nanoblades durante la noche (12-16 h) en un rotor de cucharón oscilante a 4.300 × g o a 209.490 × g en una ultracentrífuga durante 75 min a 4 °C (ver la Tabla de Materiales).
NOTA: Si las células diana pueden crecer en DMEM, es posible incubarlas directamente con el sobrenadante obtenido después del paso 3.4.2 sin concentrar las Nanoblades.
- Recolectar 9 ml del sobrenadante del medio de cultivo utilizando una pipeta de 10 ml.
- Día 5: Resuspensión y almacenamiento de Nanoblades
- Después de la centrifugación, aspire lentamente el medio y vuelva a suspender el pellet blanco con 100 μL de solución salina tamponada con fosfato (PBS) en frío. Cubra el tubo con parafilm e incube durante 1 h a 4 °C con una agitación suave antes de volver a depositar el pellet mediante pipeteo hacia arriba y hacia abajo.
NOTA: Un material viscoso blanco puede aparecer en la resuspensión; esto es normal y no afecta significativamente la eficiencia de la transducción. - Guarde las Nanoblades a 4 °C si planea usarlas dentro de las cuatro semanas. De lo contrario, congele las Nanoblades en nitrógeno líquido y guárdelas a -80 °C.
NOTA: Use gafas de protección y guantes criogénicos cuando manipule nitrógeno líquido. La congelación a presión y el almacenamiento a -80 °C conducen a una disminución significativa en la eficiencia de Nanoblade. Además, las Nanoblades descongeladas no deben congelarse nuevamente. El protocolo se puede pausar aquí.
- Después de la centrifugación, aspire lentamente el medio y vuelva a suspender el pellet blanco con 100 μL de solución salina tamponada con fosfato (PBS) en frío. Cubra el tubo con parafilm e incube durante 1 h a 4 °C con una agitación suave antes de volver a depositar el pellet mediante pipeteo hacia arriba y hacia abajo.
4. Concentración de nanocuchillas en un cojín de sacarosa
NOTA: Como alternativa a la centrifugación nocturna o ultracentrifugación (paso 3.4.3), las Nanoblades se pueden concentrar en un cojín de sacarosa. Esto produce una fracción más pura de Nanoblades, aunque la cantidad total recuperada será menor.
- Prepare una solución de sacarosa al 10% (peso a volumen) en 1x PBS y filtre a través de un filtro de jeringa de 0,2 μm (consulte la Tabla de materiales).
- Comience el proceso de concentración de las Nanoblades en el cojín de sacarosa.
- Coloque 9 ml de muestra que contenga VLP (a partir del paso 3.4.3) en un tubo de ultracentrífuga (consulte la Tabla de materiales). Usando una jeringa y una cánula de 3 ml, coloque lentamente 2,5 ml de sacarosa al 10% debajo de la muestra, tratando de no mezclar la muestra que contiene VLP y la solución de sacarosa.
- Alternativamente, coloque 2.5 ml de sacarosa al 10% en un tubo de ultracentrífuga (consulte la Tabla de materiales). Incline el tubo y agregue lentamente los 9 ml de muestra que contiene VLP (del paso 3.4.3) con un pipeteador de baja velocidad. Durante esta operación, eleve progresivamente el tubo a una posición vertical.
- Centrifugar las muestras a 209.490 × g en una ultracentrífuga durante 90 min a 4 °C.
NOTA: Esta técnica se puede adaptar para centrifugación a baja velocidad (4.300 × g) durante la noche como se describe en 12. - Después de la centrifugación, retire el sobrenadante con cuidado y coloque el tubo boca abajo sobre papel de seda para eliminar cualquier líquido restante. Después de 1 min, agregue 100 μL de 1x PBS y coloque el tubo a 4 °C con una cubierta de parafilm en un soporte de tubo en una mesa de agitación durante 1 h (consulte la Tabla de Materiales) antes de volver a suspender el pellet mediante pipeteo hacia arriba y hacia abajo.
NOTA: El protocolo se puede pausar aquí.
5. Monitoreo de la carga de Cas9 dentro de Nanoblades por punto-blot
- Prepare el tampón de dilución agregando 1 volumen de tampón de lisis que contenga un surfactante no iónico (consulte la Tabla de materiales) en 4 volúmenes de 1x PBS. Diluir 2 μL de Nanoblades concentradas en 50 μL de tampón de dilución, vórtice brevemente, y transferir 25 μL de esta mezcla a un nuevo tubo que contenga 25 μL de tampón de dilución. Repita esta operación para tener 4 tubos de diluciones de Nanoblade (pasos de dilución de 2 veces).
- Para los controles estándar, diluya 2 μL de nucleasa Cas9 recombinante (consulte la Tabla de materiales) en 50 μL de tampón de dilución, vórtice brevemente, y proceda a realizar ocho diluciones en serie (dilución de 2 veces para cada paso).
- Detecte cuidadosamente 2,5 μL de cada dilución de VLP y 2,5 μL de cada estándar en una membrana de nitrocelulosa con una pipeta multicanal (un volumen mayor puede dar lugar a manchas superpuestas).
NOTA: También se puede utilizar una membrana de polivinildifluoruro tratada con metanol. - Una vez que las partículas se absorben en la membrana, bloquee la membrana con 1 solución salina tamponada con Tris que contenga un surfactante no iónico (TBS-T) suplementado con leche seca sin grasa (5% p/v) durante 45 min a temperatura ambiente.
NOTA: El protocolo se puede pausar aquí, y la membrana se almacena a 4 °C en 1x TBS-T. - Deseche el 1x TBST suplementado con leche seca sin grasa e incube la membrana durante la noche a 4 °C con el anticuerpo Cas9-peroxidasa de rábano picante (dilución de 1/1000 en 1x TBST, 5% de leche). Lave la membrana 3 veces con TBS-T y visualice la señal utilizando un kit de sustrato quimioluminiscente mejorado.
- Cuantifique la intensidad de los puntos para las Nanoblades y las diluciones estándar recombinantes cas9 utilizando el software patentado proporcionado con la estación de imágenes de gel o imageJ13. Defina una curva lineal que vincule la intensidad de los puntos con la concentración de Cas9. Utilizando la función de la curva obtenida, extrapolar el contenido de Cas9 en cada preparación.
NOTA: La cantidad de control recombinante de la proteína Cas9 puede saturar la lectura para las muestras más concentradas del conjunto de dilución estándar (Figura 2). Por lo tanto, se aconseja, al definir la curva lineal, eliminar la lectura de las muestras sin diluir (y a veces la de los primeros pasos de dilución) si no están en el rango lineal con respecto a la concentración conocida de Cas9 que se detectó. Del mismo modo, al extrapolar la cantidad de Cas9 dentro de las muestras de Nanoblade, solo use las lecturas que están dentro del rango lineal de la curva estándar.
6. Transducción de células diana con Nanoblades (procedimiento para la transducción en una placa de 12 pocillos)
- En una placa de 12 pocillos, sembra 100,000-200,000 células (ya sean células adherentes primarias o inmortalizadas) por pozo en 1 ml del medio de cultivo celular apropiado. Permita que las células se adhieran a la superficie de la placa antes de la transducción.
- En un tubo de microcentrífuga de 1,5 ml, agregue 5-20 μL de nanocuchillas concentradas (desde el paso 3.5.1 o 4.4) a 500 μL de medio de cultivo celular, y mezcle mediante pipeteo hacia arriba y hacia abajo con un pipettor P1000. Retire el medio de las celdas y reemplácelo con los 500 μL de esta mezcla nanoblade.
NOTA: La transducción debe optimizarse para cada tipo de célula. Es importante utilizar el menor volumen posible de medio (evitando el secado de las células diana) para que las Nanoblades permanezcan altamente concentradas. Las células adherentes deben ser transducidas directamente mientras están unidas a la placa (no transducir en suspensión, ya que esto disminuirá significativamente la eficiencia de la transducción). Algunas células toleran la exposición prolongada a Nanoblades (24-48 h), mientras que otras son muy sensibles y pueden formar pequeños sincitios. En este caso, las Nanoblades deben incubarse con células solo durante 4-6 h antes de reemplazar el medio. La esinoculación14 también puede mejorar la transducción de células cultivadas en suspensión. Los adyuvantes como los polímeros catiónicos (ver la Tabla de Materiales) también pueden mejorar la eficiencia de transducción en algunos tipos de células. - Después de 4-6 h de incubación celular en un volumen bajo de medio que contenga Nanoblades, aumente el volumen del medio a la cantidad normal (1 ml si trabaja con una placa de 12 pocillos), o reemplácelo con medio fresco si las células son sensibles a las VLP.
NOTA: El medio celular que contiene Nanoblades debe inactivarse con hipoclorito de sodio al 0,5% durante 10 minutos antes de desecharlo. Use guantes y gafas protectoras cuando manipule hipoclorito de sodio. Si las nanocuchillas inducen la muerte celular, adapte la cantidad y el tiempo total de exposición para reducir la mortalidad celular.
7. Medición de la eficiencia de CRISPR en el locus objetivo mediante el ensayo de endonucleasa T7
- Diseñe cebadores de PCR para amplificar una región de par de bases (pb) de 400-700 que abarque el sitio de escisión CRISPR.
NOTA: El sitio de escisión debe estar distante del borde del amplicón en al menos 200 pb y debe desplazarse ligeramente desde el centro del amplicón para que en la escisión de la endonucleasa T7, se liberen 2 fragmentos de diferentes tamaños. - Extraer ADN genómico de células tratadas con Nanoblades dirigidas al gen de interés y de células de control tratadas con Nanoblades programadas con un sgRNA de control (ver la Tabla de Materiales).
NOTA: El protocolo se puede pausar aquí. - Utilizando 150 ng de ADN genómico como plantilla, programe una reacción de PCR de 30 μL de volumen (volumen final) siguiendo el protocolo del fabricante. Compruebe que la amplificación por PCR produce un solo amplicó del tamaño esperado ejecutando un gel de agarosa al 2% teñido con 5 μg / ml de bromuro de etidio (o una tinción de gel de ADN alternativa más segura).
NOTA: Use el equipo de protección adecuado cuando manipule bromuro de etidio, que se sospecha que causa defectos genéticos. El protocolo se puede pausar aquí. - Generación y digestión heterodúplex
- En un tubo de PCR de 0,2 ml, agregue 5 μL del tampón enzimático (provisto de la endonucleasa I T7), 20 μL de agua y 24 μL del producto de PCR a partir del paso 7.3. Permitir la formación heterodúplex calentando las muestras a 94 °C durante 3 min y luego disminuyendo la temperatura (2 °C por min) para alcanzar los 40 °C.
- Añadir 0,5 μL de T7-endonucleasa I a temperatura ambiente a cada tubo heterodúplex, incluido el control. Incubar a 37 °C durante 15 min. Cargue la reacción resultante en un gel de agarosa al 2,5% (peso/volumen) teñido por bromuro de etidio. Después de la migración, tome una imagen del gel en un transiluminador UV.
NOTA: Use el equipo de protección adecuado cuando manipule bromuro de etidio, que se sospecha que causa defectos genéticos. Use gafas de protección UV cuando use el transiluminador UV. - Mida las eficiencias de escisión analizando la imagen resultante de la reacción de digestión para cuantificar la intensidad de cada banda con el software adecuado (consulte la Tabla de materiales).
8. Medición de la eficiencia de CRISPR en el locus objetivo mediante secuenciación de Sanger y análisis TIDE
NOTA: Como alternativa al ensayo de endonucleasa T7, la eficiencia de CRISPR puede ser monitoreada mediante el análisis y la deconvolución de trazas de secuenciación de Sanger basadas en el protocolo TIDE15.
- Realice la secuenciación sanger de los amplicones de PCR a partir del paso 7.3 (incluya una condición de control correspondiente a células no tratadas) utilizando el cebador de PCR hacia adelante o hacia atrás.
- Analice los rastros de secuenciación de Sanger de la condición de control (células no tratadas) y las muestras tratadas con Nanoblade utilizando el servidor TIDE (https://tide.nki.nl) y siguiendo sus pautas de análisis.
9. Formación de complejos nanoblade con donantes ssODN para reparación dirigida a homología (procedimiento para la transducción en una placa de 12 pocillos)
NOTA: Anteriormente se han descrito directrices para el diseño de ssODN para una edición mediada por reparación dirigida a homología eficiente16.
- En una placa de 12 pocillos, sembra 100,000-200,000 células por pozo en 1 ml del medio de cultivo celular apropiado. Permita que las células se adhieran a la superficie de la placa antes de la transducción.
- Preparar 100 μL de una solución del polímero catiónico (ver la Tabla de Materiales) a 8 μg/mL en 1x PBS.
- Mezclar 19 μL de la solución de polímero catiónico con 100 pmol de la plantilla ssODN. Añadir 20 μL de Nanoblades concentradas (a partir del paso 3.5.1 o 4.4) e incubar durante 15 min sobre hielo.
- Retire las Nanoblades/ssODN complejadas del hielo y agregue 500 μL de medio de cultivo celular (a 37 °C). Retire el medio de las células objetivo (a partir del paso 9.1) y añada los 500 μL de medio que contienen las Nanoblades/ssODN complejadas. Permita que las células proliferen durante 48 h antes del genotipado.
- Extraiga ADN genómico de una fracción de la población celular utilizando un kit de extracción dedicado (consulte la Tabla de materiales).
- Diseñe imprimaciones de PCR para amplificar una región de 400-700 pb que abarca el sitio de imitación.
NOTA: Los cebadores de PCR no deben superponerse con los brazos de homología del ssODN para evitar resultados falsos positivos resultantes de la amplificación por PCR de cualquier ssODN residual aún presente dentro de las células diana. - Utilizando 150 ng de ADN genómicos de células de control (no tratadas) o células tratadas con Nanoblade como plantilla, programe una reacción de PCR de 30 μL siguiendo el protocolo del fabricante.
NOTA: las trazas ssODN pueden estar presentes en el medio celular varios días después de la transducción con el complejo. Este ssODN puede servir como una plantilla parcial para los ensayos de PCR que intentan detectar la integración correcta. Por lo tanto, es aconsejable pasar las células al menos dos veces después de la transducción, para evitar eventuales ensayos de falsos positivos. - Carga 5 μL de las reacciones de control y PCR tratadas con Nanoblade en un gel de agarosa al 1% (peso/volumen) teñido por bromuro de etidio. Después de la migración, tome una imagen del gel en un transiluminador UV.
NOTA: Si la recombinación de homología es exitosa y corresponde a la inserción de más de 1 pb de material genético, debe haber una diferencia en el peso molecular de los amplicones de PCR entre el control y la muestra tratada con Nanoblade. Como la eficiencia de HDR no alcanza el 100%, dos bandas deben ser visibles en la muestra tratada con Nanoblade (una de tamaño similar al amplicón PCR de control correspondiente al alelo sin editar y otra de mayor peso molecular correspondiente al alelo knock-in, ver Panel central de la Figura 3B).
- Diseñe imprimaciones de PCR para amplificar una región de 400-700 pb que abarca el sitio de imitación.
- Realizar la secuenciación Sanger de los amplicones de control y PCR tratados con Nanoblade.
- Cuantificar la eficiencia en cadena utilizando el protocolo TIDER17.
10. Entrega de nanoblade in vivo
- Administrar hasta 25 μL de Nanoblades concentradas desde el paso 3.5.1 a través de inyección retroorbital o hasta 100 μL a través de la inyección de vena de la cola, como se describe en18, si se trabaja con ratones.
NOTA: Todos los procedimientos que involucran la experimentación con animales (incluidas las inyecciones de Nanoblade para fines de edición del genoma) requieren un protocolo aprobado por un comité de ética local. - Para la generación de ratones transgénicos, utilice un microinyector para administrar de 1 pL a 10 pL de Nanoblades concentradas desde el paso 4.4 en el espacio perivitelino de los ovocitos de ratón como se describió anteriormente18.
NOTA: Para la inyección de perivitelino, es esencial purificar y concentrar Nanoblades en un cojín de sacarosa para evitar la obstrucción del microinyector.
Resultados
El protocolo para la preparación de Nanoblade es bastante sencillo y requiere un equipo de laboratorio simple además del acceso a una campana de cultivo de tejidos, una incubadora de CO2 y una centrífuga de cuchara oscilante o una ultracentrífuga. Sin embargo, algunos pasos requieren una atención particular, como la fuente y el manejo de las células productoras, así como las condiciones de transducción. Como se muestra en la Figura 1A, es importante sembrar células para que se distribuyan homogéneamente en la placa y alcancen ~ 70-80% de confluencia el día de la transfección (evite tener grupos de células). Veinticuatro horas después de la transfección (Figura 1B, C), las células productoras formarán sincitia que dará lugar a células de mayor tamaño con múltiples núcleos. Cuarenta horas después de la transfección (Figura 1D), la mayoría de las células de la placa habrán formado sincitia y comenzarán a desprenderse de la placa.
Esto es perfectamente normal y es causado por la expresión de la glicoproteína de la envoltura, que induce la fusión entre las células vecinas. Tras la concentración por centrifugación (o incluso directamente del sobrenadante de las células productoras), la cantidad de Cas9 cargada dentro de nanoblades se puede cuantificar de manera absoluta mediante puntos secantes en una membrana de nitrocelulosa utilizando Cas9 recombinante como referencia (Figura 2). Este paso es importante para determinar la cantidad correcta de Nanoblades para usar para la transducción de células objetivo. Al realizar el ensayo de blot de puntos, es importante considerar solo las lecturas que caen dentro del rango lineal de la curva estándar. Sin embargo, independientemente de la cantidad de Cas9 presente en Nanoblades, es esencial probar la eficiencia de la edición del genoma directamente en las células diana utilizando el ensayo de endonucleasa T7 (Figura 3) o la secuenciación de Sanger.
Como se muestra en la Figura 3, la eficiencia de las Nanoblades puede diferir de un lote a otro, aunque generalmente se correlaciona con la cantidad de Cas9. En el ejemplo que se muestra en la Figura 3, el lote del carril 1 conduce a una eficiencia de edición general del 20%, mientras que el lote del carril 3 conduce a una eficiencia del 60%. En este caso, es posible aumentar el volumen de Nanoblades utilizadas a partir del lote 1 para lograr una eficiencia de edición similar a la del lote 3. La Figura 4 muestra la máxima eficiencia de edición obtenida utilizando Nanoblades en diferentes tipos de células primarias. Es importante tener en cuenta que la eficiencia puede variar dependiendo de la secuencia del sgRNA utilizado y la accesibilidad objetivo.
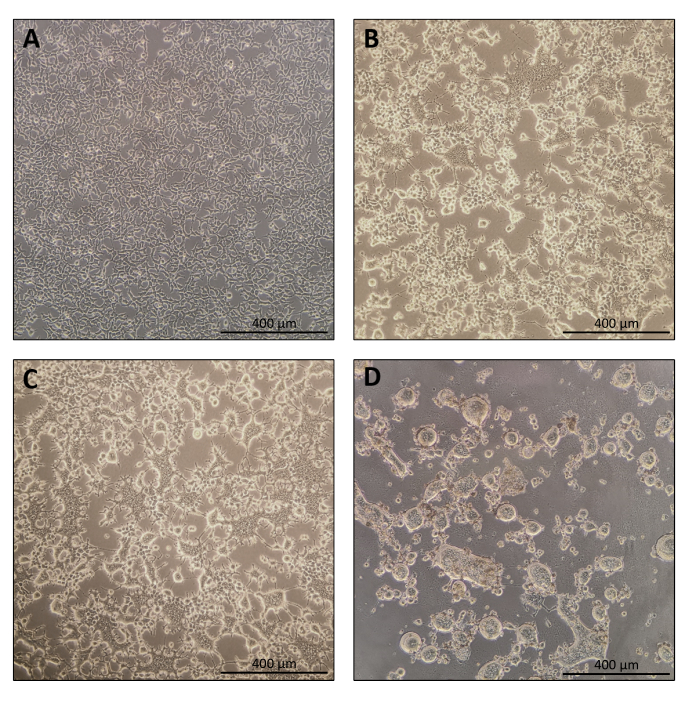
Figura 1: Morfología de las células productoras durante la producción de Nanoblade. (A) Células HEK293T al 70-80% de confluencia 24 h después del chapado. (B y C) Morfología celular HEK293T 24 h después de la transfección. (D) Morfología celular HEK293T 40 h después de la transfección. Barras de escala = 400 μm. Haga clic aquí para ver una versión más grande de esta figura.
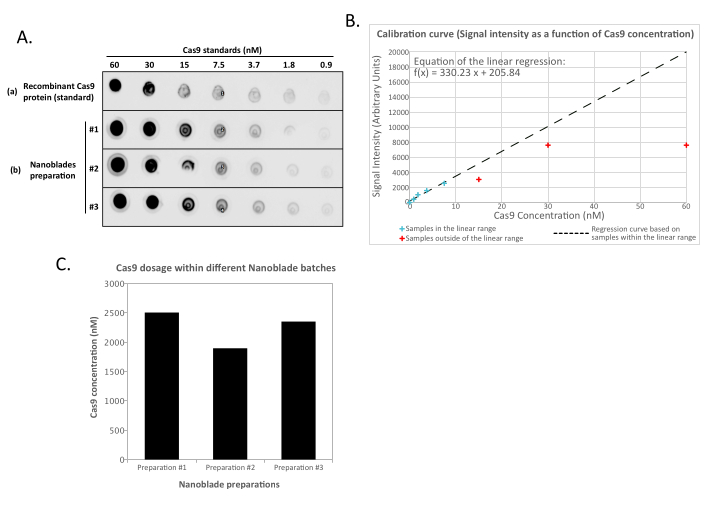
Figura 2: Cuantificación de la carga de Cas9 dentro de Nanoblades por punto-blot. (A) Cas9 recombinante o 100x concentrado (por ultracentrifugación) Las muestras de Nanoblade (#1, #2 y #3) se diluyen 2 veces secuencialmente y se manchan en una membrana de nitrocelulosa antes de incubar con anticuerpos anti-Cas9 acoplados a HRP. La señal se revela mediante una quimioluminiscencia mejorada. (B) La señal de quimioluminiscencia se adquiere y cuantifica para las diluciones recombinantes de Cas9 y la intensidad de la señal trazada contra la cantidad conocida de Cas9 detectada en la membrana de nitrocelulosa. Se calcula una curva de regresión para las diluciones que están dentro del rango lineal (ver cruces azules), excluyendo todas las concentraciones que están fuera del rango lineal (ver cruces rojas). (C) La concentración de Cas9 (nM) en cada preparación de Nanoblade se extrapoló utilizando la ecuación de la regresión lineal obtenida en (B). Para ello, es importante utilizar únicamente la señal cuantificada de las diluciones de Nanoblade que se encuentran dentro del rango lineal de la curva de regresión. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Monitoreo de la eficiencia de edición tras la transducción. (A) Ensayo de endonucleasa T7 que mide la eficiencia de escisión en células tratadas con Nanoblade. Las células transducidas con Nanoblades dirigidas al gen EMX1 fueron analizadas por ensayo de endonucleasa T7. Carril 1: Lote de preparación de Nanoblade # 1 (20% de eficiencia de escisión); Carril 2: Celdas de control; Carril 3: Lote de preparación de Nanoblade # 2 (60% de eficiencia de escisión). (B) Knock-in de la secuencia Flag-tag dentro del marco de lectura abierto DDX3 . Las nanoblades concentradas programadas con un sgRNA dirigido al locus DDX3 se produjeron a partir de diferentes clones de HEK293T (# 1, # 2) y se complejaron con dosis crecientes de una plantilla ssODN Flag-DDX3 y los complejos obtenidos utilizados para la transducción de células diana HEK293T. Tras la transducción, las células se cultivaron durante tres días antes de recolectarlas para extraer adn genómico y proteínas totales. Las proteínas Flag-DDX3 fueron inmunoprecipitadas utilizando perlas de agarosa anti-Flag seguidas de un análisis western-blot de las proteínas recuperadas utilizando un anticuerpo anti-Flag (panel superior). La inserción dirigida al sitio de la etiqueta Flag en el locus Ddx3 también se ensayó mediante PCR utilizando cebadores que flanquean el sitio de inserción (panel central) o utilizando una imprimación hacia adelante que reconoce la secuencia Flag-tag y una imprimación inversa específica para el locus Ddx3 aguas abajo del sitio de inserción Flag (panel inferior). Abreviaturas: EMX1 = Espiráculos vacíos Homeobox 1; DDX3 = HELICASA 3 de ARN de caja muerta; PCR = reacción en cadena de la polimerasa; ODN = oligodesoxinucleótido; ssODN = ODN de cadena continua; sgRNA = ARN de guía única; IP = inmunoprecipitación. Haga clic aquí para ver una versión más grande de esta figura.
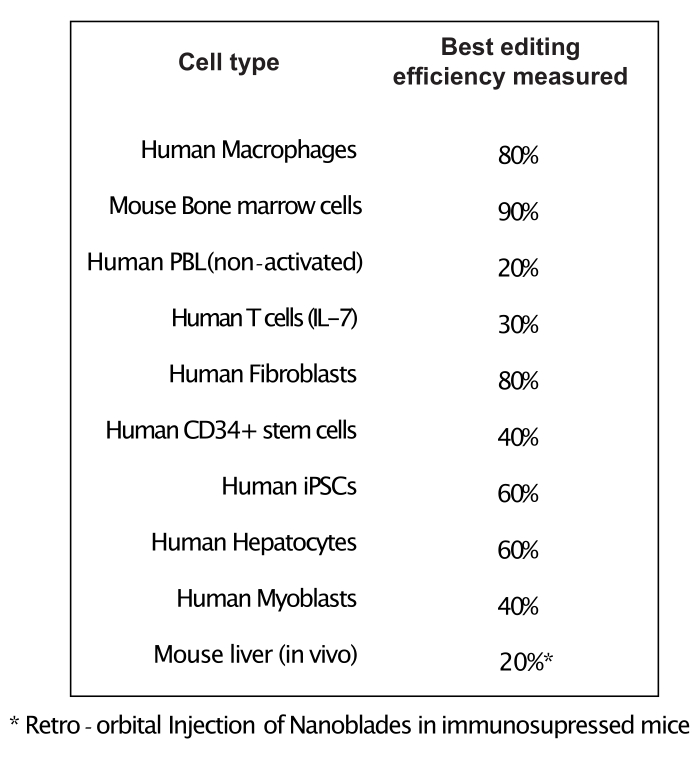
Figura 4: Eficiencia de edición lograda en diferentes tipos de células primarias utilizando Nanoblades. Abreviaturas: PBL = linfocito de sangre periférica; IL = interleucina; CD = grupo de diferenciación; iPSC = célula madre pluripotente inducida. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Las nanocuchillas permiten la administración rápida y dependiente de la dosis del complejo Cas9/sgRNA RNP en líneas celulares y células primarias. A diferencia de la transfección clásica y otros vectores de entrega viral, pero al igual que la electroporación de proteínas, las Nanoblades tienen la ventaja de la entrega transitoria del RNP Cas9 / sgRNA de una manera libre de transgenes. Las nanoblades ofrecen una plataforma altamente versátil, simple y económica para la entrega de proteínas que se puede adaptar fácil y rápidamente a la familia en constante expansión de variantes CRISPR. Las nanoblades se pueden producir en la línea celular HEK293T o sus derivados. Las líneas celulares HEK293T utilizadas aquí se han desarrollado para maximizar la producción de partículas retrovirales y lentivirales (consulte la Tabla de materiales). Sin embargo, aunque otras fuentes de células HEK293T pueden ser adecuadas, los usuarios deben probar y comparar células HEK293T de diferentes fuentes, ya que se han observado diferencias importantes en la producción de partículas dependiendo de la fuente de células HEK293T. Las células también deben ser revisadas para detectar contaminación por Mycoplasma con frecuencia y pasar cada tres días (clásicamente dilución de 1/8) para evitar la sobreconfluencia, lo que tiene un impacto negativo en la producción de partículas.
Las células no deben mantenerse durante más de 20 pasajes. Se utilizó DMEM suplementado con glucosa, penicilina/estreptomicina, glutamina y suero bovino fetal descomplementado al 10% para el cultivo celular. Como el origen del suero puede afectar la calidad de la preparación de Nanoblade, se deben probar diferentes lotes de suero antes de la producción a gran escala. Las nanoblades se pueden producir de manera eficiente en otros medios como RPMI o modificaciones sin suero del medio esencial mínimo que pueden reemplazar a DMEM el día después de la transfección. Como se indica a continuación, aunque el reemplazo medio después de la transfección con algunos reactivos de transfección de ADN es opcional, puede ser beneficioso modificar el medio en el que se liberan las VLP, especialmente para limitar los rastros séricos en la preparación de partículas. Sin embargo, el cultivo de células en un medio esencial mínimo de suero reducido el día antes de la transfección aún no se ha intentado.
Como se mencionó, las nanocuchillas se producen tras la sobreexpresión de una mezcla de plásmidos en las células productoras. La sobreexpresión parece ser necesaria para una producción óptima. De hecho, este laboratorio desarrolló una línea celular productora donde la construcción que expresa Gag-Pol se estabilizó mediante la selección de antibióticos; sin embargo, este sistema no pudo producir cantidades significativas de Nanoblades. Una observación similar se hizo cuando la construcción codificante de sgRNA se integró de manera estable en el genoma de las células productoras. Como se describe para otros sistemas de producción de partículas, una línea celular estable que exprese al menos algunas construcciones involucradas en la producción de Nanoblades puede ser útil; sin embargo, esto ciertamente requeriría el procesamiento de grandes volúmenes de sobrenadante y una técnica apropiada para purificar partículas. El protocolo anterior describe el procedimiento preferido para producir Nanoblades que explota reactivos de transfección específicos (consulte la Tabla de materiales).
Aunque los reactivos de transfección de otros fabricantes también se han probado con éxito, la gran mayoría de los resultados de este grupo con Nanoblades siguen el procedimiento descrito en este documento. La transfección de bajo costo se puede lograr utilizando reactivos de fosfato de calcio y producir una buena eficiencia de producción; sin embargo, este método requiere absolutamente el reemplazo del medio de transfección el día después de la transfección y puede dejar residuos de fosfato de calcio en la preparación de partículas sedimentadas. Consistente con la necesidad de altos niveles de expresión para los componentes de Nanoblade dentro de las células productoras es la observación de que la cantidad de sgRNAs asociados con la proteína Cas9 puede ser un factor limitante para la edición eficiente del genoma. Para mejorar la carga de sgRNA, dos enfoques técnicos han sido desarrollados recientemente por grupos independientes que utilizan vectores de entrega de proteínas similares a nanoblades. Estos se basan en el uso de la expresión citoplasmática dependiente de la polimerasa T7 de sgRNA6 o mediante la adición de una señal de encapsidación retroviral a la secuencia de sgRNA para mediar la unión a la poliproteína Gag6. Estos enfoques podrían mejorar la carga de sgRNA dentro de Nanoblades, aunque aún no se han probado.
La transducción de células diana es un paso crítico en el procedimiento. En la mayoría de las líneas celulares inmortalizadas, la transducción con Nanoblades tiene poco o ningún efecto citopático. Sin embargo, en las células primarias, la toxicidad puede ser un problema. Por lo tanto, la transducción debe optimizarse para cada tipo de célula. En concreto, el tiempo de exposición a nanoblades es un factor importante a modificar a la hora de optimizar el protocolo de transducción. Para células sensibles como neuronas primarias o células de la médula ósea, 4-6 h de incubación con Nanoblades antes de reemplazar el medio permiten una entrega eficiente de la proteína Cas9 al tiempo que minimizan la toxicidad celular. Además, los adyuvantes como los polímeros catiónicos, entre otros, pueden mejorar significativamente la eficiencia de la transducción en algunas células (ver la Tabla de Materiales). Es importante tener en cuenta que las Nanoblades son VLP y pueden inducir una respuesta inmunogénica. Esto puede ser una limitación si se trabaja con ciertos tipos de células primarias, como macrófagos o células dendríticas, en las que la incubación con Nanoblades podría inducir cambios importantes en la expresión génica y el fenotipo de las células. Si los macrófagos y las células dendríticas se derivan de precursores de células madre hematopoyéticas (como las células de médula ósea de ratón), es preferible transducir células con las Nanoblades antes de que estén completamente diferenciadas para evitar inducir una respuesta celular contra las Nanoblades. De lo contrario, la electroporación de la proteína Cas9 podría representar una alternativa viable cuando se trabaja con células inmunes diferenciadas.
Las nanocuchillas se pueden utilizar in vivo para transducir cigotos o embriones de ratón para generar animales transgénicos. Al igual que los vectores retrovirales o lentivirales clásicos, también se pueden inyectar directamente en los tejidos de animales adultos. Sin embargo, las nanocuchillas (similares a los vectores retrovirales y lentivirales) pueden ser inactivadas por la respuesta inmune del animal huésped; por lo tanto, la dosis a inyectar debe optimizarse para cada aplicación. Esta respuesta inmune también puede limitar la distribución de VLP funcionales a los tejidos cercanos al sitio de inyección. Finalmente, a diferencia de los vectores lentivirales, las Nanoblades están libres de transgenes y entregan el Cas9 en un marco de tiempo restringido. Por lo tanto, no se pueden utilizar para realizar exámenes funcionales de todo el genoma que requieren una secuenciación de alto rendimiento de sgRNAs tras la selección de células. Las nanocuchillas son útiles cuando se requiere una edición del genoma rápida, dependiente de la dosis y libre de transgenes20. Además, al igual que la electroporación de proteínas, las Nanoblades producen menos efectos fuera del objetivo que la expresión prolongada de Cas9/sgRNA a través de la transfección de ADN o vectores virales clásicos3. El desarrollo futuro de Nanoblades se centra en la incorporación de variantes cas9 para diferentes aplicaciones tecnológicas como la edición de bases y la orientación de ARN.
Divulgaciones
Philippe E. Mangeot y Emiliano P. Ricci son nombrados inventores de una patente relacionada con la tecnología Nanoblades (solicitantes de patentes: Institut National de la Santé et de la Recherche Médicale (INSERM), Centre National de la Recherche Scientifique (CNRS), Ecole Normale Superieure de Lyon, Université Claude Bernard Lyon 1, Villeurbanne Cedex; número de solicitud: WO 2017/068077 Al; estado de la patente: publicado, 27 de abril de 2017; todos los aspectos del manuscrito están cubiertos por la solicitud de patente. Los autores restantes no declaran intereses contrapuestos.
Agradecimientos
Este trabajo fue financiado por Labex Ecofect (ANR-11-LABX-0048) de la Universidad de Lyon, dentro del programa Investissements d'Avenir (ANR-11-IDEX-0007) operado por la Agencia Nacional de Investigación de Francia (ANR), Fondation FINOVI, Agence Nationale des Recherches sur le SIDA et les Hépatites Virales (ANRS-ECTZ3306) y por el Consejo Europeo de Investigación (ERC-StG-LS6-805500 a E.P.R.) en el marco de los programas de investigación e innovación Horizonte 2020 de la Unión Europea.
Materiales
| Name | Company | Catalog Number | Comments |
| 13.2 mL, Thinwall Polypropylene Tubes, 14 x 89 mm - 50Pk | Beckman Coulter Life Sciences | 331372 | Ultracentrifugation tubes for Nanoblades purification |
| Amersham Protran Premium Western blotting membranes, nitrocellulose | Merck | GE10600004 | Nitrocellulose membrane for quantifying Cas9 levels within purified Nanoblades |
| BIC-Gag-CAS9 | Addgene | 119942 | Encodes a GAG (F-MLV)-CAS9(sp) fusion. Allows the production of GAG-CAS9 Virus like particles from producer cells in association with over expressed gRNA(s) and appropriate envelopes |
| BICstim-Gag-dCAS9-VPR | Addgene | 120922 | Encodes a GAG-dCAS9-VPR fusion for targeted transcriptional activation |
| BLADE | Addgene | 134912 | Empty backbone for cloning sgRNA sequence to be used in Nanoblades system |
| BsmBI-v2 | New England Biolabs | R0739S | Restriction enzyme to digest the BLADE and SUPERBLADES vectors for sgRNA cloning |
| Cas9 (7A9-3A3) Mouse mAb (HRP Conjugate) #97982 | Cell Signaling Technology | 97982S | Anti-Cas9 antibody for Cas9 quantification by dot-blot |
| Cas9 Nuclease, S. pyogenes | New England Biolabs | M0386T | Recombinant Cas9 protein to be used as a reference for absolute quantification of the amount of Cas9 loaded within Nanoblades |
| Ethidium bromide solution (10 mg/mL in H2O) | Sigma-Aldrich | E1510-10ML | For staining agarose gels and visualize DNA |
| Fisherbrand Wave Motion Shakers | Fisher Scientific | 88-861-028 | Agitation table to resuspend Nanoblades upon centrifugation |
| gelAnalyzer | http://www.gelanalyzer.com; quantifying band intensity after digestion | ||
| Gesicle Producer 293T | Takara | 632617 | Nanoblades producer cell line |
| Gibco DMEM, high glucose, pyruvate | ThermoFisher Scientific | 41966052 | Cell culture medium for Gesicle Producer 293T cells |
| GoTaq G2 DNA Polymerase | Promega | M7848 | Taq polymerase for amplification of genomic DNA before T7 endonuclease assays |
| jetPRIME Transfection Reagent kit for DNA and DNA/siRNA | Polyplus | POL114-15 | Transfection reagent for Nanoblade production in Gesicle Producer 293T cells |
| Millex-AA, 0.80 µm, syringe filter | Millipore | SLAA033SS | Syringe filter to remove cellular debris before concentration of Nanoblades |
| Millex-GS, 0.22 µm, syringe filter | Millipore | SLGS033SS | Syringe filter to sterilise the sucrose cushion solution |
| Millex-HP, 0.45 µm, polyethersulfone, syringe filter | Millipore | SLHP033RS | Syringe filter to remove cellular debris before Nanoblades concentration |
| Monarch DNA Gel Extraction Kit | New England Biolabs | T1020L | DNA gel extraction kit for purification of the pBLADES or pSUPERBLADES plasmid fragment upon digestion with BsmBI |
| NEB Stable Competent E. coli (High Efficiency) | New England Biolabs | C3040I | Competent bacteria for plasmid transformation and amplification |
| NucleoBond Xtra Midi kit for transfection-grade plasmid DNA | Macherey-Nagel | 740410.50 | Maxipreparation kit for purification of plasmid DNA from cultured bacteria |
| Nucleospin gDNA extraction kit | Macherey-Nagel | 740952.50 | Extraction of genomic DNA from transduced cells |
| NucleoSpin Plasmid, Mini kit for plasmid DNA | Macherey-Nagel | 740588.50 | Minipreparation kit for purification of plasmid DNA from cultured bacteria |
| NucleoSpin Tissue, Mini kit for DNA from cells and tissue | Macherey-Nagel | 740952.5 | Genomic DNA extraction kit |
| Optima XE-90 | Beckman Coulter Life Sciences | A94471 | Ultracentrifuge |
| pBaEVRless | Els Verhoeyen (Inserm U1111) | Personnal requests have to be sent to: els.verhoyen@ens-lyon.fr | Baboon Endogenous retrovirus Rless glycoprotein described in Girard-Gagnepain, A. et al. Baboon envelope pseudotyped LVs outperform VSV-G-LVs for gene transfer into early-cytokine-stimulated and resting HSCs. Blood 124, 1221–1231 (2014) |
| pBS-CMV-gagpol | Addgene | 35614 | Enocdes the Murine Leukemia Virus gag and pol genes |
| pCMV-VSV-G | Addgene | 8454 | Envelope protein for producing lentiviral and MuLV retroviral particles |
| Phosphate-Buffered Saline (PBS) | ThermoFisher Scientific | 14200091 | 10X PBS to dilute in millipore water |
| Polybrene Transfection Reagent | Millipore Sigma | TR-1003-G | Cationic polymer that enhances the efficiency of retroviral transduction in specific mammalian cells. It can also allow viral-dependent entry of an Oligodeoxynucleotide (ODN) for homology-directed repair |
| Sucrose,for molecular biology, ≥99.5% (GC) | Sigma-Aldrich | S0389-5KG | Sucrose to prepare a cushion for Nanoblade purification through ultracentrifugation |
| SUPERBLADE5 | Addgene | 134913 | Empty backbone for cloning sgRNA sequence to be used in nanoblades system (Optimized for increased genome editing efficiency via Chen B et al., 2013) |
| SuperSignal West Dura Extended Duration Substrate | ThermoFisher Scientific | 34076 | Enhanced chemiluminescence (ECL) HRP substrate for Cas9 dot blots |
| SW 41 Ti Swinging-Bucket Rotor | Beckman Coulter Life Sciences | 331362 | Rotor for ultracentrifugation |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | Alternative to ethidium bromide for staining agarose gels and visualize DNA |
| T4 DNA Ligase | New England Biolabs | M0202S | DNA ligase to ligate the BLADE or SUPERBLADES vectors with the duplexed DNA oligos corresponding to the variable region of the sgRNA |
| T7 Endonuclease I | New England Biolabs | M0302S | T7 Endonuclease I recognizes and cleaves non-perfectly matched DNA. Allows to monitor the extent of genome editing at a specific locus |
| Triton-containing lysis buffer | Promega | E291A | Lysis buffer to disrupt Nanoblades and allow Cas9 quantification |
| TWEEN 20 | Sigma-Aldrich | P9416 | For the preparation of TBST |
Referencias
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zuris, J. A., et al. Cationic lipid-mediated delivery of proteins enables efficient protein-based genome-editing in vitro and in vivo. Nature Biotechnology. 33, 73-80 (2015).
- Mangeot, P. E., et al. Genome editing in primary cells and in vivo using viral-derived Nanoblades loaded with Cas9-sgRNA ribonucleoproteins. Nature Communications. 10 (1), 45 (2019).
- Cai, Y., Bak, R. O., Mikkelsen, J. G. Targeted genome editing by lentiviral protein transduction of zinc-finger and TAL-effector nucleases. eLife. 3, 01911 (2014).
- Choi, J. G., et al. Lentivirus pre-packed with Cas9 protein for safer gene editing. Gene Therapy. 23 (7), 627-633 (2016).
- Gee, P., et al. Extracellular nanovesicles for packaging of CRISPR-Cas9 protein and sgRNA to induce therapeutic exon skipping. Nature Communications. 11, 1334 (2020).
- Indikova, I., Indik, S. Highly efficient 'hit-and-run' genome editing with unconcentrated lentivectors carrying Vpr.Prot.Cas9 protein produced from RRE-containing transcripts. Nucleic Acids Research. 48 (14), 8178-8187 (2020).
- Lyu, P., Javidi-Parsijani, P., Atala, A., Lu, B. Delivering Cas9/sgRNA ribonucleoprotein (RNP) by lentiviral capsid-based bionanoparticles for efficient 'hit-and-run' genome editing. Nucleic Acids Research. 47 (17), 99 (2019).
- Gheysen, D., Jacobs, E., de Foresta, F., Thiriart, C. Assembly and release of HIV-1 precursor Pr55gag virus-like particles from recombinant baculovirus-infected insect cells. Cell. 59 (1), 103-112 (1989).
- Hanna, R. E., Doench, J. G. Design and analysis of CRISPR-Cas experiments. Nature Biotechnology. 38 (7), 813-823 (2020).
- Sambrook, J. . Molecular cloning a laboratory manual. Third edition. , (2001).
- Jiang, W., et al. An optimized method for high-titer lentivirus preparations without ultracentrifugation. Scientific Reports. 5, 13875 (2015).
- Rueden, C. T., et al. ImageJ2: ImageJ for the next generation of scientific image data. BMC Bioinformatics. 18 (1), 529 (2017).
- O'Doherty, U., Swiggard, W. J., Malim, M. H. Human immunodeficiency virus type 1 spinoculation enhances infection through virus binding. Journal of Virology. 74 (21), 10074-10080 (2000).
- Brinkman, E. K., Chen, T., Amendola, M., van Steensel, B. Easy quantitative assessment of genome editing by sequence trace decomposition. Nucleic Acids Research. 42 (22), 168 (2014).
- Bollen, Y., Post, J., Koo, B. -. K., Snippert, H. J. G. How to create state-of-the-art genetic model systems: strategies for optimal CRISPR-mediated genome editing. Nucleic Acids Research. 46 (13), 6435-6454 (2018).
- Brinkman, E. K., et al. Easy quantification of template-directed CRISPR/Cas9 editing. Nucleic Acids Research. 46 (10), 58 (2018).
- Dussaud, S., Pardanaud-Glavieux, C., Sauty-Colace, C., Ravassard, P. Lentiviral mediated production of transgenic mice: a simple and highly efficient method for direct study of founders. Journal of Visualized Experiments. (140), e57609 (2018).
- Montagna, C., et al. VSV-G-enveloped vesicles for traceless delivery of CRISPR-Cas9. Molecular Therapy. Nucleic Acids. 12, 453-462 (2018).
- Marnef, A., et al. A cohesin/HUSH- and LINC-dependent pathway controls ribosomal DNA double-strand break repair. Genes & Development. 33 (17-18), 1175-1190 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados