Method Article
Somministrazione del complesso ribonucleoproteico Cas9/sgRNA in cellule immortalizzate e primarie tramite particelle simili a virus ("Nanoblades")
In questo articolo
Riepilogo
Abbiamo sviluppato un protocollo semplice ed economico per caricare complessi di ribonucleoproteine Cas9/single-guide RNA (sgRNA) all'interno di particelle simili a virus. Queste particelle, chiamate "Nanoblades", consentono una consegna efficiente del complesso Cas9/sgRNA in cellule immortalizzate e primarie e in vivo.
Abstract
Il sistema CRISPR(Cas) a brevi ripetizioni palindrome raggruppate regolarmente intervallate ha democratizzato l'editing del genoma nelle cellule eucariotiche e ha portato allo sviluppo di numerose applicazioni innovative. Tuttavia, la consegna della proteina Cas9 e dell'RNA a guida singola (sgRNA) nelle cellule bersaglio può essere tecnicamente impegnativa. I vettori virali classici, come quelli derivati da lentivirus (LV) o virus adeno-associati (AAV), consentono una consegna efficiente di transgeni che codificano per la proteina Cas9 e il suo sgRNA associato in molte cellule primarie e in vivo. Tuttavia, questi vettori possono soffrire di inconvenienti come l'integrazione del transgene nel genoma della cellula bersaglio, una capacità di carico limitata e l'espressione a lungo termine della proteina Cas9 e dell'RNA guida nelle cellule bersaglio.
Per superare alcuni di questi problemi, è stato sviluppato un vettore di consegna basato sul virus murino della leucemia (MLV) per confezionare la proteina Cas9 e il suo RNA guida associato in assenza di qualsiasi transgene codificante. Fondendo la proteina Cas9 al C-terminus della proteina strutturale Gag da MLV, si sono formate particelle simili a virus (VLP) caricate con la proteina Cas9 e sgRNA (denominate "Nanoblades"). Le nanolame possono essere raccolte dal terreno di coltura delle cellule produttrici, purificate, quantificate e utilizzate per trasdurre le cellule bersaglio e fornire il complesso cas9 / sgRNA attivo. Le nanolame forniscono il loro carico di ribonucleoproteine (RNP) in modo transitorio e rapido in una vasta gamma di cellule primarie e immortalizzate e possono essere programmate per altre applicazioni, come l'attivazione trascrizionale transitoria di geni mirati, utilizzando proteine Cas9 modificate. Le nanolame sono in grado di modificare il genoma in vivo nel fegato di topi adulti iniettati e negli ovociti per generare animali transgenici. Infine, possono essere complessati con il DNA del donatore per una riparazione diretta all'omologia "senza trasfezione". La preparazione delle nanolame è semplice, relativamente a basso costo e può essere facilmente eseguita in qualsiasi laboratorio di biologia cellulare.
Introduzione
Rispetto ad altre nucleasi programmabili, il sistema CRISPR-Cas ha notevolmente semplificato e democratizzato la procedura di targeting e scissione del genoma specifico della sequenza nelle cellule eucariotiche. Attraverso la semplice espressione di un sgRNA, gli utenti possono programmare la proteina Cas9 (o varianti ottimizzate) per quasi tutti i locus cellulari1. In questo scenario, la somministrazione della proteina Cas9 e dell'sgRNA diventa la principale limitazione quando si esegue la mutagenesi diretta al sito. Nelle cellule immortalizzate, l'sgRNA e la proteina Cas possono essere facilmente espressi da plasmidi trasfettati per ottenere un efficiente targeting del genoma nella maggior parte delle cellule. Tuttavia, l'espressione costitutiva del complesso Cas9/sgRNA può aumentare l'attività off-target della proteina Cas9 e introdurre cambiamenti indesiderati in loci non specifici2. Nelle cellule primarie, la trasfezione del DNA può essere tecnicamente difficile da raggiungere e portare a una scarsa espressione o a una piccola percentuale di cellule trasfettate. Le alternative alla classica trasfezione del DNA comprendono l'uso di vettori virali che forniscono una codifica transgenica per cas9 e sgRNA o l'elettroporazione della proteina Cas9 ricombinante accoppiata a un sgRNA sintetico. Tuttavia, questi approcci possono portare all'integrazione transgenica all'interno del genoma dell'ospite cellulare (come nel caso dei classici vettori di espressione retrovirale e lentivirale), alla restrizione da parte di fattori cellulari e portare all'espressione costitutiva della proteina Cas9 e dell'sgRNA.
L'elettroporazione del complesso Cas9/sgRNA RNP può superare la maggior parte di questi problemi e portare a una consegna efficiente e transitoria nelle cellule primarie e in vivo, oltre a consentire una risposta dose-dipendente. Tuttavia, di solito si basa su costose attrezzature e reagenti ed è anche difficile da eseguire l'upscaling se è necessario trattare un gran numero di cellule. In alternativa alle tecniche sopra menzionate, questi autori hanno sviluppato "Nanoblades", un vettore di consegna retrovirale per la proteina Cas9 e sgRNA3 che è concettualmente simile ad altri sistemi di rilascio di proteine del capside derivate da virus4,5,6,7,8. Le nanolame sfruttano la naturale capacità della poliproteina Gag dei retrovirus di produrre, quando espresse da sole in cellule in coltura, VLP che vengono rilasciate nel mezzo extracellulare9. Fondendo la proteina Cas9 all'estremità C-terminale della poliproteina Gag del virus della leucemia murina (MLV) e co-esprimendo glicoproteine sgRNA e involucro virale, la proteina Cas9 può essere incapsulata all'interno di VLP o Nanoblade rilasciati. Al momento della purificazione, le Nanolame possono essere incubate con cellule bersaglio o iniettate in vivo per mediare la consegna rapida, transitoria e dose-dipendente del complesso RNP Cas9/sgRNA3.
Le nanolame possono essere programmate con più sgRNA per l'editing simultaneo in diversi loci o con varianti Cas9 per eseguire altre applicazioni come l'attivazione trascrizionale o la repressione specifica del bersaglio3. A differenza dell'elettroporazione proteica, che si basa sull'espressione ricombinante, le varianti Cas appena descritte dalla letteratura possono essere facilmente clonate nel vettore di espressione di fusione Gag, rendendolo una piattaforma versatile. Le nanolame possono essere ulteriormente complessate o caricate con oligodesossinucleotidi a singolo filamento e doppio filamento (ssODN) per eseguire riparazioni dirette all'omologia3. La produzione di nanolame è relativamente semplice ed economica. Inoltre, le nanolame possono essere conservate a 4 °C per molti giorni o a -80 °C per la conservazione a lungo termine. Tipicamente, le nanolame mediano l'editing del genoma efficiente e privo di transgeni nella maggior parte delle cellule immortalizzate e primarie in coltura. Tuttavia, alcune cellule primarie potrebbero essere sensibili alla presenza di particelle virali, con conseguente aumento della mortalità. Le cellule del sistema immunitario innato possono anche reagire alla presenza di Nanoblade (a causa della loro origine virale) e attivarsi. In questi casi, il protocollo di trasduzione deve essere ottimizzato per limitare il tempo di esposizione alle Nanolame e ridurre al minimo gli effetti non specifici. Le nanolame rappresentano un'alternativa praticabile e facile da implementare ad altri metodi di consegna CRISPR disponibili.
Protocollo
1. sgRNA Progettazione e clonazione
NOTA: Le linee guida per la progettazione di sgRNA possono essere ottenute da più fonti come https://blog.addgene.org/how-to-design-your-grna-for-crispr-genome-editing o da Hanna e Doench10.
- Una volta progettate le sequenze di sgRNA a 20 nucleotidi, ordinare i seguenti oligonucleotidi di DNA a singolo filamento:
- Avanti: 5' caccgNNNNNNNNNNNNNNNNNNNNNNNNNNNN 3' (N corrisponde al locus bersaglio senza la sequenza del motivo adiacente al protospacer (PAM))
- Inverso: 5' aaacNNNNNNNNNNNNNNNNNNNNNNNNNNNc 3' (N corrisponde al complemento inverso del locus bersaglio senza la sequenza PAM)
NOTA: Non sono necessarie modifiche particolari quando si ordinano gli oligonucleotidi (nessun requisito per il fosfato 5').
- Ibridare i due oligonucleotidi del DNA in un tubo di reazione a catena della polimerasi (PCR) da 0,2 mL mescolando 5 μL di tampone di ricottura (500 mM NaCl; 100 mM Tris-HCl; 100 mM MgCl2; 10 mM DTT; pH 7,9 a 25 °C), 1 μL di ciascun oligonucleotide del DNA (100 μM di soluzione madre in acqua) e 42 μL di acqua.
- Su un blocco PCR, incubare i campioni a 95 °C per 15 s e quindi diminuire la temperatura a 20 °C con una rampa di 0,5 °C/s. Conservare a temperatura ambiente o conservare a -20 °C.
NOTA: il protocollo può essere messo in pausa qui. - Digerire 10 μg dei plasmidi di espressione SGRNA BLADE o SUPERBLADE con 10 unità di enzima di restrizione BsmBI-v2 per 3 ore a 55 °C in un volume totale di reazione di 50 μL.
NOTA: il vettore digerito dovrebbe rilasciare un inserto di DNA di ~ 1,9 kb e un secondo frammento di DNA di ~ 3,3 kb. - Caricare la reazione di restrizione su un gel di agarosio all'1% colorato con 5 μg/mL di bromuro di etidio (o una colorazione alternativa più sicura del gel di DNA).
NOTA: Indossare dispositivi di protezione appropriati quando si manipola il bromuro di etidio, che è sospettato di causare difetti genetici.- Su un tavolo ultravioletto (UV) impostato a una lunghezza d'onda di 312 nm (per evitare di danneggiare il DNA), tagliare il frammento di DNA da 3,3 kb dal gel e posizionarlo in un tubo microcentrifuga da 1,5 ml.
NOTA: Indossare dispositivi di protezione appropriati (guanti e occhiali di protezione UV) quando si manipola il bromuro di etidio e si lavora sul tavolo UV. - Estrarre il DNA dal gel affettato contenente il frammento di DNA da 3,3 kb utilizzando un kit di estrazione del gel di DNA dedicato (vedi tabella dei materiali). Quantificare la quantità di DNA purificato utilizzando uno spettrofotometro.
NOTA: il protocollo può essere messo in pausa qui.
- Su un tavolo ultravioletto (UV) impostato a una lunghezza d'onda di 312 nm (per evitare di danneggiare il DNA), tagliare il frammento di DNA da 3,3 kb dal gel e posizionarlo in un tubo microcentrifuga da 1,5 ml.
- Ligate gli oligonucleotidi del DNA ibridati avanti e inverso dallo step 1.2 al vettore BLADES o SUPERBLADE digerito da BsmB1, purificato in gel dal passo 1.5.2. Per questo, aggiungere 2 μL di tampone T4 DNA ligasi, 50 ng del vettore purificato in gel (dal passo 1.5.2), 1 μL degli oligonucleotidi del DNA ibridato (dal passo 1.2), acqua per comporre il volume a 19 μL e 1 μL di T4 DNA ligasi. Incubare la reazione a 25 °C per 10 min.
- Trasformare il prodotto di legatura in batteri competenti (vedi la Tabella dei materiali) come descritto in11. Placcare i batteri trasformati su una piastra di agar Luria Bertani e incubare durante la notte a 37 °C.
- Selezionare diverse colonie isolate sulla piastra di agar per eseguire la minipreparazione del DNA11 (vedere la Tabella dei materiali) ed eseguire il sequenziamento Sanger utilizzando un primer in avanti U6 (5' GACTATCATATGCTTACCGT 3') per verificare la corretta legatura della sequenza variabile sgRNA.
NOTA: Altri plasmidi di espressione sgRNA possono essere utilizzati se non codificano per la proteina Cas9, che potrebbe interferire con la produzione di Nanoblade.
2. Preparazione del plasmide
- Eseguire la maxipreparazione (vedere la tabella dei materiali) di tutti i plasmidi richiesti e preparare aliquote da 10 μg a 1 μg/mL da conservare a -20 °C. Evitare ripetuti cicli di congelamento/scongelamento dei plasmidi; utilizzare le aliquote due volte prima di scartarle.
3. Preparazione nanoblade
- Il giorno 1, il seme tra 3,5 e 4 × 106 cellule HEK293T (vedi la tabella dei materiali) in 10 ml di mezzo Eagle modificato di Dulbecco (DMEM) contenente glucosio alto, piruvato di sodio, L-glutammina, siero bovino fetale al 10% (FBS) e penicillina / streptomicina in un piatto di coltura cellulare di 10 cm. Spostare delicatamente la piastra di 10 cm avanti e indietro, quindi da destra a sinistra (ripetere questa sequenza 5x) per distribuire le cellule in modo omogeneo sul piatto di coltura. Incubare le cellule a 37 °C in un incubatore cellulare con il 5% di CO2.
NOTA: Tutte le procedure relative alla manipolazione delle cellule in coltura e delle Nanolame devono essere eseguite sotto una cappa a flusso laminare di coltura cellulare per evitare la loro contaminazione. - Giorno 2: Trasfezione del plasmide
- Le cellule dovrebbero essere confluenti al 70-80% 24 ore dopo la placcatura (Figura 1A). Sostituire il mezzo con 10 ml di DMEM fresco contenente glucosio alto, piruvato di sodio, L-glutammina, FBS al 10% (penicillina e streptomicina possono essere omesse anche se non è obbligatorio) prima della trasfezione.
NOTA: in questa fase, è importante che le cellule non siano confluenti. In caso contrario, l'efficienza di trasfezione e la produzione di particelle potrebbero essere ridotte. - Per ogni piastra da 10 cm, preparare le seguenti quantità di plasmidi in un tubo da 1,5 mL: 0,3 μg pCMV-VSV-G, 0,7 μg pBaEVRless, 2,7 μg MLV Gag/Pol, 1,7 μg BIC-Gag-Cas9, 4,4 μg di BLADES o plasmide SUPERBLADES che codifica per lo sgRNA clonato (o 2,2 μg ciascuno se si utilizzano due sgRNA).
- Aggiungere 500 μL di tampone di trasfezione (vedere la tabella dei materiali), vortice per 10 s e quindi centrifuga per 1 s. Aggiungere 20 μL del reagente di trasfezione (vedere la tabella dei materiali), ruotare il tubo per 1 s e quindi centrifugare per 1 s.
- Incubare per 10 minuti a temperatura ambiente e aggiungere l'intera soluzione a goccia alle celle in mezzo DMEM utilizzando un pipettor P1000. Spostare delicatamente la piastra di 10 cm avanti e indietro, quindi da destra a sinistra (ripetere questa sequenza 5x) per distribuire uniformemente il reagente di trasfezione sulle cellule. Incubare le cellule a 37 °C per almeno 40 ore in un incubatore cellulare con il 5% di CO2.
NOTA: Se lo si desidera, il mezzo può essere cambiato 4 ore dopo la trasfezione.
- Le cellule dovrebbero essere confluenti al 70-80% 24 ore dopo la placcatura (Figura 1A). Sostituire il mezzo con 10 ml di DMEM fresco contenente glucosio alto, piruvato di sodio, L-glutammina, FBS al 10% (penicillina e streptomicina possono essere omesse anche se non è obbligatorio) prima della trasfezione.
- Il giorno 3, controlla la morfologia delle cellule trasfettate al microscopio.
NOTA: le celle del produttore inizieranno a fondersi. Questo è un evento normale a causa dell'espressione di involucri virali fusogeni (Figura 1B,C). - Giorno 4: Raccolta delle Nanolame
NOTA: Almeno 40 ore dopo la trasfezione, le cellule si sarebbero fuse insieme a causa dell'espressione degli involucri virali fusogeni e, a volte, le cellule sono completamente staccate dal supporto della piastra (Figura 1D).- Raccogliere 9 mL del surnatante del terreno di coltura utilizzando una pipetta da 10 ml.
NOTA: Le nanolame sono VLP in grado di fornire la proteina Cas9 e il suo sgRNA associato nelle cellule primarie e in vivo. Sebbene non siano considerati organismi geneticamente modificati in quanto privi di materiale genetico, possono indurre cambiamenti genetici. Pertanto, devono essere manipolati con cautela per evitare qualsiasi contatto con gli utenti (specialmente se sono programmati per colpire i geni oncosoppressori). Si consiglia agli utenti di seguire le loro linee guida di sicurezza locali per la manipolazione di vettori retrovirali e di lavorare in un laboratorio di livello BSL-2 durante la preparazione di VLP e l'esecuzione di esperimenti di trasduzione. Le nanolame possono essere inattivate con il 70% di etanolo o lo 0,5% di ipoclorito di sodio. Si consiglia inoltre di trattare tutti i rifiuti plastici (punte di pipette, piastre di coltura tissutale, tubi di centrifugazione) con ipoclorito di sodio allo 0,5% per almeno 10 minuti per inattivare le Nanoblade. - Centrifugare il surnatante raccolto a 500 × g per 5 minuti per rimuovere i detriti cellulari e recuperare il surnatante senza disturbare il pellet cellulare.
NOTA: se le nanolame devono essere utilizzate su cellule primarie, filtrare il surnatante utilizzando un filtro da 0,45 μm o 0,8 μm. Tieni presente che questo passaggio riduce drasticamente il titolo Nanoblade poiché una frazione significativa verrà bloccata nella membrana del filtro. - Pellet le Nanolame durante la notte (12-16 h) in un rotore a benna oscillante a 4.300 × g o a 209.490 × g in un'ultracentrifuga per 75 min a 4 °C (vedi la Tabella dei Materiali).
NOTA: Se le cellule bersaglio possono crescere in DMEM, è possibile incubarle direttamente con il surnatante ottenuto dopo il passo 3.4.2 senza concentrare le Nanolame.
- Raccogliere 9 mL del surnatante del terreno di coltura utilizzando una pipetta da 10 ml.
- Giorno 5: Risospensione e conservazione delle Nanolame
- Dopo la centrifugazione, aspirare lentamente il mezzo e risospese il pellet bianco con 100 μL di soluzione salina fredda tamponata con 1x fosfato (PBS). Coprire il tubo con parafilm e incubare per 1 ora a 4 °C con una leggera agitazione prima di risospendere il pellet mediante pipettatura su e giù.
NOTA: un materiale viscoso bianco può apparire al momento della risospensione; questo è normale e non influisce in modo significativo sull'efficienza della trasduzione. - Conservare le Nanolame a 4 °C se si prevede di utilizzarle entro quattro settimane. In caso contrario, congelare a scatto le Nanolame in azoto liquido e conservarle a -80 °C.
NOTA: Indossare occhiali di protezione e guanti criogenici quando si manipola l'azoto liquido. Il congelamento e lo stoccaggio a scatto a -80 °C portano a una significativa diminuzione dell'efficienza di Nanoblade. Inoltre, le Nanolame scongelate non dovrebbero essere nuovamente congelate. Il protocollo può essere messo in pausa qui.
- Dopo la centrifugazione, aspirare lentamente il mezzo e risospese il pellet bianco con 100 μL di soluzione salina fredda tamponata con 1x fosfato (PBS). Coprire il tubo con parafilm e incubare per 1 ora a 4 °C con una leggera agitazione prima di risospendere il pellet mediante pipettatura su e giù.
4. Concentrazione di Nanolame su un cuscino di saccarosio
NOTA: In alternativa alla centrifugazione notturna o all'ultracentrifugazione (fase 3.4.3), le Nanolame possono essere concentrate su un cuscino di saccarosio. Ciò produce una frazione più pura di Nanoblade, anche se la quantità totale recuperata sarà inferiore.
- Preparare una soluzione di saccarosio al 10% (da peso a volume) in 1x PBS e filtrarla attraverso un filtro a siringa da 0,2 μm (vedere la Tabella dei materiali).
- Inizia il processo di concentrazione delle Nanolame sul cuscino di saccarosio.
- Introdurre 9 mL di campione contenente VLP (dal punto 3.4.3) in un tubo ultracentrifuga (vedere la tabella dei materiali). Utilizzando una siringa da 3 mL e una cannula, stratificare lentamente 2,5 mL del saccarosio al 10% sotto il campione, cercando di non mescolare il campione contenente VLP e la soluzione di saccarosio.
- In alternativa, posizionare 2,5 ml di saccarosio al 10% in un tubo ultracentrifuga (vedere la tabella dei materiali). Inclinare il tubo e aggiungere lentamente i 9 mL di campione contenente VLP (dal punto 3.4.3) con un pipettor a bassa velocità. Durante questa operazione, sollevare progressivamente il tubo in posizione verticale.
- Centrifugare i campioni a 209.490 × g in un ultracentrifuga per 90 minuti a 4 °C.
NOTA: Questa tecnica può essere adattata per la centrifugazione a bassa velocità (4.300 × g) durante la notte come descritto in 12. - Dopo la centrifugazione, rimuovere accuratamente il surnatante e posizionare il tubo a testa in giù su carta velina per rimuovere il liquido rimanente. Dopo 1 min, aggiungere 100 μL di 1x PBS e posizionare il tubo a 4 °C con un coperchio parafilm in un supporto per tubi su un tavolo di agitazione per 1 ora (vedere la Tabella dei materiali) prima di riutilizzare il pellet mediante pipettaggio su e giù.
NOTA: il protocollo può essere messo in pausa qui.
5. Monitoraggio del caricamento di Cas9 all'interno di Nanoblades tramite dot-blot
- Preparare il tampone di diluizione aggiungendo 1 volume di tampone di lisi contenente un tensioattivo non ionico (vedere la Tabella dei materiali) in 4 volumi da 1x PBS. Diluire brevemente 2 μL di Nanolame concentrate in 50 μL di tampone di diluizione, vortice e trasferire 25 μL di questa miscela in un nuovo tubo contenente 25 μL di tampone di diluizione. Ripetere questa operazione per avere 4 tubi di diluizioni Nanoblade (fasi di diluizione 2 volte).
- Per i controlli standard, diluire brevemente 2 μL di nucleasi Cas9 ricombinante (vedere la Tabella dei materiali) in 50 μL di tampone di diluizione, vortice brevemente, e procedere a fare otto diluizioni seriali (diluizione 2 volte per ogni passaggio).
- Individuare con attenzione 2,5 μL di ciascuna diluizione VLP e 2,5 μL di ciascuno standard su una membrana di nitrocellulosa con un pipetto multicanale (un volume maggiore può causare punti sovrapposti).
NOTA: può essere utilizzata anche una membrana in polivinilendifluoruro trattata con metanolo. - Una volta che le particelle vengono assorbite sulla membrana, bloccare la membrana con 1x soluzione salina tamponata tris contenente un tensioattivo non ionico (TBS-T) integrato con latte secco non grasso (5% p / v) per 45 minuti a temperatura ambiente.
NOTA: il protocollo può essere messo in pausa qui e la membrana memorizzata a 4 °C in 1x TBS-T. - Scartare l'1x TBST integrato con latte secco non grasso e incubare la membrana durante la notte a 4 °C con l'anticorpo Cas9-rafano perossidasi (diluizione 1/1000 in 1x TBST, 5% latte). Lavare la membrana 3 volte con TBS-T e visualizzare il segnale utilizzando un kit di substrato chemiluminescente migliorato.
- Quantificare l'intensità dei punti per le diluizioni standard Nanoblades e Cas9 ricombinanti utilizzando il software proprietario fornito con la stazione di imaging in gel o imageJ13. Definite una curva lineare che colleghi l'intensità del punto alla concentrazione cas9. Utilizzando la funzione della curva ottenuta, estrapolare il contenuto di Cas9 in ogni preparazione.
NOTA: La quantità di controllo della proteina Cas9 ricombinante può saturare la lettura per i campioni più concentrati del set di diluizione standard (Figura 2). Si consiglia quindi, in sede di definizione della curva lineare, di rimuovere la lettura dai campioni non diluiti (e talvolta quella delle prime fasi di diluizione) se non si trovano nell'intervallo lineare rispetto alla concentrazione nota di Cas9 che è stata individuata. Allo stesso modo, quando si estrapola la quantità di Cas9 all'interno dei campioni Nanoblade, utilizzare solo le letture che si trovano all'interno dell'intervallo lineare della curva standard.
6. Trasduzione di cellule bersaglio con Nanoblades (procedura per la trasduzione in una piastra a 12 pozzetti)
- In un piatto a 12 pozzetti, seminare 100.000-200.000 cellule (cellule aderenti primarie o immortalizzate) per pozzetto in 1 mL del terreno di coltura cellulare appropriato. Lasciare che le cellule aderiscano alla superficie della piastra prima della trasduzione.
- In un tubo microcentrifuga da 1,5 mL, aggiungere 5-20 μL di Nanoblade concentrate (dal passo 3.5.1 o 4.4) a 500 μL di terreno di coltura cellulare e mescolare pipettando su e giù con un pipettatore P1000. Rimuovere il mezzo dalle cellule e sostituirlo con i 500 μL di questa miscela Nanoblade.
NOTA: la trasduzione deve essere ottimizzata per ogni tipo di cella. È importante utilizzare il minor volume possibile di mezzo (evitando l'essiccazione delle cellule bersaglio) in modo che le Nanolam rimangano altamente concentrate. Le cellule aderenti devono essere trasdotte direttamente mentre sono attaccate alla piastra (non trasdurre in sospensione in quanto ciò ridurrà significativamente l'efficienza di trasduzione). Alcune cellule tollerano l'esposizione prolungata a Nanoblades (24-48 h) mentre altre sono molto sensibili e possono formare una piccola sincizia. In questo caso, le Nanolame devono essere incubate con le cellule solo per 4-6 ore prima di sostituire il mezzo. La spinoculazione14 può anche migliorare la trasduzione per le cellule cresciute in sospensione. Gli adiuvanti come i polimeri cationici (vedi la Tabella dei materiali) possono anche migliorare l'efficienza di trasduzione in alcuni tipi di cellule. - Dopo 4-6 ore di incubazione cellulare in un basso volume di mezzo contenente Nanolame, aumentare il volume del mezzo alla quantità normale (1 mL se si lavora con una piastra a 12 pozzetti), o sostituirlo con mezzo fresco se le cellule sono sensibili ai VLP.
NOTA: Il mezzo cellulare contenente Nanoblades deve essere inattivato con ipoclorito di sodio allo 0,5% per 10 minuti prima di scartarlo. Utilizzare guanti e occhiali protettivi quando si manipola l'ipoclorito di sodio. Se le nanolame inducono la morte cellulare, adattare la quantità e il tempo totale di esposizione per ridurre la mortalità cellulare.
7. Misurazione dell'efficienza CRISPR al locus mirato mediante il test dell'endonucleasi T7
- Progettare primer PCR per amplificare una regione di 400-700 coppie di basi (bp) che comprende il sito di scissione CRISPR.
NOTA: Il sito di scissione deve essere distante dal bordo dell'amplicon di almeno 200 bp e deve essere leggermente spostato dal centro dell'amplicon in modo che alla scissione dell'endonucleasi T7, vengano rilasciati 2 frammenti di diverse dimensioni. - Estrarre dna genomico da cellule trattate con Nanoblades mirate al gene di interesse e da cellule di controllo trattate con Nanoblade programmate con un sgRNA di controllo (vedi la Tabella dei Materiali).
NOTA: il protocollo può essere messo in pausa qui. - Utilizzando 150 ng di DNA genomico come modello, programmare una reazione PCR di 30 μL di volume (volume finale) seguendo il protocollo del produttore. Verificare che l'amplificazione PCR produca un singolo amplicon della dimensione prevista eseguendo un gel di agarosio al 2% colorato con 5 μg/mL di bromuro di etidio (o una colorazione alternativa più sicura del gel di DNA).
NOTA: Indossare dispositivi di protezione appropriati quando si manipola il bromuro di etidio, che è sospettato di causare difetti genetici. Il protocollo può essere messo in pausa qui. - Generazione e digestione di eteroduplex
- In un tubo PCR da 0,2 mL, aggiungere 5 μL del tampone enzimatico (fornito con l'endonucleasi T7 I), 20 μL di acqua e 24 μL del prodotto PCR dal passaggio 7.3. Consentire la formazione di eteroduplex riscaldando i campioni a 94 °C nell'arco di 3 minuti e quindi diminuendo la temperatura (2 °C al minuto) fino a raggiungere i 40 °C.
- Aggiungere 0,5 μL di T7-endonucleasi I a temperatura ambiente a ciascun tubo eteroduplex, incluso il controllo. Incubare a 37 °C per 15 min. Caricare la reazione risultante in un gel di agarosio al 2,5% (peso/volume) colorato con bromuro di etidio. Dopo la migrazione, visualizzare il gel su un transilluminatore UV.
NOTA: Indossare dispositivi di protezione appropriati quando si manipola il bromuro di etidio, che è sospettato di causare difetti genetici. Utilizzare occhiali di protezione UV quando si utilizza il transilluminatore UV. - Misurare le efficienze di scissione analizzando l'immagine risultante dalla reazione di digestione per quantificare l'intensità di ciascuna banda con un software appropriato (vedi la Tabella dei Materiali).
8. Misurazione dell'efficienza CRISPR nel locus mirato mediante sequenziamento Sanger e analisi TIDE
NOTA: In alternativa al test dell'endonucleasi T7, l'efficienza CRISPR può essere monitorata mediante analisi e deconvoluzione delle tracce di sequenziamento Sanger basate sul protocollo TIDE15.
- Eseguire il sequenziamento Sanger degli ampliconi PCR dal passaggio 7.3 (includere una condizione di controllo corrispondente alle cellule non trattate) utilizzando il primer PCR avanti o indietro.
- Analizzare le tracce di sequenziamento Sanger della condizione di controllo (cellule non trattate) e campioni trattati con Nanoblade utilizzando il server TIDE (https://tide.nki.nl) e seguendo le loro linee guida di analisi.
9. Formazione di complessi di nanolame con donatori ssODN per la riparazione diretta all'omologia (procedura per la trasduzione in una piastra a 12 pozzetti)
NOTA: le linee guida per la progettazione di ssODN per un efficiente editing mediato dalla riparazione diretto all'omologia sono state descritte in precedenza16.
- In una piastra a 12 pozzetti, seminare 100.000-200.000 cellule per pozzetto in 1 mL del terreno di coltura cellulare appropriato. Lasciare che le cellule aderiscano alla superficie della piastra prima della trasduzione.
- Preparare 100 μL di una soluzione del polimero cationico (vedere la Tabella dei materiali) a 8 μg/mL in 1x PBS.
- Miscelare 19 μL della soluzione polimerica cationica con 100 pmol del modello ssODN. Aggiungere 20 μL di Nanoblade concentrate (dal punto 3.5.1 o 4.4) e incubare per 15 minuti sul ghiaccio.
- Rimuovere le Nanolame/ssODN complessate dal ghiaccio e aggiungere 500 μL di terreno di coltura cellulare (a 37 °C). Rimuovere il mezzo dalle cellule bersaglio (dal punto 9.1) e aggiungere i 500 μL di mezzo contenente le Nanolame/ssODN complessate. Lasciare che le cellule proliferino per 48 ore prima della genotipizzazione.
- Estrarre il DNA genomico da una frazione della popolazione cellulare utilizzando un kit di estrazione dedicato (vedi la Tabella dei Materiali).
- Progettare primer PCR per amplificare una regione di 400-700 bp che comprende il sito knock-in.
NOTA: i primer PCR non devono sovrapporsi ai bracci di omologia dell'ssODN per evitare risultati falsi positivi derivanti dall'amplificazione PCR di eventuali ssODN residui ancora presenti all'interno delle cellule bersaglio. - Utilizzando 150 ng di DNA genomici da cellule di controllo (non trattate) o cellule trattate con Nanoblade come modello, programmare una reazione PCR da 30 μL seguendo il protocollo del produttore.
NOTA: tracce di ssODN possono essere presenti nel mezzo cellulare diversi giorni dopo la trasduzione con il complesso. Questo ssODN può servire come modello parziale per i test PCR che tentano di schermare la corretta integrazione. Pertanto, è consigliabile passare le cellule almeno due volte dopo la trasduzione, per evitare eventuali test falsi positivi. - Caricare 5 μL delle reazioni PCR di controllo e trattate con Nanoblade in un gel di agarosio all'1% (peso/volume) colorato con bromuro di etidio. Dopo la migrazione, visualizzare il gel su un transilluminatore UV.
NOTA: Se la ricombinazione omologica ha esito positivo e corrisponde all'inserimento di più di 1 bp di materiale genetico, dovrebbe esserci una differenza nel peso molecolare degli ampliconi PCR tra il campione di controllo e quello trattato con Nanoblade. Poiché l'efficienza dell'HDR non raggiunge il 100%, due bande dovrebbero essere visibili nel campione trattato con Nanoblade (una di dimensioni simili all'amplicon PCR di controllo corrispondente all'allele inedito e una di peso molecolare più elevato corrispondente all'allele knock-in, vedere il pannello centrale della Figura 3B).
- Progettare primer PCR per amplificare una regione di 400-700 bp che comprende il sito knock-in.
- Eseguire il sequenziamento Sanger degli ampliconi PCR di controllo e trattati con Nanoblade.
- Quantificare l'efficienza knock-in utilizzando il protocollo TIDER17.
10. Somministrazione di nanoblade in vivo
- Fornire fino a 25 μL di Nanoblade concentrate dallo stadio 3.5.1 attraverso l'iniezione retro-orbitale o fino a 100 μL attraverso l'iniezione della vena della coda, come descritto in18, se si lavora con topi.
NOTA: Tutte le procedure che coinvolgono la sperimentazione animale (comprese le iniezioni di Nanoblade per scopi di modifica del genoma) richiedono un protocollo approvato da un comitato etico locale. - Per la generazione di topi transgenici, utilizzare un microiniettore per erogare da 1 pL a 10 pL di Nanoblades concentrati dal passo 4.4 nello spazio perivitellino degli ovociti di topo come descritto in precedenza18.
NOTA: Per l'iniezione di perivitellina, è essenziale purificare e concentrare Nanoblades su un cuscino di saccarosio per evitare l'intasamento del microiniettore.
Risultati
Il protocollo per la preparazione di Nanoblade è abbastanza semplice e richiede semplici attrezzature di laboratorio oltre all'accesso a una cappa di coltura tissutale, un incubatore a CO2 e una centrifuga a secchio oscillante o un ultracentrifuga. Tuttavia, alcuni passaggi richiedono particolare attenzione come la fonte e la gestione delle cellule produttrici, nonché le condizioni di trasduzione. Come mostrato nella Figura 1A, è importante seminare le cellule in modo che siano distribuite in modo omogeneo nella piastra e raggiungano una confluenza di ~ 70-80% il giorno della trasfezione (evitare di avere grumi di cellule). Ventiquattro ore dopo la trasfezione (Figura 1B, C), le cellule produttrici formeranno sincizia portando a cellule di dimensioni maggiori con nuclei multipli. Quaranta ore dopo la trasfezione (Figura 1D), la maggior parte delle cellule nella piastra avrà formato sincizia e inizierà a staccarsi dalla piastra.
Questo è perfettamente normale ed è causato dall'espressione della glicoproteina dell'involucro, che induce la fusione tra cellule vicine. Alla concentrazione per centrifugazione (o anche direttamente dal surnatante delle cellule produttrici), la quantità di Cas9 caricata all'interno di Nanoblade può essere quantificata in modo assoluto da dot-blot su una membrana di nitrocellulosa usando Cas9 ricombinante come riferimento (Figura 2). Questo passaggio è importante per determinare la quantità corretta di Nanoblade da utilizzare per la trasduzione delle cellule bersaglio. Quando si esegue il test dot-blot, è importante considerare solo le letture che rientrano nell'intervallo lineare della curva standard. Tuttavia, indipendentemente dalla quantità di Cas9 presente all'interno di Nanoblades, è essenziale testare l'efficienza dell'editing del genoma direttamente sulle cellule bersaglio utilizzando il test dell'endonucleasi T7 (Figura 3) o il sequenziamento Sanger.
Come mostrato nella Figura 3, l'efficienza delle Nanoblade può differire da lotto a lotto, anche se di solito è correlata alla quantità di Cas9. Nell'esempio mostrato nella Figura 3, il batch dalla corsia 1 porta al 20% di efficienza complessiva di modifica mentre il batch dalla corsia 3 porta al 60% di efficienza. In questo caso, è possibile aumentare il volume delle Nanolame utilizzate dal lotto 1 per ottenere un'efficienza di editing simile a quella del lotto 3. La Figura 4 mostra la massima efficienza di editing ottenuta utilizzando Nanoblades in diversi tipi di celle primarie. È importante notare che l'efficienza può variare a seconda della sequenza dello sgRNA utilizzato e dell'accessibilità target.
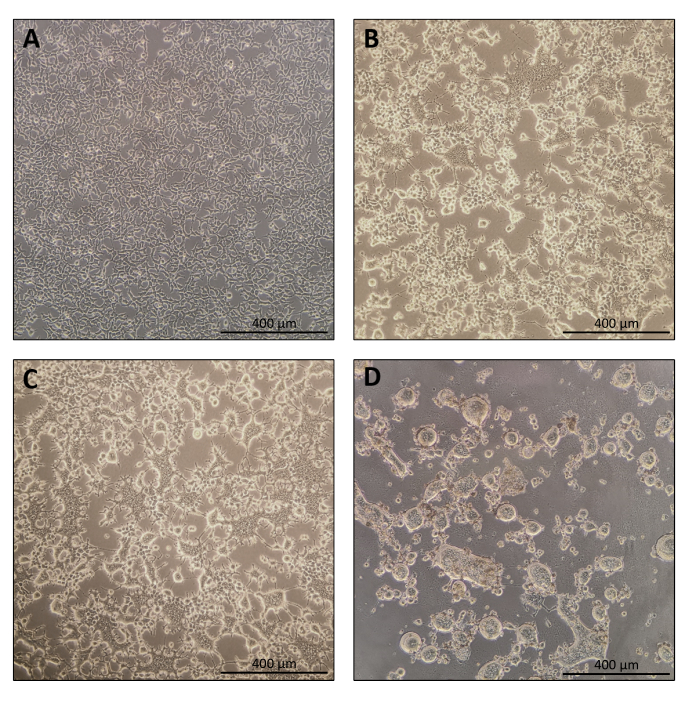
Figura 1: Morfologia delle cellule produttrici durante la produzione di Nanoblade. (A) Celle HEK293T alla confluenza del 70-80% 24 ore dopo la placcatura. (B e C) Morfologia cellulare HEK293T 24 ore dopo la trasfezione. (D) Morfologia cellulare HEK293T 40 ore dopo la trasfezione. Barre di scala = 400 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
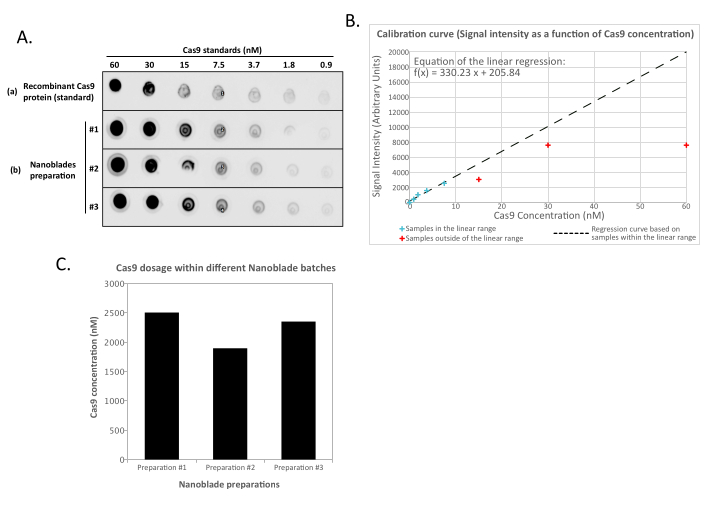
Figura 2: Quantificazione del carico Cas9 all'interno di Nanoblades mediante dot-blot. (A) Cas9 ricombinante o concentrato 100x (mediante ultracentrifugazione) I campioni di Nanoblade (#1, #2 e #3) vengono diluiti 2 volte sequenzialmente e individuati su una membrana di nitrocellulosa prima di incubare con anticorpi accoppiati HRP anti-Cas9. Il segnale è rivelato da una chemiluminescenza migliorata. (B) Il segnale di chemiluminescenza viene acquisito e quantificato per le diluizioni Cas9 ricombinanti e l'intensità del segnale tracciata rispetto alla quantità nota di Cas9 individuata sulla membrana della nitrocellulosa. Una curva di regressione viene calcolata per le diluizioni che si trovano all'interno dell'intervallo lineare (vedi croci blu), escludendo tutte le concentrazioni che sono al di fuori dell'intervallo lineare (vedi croci rosse). (C) La concentrazione di Cas9 (nM) in ogni preparazione Nanoblade è stata estrapolata usando l'equazione della regressione lineare ottenuta in (B). Per questo, è importante utilizzare solo il segnale quantificato dalle diluizioni Nanoblade che rientrano nell'intervallo lineare della curva di regressione. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 3: Monitoraggio dell'efficienza di editing sulla trasduzione. (A) Saggio di endonucleasi T7 che misura l'efficienza di scissione in cellule trattate con Nanoblade. Le cellule trasdotte con Nanoblades mirate al gene EMX1 sono state analizzate mediante test di endonucleasi T7. Corsia 1: Lotto di preparazione Nanoblade #1 (efficienza di scissione del 20%); Corsia 2: Celle di controllo; Corsia 3: Lotto di preparazione Nanoblade #2 (efficienza di scissione del 60%). (B) Knock-in della sequenza Flag-tag all'interno del frame di lettura aperto DDX3 . Le nanolame concentrate programmate con un sgRNA mirato al locus DDX3 sono state prodotte da diversi cloni hek293T (# 1, # 2) e complessate con dosi crescenti di un modello ssODN Flag-DDX3 e i complessi ottenuti utilizzati per la trasduzione delle cellule bersaglio HEK293T. Dopo la trasduzione, le cellule sono state coltivate per tre giorni prima di raccoglierle per estrarre il DNA genomico e le proteine totali. Le proteine Flag-DDX3 sono state immunoprecipitate utilizzando perline di agarosio anti-Flag seguite da un'analisi western-blot delle proteine recuperate utilizzando un anticorpo anti-Flag (pannello superiore). Anche l'inserimento diretto al sito del Flag-tag nel locus Ddx3 è stato analizzato mediante PCR utilizzando primer che fiancheggiano il sito di inserimento (pannello centrale) o utilizzando un primer in avanti che riconosce la sequenza Flag-tag e un primer inverso specifico per il locus Ddx3 a valle del sito di inserimento Flag (pannello inferiore). Abbreviazioni: EMX1 = Spiracles Vuoto Homeobox 1; DDX3 = DEAD-box RNA elicasi 3; PCR = reazione a catena della polimerasi; ODN = oligodesossinucleotide; ssODN = ODN a sing-stranded; sgRNA = RNA a guida singola; IP = immunoprecipitazione. Fare clic qui per visualizzare una versione più grande di questa figura.
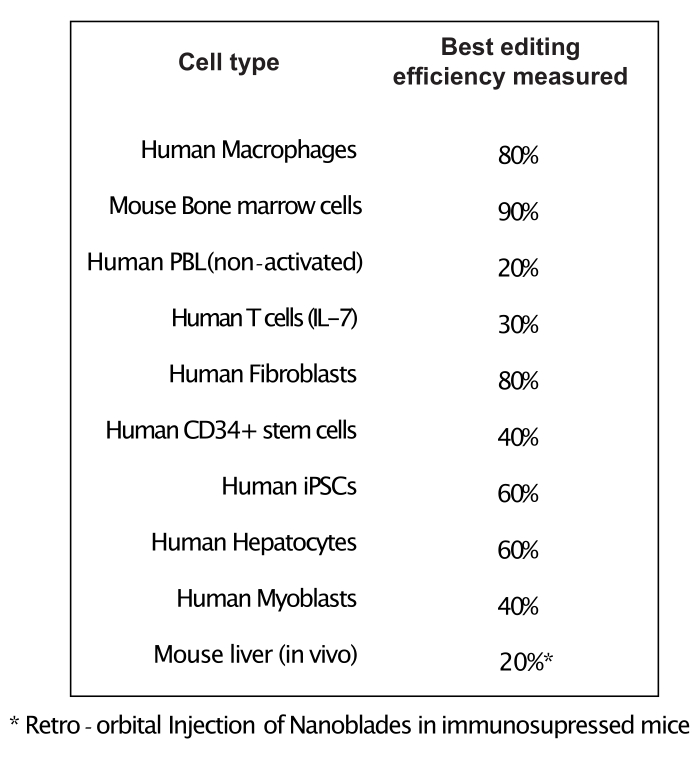
Figura 4: Efficienza di editing ottenuta in diversi tipi di cellule primarie utilizzando Nanoblades. Abbreviazioni: PBL = linfocita del sangue periferico; IL = interleuchina; CD = cluster di differenziazione; iPSC = cellule staminali pluripotenti indotte. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Le nanolame consentono la somministrazione rapida e dose-dipendente del complesso RNP Cas9/sgRNA nelle linee cellulari e nelle cellule primarie. A differenza della trasfezione classica e di altri vettori di consegna virale, ma come l'elettroporazione proteica, le nanolame hanno il vantaggio della consegna transitoria del Cas9 / sgRNA RNP in modo privo di transgene. Nanoblades offre una piattaforma altamente versatile, semplice ed economica per la somministrazione di proteine che può essere facilmente e rapidamente adattata alla famiglia in continua espansione di varianti CRISPR. Le nanolame possono essere prodotte nella linea cellulare HEK293T o nei suoi derivati. Le linee cellulari HEK293T qui utilizzate sono state sviluppate per massimizzare la produzione di particelle retrovirali e lentivirali (vedere la Tabella dei materiali). Tuttavia, sebbene altre fonti di celle HEK293T possano essere adatte, gli utenti devono testare e confrontare le cellule HEK293T da fonti diverse poiché sono state osservate importanti differenze nella produzione di particelle a seconda della sorgente cellulare HEK293T. Le cellule devono anche essere controllate frequentemente per la contaminazione da Mycoplasma e passate ogni tre giorni (classicamente 1/8 di diluizione) per evitare un'eccessiva confluenza, che ha un impatto negativo sulla produzione di particelle.
Le cellule non devono essere mantenute per oltre 20 passaggi. DMEM integrato con glucosio, penicillina / streptomicina, glutammina e il 10% di siero bovino fetale decomplementato è stato utilizzato per la coltura cellulare. Poiché l'origine del siero può influire sulla qualità del preparato Nanoblade, diversi lotti di siero devono essere testati prima della produzione su larga scala. Le nanolame possono essere prodotte in modo efficiente in altri mezzi come RPMI o modifiche prive di siero di un mezzo essenziale minimo che può sostituire DMEM il giorno successivo alla trasfezione. Come indicato di seguito, sebbene la sostituzione del mezzo dopo la trasfezione con alcuni reagenti di trasfezione del DNA sia facoltativa, può essere utile modificare il mezzo in cui vengono rilasciati i VLP, in particolare per limitare le tracce sieriche nella preparazione delle particelle. Tuttavia, la coltivazione di cellule in un mezzo essenziale minimo sierico ridotto il giorno prima della trasfezione non è stata ancora tentata.
Come accennato, le nanolame sono prodotte dopo la sovraespressione di una miscela di plasmidi nelle cellule produttrici. La sovraespressione sembra essere necessaria per una produzione ottimale. In effetti, questo laboratorio ha sviluppato una linea cellulare di produzione in cui il costrutto che esprime Gag-Pol è stato stabilizzato dalla selezione antibiotica; tuttavia, questo sistema non è riuscito a produrre quantità significative di Nanoblade. Un'osservazione simile è stata fatta quando il costrutto di codifica sgRNA è stato stabilmente integrato nel genoma delle cellule produttrici. Come descritto per altri sistemi di produzione di particelle, una linea cellulare stabile che esprima almeno alcuni costrutti coinvolti nella produzione di Nanoblades può essere utile; tuttavia, ciò richiederebbe certamente l'elaborazione di grandi volumi di surnatante e una tecnica appropriata per purificare le particelle. Il protocollo di cui sopra delinea la procedura preferita per produrre Nanoblades che sfrutta specifici reagenti di trasfezione (vedi tabella dei materiali).
Sebbene anche i reagenti di trasfezione di altri produttori siano stati testati con successo, la stragrande maggioranza dei risultati di questo gruppo con Nanoblades segue la procedura descritta nel presente documento. La trasfezione a basso costo può essere ottenuta utilizzando reagenti di fosfato di calcio e produrre una buona efficienza produttiva; tuttavia, questo metodo richiede assolutamente la sostituzione del mezzo di trasfezione il giorno successivo alla trasfezione e può lasciare residui di fosfato di calcio nella preparazione di particelle sedimentate. Coerente con la necessità di alti livelli di espressione per i componenti Nanoblade all'interno delle cellule produttrici è l'osservazione che la quantità di sgRNA associati alla proteina Cas9 può essere un fattore limitante per un efficiente editing del genoma. Per migliorare il carico di sgRNA, due approcci tecnici sono stati recentemente sviluppati da gruppi indipendenti che utilizzano vettori di consegna proteica simili a Nanoblades. Questi si basano sull'uso dell'espressione citoplasmatica T7 polimerasi-dipendente di sgRNA6 o attraverso l'aggiunta di un segnale di incapsidazione retrovirale alla sequenza sgRNA per mediare il legame con la poliproteina Gag6. Questi approcci potrebbero effettivamente migliorare il carico di sgRNA all'interno di Nanoblades anche se non sono ancora stati testati.
La trasduzione delle cellule bersaglio è un passo fondamentale nella procedura. Nella maggior parte delle linee cellulari immortalizzate, la trasduzione con Nanoblades ha poco o nessun effetto citopatico. Tuttavia, nelle cellule primarie, la tossicità può essere un problema. La trasduzione deve quindi essere ottimizzata per ogni tipo di cellula. In particolare, il tempo di esposizione alle Nanoblade è un fattore importante da modificare quando si ottimizza il protocollo di trasduzione. Per le cellule sensibili come i neuroni primari o le cellule del midollo osseo, 4-6 ore di incubazione con Nanoblades prima di sostituire il mezzo consentono una consegna efficiente della proteina Cas9 riducendo al minimo la tossicità cellulare. Inoltre, i coadiuvanti come i polimeri cationici, tra gli altri, possono migliorare significativamente l'efficienza della trasduzione in alcune celle (vedi la Tabella dei materiali). È importante notare che le nanolame sono VLP e possono indurre una risposta immunogenica. Questa può essere una limitazione se si lavora con alcuni tipi di cellule primarie, come macrofagi o cellule dendritiche, in cui l'incubazione con Nanoblades potrebbe indurre importanti cambiamenti nell'espressione genica e nel fenotipo delle cellule. Se i macrofagi e le cellule dendritiche derivano da precursori di cellule staminali ematopoietiche (come le cellule del midollo osseo del topo), è preferibile trasdurre le cellule con le Nanolame prima che siano completamente differenziate per evitare di indurre una risposta cellulare contro le Nanolame. Altrimenti, l'elettroporazione della proteina Cas9 potrebbe rappresentare una valida alternativa quando si lavora con cellule immunitarie differenziate.
Le nanolame possono essere utilizzate in vivo per trasdurre zigoti di topo o embrioni per generare animali transgenici. Simili ai classici vettori retrovirali o lentivirali, possono anche essere iniettati direttamente nei tessuti di animali adulti. Tuttavia, le nanolame (simili ai vettori retrovirali e lentivirali) possono essere inattivate dalla risposta immunitaria dell'animale ospite; pertanto, la dose da iniettare deve essere ottimizzata per ogni applicazione. Questa risposta immunitaria può anche limitare la distribuzione di VLP funzionali ai tessuti vicini al sito di iniezione. Infine, a differenza dei vettori lentivirali, le Nanolame sono prive di transgeni e consegnano il Cas9 in un lasso di tempo limitato. Pertanto, non possono essere utilizzati per eseguire screening funzionali a livello di genoma che richiedono il sequenziamento ad alto rendimento di sgRNA al momento della selezione delle cellule. Le nanolame sono utili quando è richiesta una modifica del genoma rapida, dose-dipendente e senza transgeni20. Inoltre, analogamente all'elettroporazione proteica, le nanolame portano a un minor numero di effetti fuori bersaglio rispetto all'espressione prolungata di Cas9/sgRNA attraverso la trasfezione del DNA o i vettori virali classici3. Lo sviluppo futuro di Nanoblades si concentra sull'incorporazione di varianti Cas9 per diverse applicazioni tecnologiche come l'editing di base e il targeting dell'RNA.
Divulgazioni
Philippe E. Mangeot ed Emiliano P. Ricci sono nominati inventori di un brevetto relativo alla tecnologia Nanoblades (richiedenti il brevetto: Institut National de la Santé et de la Recherche Médicale (INSERM), Centre National de la Recherche Scientifique (CNRS), Ecole Normale Superieure de Lyon, Université Claude Bernard Lyon 1, Villeurbanne Cedex; numero di domanda: WO 2017/068077 Al; status di brevetto: pubblicato, 27 aprile 2017; tutti gli aspetti del manoscritto sono coperti dalla domanda di brevetto. Gli altri autori non dichiarano interessi concorrenti.
Riconoscimenti
Questo lavoro è stato finanziato da Labex Ecofect (ANR-11-LABX-0048) dell'Université de Lyon, nell'ambito del programma Investissements d'Avenir (ANR-11-IDEX-0007) gestito dall'Agenzia nazionale francese delle ricerche (ANR), Fondation FINOVI, Agence Nationale des Recherches sur le SIDA et les Hépatites Virales (ANRS-ECTZ3306) e dal Consiglio europeo della ricerca (ERC-StG-LS6-805500 a E.P.R.) nell'ambito dei programmi di ricerca e innovazione Horizon 2020 dell'Unione europea.
Materiali
| Name | Company | Catalog Number | Comments |
| 13.2 mL, Thinwall Polypropylene Tubes, 14 x 89 mm - 50Pk | Beckman Coulter Life Sciences | 331372 | Ultracentrifugation tubes for Nanoblades purification |
| Amersham Protran Premium Western blotting membranes, nitrocellulose | Merck | GE10600004 | Nitrocellulose membrane for quantifying Cas9 levels within purified Nanoblades |
| BIC-Gag-CAS9 | Addgene | 119942 | Encodes a GAG (F-MLV)-CAS9(sp) fusion. Allows the production of GAG-CAS9 Virus like particles from producer cells in association with over expressed gRNA(s) and appropriate envelopes |
| BICstim-Gag-dCAS9-VPR | Addgene | 120922 | Encodes a GAG-dCAS9-VPR fusion for targeted transcriptional activation |
| BLADE | Addgene | 134912 | Empty backbone for cloning sgRNA sequence to be used in Nanoblades system |
| BsmBI-v2 | New England Biolabs | R0739S | Restriction enzyme to digest the BLADE and SUPERBLADES vectors for sgRNA cloning |
| Cas9 (7A9-3A3) Mouse mAb (HRP Conjugate) #97982 | Cell Signaling Technology | 97982S | Anti-Cas9 antibody for Cas9 quantification by dot-blot |
| Cas9 Nuclease, S. pyogenes | New England Biolabs | M0386T | Recombinant Cas9 protein to be used as a reference for absolute quantification of the amount of Cas9 loaded within Nanoblades |
| Ethidium bromide solution (10 mg/mL in H2O) | Sigma-Aldrich | E1510-10ML | For staining agarose gels and visualize DNA |
| Fisherbrand Wave Motion Shakers | Fisher Scientific | 88-861-028 | Agitation table to resuspend Nanoblades upon centrifugation |
| gelAnalyzer | http://www.gelanalyzer.com; quantifying band intensity after digestion | ||
| Gesicle Producer 293T | Takara | 632617 | Nanoblades producer cell line |
| Gibco DMEM, high glucose, pyruvate | ThermoFisher Scientific | 41966052 | Cell culture medium for Gesicle Producer 293T cells |
| GoTaq G2 DNA Polymerase | Promega | M7848 | Taq polymerase for amplification of genomic DNA before T7 endonuclease assays |
| jetPRIME Transfection Reagent kit for DNA and DNA/siRNA | Polyplus | POL114-15 | Transfection reagent for Nanoblade production in Gesicle Producer 293T cells |
| Millex-AA, 0.80 µm, syringe filter | Millipore | SLAA033SS | Syringe filter to remove cellular debris before concentration of Nanoblades |
| Millex-GS, 0.22 µm, syringe filter | Millipore | SLGS033SS | Syringe filter to sterilise the sucrose cushion solution |
| Millex-HP, 0.45 µm, polyethersulfone, syringe filter | Millipore | SLHP033RS | Syringe filter to remove cellular debris before Nanoblades concentration |
| Monarch DNA Gel Extraction Kit | New England Biolabs | T1020L | DNA gel extraction kit for purification of the pBLADES or pSUPERBLADES plasmid fragment upon digestion with BsmBI |
| NEB Stable Competent E. coli (High Efficiency) | New England Biolabs | C3040I | Competent bacteria for plasmid transformation and amplification |
| NucleoBond Xtra Midi kit for transfection-grade plasmid DNA | Macherey-Nagel | 740410.50 | Maxipreparation kit for purification of plasmid DNA from cultured bacteria |
| Nucleospin gDNA extraction kit | Macherey-Nagel | 740952.50 | Extraction of genomic DNA from transduced cells |
| NucleoSpin Plasmid, Mini kit for plasmid DNA | Macherey-Nagel | 740588.50 | Minipreparation kit for purification of plasmid DNA from cultured bacteria |
| NucleoSpin Tissue, Mini kit for DNA from cells and tissue | Macherey-Nagel | 740952.5 | Genomic DNA extraction kit |
| Optima XE-90 | Beckman Coulter Life Sciences | A94471 | Ultracentrifuge |
| pBaEVRless | Els Verhoeyen (Inserm U1111) | Personnal requests have to be sent to: els.verhoyen@ens-lyon.fr | Baboon Endogenous retrovirus Rless glycoprotein described in Girard-Gagnepain, A. et al. Baboon envelope pseudotyped LVs outperform VSV-G-LVs for gene transfer into early-cytokine-stimulated and resting HSCs. Blood 124, 1221–1231 (2014) |
| pBS-CMV-gagpol | Addgene | 35614 | Enocdes the Murine Leukemia Virus gag and pol genes |
| pCMV-VSV-G | Addgene | 8454 | Envelope protein for producing lentiviral and MuLV retroviral particles |
| Phosphate-Buffered Saline (PBS) | ThermoFisher Scientific | 14200091 | 10X PBS to dilute in millipore water |
| Polybrene Transfection Reagent | Millipore Sigma | TR-1003-G | Cationic polymer that enhances the efficiency of retroviral transduction in specific mammalian cells. It can also allow viral-dependent entry of an Oligodeoxynucleotide (ODN) for homology-directed repair |
| Sucrose,for molecular biology, ≥99.5% (GC) | Sigma-Aldrich | S0389-5KG | Sucrose to prepare a cushion for Nanoblade purification through ultracentrifugation |
| SUPERBLADE5 | Addgene | 134913 | Empty backbone for cloning sgRNA sequence to be used in nanoblades system (Optimized for increased genome editing efficiency via Chen B et al., 2013) |
| SuperSignal West Dura Extended Duration Substrate | ThermoFisher Scientific | 34076 | Enhanced chemiluminescence (ECL) HRP substrate for Cas9 dot blots |
| SW 41 Ti Swinging-Bucket Rotor | Beckman Coulter Life Sciences | 331362 | Rotor for ultracentrifugation |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | Alternative to ethidium bromide for staining agarose gels and visualize DNA |
| T4 DNA Ligase | New England Biolabs | M0202S | DNA ligase to ligate the BLADE or SUPERBLADES vectors with the duplexed DNA oligos corresponding to the variable region of the sgRNA |
| T7 Endonuclease I | New England Biolabs | M0302S | T7 Endonuclease I recognizes and cleaves non-perfectly matched DNA. Allows to monitor the extent of genome editing at a specific locus |
| Triton-containing lysis buffer | Promega | E291A | Lysis buffer to disrupt Nanoblades and allow Cas9 quantification |
| TWEEN 20 | Sigma-Aldrich | P9416 | For the preparation of TBST |
Riferimenti
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zuris, J. A., et al. Cationic lipid-mediated delivery of proteins enables efficient protein-based genome-editing in vitro and in vivo. Nature Biotechnology. 33, 73-80 (2015).
- Mangeot, P. E., et al. Genome editing in primary cells and in vivo using viral-derived Nanoblades loaded with Cas9-sgRNA ribonucleoproteins. Nature Communications. 10 (1), 45 (2019).
- Cai, Y., Bak, R. O., Mikkelsen, J. G. Targeted genome editing by lentiviral protein transduction of zinc-finger and TAL-effector nucleases. eLife. 3, 01911 (2014).
- Choi, J. G., et al. Lentivirus pre-packed with Cas9 protein for safer gene editing. Gene Therapy. 23 (7), 627-633 (2016).
- Gee, P., et al. Extracellular nanovesicles for packaging of CRISPR-Cas9 protein and sgRNA to induce therapeutic exon skipping. Nature Communications. 11, 1334 (2020).
- Indikova, I., Indik, S. Highly efficient 'hit-and-run' genome editing with unconcentrated lentivectors carrying Vpr.Prot.Cas9 protein produced from RRE-containing transcripts. Nucleic Acids Research. 48 (14), 8178-8187 (2020).
- Lyu, P., Javidi-Parsijani, P., Atala, A., Lu, B. Delivering Cas9/sgRNA ribonucleoprotein (RNP) by lentiviral capsid-based bionanoparticles for efficient 'hit-and-run' genome editing. Nucleic Acids Research. 47 (17), 99 (2019).
- Gheysen, D., Jacobs, E., de Foresta, F., Thiriart, C. Assembly and release of HIV-1 precursor Pr55gag virus-like particles from recombinant baculovirus-infected insect cells. Cell. 59 (1), 103-112 (1989).
- Hanna, R. E., Doench, J. G. Design and analysis of CRISPR-Cas experiments. Nature Biotechnology. 38 (7), 813-823 (2020).
- Sambrook, J. . Molecular cloning a laboratory manual. Third edition. , (2001).
- Jiang, W., et al. An optimized method for high-titer lentivirus preparations without ultracentrifugation. Scientific Reports. 5, 13875 (2015).
- Rueden, C. T., et al. ImageJ2: ImageJ for the next generation of scientific image data. BMC Bioinformatics. 18 (1), 529 (2017).
- O'Doherty, U., Swiggard, W. J., Malim, M. H. Human immunodeficiency virus type 1 spinoculation enhances infection through virus binding. Journal of Virology. 74 (21), 10074-10080 (2000).
- Brinkman, E. K., Chen, T., Amendola, M., van Steensel, B. Easy quantitative assessment of genome editing by sequence trace decomposition. Nucleic Acids Research. 42 (22), 168 (2014).
- Bollen, Y., Post, J., Koo, B. -. K., Snippert, H. J. G. How to create state-of-the-art genetic model systems: strategies for optimal CRISPR-mediated genome editing. Nucleic Acids Research. 46 (13), 6435-6454 (2018).
- Brinkman, E. K., et al. Easy quantification of template-directed CRISPR/Cas9 editing. Nucleic Acids Research. 46 (10), 58 (2018).
- Dussaud, S., Pardanaud-Glavieux, C., Sauty-Colace, C., Ravassard, P. Lentiviral mediated production of transgenic mice: a simple and highly efficient method for direct study of founders. Journal of Visualized Experiments. (140), e57609 (2018).
- Montagna, C., et al. VSV-G-enveloped vesicles for traceless delivery of CRISPR-Cas9. Molecular Therapy. Nucleic Acids. 12, 453-462 (2018).
- Marnef, A., et al. A cohesin/HUSH- and LINC-dependent pathway controls ribosomal DNA double-strand break repair. Genes & Development. 33 (17-18), 1175-1190 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon