Method Article
Entrega do Complexo cas9/sgRNA Ribonucleoproteína em Células Imortalizadas e Primárias através de Partículas semelhantes a vírus ("Nanoblades")
Neste Artigo
Resumo
Desenvolvemos um protocolo simples e barato para carregar complexos de ribonucleoproteína Cas9/guia único (sgRNA) dentro de partículas semelhantes a vírus. Essas partículas, chamadas "Nanoblades", permitem a entrega eficiente do complexo Cas9/sgRNA em células imortalizadas e primárias, bem como in vivo.
Resumo
O sistema de repetições palindômicas curtas interespaçadas (CRISPR)-Cas democratizou a edição de genomas em células eucarióticas e levou ao desenvolvimento de inúmeras aplicações inovadoras. No entanto, a entrega da proteína Cas9 e do RNA de guia único (sgRNA) em células-alvo pode ser tecnicamente um desafio. Vetores virais clássicos, como os derivados de lentivírus (LVs) ou vírus associados ao Adeno (AAVs), permitem a entrega eficiente da codificação de transgenes para a proteína Cas9 e seu sgRNA associado em muitas células primárias e in vivo. No entanto, esses vetores podem sofrer de desvantagens como a integração do transgene no genoma da célula alvo, uma capacidade de carga limitada e expressão a longo prazo da proteína Cas9 e guiar o RNA em células-alvo.
Para superar alguns desses problemas, um vetor de entrega baseado no vírus da leucemia murina (MLV) foi desenvolvido para embalar a proteína Cas9 e seu guia associado RNA na ausência de qualquer transgene de codificação. Ao fundir a proteína Cas9 ao c-terminus da proteína estrutural Gag de MLV, foram formadas partículas semelhantes a vírus (VLPs) carregadas com a proteína Cas9 e sgRNA (denominadas "Nanoblades"). Nanoblades podem ser coletados do meio de cultura de células produtoras, purificadas, quantificadas e usadas para transduzir células-alvo e fornecer o complexo cas9/sgRNA ativo. Nanoblades fornecem sua carga ribonucleoproteína (RNP) de forma transitória e rápida em uma ampla gama de células primárias e imortalizadas e podem ser programadas para outras aplicações, como a ativação transcricional transitória de genes-alvo, usando proteínas Cas9 modificadas. Nanoblades são capazes de editar genoma in vivo no fígado de camundongos adultos injetados e em oócitos para gerar animais transgênicos. Finalmente, eles podem ser complexos com DNA de doador para reparo "livre de transfecção" direcionado à escritologia. A preparação de nanoblade é simples, relativamente de baixo custo, e pode ser facilmente realizada em qualquer laboratório de biologia celular.
Introdução
Comparado com outros núcleos programáveis, o sistema CRISPR-Cas simplificou drasticamente e democratizou o procedimento de segmentação de genoma e decote específicos de sequência em células eucarióticas. Através da simples expressão de um sgRNA, os usuários podem programar a proteína Cas9 (ou variantes otimizadas) para quase qualquer lócusular1. Nesse cenário, a entrega da proteína Cas9 e do sgRNA torna-se a principal limitação na realização da mutagênese dirigida ao local. Em células imortalizadas, o sgRNA e a proteína Cas podem ser facilmente expressos a partir de plasmídeos transfectados para alcançar um alvo eficiente do genoma na maioria das células. No entanto, a expressão constitutiva do complexo Cas9/sgRNA pode aumentar a atividade fora do alvo da proteína Cas9 e introduzir alterações indesejadas em loci2 não específico. Nas células primárias, a transfecção de DNA pode ser tecnicamente difícil de alcançar e levar à má expressão ou a uma pequena porcentagem de células transfeccionadas. Alternativas à transfecção clássica do DNA compreendem o uso de vetores virais que fornecem uma codificação transgênica para o Cas9 e sgRNA ou a eletroporação de proteína Cas9 recombinante acoplado a um sgRNA sintético. No entanto, essas abordagens podem levar à integração transgênica dentro do genoma do hospedeiro celular (como é o caso dos vetores clássicos de expressão retroviral e lentiviral), restrição por fatores celulares e levar à expressão constitutiva da proteína Cas9 e do sgRNA.
A eletroporação do complexo Cas9/sgRNA RNP pode superar a maioria desses problemas e levar a uma entrega eficiente e transitória em células primárias e in vivo, bem como permitir uma resposta dependente de dose. No entanto, geralmente depende de equipamentos e reagentes caros e também é difícil de dimensionar se um grande número de células tem que ser tratada. Como alternativa às técnicas acima mencionadas, esses autores desenvolveram "Nanoblades" - um vetor de entrega retroviral para a proteína Cas9 e sgRNA3 que é conceitualmente semelhante a outros sistemas de entrega de proteína capsid derivados pelo viral4,5,6,7,8. Os nanoblados exploram a capacidade natural da poliproteína gaga de retrovírus para produzir, quando expressas sozinhas em células cultivadas, VLPs que são liberadas no meio extracelular9. Ao fundir a proteína Cas9 à extremidade terminal C do vírus da leucemia murina (MLV) Gag polyprotein e co-expressando as glicoproteínas de sgRNA e envelope viral, a proteína Cas9 pode ser encapsidada dentro de VLPs ou Nanoblades liberados. Após a purificação, os Nanoblades podem ser incubados com células-alvo ou injetados in vivo para mediar a entrega rápida, transitória e dependente de dose do complexo Cas9/sgRNA RNP3.
Nanoblades podem ser programados com vários sgRNAs para edição simultânea em diferentes loci ou com variantes Cas9 para executar outros aplicativos, como ativação transcricional específica do alvo ou repressão3. Em contraste com a eletroporação proteica, que se baseia na expressão recombinante, as variantes cas recém-descritas da literatura podem ser facilmente clonadas no vetor de expressão de fusão gag, tornando-se uma plataforma versátil. Nanoblades podem ser ainda mais complexos ou carregados com oligodeoxotídeos de fio único e duplamente encalhados (ssODNs) para realizar reparos direcionados à homologia3. A produção de nanoblade é relativamente simples e barata. Além disso, nanoblades podem ser armazenados a 4 °C por muitos dias ou a -80 °C para armazenamento a longo prazo. Normalmente, os Nanoblades mediam a edição eficiente e livre de transgenes em células mais imortalizadas e cultivadas primárias. No entanto, algumas células primárias podem ser sensíveis à presença de partículas virais, resultando em aumento da mortalidade. As células do sistema imunológico inato também podem reagir à presença de Nanoblades (por causa de sua origem viral) e se ativar. Nesses casos, o protocolo de transdução deve ser otimizado para limitar o tempo de exposição aos Nanoblades e minimizar efeitos não específicos. Os nanoblades representam uma alternativa viável e fácil de implementar para outros métodos de entrega CRISPR disponíveis.
Protocolo
1. sgRNA Design e clonagem
NOTA: As diretrizes para o design de sgRNAs podem ser obtidas de múltiplas fontes, como https://blog.addgene.org/how-to-design-your-grna-for-crispr-genome-editing ou de Hanna e Doench10.
- Uma vez que as 20 sequências de sgRNA nucleotídeos tenham sido projetadas, ordene os seguintes oligonucleotídeos de DNA de fio único:
- Avante: 5' caccgNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN 3' (N corresponde ao lócus alvo sem a sequência protoespacial-adjacente (PAM)
- Reverso: 5' aaacNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNNN
NOTA: Não são necessárias modificações especiais ao encomendar os oligonucleotídeos (sem exigência de fosfato de 5').
- Hibridize os dois oligonucleotídeos de DNA em um tubo de reação em cadeia de polimerase de 0,2 mL (PCR) misturando 5 μL de tampão de renas (500 mM NaCl; 100 mM Tris-HCl; 100 mM MgCl2; 10 mM DTT; pH 7,9 a 25 °C), 1 μL de cada oligonucleotídeo de DNA (solução de estoque de 100 μM em água) e 42 μL de água.
- Em um bloco PCR, incubar amostras a 95 °C para 15 s e, em seguida, diminuir a temperatura para 20 °C com uma rampa de 0,5 °C/s. Mantenha-se em temperatura ambiente ou armazene a -20 °C.
NOTA: O protocolo pode ser pausado aqui. - Digere 10 μg da expressão BLADE ou SUPERBLADE sgRNA com 10 unidades de enzima de restrição BsmBI-v2 por 3 h a 55 °C em um volume total de reação de 50 μL.
NOTA: O vetor digerido deve liberar uma inserção de DNA de ~1,9 kb e um segundo fragmento de DNA de ~3,3 kb. - Carregue a reação de restrição em um gel de 1% de agarose manchado com 5 μg/mL de brometo de ethidium (ou uma mancha alternativa de gel de DNA mais segura).
NOTA: Use equipamento de proteção adequado ao manipular brometo de ethidium, que é suspeito de causar defeitos genéticos.- Em uma mesa ultravioleta (UV) fixada em um comprimento de onda de 312 nm (para evitar danificar o DNA), corte o fragmento de DNA de 3,3 kb do gel e coloque-o em um tubo de microcentrifus de 1,5 mL.
NOTA: Use equipamentos de proteção adequados (luvas e óculos de proteção UV) ao manipular brometo de ethidium e trabalhar na mesa UV. - Extrair DNA do gel fatiado contendo o fragmento de DNA de 3,3 kb usando um kit dedicado de extração de gel de DNA (ver a Tabela de Materiais). Quantifique a quantidade de DNA purificado usando um espectotômetro.
NOTA: O protocolo pode ser pausado aqui.
- Em uma mesa ultravioleta (UV) fixada em um comprimento de onda de 312 nm (para evitar danificar o DNA), corte o fragmento de DNA de 3,3 kb do gel e coloque-o em um tubo de microcentrifus de 1,5 mL.
- Ligate os oligonucleotídeos de DNA hibridizados e reversos da etapa 1.2 para o blades digerido por BsmB1, purificado em gel ou vetor SUPERBLADE do passo 1.5.2. Para isso, adicione 2 μL de tampão de ligase de DNA T4, 50 ng do vetor purificado em gel (a partir da etapa 1,5.2), 1 μL dos oligonucleotídeos de DNA hibridizados (a partir do passo 1.2), água para compor o volume a 19 μL, e 1 μL de ligase de DNA T4. Incubar a reação a 25 °C por 10 min.
- Transforme o produto de ligadura em bactérias competentes (ver a Tabela de Materiais) conforme descrito em 11. Emcore as bactérias transformadas em uma placa de ágar luria Bertani ampicillin e incuba durante a noite a 37 °C.
- Selecione várias colônias isoladas na placa de ágar para realizar minipreparação de DNA11 (ver a Tabela de Materiais) e realize o sequenciamento Sanger usando um primer para frente U6 (5' GACTATCATATCCCCGT 3') para verificar se há uma ligadura correta da sequência variável sgRNA.
NOTA: Outros plasmídeos de expressão sgRNA podem ser usados se não codificarem a proteína Cas9, o que pode interferir na produção de nanoblade.
2. Preparação plasmid
- Realize a maxipreparação (veja a Tabela de Materiais) de todos os plasmídeos necessários e prepare 10 alíquotas de μg a 1 μg/mL para armazenar a -20 °C. Evite ciclos repetidos de congelamento/descongelamento dos plasmídeos; use alíquotas duas vezes antes de descartá-las.
3. Preparação de nanoblade
- No primeiro dia, semente entre 3,5 e 4 × 106 células HEK293T (ver a Tabela de Materiais) em 10 mL do meio águia modificado de Dulbecco (DMEM) contendo alta glicose, piruvato de sódio, L-glutamina, 10% de soro bovino (FBS) e penicilina/estreptomicina em uma cultura de 10 cm de célula. Mova a placa de 10 cm suavemente para trás e para frente, depois da direita para a esquerda (repita esta sequência 5x) para distribuir as células de forma homogênea sobre o prato de cultura. Incubar células a 37 °C em uma incubadora celular com 5% de CO2.
NOTA: Todos os procedimentos relacionados ao manuseio de células cultivadas e nanoblades devem ser realizados sob uma cultura celular laminar flow hood para evitar sua contaminação. - Dia 2: Transfecção plasmida
- As células devem ser 70-80% confluentes 24 h após o revestimento (Figura 1A). Substitua o meio por 10 mL de DMEM fresco contendo alta glicose, piruvato de sódio, L-glutamina, 10% FBS (penicilina e estreptomicina podem ser omitidas, embora não seja obrigatória) antes da transfecção.
NOTA: Nesta etapa, é importante que as células não sejam confluentes. Caso contrário, a eficiência da transfecção, bem como a produção de partículas poderiam ser reduzidas. - Para cada placa de 10 cm, prepare as seguintes quantidades de plasmídeos em um tubo de 1,5 mL: 0,3 μg pCMV-VSV-G, 0,7 μg pBaEVRless, 2,7 μg MLV Gag/Pol, 1,7 μg BIC-Gag-Cas9, 4,4 μg de BLADES ou SUPERBLADES plasmid codificando o sgRNA clonado (ou 2,2 μg cada se usar dois sgRNAs).
- Adicione 500 μL de tampão de transfecção (veja a Tabela de Materiais), vórtice para 10 s e, em seguida, centrífuga para 1 s. Adicione 20 μL do reagente de transfecção (veja a Tabela de Materiais), vórtice do tubo para 1 s e, em seguida, centrífuga para 1 s.
- Incubar por 10 minutos à temperatura ambiente e adicionar toda a solução dropwise às células em meio DMEM usando um pipettor P1000. Mova a placa de 10 cm suavemente para trás e para frente, depois da direita para a esquerda (repita esta sequência 5x) para distribuir uniformemente o reagente de transfecção sobre as células. Incubar células a 37 °C por pelo menos 40 h em uma incubadora celular com 5% de CO2.
NOTA: Se desejar, o meio pode ser alterado 4h após a transfecção.
- As células devem ser 70-80% confluentes 24 h após o revestimento (Figura 1A). Substitua o meio por 10 mL de DMEM fresco contendo alta glicose, piruvato de sódio, L-glutamina, 10% FBS (penicilina e estreptomicina podem ser omitidas, embora não seja obrigatória) antes da transfecção.
- No dia 3, verifique a morfologia das células transfeinadas sob o microscópio.
NOTA: As células produtoras começarão a se fundir. Esta é uma ocorrência normal devido à expressão de envelopes virais fusogênicos (Figura 1B,C). - Dia 4: Colheita de Nanoblades
NOTA: Pelo menos 40 h após a transfecção, as células teriam se fundido por causa da expressão dos envelopes virais fusogênicos, e às vezes, as células são completamente separadas do suporte da placa (Figura 1D).- Colete 9 mL do supernaente médio de cultura usando uma pipeta de 10 mL.
NOTA: Nanoblades são VLPs capazes de fornecer a proteína Cas9 e seu sgRNA associado em células primárias e in vivo. Embora não sejam considerados organismos geneticamente modificados por serem desprovidos de material genético, eles podem induzir alterações genéticas. Portanto, eles devem ser manipulados com cautela para evitar qualquer contato com os usuários (especialmente se eles são programados para atingir genes supressores de tumores). Os usuários são aconselhados a seguir suas diretrizes locais de segurança para a manipulação de vetores retrovirais e trabalhar em um laboratório de nível BSL-2 ao preparar VLPs e realizar experimentos de transdução. Nanoblades podem ser inativados com 70% de etanol ou 0,5% de hipoclorito de sódio. Também é aconselhável tratar todos os resíduos plásticos (pontas de pipeta, placas de cultura tecidual, tubos de centrifugação) com hipoclorito de sódio de 0,5% por pelo menos 10 minutos para inativar os Nanoblades. - Centrifugar o supernante coletado a 500 × g por 5 minutos para remover detritos celulares e recuperar o supernatante sem perturbar a pelota celular.
NOTA: Se os nanoblades forem usados em células primárias, filtre o supernante usando um filtro de 0,45 μm ou 0,8 μm. Esteja ciente de que este passo reduz drasticamente o título de Nanoblade, pois uma fração significativa será bloqueada na membrana do filtro. - Pelota os Nanoblades durante a noite (12-16 h) em um rotor de balde balançando a 4.300 × g ou a 209.490 × g em um ultracentrifuuge por 75 min a 4 °C (ver a Tabela de Materiais).
NOTA: Se as células-alvo podem crescer no DMEM, é possível incuba-las diretamente com o sobrenante obtido após a etapa 3.4.2 sem concentrar os Nanoblades.
- Colete 9 mL do supernaente médio de cultura usando uma pipeta de 10 mL.
- Dia 5: Resuspensão e armazenamento de Nanoblades
- Após a centrifugação, aspire lentamente o meio e resuspenque a pelota branca com 100 μL de soro fisiológico frio 1x tamponado (PBS). Cubra o tubo com parafilm e incubar por 1h a 4 °C com agitação suave antes de reutilizar a pelota, encanando para cima e para baixo.
NOTA: Um material viscoso branco pode aparecer após a ressuspensão; isso é normal e não afeta significativamente a eficiência da transdução. - Armazene os Nanoblades a 4 °C se planejar usá-los dentro de quatro semanas. Caso contrário, congele os Nanoblades em nitrogênio líquido e armazene-os a -80 °C.
NOTA: Use óculos de proteção e luvas criogênicas ao manipular nitrogênio líquido. O congelamento e o armazenamento instantâneo a -80 °C leva a uma diminuição significativa na eficiência de Nanoblade. Além disso, nanoblades descongelados não devem ser congelados novamente. O protocolo pode ser pausado aqui.
- Após a centrifugação, aspire lentamente o meio e resuspenque a pelota branca com 100 μL de soro fisiológico frio 1x tamponado (PBS). Cubra o tubo com parafilm e incubar por 1h a 4 °C com agitação suave antes de reutilizar a pelota, encanando para cima e para baixo.
4. Concentração de Nanoblades em uma almofada de sacarose
NOTA: Como alternativa à centrifugação ou ultracentrifugação noturna (passo 3.4.3), os Nanoblades podem ser concentrados em uma almofada de sacarose. Isso produz uma fração mais pura de Nanoblades, embora o valor total recuperado seja menor.
- Prepare uma solução de sacarose de 10% (peso a volume) em 1x PBS e filtre-a através de um filtro de seringa de 0,2 μm (veja a Tabela de Materiais).
- Comece o processo de concentrar os Nanoblades na almofada de sacarose.
- Coloque 9 mL de amostra contendo VLP (a partir da etapa 3.4.3) em um tubo de ultracentrifuuge (veja a Tabela de Materiais). Utilizando uma seringa de 3 mL e cânula, lentamente camada 2,5 mL da sacarose de 10% sob a amostra, tentando não misturar a amostra contendo VLP e a solução de sacarose.
- Alternativamente, coloque 2,5 mL de 10% de sacarose em um tubo ultracentrífuga (veja a Tabela de Materiais). Incline o tubo e adicione lentamente os 9 mL de amostra contendo VLP (da etapa 3.4.3) com um pipettor de baixa velocidade. Durante esta operação, eleve progressivamente o tubo para uma posição vertical.
- Centrifugar as amostras a 209.490 × g em uma ultracentrifugação por 90 min a 4 °C.
NOTA: Esta técnica pode ser adaptada para centrifugação de baixa velocidade (4.300 × g) durante a noite, conforme descrito em 12. - Após a centrifugação, remova o supernatante cuidadosamente e coloque o tubo de cabeça para baixo no papel de tecido para remover qualquer líquido restante. Depois de 1 min, adicione 100 μL de 1x PBS e coloque o tubo a 4 °C com uma tampa de parafilm em um suporte de tubo em uma mesa de agitação por 1h (veja a Tabela de Materiais) antes de reutilizar a pelota, pipetando para cima e para baixo.
NOTA: O protocolo pode ser pausado aqui.
5. Monitoramento do carregamento cas9 dentro de Nanoblades por ponto-blot
- Prepare o tampão de diluição adicionando 1 volume de tampão de lise contendo um surfactante não iônico (veja a Tabela de Materiais) em 4 volumes de 1x PBS. Diluir 2 μL de nanoblades concentrados em 50 μL de tampão de diluição, vórtice brevemente, e transferir 25 μL desta mistura para um novo tubo contendo 25 μL de tampão de diluição. Repita esta operação para ter 4 tubos de diluições de nanoblade (passos de diluição de 2 vezes).
- Para os controles padrão, diluir 2 μL de nuclease Cas9 recombinante (ver a Tabela de Materiais) em 50 μL de tampão de diluição, vórtice brevemente, e proceder a fazer oito diluições seriais (diluição de 2 vezes para cada etapa).
- Localmente cuidadosamente 2,5 μL de cada diluição VLP e 2,5 μL de cada padrão em uma membrana nitrocelulose com um tubo multicanal (um volume maior pode resultar em pontos sobrepostos).
NOTA: Uma membrana polivinilafluoreto tratada com metanol também pode ser usada. - Uma vez absorvidas as partículas na membrana, bloqueie a membrana com soro fisiológico 1x tris-tamponado contendo um surfactante não iônico (TBS-T) suplementado com leite seco não gordo (5% w/v) por 45 min à temperatura ambiente.
NOTA: O protocolo pode ser pausado aqui, e a membrana armazenada a 4 °C em 1x TBS-T. - Descarte o 1x TBST suplementado com leite seco não gordo e incubar a membrana durante a noite a 4 °C com o anticorpo peroxidase de rabanete Cas9 (1/1000 diluição em 1x TBST, 5% de leite). Lave a membrana 3x com TBS-T e visualize o sinal usando um kit de substrato quimiamminescente aprimorado.
- Quantifique a intensidade do ponto para os Nanoblades e diluições padrão Cas9 recombinantes usando o software proprietário fornecido com a estação de imagem em gel ou imagemJ13. Defina uma curva linear ligando a intensidade do ponto à concentração de Cas9. Utilizando a função da curva obtida, extrava o conteúdo Cas9 em cada preparação.
NOTA: A quantidade de controle recombinante da proteína Cas9 pode saturar a leitura para as amostras mais concentradas do conjunto de diluição padrão (Figura 2). Aconselha-se, ao definir a curva linear, a remover a leitura das amostras não diluídas (e às vezes a dos primeiros passos de diluição) se não estiverem na faixa linear em relação à concentração conhecida de Cas9 que foi avistada. Da mesma forma, ao extrapolar a quantidade de Cas9 dentro das amostras de Nanoblade, utilize apenas as leituras que estão dentro da faixa linear da curva padrão.
6. Transdução de células-alvo com Nanoblades (procedimento para transdução em uma placa de 12 poços)
- Em uma placa de 12 poços, sementes 100.000-200.000 células (células primárias ou imortalizadas) por poço em 1 mL do meio de cultura celular apropriada. Permita que as células aderam à superfície da placa antes da transdução.
- Em um tubo de microcentrifuuge de 1,5 mL, adicione 5-20 μL de Nanoblades concentrados (da etapa 3,5,1 ou 4,4) a 500 μL de meio de cultura celular, e misture por pipetar para cima e para baixo com um pipettor P1000. Remova o meio das células e substitua-o pelo 500 μL desta mistura de Nanoblade.
NOTA: A transdução deve ser otimizada para cada tipo de célula. É importante usar o menor volume possível de meio (evitando a secagem das células-alvo) para que os Nanoblades permaneçam altamente concentrados. As células aderentes devem ser transduzidas diretamente enquanto anexadas à placa (não transdugue em suspensão, pois isso diminuirá significativamente a eficiência da transdução). Algumas células toleram exposição prolongada aos Nanoblades (24-48 h), enquanto outras são muito sensíveis e podem formar pequenas sincronias. Neste caso, os nanoblades devem ser incubados com células apenas por 4-6 h antes de substituir o meio. A spinoculação14 também pode melhorar a transdução para células cultivadas em suspensão. Adjuvantes como polímeros cônicos (ver a Tabela de Materiais) também podem melhorar a eficiência da transdução em alguns tipos de células. - Após 4-6 h de incubação celular em um baixo volume de nanoblades médios contendo, aumente o volume de média para a quantidade normal (1 mL se trabalhar com uma placa de 12 poços), ou substitua-o por meio fresco se as células forem sensíveis aos VLPs.
NOTA: O meio celular contendo nanobladas deve ser inativado com hipoclorito de sódio de 0,5% por 10 minutos antes de descartá-lo. Use luvas e óculos de proteção ao manipular hipoclorito de sódio. Se nanobladas induzirem a morte celular, adapte a quantidade e o tempo total de exposição para reduzir a mortalidade celular.
7. Medir a eficiência do CRISPR no lócus-alvo pelo ensaio endonuclease T7
- Projete primers PCR para amplificar uma região de 400-700 de par base (bp) que engloba o local de decote CRISPR.
NOTA: O local do decote deve estar distante da borda amplicon por pelo menos 200 bp e deve ser ligeiramente deslocado do centro da amplicon para que após o decote endonuclease T7, 2 fragmentos de diferentes tamanhos sejam liberados. - Extrair DNA genômico de células tratadas com Nanoblades visando o gene de interesse e de células de controle tratadas com Nanoblades programadas com um sgRNA de controle (ver a Tabela de Materiais).
NOTA: O protocolo pode ser pausado aqui. - Usando 150 ng de DNA genômico como modelo, programe uma reação pcr de volume de 30 μL (volume final) seguindo o protocolo do fabricante. Verifique se a amplificação pcr produz um único amplicon do tamanho esperado executando um gel de 2% de agarose manchado com 5 μg/mL de brometo de ethidium (ou uma mancha alternativa de gel de DNA mais segura).
NOTA: Use equipamento de proteção adequado ao manipular brometo de ethidium, que é suspeito de causar defeitos genéticos. O protocolo pode ser pausado aqui. - Geração e digestão heteroduplex
- Em um tubo PCR de 0,2 mL, adicione 5 μL do tampão enzimátil (fornecido com a endonuclease T7 I), 20 μL de água e 24 μL do produto PCR a partir da etapa 7.3. Deixe a formação heteroduplex aquecendo as amostras para 94 °C ao longo de 3 minutos e, em seguida, diminuindo a temperatura (2 °C por minuto) para chegar a 40 °C.
- Adicione 0,5 μL de T7-endonuclease I à temperatura ambiente a cada tubo heteroduplex, incluindo o controle. Incubar a 37 °C por 15 min. Carregue a reação resultante em um gel de agarose de 2,5% (peso/volume) manchado por brometo de ethidium. Após a migração, imagem do gel em um transilluminator UV.
NOTA: Use equipamento de proteção adequado ao manipular brometo de ethidium, que é suspeito de causar defeitos genéticos. Use óculos de proteção UV ao usar o transilluminator UV. - Meça a eficiência do decote analisando a imagem resultante da reação de digestão para quantificar a intensidade de cada banda com software apropriado (ver a Tabela de Materiais).
8. Medir a eficiência do CRISPR no lócus-alvo por sanger sequenciamento e análise de MARÉ
NOTA: Como alternativa ao ensaio endonuclease T7, a eficiência do CRISPR pode ser monitorada pela análise e desconvolução de traços de sequenciamento de Sanger com base no protocolo TIDE15.
- Realize o sequenciamento Sanger de amplicons PCR a partir da etapa 7.3 (inclua uma condição de controle correspondente a células não tratadas) usando o primer PCR para frente ou reverso.
- Analise os traços de sequenciamento de Sanger da condição de controle (células não tratadas) e amostras tratadas com nanoblade usando o servidor TIDE (https://tide.nki.nl) e seguindo suas diretrizes de análise.
9. Formação de complexo nanoblade com doadores de SsODN para reparo direcionado à e homologia (procedimento para transdução em uma placa de 12 poços)
NOTA: As diretrizes para o projeto do SSODN para uma edição mediada de reparo direcionada à escoologia eficiente foram descritas anteriormente16.
- Em uma placa de 12 poços, sementes 100.000-200.000 células por poço em 1 mL do meio de cultura celular apropriada. Permita que as células aderam à superfície da placa antes da transdução.
- Prepare 100 μL de uma solução do polímero cationic (ver a Tabela de Materiais) a 8 μg/mL em 1x PBS.
- Misture 19 μL da solução de polímero cationic com 100 pmol do modelo ssODN. Adicione 20 μL de Nanoblades concentrados (a partir da etapa 3.5.1 ou 4.4), e incubar por 15 minutos no gelo.
- Remova os nanoblades/ssODN complexos do gelo e adicione 500 μL de cultura celular (a 37 °C). Remova o meio das células alvo (da etapa 9.1) e adicione os 500 μL de meio contendo os Nanoblades/ssODN complexos. Permita que as células se proliferem por 48 h antes da genotipagem.
- Extrair DNA genômico de uma fração da população celular usando um kit de extração dedicado (ver a Tabela de Materiais).
- Projete primers PCR para amplificar uma região de 400-700 bp que engloba o local de knock-in.
NOTA: Os primers do PCR não devem se sobrepor aos braços de escoologia do ssODN para evitar resultados falso-positivos resultantes da amplificação do PCR de qualquer ssODN residual ainda presente dentro das células-alvo. - Usando 150 ng de DNAs genômicas de células de controle (não tratadas) ou células tratadas com nanoblade como modelo, programe uma reação pcr de 30 μL seguindo o protocolo do fabricante.
NOTA: os traços de SSODN podem estar presentes no meio celular vários dias após a transdução com o complexo. Este ssODN pode servir como um modelo parcial para ensaios PCR que tentam tela para a integração correta. Assim, é aconselhável passar as células pelo menos duas vezes após a transdução, para evitar eventuais ensaios falso-positivos. - Carregar 5 μL do controle e reações pcr tratadas com nanoblade em um gel de 1% (peso/volume) manchado por brometo de ethidium. Após a migração, imagem do gel em um UV-transilluminator.
NOTA: Se a recombinação homologia for bem sucedida e corresponder à inserção de mais de 1 bp de material genético, deve haver uma diferença no peso molecular dos amplicons pcr entre o controle e a amostra tratada por Nanoblade. Como a eficiência do HDR não chega a 100%, duas bandas devem ser visíveis na amostra tratada por Nanoblade (uma de tamanho semelhante ao amplicon PCR de controle correspondente ao alelo não editada e uma de maior peso molecular correspondente ao alelo knock-in, ver painel médio da Figura 3B).
- Projete primers PCR para amplificar uma região de 400-700 bp que engloba o local de knock-in.
- Realize o sequenciamento Sanger do controle e amplicons PCR tratados com nanoblade.
- Quantifique a eficiência do knock-in usando o protocolo TIDER17.
10. Entrega de nanoblade in vivo
- Entregue até 25 μL de Nanoblades concentrados a partir da etapa 3.5.1 através de injeção retro-orbital ou até 100 μL através da injeção da veia da cauda, como descrito em 18, se trabalhar com camundongos.
NOTA: Todos os procedimentos envolvendo experimentação animal (incluindo injeções de Nanoblade para fins de edição de genomas) exigem um protocolo aprovado de um comitê de ética local. - Para a geração de camundongos transgênicos, use um microinjetivo para entregar de 1 pL a 10 pL de Nanoblades concentrados a partir da etapa 4.4 para o espaço perivitelline de oócitos de rato, como descrito anteriormente18.
NOTA: Para injeção de perivitelina, é essencial purificar e concentrar nanoblades em uma almofada de sacarose para evitar entupimento do microinjetor.
Resultados
O protocolo para a preparação de Nanoblade é bastante simples e requer equipamentos de laboratório simples além do acesso a uma capa de cultura de tecido, uma incubadora de CO2 e uma centrífuga de balde balançando ou uma ultracentrifugação. No entanto, algumas etapas requerem atenção especial, como a origem e o manuseio de células produtoras, bem como condições de transdução. Como mostrado na Figura 1A, é importante as células de sementes para que elas sejam distribuídas homogêneamente na placa e alcancem ~70-80% de confluência no dia da transfecção (evite ter aglomerados de células). Vinte e quatro horas após a transfecção (Figura 1B,C), as células produtoras formarão sincronia levando a células de maior porte com múltiplos núcleos. Quarenta horas após a transfecção (Figura 1D), a maioria das células da placa terá formado sincronia e começará a se desprender da placa.
Isso é perfeitamente normal e é causado pela expressão do envelope glicoproteína, que induz a fusão entre células vizinhas. Após a concentração por centrifugação (ou mesmo diretamente do supernacido das células produtoras), a quantidade de Cas9 carregada dentro de Nanoblades pode ser quantificada de forma absoluta por mancha de ponto em uma membrana nitrocelulose usando Cas9 recombinante como referência (Figura 2). Esta etapa é importante para determinar a quantidade correta de Nanoblades para usar para transdução de células-alvo. Ao realizar o ensaio de ponto-mancha, é importante considerar apenas as leituras que se enquadram na faixa linear da curva padrão. No entanto, independentemente da quantidade de Cas9 presente dentro de Nanoblades, é essencial testar a eficiência da edição de genoma diretamente em células-alvo usando o ensaio endonuclease T7 (Figura 3) ou sequenciamento de Sanger.
Como mostrado na Figura 3, a eficiência dos Nanoblades pode diferir de lote para lote, embora esteja geralmente correlacionada com a quantidade de Cas9. No exemplo mostrado na Figura 3, o lote da faixa 1 leva a 20% de eficiência de edição geral, enquanto o lote da faixa 3 leva a 60% de eficiência. Neste caso, é possível aumentar o volume de Nanoblades usados a partir do lote 1 para obter uma eficiência de edição semelhante à do lote 3. A Figura 4 mostra a máxima eficiência de edição obtida usando Nanoblades em diferentes tipos de células primárias. É importante notar que a eficiência pode variar dependendo da sequência do sgRNA utilizado e da acessibilidade de destino.
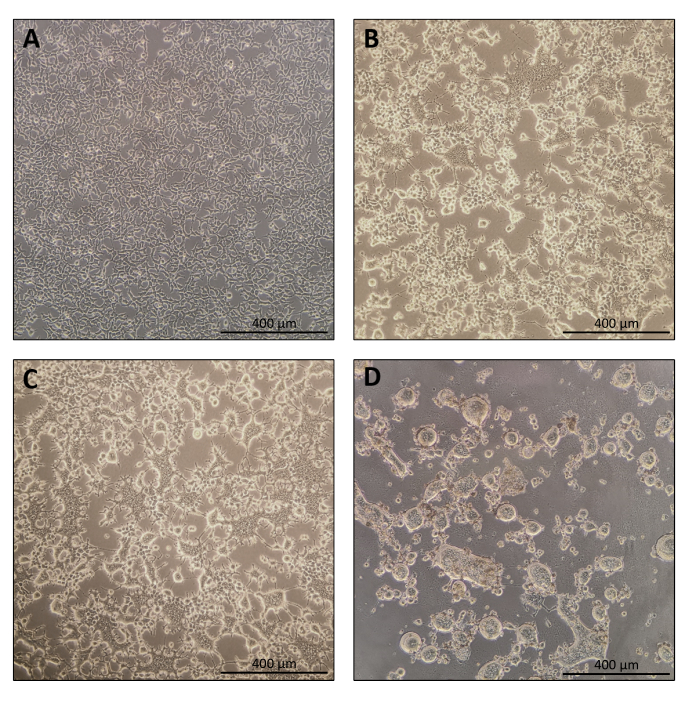
Figura 1: Morfologia das células produtoras durante a produção de Nanoblade. (A) Células HEK293T a 70-80% de confluência 24 h após o revestimento. (B e C) Morfologia celular HEK293T 24 h após a transfecção. (D) Morfologia celular HEK293T 40 h após a transfecção. Barras de escala = 400 μm. Clique aqui para ver uma versão maior desta figura.
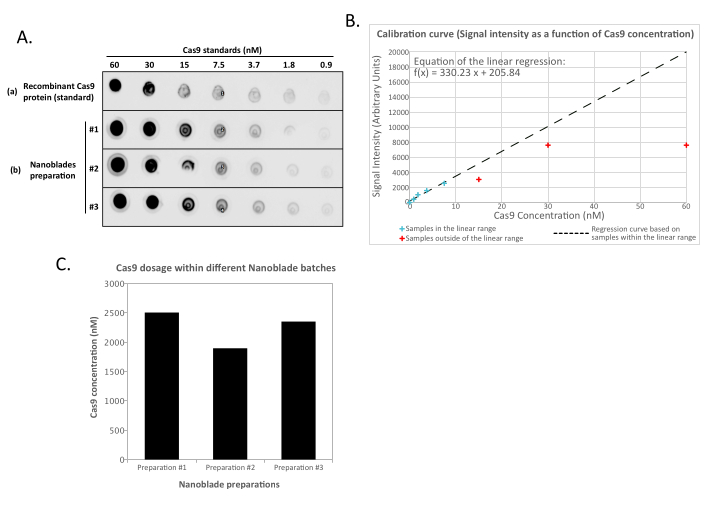
Figura 2: Quantificação do carregamento cas9 dentro de Nanoblades por ponto-blot. (A) Cas9 recombinante ou 100x concentrado (por ultracentrifugação) Amostras de nanoblade (#1, #2 e #3) são diluídas 2 vezes sequencialmente e vistas em membrana nitrocelulose antes de incubar com anticorpos anti-Cas9 HRP-acoplados. O sinal é revelado pela quimiominascência aprimorada. (B) O sinal de chemiluminescência é adquirido e quantificado para as diluições cas9 recombinantes e intensidade de sinal plotada contra a quantidade conhecida de Cas9 detectada na membrana nitrocelulose. Uma curva de regressão é calculada para as diluições que estão dentro da faixa linear (ver cruzes azuis), excluindo todas as concentrações que estão fora da faixa linear (ver cruzes vermelhas). (C) A concentração de Cas9 (nM) em cada preparação de Nanoblade foi extrapolada utilizando-se a equação da regressão linear obtida em (B). Para isso, é importante usar apenas o sinal quantificado das diluições de Nanoblade que se enquadram na faixa linear da curva de regressão. Clique aqui para ver uma versão maior desta figura.

Figura 3: Monitorando a eficiência de edição após a transdução. (A) T7 ensaio endonuclease medindo a eficiência do decote em células tratadas com nanoblade. As células transduzidas com nanoblades direcionados ao gene EMX1 foram analisadas pelo ensaio endonuclease T7. Raia 1: Lote de preparação de nanoblade #1 (20% de eficiência do decote); Faixa 2: Células de controle; Pista 3: Lote de preparação de nanoblade #2 (60% de eficiência de decote). (B) Knock-in da sequência de marca de bandeira dentro do quadro de leitura aberto DDX3 . Nanoblades concentrados programados com um sgRNA direcionado ao lócus DDX3 foram produzidos a partir de diferentes clones HEK293T (#1, #2) e complexos com doses crescentes de um modelo SsODN Flag-DDX3 e os complexos obtidos usados para a transdução de células-alvo HEK293T. Após a transdução, as células foram cultivadas por três dias antes de colhê-las para extrair DNA genômico e proteínas totais. As proteínas Flag-DDX3 foram imunoprecipitadas usando contas anti-bandeira, seguidas pela análise ocidental das proteínas recuperadas usando um anticorpo anti-Bandeira (painel superior). A inserção direcionada ao local da tag Flag no lócus Ddx3 também foi avaliada pelo PCR usando primers flanqueando o local de inserção (painel do meio) ou usando um primer para a frente que reconhece a sequência de marca de bandeira e um primer reverso específico para o lócus Ddx3 a jusante do local de inserção da bandeira (painel inferior). Abreviaturas: EMX1 = Empty Spiracles Homeobox 1; DDX3 = helicase de RNA de caixa morta 3; PCR = reação em cadeia de polimerase; ODN = oligodeoxynucleotídeo; ssODN = ODN encalhado em sing; sgRNA = RNA de guia único; IP = imunoprecipitação. Clique aqui para ver uma versão maior desta figura.
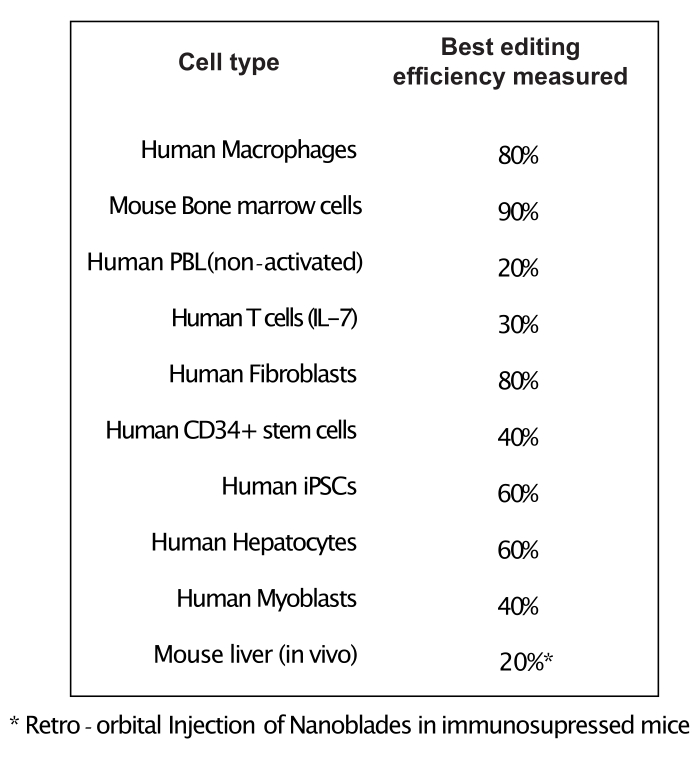
Figura 4: Eficiência de edição alcançada em diferentes tipos de células primárias usando Nanoblades. Abreviaturas: PBL = linfócito sanguíneo periférico; IL = interleucina; CD = cluster de diferenciação; iPSC = célula-tronco pluripotente induzida. Clique aqui para ver uma versão maior desta figura.
Discussão
Os nanoblados permitem a entrega rápida e dependente de doses do complexo Cas9/sgRNA RNP em linhas celulares e células primárias. Em contraste com a transfecção clássica e outros vetores de entrega viral, mas como a eletroporação de proteínas, os nanoblades têm a vantagem da entrega transitória do Cas9/sgRNA RNP de forma livre de transgenes. Os nanoblades oferecem uma plataforma altamente versátil, simples e barata para a entrega de proteínas que pode ser facilmente e rapidamente adaptada à família em constante expansão das variantes CRISPR. Nanoblades podem ser produzidos na linha celular HEK293T ou seus derivados. As linhas celulares HEK293T utilizadas aqui foram desenvolvidas para maximizar a produção de partículas retrovirais e lentivirais (ver a Tabela de Materiais). No entanto, embora outras fontes de células HEK293T possam ser adequadas, os usuários devem testar e comparar células HEK293T de diferentes fontes, pois grandes diferenças na produção de partículas foram observadas dependendo da fonte de células HEK293T. As células também devem ser verificadas quanto à contaminação do Mycoplasma com frequência e passagem a cada três dias (diluição clássica de 1/8) para evitar superconfluência, o que tem um impacto negativo na produção de partículas.
As células não devem ser mantidas por mais de 20 passagens. DMEM suplementado com glicose, penicilina/estreptomicina, glutamina e 10% soro bovino fetal descomplementado foi utilizado para a cultura celular. Como a origem sárida pode afetar a qualidade da preparação de Nanoblade, diferentes lotes de soro devem ser testados antes da produção em larga escala. Nanoblades podem ser produzidos eficientemente em outras mídias, como RPMI ou modificações sem soro do meio essencial mínimo que podem substituir o DMEM no dia seguinte à transfecção. Como indicado abaixo, embora a substituição média após a transfecção com alguns reagentes de transfecção de DNA seja opcional, pode ser benéfico modificar o meio em que os VLPs são liberados, especialmente para limitar traços de soro na preparação de partículas. No entanto, o cultivo de células em meio essencial mínimo de soro reduzido na véspera da transfecção ainda não foi tentado.
Como mencionado, nanoblades são produzidos após a superexpressão de uma mistura de plasmídeos em células produtoras. A superexpressão parece ser necessária para uma produção ideal. De fato, este laboratório desenvolveu uma linha celular produtora onde a construção de gag-pol-expressa foi estabilizada por seleção de antibióticos; no entanto, este sistema falhou em produzir quantidades significativas de Nanoblades. Uma observação semelhante foi feita quando a construção de codificação sgRNA foi firmemente integrada ao genoma das células produtoras. Como descrito para outros sistemas de produção de partículas, uma linha celular estável expressando pelo menos alguns construtos envolvidos na produção de Nanoblades pode ser útil; no entanto, isso certamente exigiria o processamento de grandes volumes de supernaspentes e uma técnica apropriada para purificar partículas. O protocolo acima descreve o procedimento preferido para produzir Nanoblades que explora reagentes de transfecção específicos (ver a Tabela de Materiais).
Embora os reagentes de transfecção de outros fabricantes também tenham sido testados com sucesso, a grande maioria dos resultados deste grupo com nanoblades seguem o procedimento aqui descrito. A transfecção de baixo custo pode ser alcançada usando reagentes de fosfato de cálcio e produzir boa eficiência de produção; no entanto, este método requer absolutamente a substituição do meio de transfecção no dia seguinte à transfecção e pode deixar resíduos de fosfato de cálcio na preparação de partículas sedimentadas. Consistente com a necessidade de altos níveis de expressão para componentes de Nanoblade dentro das células produtoras é a observação de que a quantidade de sgRNAs associadas à proteína Cas9 pode ser um fator limitante para a edição eficiente do genoma. Para melhorar o carregamento de sgRNA, duas abordagens técnicas foram recentemente desenvolvidas por grupos independentes usando vetores de entrega de proteínas semelhantes aos Nanoblades. Estes dependem do uso da expressão citoplasmática dependente de polimerase T7 do sgRNA6 ou através da adição de um sinal de encapsidation retroviral à sequência sgRNA para mediar a ligação à poliproteína gag6. Essas abordagens poderiam de fato melhorar o carregamento de sgRNA dentro de Nanoblades, embora ainda não tenham sido testadas.
A transdução de células-alvo é um passo crítico no procedimento. Na maioria das linhas celulares imortalizadas, a transdução com Nanoblades tem pouco ou nenhum efeito citopático. No entanto, nas células primárias, a toxicidade pode ser um problema. A transdução tem, portanto, de ser otimizada para cada tipo de célula. Especificamente, o tempo de exposição aos Nanoblades é um fator importante a ser modificado ao otimizar o protocolo de transdução. Para células sensíveis, como neurônios primários ou células de medula óssea, 4-6 h de incubação com Nanoblades antes de substituir o meio permite uma entrega eficiente da proteína Cas9, minimizando a toxicidade celular. Além disso, adjuvantes como polímeros cationicos, entre outros, podem melhorar significativamente a eficiência da transdução em algumas células (ver a Tabela de Materiais). É importante notar que nanoblades são VLPs e podem induzir uma resposta imunogênica. Isso pode ser uma limitação se trabalhar com certos tipos de células primárias, como macrófagos ou células dendríticas, nas quais a incubação com Nanoblades poderia induzir mudanças importantes na expressão genética e no fenótipo das células. Se macrófagos e células dendríticas são derivados de precursores de células-tronco hematopoiéticas (como células de medula óssea do rato), é preferível transduzir células com os Nanoblades antes que elas sejam totalmente diferenciadas para evitar induzir uma resposta celular contra os Nanoblades. Caso contrário, a eletroporação de proteínas Cas9 poderia representar uma alternativa viável ao trabalhar com células imunes diferenciadas.
Nanoblades podem ser usados in vivo para transduzir zigotes de camundongos ou embriões para gerar animais transgênicos. Semelhantes aos vetores retrovirais clássicos ou lentivirais, eles também podem ser injetados diretamente em tecidos de animais adultos. No entanto, nanoblades (semelhantes aos vetores retrovirais e lentivirais) podem ser inativados pela resposta imune do animal hospedeiro; portanto, a dose a ser injetada tem que ser otimizada para cada aplicação. Essa resposta imune também pode limitar a distribuição de VLPs funcionais a tecidos próximos ao local da injeção. Finalmente, ao contrário dos vetores lentivirais, os Nanoblades são livres de transgenes e entregam o Cas9 em um prazo restrito. Portanto, eles não podem ser usados para realizar triagens funcionais em todo o genoma que requerem sequenciamento de alto rendimento de sgRNAs após a seleção de células. Nanoblades são úteis quando é necessária edição rápida, dependente de doses e transgene-free de genoma20. Além disso, semelhante à eletroporação de proteínas, os nanoblades levam a menos efeitos fora do alvo do que a expressão prolongada de Cas9/sgRNA através da transfecção de DNA ou vetores virais clássicos3. O desenvolvimento futuro da Nanoblades está focado em incorporar variantes Cas9 para diferentes aplicações tecnológicas, como edição de base e segmentação de RNA.
Divulgações
Philippe E. Mangeot e Emiliano P. Ricci são nomeados como inventores de uma patente relacionada à tecnologia Nanoblades (requerentes de patente: Institut National de la Santé et de la Recherche Médicale (INSERM), Centre National de la Recherche Scientifique (CNRS), Ecole Normale Superieure de Lyon, Université Claude Bernard Lyon 1, Villeurbanne Cedex; número de inscrição: WO 2017/068077 Al; status de patente: status de publicado, 27 de abril de 2017; todos os aspectos do manuscrito são cobertos pelo pedido de patente. Os demais autores não declaram interesses concorrentes.
Agradecimentos
Este trabalho foi financiado pela Labex Ecofect (ANR-11-LABX-0048) da Université de Lyon, dentro do programa Investissements d'Avenir (ANR-11-IDEX-0007) operado pela Agência Nacional de Pesquisa Francesa (ANR), Fondation FINOVI, Agence Nationale des Recherches sur le SIDA et les Hépatites Virales (ANRS-ECTZ3306) e pelo European Research Council (ERC-StG-LS6-805500 to E.P.R.) sob os programas de pesquisa e inovação Horizon 2020 da União Europeia.
Materiais
| Name | Company | Catalog Number | Comments |
| 13.2 mL, Thinwall Polypropylene Tubes, 14 x 89 mm - 50Pk | Beckman Coulter Life Sciences | 331372 | Ultracentrifugation tubes for Nanoblades purification |
| Amersham Protran Premium Western blotting membranes, nitrocellulose | Merck | GE10600004 | Nitrocellulose membrane for quantifying Cas9 levels within purified Nanoblades |
| BIC-Gag-CAS9 | Addgene | 119942 | Encodes a GAG (F-MLV)-CAS9(sp) fusion. Allows the production of GAG-CAS9 Virus like particles from producer cells in association with over expressed gRNA(s) and appropriate envelopes |
| BICstim-Gag-dCAS9-VPR | Addgene | 120922 | Encodes a GAG-dCAS9-VPR fusion for targeted transcriptional activation |
| BLADE | Addgene | 134912 | Empty backbone for cloning sgRNA sequence to be used in Nanoblades system |
| BsmBI-v2 | New England Biolabs | R0739S | Restriction enzyme to digest the BLADE and SUPERBLADES vectors for sgRNA cloning |
| Cas9 (7A9-3A3) Mouse mAb (HRP Conjugate) #97982 | Cell Signaling Technology | 97982S | Anti-Cas9 antibody for Cas9 quantification by dot-blot |
| Cas9 Nuclease, S. pyogenes | New England Biolabs | M0386T | Recombinant Cas9 protein to be used as a reference for absolute quantification of the amount of Cas9 loaded within Nanoblades |
| Ethidium bromide solution (10 mg/mL in H2O) | Sigma-Aldrich | E1510-10ML | For staining agarose gels and visualize DNA |
| Fisherbrand Wave Motion Shakers | Fisher Scientific | 88-861-028 | Agitation table to resuspend Nanoblades upon centrifugation |
| gelAnalyzer | http://www.gelanalyzer.com; quantifying band intensity after digestion | ||
| Gesicle Producer 293T | Takara | 632617 | Nanoblades producer cell line |
| Gibco DMEM, high glucose, pyruvate | ThermoFisher Scientific | 41966052 | Cell culture medium for Gesicle Producer 293T cells |
| GoTaq G2 DNA Polymerase | Promega | M7848 | Taq polymerase for amplification of genomic DNA before T7 endonuclease assays |
| jetPRIME Transfection Reagent kit for DNA and DNA/siRNA | Polyplus | POL114-15 | Transfection reagent for Nanoblade production in Gesicle Producer 293T cells |
| Millex-AA, 0.80 µm, syringe filter | Millipore | SLAA033SS | Syringe filter to remove cellular debris before concentration of Nanoblades |
| Millex-GS, 0.22 µm, syringe filter | Millipore | SLGS033SS | Syringe filter to sterilise the sucrose cushion solution |
| Millex-HP, 0.45 µm, polyethersulfone, syringe filter | Millipore | SLHP033RS | Syringe filter to remove cellular debris before Nanoblades concentration |
| Monarch DNA Gel Extraction Kit | New England Biolabs | T1020L | DNA gel extraction kit for purification of the pBLADES or pSUPERBLADES plasmid fragment upon digestion with BsmBI |
| NEB Stable Competent E. coli (High Efficiency) | New England Biolabs | C3040I | Competent bacteria for plasmid transformation and amplification |
| NucleoBond Xtra Midi kit for transfection-grade plasmid DNA | Macherey-Nagel | 740410.50 | Maxipreparation kit for purification of plasmid DNA from cultured bacteria |
| Nucleospin gDNA extraction kit | Macherey-Nagel | 740952.50 | Extraction of genomic DNA from transduced cells |
| NucleoSpin Plasmid, Mini kit for plasmid DNA | Macherey-Nagel | 740588.50 | Minipreparation kit for purification of plasmid DNA from cultured bacteria |
| NucleoSpin Tissue, Mini kit for DNA from cells and tissue | Macherey-Nagel | 740952.5 | Genomic DNA extraction kit |
| Optima XE-90 | Beckman Coulter Life Sciences | A94471 | Ultracentrifuge |
| pBaEVRless | Els Verhoeyen (Inserm U1111) | Personnal requests have to be sent to: els.verhoyen@ens-lyon.fr | Baboon Endogenous retrovirus Rless glycoprotein described in Girard-Gagnepain, A. et al. Baboon envelope pseudotyped LVs outperform VSV-G-LVs for gene transfer into early-cytokine-stimulated and resting HSCs. Blood 124, 1221–1231 (2014) |
| pBS-CMV-gagpol | Addgene | 35614 | Enocdes the Murine Leukemia Virus gag and pol genes |
| pCMV-VSV-G | Addgene | 8454 | Envelope protein for producing lentiviral and MuLV retroviral particles |
| Phosphate-Buffered Saline (PBS) | ThermoFisher Scientific | 14200091 | 10X PBS to dilute in millipore water |
| Polybrene Transfection Reagent | Millipore Sigma | TR-1003-G | Cationic polymer that enhances the efficiency of retroviral transduction in specific mammalian cells. It can also allow viral-dependent entry of an Oligodeoxynucleotide (ODN) for homology-directed repair |
| Sucrose,for molecular biology, ≥99.5% (GC) | Sigma-Aldrich | S0389-5KG | Sucrose to prepare a cushion for Nanoblade purification through ultracentrifugation |
| SUPERBLADE5 | Addgene | 134913 | Empty backbone for cloning sgRNA sequence to be used in nanoblades system (Optimized for increased genome editing efficiency via Chen B et al., 2013) |
| SuperSignal West Dura Extended Duration Substrate | ThermoFisher Scientific | 34076 | Enhanced chemiluminescence (ECL) HRP substrate for Cas9 dot blots |
| SW 41 Ti Swinging-Bucket Rotor | Beckman Coulter Life Sciences | 331362 | Rotor for ultracentrifugation |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | Alternative to ethidium bromide for staining agarose gels and visualize DNA |
| T4 DNA Ligase | New England Biolabs | M0202S | DNA ligase to ligate the BLADE or SUPERBLADES vectors with the duplexed DNA oligos corresponding to the variable region of the sgRNA |
| T7 Endonuclease I | New England Biolabs | M0302S | T7 Endonuclease I recognizes and cleaves non-perfectly matched DNA. Allows to monitor the extent of genome editing at a specific locus |
| Triton-containing lysis buffer | Promega | E291A | Lysis buffer to disrupt Nanoblades and allow Cas9 quantification |
| TWEEN 20 | Sigma-Aldrich | P9416 | For the preparation of TBST |
Referências
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zuris, J. A., et al. Cationic lipid-mediated delivery of proteins enables efficient protein-based genome-editing in vitro and in vivo. Nature Biotechnology. 33, 73-80 (2015).
- Mangeot, P. E., et al. Genome editing in primary cells and in vivo using viral-derived Nanoblades loaded with Cas9-sgRNA ribonucleoproteins. Nature Communications. 10 (1), 45 (2019).
- Cai, Y., Bak, R. O., Mikkelsen, J. G. Targeted genome editing by lentiviral protein transduction of zinc-finger and TAL-effector nucleases. eLife. 3, 01911 (2014).
- Choi, J. G., et al. Lentivirus pre-packed with Cas9 protein for safer gene editing. Gene Therapy. 23 (7), 627-633 (2016).
- Gee, P., et al. Extracellular nanovesicles for packaging of CRISPR-Cas9 protein and sgRNA to induce therapeutic exon skipping. Nature Communications. 11, 1334 (2020).
- Indikova, I., Indik, S. Highly efficient 'hit-and-run' genome editing with unconcentrated lentivectors carrying Vpr.Prot.Cas9 protein produced from RRE-containing transcripts. Nucleic Acids Research. 48 (14), 8178-8187 (2020).
- Lyu, P., Javidi-Parsijani, P., Atala, A., Lu, B. Delivering Cas9/sgRNA ribonucleoprotein (RNP) by lentiviral capsid-based bionanoparticles for efficient 'hit-and-run' genome editing. Nucleic Acids Research. 47 (17), 99 (2019).
- Gheysen, D., Jacobs, E., de Foresta, F., Thiriart, C. Assembly and release of HIV-1 precursor Pr55gag virus-like particles from recombinant baculovirus-infected insect cells. Cell. 59 (1), 103-112 (1989).
- Hanna, R. E., Doench, J. G. Design and analysis of CRISPR-Cas experiments. Nature Biotechnology. 38 (7), 813-823 (2020).
- Sambrook, J. . Molecular cloning a laboratory manual. Third edition. , (2001).
- Jiang, W., et al. An optimized method for high-titer lentivirus preparations without ultracentrifugation. Scientific Reports. 5, 13875 (2015).
- Rueden, C. T., et al. ImageJ2: ImageJ for the next generation of scientific image data. BMC Bioinformatics. 18 (1), 529 (2017).
- O'Doherty, U., Swiggard, W. J., Malim, M. H. Human immunodeficiency virus type 1 spinoculation enhances infection through virus binding. Journal of Virology. 74 (21), 10074-10080 (2000).
- Brinkman, E. K., Chen, T., Amendola, M., van Steensel, B. Easy quantitative assessment of genome editing by sequence trace decomposition. Nucleic Acids Research. 42 (22), 168 (2014).
- Bollen, Y., Post, J., Koo, B. -. K., Snippert, H. J. G. How to create state-of-the-art genetic model systems: strategies for optimal CRISPR-mediated genome editing. Nucleic Acids Research. 46 (13), 6435-6454 (2018).
- Brinkman, E. K., et al. Easy quantification of template-directed CRISPR/Cas9 editing. Nucleic Acids Research. 46 (10), 58 (2018).
- Dussaud, S., Pardanaud-Glavieux, C., Sauty-Colace, C., Ravassard, P. Lentiviral mediated production of transgenic mice: a simple and highly efficient method for direct study of founders. Journal of Visualized Experiments. (140), e57609 (2018).
- Montagna, C., et al. VSV-G-enveloped vesicles for traceless delivery of CRISPR-Cas9. Molecular Therapy. Nucleic Acids. 12, 453-462 (2018).
- Marnef, A., et al. A cohesin/HUSH- and LINC-dependent pathway controls ribosomal DNA double-strand break repair. Genes & Development. 33 (17-18), 1175-1190 (2019).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados