Method Article
Abgabe des Cas9/sgRNA Ribonukleoprotein-Komplexes in Immortalisierte und Primärzellen über virusähnliche Partikel ("Nanoblades")
In diesem Artikel
Zusammenfassung
Wir haben ein einfaches und kostengünstiges Protokoll entwickelt, um Cas9/Single-Guide-RNA (sgRNA)-Ribonukleoproteinkomplexe in virusähnliche Partikel zu laden. Diese Partikel, "Nanoblades" genannt, ermöglichen eine effiziente Abgabe des Cas9/sgRNA-Komplexes in immortalisierten und primären Zellen sowie in vivo.
Zusammenfassung
Das clustered regularly interspaced short palindromic repeats (CRISPR)-Cas-System hat die Genom-Editierung in eukaryotischen Zellen demokratisiert und zur Entwicklung zahlreicher innovativer Anwendungen geführt. Die Abgabe des Cas9-Proteins und der Single-Guide-RNA (sgRNA) in Zielzellen kann jedoch eine technische Herausforderung darstellen. Klassische virale Vektoren, wie sie von Lentiviren (LVs) oder Adeno-assoziierten Viren (AAVs) abgeleitet werden, ermöglichen eine effiziente Abgabe von Transgenen, die für das Cas9-Protein und die zugehörige sgRNA in vielen Primärzellen und in vivo kodieren. Dennoch können diese Vektoren nachteile wie die Integration des Transgens in das Zielzellgenom, eine begrenzte Ladekapazität und die langfristige Expression des Cas9-Proteins und der Guide-RNA in Zielzellen aufweisen.
Um einige dieser Probleme zu überwinden, wurde ein auf dem murinen Leukämievirus (MLV) basierender Verabreichungsvektor entwickelt, um das Cas9-Protein und die zugehörige Guide-RNA in Abwesenheit eines kodierenden Transgens zu verpacken. Durch die Verschmelzung des Cas9-Proteins mit dem C-Terminus des Strukturproteins Gag aus MLV wurden virusähnliche Partikel (VLPs) gebildet, die mit dem Cas9-Protein und sgRNA (genannt "Nanoblades") beladen waren. Nanoblades können aus dem Kulturmedium von Produzentenzellen gesammelt, gereinigt, quantifiziert und verwendet werden, um Zielzellen zu transduzieren und den aktiven Cas9 / sgRNA-Komplex abzugeben. Nanoblades liefern ihre Ribonukleoprotein (RNP) -Ladung vorübergehend und schnell in eine Vielzahl von primären und immortalisierten Zellen und können für andere Anwendungen programmiert werden, z. B. die vorübergehende transkriptionelle Aktivierung von Zielgenen unter Verwendung modifizierter Cas9-Proteine. Nanoblades sind in der Lage, in vivo Genome-Editing in der Leber von injizierten erwachsenen Mäusen und in Eizellen zu bearbeiten, um transgene Tiere zu erzeugen. Schließlich können sie mit Donor-DNA für eine "transfektionsfreie" homologiegesteuerte Reparatur komplexiert werden. Die Nanoklingenpräparation ist einfach, relativ kostengünstig und kann problemlos in jedem zellbiologischen Labor durchgeführt werden.
Einleitung
Im Vergleich zu anderen programmierbaren Nukleasen vereinfachte und demokratisierte das CRISPR-Cas-System das Verfahren des sequenzspezifischen Genom-Targetings und der Spaltung in eukaryotischen Zellen dramatisch. Durch die einfache Expression einer sgRNA können Anwender das Cas9-Protein (oder optimierte Varianten) für fast jeden zellulären Locus programmieren1. In diesem Szenario wird die Abgabe des Cas9-Proteins und der sgRNA zur Haupteinschränkung bei der Durchführung einer ortsgerichteten Mutagenese. In immortalisierten Zellen können die sgRNA und das Cas-Protein leicht aus transfizierten Plasmiden exprimiert werden, um in den meisten Zellen ein effizientes Genom-Targeting zu erreichen. Die konstitutive Expression des Cas9/sgRNA-Komplexes kann jedoch die Off-Target-Aktivität des Cas9-Proteins erhöhen und unerwünschte Veränderungen des unspezifischen Loci2 verursachen. In Primärzellen kann die DNA-Transfektion technisch schwierig zu erreichen sein und zu einer schlechten Expression oder einem kleinen Prozentsatz transfizierter Zellen führen. Alternativen zur klassischen DNA-Transfektion umfassen die Verwendung viraler Vektoren, die ein Transgen liefern, das für die Cas9- und sgRNA kodiert, oder die Elektroporation eines rekombinanten Cas9-Proteins, das an eine synthetische sgRNA gekoppelt ist. Diese Ansätze können jedoch zu einer transgenen Integration innerhalb des Zellwirtsgenoms führen (wie es bei klassischen retroviralen und lentiviralen Expressionsvektoren der Fall ist), zu einer Einschränkung durch zelluläre Faktoren und zu einer konstitutiven Expression des Cas9-Proteins und der sgRNA.
Die Elektroporation des Cas9/sgRNA-RNP-Komplexes kann die meisten dieser Probleme überwinden und zu einer effizienten und vorübergehenden Abgabe in Primärzellen und in vivo sowie zu einer dosisabhängigen Reaktion führen. Dennoch ist es in der Regel auf teure Geräte und Reagenzien angewiesen und auch schwierig zu skalieren, wenn eine große Anzahl von Zellen behandelt werden muss. Als Alternative zu den oben genannten Techniken haben diese Autoren "Nanoblades" entwickelt - einen retroviralen Abgabevektor für das Cas9-Protein und sgRNA3, der konzeptionell anderen viralen Kapsidprotein-Abgabesystemen ähnelt4,5,6,7,8. Nanoblades nutzen die natürliche Fähigkeit des Gag-Polyproteins aus Retroviren, VLPs zu produzieren, die im extrazellulären Medium freigesetzt werden, wenn sie allein in kultivierten Zellen exprimiert werden9. Durch die Fusion des Cas9-Proteins mit dem C-terminalen Ende des Murinen Leukämievirus (MLV) Gag-Polyproteins und die Co-Exprimierung der sgRNA- und viralen Hüllglykoproteine kann das Cas9-Protein in freigesetzten VLPs oder Nanoblades verkapselt werden. Nach der Reinigung können die Nanoblades mit Zielzellen inkubiert oder in vivo injiziert werden, um eine schnelle, vorübergehende und dosisabhängige Abgabe des Cas9/sgRNA-RNP-Komplexes zu vermitteln3.
Nanoblades können mit mehreren sgRNAs für die gleichzeitige Bearbeitung an verschiedenen Orten oder mit Cas9-Varianten programmiert werden, um andere Anwendungen wie zielspezifische transkriptionelle Aktivierung oder Repression durchzuführen3. Im Gegensatz zur Proteinelektroporation, die auf rekombinanter Expression beruht, können neu beschriebene Cas-Varianten aus der Literatur leicht in den Gag-Fusionsexpressionsvektor kloniert werden, was ihn zu einer vielseitigen Plattform macht. Nanoblades können weiter komplexiert oder mit einzelsträngigen und doppelsträngigen Oligodeoxynukleotiden (ssODNs) beladen werden, um homologiegesteuerte Reparaturen durchzuführen3. Die Nanoklingenproduktion ist relativ einfach und billig. Darüber hinaus können Nanoblades bei 4 °C für viele Tage oder bei -80 °C für die Langzeitlagerung gelagert werden. Typischerweise vermitteln Nanoblades eine effiziente, transgenfreie Genom-Editierung in den meisten immortalisierten und primär kultivierten Zellen. Einige Primärzellen können jedoch empfindlich auf das Vorhandensein von Viruspartikeln reagieren, was zu einer erhöhten Mortalität führt. Zellen des angeborenen Immunsystems können auch auf das Vorhandensein von Nanoblades (aufgrund ihres viralen Ursprungs) reagieren und aktiviert werden. In diesen Fällen muss das Transduktionsprotokoll optimiert werden, um die Expositionszeit gegenüber Nanoblades zu begrenzen und unspezifische Effekte zu minimieren. Nanoblades stellen eine praktikable und einfach zu implementierende Alternative zu anderen verfügbaren CRISPR-Verabreichungsmethoden dar.
Protokoll
1. sgRNA-Design und Klonen
HINWEIS: Richtlinien für das Design von sgRNAs können aus mehreren Quellen wie https://blog.addgene.org/how-to-design-your-grna-for-crispr-genome-editing oder von Hanna und Doench10 bezogen werden.
- Sobald die 20 Nukleotid-sgRNA-Sequenzen entworfen wurden, bestellen Sie die folgenden einzelsträngigen DNA-Oligonukleotide:
- Vorwärts: 5' caccgNNNNNNNNNNNNNNNNNNN 3' (N entspricht dem Zielort ohne die Protospacer-adjacent Motif (PAM) Sequenz)
- Reverse: 5' aaacNNNNNNNNNNNNNNNNNNnc 3' (N entsprechen dem Reverse-Komplement des Zielortes ohne die PAM-Sequenz)
HINWEIS: Bei der Bestellung der Oligonukleotide sind keine besonderen Modifikationen erforderlich (keine Anforderung für 5'-Phosphat).
- Hybridisieren Sie die beiden DNA-Oligonukleotide in einem 0,2 ml Polymerase-Kettenreaktionsröhrchen (PCR), indem Sie 5 μL Glühpuffer (500 mM NaCl; 100 mM Tris-HCl; 100 mM MgCl2; 10 mM DTT; pH 7,9 bei 25 °C), 1 μL jedes DNA-Oligonukleotids (100 μM Stammlösung in Wasser) und 42 μL Wasser mischen.
- Auf einem PCR-Block die Proben bei 95 °C für 15 s inkubieren und dann die Temperatur mit einer Rampe von 0,5 °C/s auf 20 °C senken. Bei Raumtemperatur aufbewahren oder bei -20 °C lagern.
HINWEIS: Das Protokoll kann hier angehalten werden. - 10 μg der BLADE- oder SUPERBLADE sgRNA-Expressionsplasmide mit 10 Einheiten BsmBI-v2-Restriktionsenzym für 3 h bei 55 °C in einem Gesamtreaktionsvolumen von 50 μL verdauen.
HINWEIS: Der verdaute Vektor sollte einen DNA-Insert von ~1,9 kb und ein zweites DNA-Fragment von ~3,3 kb freisetzen. - Laden Sie die Restriktionsreaktion auf ein 1% iges Agarosegel, das mit 5 μg / ml Ethidiumbromid (oder einer sichereren alternativen DNA-Gelfärbung) gefärbt ist.
HINWEIS: Tragen Sie bei der Manipulation von Ethidiumbromid, das im Verdacht steht, genetische Defekte zu verursachen, eine geeignete Schutzausrüstung.- Schneiden Sie auf einem ultravioletten (UV) Tisch mit einer Wellenlänge von 312 nm (um die DNA nicht zu beschädigen) das 3,3 kb große DNA-Fragment aus dem Gel und legen Sie es in ein 1,5 ml Mikrozentrifugenröhrchen.
HINWEIS: Tragen Sie geeignete Schutzausrüstung (Handschuhe und UV-Schutzbrille), wenn Sie Ethidiumbromid manipulieren und am UV-Tisch arbeiten. - Extrahieren Sie DNA aus dem geschnittenen Gel, das das 3,3 kb DNA-Fragment enthält, mit einem speziellen DNA-Gel-Extraktionskit (siehe Materialtabelle). Quantifizieren Sie die Menge an gereinigter DNA mit einem Spektralphotometer.
HINWEIS: Das Protokoll kann hier angehalten werden.
- Schneiden Sie auf einem ultravioletten (UV) Tisch mit einer Wellenlänge von 312 nm (um die DNA nicht zu beschädigen) das 3,3 kb große DNA-Fragment aus dem Gel und legen Sie es in ein 1,5 ml Mikrozentrifugenröhrchen.
- Ligatieren Sie die hybridisierten Vorwärts- und Rückwärts-DNA-Oligonukleotide aus Schritt 1.2 zum BsmB1-verdauten, gelgereinigten BLADES- oder SUPERBLADE-Vektor aus Schritt 1.5.2. Dazu fügen Sie 2 μL T4-DNA-Ligasepuffer, 50 ng des gelgereinigten Vektors (aus Schritt 1.5.2), 1 μL der hybridisierten DNA-Oligonukleotide (aus Schritt 1.2), Wasser hinzu, um das Volumen auf 19 μL zu erhöhen, und 1 μL T4-DNA-Ligase. Inkubieren Sie die Reaktion bei 25 °C für 10 min.
- Wandeln Sie das Ligationsprodukt in kompetente Bakterien um (siehe Materialtabelle), wie in11 beschrieben. Die transformierten Bakterien werden auf einer Ampicillin-Luria-Bertani-Agarplatte plattiert und über Nacht bei 37 °C inkubiert.
- Wählen Sie mehrere isolierte Kolonien auf der Agarplatte aus, um eine DNA-Minivorbereitung11 durchzuführen (siehe Materialtabelle), und führen Sie eine Sanger-Sequenzierung mit einem U6-Vorwärtsprimer (5' GACTATCATATGCTTACCGT 3') durch, um die korrekte Ligatur der variablen SgRNA-Sequenz zu überprüfen.
HINWEIS: Andere sgRNA-Expressionsplasmide können verwendet werden, wenn sie nicht für das Cas9-Protein kodieren, was die Nanoblade-Produktion stören könnte.
2. Plasmidpräparation
- Führen Sie eine Maxivorbereitung (siehe Materialtabelle) aller erforderlichen Plasmide durch und bereiten Sie 10 μg Aliquots bei 1 μg/ml vor, um sie bei -20 °C zu lagern. Vermeiden Sie wiederholte Gefrier- / Auftauzyklen der Plasmide; Verwenden Sie Aliquots zweimal, bevor Sie sie entsorgen.
3. Nanoblade-Vorbereitung
- An Tag 1 samen Sie zwischen 3,5 und 4 × 106 HEK293T-Zellen (siehe Materialtabelle) in 10 ml modifiziertem Eagle-Medium (DMEM) von Dulbecco, das hohe Glukose, Natriumpyruvat, L-Glutamin, 10% fötales Rinderserum (FBS) und Penicillin / Streptomycin in einer 10 cm langen Zellkulturschale enthält. Bewegen Sie den 10 cm großen Teller vorsichtig vor und zurück, dann von rechts nach links (wiederholen Sie diese Sequenz 5x), um die Zellen homogen über die Kulturschale zu verteilen. Inkubieren Sie Zellen bei 37 °C in einem Zellinkubator mit 5% CO2.
HINWEIS: Alle Verfahren im Zusammenhang mit der Handhabung von kultivierten Zellen und Nanoblades sollten unter einer Zellkultur-Laminar-Flow-Haube durchgeführt werden, um deren Kontamination zu vermeiden. - Tag 2: Plasmidtransfektion
- Die Zellen sollten 24 h nach dem Plattieren zu 70-80% konfluent sein (Abbildung 1A). Ersetzen Sie das Medium vor der Transfektion durch 10 ml frisches DMEM mit hohem Glukosegehalt, Natriumpyruvat, L-Glutamin, 10% FBS (Penicillin und Streptomycin können weggelassen werden, obwohl es nicht obligatorisch ist).
HINWEIS: Bei diesem Schritt ist es wichtig, dass die Zellen nicht konfluent sind. Andernfalls könnte die Transfektionseffizienz sowie die Partikelproduktion reduziert werden. - Bereiten Sie für jede 10 cm Platte die folgenden Mengen an Plasmiden in einem 1,5 ml Röhrchen vor: 0,3 μg pCMV-VSV-G, 0,7 μg pBaEVRless, 2,7 μg MLV Gag/Pol, 1,7 μg BIC-Gag-Cas9, 4,4 μg BLADES- oder SUPERBLADES-Plasmid, das die geklonte sgRNA kodiert (oder jeweils 2,2 μg bei Verwendung von zwei sgRNAs).
- Fügen Sie 500 μL Transfektionspuffer (siehe Materialtabelle), Wirbel für 10 s hinzu und zentrifugieren Sie dann für 1 s. Fügen Sie 20 μL des Transfektionsreagenzes hinzu (siehe Materialtabelle), wirbeln Sie das Röhrchen für 1 s und zentrifugieren Sie es dann für 1 s.
- Inkubieren Sie für 10 min bei Raumtemperatur und geben Sie die gesamte Lösung tropfenweise zu den Zellen in DMEM-Medium mit einem P1000-Pipettor. Bewegen Sie die 10 cm große Platte vorsichtig vor und zurück, dann von rechts nach links (wiederholen Sie diese Sequenz 5x), um das Transfektionsreagenz gleichmäßig über die Zellen zu verteilen. Inkubieren Sie Zellen bei 37 °C für mindestens 40 h in einem Zellinkubator mit 5% CO2.
HINWEIS: Falls gewünscht, kann das Medium 4 h nach der Transfektion gewechselt werden.
- Die Zellen sollten 24 h nach dem Plattieren zu 70-80% konfluent sein (Abbildung 1A). Ersetzen Sie das Medium vor der Transfektion durch 10 ml frisches DMEM mit hohem Glukosegehalt, Natriumpyruvat, L-Glutamin, 10% FBS (Penicillin und Streptomycin können weggelassen werden, obwohl es nicht obligatorisch ist).
- Überprüfen Sie an Tag 3 die Morphologie der transfizierten Zellen unter dem Mikroskop.
HINWEIS: Die Produzentenzellen beginnen zu verschmelzen. Dies ist aufgrund der Expression von fusogenen Virushüllen ein normales Ereignis (Abbildung 1B,C). - Tag 4: Ernte von Nanoblades
HINWEIS: Mindestens 40 h nach der Transfektion wären die Zellen aufgrund der Expression der fusogenen Virushüllen miteinander verschmolzen, und manchmal sind die Zellen vollständig von der Plattenhalterung getrennt (Abbildung 1D).- Sammeln Sie 9 ml des Mittleren Überstandes der Kultur mit einer 10 ml Pipette.
HINWEIS: Nanoblades sind VLPs, die in der Lage sind, das Cas9-Protein und die zugehörige sgRNA in Primärzellen und in vivo zu liefern. Obwohl sie nicht als genetisch veränderte Organismen gelten, da sie frei von genetischem Material sind, können sie genetische Veränderungen induzieren. Daher müssen sie mit Vorsicht manipuliert werden, um jeglichen Kontakt mit Benutzern zu vermeiden (insbesondere wenn sie so programmiert sind, dass sie auf Tumorsuppressorgene abzielen). Den Anwendern wird empfohlen, bei der Vorbereitung von VLPs und der Durchführung von Transduktionsexperimenten ihre lokalen Sicherheitsrichtlinien für die Manipulation retroviraler Vektoren zu befolgen und in einem BSL-2-Labor zu arbeiten. Nanoblades können mit 70% Ethanol oder 0,5% Natriumhypochlorit inaktiviert werden. Es ist auch ratsam, alle Kunststoffabfälle (Pipettenspitzen, Gewebekulturplatten, Zentrifugationsröhrchen) mit 0,5% Natriumhypochlorit für mindestens 10 min zu behandeln, um die Nanoblades zu inaktivieren. - Zentrifugieren Sie den gesammelten Überstand bei 500 × g für 5 min, um Zelltrümmer zu entfernen und den Überstand zurückzugewinnen, ohne das Zellpellet zu stören.
HINWEIS: Wenn Nanoblades für die Verwendung auf Primärzellen bestimmt sind, filtern Sie den Überstand mit einem 0,45 μm- oder 0,8 μm-Filter. Beachten Sie, dass dieser Schritt den Nanoblade-Titer drastisch reduziert, da ein erheblicher Anteil in der Filtermembran blockiert wird. - Pelletieren Sie die Nanoblades über Nacht (12-16 h) in einem schwingenden Eimerrotor bei 4.300 × g oder bei 209.490 × g in einer Ultrazentrifuge für 75 min bei 4 °C (siehe Materialtabelle).
HINWEIS: Wenn Zielzellen in DMEM wachsen können, ist es möglich, sie direkt mit dem nach Schritt 3.4.2 erhaltenen Überstand zu inkubieren, ohne die Nanoblades zu konzentrieren.
- Sammeln Sie 9 ml des Mittleren Überstandes der Kultur mit einer 10 ml Pipette.
- Tag 5: Resuspension und Lagerung von Nanoblades
- Nach der Zentrifugation das Medium langsam absaugen und das weiße Pellet mit 100 μL kalter 1x phosphatgepufferter Kochsalzlösung (PBS) resuspendieren. Das Röhrchen mit Parafilm abdecken und 1 h bei 4 °C mit sanfter Bewegung inkubieren, bevor das Pellet durch Pipettieren auf und ab wiederverwendet wird.
HINWEIS: Bei der Wiederverwendung kann ein weißes viskoses Material auftreten; Dies ist normal und wirkt sich nicht wesentlich auf die Effizienz der Transduktion aus. - Lagern Sie die Nanoblades bei 4 °C, wenn Sie planen, sie innerhalb von vier Wochen zu verwenden. Andernfalls frieren Sie die Nanoblades in flüssigem Stickstoff ein und lagern sie bei -80 °C.
HINWEIS: Tragen Sie eine Schutzbrille und kryogene Handschuhe, wenn Sie flüssigen Stickstoff manipulieren. Snap-Freezing und Lagerung bei -80 °C führt zu einer signifikanten Abnahme der Nanoblade-Effizienz. Außerdem sollten aufgetaute Nanoblades nicht wieder eingefroren werden. Das Protokoll kann hier pausiert werden.
- Nach der Zentrifugation das Medium langsam absaugen und das weiße Pellet mit 100 μL kalter 1x phosphatgepufferter Kochsalzlösung (PBS) resuspendieren. Das Röhrchen mit Parafilm abdecken und 1 h bei 4 °C mit sanfter Bewegung inkubieren, bevor das Pellet durch Pipettieren auf und ab wiederverwendet wird.
4. Konzentration von Nanoblades auf einem Saccharose-Kissen
HINWEIS: Alternativ zur Zentrifugation über Nacht oder Ultrazentrifugation (Schritt 3.4.3) können die Nanoblades auf einem Saccharosekissen konzentriert werden. Dies ergibt einen reineren Anteil an Nanoblades, obwohl die Gesamtmenge, die zurückgewonnen wird, niedriger sein wird.
- Bereiten Sie eine 10% ige Saccharoselösung (Gewicht zu Volumen) in 1x PBS vor und filtern Sie sie durch einen 0,2 μm Spritzenfilter (siehe Materialtabelle).
- Beginnen Sie mit der Konzentration der Nanoblades auf dem Saccharosekissen.
- 9 ml VLP-haltige Probe (ab Schritt 3.4.3) in ein Ultrazentrifugenröhrchen geben (siehe Materialtabelle). Mit einer 3-ml-Spritze und einer Kanüle langsam 2,5 ml der 10% igen Saccharose unter die Probe schichten und versuchen, die VLP-haltige Probe und die Saccharoselösung nicht zu mischen.
- Alternativ können Sie 2,5 ml 10% Saccharose in ein Ultrazentrifugenröhrchen geben (siehe Materialtabelle). Kippen Sie das Röhrchen und fügen Sie langsam die 9 ml VLP-haltige Probe (aus Schritt 3.4.3) mit einem Pipettiertor mit niedriger Geschwindigkeit hinzu. Heben Sie während dieser Operation das Rohr progressiv in eine vertikale Position.
- Zentrifugieren Sie die Proben bei 209.490 × g in einer Ultrazentrifuge für 90 min bei 4 °C.
HINWEIS: Diese Technik kann für die Zentrifugation mit niedriger Geschwindigkeit (4.300 × g) über Nacht angepasst werden, wie in 12 beschrieben. - Entfernen Sie nach der Zentrifugation den Überstand vorsichtig und legen Sie den Schlauch kopfüber auf Seidenpapier, um die verbleibende Flüssigkeit zu entfernen. Nach 1 min 100 μL 1x PBS hinzufügen und das Röhrchen bei 4 °C mit einer Parafilmabdeckung in einem Röhrchenhalter für 1 h auf einen Rührtisch stellen (siehe Materialtabelle), bevor das Pellet durch Pipettieren auf und ab wiederverwendet wird.
HINWEIS: Das Protokoll kann hier angehalten werden.
5. Überwachung der Cas9-Belastung in Nanoblades durch Dot-Blot
- Bereiten Sie den Verdünnungspuffer vor, indem Sie 1 Volumen Lysepuffer mit einem nichtionischen Tensid (siehe Materialtabelle) in 4 Bänden von 1x PBS hinzufügen. Verdünnen Sie 2 μL konzentrierte Nanoblades in 50 μL Verdünnungspuffer, wirbeln Sie kurz und übertragen Sie 25 μL dieser Mischung in ein neues Röhrchen mit 25 μL Verdünnungspuffer. Wiederholen Sie diesen Vorgang, um 4 Röhrchen Nanoblade-Verdünnungen (2-fache Verdünnungsschritte) zu erhalten.
- Für die Standardkontrollen verdünnen Sie 2 μL rekombinante Cas9-Nuklease (siehe Materialtabelle) in 50 μL Verdünnungspuffer, Wirbel kurz und fahren Sie fort, acht serielle Verdünnungen (2-fache Verdünnung für jeden Schritt) zu machen.
- Markieren Sie vorsichtig 2,5 μL jeder VLP-Verdünnung und 2,5 μL jedes Standards auf einer Nitrocellulosemembran mit einem Mehrkanalpipetett (ein größeres Volumen kann zu überlappenden Stellen führen).
HINWEIS: Eine methanolbehandelte Polyvinyldifluoridmembran kann ebenfalls verwendet werden. - Sobald die Partikel auf der Membran absorbiert sind, blockieren Sie die Membran mit 1x Tris-gepufferter Kochsalzlösung, die ein nichtionisches Tensid (TBS-T) enthält, das mit fettfreier Trockenmilch (5% w/v) für 45 min bei Raumtemperatur ergänzt wird.
HINWEIS: Hier kann das Protokoll pausiert und die Membran bei 4 °C in 1x TBS-T gelagert werden. - Verwerfen Sie die 1x TBST, die mit fettfreier Trockenmilch ergänzt wird, und inkubieren Sie die Membran über Nacht bei 4 °C mit dem Cas9-Meerrettichperoxidase-Antikörper (1/1000 Verdünnung in 1x TBST, 5% Milch). Waschen Sie die Membran 3x mit TBS-T und visualisieren Sie das Signal mit einem verbesserten Chemilumineszenz-Substrat-Kit.
- Quantifizieren Sie die Punktintensität für die Nanoblades und rekombinanten Cas9-Standardverdünnungen mit der proprietären Software, die mit der Gel-Bildgebungsstation oder imageJ13 geliefert wird. Definieren Sie eine lineare Kurve, die die Punktintensität mit der Cas9-Konzentration verknüpft. Unter Verwendung der Funktion der erhaltenen Kurve wird der Cas9-Gehalt in jeder Zubereitung extrapoliert.
HINWEIS: Die Menge an rekombinanter Cas9-Proteinkontrolle kann den Messwert für die konzentriertesten Proben des Standardverdünnungssatzes sättigen (Abbildung 2). Es wird daher empfohlen, bei der Definition der linearen Kurve den Messwert aus den unverdünnten Proben (und manchmal auch aus den ersten Verdünnungsschritten) zu entfernen, wenn sie nicht im linearen Bereich in Bezug auf die bekannte Konzentration von Cas9 liegen, die entdeckt wurde. Verwenden Sie bei der Extrapolation der Cas9-Menge innerhalb der Nanoblade-Samples nur die Messwerte, die innerhalb des linearen Bereichs der Standardkurve liegen.
6. Transduktion von Zielzellen mit Nanoblades (Verfahren zur Transduktion in einer 12-Well-Platte)
- In einer 12-Well-Platte säen Sie 100.000-200.000 Zellen (entweder primäre oder immortalisierte adhärente Zellen) pro Vertiefung in 1 ml des entsprechenden Zellkulturmediums. Lassen Sie die Zellen vor der Transduktion an der Plattenoberfläche haften.
- In einem 1,5 mL Mikrozentrifugenröhrchen 5-20 μL konzentrierte Nanoblades (aus Schritt 3.5.1 oder 4.4) zu 500 μL Zellkulturmedium geben und durch Pipettieren mit einem P1000-Pipettor auf und ab mischen. Entfernen Sie das Medium aus den Zellen und ersetzen Sie es durch die 500 μL dieser Nanoblade-Mischung.
HINWEIS: Die Transduktion muss für jeden Zelltyp optimiert werden. Es ist wichtig, ein möglichst kleines Mediumvolumen zu verwenden (unter Vermeidung der Trocknung der Zielzellen), damit die Nanoblades hochkonzentriert bleiben. Adhärente Zellen müssen direkt transduziert werden, während sie an die Platte gebunden sind (nicht in Suspension transduzieren, da dies die Transduktionseffizienz signifikant verringert). Einige Zellen tolerieren eine längere Exposition gegenüber Nanoblades (24-48 h), während andere sehr empfindlich sind und kleine Synzytien bilden können. In diesem Fall müssen Nanoblades nur für 4-6 h mit Zellen inkubiert werden, bevor das Medium ausgetauscht wird. Spinokulation14 kann auch die Transduktion für in Suspension gezüchtete Zellen verbessern. Adjuvantien wie kationische Polymere (siehe Materialtabelle) können auch die Transduktionseffizienz in einigen Zelltypen verbessern. - Nach 4-6 h Zellinkubation in einem geringen Volumen von Medium, das Nanoblades enthält, erhöhen Sie das Volumen des Mediums auf die normale Menge (1 ml, wenn Sie mit einer 12-Well-Platte arbeiten) oder ersetzen Sie es durch frisches Medium, wenn die Zellen empfindlich auf VLPs reagieren.
HINWEIS: Zellmedium, das Nanoblades enthält, muss 10 Minuten lang mit 0,5% Natriumhypochlorit inaktiviert werden, bevor es verworfen wird. Verwenden Sie Handschuhe und Schutzbrille, wenn Sie Natriumhypochlorit manipulieren. Wenn Nanoblades den Zelltod induzieren, passen Sie die Menge und die Gesamtzeit der Exposition an, um die Zellsterblichkeit zu reduzieren.
7. Messung der CRISPR-Effizienz am Zielort mittels T7-Endonuklease-Assay
- Entwerfen Sie PCR-Primer, um eine 400-700 Base-Pair-Region (bp) zu verstärken, die die CRISPR-Spaltungsstelle umfasst.
HINWEIS: Die Spaltstelle sollte um mindestens 200 bp von der Amplikonkante entfernt sein und sollte leicht von der Mitte des Amplikons verschoben werden, so dass bei der T7-Endonuklease-Spaltung 2 Fragmente unterschiedlicher Größe freigesetzt werden. - Extrahieren Sie genomische DNA aus Zellen, die mit Nanoblades behandelt wurden, die auf das interessierende Gen abzielen, und aus Kontrollzellen, die mit Nanoblades behandelt wurden, die mit einer Kontroll-sgRNA programmiert sind (siehe Materialtabelle).
HINWEIS: Das Protokoll kann hier angehalten werden. - Unter Verwendung von 150 ng genomischer DNA als Vorlage programmieren Sie eine PCR-Reaktion von 30 μL Volumen (Endvolumen), indem Sie dem Protokoll des Herstellers folgen. Überprüfen Sie, ob die PCR-Amplifikation ein einzelnes Amplikon der erwarteten Größe ergibt, indem Sie ein 2% iges Agarosegel verwenden, das mit 5 μg / ml Ethidiumbromid (oder einer sichereren alternativen DNA-Gelfärbung) gefärbt ist.
HINWEIS: Tragen Sie bei der Manipulation von Ethidiumbromid, das im Verdacht steht, genetische Defekte zu verursachen, eine geeignete Schutzausrüstung. Das Protokoll kann hier pausiert werden. - Heteroduplex-Erzeugung und -Verdauung
- In einem 0,2-ml-PCR-Röhrchen werden 5 μL des Enzympuffers (mit der T7-Endonuklease I bereitgestellt), 20 μL Wasser und 24 μL des PCR-Produkts aus Schritt 7.3 zugegeben. Heteroduplexbildung zulassen, indem die Proben über 3 min auf 94 °C erhitzt und dann die Temperatur (2 °C pro min) auf 40 °C gesenkt wird.
- Jedem Heteroduplexröhrchen, einschließlich der Kontrolle, werden 0,5 μL T7-Endonuklease I bei Raumtemperatur zugegeben. Bei 37 °C für 15 min inkubieren. Laden Sie die resultierende Reaktion in ein 2,5% (Gewicht/Volumen) Agarosegel, das mit Ethidiumbromid gefärbt ist. Stellen Sie das Gel nach der Migration auf einem UV-Transilluminator dar.
HINWEIS: Tragen Sie bei der Manipulation von Ethidiumbromid, das im Verdacht steht, genetische Defekte zu verursachen, eine geeignete Schutzausrüstung. Verwenden Sie eine UV-Schutzbrille, wenn Sie den UV-Transilluminator verwenden. - Messen Sie die Spaltungseffizienz, indem Sie das Bild analysieren, das sich aus der Aufschlussreaktion ergibt, um die Intensität jedes Bandes mit geeigneter Software zu quantifizieren (siehe Materialtabelle).
8. Messung der CRISPR-Effizienz am Zielort durch Sanger-Sequenzierung und TIDE-Analyse
HINWEIS: Als Alternative zum T7-Endonuklease-Assay kann die CRISPR-Effizienz durch Analyse und Dekonvolution von Sanger-Sequenzierungsspuren basierend auf dem TIDE-Protokoll überwacht werden15.
- Führen Sie eine Sanger-Sequenzierung von PCR-Amplikonen aus Schritt 7.3 (einschließlich einer Kontrollbedingung, die unbehandelten Zellen entspricht) entweder mit dem Vorwärts- oder Rückwärts-PCR-Primer durch.
- Analysieren Sie die Sanger-Sequenzierungsspuren der Kontrollbedingung (unbehandelte Zellen) und mit Nanoblade behandelte Proben mit dem TIDE-Server (https://tide.nki.nl) und befolgen Sie deren Analyserichtlinien.
9. Nanoblade-Komplexbildung mit ssODN-Donatoren zur homologiegesteuerten Reparatur (Verfahren zur Transduktion in einer 12-Well-Platte)
HINWEIS: Richtlinien für den Entwurf von ssODN für eine effiziente homologiegesteuerte reparaturvermittelte Bearbeitung wurden bereits beschrieben16.
- In einer 12-Well-Platte säen Sie 100.000-200.000 Zellen pro Vertiefung in 1 ml des entsprechenden Zellkulturmediums. Lassen Sie die Zellen vor der Transduktion an der Plattenoberfläche haften.
- 100 μL einer Lösung des kationischen Polymers (siehe Materialtabelle) bei 8 μg/ml in 1x PBS herstellen.
- Mischen Sie 19 μL der kationischen Polymerlösung mit 100 pmol der ssODN-Schablone. 20 μL konzentrierte Nanoblades (ab Schritt 3.5.1 oder 4.4) zugeben und 15 min auf Eis inkubieren.
- Entfernen Sie die komplexierten Nanoblades/ssODN aus dem Eis und fügen Sie 500 μL Zellkulturmedium (bei 37 °C) hinzu. Entfernen Sie das Medium aus den Zielzellen (ab Schritt 9.1) und fügen Sie das 500 μL Medium hinzu, das die komplexierten Nanoblades/ssODN enthält. Lassen Sie die Zellen vor der Genotypisierung 48 h lang proliferieren.
- Extrahieren Sie genomische DNA aus einem Bruchteil der Zellpopulation mit einem speziellen Extraktionskit (siehe Materialtabelle).
- Entwerfen Sie PCR-Primer, um eine 400-700 bp-Region zu verstärken, die die Knock-in-Site umfasst.
HINWEIS: PCR-Primer sollten sich nicht mit den Homologiearmen des ssODN überschneiden, um falsch-positive Ergebnisse zu vermeiden, die sich aus der PCR-Amplifikation aller noch in den Zielzellen vorhandenen Rest-ssODN ergeben. - Programmieren Sie anhand von 150 ng genomischer DNAs aus Kontrollzellen (unbehandelt) oder mit Nanoblade behandelten Zellen als Vorlage eine 30 μL PCR-Reaktion nach dem Protokoll des Herstellers.
HINWEIS: ssODN-Spuren können einige Tage nach der Transduktion mit dem Komplex im Zellmedium vorhanden sein. Dieses ssODN kann als Teilvorlage für PCR-Assays dienen, die versuchen, nach der korrekten Integration zu suchen. Daher ist es ratsam, die Zellen nach der Transduktion mindestens zweimal zu passieren, um eventuelle falsch-positive Assays zu vermeiden. - Belastung 5 μL der Kontroll- und Nanoblade-behandelten PCR-Reaktionen in einem 1% (Gewicht/Volumen) Agarosegel, das mit Ethidiumbromid gefärbt ist. Stellen Sie das Gel nach der Migration auf einem UV-Transilluminator dar.
HINWEIS: Wenn die Homologie-Rekombination erfolgreich ist und der Insertion von mehr als 1 bp genetischem Material entspricht, sollte das Molekulargewicht der PCR-Amplikone zwischen der Kontrollprobe und der mit Nanoblade behandelten Probe unterschiedlich sein. Da die Effizienz von HDR nicht 100% erreicht, sollten in der mit Nanoblade behandelten Probe zwei Bandbreiten sichtbar sein (eines von ähnlicher Größe wie das Kontroll-PCR-Amplikon, das dem unbearbeiteten Allel entspricht, und eines mit höherem Molekulargewicht, das dem Knock-in-Allel entspricht, siehe Abbildung 3B mittleres Feld).
- Entwerfen Sie PCR-Primer, um eine 400-700 bp-Region zu verstärken, die die Knock-in-Site umfasst.
- Führen Sie eine Sanger-Sequenzierung der Kontroll- und Nanoblade-behandelten PCR-Amplikone durch.
- Quantifizieren Sie die Knock-in-Effizienz mit dem TIDER-Protokoll17.
10. Nanoblade-Abgabe in vivo
- Abgabe von bis zu 25 μL konzentrierter Nanoblades ab Schritt 3.5.1 durch retroorbitale Injektion oder bis zu 100 μL durch Schwanzveneninjektion, wie in18 beschrieben, wenn Sie mit Mäusen arbeiten.
HINWEIS: Alle Verfahren, die Tierversuche beinhalten (einschließlich Nanoblade-Injektionen für Genom-Editing-Zwecke), erfordern ein von einer lokalen Ethikkommission genehmigtes Protokoll. - Verwenden Sie für die Erzeugung transgener Mäuse einen Mikroinjektor, um 1 pL bis 10 pL konzentrierte Nanoblasen aus Schritt 4.4 in den perivitellinen Raum von Mausoozyten abzugeben, wie zuvor beschrieben18.
HINWEIS: Für die Perivitellin-Injektion ist es wichtig, Nanoblades auf einem Saccharosekissen zu reinigen und zu konzentrieren, um eine Verstopfung des Mikroinjektors zu vermeiden.
Ergebnisse
Das Protokoll für die Nanoblade-Vorbereitung ist ziemlich einfach und erfordert neben dem Zugang zu einer Gewebekulturhaube, einem CO2-Inkubator und einer schwingenden Eimerzentrifuge oder einer Ultrazentrifuge eine einfache Laborausrüstung. Einige Schritte erfordern jedoch besondere Aufmerksamkeit, wie z. B. die Quelle und Handhabung von Produzentenzellen sowie transduktionsbedingungen. Wie in Abbildung 1A gezeigt, ist es wichtig, Zellen so zu säen, dass sie homogen in der Platte verteilt sind und am Tag der Transfektion ~ 70-80% Konfluenz erreichen (vermeiden Sie Zellklumpen). Vierundzwanzig Stunden nach der Transfektion (Abbildung 1B,C) bilden die Produzentenzellen eine Synzytie, die zu Zellen größerer Größe mit mehreren Kernen führt. Vierzig Stunden nach der Transfektion (Abbildung 1D) haben die meisten Zellen in der Platte synzytiös gebildet und beginnen sich von der Platte zu lösen.
Dies ist völlig normal und wird durch die Expression des Hüllglykoproteins verursacht, das die Fusion zwischen benachbarten Zellen induziert. Bei Konzentration durch Zentrifugation (oder sogar direkt aus dem Überstand der Produzentenzellen) kann die in Nanoblades geladene Cas9-Menge absolut durch Dot-Blot auf einer Nitrocellulosemembran unter Verwendung des rekombinanten Cas9 als Referenz quantifiziert werden (Abbildung 2). Dieser Schritt ist wichtig, um die richtige Menge an Nanoblades für die Transduktion von Zielzellen zu bestimmen. Bei der Durchführung des Dot-Blot-Assays ist es wichtig, nur die Messwerte zu berücksichtigen, die in den linearen Bereich der Standardkurve fallen. Unabhängig von der Menge an Cas9, die in Nanoblades vorhanden ist, ist es jedoch wichtig, die Effizienz der Genom-Editierung direkt an Zielzellen mit dem T7-Endonuklease-Assay (Abbildung 3) oder der Sanger-Sequenzierung zu testen.
Wie in Abbildung 3 gezeigt, kann die Effizienz von Nanoblades von Charge zu Charge unterschiedlich sein, obwohl sie normalerweise mit der Menge an Cas9 korreliert. In dem in Abbildung 3 gezeigten Beispiel führt die Charge aus Bahn 1 zu einer Gesamtbearbeitungseffizienz von 20 %, während die Charge aus Bahn 3 zu einer Effizienz von 60 % führt. In diesem Fall ist es möglich, das Volumen der verwendeten Nanoblades aus Charge 1 zu erhöhen, um eine Bearbeitungseffizienz zu erreichen, die der von Charge 3 ähnelt. Abbildung 4 zeigt die maximale Bearbeitungseffizienz, die mit Nanoblades in verschiedenen Arten von Primärzellen erzielt wurde. Es ist wichtig zu beachten, dass die Effizienz in Abhängigkeit von der Sequenz der verwendeten sgRNA und der Zielzugänglichkeit variieren kann.
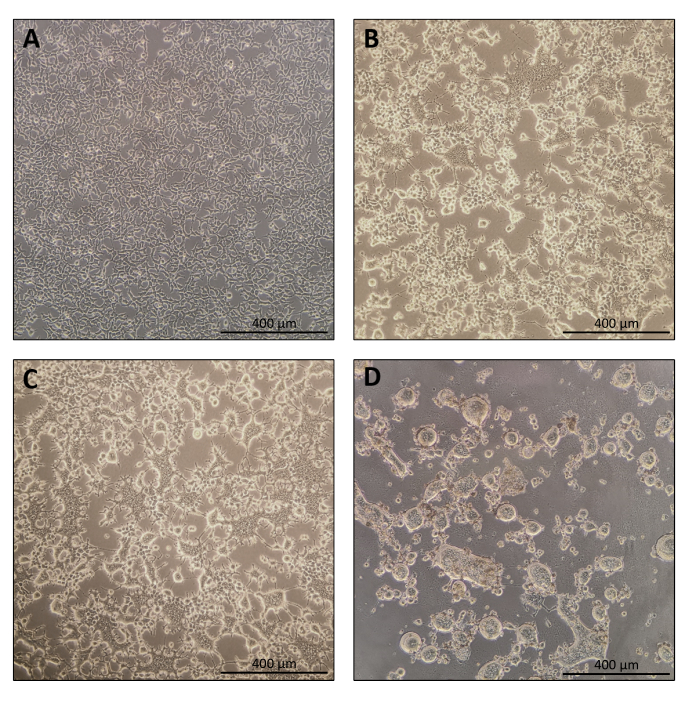
Abbildung 1: Morphologie der Produzentenzellen während der Nanoblade-Produktion. (A) HEK293T-Zellen bei 70-80% Konfluenz 24 h nach der Beschichtung. (B und C) HEK293T Zellmorphologie 24 h nach Transfektion. (D) HEK293T Zellmorphologie 40 h nach Transfektion. Maßstabsbalken = 400 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
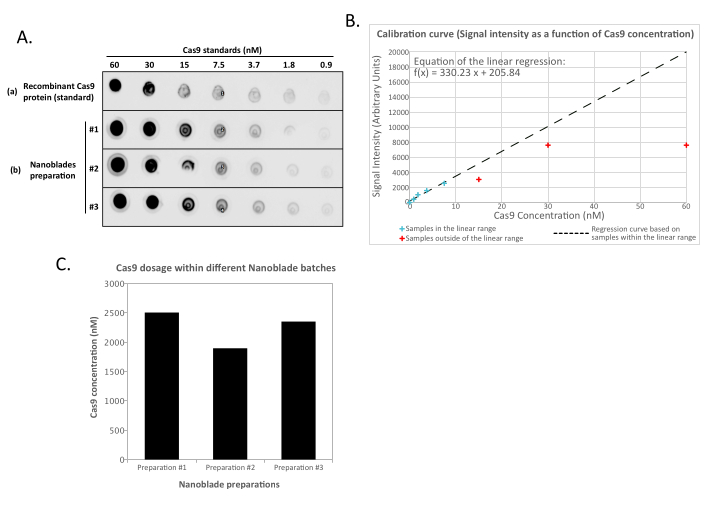
Abbildung 2: Quantifizierung der Cas9-Belastung innerhalb von Nanoblades durch Dot-Blot. (A) Rekombinante Cas9- oder 100-fache konzentrierte (durch Ultrazentrifugation) Nanoblade-Proben (#1, #2 und #3) werden 2-fach sequentiell verdünnt und vor der Inkubation mit Anti-Cas9-HRP-gekoppelten Antikörpern auf einer Nitrocellulosemembran gefleckt. Das Signal wird durch eine verstärkte Chemilumineszenz aufgedeckt. (B) Das Chemilumineszenzsignal wird erfasst und quantifiziert für die rekombinanten Cas9-Verdünnungen und die Signalintensität, die gegen die bekannte Menge an Cas9 aufgetragen werden, die auf der Nitrocellulosemembran entdeckt wurde. Für die Verdünnungen, die innerhalb des linearen Bereichs liegen (siehe blaue Kreuze), wird eine Regressionskurve berechnet, wobei alle Konzentrationen ausgeschlossen sind, die außerhalb des linearen Bereichs liegen (siehe rote Kreuze). (C) Die Cas9-Konzentration (nM) in jeder Nanoblade-Zubereitung wurde unter Verwendung der Gleichung aus der linearen Regression in (B) extrapoliert. Dazu ist es wichtig, nur das quantifizierte Signal der Nanoblade-Verdünnungen zu verwenden, die in den linearen Bereich der Regressionskurve fallen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Überwachung der Bearbeitungseffizienz bei transduktion. (A) T7-Endonuklease-Assay zur Messung der Spaltungseffizienz in mit Nanoblade behandelten Zellen. Zellen, die mit Nanoblades transduziert wurden, die auf das EMX1-Gen abzielen, wurden mittels T7-Endonuklease-Assay analysiert. Bahn 1: Nanoblade-Präparationscharge #1 (20% Spaltungseffizienz); Spur 2: Steuerzellen; Bahn 3: Nanoblade-Präparationscharge #2 (60% Spaltungseffizienz). (B) Knock-in der Flag-Tag-Sequenz innerhalb des offenen DDX3-Leserahmens . Konzentrierte Nanoblades, die mit einer sgRNA programmiert wurden, die auf den DDX3-Locus abzielt, wurden aus verschiedenen HEK293T-Klonen (#1, #2) hergestellt und mit steigenden Dosen einer Flag-DDX3 ssODN-Vorlage und der erhaltenen Komplexe, die für die Transduktion von HEK293T-Zielzellen verwendet werden, komplexiert. Nach der Transduktion wurden die Zellen drei Tage lang gezüchtet, bevor sie gesammelt wurden, um genomische DNA und Gesamtproteine zu extrahieren. Flag-DDX3-Proteine wurden mit Anti-Flag-Agaroseperlen immunpräzipitiert, gefolgt von einer Western-Blot-Analyse der gewonnenen Proteine mit einem Anti-Flag-Antikörper (oberes Bild). Die standortgesteuerte Insertion des Flag-Tags in den Ddx3-Locus wurde ebenfalls mittels PCR untersucht, wobei entweder Primer, die die Insertionsstelle flankieren (mittleres Panel), oder unter Verwendung eines Forward-Primers, der die Flag-Tag-Sequenz erkennt, und eines Reverse-Primers, der für den Ddx3-Locus stromabwärts der Flag-Insertionsstelle (unteres Feld) spezifisch ist, untersucht wurde. Abkürzungen: EMX1 = Empty Spiracles Homeobox 1; DDX3 = DEAD-Box-RNA-Helikase 3; PCR = Polymerase-Kettenreaktion; ODN = Oligodesoxynukleotid; ssODN = singsträngiges ODN; sgRNA = Single-Guide-RNA; IP = Immunpräzipitation. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
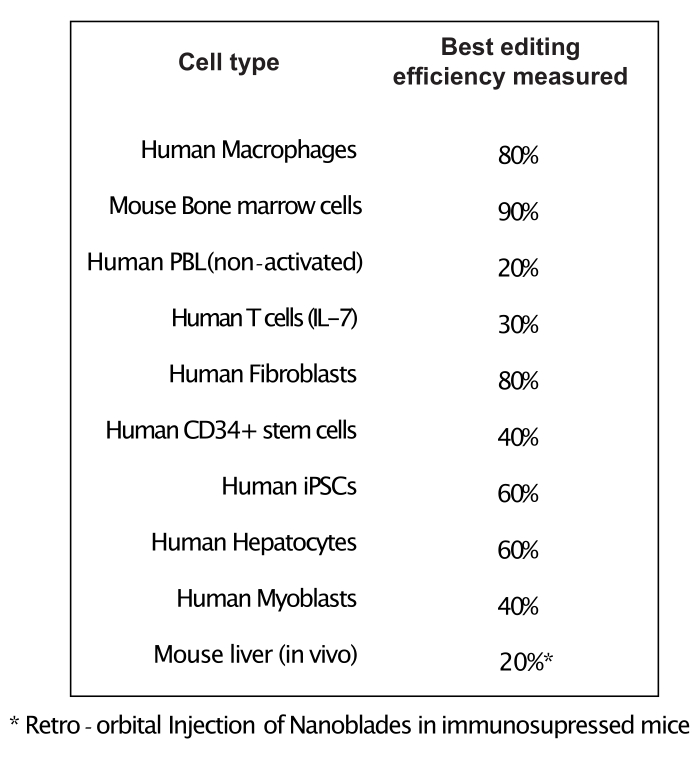
Abbildung 4: Bearbeitungseffizienz in verschiedenen Primärzelltypen mit Nanoblades. Abkürzungen: PBL = periphere Blutlymphozyten; IL = Interleukin; CD = Cluster der Differenzierung; iPSC = induzierte pluripotente Stammzelle. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Nanoblades ermöglichen eine schnelle und dosisabhängige Abgabe des Cas9/sgRNA-RNP-Komplexes in Zelllinien und Primärzellen. Im Gegensatz zur klassischen Transfektion und anderen viralen Abgabevektoren, aber wie die Proteinelektroporation, haben Nanoblades den Vorteil einer transienten Abgabe des Cas9/sgRNA RNP in transgenfreier Weise. Nanoblades bieten eine äußerst vielseitige, einfache und kostengünstige Plattform für die Proteinabgabe, die einfach und schnell an die ständig wachsende Familie der CRISPR-Varianten angepasst werden kann. Nanoblades können in der Zelllinie HEK293T oder deren Derivaten hergestellt werden. Die hier verwendeten HEK293T-Zelllinien wurden entwickelt, um die retrovirale und lentivirale Partikelproduktion zu maximieren (siehe Materialtabelle). Obwohl andere Quellen von HEK293T-Zellen geeignet sein können, müssen Benutzer HEK293T-Zellen aus verschiedenen Quellen testen und vergleichen, da je nach HEK293T-Zellquelle große Unterschiede in der Partikelproduktion beobachtet wurden. Die Zellen müssen auch häufig auf Mykoplasmenkontamination untersucht und alle drei Tage (klassisch 1/8 Verdünnung) durchquert werden, um einen Überfluss zu vermeiden, der sich negativ auf die Partikelproduktion auswirkt.
Zellen sollten nicht für mehr als 20 Passagen gehalten werden. DMEM, ergänzt mit Glukose, Penicillin / Streptomycin, Glutamin und 10% dekomplementiertem fötalem Rinderserum, wurde für die Zellkultur verwendet. Da die Serumherkunft die Qualität des Nanoblade-Präparats beeinträchtigen kann, sollten vor der Großproduktion verschiedene Serumchargen getestet werden. Nanoblades können effizient in anderen Medien wie RPMI oder serumfreien Modifikationen des minimal essentiellen Mediums hergestellt werden, die DMEM am Tag nach der Transfektion ersetzen können. Wie unten angegeben, kann es, obwohl der Mediumaustausch nach der Transfektion mit einigen DNA-Transfektionsreagenzien optional ist, vorteilhaft sein, das Medium, in das die VLPs freigesetzt werden, zu modifizieren, insbesondere um Serumspuren in der Partikelzubereitung zu begrenzen. Die Kultivierung von Zellen in reduziertem Serum-Minimum-essentiellem Medium am Tag vor der Transfektion wurde jedoch noch nicht versucht.
Wie bereits erwähnt, werden Nanoblades bei Überexpression einer Mischung von Plasmiden in Produzentenzellen hergestellt. Die Überexpression scheint für eine optimale Produktion erforderlich zu sein. Tatsächlich entwickelte dieses Labor eine Produzentenzelllinie, bei der das Gag-Pol-exprimierende Konstrukt durch Antibiotika-Selektion stabilisiert wurde; Dieses System produzierte jedoch keine signifikanten Mengen an Nanoblades. Eine ähnliche Beobachtung wurde gemacht, als das sgRNA-kodierende Konstrukt stabil in das Genom von Produzentenzellen integriert wurde. Wie für andere Partikelproduktionssysteme beschrieben, kann eine stabile Zelllinie, die zumindest einige Konstrukte exprimiert, die an der Nanoblades-Produktion beteiligt sind, nützlich sein; Dies würde jedoch sicherlich die Verarbeitung großer Mengen an Überstand und eine geeignete Technik zur Reinigung von Partikeln erfordern. Das obige Protokoll beschreibt das bevorzugte Verfahren zur Herstellung von Nanoblades, das spezifische Transfektionsreagenzien nutzt (siehe Materialtabelle).
Obwohl auch Transfektionsreagenzien anderer Hersteller mit Erfolg getestet wurden, folgt die überwiegende Mehrheit der Ergebnisse dieser Gruppe mit Nanoblades dem hier beschriebenen Verfahren. Eine kostengünstige Transfektion kann mit Calciumphosphatreagenzien erreicht werden und führt zu einer guten Produktionseffizienz; Diese Methode erfordert jedoch unbedingt den Austausch des Transfektionsmediums am Tag nach der Transfektion und kann Calciumphosphatrückstände in der sedimentierten Partikelzubereitung hinterlassen. Im Einklang mit der Notwendigkeit hoher Expressionsniveaus für Nanoblade-Komponenten in Produzentenzellen steht die Beobachtung, dass die Menge an sgRNAs, die mit dem Cas9-Protein assoziiert sind, ein limitierender Faktor für eine effiziente Genom-Editierung sein kann. Um die sgRNA-Beladung zu verbessern, wurden kürzlich zwei technische Ansätze von unabhängigen Gruppen entwickelt, die Proteinliefervektoren ähnlich nanoblades verwenden. Diese beruhen auf der Verwendung der T7-Polymerase-abhängigen zytoplasmatischen Expression von sgRNA6 oder durch die Zugabe eines retroviralen Verkapselungssignals an die sgRNA-Sequenz, um die Bindung an das Gag-Polyprotein6 zu vermitteln. Diese Ansätze könnten in der Tat die sgRNA-Beladung in Nanoblades verbessern, obwohl sie noch nicht getestet wurden.
Die Transduktion von Zielzellen ist ein kritischer Schritt im Verfahren. In den meisten immortalisierten Zelllinien hat die Transduktion mit Nanoblades wenig oder keine zytopathische Wirkung. In Primärzellen kann die Toxizität jedoch ein Problem darstellen. Die Transduktion muss daher für jeden Zelltyp optimiert werden. Insbesondere die Expositionszeit gegenüber Nanoblades ist ein wichtiger Faktor, der bei der Optimierung des Transduktionsprotokolls geändert werden muss. Für empfindliche Zellen wie primäre Neuronen oder Knochenmarkzellen ermöglichen 4-6 h Inkubation mit Nanoblades vor dem Ersetzen des Mediums eine effiziente Abgabe des Cas9-Proteins bei gleichzeitiger Minimierung der Zelltoxizität. Darüber hinaus können Adjuvantien wie unter anderem kationische Polymere die Effizienz der Transduktion in einigen Zellen signifikant verbessern (siehe Materialtabelle). Es ist wichtig zu beachten, dass Nanoblades VLPs sind und eine immunogene Reaktion induzieren können. Dies kann eine Einschränkung darstellen, wenn mit bestimmten Arten von Primärzellen wie Makrophagen oder dendritischen Zellen gearbeitet wird, bei denen die Inkubation mit Nanoblades wichtige Veränderungen der Genexpression und des Phänotyps der Zellen induzieren könnte. Wenn Makrophagen und dendritische Zellen aus hämatopoetischen Stammzellvorläufern (wie Knochenmarkzellen der Maus) gewonnen werden, ist es vorzuziehen, Zellen mit den Nanoblades zu transduzieren, bevor sie vollständig differenziert sind, um eine zelluläre Reaktion gegen die Nanoblades zu vermeiden. Andernfalls könnte die Cas9-Proteinelektroporation eine praktikable Alternative bei der Arbeit mit differenzierten Immunzellen darstellen.
Nanoblades können in vivo verwendet werden, um Mauszygoten oder Embryonen zu transgenen Tieren zu transduzieren. Ähnlich wie klassische retrovirale oder lentivirale Vektoren können sie auch direkt in Gewebe von erwachsenen Tieren injiziert werden. Nanoblades (ähnlich retroviralen und lentiviralen Vektoren) können jedoch durch die Immunantwort des Wirtstieres inaktiviert werden; Daher muss die zu injizierende Dosis für jede Anwendung optimiert werden. Diese Immunantwort kann auch die Verteilung von funktionellen VLPs auf Gewebe in der Nähe der Injektionsstelle begrenzen. Schließlich sind Nanoblades im Gegensatz zu lentiviralen Vektoren transgenfrei und liefern das Cas9 in einem begrenzten Zeitrahmen. Daher können sie nicht verwendet werden, um genomweite funktionelle Screenings durchzuführen, die eine Hochdurchsatzsequenzierung von sgRNAs bei der Auswahl von Zellen erfordern. Nanoblades sind nützlich, wenn eine schnelle, dosisabhängige und transgenfreie Genom-Editierung erforderlich ist20. Darüber hinaus führen Nanoblades ähnlich wie die Proteinelektroporation zu weniger Off-Target-Effekten als eine verlängerte Expression von Cas9/sgRNA durch DNA-Transfektion oder klassische virale Vektoren3. Die zukünftige Entwicklung von Nanoblades konzentriert sich auf die Integration von Cas9-Varianten für verschiedene technologische Anwendungen wie Base-Editing und RNA-Targeting.
Offenlegungen
Philippe E. Mangeot und Emiliano P. Ricci werden als Erfinder eines Patents im Zusammenhang mit der Nanoblades-Technologie genannt (Patentanmelder: Institut National de la Santé et de la Recherche Médicale (INSERM), Centre National de la Recherche Scientifique (CNRS), Ecole Normale Superieure de Lyon, Université Claude Bernard Lyon 1, Villeurbanne Cedex; Anmeldenummer: WO 2017/068077 Al; Patentstatus: veröffentlicht, 27. April 2017; alle Aspekte des Manuskripts sind durch die Patentanmeldung abgedeckt. Die übrigen Autoren erklären keine konkurrierenden Interessen.
Danksagungen
Diese Arbeit wurde von Labex Ecofect (ANR-11-LABX-0048) der Université de Lyon im Rahmen des Programms Investissements d'Avenir (ANR-11-IDEX-0007) der französischen Nationalen Forschungsagentur (ANR), der Fondation FINOVI, der Agence Nationale des Recherches sur le SIDA et les Hépatites Virales (ANRS-ECTZ3306) und des Europäischen Forschungsrats (ERC-StG-LS6-805500 to E.P.R.) im Rahmen der Forschungs- und Innovationsprogramme Horizon 2020 der Europäischen Union finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| 13.2 mL, Thinwall Polypropylene Tubes, 14 x 89 mm - 50Pk | Beckman Coulter Life Sciences | 331372 | Ultracentrifugation tubes for Nanoblades purification |
| Amersham Protran Premium Western blotting membranes, nitrocellulose | Merck | GE10600004 | Nitrocellulose membrane for quantifying Cas9 levels within purified Nanoblades |
| BIC-Gag-CAS9 | Addgene | 119942 | Encodes a GAG (F-MLV)-CAS9(sp) fusion. Allows the production of GAG-CAS9 Virus like particles from producer cells in association with over expressed gRNA(s) and appropriate envelopes |
| BICstim-Gag-dCAS9-VPR | Addgene | 120922 | Encodes a GAG-dCAS9-VPR fusion for targeted transcriptional activation |
| BLADE | Addgene | 134912 | Empty backbone for cloning sgRNA sequence to be used in Nanoblades system |
| BsmBI-v2 | New England Biolabs | R0739S | Restriction enzyme to digest the BLADE and SUPERBLADES vectors for sgRNA cloning |
| Cas9 (7A9-3A3) Mouse mAb (HRP Conjugate) #97982 | Cell Signaling Technology | 97982S | Anti-Cas9 antibody for Cas9 quantification by dot-blot |
| Cas9 Nuclease, S. pyogenes | New England Biolabs | M0386T | Recombinant Cas9 protein to be used as a reference for absolute quantification of the amount of Cas9 loaded within Nanoblades |
| Ethidium bromide solution (10 mg/mL in H2O) | Sigma-Aldrich | E1510-10ML | For staining agarose gels and visualize DNA |
| Fisherbrand Wave Motion Shakers | Fisher Scientific | 88-861-028 | Agitation table to resuspend Nanoblades upon centrifugation |
| gelAnalyzer | http://www.gelanalyzer.com; quantifying band intensity after digestion | ||
| Gesicle Producer 293T | Takara | 632617 | Nanoblades producer cell line |
| Gibco DMEM, high glucose, pyruvate | ThermoFisher Scientific | 41966052 | Cell culture medium for Gesicle Producer 293T cells |
| GoTaq G2 DNA Polymerase | Promega | M7848 | Taq polymerase for amplification of genomic DNA before T7 endonuclease assays |
| jetPRIME Transfection Reagent kit for DNA and DNA/siRNA | Polyplus | POL114-15 | Transfection reagent for Nanoblade production in Gesicle Producer 293T cells |
| Millex-AA, 0.80 µm, syringe filter | Millipore | SLAA033SS | Syringe filter to remove cellular debris before concentration of Nanoblades |
| Millex-GS, 0.22 µm, syringe filter | Millipore | SLGS033SS | Syringe filter to sterilise the sucrose cushion solution |
| Millex-HP, 0.45 µm, polyethersulfone, syringe filter | Millipore | SLHP033RS | Syringe filter to remove cellular debris before Nanoblades concentration |
| Monarch DNA Gel Extraction Kit | New England Biolabs | T1020L | DNA gel extraction kit for purification of the pBLADES or pSUPERBLADES plasmid fragment upon digestion with BsmBI |
| NEB Stable Competent E. coli (High Efficiency) | New England Biolabs | C3040I | Competent bacteria for plasmid transformation and amplification |
| NucleoBond Xtra Midi kit for transfection-grade plasmid DNA | Macherey-Nagel | 740410.50 | Maxipreparation kit for purification of plasmid DNA from cultured bacteria |
| Nucleospin gDNA extraction kit | Macherey-Nagel | 740952.50 | Extraction of genomic DNA from transduced cells |
| NucleoSpin Plasmid, Mini kit for plasmid DNA | Macherey-Nagel | 740588.50 | Minipreparation kit for purification of plasmid DNA from cultured bacteria |
| NucleoSpin Tissue, Mini kit for DNA from cells and tissue | Macherey-Nagel | 740952.5 | Genomic DNA extraction kit |
| Optima XE-90 | Beckman Coulter Life Sciences | A94471 | Ultracentrifuge |
| pBaEVRless | Els Verhoeyen (Inserm U1111) | Personnal requests have to be sent to: els.verhoyen@ens-lyon.fr | Baboon Endogenous retrovirus Rless glycoprotein described in Girard-Gagnepain, A. et al. Baboon envelope pseudotyped LVs outperform VSV-G-LVs for gene transfer into early-cytokine-stimulated and resting HSCs. Blood 124, 1221–1231 (2014) |
| pBS-CMV-gagpol | Addgene | 35614 | Enocdes the Murine Leukemia Virus gag and pol genes |
| pCMV-VSV-G | Addgene | 8454 | Envelope protein for producing lentiviral and MuLV retroviral particles |
| Phosphate-Buffered Saline (PBS) | ThermoFisher Scientific | 14200091 | 10X PBS to dilute in millipore water |
| Polybrene Transfection Reagent | Millipore Sigma | TR-1003-G | Cationic polymer that enhances the efficiency of retroviral transduction in specific mammalian cells. It can also allow viral-dependent entry of an Oligodeoxynucleotide (ODN) for homology-directed repair |
| Sucrose,for molecular biology, ≥99.5% (GC) | Sigma-Aldrich | S0389-5KG | Sucrose to prepare a cushion for Nanoblade purification through ultracentrifugation |
| SUPERBLADE5 | Addgene | 134913 | Empty backbone for cloning sgRNA sequence to be used in nanoblades system (Optimized for increased genome editing efficiency via Chen B et al., 2013) |
| SuperSignal West Dura Extended Duration Substrate | ThermoFisher Scientific | 34076 | Enhanced chemiluminescence (ECL) HRP substrate for Cas9 dot blots |
| SW 41 Ti Swinging-Bucket Rotor | Beckman Coulter Life Sciences | 331362 | Rotor for ultracentrifugation |
| SYBR Safe DNA Gel Stain | ThermoFisher Scientific | S33102 | Alternative to ethidium bromide for staining agarose gels and visualize DNA |
| T4 DNA Ligase | New England Biolabs | M0202S | DNA ligase to ligate the BLADE or SUPERBLADES vectors with the duplexed DNA oligos corresponding to the variable region of the sgRNA |
| T7 Endonuclease I | New England Biolabs | M0302S | T7 Endonuclease I recognizes and cleaves non-perfectly matched DNA. Allows to monitor the extent of genome editing at a specific locus |
| Triton-containing lysis buffer | Promega | E291A | Lysis buffer to disrupt Nanoblades and allow Cas9 quantification |
| TWEEN 20 | Sigma-Aldrich | P9416 | For the preparation of TBST |
Referenzen
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Zuris, J. A., et al. Cationic lipid-mediated delivery of proteins enables efficient protein-based genome-editing in vitro and in vivo. Nature Biotechnology. 33, 73-80 (2015).
- Mangeot, P. E., et al. Genome editing in primary cells and in vivo using viral-derived Nanoblades loaded with Cas9-sgRNA ribonucleoproteins. Nature Communications. 10 (1), 45 (2019).
- Cai, Y., Bak, R. O., Mikkelsen, J. G. Targeted genome editing by lentiviral protein transduction of zinc-finger and TAL-effector nucleases. eLife. 3, 01911 (2014).
- Choi, J. G., et al. Lentivirus pre-packed with Cas9 protein for safer gene editing. Gene Therapy. 23 (7), 627-633 (2016).
- Gee, P., et al. Extracellular nanovesicles for packaging of CRISPR-Cas9 protein and sgRNA to induce therapeutic exon skipping. Nature Communications. 11, 1334 (2020).
- Indikova, I., Indik, S. Highly efficient 'hit-and-run' genome editing with unconcentrated lentivectors carrying Vpr.Prot.Cas9 protein produced from RRE-containing transcripts. Nucleic Acids Research. 48 (14), 8178-8187 (2020).
- Lyu, P., Javidi-Parsijani, P., Atala, A., Lu, B. Delivering Cas9/sgRNA ribonucleoprotein (RNP) by lentiviral capsid-based bionanoparticles for efficient 'hit-and-run' genome editing. Nucleic Acids Research. 47 (17), 99 (2019).
- Gheysen, D., Jacobs, E., de Foresta, F., Thiriart, C. Assembly and release of HIV-1 precursor Pr55gag virus-like particles from recombinant baculovirus-infected insect cells. Cell. 59 (1), 103-112 (1989).
- Hanna, R. E., Doench, J. G. Design and analysis of CRISPR-Cas experiments. Nature Biotechnology. 38 (7), 813-823 (2020).
- Sambrook, J. . Molecular cloning a laboratory manual. Third edition. , (2001).
- Jiang, W., et al. An optimized method for high-titer lentivirus preparations without ultracentrifugation. Scientific Reports. 5, 13875 (2015).
- Rueden, C. T., et al. ImageJ2: ImageJ for the next generation of scientific image data. BMC Bioinformatics. 18 (1), 529 (2017).
- O'Doherty, U., Swiggard, W. J., Malim, M. H. Human immunodeficiency virus type 1 spinoculation enhances infection through virus binding. Journal of Virology. 74 (21), 10074-10080 (2000).
- Brinkman, E. K., Chen, T., Amendola, M., van Steensel, B. Easy quantitative assessment of genome editing by sequence trace decomposition. Nucleic Acids Research. 42 (22), 168 (2014).
- Bollen, Y., Post, J., Koo, B. -. K., Snippert, H. J. G. How to create state-of-the-art genetic model systems: strategies for optimal CRISPR-mediated genome editing. Nucleic Acids Research. 46 (13), 6435-6454 (2018).
- Brinkman, E. K., et al. Easy quantification of template-directed CRISPR/Cas9 editing. Nucleic Acids Research. 46 (10), 58 (2018).
- Dussaud, S., Pardanaud-Glavieux, C., Sauty-Colace, C., Ravassard, P. Lentiviral mediated production of transgenic mice: a simple and highly efficient method for direct study of founders. Journal of Visualized Experiments. (140), e57609 (2018).
- Montagna, C., et al. VSV-G-enveloped vesicles for traceless delivery of CRISPR-Cas9. Molecular Therapy. Nucleic Acids. 12, 453-462 (2018).
- Marnef, A., et al. A cohesin/HUSH- and LINC-dependent pathway controls ribosomal DNA double-strand break repair. Genes & Development. 33 (17-18), 1175-1190 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten