Method Article
Recopilación de datos microscópicos de iluminación crioestructurada de células preservadas criogénicamente
* Estos autores han contribuido por igual
En este artículo
Resumen
Este protocolo demuestra cómo obtener imágenes de muestras criopreservas biológicas utilizando microscopía de iluminación crioestructurada. Demostramos la metodología mediante la obtención de imágenes del citoesqueleto de las células U2OS.
Resumen
La microscopía de iluminación estructurada (SIM) tridimensional (3D) permite obtener imágenes de estructuras celulares etiquetadas fluorescentemente a una resolución más alta que la microscopía de fluorescencia convencional. Esta técnica de superresión (SR) permite la visualización de procesos moleculares en células enteras y tiene el potencial de ser utilizada junto con la microscopía electrónica y la tomografía de rayos X para correlacionar la información estructural y funcional. Recientemente se ha encargado un microscopio SIM para muestras criogénicamente preservadas (cryoSIM) en la línea de haz de crioimágenes correlativa B24 en el sincrotrón del Reino Unido.
Fue diseñado específicamente para imágenes 3D de muestras biológicas a temperaturas criogénicas de una manera compatible con imágenes posteriores de las mismas muestras mediante métodos de microscopía de rayos X, como la tomografía de rayos X criosumos. Este artículo de video proporciona métodos y protocolos detallados para obtener imágenes exitosas utilizando la crioSIM. Además de las instrucciones sobre el funcionamiento del microscopio crioSIM, se han incluido recomendaciones con respecto a la elección de muestras, fluoróforos y ajustes de parámetros. El protocolo se demuestra en muestras de células U2OS cuyas mitocondrias y tubulina han sido etiquetadas fluorescentemente.
Introducción
Las técnicas de imagen SR se han vuelto ampliamente accesibles para los biólogos en la última década1. Permiten imágenes de alta resolución de muestras marcadas fluorescentemente más allá del límite de difracción. Sin embargo, ha sido difícil adaptar los métodos de microscopía SR para trabajar con muestras a temperaturas criogénicas2. Esto sería ventajoso para las imágenes correlativas en combinación con la tomografía electrónica o de rayos X. Recientemente, SIM se ha adaptado para su uso con muestras criogénicas y se ha demostrado con éxito que permite estudios correlativos de células biológicas junto con la tomografía de rayos X suave (SXT)3 en la línea de haz de crio-imagen correlativa B24 en el Sincrotrón diamond light source (https://www.diamond.ac.uk/Instruments/Biological-Cryo-Imaging/B24.html). SIM puede duplicar la resolución de la microscopía convencional de campo amplio iluminando la muestra con patrones de luz rayados (flecos de Moiré) en tres ángulos y en cinco fases. La interferencia entre estos patrones de luz y la fluorescencia de la muestra se puede utilizar para descubrir computacionalmente información adicional sobre estructuras de subdiffracción4,5.
Hay varias ventajas de SIM sobre otras técnicas de SR para aplicaciones criogénicas. Primero, puede funcionar sin fluoróforos parpadeantes especialmente diseñados; se pueden utilizar fluoróforos convencionales, dando acceso a una gama más amplia de posibles agentes de marcado fluorescente6. Además, solo requiere 15 imágenes por rebanada z (en 3D; 9 imágenes para 2D), mientras que otros métodos SR toman aproximadamente 1000 imágenes por rebanada, lo que aumenta la posibilidad de que la muestra se caliente y, por lo tanto, aumenta el riesgo de formación de cristales de hielo, lo que puede causar artefactos. Finalmente, esta técnica puede obtener imágenes de muestras biológicas más gruesas de más de 10 μm, lo que permite obtener imágenes de células enteras en su estado casi nativo6. La crioSIM se ha construido utilizando componentes ópticos estándar y con software de acceso abierto para imágenes, lo que facilita la documentación y el duplicado si se desea6. La crioSIM tiene un objetivo de apertura numérica de 100x/0.9 (ver la Tabla de Materiales); Phillips et al.6 Aquí, este protocolo demuestra cómo usar el microscopio crioSIM, incluida la forma de cargar y descargar muestra muestras en la etapa criogénica, cómo recopilar datos en el microscopio y cómo reconstruir las imágenes SIM.
Protocolo
NOTA: Este protocolo se refiere a muestras que contienen células cultivadas o depositadas en rejillas planas de oro planas de 3 mm de microscopía electrónica de transmisión (TEM) con una película de soporte de carbono perforada que han sido vitrificadas por congelación por inmersión o congelación a alta presión. Este protocolo asume que las muestras ya han sido fotografiados utilizando un microscopio convencional de epifluorescencia y campo brillante para mapear ubicaciones de interés para la obtención de imágenes en crioSIM. Consulte la Figura 1 para obtener información general sobre todo el protocolo.
1. Preparación de la crioescenda
- Prepare nitrógeno líquido sin hielo (LN2)pasando LN2 a través de un embudo forrado con cualquier toallita de papel seco científico estándar.
NOTA: Como LN2 causa quemaduras, use el equipo de protección personal adecuado, incluidos guantes y gafas crioprotectores, cuando lo manipule. Tales líquidos pueden desplazar el oxígeno y causar asfixia rápida y deben manipularse en un área adecuadamente ventilada. - Retire el polvo de la superficie de la etapa crio-stage con aire presurizado. Expulse el líquido del recipiente de aire presurizado antes de usarlo.
- Asegúrese de que el cartucho de carga de muestras esté en su lugar con el soporte de muestras con cámara apropiado (verifique que no quede ninguna rejilla en el soporte de muestras de un experimento anterior) (Figura 2).
- Retire la tapa del dewar externo de la etapa crio-etapa y vierta LN2 filtrado hasta aproximadamente 1/4de lleno. Espere hasta que la ebullición inicial disminuya antes de verter más; llene el recipiente a aproximadamente 2/3de lleno. Reemplace la tapa con cuidado, apuntando la boquilla lejos del manipulador y la etapa / óptica mientras LN2 hierve desde la salida.
- Una vez que LN2 haya dejado de salir de la salida, coloque la tubería de salida sobre el escenario dewar en el crioescénico.
- Conecte la fuente de alimentación del escenario y conecte el cable USB al crioescéfalo. Asegúrese de que la tapa de la cámara de muestras calentada esté enchufada. Conecte el dewar externo al escenario.
NOTA: No enchufe el dewar externo hasta que el tubo de salida se haya colocado sobre el dewar de etapa en el escenario crioescépico. Si LN2 se desborda (o para evitar que se desborde), extraiga el cable USB durante ~ 10 s, permita que se equilibre y vuelva a conectar el cable USB para reactivar el sensor. - Después de que LN2 se haya entregado en el dewar de etapa, presione el botón de liberación en la etapa crio-etapa para permitir que LN2 ingrese a la cámara de muestra.
NOTA: No deje el sistema desatendido mientras LN2 está llenando la cámara. - Espere ~ 30-45 minutos para permitir que el sistema se enfríe y estabilice antes de comenzar la adquisición de imágenes. Revise periódicamente el dewar externo y rellene con LN2 filtrado si está menos de un cuarto lleno (aproximadamente cada hora).
2. Transferencia de la caja de almacenamiento de muestras a la etapa crio-stage
NOTA: Sumerja la caja de almacenamiento de muestras, el soporte y las puntas de cualquier instrumento (por ejemplo, fórceps) dentro del LN2 filtrado para enfriarlos antes de tocar cualquier superficie fría como la muestra o cualquier objeto dentro de la cámara de muestra. Use una bata de laboratorio y guantes cuando manipule muestras biológicas.
- Asegúrese de que las muestras vitrificadas estén en un recipiente criocompatible y llévalas al microscopio. Pulse el botón correspondiente en el crioescépico para encender la luz de la cámara de muestra.
- Utilice la llave hexagonales de la herramienta de casete para abrir las dos placas del casete de transferencia de muestras. Abra las placas lo suficientemente anchas como para caer en la rejilla entre las dos placas, pero evite abrirse a la posición de apertura máxima para evitar que la rejilla caiga por el otro lado.
- Use forrórceps largos para levantar la caja de rejilla de muestra del LN2,gire donde la muesca se alinea con la posición de la posición de almacenamiento dentro del escenario y colóquela en el escenario. Utilice el dispositivo adecuado (por ejemplo, un destornillador) para abrir la tapa de la caja de almacenamiento a la posición correcta de la muestra.
- Usando fórceps invertidos (o cualquier fórceps quirúrgico de punta fina), retire la rejilla TEM del soporte de la muestra, sumérjala dentro del LN2y colóquela en su posición en el cassette de transferencia de muestras, manteniéndose cerca de la LN2 durante el proceso de transferencia. Asegúrese de que el lado de la película de carbono esté colocado de modo que finalmente se enfrente al objetivo en el puente de muestra.
- Cierre el cartucho de muestra con la tecla hexagonl de la herramienta de casete. Cierre y retire la caja de almacenamiento junto con las muestras restantes.
- Utilice el punto magnético de la herramienta de cassette para levantar y montar el cartucho que contiene la rejilla en el puente de muestra. Manténgalo inmerso/cerca del LN2 durante el proceso de transferencia. Asegúrese de que la orientación sea adecuada para el puente (los dos imanes harán contacto con los imanes en el puente). Coloque el cassette plano dentro de los pasadores de posicionamiento del puente y empuje suavemente para asegurarse de que esté fijo.
NOTA: Un casete de muestra para rejillas recortadas que se han preparado para el posterior fresado de vigas de iones enfocados también está disponible en las instalaciones de CryoSIM en beamline B24.
3. Acoplamiento y enfoque del escenario
- Mueva la abertura de la tapa de la etapa crio-etapa a la posición de imagen y apague la luz de la cámara de muestra. Desliza el escenario hacia la óptica para alinearlo bajo la lente del objetivo. Coloque suavemente el objetivo en su posición con la palanca, asegurándose de que descanse dentro de la tapa de la etapa crio-etapa, pero no lo toque.
NOTA: Asegúrese de que la tubería de salida externa de Dewar no toque el dewar de etapa en la etapa crio-etapa en ningún momento durante la recopilación de datos. - Cubre el escenario y la óptica con una cortina negra opaca. Inicie el software de control Cockpit en el PC crioSIM(Figura 3 y Figura 4).
- Haga clic en el botón de modo de lectura para cada cámara y configréelo en CONV 3 MHz. Compruebe que la temperatura de cada cámara es de -80 °C y que el ventilador de la cámara está apagado.
- Encienda la cámara reflejada. En Luces,elija ambiente (exposición 20 ms), y en linkam,haga clic en condensador. Haga clic en el botón Modo de video.
- En la ventana Vista de mosaico, alejo (desplazamiento del ratón) para ver el contorno de la cuadrícula. Haga clic en Buscar etapa si no se puede ver. Centra la cuadrícula haciendo doble clic izquierdo en el centro del círculo.
- Enfoque la muestra hasta que la película de soporte de rejilla (o cualquier otra característica de muestra relevante) esté enfocada, usando las teclas arriba y abajo para mover la crioescépda hacia arriba y hacia abajo, y usando las teclas 9 y 3 en el teclado numérico para cambiar el paso z (configúralo a 20 μm para el enfoque inicial).
NOTA: Si el usuario se queda sin recorrido en z, la etapa se puede mover manualmente hacia arriba o hacia abajo usando la rueda debajo de la etapa crio- . Gire una muesca a la vez y verifique si la muestra está dentro del rango de visión. Altera la dirección de nuevo si el enfoque empeora.
4. Adquisición de mosaico Brightfield
- Una vez que el escenario esté centrado, desactive el modo de videoy recoja un mosaico de luz visible (haga clic en Ejecutar mosaico en la vista Mosaico para producir mosaicos de imágenes de luz visible que se espiralicen hacia afuera desde el centro). Si la cuadrícula está doblada, pruebe diferentes posiciones en la cuadrícula (haga doble clic izquierdo en la vista Mosaico),vuelva a enfocar y haga clic en Ejecutar mosaico nuevamente para recopilar mosaicos parciales. Alternativamente, coloque una imagen enfocada en la parte superior del mosaico(Figura 3).
- Guarde la vista haciendo clic en Guardar mosaico. Así que le dé un nombre de archivo corto que contenga información sobre el cuadro de almacenamiento y el número de cuadrícula respectivo (se anexará automáticamente una marca de tiempo al nombre del archivo).
5. Identificación de áreas de interés
- Inspeccione el mosaico de campo brillante junto con cualquier imagen de "mapa" de fluorescencia anterior para ver dónde se han ubicado previamente las células o las características biológicas de interés. Compruebe si esas áreas producen fluorescencia adecuada.
- Apague la luz ambiental y el condensador, así como el modo de video si está activo. Encienda el láser de excitación requerido (405, 488, 561 o 647) y elija la cámara y el filtrocorrespondientes, inicialmente a 50 mW, para un tiempo de exposición de 50 ms.
NOTA: Aumente/disminuya estos ajustes de cámara y filtro en función de la señal de fluorescencia. - Pulse 0 para ajustar una imagen y * para contrastar automáticamente. Alternativamente, ajuste manualmente el contraste utilizando el control deslizante en la parte inferior de la imagen.
NOTA: Encienda el letrero de encendido láser para la habitación. - Una vez que se hayan encontrado células biológicamente interesantes con fluorescencia adecuada, marque sus posiciones usando el botón Marcar sitio en la vista Mosaico.
NOTA: Estos sitios marcados aparecerán en la lista adjunta y se puede acceder haciendo doble clic en las coordenadas. Cuando regrese a un sitio marcado, haga zoom en el área antes de hacer doble clic. - Continúe marcando todos los sitios potenciales antes de comenzar la adquisición de imágenes. Vuelva a guardar el mosaico con los sitios marcados; haga clic en Guardar sitios en archivo.
- Para unir las imágenes de mosaico utilizando el software StitchM (desarrollado internamente en beamline B24), arrastre y suelte el archivo de mosaico .txt en el archivo StitchM con extensión .bat y guarde la imagen tiff combinada de los mosaicos en la misma carpeta. Para guardar una imagen con los sitios marcados, arrastre y suelte el mosaico.txt archivo y el archivo de marcadores.txt en el icono al mismo tiempo.
NOTA: Equilibre la recopilación de datos con las necesidades del proyecto (por ejemplo, si se realizarán imágenes correlativas, verifique cuántas imágenes se pueden tomar con la modalidad de imágenes asociadas y elija el número apropiado de sitios para la imagen crioSIM). Además, si el dewar externo requiere rellenarse con LN2, el sistema de crio-etapa cambiará de posición y es probable que los sitios marcados no regresen a los mismos lugares; por lo tanto, tenga esto en cuenta al elegir el número de sitios que se desea obtener una imagen.
- Para unir las imágenes de mosaico utilizando el software StitchM (desarrollado internamente en beamline B24), arrastre y suelte el archivo de mosaico .txt en el archivo StitchM con extensión .bat y guarde la imagen tiff combinada de los mosaicos en la misma carpeta. Para guardar una imagen con los sitios marcados, arrastre y suelte el mosaico.txt archivo y el archivo de marcadores.txt en el icono al mismo tiempo.
- Apague la luz ambiental y el condensador, así como el modo de video si está activo. Encienda el láser de excitación requerido (405, 488, 561 o 647) y elija la cámara y el filtrocorrespondientes, inicialmente a 50 mW, para un tiempo de exposición de 50 ms.
6. Estrategia de recopilación de datos
- Establezca el tiempo de exposición del láser en función de los recuentos en el rango dinámico en la imagen de fluorescencia (en la parte inferior de la ventana de vistas de la cámara). Elija qué filtro aplicar y optimice estos ajustes para cada longitud de onda de luz de excitación que se utilizará, encendiendo cada láser por separado.
NOTA: Aunque los recuentos de 10,000-20,000 son óptimos, los recuentos más bajos son aceptables si hay un buen contraste. Idealmente, verifique la configuración del filtro antes de adquirir cada pila de imágenes porque las celdas en diferentes áreas de la cuadrícula podrían tener niveles de fluorescencia variables.
7. Recopilación de datos
- Haga clic en ambas cámaras para encenderlas. Regrese a uno de los sitios marcados y concéntrese nuevamente en la profundidad deseada. Una vez enfocado en un área de interés, desenfocarse hacia arriba (↑ tecla) en la ventana XY en la Etapa Macro para elegir la altura de la pila z a adquirir, y haga clic en Guardar arriba. Muévase fuera de foco hacia abajo (tecla ↓ ), haga clic en Guardar abajo y luego en Ir al centro, y verifique que la imagen todavía esté enfocada.
NOTA: La altura de la muestra se mostrará en la ventana. - Haga clic con el botón derecho en slm (modulador de luz espacial) en la ventana Cockpit y asegúrese de que el ángulo esté establecido en 0.41. En la ventana Cabina, seleccione Experimento de un solo sitio.
NOTA: No haga clic en el slm en; La cabina lo encenderá automáticamente durante la adquisición de imágenes.- En la lista desplegable, seleccione Iluminación estructurada. Modifique la altura de la pila para que sea igual a la altura z + 1 μm.
NOTA: La adición de 1 μm asegura la captura de toda la muestra en z y minimiza los artefactos de reconstrucción. - Introduzca los tiempos de exposición (ms) para los láseres de 405 nm y 488 nm en la fila superior (para la cámara reflejada) y los tiempos de exposición para los láseres de 561 nm y 647 nm en la fila inferior (para la cámara transmitida).
NOTA: Los valores de tiempo de exposición deben coincidir con los valores decididos anteriormente y mostrados en la ventana principal de cockpit. - Ingrese un nombre de archivo (convención de nomenclatura: cuadro number_grid area_filters_FL) (FL (fluorescencia) o BF (campo brillante) dependiendo del tipo de imagen que se esté realizando). Haga clic en Actualizar para producir un nuevo archivo que contenga la fecha y la hora sin sobrescribir los archivos anteriores. Luego, haga clic en Inicio.
- En la lista desplegable, seleccione Iluminación estructurada. Modifique la altura de la pila para que sea igual a la altura z + 1 μm.
- Compruebe la vista de la cámara mientras se recopilan los datos. Vuelva a tomar las imágenes si hay algún desplazamiento xy. Si el LN2 dewar rellena la etapa crio-stage durante la adquisición de la imagen, aborte el proceso haciendo clic en el botón Abort en el software Cockpit.
- Una vez que el dewar externo haya terminado de rellenar el dewar de etapa, repita el experimento porque la recarga desplaza la muestra verticalmente. Vuelva a enfocar la imagen para volver a centrarla en z y repita el experimento de sitio único. Repita el experimento de un solo sitio para todas las combinaciones de láseres y filtros necesarios.
NOTA: Se tarda aproximadamente 30 s-1 min en terminar de rellenar. Durante la toma de imágenes de una rejilla, el relleno ocurrirá ~ 4-8x.
- Una vez que el dewar externo haya terminado de rellenar el dewar de etapa, repita el experimento porque la recarga desplaza la muestra verticalmente. Vuelva a enfocar la imagen para volver a centrarla en z y repita el experimento de sitio único. Repita el experimento de un solo sitio para todas las combinaciones de láseres y filtros necesarios.
- En cada posición, recoge una pila z con luz visible.
- Apague los láseres y encienda la luz ambiental y el condensador. En Experimento de sitio único,seleccione Pila Z,establezca la luz ambiental en 20 ms de exposición y mantenga la altura z como se indica arriba.
- Repita los pasos 7.2 a 7.4 para todos los sitios marcados en la cuadrícula.
- Antes de pasar a otra muestra, elimine el mosaico haciendo clic en Eliminar iconos.
- Dibuja un cuadrado alrededor de todos los mosaicos para eliminarlos. Elimine los marcadores también seleccionándolos todos en la lista y luego haciendo clic en Eliminar sitios seleccionados.
- Apague la luz ambiental y el condensador antes de proceder a cambiar la rejilla. Siga los pasos de la sección 4 en orden inverso para desacoplar el escenario de debajo del objetivo y cambiar la cuadrícula.
8. Después de la obtención de imágenes
- Una vez finalizada la imagen, desacople el escenario y retire todas las muestras. Apague la luz de la cámara de muestra. Desenchufe el dewar externo y decante cualquier LN2 restante en otro recipiente criocompatible, lo que permite que el dewar vuelva de forma segura a una temperatura normal.
- Coloque el tapón de la tapa en el crioescéfalo. Espere hasta que la opción de hornear la pantalla de la etapa crio-etapa esté disponible después de que no quede más LN2 en la etapa dewar. Presione el botón de horneado para ingresar al modo de calentamiento y elimine la humedad de la etapa crio-stage para evitar la formación de hielo.
9. Reconstrucción
- Transfiera archivos de datos SIM sin procesar a la estación de trabajo adecuada para la reconstrucción. Ejecute el procesamiento en lotes a través de la ventana del generador de tareas de procesamiento utilizando funciones de transferencia óptica específicas del canal (OTFS) (calculadas a partir de funciones de dispersión de puntos de perlas multiconfesantes) y ángulos K0 (0.29278, -1.8028, 2.3786) con un filtro Wiener constante para todos los canales de 0.004 y un desplazamiento de sesgo de 200.
- En el panel Opciones adicionales, asegúrese de que se descartan las intensidades negativas manteniendo otras opciones sin marcar. Guarde las imágenes SIR en una carpeta denominada "processed".
NOTA: El software comercial de reconstrucción de SIM generalmente produce datos SIM reconstruidos y conserva el nombre del archivo, pero agrega SIR.dv al final. También se crea un archivo de registro que contiene el protocolo de procesamiento, los pasos y la información estadística sobre el éxito de la reconstrucción.
10. Corrección de desplazamiento cromático
- Descargue el software Chromagnon7 para corregir el desplazamiento cromático.
- Utilice la matriz de referencia de desplazamiento cromático que corresponde a las longitudes de onda de fluorescencia de los datos recopilados (proporcionados por la línea de haz).
NOTA: El archivo de referencia es un archivo 'chromagnon.csv', que contiene los parámetros de alineación y se ha obtenido a partir de imágenes de calibración utilizando nanopartículas multi-fluorescentes. Se puede utilizar para procesar por lotes varios conjuntos de datos a la vez.- Elija el archivo de referencia apropiado que coincida con la longitud de onda del láser y el filtro utilizado para obtener imágenes de la muestra, y agréguelo al campo de referencia. Agregue los datos SIR reconstruidos para alinearlos en el campo de origen y haga clic en Ejecutar.
- Compruebe que la señal de fluorescencia ahora está alineada en las imágenes. Para el procesamiento por lotes, coloque todas las imágenes SIR que se alinean en el campo de origen y el archivo de referencia en el campo de referencia, y presione Ejecutar todo.
Resultados
Una muestra que contenía células U2OS se tiñó con una mezcla de colorante de citoesqueleto de microtúbulos verdes y tinte de mitocondrias rojas, lo que resultó en la tinción del componente de microtúbulos del citoesqueleto (verde) y las mitocondrias (rojo). Las imágenes posteriores mostraron la localización de las mitocondrias dentro de la célula, así como la disposición de los microtúbulos, destacando el marco estructural que proporcionan a la célula y el ensamblaje del citoesqueleto alrededor de orgánulos como el núcleo. La resolución en crioSIM es significativamente mayor que en la microscopía de epifluorescencia estándar (Figura 5). La Figura 6 muestra cómo el "mapa" fluorescente de un microscopio de epifluorescencia convencional se puede utilizar para localizar áreas de interés para la obtención de imágenes y la imagen correspondiente reconstruida por crioSIM desde una ubicación en la cuadrícula.
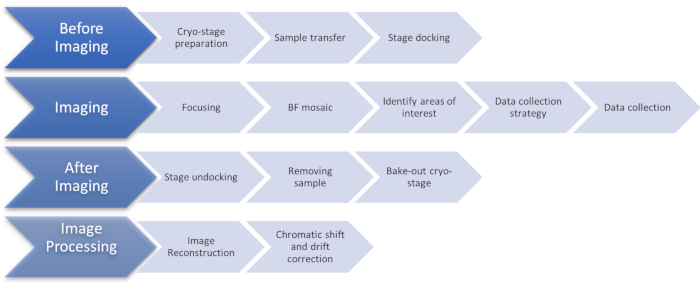
Figura 1: Diagrama de flujo que muestra las etapas del protocolo de imágenes cryoSIM. Abreviatura: cryoSIM = Microscopía de iluminación crioestructurada. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: La etapa crio- (A) La configuración de la crioescé etapa. (B) Una cuadrícula de muestra que se muestra sostenida por fórceps invertidos. (C) Componentes de la crio-etapa. Los puertos de conexión están etiquetados, con los colores correspondientes al naranja: fuente de alimentación, amarillo: tapa de escenario calentada, azul: dewar externo, verde: conexión a PC. Abreviaturas: PC = computadora personal; LN2 = nitrógeno líquido. Haga clic aquí para ver una versión más grande de esta figura.
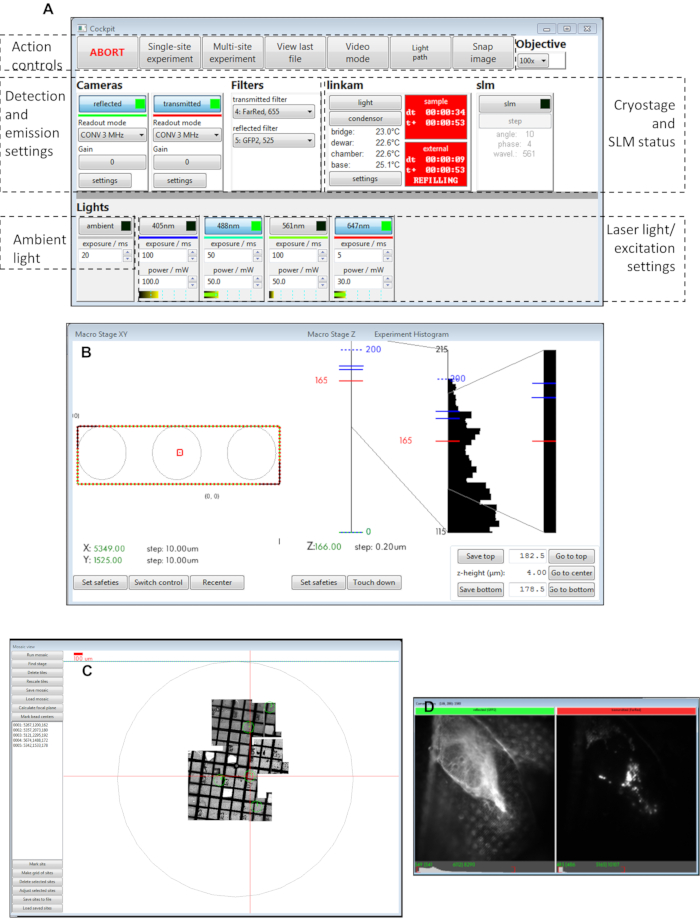
Figura 3: Vistas de los paneles de software de la cabina. (A) Panel principal, (B) macro etapa XY, (C) vista de mosaico, (D) vistas de cámara. Abreviatura: SLM = modulador de luz espacial. Haga clic aquí para ver una versión más grande de esta figura.
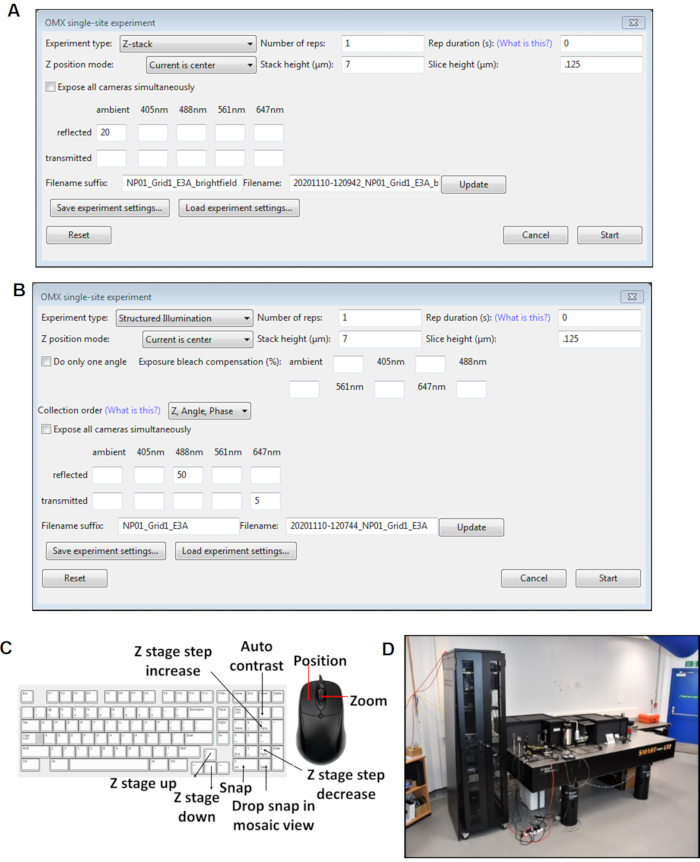
Figura 4: Vistas de los paneles de software Cockpit. (A) Experimento de un solo sitio de pila Z. (B) Experimento de sitio único si. (C) Métodos abreviados de teclado para el software de cabina utilizado durante la adquisición de imágenes. (D) El microscopio crioSIM está in situ en la línea de haz B24 en el sincrotrón Diamond Light Source. Abreviatura: cryoSIM = Microscopía de iluminación crioestructurada. Haga clic aquí para ver una versión más grande de esta figura.
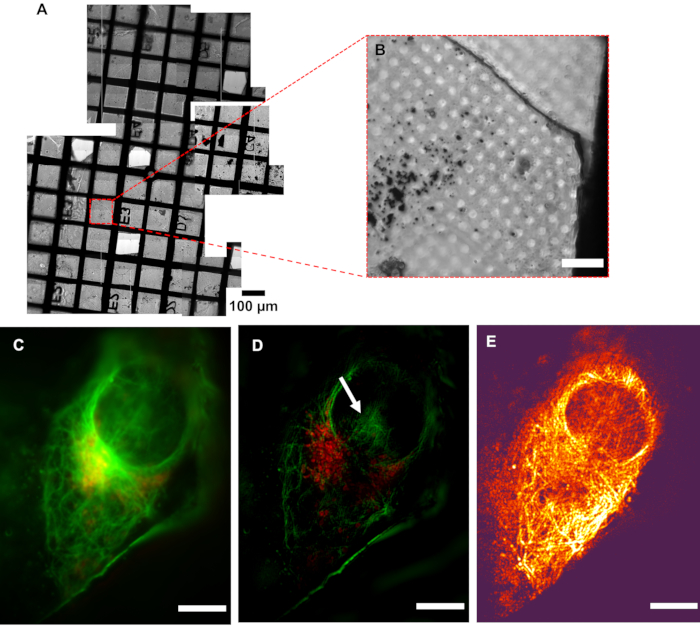
Figura 5: Resolución de la crioSIM. (A) Vista en mosaico de una cuadrícula bajo examen. (B) Imagen de campo brillante de un área de interés (AOI). (C) Imagen pseudo-widefield en comparación con su imagen SIM (D) que muestra el aumento de la resolución. La flecha blanca indica los artefactos de reconstrucción de SIM. (E) Mapa de contraste de modulación que combina la información de intensidad de píxeles de la imagen reconstruida con la información de color de los respectivos valores de relación contraste-ruido de modulación (MCNR) de los datos brutos generados por SIMCheck2. Las regiones brillantes y oscuras muestran un contraste alto y bajo, respectivamente. Barra de escala = 10 μm. Ajustes de imagen cryoSIM: longitudes de onda de excitación/emisión: 488/525 nm, potencia láser de 50 mW, tiempo de exposición de 50 ms y 647/655 nm, potencia láser de 20 mW, tiempo de exposición de 5 ms. Abreviatura: cryoSIM = Microscopía de iluminación crioestructurada. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Reconstrucción de imágenes en crioSIM desde una ubicación en la cuadrícula en un mapa de fluorescencia a partir de un mapa de epifluorescencia convencional. (A, B) Superposición de mapas de imágenes de campo brillante y fluorescencia generados con un microscopio de epifluorescencia convencional. Este mapa se utiliza para localizar regiones de interés para posteriormente obtener una imagen en crioSIM. (C) La imagen crioSIM reconstruida obtenida en la ubicación que se muestra en (B). Abreviatura: cryoSIM = Microscopía de iluminación crioestructurada. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
La SIM 3D a temperaturas criogénicas tiene muchas ventajas sobre otras técnicas de imagen SR para obtener imágenes de material biológico vitrificado. Requiere significativamente menos imágenes por rebanada z en comparación con otros métodos de SR, lo que resulta en menos irradiación y una menor probabilidad de formación de cristales de hielo para muestras vitrificadas. También es capaz de obtener imágenes de células enteras y se puede correlacionar con la tomografía de rayos X para hacer coincidir la estructura con la función. Curiosamente, la mayoría de los fluoróforos y etiquetas de fluorescencia disponibles comercialmente se blanquean menos en condiciones criogénicas que a temperatura ambiente. Sin embargo, dado el alto rendimiento cuántico de los fluoróforos más comunes a temperatura ambiente (más del 80% en algunos casos), la ganancia absoluta detectada en los fotones no se debe a cambios en el rendimiento cuántico, sino a una reducción en los complejos procesos de blanqueo. Más información sobre el rendimiento de los fluoróforos a temperaturas criogénicas se puede encontrar en 8.
Es fundamental que las muestras que llegan a la crioSIM hayan sido preaplicadas utilizando un microscopio de criofluorescencia convencional con capacidad de campo brillante para producir un "mapa" de cuadrícula que incluya el resaltado de todas las AOI potenciales para obtener imágenes adicionales(Figura 5). El tiempo de acceso en la crioSIM se asigna a través de un proceso competitivo que implica la presentación de una propuesta, que posteriormente se evalúa para determinar la viabilidad de la técnica y el impacto biomédico. El tiempo en el equipo, por lo tanto, siempre es "escaso", y las redes premaptadas permiten el uso más eficiente de una asignación. También es esencial que la muestra se mantenga vitrificada, especialmente durante la transferencia de la muestra desde el soporte de la muestra a la plataforma de imágenes, para minimizar la formación de cristales de hielo y el daño posterior de la muestra. La muestra debe ser de buena calidad para producir las mejores imágenes SIM. Una muestra bien preparada se caracterizará por las siguientes características: (a) no tendrá contaminación por cristal de hielo, (b) la rejilla utilizada será una rejilla de búsqueda, (c) el portador será plano, (d) la malla de la rejilla y la superficie del sustrato no serán auto-fluorescentes, y (e) no habrá roturas en la membrana de soporte. Estos requisitos previos se pueden lograr mediante un manejo cuidadoso de las muestras y asegurando que las muestras siempre permanezcan vitrificadas.
Es importante verificar de antemano si los fluoróforos de muestra propuestos darán suficiente señal en el microscopio crioSIM. Herramientas como SPEKCheck9 pueden ayudar a elegir las combinaciones óptimas de fluoróforos y filtros. Si hay problemas con la recopilación de datos sin procesar o el proceso de reconstrucción, los artefactos pueden aparecer en las imágenes después de la reconstrucción. Ejemplos de varios artefactos han sido documentados por Demmerle et al.10 Los parámetros de reconstrucción de imagen se pueden revisar en el archivo de registro de SoftWoRx si la reconstrucción no es óptima abriendo el archivo de resumen de reconstrucción. Debe haber un interlineado consistente a través de ángulos en un canal dado y una amplitud relativamente consistente. La variación de más del 30% y los valores significativamente superiores a 1 (si se aplica la compensación del tamaño de la perla) deben investigarse más de cerca y es probable que indiquen reconstrucciones fallidas. Además, el software SIMcheck2 en Fiji también se puede utilizar para realizar varias comprobaciones de los datos en bruto y reconstruidos para diagnosticar la causa de los errores en la configuración de los parámetros de imagen o reconstrucción. SIM-check y su mapa de contraste de modulación también pueden ayudar en la evaluación de la calidad de los datos reconstruidos al interpretar qué áreas de una imagen probablemente sean estructuras reales frente a artefactos.
El bajo contraste de modulación (mostrado por el color oscuro, en la Figura 5E)dentro del área nuclear significa que esta región va a ser más susceptible a los artefactos de reconstrucción, lo que implica que los patrones hash mostrados en el núcleo podrían clasificarse(Figura 5D)como un artefacto. Es más probable que las áreas de señal de fluorescencia fuertes reflejen con precisión las estructuras nativas en los datos procesados. En áreas de señal débil donde los fluoróforos se distribuyen en áreas más amplias, como la superficie total de una vesícula, es probable que la señal real coexista con los artefactos de procesamiento, y se debe tener cuidado en la interpretación de esos datos. Después de la inspección de los datos reconstruidos de rango completo para garantizar que no haya artefactos extraños, y que el fondo sea generalmente gaussiano y centrado cerca de cero, los datos generalmente se recortan a cero, o el valor modal, el pico de la señal de fondo, debería estar muy cerca de cero. Esto garantiza que el rango dinámico de la imagen mostrada no esté dominado por artefactos de fondo negativos. Cuando se espera una señal más débil, se debe tener especial cuidado al analizar las características y asegurarse de que sean estructuras reales en lugar de artefactos de reconstrucción.
Existen algunas limitaciones del sistema de imágenes. Debido a que la etapa de muestra es plana, las muestras con espesor variable o rejillas que no son planas no son sujetos ideales para la obtención de imágenes. Además, si las imágenes correlativas se realizarán utilizando tomografía de rayos X suave, las células cerca de los límites de la cuadrícula no deben ser fotografiadas, ya que no serán visibles en el microscopio de rayos X durante la adquisición de la serie de inclinación. Finalmente, la cantidad de borrado de la muestra antes de la congelación por inmersión tiene un impacto significativo en la calidad de la imagen; muy poca sección da como resultado muestras demasiado gruesas, lo que proporciona imágenes SIM subóptimas con un alto ruido de fondo, mientras que demasiada secreción puede hacer que las células se desforman y, por lo tanto, sean más susceptibles al daño por calor del rayo láser incidente. En resumen, cryoSIM es una poderosa herramienta de microscopía de fluorescencia para obtener imágenes de muestras biológicas en 3D en una etapa casi nativa y tiene una amplia gama de aplicaciones en muchas áreas.
Divulgaciones
Los autores no declaran intereses financieros contrapuestos.
Agradecimientos
Este proyecto ha recibido financiación del proyecto iNEXT-Discovery Horizonte 2020 de la Comisión Europea. I.M. Dobbie reconoce la financiación del Wellcome Trust (107457/Z/15/Z). Este trabajo se llevó a cabo con el apoyo de la Fuente de Luz Diamante, instrumento B24 (propuesta BI25512). Nuestro agradecimiento al personal de Micron y a todos nuestros excelentes usuarios y colaboradores por ayudarnos a establecer la crioSIM y su potencial correlativo.
Materiales
| Name | Company | Catalog Number | Comments |
| Auto grids | FEI | ||
| Autogrids | Thermo Fisher Scientific | 1036173 | |
| BioTracker 488 Green Microtubule Cytoskeleton Dye | Sigma-Aldrich | SCT142 | |
| Cockpit Software | Oxford University | https://github.com/MicronOxford/cockpit | |
| cryo compatible polyurethane container | Jena bioscience | CC-FD-800 | |
| Cryo TEM grid storage box | Thermo Fisher Scientific | Model#AutoGrid | |
| cryo-SIM microscope | Custom made | N/A | Custom made, see following reference for the design: Michael A. Phillips, Maria Harkiolaki, David Miguel Susano Pinto, Richard M. Parton, Ana Palanca, Manuel Garcia-Moreno, Ilias Kounatidis, John W. Sedat, David I. Stuart, Alfredo Castello, Martin J. Booth, Ilan Davis, and Ian M. Dobbie, "CryoSIM: super-resolution 3D structured illumination cryogenic fluorescence microscopy for correlated ultrastructural imaging," Optica 7, 802-812 (2020). Has a Nikon TU Plan Apo 100x/0.9 NA. |
| Cryostage system | Linkam Scientific Instruments | CMS196 | |
| Fine tip surgical forceps | Ted Pella | 38125 | |
| MitoTracker Deep Red FM | Thermo Fisher Scientific | M22426 | |
| Python Microscope Software | Oxford University | https://www.python-microscope.org/ | |
| Scientific dry paper wipes | Kimberly-Clark 7551 | 2 | |
| SIM Reconstruction Software | softWoRx, GE Healthcare | Version 6.5.2 | |
| StitchM Software | Diamond Light Source | https://github.com/DiamondLightSource/StitchM | |
| TEM grids for samples | Quantifoil Micro Tools | G200F1 |
Referencias
- Schermelleh, L., et al. Super-resolution microscopy demystified. Nature Cell Biology. 21 (1), 72-84 (2019).
- Ball, G., Demmerle, J., Kaufmann, R., Davis, I., Dobbie, I. M., Schermelleh, L. SIMcheck: A toolbox for successful super-resolution structured illumination microscopy. Scientific Reports. 5 (1), 15915 (2015).
- Kounatidis, I., et al. 3D Correlative cryo-structured illumination fluorescence and soft X-ray microscopy elucidates reovirus intracellular release pathway. Cell. 182 (2), 515-530 (2020).
- Gustafsson, M. G. L. Surpassing the lateral resolution limit by a factor of two using structured illumination microscopy. Journal of Microscopy. 198 (2), 82-87 (2000).
- Rego, E. H., Shao, L., Rego, E. H. Practical structured illumination microscopy. Methods in Molecular Biology. 1251, 1251 (2015).
- Phillips, M. A., et al. CryoSIM: super-resolution 3D structured illumination cryogenic fluorescence microscopy for correlated ultrastructural imaging. Optica. 7 (7), 802 (2020).
- Matsuda, A., Schermelleh, L., Hirano, Y., Haraguchi, T., Hiraoka, Y. Accurate and fiducial-marker-free correction for three-dimensional chromatic shift in biological fluorescence microscopy. Scientific Reports. 8, 7583 (2018).
- Kaufmann, R., Hagen, C., Grünewald, K. Fluorescence cryo-microscopy: current challenges and prospects. Current Opinion in Chemical Biology. 20, 86-91 (2014).
- Phillips, M. A., Pinto, D. M. S., Dobbie, I. M. SPEKcheck-fluorescence microscopy spectral visualisation and optimisation: a web application, javascript library, and data resource. Wellcome Open Research. 3, 92 (2018).
- Demmerle, J., et al. Strategic and practical guidelines for successful structured illumination microscopy. Nature Protocols. 12 (5), 988-1010 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados