Method Article
Coleta de dados microscópicos de iluminação crioesticamente estruturadas de células criogenicamente preservadas
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Este protocolo demonstra como imaginar amostras biológicas conservadas por crio-preservados usando microscopia de iluminação estruturada crioes. Demonstramos a metodologia ao fotografar o citoesqueleto das células U2OS.
Resumo
A microscopia estruturada de iluminação tridimensional (3D) (SIM) permite imagens de estruturas celulares fluorescentes com maior resolução do que a microscopia de fluorescência convencional. Esta técnica de super-resolução (RS) permite a visualização de processos moleculares em células inteiras e tem o potencial de ser usada em conjunto com microscopia eletrônica e tomografia de raios-X para correlacionar informações estruturais e funcionais. Um microscópio SIM para amostras criogenicamente preservadas (crioSIM) foi recentemente encomendado na linha de feixe de crio-imagem correlativa B24 no síncrotron do Reino Unido.
Foi projetado especificamente para imagens 3D de amostras biológicas a temperaturas criogênicas de forma compatível com imagens subsequentes das mesmas amostras por métodos de microscopia de raios-X, como a tomografia de raios-X crio-macio. Este artigo de vídeo fornece métodos e protocolos detalhados para imagens bem sucedidas usando o crioSIM. Além das instruções sobre o funcionamento do microscópio crioSIM, foram incluídas recomendações sobre a escolha de amostras, fluoroforos e configurações de parâmetros. O protocolo é demonstrado em amostras de células U2OS cujas mitocôndrias e tubulinas foram rotuladas fluorescentemente.
Introdução
As técnicas de imagem de SR tornaram-se amplamente acessíveis aos biólogos na última década1. Eles permitem imagens de alta resolução de amostras marcadas fluorescentes além do limite de difração. No entanto, tem sido desafiador adaptar os métodos de microscopia de RS para trabalhar com amostras a temperaturas criogênicas2. Isso seria vantajoso para imagens correlativas em combinação com tomografia eletrônica ou de raios-X. Recentemente, o SIM foi adaptado para uso com amostras criogênicas e tem sido demonstrado com sucesso para permitir estudos correlativos de células biológicas em conjunto com a tomografia de raios-X macia (SXT)3 na linha de feixe de crio-imagem correlativa B24 no Diamond Light Source Síncrotron (https://www.diamond.ac.uk/Instruments/Biological-Cryo-Imaging/B24.html). O SIM pode dobrar a resolução da microscopia convencional de campo largo iluminando a amostra com padrões listrados de luz (franjas moiré) em três ângulos e em cinco fases. A interferência entre esses padrões de luz e a fluorescência amostral pode ser usada para descobrir computacionalmente informações extras sobre estruturas de sub-difração4,5.
Existem várias vantagens do SIM em relação a outras técnicas de RS para aplicações criogênicas. Primeiro, pode funcionar sem fluoroforos piscando especialmente projetados; fluoroforos convencionais podem ser usados, dando acesso a uma gama mais ampla de potenciais agentes fluorescentes de marcaçãofluorescente 6. Além disso, requer apenas 15 imagens por fatia z (em 3D; 9 imagens para 2D), enquanto outros métodos de RS tiram aproximadamente 1000 imagens por fatia, aumentando a chance da amostra ser aquecida e, portanto, aumentando o risco de formação de cristais de gelo, o que pode causar artefatos. Finalmente, essa técnica pode imaginar amostras biológicas mais espessas de mais de 10 μm, permitindo que células inteiras sejam imagens em seu estado quase nativo6. O crioSIM foi construído usando componentes ópticos padrão e com software de acesso aberto para imagem, facilitando a documentação e duplicação se desejar6. O crioSIM tem um objetivo de abertura numérica 100x/0.9 (ver a Tabela de Materiais); mais informações sobre seus componentes ópticos, parâmetros de design e desempenho foram descritas por Phillips et al.6 Aqui, este protocolo demonstra como usar o microscópio crioSIM, incluindo como carregar e descarregar amostras no estágio criogênico, como coletar dados sobre o microscópio e como reconstruir as imagens SIM.
Protocolo
NOTA: Este protocolo diz respeito a amostras contendo células cultivadas ou depositadas em redes de ouro planas de 3 mm de elétrons de transmissão (TEM) com uma película de suporte de carbono furada que foram vitrificadas por congelamento de mergulho ou congelamento de alta pressão. Este protocolo pressupõe que as amostras já foram imagens usando uma epifluorescência convencional e microscópio brightfield para mapear locais de interesse para imagens em crioSIM. Consulte a Figura 1 para uma visão geral de todo o protocolo.
1. Preparação da crio-etapa
- Prepare nitrogênio líquido sem gelo (LN2) passando LN2 através de um funil forrado com quaisquer lenços de papel seco científico padrão.
NOTA: Como a LN2 causa queimaduras, use equipamentos de proteção individual adequados, incluindo luvas e óculos crioprotetos, ao manuseá-lo. Esses líquidos podem deslocar oxigênio e causar sufocamento rápido e devem ser manuseados em uma área devidamente ventilada. - Remova o pó superficial do crio-estágio com ar pressurizado. Expulse o líquido do recipiente de ar pressurizado antes de usá-lo.
- Certifique-se de que o cartucho de carregamento de amostras esteja no lugar com o suporte de amostra em câmara apropriado (verifique se não há grade deixada no suporte da amostra de um experimento anterior)(Figura 2).
- Retire a tampa da dewar externa do estágio crio-e despeje lN2 filtrada até aproximadamente 1/4de completo. Aguarde até que a ebulição inicial diminua antes de derramar mais; encha o recipiente para cerca de 2/3completo. Substitua a tampa cuidadosamente, apontando o bocal para longe do manipulador e do estágio/óptica enquanto a LN2 ferve para fora da tomada.
- Uma vez que a LN2 parou de sair da tomada, coloque o tubo de saída sobre o palco de guerra no crio-palco.
- Conecte a fonte de alimentação do estágio e conecte o cabo USB ao estágio criográfico. Certifique-se de que a tampa da câmara de amostra aquecida está conectada. Ligue a de guerra externa ao palco.
NOTA: Não conecte a dewar externa até que o tubo de saída tenha sido posicionado sobre o estágio de guerra no estágio crio-stage. Se a LN2 transbordar (ou para evitar que ela transborda), puxe o cabo USB para ~10 s, permita que ele se equilibre e reconecte o cabo USB para reativar o sensor. - Depois que a LN2 for entregue no estágio de guerra, pressione o botão de liberação no crio-palco para permitir que lN2 entre na câmara de amostra.
NOTA: Não deixe o sistema sozinho enquanto a LN2 estiver enchendo a câmara. - Aguarde ~30-45 min para permitir que o sistema esfrie e estabilize antes de iniciar a aquisição de imagens. Verifique periodicamente o dewar externo e o top-up com LN2 filtrado se estiver com menos de um quarto de cheio (aproximadamente a cada hora).
2. Transferência da caixa de armazenamento de amostras para o estágio criográfico
NOTA: Mergulhe a caixa de armazenamento da amostra, o suporte e as pontas de quaisquer instrumentos (por exemplo, fórceps) dentro filtrados da LN2 para esfriá-los antes de tocar em superfícies frias, como a amostra ou quaisquer objetos dentro da câmara de amostra. Use um casaco de laboratório e luvas ao manusear amostras biológicas.
- Certifique-se de que as amostras vitrificadas estejam em um recipiente compatível com criogeno e leve-as ao microscópio. Pressione o botão correspondente no crio-palco para acender a luz na câmara de amostra.
- Use a chave hexa, na ferramenta para abrir as duas placas do de transferência de amostra. Abra as placas largas o suficiente para cair na grade entre as duas placas, mas evite abrir para a posição máxima aberta para evitar que a grade caia pelo outro lado.
- Use fórceps longos para levantar a caixa de grade de amostra para fora da LN2,gire-a onde o entalhe se alinha com a posição da posição de armazenamento dentro do palco e coloque-a no palco. Use o dispositivo apropriado (por exemplo, uma chave de fenda) para abrir a tampa da caixa de armazenamento à posição correta da amostra.
- Utilizando fórceps invertidos (ou quaisquer fórceps cirúrgicos de ponta fina), remova a grade TEM do suporte da amostra, mergulhe-a dentro da LN2e solte-a em posição no de transferência de amostra, mantendo-a próxima da LN2 durante o processo de transferência. Certifique-se de que o lado do filme de carbono seja colocado para que ele esteja, em última análise, voltado para o objetivo na ponte amostral.
- Feche o cartucho de amostra usando a tecla hexax na ferramenta. Feche e remova a caixa de armazenamento junto com todas as amostras restantes.
- Use o ponto de ímã na ferramenta de para levantar e montar o cartucho contendo a grade na ponte de amostra. Mantenha-o imerso/próximo ao LN2 durante o processo de transferência. Certifique-se de que a orientação é apropriada para a ponte (os dois ímãs farão contato com os ímãs na ponte). Coloque o plano dentro dos pinos de posicionamento da ponte e cutuque suavemente para garantir que ele esteja fixo.
NOTA: Um de amostra para grades cortadas que foram preparadas para fresagem posterior de feixe de íons focalados também está disponível na instalação CryoSIM na linha de trave B24.
3. Acoplamento e foco do palco
- Mova a tampa do estágio crioco para a posição de imagem e desligue a luz da câmara de amostra. Deslize o palco em direção à óptica para alinhá-lo sob a lente objetiva. Abaixe suavemente o objetivo na posição usando a alavanca, garantindo que ela repouse dentro da tampa do crio-estágio, mas não a toque.
NOTA: Certifique-se de que o tubo de saída externo de Dewar não toque no estágio de guerra no estágio crioque em qualquer momento durante a coleta de dados. - Cubra o palco e a óptica com uma cortina preta opaca. Inicie o software de controle Cockpit no CRYOSIM PC (Figura 3 e Figura 4).
- Clique no botão do modo de leitura para cada câmera e ajuste-a para CONV 3 MHz. Verifique se a temperatura de cada câmera é de -80 °C, e o ventilador da câmera está desligado.
- Ligue a câmera refletida. Em Lights, escolha ambiente (exposição 20 ms) e em linkam, clique no condensador. Clique no botão modo Vídeo.
- Na janela de exibição mosaico, amplie (pergaminho do mouse) para ver o contorno da grade. Clique no find stage se ele não puder ser visto. Centralizar a grade clicando duas vezes no meio do círculo.
- Concentre a amostra até que o filme de suporte à grade (ou qualquer outro recurso de amostra relevante) esteja em foco, usando as teclas para cima e para baixo para mover o crio-palco para cima e para baixo, e usando as teclas 9 e 3 na almofada numérica para alterar o passo z (coloque-o em 20 μm para foco inicial).
NOTA: Se o usuário ficar sem viagem em z, o estágio pode ser movido manualmente para cima ou para baixo usando a roda sob o estágio criográfico. Gire-o por um entalhe de cada vez e verifique se a amostra está dentro do intervalo de exibição. Altere a direção novamente se o foco piorar.
4. Aquisição de mosaico brightfield
- Uma vez que o palco esteja centrado, desligue o modo de vídeoe colete um mosaico de luz visível (clique em Executar mosaico na vista do Mosaico para produzir telhas de imagens de luz visível que espiral para fora do centro). Se a grade estiver dobrada, experimente diferentes posições na grade (clique duplo à esquerda na vista do Mosaico),refoque e clique em Executar mosaico novamente para coletar mosaicos parciais. Alternativamente, drop-in uma imagem focada no topo do mosaico (Figura 3).
- Salve a exibição clicando em Salvar mosaico. Dê-lhe um nome de arquivo curto contendo informações na caixa de armazenamento e no respectivo número da grade (um boletim de tempos será anexado automaticamente ao nome do arquivo).
5. Identificação de áreas de interesse
- Inspecione o mosaico brightfield ao lado de qualquer imagen anterior de "mapa" de fluorescência para onde células ou características biológicas de interesse tenham sido previamente localizadas. Verifique se essas áreas produzem fluorescência adequada.
- Desligue a luz ambiente e o condensador, bem como o modo de vídeo, se estiver ativo. Ligue o laser de excitação necessário (405, 488, 561 ou 647), e escolha a câmera e o filtrocorrespondentes , inicialmente a 50 mW, para tempo de exposição de 50 ms.
NOTA: Aumente/diminua essas configurações da câmera e do filtro, dependendo do sinal de fluorescência. - Pressione 0 para tirar uma imagem e * para auto-contraste. Alternativamente, ajuste manualmente o contraste usando o controle deslizante na parte inferior da imagem.
NOTA: Ligue o sinal laser para a sala. - Uma vez que células biologicamente interessantes com fluorescência adequada foram encontradas, marque suas posições usando o botão mark site na vista do Mosaico.
NOTA: Esses sites marcados aparecerão na lista fechada e podem ser acessados clicando duas vezes nas coordenadas. Ao retornar a um site marcado, amplie a área antes de clicar duas vezes. - Continue marcando todos os sites potenciais antes de iniciar a aquisição de imagens. Ressusar o mosaico com os locais marcados; clique em Salvar sites para arquivar.
- Para costurar as imagens do mosaico usando o software StitchM (desenvolvido dentro na linha de trave B24), arraste e solte o arquivo de mosaico .txt no arquivo StitchM com .bat de extensão e salve a imagem combinada de tiff das telhas de mosaico na mesma pasta. Para salvar uma imagem com os sites marcados, arraste e solte o arquivo .txt mosaico e os marcadores.txt arquivar no ícone ao mesmo tempo.
NOTA: Equilibrar a coleta de dados contra as necessidades do projeto (por exemplo, se a imagem correlativa for feita, verifique quantas imagens podem ser tiradas com a modalidade de imagem de parceiro e escolha o número adequado de locais para imagens crioSIM). Além disso, se o dewar externo exigir reabastecimento com LN2, o sistema crio-estágio mudará de posição e os locais marcados provavelmente não retornarão aos mesmos locais; portanto, considere isso ao escolher o número de sites a serem imagens.
- Para costurar as imagens do mosaico usando o software StitchM (desenvolvido dentro na linha de trave B24), arraste e solte o arquivo de mosaico .txt no arquivo StitchM com .bat de extensão e salve a imagem combinada de tiff das telhas de mosaico na mesma pasta. Para salvar uma imagem com os sites marcados, arraste e solte o arquivo .txt mosaico e os marcadores.txt arquivar no ícone ao mesmo tempo.
- Desligue a luz ambiente e o condensador, bem como o modo de vídeo, se estiver ativo. Ligue o laser de excitação necessário (405, 488, 561 ou 647), e escolha a câmera e o filtrocorrespondentes , inicialmente a 50 mW, para tempo de exposição de 50 ms.
6. Estratégia de coleta de dados
- Defina o tempo de exposição a laser com base nas contagens no alcance dinâmico da imagem de fluorescência (na parte inferior da janela de visualização da câmera). Escolha qual filtro aplicar e otimize essas configurações para cada comprimento de onda de luz de excitação a ser usada, ligando cada laser separadamente.
NOTA: Embora as contagens de 10.000-20.000 sejam ótimas, as contagens mais baixas são aceitáveis se houver um bom contraste. O ideal é verificar as configurações do filtro antes que cada pilha de imagens seja adquirida porque as células em diferentes áreas da grade podem ter níveis variáveis de fluorescência.
7. Coleta de dados
- Clique em ambas as câmeras para ligá-las. Retorne a um dos locais marcados e foque na profundidade desejada novamente. Uma vez em foco em uma área de interesse, saia do foco para cima(↑ tecla) na janela XY no Estágio Macro para escolher a altura da pilha z para adquirir e clique em Salvar superior. Saia do foco para baixo(tecla ◗ ), clique em Salvar a parte inferior e, em seguida, vá para o centro, e verifique se a imagem ainda está em foco.
NOTA: A altura da amostra será mostrada na janela. - Clique com o botão direito do mouse no slm (modulador de luz espacial) na janela Cockpit e certifique-se de que o ângulo está definido em 0,41. Na janela Cockpit, selecione experimento de um único local.
NOTA: Não clique no slm; O cockpit irá ligá-lo automaticamente durante a aquisição de imagens.- Na lista suspensa, selecione Iluminação Estruturada. Altere a altura do Stack de modo que seja igual à altura z + 1 μm.
NOTA: A adição de 1 μm garante a captura de toda a amostra em z e minimiza artefatos de reconstrução. - Digite os tempos de exposição (ms) para os lasers de 405 nm e 488 nm na linha superior (para a câmera refletida) e os tempos de exposição para os lasers de 561 nm e 647 nm na linha inferior (para a câmera transmitida).
NOTA: Os valores para o tempo de exposição devem corresponder aos valores decididos anteriormente e mostrados na janela principal do Cockpit. - Insira um nome de arquivo (convenção de nomeação: caixa number_grid area_filters_FL) (FL (fluorescência) ou BF (brightfield) dependendo do tipo de imagem que está sendo feito). Clique em Atualizar para produzir um novo arquivo contendo a data e a hora sem substituir arquivos anteriores. Em seguida, clique em Iniciar.
- Na lista suspensa, selecione Iluminação Estruturada. Altere a altura do Stack de modo que seja igual à altura z + 1 μm.
- Verifique a visualização da câmera enquanto os dados estão sendo coletados. Re-tirar as imagens se houver algum deslocamento de xy. Se o LN2 dewar reabastecer o estágio criográfico durante a aquisição de imagem, aborte o processo clicando no botão Abortar no software Cockpit.
- Uma vez que a dewar externa tenha terminado de reabastecer o estágio de guerra, repita o experimento porque a recarga desloca a amostra verticalmente. Refoque a imagem para centralizar novamente em z e repetir o experimento single-site. Repita o experimento de um único local para todas as combinações de lasers e filtros necessários.
NOTA: Leva aproximadamente 30 s-1 min para terminar o reabastemento. Durante a imagem de uma grade, o reabastecimento acontecerá ~4-8x.
- Uma vez que a dewar externa tenha terminado de reabastecer o estágio de guerra, repita o experimento porque a recarga desloca a amostra verticalmente. Refoque a imagem para centralizar novamente em z e repetir o experimento single-site. Repita o experimento de um único local para todas as combinações de lasers e filtros necessários.
- Em cada posição, colete uma pilha z usando luz visível.
- Desligue os lasers e ligue a luz ambiente e o condensador. Em experimento de um único local,selecione Z-stack,defina a luz ambiente para exposição de 20 ms e mantenha a altura z como acima.
- Repita as etapas 7.2 a 7.4 para todos os locais marcados na grade.
- Antes de passar para outra amostra, exclua o mosaico clicando em Excluir telhas.
- Desenhe um quadrado em torno de todas as telhas para excluí-las. Exclua os marcadores também selecionando todos eles na lista e, em seguida, clicando em Excluir sites selecionados.
- Desligue a luz ambiente e o condensador antes de continuar a mudar a grade. Siga os passos na seção 4 em ordem inversa para desacorrer a etapa sob o objetivo e alterar a grade.
8. Após a imagem
- Depois que a imagem estiver terminada, desencaixe o estágio e remova todas as amostras. Desligue a luz da câmara de amostra. Desligue o dewar externo e decante qualquer LN2 restante em outro recipiente compatível com criogeno, permitindo que a dewar retorne com segurança a uma temperatura normal.
- Coloque a tampa no palco criogeno. Aguarde até que a opção de assar o display crio-palco fique disponível depois que não permanecer mais LN2 no estágio de guerra. Pressione o botão de assa para entrar no modo de aquecimento e remova qualquer umidade do estágio crioque para evitar a formação de gelo.
9. Reconstrução
- Transfira arquivos de dados SIM brutos para a estação de trabalho apropriada para reconstrução. Executar o processamento em lotes através da janela de construção de tarefas de processamento usando funções de transferência óptica específicas do canal (OTFS) (calculadas a partir de funções de spread de ponto de contas multi-fluorescentes) e ângulos K0 (0,29278, -1.8028, 2,3786) com um filtro Wiener constante para todos os canais de 0,004 e um deslocamento de viés de 200.
- No painel Opções adicionais, certifique-se de que as intensidades negativas sejam descartadas mantendo outras opções desmarcadas. Salve as imagens SIR em uma pasta chamada "processada".
NOTA: O software comercial de reconstrução do SIM geralmente produz dados SIM reconstruídos e retém o nome do arquivo, mas anexa SIR.dv no final. Também é criado um arquivo de registro que contém o protocolo de processamento, etapas e informações estatísticas sobre o sucesso da reconstrução.
10. Correção de turno cromático
- Baixe o software Chromagnon7 para corrigir para o turno cromático.
- Use a matriz de referência de mudança cromática que corresponde aos comprimentos de onda de fluorescência dos dados coletados (fornecidos pela linha de trave).
NOTA: O arquivo de referência é um arquivo 'chromagnon.csv', que contém os parâmetros de alinhamento e foi obtido a partir de imagens de calibração usando nanopartículas multi-fluorescentes. Ele pode ser usado para processar em lote vários conjuntos de dados ao mesmo tempo.- Escolha o arquivo de referência apropriado que corresponda ao comprimento de onda do laser e ao filtro usado para a imagem da amostra e adicione-o ao campo de referência. Adicione os dados SIR reconstruídos a serem alinhados no campo de origem e clique em Executar.
- Verifique se o sinal de fluorescência está agora alinhado nas imagens. Para processamento em lote, coloque todas as imagens SIR a serem alinhadas no campo de origem e no arquivo de referência no campo de referência e pressione Executar todas.
Resultados
Uma amostra contendo células U2OS foi manchada com uma mistura de corante de citoesqueleto de microtúbulo verde e corante de mitocôndrias vermelhas, resultando na coloração do componente microtúbulo do citoesqueleto (verde) e das mitocôndrias (vermelho). Imagens subsequentes mostraram a localização de mitocôndrias dentro da célula, bem como o arranjo dos microtúbulos, destacando a estrutura estrutural que eles fornecem à célula e a montagem do citoesqueleto em torno de organelas como o núcleo. A resolução no crioSIM é significativamente maior do que a da microscopia de epifluorescência padrão(Figura 5). A Figura 6 demonstra como o "mapa" fluorescente de um microscópio convencional de epifluorescência pode ser usado para localizar áreas de interesse para imagens e a imagem reconstruída por crioSIM correspondente a partir de um local na grade.
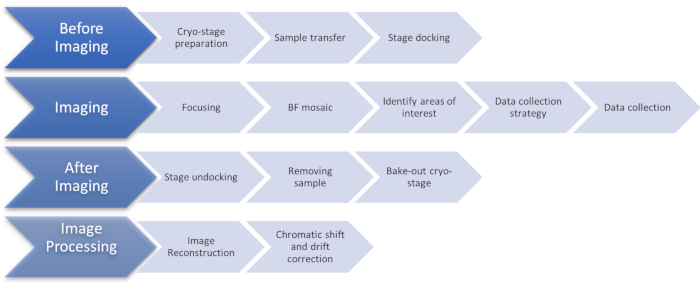
Figura 1: Gráfico de fluxo mostrando os estágios do protocolo de imagem crioSIM. Abreviação: crioSIM = Microscopia de iluminação estruturada por crio. Clique aqui para ver uma versão maior desta figura.

Figura 2: O crio-palco. (A) A configuração crio-palco. (B) Uma grade de amostra mostrada por fórceps invertidos. (C) Componentes do crio-palco. As portas de conexão são rotuladas, com as cores correspondentes ao laranja: alimentação, amarelo: tampa de palco aquecida, azul: de guerra externa, verde: conexão com PC. Abreviaturas: PC = computador pessoal; LN2 = nitrogênio líquido. Clique aqui para ver uma versão maior desta figura.
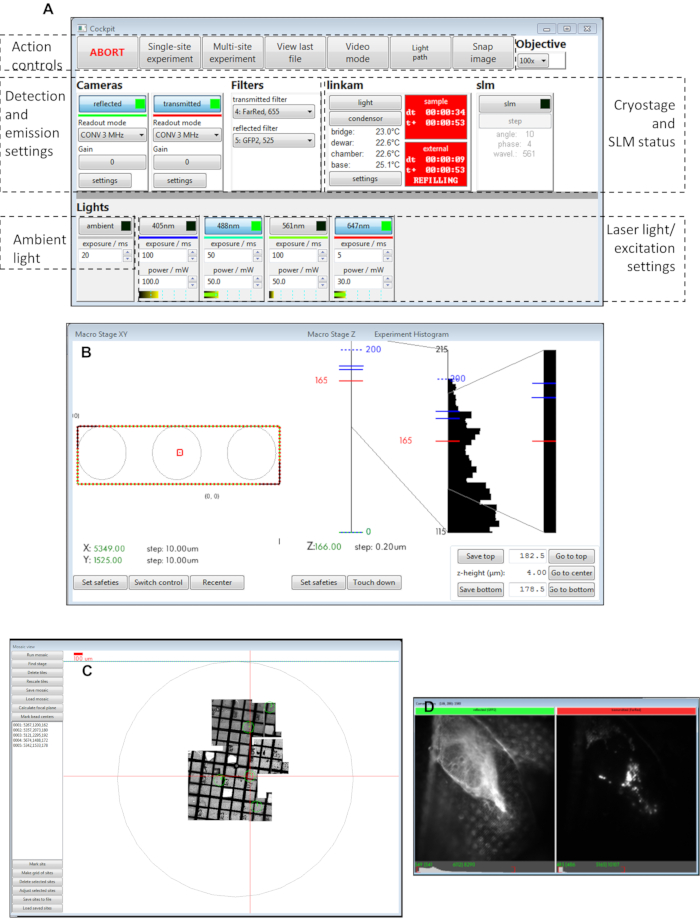
Figura 3: Visualizações dos painéis de software do Cockpit. (A) Painel principal,(B) macro estágio XY, (C) vista da câmera do mosaico,(D). Abreviação: SLM = modulador de luz espacial. Clique aqui para ver uma versão maior desta figura.
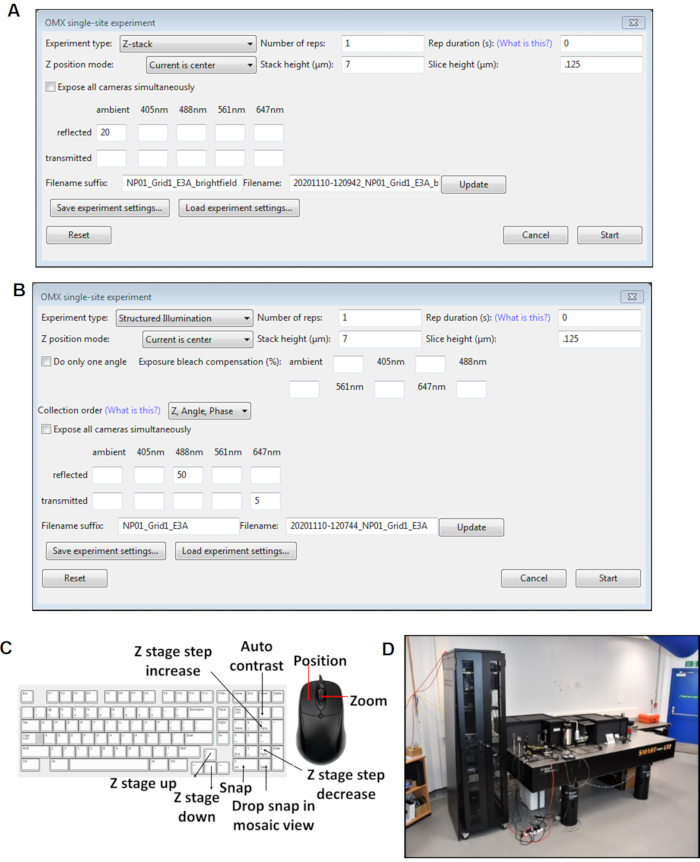
Figura 4: Visualizações dos painéis de software do Cockpit. (A) Z empilhar experiência de local único. (B) Si experiência de local único. (C) Atalhos de teclado para o software do cockpit usado durante a aquisição de imagens. (D) O microscópio crioSIM está no local no feixe B24 no síncrotron Diamond Light Source. Abreviação: crioSIM = Microscopia de iluminação estruturada por crio. Clique aqui para ver uma versão maior desta figura.
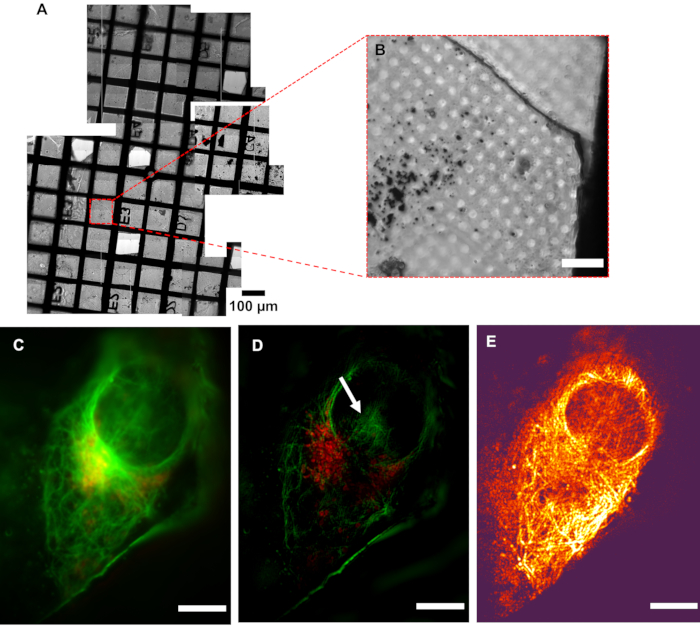
Figura 5: Resolução de crioSIM. (A) Visão mosaica de uma grade em análise. (B) Imagem brightfield de uma Área de Interesse (AOI). (C) Imagem pseudo-widefield em comparação com sua(D) imagem SIM mostrando o aumento da resolução. A seta branca indica artefatos de reconstrução SIM. (E) Mapa de contraste modulação combinando as informações de intensidade de pixel da imagem reconstruída com as informações coloridas dos respectivos valores de relação contraste-ruído (MCNR) de modulação dos dados brutos gerados pelo SIMCheck2. As regiões brilhantes e escuras apresentam alto e baixo contraste, respectivamente. Barra de escala = 10 μm. Configurações de imagem CryoSIM: comprimentos de onda de excitação/emissão: 488/525 nm, 50 mW de potência laser, 50 ms de tempo de exposição e 647/655 nm, 20 mW de potência laser, 5 ms de tempo de exposição. Abreviação: crioSIM = Microscopia de iluminação estruturada por crio. Clique aqui para ver uma versão maior desta figura.

Figura 6: Reconstrução de imagem no crioSIM a partir de um local na grade em um mapa de fluorescência a partir de um mapa de epifluorescência convencional. (A,B) Sobreposição de mapas de imagem de campo brilhante e fluorescência gerados com um microscópio de epifluorescência convencional. Este mapa é usado para localizar regiões de interesse para posteriormente imagem em crioSIM. (C) A imagem crioSIM reconstruída obtida no local mostrado em (B). Abreviação: crioSIM = Microscopia de iluminação estruturada por crio. Clique aqui para ver uma versão maior desta figura.
Discussão
O SIM 3D em temperaturas criogênicas tem muitas vantagens sobre outras técnicas de imagem de RS para material biológico vitrificado por imagem. Requer significativamente menos imagens por fatia z em comparação com outros métodos de RS, resultando em menos irradiação e menor chance de formação de cristais de gelo para amostras vitrificadas. Ele também é capaz de imagem células inteiras e pode ser correlacionado com tomografia de raios-X para combinar estrutura com função. Curiosamente, a maioria das marcas de fluoroforos e fluorescência disponíveis comercialmente alveja menos sob condições criogênicas do que à temperatura ambiente. No entanto, dado o alto rendimento quântico dos fluoroforos mais comuns à temperatura ambiente (mais de 80% em alguns casos), o ganho absoluto detectado em fótons não se deve a mudanças no rendimento quântico, mas devido a uma redução nos processos complexos de branqueamento. Mais informações sobre o rendimento de fluoroforos a temperaturas criogênicas podem ser encontradas em 8.
É fundamental que as amostras que chegam ao crioSIM tenham sido pré-mapeadas usando um microscópio de criofluorescência convencional com capacidade de campo brilhante para produzir um "mapa" de grade que inclui o destaque de todas as POSSÍVEIS AOIs para imagens posteriores(Figura 5). O tempo de acesso no crioSIM é alocado por meio de um processo competitivo que envolve a apresentação de uma proposta, que é posteriormente avaliada para viabilidade técnica e impacto biomédico. O tempo no equipamento, portanto, é sempre "premium", e as grades pré-mapeadas permitem o uso mais eficiente de uma alocação. Também é essencial que a amostra seja mantida vitrificada, especialmente durante a transferência amostral do porta-amostras para a plataforma de imagem, para minimizar a formação de cristais de gelo e danos amostrais subsequentes. A amostra deve ser de boa qualidade para produzir as melhores imagens SIM. Uma amostra bem preparada será caracterizada pelas seguintes características: (a) não terá contaminação por cristal de gelo, (b) a grade utilizada será uma grade de localizador, (c) o portador será plano, (d) a malha de grade e a superfície do substrato não serão auto-fluorescentes, e (e) não haverá quebras na membrana de suporte. Esses pré-requisitos podem ser alcançados através do manuseio cuidadoso da amostra e garantindo que as amostras permaneçam sempre vitrificadas.
É importante verificar com antecedência se os fluoroforos de amostra propostos darão sinal suficiente no microscópio crioSIM. Ferramentas como SPEKCheck9 podem ajudar na escolha das combinações ideais de fluoróforo e filtro. Se houver problemas com a coleta de dados brutos ou o processo de reconstrução, artefatos podem aparecer nas imagens após a reconstrução. Exemplos de vários artefatos foram documentados por Demmerle et al.10 Os parâmetros de reconstrução da imagem podem ser revisados no arquivo de registro SoftWoRx se a reconstrução não for ideal abrindo o arquivo de resumo de reconstrução. Deve haver um espaçamento de linha consistente entre ângulos em um determinado canal e amplitude relativamente consistente. A variação de mais de 30% e valores significativamente acima de 1 (se a compensação do tamanho das contas for aplicada) deve ser mais investigada e provavelmente indicar reconstruções fracassadas. Além disso, o software SIMcheck2 em Fiji também pode ser usado para realizar várias verificações nos dados brutos e reconstruídos para diagnosticar a causa de erros nas configurações do parâmetro de imagem ou reconstrução. A verificação do SIM e seu mapa de contraste de modulação também podem auxiliar na avaliação da qualidade dos dados reconstruídos, interpretando quais áreas de uma imagem provavelmente serão estruturas reais versus artefatos.
O baixo contraste de modulação (mostrado pela cor escura, na Figura 5E) dentro da área nuclear significa que esta região será mais suscetível a artefatos de reconstrução, implicando, portanto, que os padrões de hash mostrados no núcleo poderiam ser classificados(Figura 5D) como um artefato. Áreas fortes de sinal de fluorescência são mais propensas a refletir com precisão estruturas nativas nos dados processados. Em áreas de sinal fraco onde fluoroforos são distribuídos em áreas mais amplas, como a superfície total de uma vesícula, é provável que o sinal real coexista com artefatos de processamento, e deve-se tomar cuidado na interpretação desses dados. Após a inspeção dos dados reconstruídos de longo alcance para garantir que não haja artefatos estranhos, e que o fundo é geralmente gaussiano e centrado perto de zero, os dados são geralmente cortados a zero, ou o valor modal - o pico do sinal de fundo - deve ser muito próximo de zero. Isso garante que o alcance dinâmico da imagem exibida não seja dominado por artefatos de fundo negativos. Quando um sinal mais fraco é esperado, deve-se tomar cuidado extra na análise das características e na garantia de que sejam estruturas reais em vez de artefatos de reconstrução.
Existem algumas limitações do sistema de imagem. Como o estágio amostral é plano, amostras com espessura variável ou grades que não são planas não são objetos ideais para imagem. Além disso, se a imagem correlativa for feita usando tomografia de raios-X macia, as células próximas aos limites da grade não devem ser imagens, pois estas não serão visíveis no microscópio de raios-X durante a aquisição da série de inclinação. Finalmente, a quantidade de manchas da amostra antes do congelamento do mergulho tem um impacto significativo na qualidade da imagem; muito pouco blotting resulta em amostras que são muito grossas, dando imagens sim subótimas com alto ruído de fundo, enquanto muita mancha pode fazer com que as células fiquem desaformadas e, portanto, mais suscetíveis a danos térmicos do raio laser incidente. Em resumo, o crioSIM é uma poderosa ferramenta de microscopia de fluorescência para imagens biológicas em 3D em um estágio quase nativo e tem aplicações abrangentes em muitas áreas.
Divulgações
Os autores não declaram interesses financeiros concorrentes.
Agradecimentos
Este projeto recebeu financiamento do projeto iNEXT-Discovery da Comissão Europeia Horizon 2020. I.M. Dobbie reconhece financiamento do Wellcome Trust (107457/Z/15/Z). Este trabalho foi realizado com o apoio da Fonte de Luz Diamante, instrumento B24 (proposta BI25512). Nossos agradecimentos à equipe da Micron e a todos os nossos excelentes usuários e colaboradores por nos ajudar a estabelecer o crioSIM e seu potencial correlativo.
Materiais
| Name | Company | Catalog Number | Comments |
| Auto grids | FEI | ||
| Autogrids | Thermo Fisher Scientific | 1036173 | |
| BioTracker 488 Green Microtubule Cytoskeleton Dye | Sigma-Aldrich | SCT142 | |
| Cockpit Software | Oxford University | https://github.com/MicronOxford/cockpit | |
| cryo compatible polyurethane container | Jena bioscience | CC-FD-800 | |
| Cryo TEM grid storage box | Thermo Fisher Scientific | Model#AutoGrid | |
| cryo-SIM microscope | Custom made | N/A | Custom made, see following reference for the design: Michael A. Phillips, Maria Harkiolaki, David Miguel Susano Pinto, Richard M. Parton, Ana Palanca, Manuel Garcia-Moreno, Ilias Kounatidis, John W. Sedat, David I. Stuart, Alfredo Castello, Martin J. Booth, Ilan Davis, and Ian M. Dobbie, "CryoSIM: super-resolution 3D structured illumination cryogenic fluorescence microscopy for correlated ultrastructural imaging," Optica 7, 802-812 (2020). Has a Nikon TU Plan Apo 100x/0.9 NA. |
| Cryostage system | Linkam Scientific Instruments | CMS196 | |
| Fine tip surgical forceps | Ted Pella | 38125 | |
| MitoTracker Deep Red FM | Thermo Fisher Scientific | M22426 | |
| Python Microscope Software | Oxford University | https://www.python-microscope.org/ | |
| Scientific dry paper wipes | Kimberly-Clark 7551 | 2 | |
| SIM Reconstruction Software | softWoRx, GE Healthcare | Version 6.5.2 | |
| StitchM Software | Diamond Light Source | https://github.com/DiamondLightSource/StitchM | |
| TEM grids for samples | Quantifoil Micro Tools | G200F1 |
Referências
- Schermelleh, L., et al. Super-resolution microscopy demystified. Nature Cell Biology. 21 (1), 72-84 (2019).
- Ball, G., Demmerle, J., Kaufmann, R., Davis, I., Dobbie, I. M., Schermelleh, L. SIMcheck: A toolbox for successful super-resolution structured illumination microscopy. Scientific Reports. 5 (1), 15915 (2015).
- Kounatidis, I., et al. 3D Correlative cryo-structured illumination fluorescence and soft X-ray microscopy elucidates reovirus intracellular release pathway. Cell. 182 (2), 515-530 (2020).
- Gustafsson, M. G. L. Surpassing the lateral resolution limit by a factor of two using structured illumination microscopy. Journal of Microscopy. 198 (2), 82-87 (2000).
- Rego, E. H., Shao, L., Rego, E. H. Practical structured illumination microscopy. Methods in Molecular Biology. 1251, 1251 (2015).
- Phillips, M. A., et al. CryoSIM: super-resolution 3D structured illumination cryogenic fluorescence microscopy for correlated ultrastructural imaging. Optica. 7 (7), 802 (2020).
- Matsuda, A., Schermelleh, L., Hirano, Y., Haraguchi, T., Hiraoka, Y. Accurate and fiducial-marker-free correction for three-dimensional chromatic shift in biological fluorescence microscopy. Scientific Reports. 8, 7583 (2018).
- Kaufmann, R., Hagen, C., Grünewald, K. Fluorescence cryo-microscopy: current challenges and prospects. Current Opinion in Chemical Biology. 20, 86-91 (2014).
- Phillips, M. A., Pinto, D. M. S., Dobbie, I. M. SPEKcheck-fluorescence microscopy spectral visualisation and optimisation: a web application, javascript library, and data resource. Wellcome Open Research. 3, 92 (2018).
- Demmerle, J., et al. Strategic and practical guidelines for successful structured illumination microscopy. Nature Protocols. 12 (5), 988-1010 (2017).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados