Method Article
Collecte de données microscopiques d’éclairage cryostructuré à partir de cellules cryogéniquement préservées
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole montre comment imager des échantillons biologiques cryo-préservés à l’aide de la microscopie à éclairage cryo-structuré. Nous démontrons la méthodologie en imageant le cytosquelette des cellules U2OS.
Résumé
La microscopie à éclairage structuré (SIM) tridimensionnelle (3D) permet l’imagerie de structures cellulaires marquées par fluorescence à une résolution plus élevée que la microscopie à fluorescence conventionnelle. Cette technique de super-résolution (SR) permet de visualiser les processus moléculaires dans des cellules entières et a le potentiel d’être utilisée conjointement avec la microscopie électronique et la tomographie aux rayons X pour corréler les informations structurelles et fonctionnelles. Un microscope SIM pour échantillons cryogéniquement conservés (cryoSIM) a récemment été mis en service sur la ligne de faisceau corrélative de cryo-imagerie B24 du synchrotron britannique.
Il a été conçu spécifiquement pour l’imagerie 3D d’échantillons biologiques à des températures cryogéniques d’une manière compatible avec l’imagerie ultérieure des mêmes échantillons par des méthodes de microscopie à rayons X telles que la tomographie aux rayons X cryo-mous. Cet article vidéo fournit des méthodes et des protocoles détaillés pour une imagerie réussie à l’aide du cryoSIM. En plus des instructions sur le fonctionnement du microscope cryoSIM, des recommandations ont été incluses concernant le choix des échantillons, des fluorophores et des paramètres. Le protocole est démontré dans des échantillons de cellules U2OS dont les mitochondries et la tubuline ont été marquées par fluorescence.
Introduction
Les techniques d’imagerie SR sont devenues largement accessibles aux biologistes au cours de la dernière décennie1. Ils permettent l’imagerie à haute résolution d’échantillons marqués par fluorescence au-delà de la limite de diffraction. Cependant, il a été difficile d’adapter les méthodes de microscopie SR pour travailler avec des échantillons à des températures cryogéniques2. Cela serait avantageux pour l’imagerie corrélative en combinaison avec la tomographie par électrons ou rayons X. Récemment, sim a été adapté pour une utilisation avec des échantillons cryogéniques et a été démontré avec succès pour permettre des études corrélatives de cellules biologiques en conjonction avec la tomographie par rayons X mous (SXT)3 à la ligne de faisceau de cryo-imagerie corrélative B24 au synchrotron de source lumineuse Diamond (https://www.diamond.ac.uk/Instruments/Biological-Cryo-Imaging/B24.html). SIM peut doubler la résolution de la microscopie à grand champ conventionnelle en éclairant l’échantillon avec des motifs de lumière rayés (franges Moiré) à trois angles et en cinq phases. L’interférence entre ces motifs lumineux et la fluorescence de l’échantillon peut être utilisée pour découvrir informatiquement des informations supplémentaires sur les structures de sous-diffraction4,5.
Il existe plusieurs avantages de la carte SIM par rapport aux autres techniques SR pour les applications cryogéniques. Tout d’abord, il peut fonctionner sans fluorophores clignotants spécialement conçus; les fluorophores conventionnels peuvent être utilisés, donnant accès à une gamme plus large d’agents de marquage fluorescents potentiels6. De plus, il ne nécessite que 15 images par tranche z (en 3D; 9 images pour la 2D), alors que d’autres méthodes SR prennent environ 1000 images par tranche, ce qui augmente les chances que l’échantillon soit chauffé et augmente donc le risque de formation de cristaux de glace, ce qui peut causer des artefacts. Enfin, cette technique permet d’imager des échantillons biologiques plus épais de plus de 10 μm, permettant d’imager des cellules entières dans leur état quasi natif6. Le cryoSIM a été construit à l’aide de composants optiques standard et avec un logiciel en libre accès pour l’imagerie, ce qui facilite la documentation et la duplication si vous le souhaitez6. Le cryoSIM a un objectif à ouverture numérique 100x/0,9 (voir le Tableau des matériaux); Phillips et al.6 Ici, ce protocole montre comment utiliser le microscope cryoSIM, y compris comment charger et décharger des échantillons sur la scène cryogénique, comment collecter des données au microscope et comment reconstruire les images SIM.
Protocole
REMARQUE: Ce protocole concerne les échantillons contenant des cellules cultivées ou déposées sur des grilles d’or plates de 3 mm en microscopie électronique à transmission (TEM) avec un film de support de carbone troué qui ont été vitrifiées par congélation par plongeon ou congélation à haute pression. Ce protocole suppose que les échantillons ont déjà été imagés à l’aide d’un microscope conventionnel à épifluorescence et à champ clair pour cartographier les emplacements d’intérêt pour l’imagerie dans cryoSIM. Reportez-vous à la figure 1 pour une vue d’ensemble de l’ensemble du protocole.
1. Préparation de l’étape cryogénique
- Préparez de l’azote liquide libre de glace (LN2)en faisant passer LN2 à travers un entonnoir tapissé de n’importe quelle lingette scientifique à papier sec standard.
REMARQUE: Comme LN2 provoque des brûlures, portez un équipement de protection individuelle approprié, y compris des gants et des lunettes de cryo-protection, lorsque vous le manipulez. Ces liquides peuvent déplacer l’oxygène et provoquer une suffocation rapide et doivent être manipulés dans un endroit correctement ventilé. - Enlevez la poussière de surface de l’étage cryogénique avec de l’air sous pression. Expulser le liquide du récipient d’air sous pression avant de l’utiliser.
- Assurez-vous que la cartouche de chargement de l’échantillon est en place avec le porte-échantillon chambré approprié (vérifiez qu’il ne reste pas de grille dans le porte-échantillon d’une expérience précédente) (Figure 2).
- Retirez le couvercle du dewar externe de l’étage cryogénique et versez le LN2 filtré jusqu’à environ 1/4 de lacyème. Attendez que l’ébullition initiale disparaisse avant d’en verser plus; remplir le récipient à environ 2/3de remplissage. Remplacez soigneusement le couvercle en pointant la buse loin du manipulateur et de la scène/optique pendant que LN2 bout à partir de la sortie.
- Une fois que LN2 a cessé de sortir de la sortie, placez le tuyau de sortie au-dessus de l’étage dewar sur l’étage cryogénique.
- Branchez la source d’alimentation de la scène et connectez le câble USB à la cryo-scène. Assurez-vous que le couvercle de la chambre d’échantillonnage chauffée est branché. Branchez le dewar externe à la scène.
REMARQUE: Ne branchez pas le dewar externe tant que le tuyau de sortie n’a pas été positionné sur le dewar de l’étage sur le cryo-étage. Si LN2 déborde (ou pour l’empêcher de déborder), retirez le câble USB pendant environ 10 s, laissez-le s’équilibrer et reconnectez le câble USB pour réactiver le capteur. - Une fois que LN2 a été livré dans le dewar de scène, appuyez sur le bouton de déverrouillage de l’étage cryogénique pour permettre à LN2 d’entrer dans la chambre d’échantillonnage.
REMARQUE: Ne laissez pas le système sans surveillance pendant que LN2 remplit la chambre. - Attendez environ 30-45 minutes pour permettre au système de refroidir et de se stabiliser avant de commencer l’acquisition d’image. Vérifiez périodiquement le dewar externe et rechargez-le avec du LN2 filtré s’il est rempli de moins d’un quart (environ toutes les heures).
2. Transfert de la boîte de stockage de l’échantillon dans la cryo-étape
REMARQUE: Immergez la boîte de stockage de l’échantillon, le support et les extrémités de tout instrument (par exemple, les pinces) à l’intérieur du LN2 filtré pour les refroidir avant de toucher des surfaces froides telles que l’échantillon ou tout objet à l’intérieur de la chambre d’échantillonnage. Portez un manteau de laboratoire et des gants lorsque vous manipulez des échantillons biologiques.
- Assurez-vous que les échantillons vitrifiés sont dans un récipient cryocompatible et apportez-les au microscope. Appuyez sur le bouton correspondant de l’étage cryogénique pour allumer la lumière dans la chambre d’échantillonnage.
- Utilisez la touche hexadétienne de l’outil cassette pour ouvrir les deux plaques de la cassette de transfert d’échantillons. Ouvrez les plaques assez largement pour tomber dans la grille entre les deux plaques, mais évitez de les ouvrir à la position ouverte maximale pour éviter que la grille ne tombe de l’autre côté.
- Utilisez de longues pinces pour soulever la boîte de grille d’échantillon hors du LN2,tournez-la à l’endroit où l’encoche s’aligne sur la position de la position de stockage à l’intérieur de la scène et placez-la sur la scène. Utilisez l’appareil approprié (p. ex., un tournevis) pour ouvrir le couvercle de la boîte de rangement à la bonne position de l’échantillon.
- À l’aide d’une pince inversée (ou de toute pince chirurgicale à pointe fine), retirez la grille TEM du porte-échantillon, immergez-la à l’intérieur du LN2et près de la cassette de transfert d’échantillon, en restant proche du LN2 pendant le processus de transfert. Assurez-vous que le côté du film de carbone est placé de manière à ce qu’il soit finalement orienté vers l’objectif sur le pont d’échantillonnage.
- Fermez la cartouche d’échantillon à l’aide de la touche hexadétiste de l’outil cassette. Fermez et retirez la boîte de stockage ainsi que les échantillons restants.
- Utilisez le point magnétique de l’outil de cassette pour soulever et monter la cartouche contenant la grille sur le pont d’échantillonnage. Gardez-le immergé / proche du LN2 pendant le processus de transfert. Assurez-vous que l’orientation est appropriée pour le pont (les deux aimants entreront en contact avec les aimants sur le pont). Placez la cassette à plat dans les broches de positionnement du pont et poussez doucement pour vous assurer qu’elle est fixée.
REMARQUE: Une cassette d’échantillon pour les grilles clipsées qui ont été préparées pour le fraisage ultérieur par faisceau d’ions focalisé est également disponible à l’installation CryoSIM à la ligne de faisceau B24.
3. Amarrage et mise au point de la scène
- Déplacez l’ouverture du couvercle de l’étage cryogénique vers la position d’imagerie et éteignez la lumière de la chambre d’échantillonnage. Faites glisser la scène vers l’optique pour l’aligner sous l’objectif. Laissez doucement tomber l’objectif en position à l’aide du levier, en vous assurant qu’il repose dans le couvercle du cryo-étage, mais ne le touche pas.
REMARQUE: Assurez-vous que le tuyau de sortie externe de Dewar ne touche pas le dewar de scène sur le cryo-étage à aucun moment pendant la collecte de données. - Couvrez la scène et l’optique avec un rideau noir opaque. Démarrez le logiciel de contrôle Cockpit sur le PC cryoSIM (Figure 3 et Figure 4).
- Cliquez sur le bouton du mode de lecture pour chaque caméra et réglez-le sur CONV 3 MHz. Vérifiez que la température de chaque caméra est de -80 °C et que le ventilateur de la caméra est éteint.
- Allumez la caméra réfléchie. Sous Lumières,choisissez ambiant (exposition 20 ms), et sous linkam,cliquez sur condenseur. Cliquez sur le bouton Mode vidéo.
- Dans la fenêtre mosaïque, effectuez un zoom arrière (défilement de la souris) pour afficher le contour de la grille. Cliquez sur Localiser l’étape si elle ne peut pas être vue. Centrez la grille en double-cliquant avec le bouton gauche au milieu du cercle.
- Focalisation de l’échantillon jusqu’à ce que le film de support de grille (ou toute autre caractéristique pertinente de l’échantillon) soit mise au point, en utilisant les touches haut et bas pour déplacer l’étage cryogénique vers le haut et vers le bas, et en utilisant les touches 9 et 3 du pavé numérique pour changer le z-step (réglez-le sur 20 μm pour la mise au point initiale).
REMARQUE: Si l’utilisateur manque de déplacement en z, la scène peut être déplacée manuellement vers le haut ou vers le bas à l’aide de la roue sous la cryo-étape. Tournez-le d’un cran à la fois et vérifiez si l’échantillon se trouve dans la plage de vue. Modifiez à nouveau la direction si la mise au point s’aggrave.
4. Acquisition de mosaïques en champ clair
- Une fois la scène centrée, désactivez le mode vidéoet collectez une mosaïque de lumière visible (cliquez sur Exécuter la mosaïque dans la vue Mosaïque pour produire des tuiles d’images de lumière visible qui s’enroulent vers l’extérieur à partir du centre). Si la grille est pliée, essayez différentes positions sur la grille (double-cliquez avec le bouton gauche dans la vue Mosaïque), recentrez-vous et cliquez à nouveau sur Exécuter la mosaïque pour collecter des mosaïques partielles. Vous pouvez également déposer une image focale au-dessus de la mosaïque(Figure 3).
- Enregistrez la vue en cliquant sur Enregistrer la mosaïque. Donnez-lui un nom de fichier court contenant des informations sur la boîte de stockage et le numéro de grille respectif (un horodatage sera automatiquement ajouté au nom du fichier).
5. Identification des domaines d’intérêt
- Inspectez la mosaïque de champ lumineux à côté de toutes les images de « carte » de fluorescence précédentes pour savoir où les cellules ou les caractéristiques biologiques d’intérêt ont déjà été localisées. Vérifiez si ces zones produisent une fluorescence appropriée.
- Éteignez la lumière ambiante et le condenseur ainsi que le mode vidéo s’il est actif. Allumez le laser d’excitation requis (405, 488, 561 ou 647) et choisissez la caméra et le filtrecorrespondants, initialement à 50 mW, pour un temps d’exposition de 50 ms.
REMARQUE: Augmentez / diminuez ces paramètres de caméra et de filtre en fonction du signal de fluorescence. - Appuyez sur 0 pour prendre une image et sur * pour contraster automatiquement. Vous pouvez également ajuster manuellement le contraste à l’aide du curseur en bas de l’image.
REMARQUE: Allumez le panneau Laser on pour la pièce. - Une fois que des cellules biologiquement intéressantes avec une fluorescence appropriée ont été trouvées, marquez leurs positions à l’aide du bouton Marquer le site dans la vue Mosaïque.
REMARQUE: Ces sites marqués apparaîtront dans la liste ci-jointe et sont accessibles en double-cliquant sur les coordonnées. Lorsque vous revenez à un site marqué, effectuez un zoom sur la zone avant de double-cliquer. - Continuez à marquer tous les sites potentiels avant de commencer l’acquisition d’images. Sauvez à nouveau la mosaïque avec les sites marqués; cliquez sur Enregistrer les sites dans le fichier.
- Pour assembler les images de mosaïque à l’aide du logiciel StitchM (développé en interne à la ligne de faisceau B24), faites glisser et déposez le fichier de mosaïque .txt dans le fichier StitchM avec l’extension .bat et enregistrez l’image tiff combinée des carreaux de mosaïque dans le même dossier. Pour enregistrer une image avec les sites marqués, faites glisser et déposez la mosaïque.txt fichier et les marqueurs.txt fichier dans l’icône en même temps.
REMARQUE : Équilibrer la collecte de données par rapport aux besoins du projet (p. ex., si l’imagerie corrélative doit être effectuée, vérifier combien d’images peuvent être prises avec la modalité d’imagerie du partenaire et choisir le nombre approprié de sites pour l’imagerie cryoSIM). De plus, si le dewar externe nécessite un remplissage avec LN2 , le système cryo-étage changera de position et les sites marqués ne reviendront probablement pas aux mêmes endroits; tenez donc compte de cela lors du choix du nombre de sites à imager.
- Pour assembler les images de mosaïque à l’aide du logiciel StitchM (développé en interne à la ligne de faisceau B24), faites glisser et déposez le fichier de mosaïque .txt dans le fichier StitchM avec l’extension .bat et enregistrez l’image tiff combinée des carreaux de mosaïque dans le même dossier. Pour enregistrer une image avec les sites marqués, faites glisser et déposez la mosaïque.txt fichier et les marqueurs.txt fichier dans l’icône en même temps.
- Éteignez la lumière ambiante et le condenseur ainsi que le mode vidéo s’il est actif. Allumez le laser d’excitation requis (405, 488, 561 ou 647) et choisissez la caméra et le filtrecorrespondants, initialement à 50 mW, pour un temps d’exposition de 50 ms.
6. Stratégie de collecte de données
- Réglez le temps d’exposition laser en fonction des nombres dans la plage dynamique de l’image de fluorescence (en bas de la fenêtre des vues de la caméra). Choisissez le filtre à appliquer et optimisez ces paramètres pour chaque longueur d’onde de lumière d’excitation à utiliser, en allumant chaque laser séparément.
REMARQUE: Bien que 10 000 à 20 000 comptes soient optimaux, des comptes plus faibles sont acceptables s’il y a un bon contraste. Idéalement, vérifiez les paramètres de filtre avant que chaque pile d’images ne soit acquise, car les cellules situées à différentes zones de la grille peuvent avoir des niveaux de fluorescence variables.
7. Collecte de données
- Cliquez sur les deux caméras pour les allumer. Revenez à l’un des sites marqués et concentrez-vous à nouveau sur la profondeur souhaitée. Une fois au point dans une zone d’intérêt, déplacez-vous vers le haut (touche ↑) dans la fenêtre XY de la macro-étape pour choisir la hauteur de la pile z à acquérir, puis cliquez sur Enregistrer en haut. Déplacez-vous vers le bas ( touche↓), cliquez sur Enregistrer en bas, puis sur Aller au centre, et vérifiez que l’image est toujours au point.
REMARQUE: La hauteur de l’échantillon sera affichée dans la fenêtre. - Cliquez avec le bouton droit de la souris sur slm (modulateur de lumière spatial) dans la fenêtre Cockpit et assurez-vous que l’angle est réglé à 0,41. Dans la fenêtre Cockpit, sélectionnez Expérience sur un seul site.
REMARQUE: Ne cliquez pas sur le slm; Cockpit l’allumera automatiquement lors de l’acquisition d’images.- Dans la liste déroulante, sélectionnez Éclairage structuré. Modifiez la hauteur de la pile pour qu’elle soit égale à la hauteur z + 1 μm.
REMARQUE: L’ajout de 1 μm assure la capture de l’échantillon entier en z et minimise les artefacts de reconstruction. - Entrez les temps d’exposition (ms) pour les lasers 405 nm et 488 nm dans la rangée supérieure (pour la caméra réfléchie) et les temps d’exposition pour les lasers 561 nm et 647 nm dans la rangée inférieure (pour la caméra transmise).
REMARQUE: Les valeurs du temps d’exposition doivent correspondre aux valeurs décidées précédemment et affichées dans la fenêtre principale du cockpit. - Entrez un nom de fichier (convention de nommage : boîte number_grid area_filters_FL) (FL (fluorescence) ou BF (champ lumineux) selon le type d’imagerie en cours). Cliquez sur Mettre à jour pour produire un nouveau fichier contenant la date et l’heure sans écraser les fichiers précédents. Ensuite, cliquez sur Démarrer.
- Dans la liste déroulante, sélectionnez Éclairage structuré. Modifiez la hauteur de la pile pour qu’elle soit égale à la hauteur z + 1 μm.
- Vérifiez la vue Caméra pendant la collecte des données. Repaitez les images s’il y a un déplacement xy. Si le dewar LN2 remplit l’étage cryogénique lors de l’acquisition de l’image, abandonnez le processus en cliquant sur le bouton Abandonner du logiciel Cockpit.
- Une fois que le dewar externe a fini de remplir le dewar d’étage, répétez l’expérience car la recharge déplace l’échantillon verticalement. Recentrez l’image pour la recentrer en z et répétez l’expérience Sur un seul site. Répétez l’expérience sur un seul site pour toutes les combinaisons de lasers et de filtres nécessaires.
REMARQUE: Il faut environ 30 s-1 min pour terminer le remplissage. Lors de l’imagerie d’une grille, le remplissage aura lieu ~ 4-8x.
- Une fois que le dewar externe a fini de remplir le dewar d’étage, répétez l’expérience car la recharge déplace l’échantillon verticalement. Recentrez l’image pour la recentrer en z et répétez l’expérience Sur un seul site. Répétez l’expérience sur un seul site pour toutes les combinaisons de lasers et de filtres nécessaires.
- À chaque position, collectez une pile z à l’aide de la lumière visible.
- Éteignez les lasers et allumez la lumière ambiante et le condenseur. Sous Expérience sur un seul site,sélectionnez Z-stack, réglez la lumière ambiante sur une exposition de 20 ms et conservez la hauteur z comme ci-dessus.
- Répétez les étapes 7.2 à 7.4 pour tous les sites marqués sur la grille.
- Avant de passer à un autre exemple, supprimez la mosaïque en cliquant sur Supprimer les vignettes.
- Dessinez un carré autour de toutes les tuiles pour les supprimer. Supprimez également les marqueurs en les sélectionnant tous dans la liste, puis en cliquant sur Supprimer les sites sélectionnés.
- Éteignez la lumière ambiante et le condenseur avant de procéder au changement de grille. Suivez les étapes de la section 4 dans l’ordre inverse pour détacher l’étape de l’objectif et modifier la grille.
8. Après imagerie
- Une fois l’imagerie terminée, désamarrer la scène et retirer tous les échantillons. Éteignez le voyant de la chambre d’échantillonnage. Débranchez le dewar externe et décantez tout LN2 restant dans un autre récipient cryo-compatible, ce qui permet au dewar de revenir en toute sécurité à une température normale.
- Placez le bouchon du couvercle sur la cryo-scène. Attendez que l’option de cuisson de l’écran cryo-étage soit disponible après qu’il ne reste plus de LN2 dans le dewar de scène. Appuyez sur le bouton de cuisson pour entrer en mode chauffage et retirez toute humidité de la cryo-étape pour éviter la formation de glace.
9. Reconstruction
- Transférez les fichiers de données SIM brutes vers le poste de travail approprié pour la reconstruction. Exécutez le traitement par lots via la fenêtre Du générateur de tâches de traitement à l’aide de fonctions de transfert optique spécifiques au canal (OTFS) (calculées à partir de fonctions d’étalement de points de billes multifluorées) et d’angles K0 (0,29278, -1,8028, 2,3786) avec un filtre Wiener constant pour tous les canaux de 0,004 et un décalage de biais de 200.
- Dans le panneau Options supplémentaires, assurez-vous que les intensités négatives sont ignorées en ne cochant pas les autres options. Enregistrez les images SIR dans un dossier nommé "traité« .
REMARQUE: Les logiciels de reconstruction SIM commerciaux produisent généralement des données SIM reconstruites et conservent le nom du fichier, mais ajoutent SIR.dv à la fin. Un fichier journal contenant le protocole de traitement, les étapes et les informations statistiques sur la réussite de la reconstruction est également créé.
10. Correction chromatique du décalage
- Téléchargez le logiciel Chromagnon7 pour corriger le décalage chromatique.
- Utilisez la matrice de référence du décalage chromatique qui correspond aux longueurs d’onde de fluorescence des données collectées (fournies par la ligne de faisceau).
REMARQUE: Le fichier de référence est un fichier « chromagnon.csv », qui contient les paramètres d’alignement et a été obtenu à partir d’images d’étalonnage à l’aide de nanoparticules multifluorées. Il peut être utilisé pour traiter par lots plusieurs ensembles de données à la fois.- Choisissez le fichier de référence approprié qui correspond à la longueur d’onde laser et au filtre utilisés pour l’imagerie de l’échantillon, puis ajoutez-le au champ de référence. Ajoutez les données SIR reconstruites à aligner dans le champ source, puis cliquez sur Exécuter.
- Vérifiez que le signal de fluorescence est maintenant aligné dans les images. Pour le traitement par lots, placez toutes les images SIR à aligner dans le champ source et le fichier de référence dans le champ de référence, puis appuyezsur Exécuter tout .
Résultats
Un échantillon contenant des cellules U2OS a été coloré avec un mélange de colorant de cytosquelette de microtubules verts et de colorant de mitochondries rouges, ce qui a entraîné la coloration du composant microtubule du cytosquelette (vert) et des mitochondries (rouge). Des images ultérieures ont montré la localisation des mitochondries dans la cellule ainsi que la disposition des microtubules, mettant en évidence le cadre structurel qu’ils fournissent à la cellule et l’assemblage du cytosquelette autour d’organites tels que le noyau. La résolution dans cryoSIM est significativement plus élevée que celle en microscopie à épifluorescence standard(Figure 5). La figure 6 montre comment la « carte » fluorescente d’un microscope à épifluorescence conventionnel peut être utilisée pour localiser les zones d’intérêt pour l’imagerie et l’image reconstruite cryoSIM correspondante à partir d’un emplacement sur la grille.
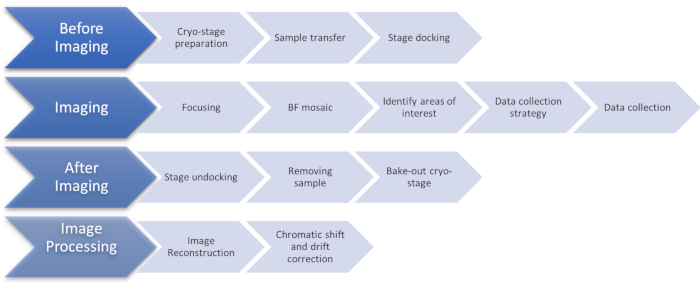
Figure 1: Organigramme montrant les étapes du protocole d’imagerie cryoSIM. Abréviation : cryoSIM = Cryo-structured illumination microscopy. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2: L’étage cryogénique. (A) La configuration du cryo-étage. (B) Grille d’échantillonnage représentée par des pinces inversées. (C) Composants de l’étage cryogénique. Les ports de connexion sont étiquetés, avec les couleurs correspondant à l’orange: alimentation, jaune: couvercle de scène chauffé, bleu: dewar externe, vert: connexion au PC. Abréviations : PC = ordinateur personnel; LN2 = azote liquide. Veuillez cliquer ici pour voir une version agrandie de cette figure.
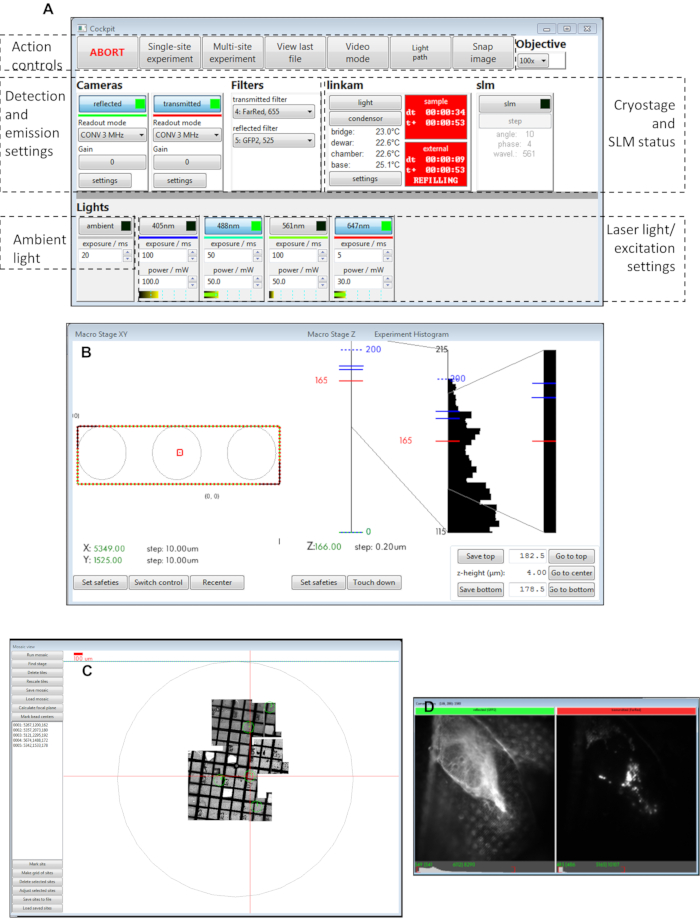
Figure 3: Vues des panneaux logiciels du cockpit. (A) Panneau principal, (B) macro étape XY, (C) vue mosaïque, (D) vues de la caméra. Abréviation : SLM = modulateur de lumière spatiale. Veuillez cliquer ici pour voir une version agrandie de cette figure.
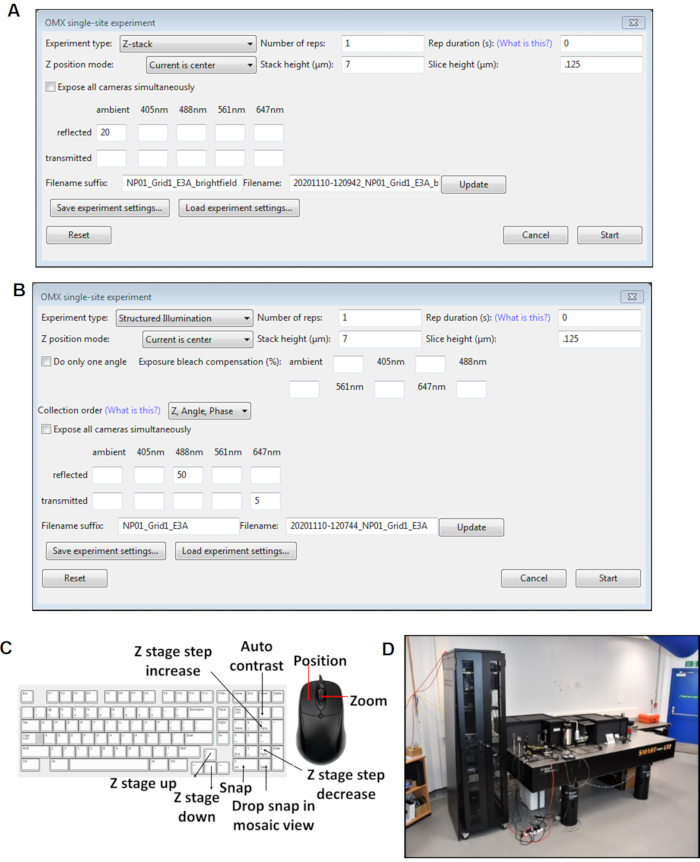
Figure 4: Vues des panneaux logiciels Cockpit. (A) Expérience sur un seul site de pile Z. (B) Expérience sur site unique SI. (C) Raccourcis clavier pour le logiciel de cockpit utilisé lors de l’acquisition d’images. (D) Le microscope cryoSIM est sur place à la ligne de faisceau B24 du synchrotron Diamond Light Source. Abréviation : cryoSIM = Cryo-structured illumination microscopy. Veuillez cliquer ici pour voir une version agrandie de cette figure.
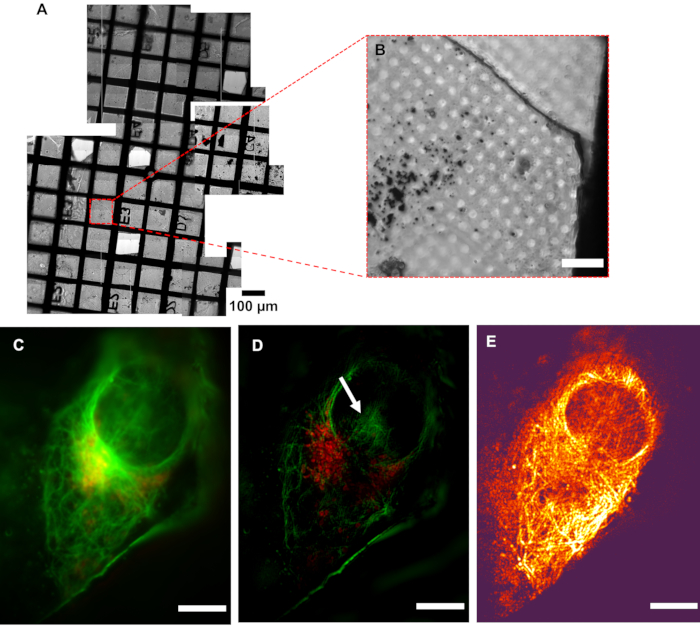
Figure 5: Résolution du cryoSIM. (A) Vue en mosaïque d’une grille à l’étude. (B) Image en champ lumineux d’une zone d’intérêt (AOI). (C) Image pseudo-grand champ par rapport à son (D) image SIM montrant l’augmentation de la résolution. La flèche blanche indique les artefacts de reconstruction SIM. (E) Carte de contraste de modulation combinant les informations d’intensité en pixels de l’image reconstruite avec les informations de couleur des valeurs respectives du rapport contraste/bruit de modulation (MCNR) des données brutes générées par SIMCheck2. Les régions claires et sombres présentent un contraste élevé et faible, respectivement. Barre d’échelle = 10 μm. Paramètres d’imagerie CryoSIM : longueurs d’onde d’excitation/émission : 488/525 nm, puissance laser de 50 mW, temps d’exposition de 50 ms et 647/655 nm, puissance laser de 20 mW, temps d’exposition de 5 ms. Abréviation : cryoSIM = Cryo-structured illumination microscopy. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 6: Reconstruction d’images dans cryoSIM à partir d’un emplacement sur la grille dans une carte de fluorescence à partir d’une carte d’épifluorescence conventionnelle. (A, B) Superposition de cartes d’images en champ clair et en fluorescence générées avec un microscope d’épifluorescence conventionnel. Cette carte est utilisée pour localiser les régions d’intérêt à imager ultérieurement dans cryoSIM. (C) L’image cryoSIM reconstruite obtenue à l’emplacement indiqué au point (B). Abréviation : cryoSIM = Cryo-structured illumination microscopy. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
La carte SIM 3D à des températures cryogéniques présente de nombreux avantages par rapport aux autres techniques d’imagerie SR pour l’imagerie de matériel biologique vitrifié. Il nécessite beaucoup moins d’images par tranche z par rapport aux autres méthodes SR, ce qui entraîne moins d’irradiation et moins de risque de formation de cristaux de glace pour les échantillons vitrifiés. Il est également capable d’imager des cellules entières et peut être corrélé avec la tomographie aux rayons X pour faire correspondre la structure à la fonction. Fait intéressant, la plupart des fluorophores et des étiquettes de fluorescence disponibles dans le commerce blanchissent moins dans des conditions cryogéniques qu’à température ambiante. Cependant, étant donné le rendement quantique élevé des fluorophores les plus courants à température ambiante (plus de 80% dans certains cas), le gain absolu détecté en photons n’est pas dû à des changements dans le rendement quantique, mais à une réduction des processus complexes de blanchiment. Plus d’informations sur le rendement des fluorophores à des températures cryogéniques peuvent être trouvées dans 8.
Il est essentiel que les échantillons arrivant au cryoSIM aient été prémélectionnés à l’aide d’un microscope à cryofluorescence conventionnel doté d’une capacité de champ lumineux pour produire une « carte » en grille qui comprend la mise en évidence de toutes les AOI potentielles pour une imagerie ultérieure(Figure 5). Le temps d’accès au cryoSIM est attribué via un processus concurrentiel qui implique la soumission d’une proposition, qui est ensuite évaluée pour la faisabilité de la technique et l’impact biomédical. Le temps passé à l’équipement est donc toujours « payant », et les grilles préappées permettent l’utilisation la plus efficace d’une allocation. Il est également essentiel que l’échantillon reste vitrifié, en particulier lors du transfert de l’échantillon du porte-échantillon à la plate-forme d’imagerie, afin de minimiser la formation de cristaux de glace et les dommages ultérieurs à l’échantillon. L’échantillon doit être de bonne qualité pour produire les meilleures images SIM. Un échantillon bien préparé sera caractérisé par les caractéristiques suivantes: (a) il n’aura pas de contamination par des cristaux de glace, (b) la grille utilisée sera une grille de recherche, (c) le support sera plat, (d) le maillage de la grille et la surface du substrat ne seront pas auto-fluorescents, et (e) il n’y aura pas de ruptures dans la membrane de support. Ces conditions préalables peuvent être obtenues en manipulant soigneusement les échantillons et en veillant à ce que les échantillons restent toujours vitrifiés.
Il est important de vérifier à l’avance si les fluorophores d’échantillon proposés donneront suffisamment de signal dans le microscope cryoSIM. Des outils tels que SPEKCheck9 peuvent aider à choisir les combinaisons optimales de fluorophores et de filtres. S’il y a des problèmes avec la collecte de données brutes ou le processus de reconstruction, des artefacts peuvent apparaître dans les images après la reconstruction. Des exemples de divers artefacts ont été documentés par Demmerle et al.10 Les paramètres de reconstruction d’image peuvent être examinés dans le fichier journal SoftWoRx si la reconstruction n’est pas optimale en ouvrant le fichier récapitulatif de reconstruction. Il doit y avoir un interligne cohérent entre les angles d’un canal donné et une amplitude relativement constante. Une variation de plus de 30 % et des valeurs significativement supérieures à 1 (si une compensation de la taille des billes est appliquée) devraient être étudiées de plus près et sont susceptibles d’indiquer l’échec des reconstructions. En outre, le logiciel SIMcheck2 aux Fidji peut également être utilisé pour effectuer diverses vérifications sur les données brutes et reconstruites afin de diagnostiquer la cause des erreurs dans les paramètres d’imagerie ou de reconstruction. SIM-check et sa carte de contraste de modulation peuvent également aider à évaluer la qualité des données reconstruites en interprétant quelles zones d’une image sont susceptibles d’être des structures réelles par rapport à des artefacts.
Un faible contraste de modulation (représenté par la couleur sombre, dans la figure 5E)dans la zone nucléaire signifie que cette région sera plus sensible aux artefacts de reconstruction, ce qui implique que les modèles de hachage montrés dans le noyau pourraient être classés(figure 5D)comme un artefact. Les zones de signal de fluorescence forte sont plus susceptibles de refléter avec précision les structures natives dans les données traitées. Dans les zones de signal faible où les fluorophores sont répartis sur des zones plus larges, telles que la surface totale d’une vésicule, il est probable que le signal réel coexiste avec les artefacts de traitement, et il convient de faire attention à l’interprétation de ces données. Après inspection des données reconstruites sur toute la gamme pour s’assurer qu’il n’y a pas d’artefacts étranges et que l’arrière-plan est généralement gaussien et centré près de zéro, les données sont généralement coupées à zéro, ou la valeur modale - le pic du signal de fond - devrait être très proche de zéro. Cela garantit que la plage dynamique de l’image affichée n’est pas dominée par des artefacts d’arrière-plan négatifs. Lorsqu’un signal plus faible est attendu, des précautions supplémentaires doivent être prises pour analyser les caractéristiques et s’assurer qu’il s’agit de structures réelles plutôt que d’artefacts de reconstruction.
Il existe certaines limites du système d’imagerie. Étant donné que l’étage de l’échantillon est plat, les échantillons d’épaisseur variable ou les grilles qui ne sont pas plates ne sont pas des sujets idéaux pour l’imagerie. De plus, si l’imagerie corrélative est effectuée à l’aide de la tomographie par rayons X mous, les cellules situées près des limites de la grille ne doivent pas être imagées car elles ne seront pas visibles au microscope à rayons X lors de l’acquisition de la série d’inclinaison. Enfin, la quantité de buvardage de l’échantillon avant la congélation en plongée a un impact significatif sur la qualité de l’imagerie; trop peu de buvardage entraîne des échantillons trop épais, ce qui donne des images SIM sous-optimales avec un bruit de fond élevé, tandis qu’un trop grand nombre de buvard peut rendre les cellules déformées et donc plus sensibles aux dommages causés par la chaleur du faisceau laser incident. En résumé, cryoSIM est un puissant outil de microscopie à fluorescence pour l’imagerie d’échantillons biologiques en 3D à un stade quasi natif et a de nombreuses applications dans de nombreux domaines.
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt financier concurrent.
Remerciements
Ce projet a reçu un financement du projet Horizon 2020 iNEXT-Discovery de la Commission européenne. I.M. Dobbie reconnaît le financement du Wellcome Trust (107457/Z/15/Z). Ce travail a été réalisé avec le soutien de la source lumineuse Diamond, instrument B24 (proposition BI25512). Nous remercions le personnel de Micron et tous nos excellents utilisateurs et collaborateurs de nous avoir aidés à établir le cryoSIM et son potentiel corrélatif.
matériels
| Name | Company | Catalog Number | Comments |
| Auto grids | FEI | ||
| Autogrids | Thermo Fisher Scientific | 1036173 | |
| BioTracker 488 Green Microtubule Cytoskeleton Dye | Sigma-Aldrich | SCT142 | |
| Cockpit Software | Oxford University | https://github.com/MicronOxford/cockpit | |
| cryo compatible polyurethane container | Jena bioscience | CC-FD-800 | |
| Cryo TEM grid storage box | Thermo Fisher Scientific | Model#AutoGrid | |
| cryo-SIM microscope | Custom made | N/A | Custom made, see following reference for the design: Michael A. Phillips, Maria Harkiolaki, David Miguel Susano Pinto, Richard M. Parton, Ana Palanca, Manuel Garcia-Moreno, Ilias Kounatidis, John W. Sedat, David I. Stuart, Alfredo Castello, Martin J. Booth, Ilan Davis, and Ian M. Dobbie, "CryoSIM: super-resolution 3D structured illumination cryogenic fluorescence microscopy for correlated ultrastructural imaging," Optica 7, 802-812 (2020). Has a Nikon TU Plan Apo 100x/0.9 NA. |
| Cryostage system | Linkam Scientific Instruments | CMS196 | |
| Fine tip surgical forceps | Ted Pella | 38125 | |
| MitoTracker Deep Red FM | Thermo Fisher Scientific | M22426 | |
| Python Microscope Software | Oxford University | https://www.python-microscope.org/ | |
| Scientific dry paper wipes | Kimberly-Clark 7551 | 2 | |
| SIM Reconstruction Software | softWoRx, GE Healthcare | Version 6.5.2 | |
| StitchM Software | Diamond Light Source | https://github.com/DiamondLightSource/StitchM | |
| TEM grids for samples | Quantifoil Micro Tools | G200F1 |
Références
- Schermelleh, L., et al. Super-resolution microscopy demystified. Nature Cell Biology. 21 (1), 72-84 (2019).
- Ball, G., Demmerle, J., Kaufmann, R., Davis, I., Dobbie, I. M., Schermelleh, L. SIMcheck: A toolbox for successful super-resolution structured illumination microscopy. Scientific Reports. 5 (1), 15915 (2015).
- Kounatidis, I., et al. 3D Correlative cryo-structured illumination fluorescence and soft X-ray microscopy elucidates reovirus intracellular release pathway. Cell. 182 (2), 515-530 (2020).
- Gustafsson, M. G. L. Surpassing the lateral resolution limit by a factor of two using structured illumination microscopy. Journal of Microscopy. 198 (2), 82-87 (2000).
- Rego, E. H., Shao, L., Rego, E. H. Practical structured illumination microscopy. Methods in Molecular Biology. 1251, 1251 (2015).
- Phillips, M. A., et al. CryoSIM: super-resolution 3D structured illumination cryogenic fluorescence microscopy for correlated ultrastructural imaging. Optica. 7 (7), 802 (2020).
- Matsuda, A., Schermelleh, L., Hirano, Y., Haraguchi, T., Hiraoka, Y. Accurate and fiducial-marker-free correction for three-dimensional chromatic shift in biological fluorescence microscopy. Scientific Reports. 8, 7583 (2018).
- Kaufmann, R., Hagen, C., Grünewald, K. Fluorescence cryo-microscopy: current challenges and prospects. Current Opinion in Chemical Biology. 20, 86-91 (2014).
- Phillips, M. A., Pinto, D. M. S., Dobbie, I. M. SPEKcheck-fluorescence microscopy spectral visualisation and optimisation: a web application, javascript library, and data resource. Wellcome Open Research. 3, 92 (2018).
- Demmerle, J., et al. Strategic and practical guidelines for successful structured illumination microscopy. Nature Protocols. 12 (5), 988-1010 (2017).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon