Method Article
Caracterización rápida de partes genéticas con sistemas libres de células
En este artículo
Resumen
Las partes genéticas bien caracterizadas son necesarias para el diseño de nuevos circuitos genéticos. Aquí describimos un método rentable y de alto rendimiento para caracterizar rápidamente las partes genéticas. Nuestro método reduce el costo y el tiempo al combinar lisados libres de células, ADN lineal para evitar la clonación y manejo acústico de líquidos para aumentar el rendimiento y reducir los volúmenes de reacción.
Resumen
La caracterización y catalogación de partes genéticas es fundamental para el diseño de circuitos genéticos útiles. Tener partes bien caracterizadas permite el ajuste fino de los circuitos genéticos, de modo que su función resulte en resultados predecibles. Con el crecimiento de la biología sintética como campo, ha habido una explosión de circuitos genéticos que se han implementado en microbios para ejecutar funciones relacionadas con la detección, la alteración metabólica y la computación celular. Aquí, mostramos un método rápido y rentable para caracterizar partes genéticas. Nuestro método utiliza lisado libre de células, preparado internamente como medio para evaluar partes a través de la expresión de una proteína reportera. El ADN de plantilla se prepara mediante amplificación por PCR utilizando cebadores de bajo costo para agregar partes variantes al gen reportero, y la plantilla se agrega a la reacción como ADN lineal sin clonación. Las partes que se pueden agregar de esta manera incluyen promotores, operadores, sitios de unión de ribosomas, aislantes y terminadores. Este enfoque, combinado con la incorporación de un manipulador de líquidos acústico y placas de 384 pocillos, permite al usuario llevar a cabo evaluaciones de alto rendimiento de partes genéticas en un solo día. En comparación, los enfoques de detección basados en células requieren una clonación que consume mucho tiempo y tienen tiempos de prueba más largos debido a los pasos nocturnos de normalización del cultivo y la densidad de cultivo. Además, trabajar en lisado libre de células permite al usuario ejercer un control más estricto sobre las condiciones de expresión mediante la adición de componentes exógenos y ADN en concentraciones precisas. Los resultados obtenidos del cribado libre de células se pueden utilizar directamente en aplicaciones de sistemas libres de células o, en algunos casos, como una forma de predecir la función en células enteras.
Introducción
Un esfuerzo central de la biología sintética es desarrollar kits de herramientas genéticas que contengan partes bien caracterizadas, que puedan usarse para construir circuitos genéticos1 que lleven a cabo funciones útiles cuando se despliegan en microbios o lisados libres de células. Las áreas en las que tales circuitos genéticos han ganado tracción son la detección 2,3,4, el rendimiento humano5,6, los biocombustibles 7,8, la producción de materiales 9,10 y la computación celular 11. Se han establecido registros de partes genéticas estandarizadas12 para catalogar partes nuevas y existentes en categorías tales como promotores, operadores, secuencias de codificación y terminadores, por nombrar solo algunos. Esfuerzos como el concurso internacional de máquinas modificadas genéticamente (iGEM (International Genetically Engineered Machines)13 han sido fundamentales para caracterizar y catalogar estas partes genéticas. Muchos métodos han sido desarrollados para facilitar el ensamblaje rápido de estas partes en circuitos genéticos útiles14,15. Incluso se ha desarrollado un software para automatizar la composición de piezas bien caracterizadas en circuitos que logran una función deseada16. Sin embargo, el ensamblaje de circuitos genéticos útiles con funciones predecibles se basa en la presunción de que los kits de herramientas genéticas contienen partes genéticas bien caracterizadas. Debido a la necesidad de estos kits de herramientas hacia el avance de la biología sintética, se han descrito numerosos esfuerzos para catalogar mejor los circuitos y las piezas con datos de caracterización apropiados 17,18,19,20,21.
Un enfoque para caracterizar los componentes genéticos hace uso de sistemas de síntesis de proteínas libres de células (CFPS), que reconstituyen funciones celulares como la transcripción y la traducción ex vivo22. Varios estudios han demostrado el potencial de CFPS para la creación de prototipos de componentes genéticos 23,24,25,26,27,28,29,30,31,32 ya sea para aplicaciones directas en sistemas libres de células o para predecir la función de construcciones genéticas en células, como la actividad relativa de partes dentro de una biblioteca 29 , optimización de la vía metabólica27 y carga celular30. Las ventajas de la creación de prototipos en CFPS frente a las células destacadas por estos estudios incluyen evitar la clonación que consume mucho tiempo, un control preciso sobre la concentración de ADN y otros componentes de la reacción, y la capacidad de mezclar y combinar fácilmente múltiples construcciones de ADN. La ventaja de evitar la clonación es especialmente evidente cuando se utilizan plantillas de ADN lineal, lo que permite ensamblar nuevas construcciones mediante métodos in vitro que toman horas en lugar de días33. La capacidad de manipular la concentración de construcciones de ADN y otros componentes simplemente pipeteando hace que el enfoque sea aún más atractivo al permitir la experimentación de alto rendimiento impulsada por robots de manejo de líquidos34,35. Si bien se han reportado éxitos en el uso de CFPS para la creación de prototipos, es importante tener en cuenta que queda por ver en qué contextos los resultados de CFPS pueden predecir de manera confiable la funcionalidad en las células.
Aquí, presentamos un método para la creación de prototipos CFPS que enfatiza las ventajas en velocidad, rendimiento y costo en comparación con los enfoques tradicionales basados en células. El enfoque se deriva de nuestro trabajo anterior en el que utilizamos CFPS para caracterizar rápidamente una biblioteca de variantes de promotores T7 reguladas por el factor de transcripción TetR32, ampliando significativamente el pequeño puñado de variantes reguladas de promotores T7 que estaban disponibles en la literatura en ese momento36,37. Desde entonces, otros han ampliado aún más la gama de tales promotores38. En nuestro método, el ensamblaje de la construcción genética se acelera mediante el uso de PCR para amplificar el ADN de la plantilla a través de cebadores que agregan partes genéticas variantes a un gen reportero. El manejo acústico de líquidos en placas de 384 pocillos se utiliza para aumentar el rendimiento y disminuir el volumen de materiales requeridos. Trabajos anteriores han demostrado el uso exitoso del manejo acústico de líquidos en volúmenes significativamente más bajos39,40 con una variabilidad comparable al pipeteo manual de volúmenes más grandes 41. Además del método, proporcionamos información de solución de problemas y una evaluación de posibles ahorros de costos y tiempo. Tenga en cuenta que si bien incluimos un protocolo para producir lisados libres de células basado en Sun et al.42 aquí, muchos otros kits y protocolos comerciales43,44 también deberían funcionar. Del mismo modo, mientras demostramos el método para la caracterización de las variantes promotoras32, otras partes pueden intercambiarse mediante amplificación por PCR, como riboreguladores, sitios de unión a ribosomas (RBS), aisladores, etiquetas de proteínas y terminadores. Esperamos que esta metodología pueda ayudar a la comunidad de biología sintética a continuar aumentando el número de piezas caracterizadas para el ensamblaje de circuitos genéticos predecibles con función útil.
Protocolo
1. Preparación del extracto celular
- Preparación de medios
- Para medios 2xYT: Agregue 16 g de triptona, 10 g de extracto de levadura y 5 g de NaCl a 900 ml de agua desionizada y ajuste el pH a 7.0 con 5 M NaOH. Elevar el volumen de la solución a 1 L con agua desionizada y esterilizar en autoclave o filtro. Alternativamente, compre medios 2xYT.
- Para tampón S30B: Prepare una solución de 14 mM de Mg-glutamato, 60 mM de K-glutamato y 5 mM de Tris en 2 L de agua desionizada. Utilice 2 M Tris para ajustar el pH a 8.2, autoclave y almacenar a 4 °C. Complete la solución agregando ditiothreitol (DTT) a una concentración final de 1 mM justo antes de su uso.
- Preparación de células
- Rayar células Rosetta2 de Escherichia coli BL21(DE3) u otra línea celular de elección (ver Cole et al.44 para una revisión exhaustiva reciente) en una placa de agar LB (caldo de lisogenia) e incubar a 37 °C durante 10-14 h.
- Utilice una sola colonia de E. coli para inocular 3 ml de medio 2xYT en un tubo de cultivo de 10 ml. Incubar este tubo a 37 °C agitando a 250 rpm durante 8 h.
- Utilizar 50 μL del cultivo de 3 ml para inocular 50 ml de medio 2xYT en un matraz de 500 ml. Incubar este matraz a 37 °C agitando a 250 rpm durante 8 h.
- Utilice 7,5 ml del cultivo de 50 ml para inocular cada uno de los cuatro matraces deflectores de 4 L que contienen 0,75 L de medio 2xYT. Incubar estos matraces a 37 °C con agitación a 220 rpm hasta que hayan alcanzado una densidad óptica a 600 nm de 2 a 4, después de aproximadamente 3-4 h.
- Recolectar las células de cada matraz transfiriéndolas a recipientes de 1 L y centrifugando a 5.000 g durante 12 min a 4 °C. Desechar el sobrenadante decantándolo en un contenedor de residuos.
- Lave cada pellet de celda con 150 ml de tampón S30B helado resuspendiéndolos completamente con una pipeta para interrumpir la masa celular, y luego recoja las células nuevamente por centrifugación a 5,000 x g durante 12 min. Deseche el sobrenadante.
- Lave cada pellet de celda nuevamente en 40 ml de tampón S30B helado resuspendiéndolos completamente e interrumpiendo la masa celular con una pipeta. Transfiera las células a tubos cónicos de 50 ml previamente pesados y recoja las células de nuevo por centrifugación a 2.000 x g durante 8 min a 4 °C. Desechar el sobrenadante por decantación.
- Pesar los gránulos de células húmedas. Congelar rápidamente los gránulos de células colocando los tubos directamente en nitrógeno líquido y almacenar a -80 °C.
- Lisis celular
- Descongele los gránulos celulares en hielo.
- Resuspender cada pellet celular en 1,4 ml de tampón S30B por 1 g del pellet celular mediante vórtice.
- Lise las células mediante una célula de presión francesa a 640 psi a 4 °C. Recoger el lisado en tubos de microcentrífuga sobre hielo y añadir 3 μL de 1 M DTT por 1 ml de lisado inmediatamente después de la lisis.
NOTA: Es mejor golpear la válvula de liberación de prensa francesa con una pequeña varilla de metal para mantener una presión uniforme y evitar caídas repentinas de presión. - Eliminar el lisado por centrifugación a 30.000 x g durante 30 min a 4 °C y desechar el pellet después de pipetear el sobrenadante a un nuevo tubo de microcentrífuga helada, teniendo cuidado de no interrumpir el pellet.
- Centrifugar el sobrenadante por segunda vez a 30.000 x g durante 30 min a 4 °C. Pipetear el sobrenadante resultante en un tubo de microcentrífuga helado. Deseche el pellet.
- Incubar el sobrenadante en un baño maría a 37 °C durante 1 h.
- Limpiar el sobrenadante por centrifugación a 15.000 x g durante 15 min a 4 °C y transferir el sobrenadante resultante a un tubo de microcentrífuga helada, teniendo cuidado de no interrumpir el pellet.
- Centrifugar el sobrenadante por segunda vez a 15.000 x g durante 15 min a 4 °C y transferir el sobrenadante resultante a un tubo de microcentrífuga helado, teniendo cuidado de no interrumpir ningún pellet restante.
- Distribuir el sobrenadante en alícuotas de 100 μL en tubos de microcentrífuga de 1,5 ml y congelarlos rápidamente colocándolos directamente en nitrógeno líquido. Conservar el sobrenadante a -80 °C.
2. Preparación lineal de plantillas
- Diseño de imprimación
- Elija una secuencia central como plantilla de PCR. Incluya como mínimo una secuencia reportera, como sfGFP (supercarpeta Green Fluorescent Protein), LacZ o Spinach aptamer. Incluya otras partes que se fijarán en las variantes filtradas, como terminators, promotores o RBS, según corresponda para el diseño.
NOTA: La inclusión de un terminador no siempre es necesaria para la expresión de ADN lineal en sistemas libres de células. - Para los cebadores directos, elija un mínimo de 20 pb que coincida con el extremo de 5' de la secuencia del núcleo como el extremo de 3' del cebador. Si agrega partes al extremo 5' de la construcción, diseñe el resto del extremo 5' del cebador para agregar las partes genéticas de interés a la secuencia central mediante amplificación por PCR (Figura 1A y Figura 2).
NOTA: Dado que los cebadores por encima de ~ 60 pb con frecuencia aumentan dramáticamente en costo, se pueden diseñar múltiples cebadores superpuestos para agregar secuencias más largas o múltiples partes. Si bien se pueden usar múltiples cebadores en una sola reacción de PCR, se recomienda realizar múltiples rondas de PCR. - Para los cebadores inversos, elija un mínimo de 20 pb para que coincida con el extremo 3' de la secuencia del núcleo como el extremo 3' del cebador. Si se agregan partes al extremo 3' de la construcción, diseñe el resto del extremo 5' del cebador para agregar las partes genéticas de interés a la secuencia central mediante amplificación por PCR (Figura 1A y Figura 2). Asegúrese de que la temperatura de recocido del cebador inverso esté dentro de los 5 °C de la temperatura de recocido de todo el cebador delantero.
- Elija una secuencia central como plantilla de PCR. Incluya como mínimo una secuencia reportera, como sfGFP (supercarpeta Green Fluorescent Protein), LacZ o Spinach aptamer. Incluya otras partes que se fijarán en las variantes filtradas, como terminators, promotores o RBS, según corresponda para el diseño.
- Amplificación lineal de plantillas
- Determine el número de reacciones de PCR que se realizarán en función del número de secuencias de núcleos y calcule la cantidad de cada componente requerido utilizando la Tabla 1.
- Prepare la mezcla maestra de acuerdo con la Tabla 1 y guárdela en hielo. Alícuota 30 o 40 μL (ver Tabla 1) de la mezcla maestra en el número determinado de tubos de PCR y añadir 10 μL de cada cebador variable (es decir, cebadores que codifican un cambio de pieza, ver Tabla 1) a 5 μM a tubos de PCR debidamente etiquetados.
- Coloque los tubos de PCR en el termociclador y ejecute el siguiente programa de PCR: 98 °C durante 3 min; 30 ciclos de 98 °C durante 15 s, XX °C durante 20 s, 72 °C durante YY min; Ampliación final a 72 °C durante 10 min. A continuación, mantenga la reacción a 4 °C.
Donde, XX representa la temperatura de recocido para el cebador con la temperatura de recocido más baja y YY representa el tiempo de extensión calculado para la longitud del amplicón basado en las recomendaciones del fabricante para la polimerasa de alta fidelidad utilizada. Optimice estas condiciones según sea necesario para diferentes imprimaciones y/o plantillas. - (Opcional) Agregue 1 μL de enzima de restricción DpnI para digerir la plantilla original. Incubar la reacción a 37 °C durante 1 h. Realice este paso solo si la plantilla original es ADN plásmido.
- Analizar 5 μL de cada producto de PCR mediante electroforesis en gel. Separar el producto con un gel de agarosa al 1% a 180 V durante 20 min. Compruebe el tamaño de banda correcto, que variará con la secuencia de núcleo elegida y la longitud de las piezas añadidas.
- Purifique la plantilla lineal utilizando un kit de purificación de PCR comercial o con el método de limpieza de PCR preferido. Si hubo múltiples bandas presentes mediante el análisis de electroforesis en gel, optimice las condiciones de PCR o purifique las bandas de peso molecular correctas utilizando un kit de extracción de gel comercial según la recomendación del fabricante.
- Cuantifique cada plantilla de ADN usando un espectrofotómetro. Evalúe la calidad de la plantilla de ADN comprobando que la relación de 260 nm/280 nm sea de aproximadamente 1,8.
- (Opcional) Nuevamente separe una porción de la plantilla de ADN usando un gel de agarosa al 1% a 180 V durante 20 minutos y asegúrese de que las bandas no deseadas se eliminen durante la purificación de la plantilla.
- Utilice plantillas de ADN purificado inmediatamente o guárdelas a -20 °C.
3. Preparación de proteínas purificadas
- Expresión de proteínas
- Para cada proteína que se exprese, ensamble una construcción de expresión apropiada. Codón-optimizar el gen para la expresión en E. coli. Insertar el gen en un vector de expresión de pET-22b u otro vector de expresión apropiado a través del método de ensamblaje de plásmidos preferido. Transformar el plásmido de expresión en células de expresión de Rosetta2 BL21(DE3) u otra línea celular apropiada.
- Para cada proteína, use una sola colonia para inocular 3 ml de medio LB en un tubo de cultivo de 10 ml. Incubar estos tubos a 37 °C agitando a 250 rpm durante la noche.
- Inocular un matraz de 2 L que contenga 750 ml de medio LB con 1 ml de cultivo nocturno. Incubar estos matraces a 37 °C con agitación a 250 rpm hasta que alcancen un OD600 de 0,6-1,0.
- Inducir la expresión de proteínas añadiendo 0,75 ml de isopropil 1 M β-D-1-tiogalactopiranósido (IPTG) en agua a cada matraz y continuar incubando estos matraces a 37 °C agitando a 250 rpm durante 4 h.
- Cosechar las células de cada matraz, utilizando un frasco de centrífuga de 1 L, por centrifugación a 5.000 g durante 12 min. Deseche el sobrenadante.
- Transfiera los gránulos a un tubo cónico de 50 ml y pese cada pellet. Congele rápidamente las células en nitrógeno líquido y almacénelas a -80 °C o continúe con el paso 3.2.
NOTA: Se espera que 2-5 g de células por 0,75 ml resulten de este paso.
- Purificación de proteínas por cromatografía de columna de afinidad de níquel
- Prepare el tampón de lisis combinando 50 mM de Tris-Cl, 500 mM de NaCl y 5 mM de imidazol. Ajuste a pH 8.0.
- Prepare el tampón de lavado combinando 50 mM de Tris-Cl, 500 mM de NaCl y 25 mM de imidazol. Ajuste a pH 8.0.
- Prepare el tampón de elución combinando 50 mM de Tris-Cl, 500 mM de NaCl y 250 mM de imidazol. Ajuste a pH 8.0.
- Prepare el tampón de diálisis combinando 50 mM NaHPO4, 100 mM NaCl y 2% de DMSO. Ajustar a pH 7.5.
- Descongele los gránulos celulares colocando los tubos en agua a temperatura ambiente. Añadir 5 ml del tampón de lisis por 1 g del pellet celular y resuspender por vórtice.
- Lise las células por sonicación. Separe el homogeneizado de la celda para que no haya más de 30 ml por tubo cónico de 50 ml y mantenga cada tubo en hielo. Lise las células usando un sonicador con una sonda de 0,16 cm de diámetro en rondas de 15 s con roturas de 30 s, 10 veces.
NOTA: Evite la formación de espuma, ya que esto desnaturaliza la proteína. La formación de espuma se puede evitar manteniendo la punta del sonicador al menos 2/3 sumergida en el lisado mientras está operativo. Otros métodos de lisis celular además de la sonicación también son posibles44. - Eliminar el lisado por centrifugación a 15.000 x g durante 30 min a 4 °C y colocar el sobrenadante en un nuevo tubo cónico de 50 ml.
- Agregue 1 ml de resina de ácido nitrilotriacético (Ni-NTA) por cada 5 ml de sobrenadante. Divida el lisado celular/suspensión Ni-NTA de modo que no haya más de 36 ml por tubo cónico de 50 ml. Incubar a 4 °C en un rotador de tubo a 10 rpm durante 1 h.
- Cargue la resina decantando el lisado celular / suspensión Ni-NTA en una columna de cromatografía de volumen de lecho de 2 ml y recoja el eluyente si es necesario para su posterior análisis, de lo contrario, deseche. Lave la resina con 10 volúmenes de lecho de resina de tampón de lavado.
- Recolectar la proteína agregando tres volúmenes de lecho de resina de tampón de elución a la columna y concentrar el volumen a 1.5 ml usando un concentrador centrífugo con la membrana de corte de peso molecular apropiada para cada proteína.
- Dializar la proteína contra 2 L de tampón de diálisis a 4 °C durante 1 h. Dializar la proteína de nuevo contra 2 L de tampón de diálisis durante la noche a 4 °C.
- Cuantificar la proteína utilizando su coeficiente de extinción molar y absorbancia a 280 nm. Analice la pureza de la proteína separándola mediante electroforesis en gel de poliacrilamida de dodecil sulfato de sodio (SDS-PAGE). Almacenar la proteína a -80 °C.
4. Síntesis de proteínas libres de células
- Preparación de la mezcla de reacción CFPS
- Prepare la mezcla de suplementos siguiendo los pasos de preparación de solución de aminoácidos, preparación de solución energética y preparación de tampón en Sun et al.42. Conservar por separado o combinado a -80 °C en alícuotas. Asegúrese de que las concentraciones finales coincidan con las descritas en Sun et al.42 en la sección Ejecución experimental de una reacción TX-TL.
- Prepare un aditivo para proteger el ADN lineal de la degradación. Si utiliza GamS33,45, prepárese a través de los pasos de la sección 3 anterior, u obtenga de un proveedor comercial; Para otros enfoques, consultar la literatura correspondiente46,47,48. Alternativamente, utilice un sistema CFPS que no requiera aditivos49.
- Prepare la polimerasa T7, las proteínas represoras y otros aditivos siguiendo los pasos de la sección 3 anterior u obtenga de un proveedor comercial.
- Determine el número de reacciones CFPS a realizar y calcule la cantidad de cada componente requerido utilizando la Tabla 2. Modificar las concentraciones de los componentes, incluida la adición o eliminación de componentes según sea necesario, y ajustar la cantidad de agua de modo que el volumen final de cada mezcla de reacción sea siempre de 10 μL. Del mismo modo, modifique la mezcla maestra para facilitar la dispensación de otros componentes mediante el manejo acústico de líquidos según lo desee (consulte la sección Discusión ).
- Descongele todos los componentes en hielo y prepare una mezcla maestra mezclando cada componente como se calculó anteriormente. Mezclar bien todos los componentes con una pipeta. Preste mucha atención para evitar la precipitación, especialmente para la mezcla de aminoácidos. Mantenga la mezcla maestra en hielo.
- Enfriar una placa de 384 pocillos sobre hielo y distribuir la mezcla maestra en alícuotas de 9 μL en cada pocillo.
NOTA: Es posible distribuir estos componentes mediante la manipulación acústica de líquidos, aunque se debe tener cuidado para garantizar una dispensación adecuada (consulte la sección Discusión para la solución de problemas).
- Distribución de componentes adicionales por manipulación acústica de líquidos
- Calcule la cantidad de proteína represora (y otros componentes opcionales) requerida para todas las reacciones CFPS.
- Descongele la proteína represora en hielo y distribúyala en una placa fuente de manejo de líquido acústico u otra placa apropiada. Asegúrese de que se incluye la cantidad adecuada de volumen muerto requerido para el tipo de placa de origen utilizada.
- Distribuir la proteína represora en volúmenes de 1 μL en los pocillos apropiados a través del manipulador de líquidos. Para obtener más información sobre la solución de problemas de distribución, consulte la sección Discusión .
- Curvas estándar
- Incluir en la placa una dilución seriada del informador purificado (ver sección 3 para la purificación de proteínas)41 o la norma química50 apropiada para permitir la comparación de los resultados con otros estudios y otros laboratorios. Elija un rango de concentraciones apropiado para el informador utilizado y el rango de expresión esperado de los experimentos.
- Ejecución de reacciones CFPS
- Precaliente el lector de placas a 30 °C. Configure el lector de placas para que lea en la configuración apropiada para el reportero utilizado en la secuencia central sin agitar los pasos.
NOTA: Mientras que 30 ° C se utiliza aquí, 29 ° C y 37 ° C también se utilizan comúnmente y funcionan bien con este protocolo. Se pueden preferir otras temperaturas para preparaciones alternativas de reacción libre de células. Para intervalos de lectura, 10 minutos es suficiente para lograr una buena resolución para los datos representativos presentados aquí; sin embargo, otras resoluciones pueden ser mejores dependiendo de la proteína reportera y la receta particular de CFPS. - (Opcional) Ejecute primero una reacción de prueba para establecer la ganancia o la configuración de sensibilidad adecuadas para capturar el cambio en la fluorescencia sin desbordamiento de señal.
- Selle la placa de 384 pocillos con una tapa impermeable de plástico sellable para evitar la evaporación. Si es posible, en el instrumento, ajuste un gradiente de temperatura vertical de 1 °C para limitar la condensación en el sello. Coloque la placa de 384 pocillos en el soporte de la placa y comience a leer.
- Precaliente el lector de placas a 30 °C. Configure el lector de placas para que lea en la configuración apropiada para el reportero utilizado en la secuencia central sin agitar los pasos.
Resultados
Para demostrar la utilidad de nuestros métodos, presentamos resultados que describen los efectos de la proximidad de la secuencia tetO al promotor T7 en la regulación de la expresión impulsada por Rnap T7. Los resultados completos y sus implicaciones se pueden encontrar en el trabajo de McManus et al.32. El flujo de trabajo se describe en la figura 1. Se prepararon quince plantillas lineales, que varían solo en la distancia del promotor T7 en relación con la secuencia tetO , amplificando por PCR el reportero sfGFP con cebadores diseñados para agregar cada variante del promotor (Figura 2) como se describe en la sección 2 del protocolo. Los componentes y reacciones de la reacción CFPS se prepararon siguiendo el protocolo. La expresión de sfGFP se midió a partir de cada plantilla con una titulación de 12 concentraciones diferentes de la proteína TetR, por triplicado, utilizando un manipulador de líquidos acústico. A 36 reacciones CFPS por plantilla y 15 plantillas, se realizaron un total de 540 reacciones para todo el conjunto de combinaciones T7-tetO . Toda la evaluación se llevó a cabo en dos placas en dos lectores de placas. El análisis de estos datos mostró que el RNAP T7 regula a la baja la expresión impulsada por T7 por igual hasta 13 pb aguas abajo desde el inicio de la transcripción T7 (Figura 3). Este resultado tiene implicaciones para el diseño futuro de circuitos genéticos regulables impulsados por T7 al describir una ventana putativa para una represión efectiva de T7 por parte de otros represores. La comparación de los resultados del protocolo descrito aquí con el ADN preparado por clonación tradicional reveló una diferencia pequeña pero estadísticamente significativa en el grado de represión de TetR entre formatos. Planteamos la hipótesis de que la unión no específica de TetR al ADN vectorial podría explicar la diferencia observada. Los resultados experimentales mostraron que la adición de ADN vectorial lineal a las reacciones con ADN de plantilla lineal redujo la diferencia a significación no estadística, aunque no descartó contribuciones de otros factores, como las diferencias en la periodicidad de la hélice del ADN para formatos lineales vs. circulares, que, a su vez, podrían afectar la unión de TetR. Dependiendo de la aplicación, el uso de la plantilla lineal puede requerir una validación adicional.
Además, incluimos datos representativos sobre posibles problemas con la dispensación precisa mediante el manejo acústico de líquidos (Figura 4). Se utilizó una solución de solución salina tamponada con fosfato (PBS), pH 7,4 que contenía 0,25 mM de colorante tartrazina para evaluar dos métodos de programación de un manipulador de líquidos acústico para dispensar volúmenes >1 μL. Después de la dispensación de líquido, la placa de destino se selló y centrifugó a 1.500 x g durante 1 min, y la absorbancia a 425 nm se midió con un lector de placas. Se muestran resultados representativos de nueve experimentos y demuestran una dispensación más consistente en toda la serie de ocho pocillos de destino cuando la transferencia de 5 μL se divide en dispensaciones separadas de 1 μL. Sobre la base de estas observaciones, se recomienda que las transferencias >1 μL se dividan en transferencias múltiples de ≤1 μL. Consulte la sección Discusión para obtener más detalles sobre la solución de problemas de este importante aspecto del protocolo.
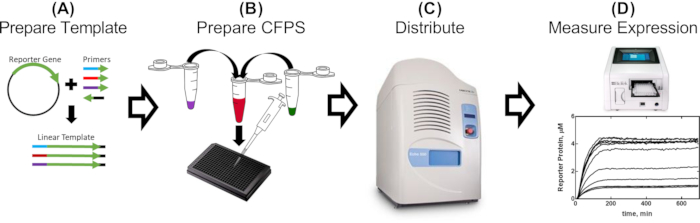
Figura 1: Flujo de trabajo de un solo día para la evaluación de partes promotoras en extracto libre de células. (A) Un informante es amplificado por PCR utilizando cebadores que contienen partes genéticas para ser evaluadas (2-5 h). (B) La mezcla de reacción libre de células se prepara como se detalla en el protocolo y se distribuye en una placa de 384 pocillos con las plantillas amplificadas por PCR (30 min). (C) El manejo acústico de líquidos se utiliza para distribuir componentes adicionales, que pueden incluir proteínas represoras, moléculas efectoras y cualquier otro efector condicional (10 min). (D) La expresión de la proteína reportera de cada reacción se mide en un lector de placas (2-16 h, dependiendo de la receta y la construcción de CFPS). Haga clic aquí para ver una versión más grande de esta figura.
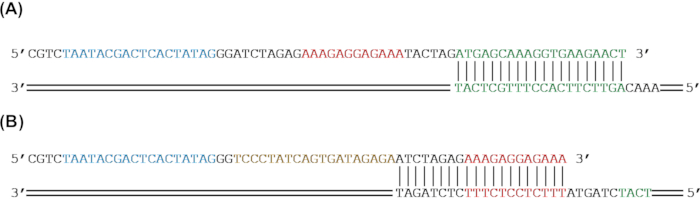
Figura 2: Diseño del cebador para agregar partes genéticas a un gen reportero mediante amplificación por PCR. (A) El gen reportero sfGFP (verde) se amplificará para agregar un RBS (rojo) y un promotor T7 (azul) por PCR. (B) El sfGFP (verde) y un RBS (rojo) se amplificarán para agregar una secuencia tetO (oro) y un promotor T7 (azul) por PCR. Haga clic aquí para ver una versión más grande de esta figura.
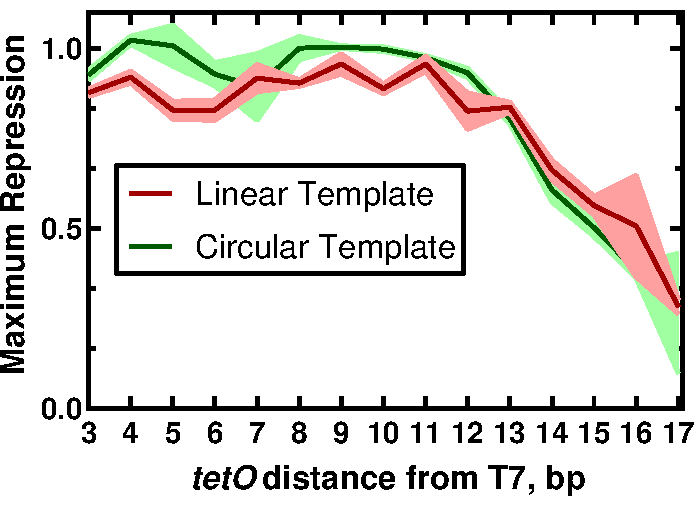
Figura 3: El efecto de la posición tetO en la regulación de una expresión impulsada por T7. Valores máximos de represión normalizados para plantillas lineales y circulares en función de la posición tetO . Las trazas representan la media y las desviaciones estándar para tres réplicas. Esta figura ha sido modificada de McManus et al.32 bajo una licencia Creative Commons CC-BY. Haga clic aquí para ver una versión más grande de esta figura.
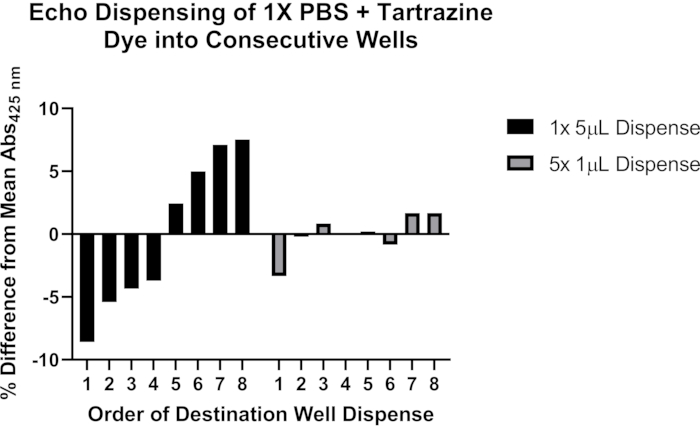
Figura 4: Uso de colorante tartrazina para validar la dispensación de líquidos con un manipulador de líquidos acústico. Las barras negras indican la dispensación de 5 μL de solución de tartrazina de un solo pocillo de fuente en cada uno de los ocho pocillos de destino consecutivos de una placa de 384 pocillos utilizando un solo comando de programación. Las barras grises indican la dispensación de 1 μL desde un solo pozo de fuente en cada uno de los ocho pozos de destino consecutivos utilizando un solo comando de programación, y luego repetir este paso cuatro veces para un total de 5 μL dispensados en cada pocillo de destino. Haga clic aquí para ver una versión más grande de esta figura.
| Nombre del componente | Volumen para 1 reacción (μL) | Volumen para el 110% del número X de reacciones (μL) |
| Premezcla Q5 PCR | 25 | |
| Agua | 4 | |
| Plantilla (1–3 ng/μL) | 1 | |
| (si se ha fijado1) Cebador directo (5 μM) | 0 o 10 | |
| (si se ha fijado1) Cebador inverso (5 μM) | 0 o 10 | |
| Master Mix Total: | 30 o 40 | |
| (si es variable1) Cebador directo (5 μM) | 0 o 10 | |
| (si es variable1) Cebador inverso (5 μM) | 0 o 10 |
Tabla 1: Hoja de trabajo para la preparación de reactivos para reacciones de PCR. Los valores de la columna situada más a la derecha pueden ser rellenados por los usuarios en función del número previsto de reacciones. 1 Los cebadores variables contienen una parte específica que se agregará en la reacción de PCR y pueden ser el cebador directo, el cebador inverso o ambos. Los cebadores fijos no agregan una pieza y pueden ser el cebador delantero o inverso, pero no ambos.
| Nombre del componente | Volumen para 1 reacción (μL) | Volumen para el 110% del número X de reacciones (μL) |
| Extracto celular | 4.2 | |
| Mezcla de suplementos | 3.3 | |
| Proteína GamS (207 μM) | 0.15 | |
| ADN plantilla (20 nM) | 1 | |
| T7 Polimerasa (13 mg/ml) | 0.12 | |
| Agua | 0.73 (este número puede variar) | |
| Master Mix Total: | 9 | |
| Proteína represora: | 1 |
Tabla 2: Hoja de trabajo para la preparación de reactivos para reacciones CFPS. Los valores de la columna situada más a la derecha pueden ser rellenados por los usuarios en función del número previsto de reacciones.
Cuadro suplementario. Haga clic aquí para descargar esta tabla.
Discusión
Los protocolos descritos aquí proporcionan un medio rentable y rápido para detectar partes genéticas a través de la expresión de una proteína reportera por CFPS. Las partes genéticas bien caracterizadas son cruciales para el diseño de circuitos genéticos predecibles con una función útil. Esta metodología aumenta el rendimiento y disminuye el tiempo necesario para detectar nuevas partes genéticas al eliminar el requisito de trabajar en células vivas, al tiempo que conserva la funcionalidad que refleja el entorno celular al retener el proceso metabólico de la expresión de proteínas en el lisado celular. Nuestro protocolo se puede realizar en 1 día después de recibir los cebadores (~ 2.5-6 h para la preparación de la reacción, 2-16 h para la reacción CFPS; Figura 1), en comparación con al menos 3 días para la clonación tradicional (1 día cada uno para el ensamblaje y transformación de constructos, la verificación de la secuencia de clones y el cultivo de células para la evaluación). Estimamos además que el costo por constructo utilizando ADN lineal es aproximadamente un tercio de la clonación tradicional ($78 vs. $237; Cuadro complementario 1) métodos. Los servicios de síntesis comerciales actualmente cotizan un mínimo de 4 días hábiles dependiendo del tamaño, aunque tendrían costos similares a nuestro método si los fragmentos lineales se criban directamente en CFPS ($ 78 vs. $ 91); No hemos verificado este enfoque. El costo de evaluar una pieza con CFPS es pequeño en comparación con la generación del ADN de la plantilla ($ 0.05 / reacción22 frente a $ 78 por plantilla), aunque debe tenerse en cuenta que los costos iniciales de reactivos a granel y equipos de lisis son de al menos varios miles de dólares. El uso de un manipulador de líquidos acústico solo mejora marginalmente los costos al permitir volúmenes más pequeños hasta 0.5 μL40; La ventaja más significativa es la reducción del tiempo para preparar las reacciones (~ 10 min frente a hasta 1 h, dependiendo del número de reacciones), especialmente cuando la preparación de un gran número de reacciones plantea preocupaciones de que la reacción preparada permanezca durante largos tiempos antes de la incubación.
Si bien es rápido y rentable, las limitaciones sobre cuándo la creación de prototipos CFPS predice adecuadamente la función in vivo aún están por verse. Por ejemplo, cualquier reactividad cruzada con el ADN genómico no se detectará debido a la eliminación del genoma del huésped durante la producción del sistema CFPS. Además, las concentraciones de componentes pueden ser 1-2 órdenes de magnitud más bajas en CFPS que en las células51, lo que probablemente afecte el comportamiento de algunas partes como resultado de diferentes condiciones de hacinamiento macromolecular. Además, la capacidad del ADN lineal para predecir la función in vivo puede ser limitada, por ejemplo, cuando la estructura secundaria del ADN juega un papel importante. Una limitación final es que las construcciones no se verifican en secuencia antes de probar las funciones. Puede haber casos en los que la parte caracterizada no esté realmente alineada con la secuencia teórica prevista. Todas estas limitaciones pueden mitigarse validando un subconjunto de las piezas examinadas por este método en la aplicación in vivo prevista.
Originalmente desarrollamos esta metodología para investigar los efectos del cambio de la posición del operador en promotores híbridos T7-tetO 32. Hemos presentado los protocolos aquí en un formato más genérico, de modo que se pueden aplicar a promotores, operadores, secuencias de unión a ribosomas, aislantes y terminadores. Estas partes genéticas se pueden agregar al extremo 5' o 3' del gen reportero mediante PCR utilizando cebadores para cada diseño, obviando la necesidad de síntesis o clonación de cada variante a probar. Los productos de PCR resultantes sirven como ADN de plantilla para la evaluación a través de la expresión de una proteína reportera. En nuestro trabajo, el protocolo de purificación de afinidad proporcionado aquí se utilizó para TetR y GamS. El mismo procedimiento se puede utilizar para la expresión y purificación de otros represores, activadores, polimerasas, factores sigma y otras proteínas afines a una parte genética de interés, aunque pueden ser necesarias modificaciones para la proteína deseada que se expresa. La purificación y titulación de estas proteínas en reacciones CFPS permite una caracterización más detallada de una parte genética particular. Por último, existen numerosos protocolos alternativos de CFPS y cada uno de ellos debería ser susceptible de la parte de selección de piezas de la metodología. Como ejemplo, no incluimos un paso de diálisis en este protocolo, que otros han encontrado importante para la expresión de promotores bacterianos nativos22. También es posible variar las concentraciones de los componentes constitutivos subyacentes de la PPC. El uso de la manipulación de líquidos mejora la capacidad de probar la miríada de condiciones al aumentar el rendimiento y disminuir los materiales requeridos34,35.
Un área que puede requerir una solución de problemas significativa es la optimización del manejador de líquidos acústico. La dispensación acústica del manipulador de líquidos debe optimizarse para cada componente que se transfiere y se recomienda encarecidamente ejecutar controles para verificar la distribución adecuada y la reproducibilidad antes de recopilar datos. El tipo de placa de origen ideal y la configuración de la clase de líquido dependerán del líquido específico que se dispensará y sus componentes. No se recomienda usar placas recubiertas de aminas para dispensar ADN, ya que el recubrimiento de amina puede interactuar con el ADN. También debe tenerse en cuenta que la capacidad de dispensar concentraciones más altas de ciertos componentes puede depender del modelo de manipulador de líquidos acústico. Se puede realizar una transferencia de líquido de prueba dispensando en un sello de placa de aluminio para visualizar la formación exitosa de gotas; Sin embargo, esta prueba proporciona información limitada y las gotas de diferentes configuraciones pueden parecer idénticas. El uso de un colorante soluble en agua, como la tartrazina, se puede utilizar para verificar con mayor precisión que se dispensa el volumen correcto con un entorno o flujo de trabajo determinado (consulte Resultados representativos). La programación óptima de las transferencias de líquidos también puede influir en la precisión y consistencia de los datos generados; para transferencias >1 μL de un pozo fuente a un pozo de destino, hemos encontrado que las transferencias secuenciales de ≤1 μL deben programarse para reducir la variabilidad sistemática de pozo a pozo (Figura 4). Por último, los volúmenes muertos de pozo fuente teóricos y reales pueden variar drásticamente según el tipo de placa fuente, la configuración de la clase de líquido y los componentes del líquido específico; El uso de la función de inspección acústica de manejo de líquidos para evaluar los volúmenes del pozo antes de ejecutar un programa puede ayudar a medir con qué precisión el instrumento puede medir un líquido en particular.
El rendimiento de la reacción CFPS puede variar al comparar los resultados entre diferentes usuarios, lotes de materiales, lectores de placas y laboratorios41. Para los casos en que se requieren tales comparaciones al crear prototipos de circuitos genéticos, recomendamos incluir reacciones de control interno con promotores constitutivos estándar en cada placa de reacción para ayudar a normalizar los resultados en configuraciones experimentales. El método de preparación del ADN también puede contribuir en gran medida a la actividad de CFPS; Se recomienda la inclusión de un paso de precipitación de etanol. Además, la composición óptima de la reacción puede variar según el lote de extracto34. Se ha demostrado que las concentraciones óptimas de glutamato de magnesio y glutamato de potasio, en particular, varían según el lote42 o con la proteína promotora o informadora utilizada24. Las concentraciones de estos componentes deben optimizarse mediante el cribado a través de varias concentraciones de cada componente por construcción genética y por preparación de extracto celular para determinar las condiciones óptimas para la expresión de proteínas. Finalmente, las mejores prácticas para un rendimiento de reacción CFPS consistente incluyen una mezcla completa, un pipeteo cuidadoso y consistencia en la preparación de cada componente del reactivo.
Más allá de la caracterización de partes individuales, el mismo método se puede utilizar para filtrar combinaciones de partes que forman circuitos complejos, como circuitos lógicos16 u osciladores52,53. Este método también se puede aplicar a la detección y optimización de biosensores para aplicaciones en diagnóstico epidemiológico 54,55,56,57 o detección y cuantificación de peligros 3,58,59. La aplicación de técnicas impulsadas por IA, como el aprendizaje activo34, también se puede combinar con la naturaleza de alto rendimiento de este método para impulsar la exploración rápida de espacios de diseño biológico complejos. En última instancia, prevemos que este enfoque apoye tiempos de desarrollo acelerados para nuevos diseños genéticos en biología sintética.
Divulgaciones
RMM tiene una participación financiera en Tierra Biosciences, una empresa privada que hace uso de tecnologías libres de células como las descritas en este artículo para la expresión y detección de proteínas.
Los otros autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue posible gracias al programa de Investigación Aplicada para el Avance de las Prioridades de Ciencia y Tecnología de la Oficina del Secretario de Defensa. Agradecemos a Scott Walper (Laboratorio de Investigación Naval) por proporcionar el stock de sfGFP utilizado, y a Zachary Sun y Abel Chiao (Tierra Biosciences) por las fructíferas discusiones relacionadas con la creación de prototipos con sistemas libres de células y la solución de problemas relacionados con el manejo de líquidos acústicos.
Materiales
| Name | Company | Catalog Number | Comments |
| 2x YT medium | Sigma-Aldrich | Y2377-250G | Alternative to making 2xYT media |
| Agar | Bacto | 214010 | For plating cells |
| Chromatography column (5 cm diameter) | BIO-RAD | 731-1550 | Used for protein purification. |
| Destination plate | Thermo Scientific Nunc plate | 142761 | For CFPS reactions |
| DMSO | Sigma-Aldrich | D2650 | For dialysis buffer |
| DpnI | NEB | R0176L | For digestion of plasmid templates |
| DTT | Roche | 20871723 | S30 Buffer B |
| E. coli BL21(DE3) Rosetta2 | Novagen | 70954 | Cell line used for production of lysate and purified proteins |
| Echo acoustic liquid handler | Labcyte | 525 | Acoustic liquid handler |
| French pressure cell | Thermo Spectronic | FA-078 | For lysing cells for CFPS |
| Imidazole | Sigma-Aldrich | 56750 | For buffers |
| Impermeable plastic sealable lid | Thermo | 232702 | Plate seal |
| IPTG | RPI | I56000-25.0 | Used for protein induction. |
| K-Glu | Sigma-Aldrich | g1501-500G | S30 Buffer B |
| Labcyte Echo source plate | Labcyte | PL-05525 | For use with Echo acoustic liquid handler |
| Mg-Glu | Sigma-Aldrich | 49605-250G | S30 Buffer B |
| NaCl | Sigma-Aldrich | S7653-250G | For buffers |
| NaHPO4 | Sigma-Aldrich | 71505 | For dialysis buffer |
| NaOH | Mallinckrodt Chemicals | 7708-10 | For making 2xYT media. Currently not produced by Mallinckrodt. Alternate: Sigma-Aldrich S0899 |
| Ni-NTA resin | Invitrogen | R901-15 | For production of purified proteins |
| PCR H2O | Ambion | AM9937 | PCR of linear templates |
| Plate Reader | BioTek | H10 | Plate reader used |
| Q5 PCR Master Mix | NEB | M0494S | PCR of linear templates |
| QIAquick Gel Extraction Kit | Qiagen | 28606 | PCR of linear templates |
| QIAquick PCR Purification Kit | Qiagen | 28004 | PCR of linear templates |
| QSonica Ultrasonic Processor | Qsonica | Q700 | Cell disruption during protein purification |
| RTS Amino Acid Sampler | biotechrabbit | BR1401801 | Updated supplier from Sun et al. |
| Tris | MP | 819623 | S30 Buffer B |
| Tris-Cl | Sigma-Aldrich | T5941 | For buffers |
| Tryptone | Fluka | T7293 | For making 2xYT media |
| Yeast Extract | Bacto | 212750 | For making 2xYT media |
Referencias
- Purnick, P. E. M., Weiss, R. The second wave of synthetic biology: from modules to systems. Nature Reviews. Molecular Cell Biology. 10 (6), 410-422 (2009).
- Saltepe, B., Kehribar, E. &. #. 3. 5. 0. ;., Su Yirmibeşoǧlu, S. S., Şafak Şeker, U. &. #. 2. 1. 4. ;. Cellular biosensors with engineered genetic circuits. ACS Sensors. 3 (1), 13-26 (2018).
- Bereza-Malcolm, L. T., Mann, G., Franks, A. E. Environmental sensing of heavy metals through whole cell microbial biosensors: A synthetic biology approach. ACS Synthetic Biology. 4 (5), 535-546 (2015).
- Mimee, M., et al. An ingestible bacterial-electronic system to monitor gastrointestinal health. Science. 360 (6391), 915-918 (2018).
- Isabella, V. M., et al. Development of a synthetic live bacterial therapeutic for the human metabolic disease phenylketonuria. Nature Biotechnology. 36 (9), 857-867 (2018).
- Healy, C. P., Deans, T. L. Genetic circuits to engineer tissues with alternative functions. Journal of Biological Engineering. 13, 39 (2019).
- Kaushik, A., Tiwari, S., Dev Jayant, R., Marty, A., Nair, M. Towards detection and diagnosis of Ebola virus disease at point-of-care. Biosensors and Bioelectronics. 75, 254-272 (2016).
- Jagadevan, S., et al. Recent developments in synthetic biology and metabolic engineering in microalgae towards biofuel production. Biotechnology for Biofuels. 11, 185 (2018).
- Keating, K. W., Young, E. M. Synthetic biology for bio-derived structural materials. Current Opinion in Chemical Engineering. 24, 107-114 (2019).
- Smanski, M. J., et al. Synthetic biology to access and expand nature's chemical diversity. Nature Reviews Microbiology. 14 (3), 135-149 (2016).
- Xiang, Y., Dalchau, N., Wang, B. Scaling up genetic circuit design for cellular computing: advances and prospects. Natural Computing. 17 (4), 833-853 (2018).
- Galdzicki, M., Rodriguez, C., Chandran, D., Sauro, H. M., Gennari, J. H. Standard biological parts knowledgebase. PLoS One. 6 (2), 17005 (2011).
- Kelwick, R., Bowater, L., Yeoman, K. H., Bowater, R. P. Promoting microbiology education through the iGEM synthetic biology competition. FEMS Microbiology Letters. 362 (16), (2015).
- Torella, J. P., et al. Unique nucleotide sequence-guided assembly of repetitive DNA parts for synthetic biology applications. Nature Protocols. 9 (9), 2075-2089 (2014).
- Halleran, A. D., Swaminathan, A., Murray, R. M. Single day construction of multigene circuits with 3G assembly. ACS Synthetic Biology. 7 (5), 1477-1480 (2018).
- Nielsen, A. K., et al. Genetic circuit design automation. Science. 352 (6281), (2016).
- Canton, B., Labno, A., Endy, D. Refinement and standardization of synthetic biological parts and devices. Nature Biotechnology. 26 (7), 787-793 (2008).
- Boehm, C. R., Bock, R. Recent advances and current challenges in synthetic biology of the plastid genetic system and metabolism. Plant Physiology. 179 (3), 794-802 (2019).
- Takahashi, M. K., et al. Characterizing and prototyping genetic networks with cell-free transcription-translation reactions. Methods. 86, 60-72 (2015).
- Chappell, J., Watters, K. E., Takahashi, M. K., Lucks, J. B. A renaissance in RNA synthetic biology: New mechanisms, applications and tools for the future. Current Opinion in Chemical Biology. 28, 47-56 (2015).
- Endy, D. Foundations for engineering biology. Nature. 438 (7067), 449-453 (2005).
- Silverman, A. D., Karim, A. S., Jewett, M. C. Cell-free gene expression: an expanded repertoire of applications. Nat. Reviews. Genetics. 21 (3), 151-170 (2020).
- Dopp, J. L., Rothstein, S. M., Mansell, T. J., Reuel, N. F. Rapid prototyping of proteins: Mail order gene fragments to assayable proteins within 24 hours. Biotechnology and Bioengineering. 116 (3), 667-676 (2019).
- Garamella, J., Marshall, R., Rustad, M., Noireaux, V. The all E. coli TX-TL toolbox 2.0: A platform for cell-free synthetic biology. ACS Synthetic Biology. 5 (4), 344-355 (2016).
- Kopniczky, M. B., et al. Cell-free protein synthesis as a prototyping platform for mammalian synthetic biology. ACS Synthetic Biology. 9 (1), 144-156 (2020).
- Kelwick, R., et al. Cell-free prototyping strategies for enhancing the sustainable production of polyhydroxyalkanoates bioplastics. Synthetic Biology. 3 (1), (2018).
- Karim, A. S., et al. In vitro prototyping and rapid optimization of biosynthetic enzymes for cell design. Nature Chemical Biology. 16 (8), 912-919 (2020).
- Takahashi, M. K., et al. Rapidly characterizing the fast dynamics of RNA genetic circuitry with cell-free Transcription-Translation (TX-TL) systems. ACS Synthetic Biology. 4 (5), 503-515 (2015).
- Chappell, J., Jensen, K., Freemont, P. S. Validation of an entirely in vitro approach for rapid prototyping of DNA regulatory elements for synthetic biology. Nucleic Acids Research. 41 (5), 3471-3481 (2013).
- Borkowski, O., et al. Cell-free prediction of protein expression costs for growing cells. Nature Communications. 9 (1), 1457 (2018).
- Dudley, Q. M., Karim, A. S., Nash, C. J., Jewett, M. C. In vitro prototyping of limonene biosynthesis using cell-free protein synthesis. Metabolic Engineering. 61, 251-260 (2020).
- McManus, J. B., Emanuel, P. A., Murray, R. M., Lux, M. W. A method for cost-effective and rapid characterization of engineered T7-based transcription factors by cell-free protein synthesis reveals insights into the regulation of T7 RNA polymerase-driven expression. Archives of Biochemistry and Biophysics. 674, 108045 (2019).
- Sun, Z. Z., Yeung, E., Hayes, C. A., Noireaux, V., Murray, R. M. Linear DNA for rapid prototyping of synthetic biological circuits in an escherichia coli based TX-TL cell-free system. ACS Synthetic Biology. 3 (6), 387-397 (2014).
- Borkowski, O., et al. Large scale active-learning-guided exploration for in vitro protein production optimization. Nature Communications. 11 (1), 1872 (2020).
- Caschera, F., et al. High-throughput optimization cycle of a cell-free ribosome assembly and protein synthesis system. ACS Synthetic Biology. 7 (12), 2841-2853 (2018).
- Iyer, S., Karig, D. K., Norred, S. E., Simpson, M. L., Doktycz, M. J. Multi-input regulation and logic with T7 promoters in cells and cell-free systems. PLoS One. 8 (10), 78442 (2013).
- Karig, D. K., Iyer, S., Simpson, M. L., Doktycz, M. J. Expression optimization and synthetic gene networks in cell-free systems. Nucleic Acids Research. 40 (8), 3763-3774 (2012).
- Jung, J. K., et al. Cell-free biosensors for rapid detection of water contaminants. Nature Biotechnology. 38 (12), 1451-1459 (2020).
- Bailey, J., Eggenstein, E., Lesnick, J. Miniaturization and rapid processing of TXTL reactions using acoustic liquid handling. Labcyte Technical Note. , 1-12 (2018).
- Marshall, R., Garamella, J., Noireaux, V., Pierson, A. High-throughput microliter-sized cell-free transcription-translation reactions for synthetic biology applications using the echo 550 liquid handler. Labcyte Application Note. , 1-6 (2018).
- Cole, S. D., et al. Quantification of interlaboratory cell-free protein synthesis variability. ACS Synthetic Biology. 8 (9), 2080-2091 (2019).
- Sun, Z. Z., et al. Protocols for implementing an Escherichia coli based TX-TL cell-free expression system for synthetic biology. Journal of Visualized Experiments: JoVE. (79), e50762 (2013).
- Dopp, J. L., Tamiev, D. D., Reuel, N. F. Cell-free supplement mixtures: Elucidating the history and biochemical utility of additives used to support in vitro protein synthesis in E. coli extract. Biotechnology Advances. 37 (1), 246-258 (2019).
- Cole, S. D., Miklos, A. E., Chiao, A. C., Sun, Z. Z., Lux, M. W. Methodologies for preparation of prokaryotic extracts for cell-free expression systems. Synthetic and Systems Biotechnology. 5 (4), 252-267 (2020).
- Sitaraman, K., et al. A novel cell-free protein synthesis system. Journal of Biotechnology. 110 (3), 257-263 (2004).
- Marshall, R., Maxwell, C. S., Collins, S. P., Beisel, C. L., Noireaux, V. Short DNA containing χ sites enhances DNA stability and gene expression in E. coli cell-free transcription-translation systems. Biotechnology and Bioengineering. 114 (9), 2137-2141 (2017).
- Yim, S. S., Johns, N. I., Noireaux, V., Wang, H. H. Protecting linear DNA templates in cell-free expression systems from diverse bacteria. ACS Synthetic Biology. 9 (10), 2851-2855 (2020).
- Zhu, B., et al. Increasing cell-free gene expression yields from linear templates in Escherichia coli and Vibrio natriegens extracts by using DNA-binding proteins. Biotechnology and Bioengineering. 117 (12), 3849-3857 (2020).
- Tuckey, C., Asahara, H., Zhou, Y., Chong, S. Protein synthesis using a reconstituted cell-free system. Current Protocols in Molecular Biology. 108, 1-22 (2014).
- Hoffman, R. A., Wang, L., Bigos, M., Nolan, J. P. NIST/ISAC standardization study: Variability in assignment of intensity values to fluorescence standard beads and in cross calibration of standard beads to hard dyed beads. Cytometry. Part A. 81 (9), 785-796 (2012).
- Karzbrun, E., Shin, J., Bar-Ziv, R. H., Noireaux, V. Coarse-grained dynamics of protein synthesis in a cell-free system. Physical Review Letters. 106 (4), 048104 (2011).
- Rossi, N. A., Dunlop, M. J. Making waves with synthetic oscillators. Cell Systems. 6 (4), 406-407 (2018).
- Niederholtmeyer, H., et al. Rapid cell-free forward engineering of novel genetic ring oscillators. eLife. 4, 09771 (2015).
- Chang, H., Voyvodic, P. L., Structurale, C. D. B. Microbially derived biosensors for diagnosis, monitoring and epidemiology. Microbial Biotechnology. 10 (5), 1031-1035 (2017).
- Pardee, K., et al. low-cost detection of Zika virus using programmable biomolecular components. Cell. 165 (5), 1255-1266 (2016).
- Pardee, K., et al. Portable, on-demand biomolecular manufacturing. Cell. 167 (1), 248-259 (2016).
- Pardee, K., et al. Paper-based synthetic gene networks. Cell. 159 (4), 940-954 (2014).
- Liu, X., Germaine, K. J., Ryan, D., Dowling, D. N. Whole-cell fluorescent biosensors for bioavailability and biodegradation of polychlorinated biphenyls. Sensors. 10 (2), 1377-1398 (2010).
- Gautam, P., Suniti, S., Amrita, K., Madathil, D., Nair, B. A Review on Recent Advances in Biosensors for Detection of Water Contamination. International Journal of Environmental Sciences. 2 (3), 1565-1574 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados