Method Article
Caratterizzazione rapida di parti genetiche con sistemi cell-free
In questo articolo
Riepilogo
Parti genetiche ben caratterizzate sono necessarie per la progettazione di nuovi circuiti genetici. Qui descriviamo un metodo economico e ad alto rendimento per caratterizzare rapidamente le parti genetiche. Il nostro metodo riduce costi e tempi combinando lisati privi di cellule, DNA lineare per evitare la clonazione e manipolazione acustica dei liquidi per aumentare la produttività e ridurre i volumi di reazione.
Abstract
La caratterizzazione e la catalogazione delle parti genetiche sono fondamentali per la progettazione di circuiti genetici utili. Avere parti ben caratterizzate consente la messa a punto dei circuiti genetici, in modo tale che la loro funzione si traduca in risultati prevedibili. Con la crescita della biologia sintetica come campo, c'è stata un'esplosione di circuiti genetici che sono stati implementati nei microbi per eseguire funzioni relative al rilevamento, all'alterazione metabolica e al calcolo cellulare. Qui, mostriamo un metodo rapido ed economico per caratterizzare le parti genetiche. Il nostro metodo utilizza lisato privo di cellule, preparato internamente come mezzo per valutare le parti attraverso l'espressione di una proteina reporter. Il DNA modello viene preparato mediante amplificazione PCR utilizzando primer economici per aggiungere parti varianti al gene reporter e il modello viene aggiunto alla reazione come DNA lineare senza clonazione. Le parti che possono essere aggiunte in questo modo includono promotori, operatori, siti di legame dei ribosomi, isolanti e terminatori. Questo approccio, combinato con l'incorporazione di un manipolatore di liquidi acustico e piastre a 384 pozzetti, consente all'utente di effettuare valutazioni ad alta produttività delle parti genetiche in un solo giorno. In confronto, gli approcci di screening basati su cellule richiedono una clonazione dispendiosa in termini di tempo e hanno tempi di test più lunghi a causa delle fasi di normalizzazione della coltura e della densità della coltura durante la notte. Inoltre, lavorare in lisato privo di cellule consente all'utente di esigere un controllo più stretto sulle condizioni di espressione attraverso l'aggiunta di componenti esogeni e DNA a concentrazioni precise. I risultati ottenuti dallo screening cell-free possono essere utilizzati direttamente nelle applicazioni di sistemi cell-free o, in alcuni casi, come un modo per prevedere la funzione in cellule intere.
Introduzione
Uno sforzo fondamentale della biologia sintetica è quello di sviluppare kit di strumenti genetici contenenti parti ben caratterizzate, che possono essere utilizzate per costruire circuiti genetici1 che svolgono funzioni utili quando vengono distribuiti in microbi o lisati privi di cellule. Le aree in cui tali circuiti genetici hanno guadagnato trazione sono il rilevamento 2,3,4, le prestazioni umane5,6, i biocarburanti 7,8, la produzione di materiali 9,10 e il calcolo cellulare 11. I registri delle parti genetiche standardizzate sono stati istituiti12 per catalogare parti nuove ed esistenti in categorie come promotori, operatori, sequenze di codifica e terminatori, per citarne solo alcuni. Sforzi come il concorso internazionale iGEM (International Geneticamente Engineered Machines)13 sono stati determinanti nella caratterizzazione e catalogazione di queste parti genetiche. Molti metodi sono stati sviluppati per facilitare il rapido assemblaggio di queste parti in circuiti genetici utili14,15. È stato persino sviluppato un software per automatizzare la composizione di parti ben caratterizzate in circuiti che raggiungono una funzione desiderata16. Tuttavia, l'assemblaggio di circuiti genetici utili con funzioni prevedibili si basa sulla presunzione che i kit di strumenti genetici contengano parti genetiche ben caratterizzate. A causa della necessità di questi kit di strumenti per il progresso della biologia sintetica, sono stati descritti numerosi sforzi per catalogare meglio circuiti e parti con dati di caratterizzazione appropriati 17,18,19,20,21.
Un approccio alla caratterizzazione delle componenti genetiche fa uso di sistemi di sintesi proteica libera da cellule (CFPS), che ricostituiscono funzioni cellulari come la trascrizione e la traduzione ex vivo22. Diversi studi hanno dimostrato il potenziale della CFPS per la prototipazione di componenti genetici 23,24,25,26,27,28,29,30,31,32 sia per applicazioni dirette in sistemi privi di cellule o per prevedere la funzione dei costrutti genetici nelle cellule, come l'attività relativa delle parti all'interno di una biblioteca 29 , ottimizzazione della via metabolica27 e carico cellulare30. I vantaggi della prototipazione in CFPS rispetto alle cellule evidenziati da questi studi includono evitare la clonazione dispendiosa in termini di tempo, un controllo preciso sulla concentrazione di DNA e altri componenti della reazione e la capacità di mescolare e abbinare facilmente più costrutti di DNA. Il vantaggio di evitare la clonazione è particolarmente evidente quando si utilizzano modelli lineari di DNA, che consentono di assemblare nuovi costrutti con metodi in vitro che richiedono ore anziché giorni33. La capacità di manipolare la concentrazione di costrutti di DNA e altri componenti semplicemente mediante pipettaggio rende l'approccio ancora più attraente consentendo una sperimentazione ad alto rendimento alimentata da robot per la movimentazione dei liquidi34,35. Sebbene siano stati riportati successi nell'utilizzo di CFPS per la prototipazione, è importante notare che resta da vedere in quali contesti i risultati di CFPS possono prevedere in modo affidabile la funzionalità nelle cellule.
In questo articolo, presentiamo un metodo per la prototipazione CFPS che sottolinea i vantaggi in termini di velocità, produttività e costi rispetto ai tradizionali approcci basati su celle. L'approccio deriva dal nostro lavoro precedente in cui abbiamo utilizzato CFPS per caratterizzare rapidamente una libreria di varianti del promotore T7 regolate dal fattore di trascrizione TetR32, espandendo significativamente la piccola manciata di varianti regolate del promotore T7 che erano disponibili in letteratura all'epoca36,37. Da allora, altri hanno ulteriormente ampliato la gamma di tali promotori38. Nel nostro metodo, l'assemblaggio del costrutto genetico viene accelerato utilizzando la PCR per amplificare il DNA modello tramite primer che aggiungono parti genetiche varianti a un gene reporter. La movimentazione acustica dei liquidi in piastre a 384 pozzetti viene utilizzata per aumentare la produttività e ridurre il volume dei materiali richiesti. Lavori precedenti hanno dimostrato il successo dell'uso della manipolazione acustica dei liquidi a volumi significativamente inferiori39,40 con una variabilità paragonabile al pipettaggio manuale di volumi più grandi 41. Oltre al metodo, forniamo informazioni sulla risoluzione dei problemi e una valutazione dei potenziali risparmi di costi e tempo. Si noti che mentre includiamo un protocollo per la produzione di lisati privi di cellule basato su Sun et al.42 qui, numerosi altri kit e protocolli commerciali43,44 dovrebbero funzionare. Allo stesso modo, mentre dimostriamo il metodo per la caratterizzazione delle varianti del promotore32, altre parti possono essere scambiate mediante amplificazione PCR, come riboregolatori, siti di legame ribosomiale (RBS), isolanti, tag proteici e terminatori. Speriamo che questa metodologia possa aiutare la comunità della biologia sintetica a continuare a far crescere il numero di parti caratterizzate per l'assemblaggio di circuiti genetici prevedibili con funzioni utili.
Protocollo
1. Preparazione dell'estratto cellulare
- Preparazione dei media
- Per i terreni 2xYT: aggiungere 16 g di triptone, 10 g di estratto di lievito e 5 g di NaCl a 900 ml di acqua deionizzata e regolare il pH a 7,0 con 5 M NaOH. Aumentare il volume della soluzione a 1 L utilizzando acqua deionizzata e sterilizzare in autoclave o filtro. In alternativa, acquista supporti 2xYT.
- Per tampone S30B: preparare una soluzione di 14 mM Mg-glutammato, 60 mM K-glutammato e 5 mM Tris in 2 L di acqua deionizzata. Utilizzare 2 M Tris per regolare il pH a 8,2, in autoclave e conservare a 4 °C. Completare la soluzione aggiungendo ditiotreitolo (DTT) ad una concentrazione finale di 1 mM appena prima dell'uso.
- Preparazione delle cellule
- Striare le cellule di Escherichia coli BL21(DE3) Rosetta2 o altra linea cellulare di scelta (vedi Cole et al.44 per una recente revisione completa) su una piastra di agar LB (Lysogeny Broth) e incubare a 37 °C per 10-14 ore.
- Utilizzare una singola colonia di E. coli per inoculare 3 ml di terreno 2xYT in una provetta di coltura da 10 ml. Incubare questo tubo a 37 °C agitando a 250 giri/min per 8 ore.
- Utilizzare 50 μL dalla coltura da 3 mL per inoculare 50 mL di terreno 2xYT in un matraccio da 500 ml. Incubare il matraccio a 37 °C agitando a 250 giri/min per 8 ore.
- Utilizzare 7,5 mL dalla coltura da 50 mL per inoculare ciascuno dei quattro palloni da 4 L deflettori contenenti 0,75 L di terreno 2xYT. Incubare questi matraccio a 37 °C agitando a 220 giri/min fino a raggiungere una densità ottica a 600 nm di 2-4, dopo circa 3-4 ore.
- Raccogliere le cellule da ciascun matraccio trasferendole in contenitori da 1 L e centrifugandole a 5.000 x g per 12 minuti a 4 °C. Scartare il surnatante decantando in un contenitore per rifiuti.
- Lavare ogni pellet cellulare con 150 ml di tampone S30B ghiacciato risospendendoli completamente usando una pipetta per interrompere la massa cellulare, quindi raccogliere nuovamente le cellule mediante centrifugazione a 5.000 x g per 12 minuti. Scartare il surnatante.
- Lavare nuovamente ogni pellet cellulare in 40 ml di tampone S30B ghiacciato risospendendoli completamente e interrompendo la massa cellulare usando una pipetta. Trasferire le celle in provette coniche prepesate da 50 ml e raccogliere nuovamente le celle mediante centrifugazione a 2.000 x g per 8 minuti a 4 °C. Scartare il surnatante mediante decantazione.
- Pesare i pellet a celle umide. Congelare i pellet cellulari posizionando i tubi direttamente nell'azoto liquido e conservarli a -80 °C.
- Lisi cellulare
- Scongelare i pellet cellulari sul ghiaccio.
- Risospendere ogni pellet cellulare in 1,4 ml di tampone S30B per 1 g di pellet cellulare mediante vortice.
- Lisare le celle mediante cella a pressione francese a 640 psi a 4 °C. Raccogliere il lisato in provette da microcentrifuga su ghiaccio e aggiungere 3 μL di 1 M DTT per 1 mL di lisato immediatamente dopo la lisi.
NOTA: è meglio picchiettare la valvola di rilascio stampa francese con una piccola asta metallica per mantenere una pressione uniforme ed evitare improvvisi cali di pressione. - Pulire il lisato mediante centrifugazione a 30.000 x g per 30 minuti a 4 °C e scartare il pellet dopo aver pipettato il surnatante in una nuova provetta di microcentrifuga ghiacciata, facendo attenzione a non disturbare il pellet.
- Centrifugare il surnatante una seconda volta a 30.000 x g per 30 minuti a 4 °C. Pipettare il surnatante risultante in una provetta di microcentrifuga ghiacciata. Scartare il pellet.
- Incubare il surnatante a bagnomaria a 37 °C per 1 ora.
- Eliminare il surnatante mediante centrifugazione a 15.000 x g per 15 minuti a 4 °C e trasferire il surnatante risultante in una provetta di microcentrifuga ghiacciata, facendo attenzione a non disturbare il pellet.
- Centrifugare il surnatante una seconda volta a 15.000 x g per 15 minuti a 4 °C e trasferire il surnatante risultante in una provetta di microcentrifuga ghiacciata, facendo attenzione a non disturbare il pellet residuo.
- Distribuire il surnatante in aliquote da 100 μL in provette da microcentrifuga da 1,5 mL e congelarle istantaneamente mettendole direttamente in azoto liquido. Conservare il surnatante a -80 °C.
2. Preparazione del modello lineare
- Design del primer
- Scegli una sequenza di base come modello PCR. Includi almeno una sequenza reporter, come sfGFP (superfolder Green Fluorescent Protein), LacZ o Aptamer di spinaci. Includere altre parti che verranno fissate tra le varianti schermate, come terminatori, promotori o RBS, a seconda della progettazione.
NOTA: L'inclusione di un terminatore non è sempre richiesta per l'espressione da DNA lineare in sistemi privi di cellule. - Per i primer in avanti, scegliere un minimo di 20 bp corrispondente all'estremità 5ʹ della sequenza del nucleo come estremità 3ʹ del primer. Se si aggiungono parti all'estremità 5′ del costrutto, progettare il resto dell'estremità 5ʹ del primer per aggiungere le parti genetiche di interesse alla sequenza centrale tramite amplificazione PCR (Figura 1A e Figura 2).
NOTA: Poiché i primer superiori a ~ 60 bp aumentano spesso notevolmente in termini di costo, è possibile progettare più primer sovrapposti per aggiungere sequenze più lunghe o più parti. Mentre più primer possono essere utilizzati in una singola reazione PCR, si raccomanda di eseguire più cicli di PCR. - Per i primer inversi, scegliete un minimo di 20 bp per abbinare l'estremità 3′ della sequenza principale come l'estremità 3ʹ del primer. Se si aggiungono parti all'estremità 3′ del costrutto, progettare il resto dell'estremità 5ʹ del primer per aggiungere le parti genetiche di interesse alla sequenza centrale tramite amplificazione PCR (Figura 1A e Figura 2). Assicurarsi che la temperatura di ricottura del primer inverso sia entro 5 °C dalla temperatura di ricottura dell'intero primer anteriore.
- Scegli una sequenza di base come modello PCR. Includi almeno una sequenza reporter, come sfGFP (superfolder Green Fluorescent Protein), LacZ o Aptamer di spinaci. Includere altre parti che verranno fissate tra le varianti schermate, come terminatori, promotori o RBS, a seconda della progettazione.
- Amplificazione lineare del modello
- Determinare il numero di reazioni PCR da eseguire in base al numero di sequenze core e calcolare la quantità di ciascun componente richiesto utilizzando la Tabella 1.
- Preparare il master mix secondo la Tabella 1 e conservarlo su ghiaccio. Aliquot 30 o 40 μL (vedi Tabella 1) della miscela master nel numero determinato di provette PCR e aggiungere 10 μL di ciascun primer variabile (cioè primer che codificano un cambiamento di parte, vedi Tabella 1) a 5 μM a tubi PCR opportunamente etichettati.
- Posizionare i tubi PCR nel termociclatore ed eseguire il seguente programma PCR: 98 °C per 3 minuti; 30 cicli di 98 °C per 15 s, XX °C per 20 s, 72 °C per YY min; estensione finale a 72 °C per 10 min. Quindi, mantenere la reazione a 4 °C.
Dove, XX rappresenta la temperatura di ricottura per il primer con la temperatura di ricottura inferiore e YY rappresenta il tempo di estensione calcolato per la lunghezza dell'amplicone in base alle raccomandazioni del produttore per la polimerasi ad alta fedeltà utilizzata. Ottimizza queste condizioni secondo necessità per diversi primer e/o modelli. - (Facoltativo) Aggiungere 1 μL di enzima di restrizione DpnI per digerire il modello originale. Incubare la reazione a 37 °C per 1 ora. Eseguire questo passaggio solo se il modello originale è DNA plasmide.
- Analizzare 5 μL di ciascun prodotto PCR mediante elettroforesi su gel. Separare il prodotto con un gel di agarosio all'1% a 180 V per 20 min. Verificare la dimensione della banda corretta, che varierà in base alla sequenza di nucleo scelta e alla lunghezza delle parti aggiunte.
- Purificare la dima lineare utilizzando un kit di purificazione PCR commerciale o con il metodo di pulizia PCR preferito. Se fossero presenti più bande dall'analisi dell'elettroforesi su gel, ottimizzare le condizioni di PCR o purificare le bande di peso molecolare corrette utilizzando un kit di estrazione del gel commerciale secondo la raccomandazione del produttore.
- Quantificare ogni modello di DNA utilizzando uno spettrofotometro. Valutare la qualità del modello di DNA controllando che il rapporto 260 nm/280 nm sia approssimativamente 1,8.
- (Facoltativo) Separare nuovamente una porzione del modello di DNA utilizzando un gel di agarosio all'1% a 180 V per 20 minuti e assicurarsi che eventuali bande indesiderate siano state rimosse durante la purificazione del modello.
- Utilizzare immediatamente modelli di DNA purificato o conservare a -20 °C.
3. Preparazione proteica purificata
- Espressione proteica
- Per ogni proteina da esprimere, assemblare un costrutto di espressione appropriato. Codon-ottimizzare il gene per l'espressione in E. coli. Inserire il gene in un vettore di espressione di pET-22b o in un altro vettore di espressione appropriato tramite il metodo di assemblaggio plasmidico preferito. Trasformare il plasmide di espressione in cellule di espressione di Rosetta2 BL21(DE3) o in altre linee cellulari appropriate.
- Per ogni proteina, utilizzare una singola colonia per inoculare 3 ml di terreno LB in una provetta da 10 mL. Incubare questi tubi a 37 °C agitando a 250 giri/min durante la notte.
- Inoculare un matraccio da 2 L contenente 750 mL di terreno LB con 1 mL di coltura notturna. Incubare questi matraccio a 37 °C agitando a 250 giri/min fino a raggiungere un OD600 di 0,6-1,0.
- Indurre l'espressione proteica aggiungendo 0,75 mL di β-D-1-tiogalattopiranoside (IPTG) isopropilico 1 M in acqua a ciascun matraccio e continuare a incubare questi matraccio a 37 °C agitando a 250 giri/min per 4 ore.
- Raccogliere le cellule da ciascun pallone, utilizzando un flacone da centrifuga da 1 L, mediante centrifugazione a 5.000 x g per 12 minuti. Scartare il surnatante.
- Trasferire il pellet in un tubo conico da 50 ml e pesare ogni pellet. Congelare le cellule in azoto liquido e conservarle a -80 °C oppure procedere al punto 3.2.
NOTA: da questo passaggio si prevedono 2-5 g di cellule per 0,75 ml.
- Purificazione delle proteine mediante cromatografia su colonna di affinità di nichel
- Preparare il tampone di lisi combinando 50 mM Tris-Cl, 500 mM NaCl e 5 mM imidazolo. Regolare a pH 8,0.
- Preparare il tampone di lavaggio combinando 50 mM Tris-Cl, 500 mM NaCl e 25 mM imidazolo. Regolare a pH 8,0.
- Preparare il tampone di eluizione combinando 50 mM Tris-Cl, 500 mM NaCl e 250 mM imidazolo. Regolare a pH 8,0.
- Preparare il tampone di dialisi combinando 50 mM NaHPO4, 100 mM NaCl e 2% DMSO. Regolare a pH 7,5.
- Scongelare i pellet cellulari mettendo i tubi in acqua a temperatura ambiente. Aggiungere 5 ml di tampone di lisi per 1 g di pellet cellulare e risospendere per vortice.
- Lisare le cellule mediante sonicazione. Separare l'omogenato cellulare in modo che non ci siano più di 30 ml per tubo conico da 50 mL e mantenere ogni tubo sul ghiaccio. Lisare le celle utilizzando un sonicatore con una sonda di 0,16 cm di diametro in 15 s colpi con 30 s break, 10 volte.
NOTA: Evitare la formazione di schiuma, poiché ciò denatura la proteina. La formazione di schiuma può essere evitata mantenendo la punta del sonicatore almeno 2/3 immersa nel lisato mentre è operativa. Altri metodi di lisi cellulare oltre alla sonicazione sono anche possibili44. - Eliminare il lisato mediante centrifugazione a 15.000 x g per 30 minuti a 4 °C e introdurre il surnatante in una nuova provetta conica da 50 ml.
- Aggiungere 1 mL di resina di acido nichel-nitrilotriacetico (Ni-NTA) per ogni 5 mL di surnatante. Dividere il lisato cellulare/Ni-NTA liquame in modo che non ci siano più di 36 ml per tubo conico da 50 ml. Incubare a 4 °C su un rotatore di tubi a 10 giri/min per 1 ora.
- Caricare la resina decantando il lisato cellulare/Ni-NTA in una colonna cromatografica a letto di 2 ml e raccogliere l'eluente se necessario per ulteriori analisi, altrimenti scartare. Lavare la resina con 10 volumi di letto resina di tampone di lavaggio.
- Raccogliere la proteina aggiungendo tre volumi di letto di resina di tampone di eluizione alla colonna e concentrare il volume a 1,5 ml utilizzando un concentratore centrifugo con la membrana di cut-off del peso molecolare appropriata per ciascuna proteina.
- Dializzare la proteina contro 2 L di tampone dialitico a 4 °C per 1 ora. Dializzare nuovamente la proteina contro 2 L di tampone dialitico per una notte a 4 °C.
- Quantificare la proteina utilizzando il suo coefficiente di estinzione molare e l'assorbanza a 280 nm. Analizzare la proteina per la purezza separandola usando l'elettroforesi su gel di sodio dodecilsolfato poliacrilammide (SDS-PAGE). Conservare la proteina a -80 °C.
4. Sintesi proteica priva di cellule
- Preparazione della miscela di reazione CFPS
- Preparare il mix di integratori seguendo le fasi di preparazione della soluzione di aminoacidi, preparazione della soluzione energetica e preparazione del tampone in Sun et al.42. Conservare separatamente o in combinazione a -80 °C in aliquote. Assicurarsi che le concentrazioni finali corrispondano a quelle descritte in Sun et al.42 nella sezione Esecuzione sperimentale di una reazione TX-TL.
- Preparare un additivo per proteggere il DNA lineare dalla degradazione. Se si utilizza GamS33,45, prepararsi tramite i passaggi nella sezione 3 di cui sopra, o ottenere da un fornitore commerciale; Per altri approcci, consultare la letteratura corrispondente46,47,48. In alternativa, utilizzare un sistema CFPS che non richieda additivi49.
- Preparare la polimerasi T7, le proteine repressori e altri additivi utilizzando i passaggi nella sezione 3 sopra o ottenere da un fornitore commerciale.
- Determinare il numero di reazioni CFPS da eseguire e calcolare la quantità di ciascun componente richiesto utilizzando la Tabella 2. Modificare le concentrazioni dei componenti, compresa l'aggiunta o la rimozione di componenti secondo necessità, e regolare la quantità di acqua in modo tale che il volume finale di ciascuna miscela di reazione sia sempre di 10 μL. Allo stesso modo, modificare la miscela master per facilitare l'erogazione di altri componenti mediante la manipolazione acustica del liquido come desiderato (vedere la sezione Discussione ).
- Scongelare tutti i componenti sul ghiaccio e preparare un master mix mescolando ogni componente come calcolato sopra. Miscelare accuratamente tutti i componenti mediante pipetta. Prestare molta attenzione per evitare precipitazioni, soprattutto per la miscela di aminoacidi. Mantenere il master mix sul ghiaccio.
- Raffreddare una piastra da 384 pozzetti sul ghiaccio e distribuire la miscela master in aliquote da 9 μL in ciascun pozzetto.
NOTA: è possibile distribuire questi componenti mediante manipolazione acustica dei liquidi, anche se è necessario prestare attenzione per garantire una corretta erogazione (vedere la sezione Discussione per la risoluzione dei problemi).
- Distribuzione di componenti aggiuntivi mediante manipolazione acustica dei liquidi
- Calcolare la quantità di proteina repressore (e altri componenti opzionali) necessaria per tutte le reazioni CFPS.
- Scongelare la proteina repressore sul ghiaccio e distribuirla in una piastra sorgente acustica di manipolazione del liquido o in un'altra piastra appropriata. Assicurarsi che sia inclusa la quantità appropriata di volume morto richiesto per il tipo di piastra sorgente utilizzata.
- Distribuire la proteina repressore in volumi di 1 μL nei pozzetti appropriati tramite il manipolatore di liquidi. Per ulteriori informazioni sulla risoluzione dei problemi di distribuzione, vedere la sezione Discussione .
- Curve standard
- Includere una diluizione seriale del reporter purificato (vedere sezione 3 per la purificazione delle proteine)41 o uno standard chimico appropriato50 sulla piastra per consentire il confronto dei risultati con altri studi e altri laboratori. Scegliere un intervallo di concentrazioni appropriato per il reporter utilizzato e l'intervallo di espressione previsto degli esperimenti.
- Esecuzione di reazioni CFPS
- Preriscaldare il lettore di piastre a 30 °C. Impostare il lettore di lastre in modo che legga alle impostazioni appropriate per il reporter utilizzato nella sequenza principale senza oscillare i passaggi.
NOTA: Mentre 30 °C è usato qui, 29 °C e 37 °C sono anche comunemente usati e funzionano bene con questo protocollo. Altre temperature possono essere preferite per preparazioni alternative di reazione prive di cellule. Per gli intervalli di lettura, 10 minuti sono sufficienti per ottenere una buona risoluzione per i dati rappresentativi qui presentati; tuttavia, altre risoluzioni possono essere migliori a seconda della proteina reporter e della particolare ricetta CFPS. - (Facoltativo) Eseguire prima una reazione di prova per impostare l'impostazione appropriata di guadagno o sensibilità per catturare la variazione di fluorescenza senza overflow del segnale.
- Sigillare la piastra a 384 pozzetti con un coperchio sigillabile in plastica impermeabile per evitare l'evaporazione. Se possibile, sullo strumento, impostare un gradiente di temperatura verticale di 1 °C per limitare la formazione di condensa sulla guarnizione. Posizionare la piastra a 384 pozzetti sul supporto della piastra e iniziare a leggere.
- Preriscaldare il lettore di piastre a 30 °C. Impostare il lettore di lastre in modo che legga alle impostazioni appropriate per il reporter utilizzato nella sequenza principale senza oscillare i passaggi.
Risultati
Per dimostrare l'utilità dei nostri metodi, presentiamo risultati che descrivono gli effetti della vicinanza della sequenza tetO al promotore T7 sulla regolazione dell'espressione guidata da RNAP T7. I risultati completi e le loro implicazioni possono essere trovati nel lavoro di McManus et al.32. Il flusso di lavoro è descritto nella Figura 1. Quindici modelli lineari, variabili solo nella distanza del promotore T7 rispetto alla sequenza tetO , sono stati preparati amplificando con PCR il reporter sfGFP con primer progettati per aggiungere ogni variante del promotore (Figura 2) come descritto nella sezione 2 del protocollo. I componenti e le reazioni della reazione CFPS sono stati preparati seguendo il protocollo. L'espressione di sfGFP è stata misurata da ciascun modello con una titolazione di 12 diverse concentrazioni della proteina TetR, in triplice copia, utilizzando un gestore acustico di liquidi. A 36 reazioni CFPS per modello e 15 modelli, sono state eseguite un totale di 540 reazioni per l'intero set di combinazioni T7-tetO . L'intera valutazione è stata effettuata su due piastre in due lettori di piastre. L'analisi di questi dati ha mostrato che l'RNAP T7 sottoregola l'espressione guidata da T7 in modo uguale fino a 13 bp a valle dall'inizio del trascritto T7 (Figura 3). Questo risultato ha implicazioni per la futura progettazione di circuiti genici regolabili guidati da T7 descrivendo una finestra putativa per un'efficace repressione di T7 da parte di altri repressori. Il confronto dei risultati del protocollo qui descritto con il DNA preparato mediante clonazione tradizionale ha rivelato una piccola ma statisticamente significativa differenza nel grado di repressione TetR tra i formati. Abbiamo ipotizzato che il legame non specifico di TetR al DNA vettore potrebbe spiegare la differenza osservata. I risultati sperimentali hanno mostrato che l'aggiunta di DNA vettoriale lineare alle reazioni con DNA modello lineare ha ridotto la differenza a significato non statistico, sebbene non abbia escluso contributi da altri fattori, come le differenze nella periodicità dell'elica del DNA per i formati lineari rispetto a quelli circolari, che, a loro volta, potrebbero influenzare il legame con TetR. A seconda dell'applicazione, l'uso del modello lineare può richiedere un'ulteriore convalida.
Includiamo inoltre dati rappresentativi su potenziali problemi con un'erogazione accurata utilizzando la gestione acustica dei liquidi (Figura 4). Una soluzione di 1x soluzione salina tamponata fosfato (PBS), pH 7,4 contenente 0,25 mM di colorante tartrazina è stata utilizzata per valutare due metodi di programmazione di un manipolatore di liquidi acustico per erogare volumi >1 μL. Dopo l'erogazione del liquido, la piastra di destinazione è stata sigillata e centrifugata a 1.500 x g per 1 minuto e l'assorbanza a 425 nm misurata con un lettore di piastre. I risultati rappresentativi di nove esperimenti sono mostrati e dimostrano un'erogazione più coerente attraverso la serie di otto pozzi di destinazione quando il trasferimento di 5 μL è diviso in erogazioni separate da 1 μL. Sulla base di queste osservazioni, si raccomanda di suddividere i trasferimenti di >1 μL in trasferimenti multipli di ≤1 μL. Vedere la sezione Discussione per ulteriori dettagli sulla risoluzione dei problemi relativi a questo importante aspetto del protocollo.
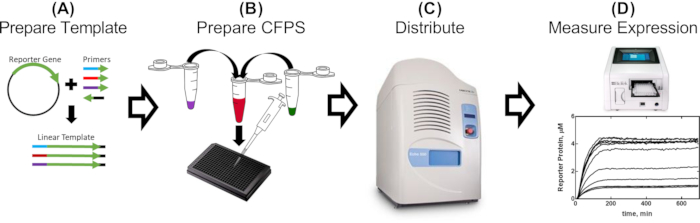
Figura 1: Flusso di lavoro di un giorno per la valutazione delle parti promotrici in estratto privo di cellule. (A) Un reporter viene amplificato con PCR utilizzando primer contenenti parti genetiche da valutare (2-5 ore). (B) La miscela di reazioni cell-free viene preparata come dettagliato nel protocollo e distribuita in una piastra a 384 pozzetti con i modelli amplificati da PCR (30 min). (C) La manipolazione acustica dei liquidi viene utilizzata per distribuire componenti aggiuntivi, che possono includere proteine repressorie, molecole effettrici e qualsiasi altro effettore condizionale (10 min). (D) L'espressione proteica di Reporter da ogni reazione viene misurata in un lettore di piastre (2-16 ore, a seconda della ricetta e del costrutto CFPS). Fare clic qui per visualizzare una versione ingrandita di questa figura.
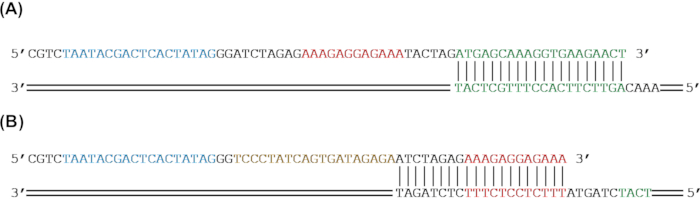
Figura 2: Progettazione di primer per l'aggiunta di parti genetiche a un gene reporter mediante amplificazione PCR . (A) Il gene reporter sfGFP (verde) sarà amplificato per aggiungere un RBS (rosso) e un promotore T7 (blu) mediante PCR. (B) L'sfGFP (verde) e un RBS (rosso) saranno amplificati per aggiungere una sequenza tetO (oro) e un promotore T7 (blu) mediante PCR. Fare clic qui per visualizzare una versione ingrandita di questa figura.
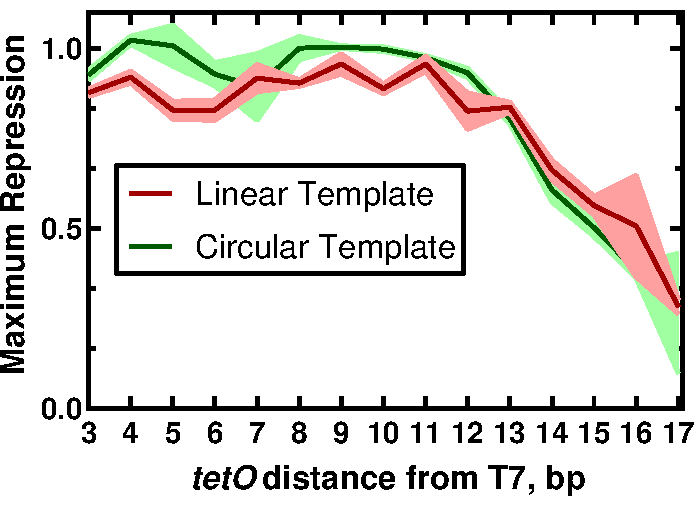
Figura 3: L'effetto della posizione tetO sulla regolazione di un'espressione guidata da T7. Valori di repressione massima normalizzati per template lineare e circolare in funzione della posizione tetO . Le tracce rappresentano la media e le deviazioni standard per tre repliche. Questa cifra è stata modificata da McManus et al.32 sotto una licenza Creative Commons CC-BY. Fare clic qui per visualizzare una versione ingrandita di questa figura.
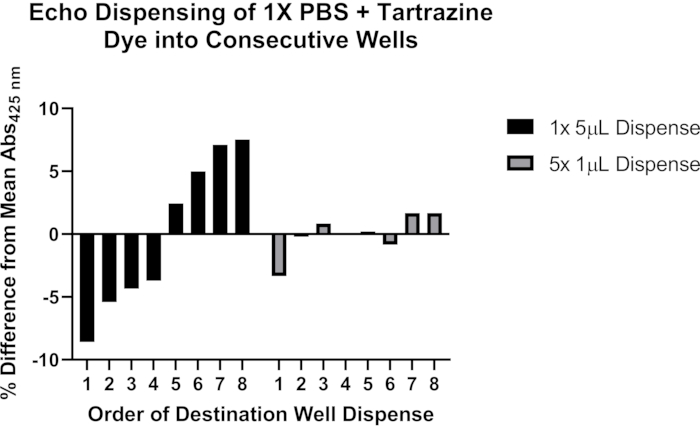
Figura 4: Utilizzo del colorante tartrazina per convalidare l'erogazione di liquidi con un manipolatore di liquidi acustico. Le barre nere indicano l'erogazione di 5 μL di soluzione di tartrazina da un singolo pozzo sorgente in ciascuno degli otto pozzetti di destinazione consecutivi di una piastra a 384 pozzetti utilizzando un singolo comando di programmazione. Le barre grigie indicano l'erogazione di 1 μL da un singolo pozzo sorgente in ciascuno degli otto pozzetti di destinazione consecutivi utilizzando un singolo comando di programmazione, e quindi ripetendo questo passaggio quattro volte per un totale di 5 μL erogati in ciascun pozzetto di destinazione. Fare clic qui per visualizzare una versione ingrandita di questa figura.
| Nome del componente | Volume per 1 reazione (μL) | Volume per il 110% del numero X di reazioni (μL) |
| Premiscela PCR Q5 | 25 | |
| Acqua | 4 | |
| Modello (1–3 ng/μL) | 1 | |
| (se fisso1) Primer anteriore (5 μM) | 0 o 10 | |
| (se fisso1) Primer inverso (5 μM) | 0 o 10 | |
| Master Mix Totale: | 30 o 40 | |
| (se variabile1) Primer anteriore (5 μM) | 0 o 10 | |
| (se variabile1) Primer inverso (5 μM) | 0 o 10 |
Tabella 1: Foglio di lavoro per la preparazione dei reagenti per le reazioni PCR. I valori nella colonna più a destra possono essere compilati dagli utenti a seconda del numero previsto di reazioni. 1 I primer variabili contengono una parte specifica da aggiungere nella reazione PCR e possono essere il primer in avanti, il primer inverso o entrambi. I primer fissi non aggiungono una parte e possono essere il primer forward o reverse primer ma non entrambi.
| Nome del componente | Volume per 1 reazione (μL) | Volume per il 110% del numero X di reazioni (μL) |
| Estratto cellulare | 4.2 | |
| Mix di integratori | 3.3 | |
| Proteina GamS (207 μM) | 0.15 | |
| Modello DNA (20 nM) | 1 | |
| T7 polimerasi (13 mg/ml) | 0.12 | |
| Acqua | 0.73 (questo numero può variare) | |
| Master Mix Totale: | 9 | |
| Proteina repressore: | 1 |
Tabella 2: Scheda di lavoro per la preparazione dei reagenti per le reazioni CFPS. I valori nella colonna più a destra possono essere compilati dagli utenti a seconda del numero previsto di reazioni.
Tabella supplementare. Clicca qui per scaricare questa tabella.
Discussione
I protocolli qui descritti forniscono un mezzo economico e rapido per lo screening di parti genetiche attraverso l'espressione di una proteina reporter da parte di CFPS. Le parti genetiche ben caratterizzate sono cruciali per la progettazione di circuiti genetici prevedibili con funzioni utili. Questa metodologia aumenta la produttività e diminuisce il tempo necessario per lo screening di nuove parti genetiche eliminando la necessità di lavorare nelle cellule viventi, pur mantenendo la funzionalità che rispecchia l'ambiente cellulare mantenendo il processo metabolico di espressione proteica nel lisato cellulare. Il nostro protocollo può essere eseguito in 1 giorno dopo il ricevimento dei primer (~ 2,5-6 h per la preparazione della reazione, 2-16 h per la reazione CFPS; Figura 1), rispetto ad almeno 3 giorni per la clonazione tradizionale (1 giorno ciascuno per l'assemblaggio e la trasformazione del costrutto, la verifica della sequenza dei cloni e la coltura delle cellule per la valutazione). Stimiamo inoltre che il costo per costrutto utilizzando DNA lineare sia circa un terzo della clonazione tradizionale ($ 78 contro $ 237; Tabella supplementare 1) metodi. I servizi di sintesi commerciale attualmente quotano un minimo di 4 giorni lavorativi a seconda delle dimensioni, anche se avrebbero costi simili al nostro metodo se i frammenti lineari fossero sottoposti a screening direttamente in CFPS ($ 78 contro $ 91); Non abbiamo verificato questo approccio. Il costo per valutare una parte con CFPS è piccolo rispetto alla generazione del DNA del modello ($ 0,05 / reazione22 contro $ 78 per modello), anche se va notato che i costi di avvio per i reagenti di massa e le apparecchiature di lisi sono almeno diverse migliaia di dollari. L'uso di un manipolatore acustico di liquidi migliora solo marginalmente i costi consentendo volumi più piccoli fino a 0,5 μL40; Il vantaggio più significativo è la riduzione del tempo per preparare le reazioni (~ 10 minuti rispetto a 1 ora, a seconda del numero di reazioni), specialmente quando la preparazione di un gran numero di reazioni solleva preoccupazioni di reazione preparata seduta per lunghi tempi prima dell'incubazione.
Sebbene rapido ed economico, restano da vedere i limiti su quando la prototipazione CFPS prevede adeguatamente la funzione in vivo . Ad esempio, qualsiasi cross-reattività con DNA genomico non sarà rilevata a causa della rimozione del genoma ospite durante la produzione del sistema CFPS. Inoltre, le concentrazioni dei componenti possono essere inferiori di 1-2 ordini di grandezza nella CFPS rispetto alle cellule51, il che è probabile che influenzi il comportamento di alcune parti a causa di diverse condizioni di affollamento macromolecolare. Inoltre, la capacità del DNA lineare di predire la funzione in vivo può essere limitata, ad esempio, quando la struttura secondaria del DNA gioca un ruolo importante. Un'ultima limitazione è che i costrutti non vengono verificati in sequenza prima di testare le funzioni. Ci possono essere casi in cui la parte caratterizzata non è effettivamente allineata con la sequenza teorica prevista. Tutte queste limitazioni possono essere mitigate convalidando un sottoinsieme delle parti vagliate con questo metodo nell'applicazione in vivo prevista.
Abbiamo originariamente sviluppato questa metodologia per studiare gli effetti del cambiamento della posizione dell'operatore sui promotori ibridi T7-tetO 32. Abbiamo presentato i protocolli qui in un formato più generico, in modo tale che possano essere applicati a promotori, operatori, sequenze di legame dei ribosomi, isolanti e terminatori. Queste parti genetiche possono essere aggiunte all'estremità 5ʹ o 3ʹ del gene reporter mediante PCR utilizzando primer per ogni disegno, ovviando alla necessità di sintesi o clonazione di ogni variante da testare. I prodotti PCR risultanti fungono da DNA modello per la valutazione tramite l'espressione di una proteina reporter. Nel nostro lavoro, il protocollo di purificazione dell'affinità fornito qui è stato utilizzato per TetR e GamS. La stessa procedura può essere utilizzata per l'espressione e la purificazione di altri repressori, attivatori, polimerasi, fattori sigma e altre proteine affini ad una parte genetica di interesse, sebbene possano essere necessarie modifiche per la proteina desiderata espressa. La purificazione e la titolazione di queste proteine in reazioni CFPS consente una caratterizzazione più dettagliata di una particolare parte genetica. Infine, esistono numerosi protocolli CFPS alternativi e ciascuno dovrebbe essere suscettibile di parte della metodologia di screening. Ad esempio, non includiamo una fase di dialisi in questo protocollo, che altri hanno trovato importante per l'espressione da promotori batterici nativi22. È anche possibile variare le concentrazioni dei componenti costitutivi sottostanti della CFPS. L'uso della manipolazione dei liquidi migliora la capacità di testare la miriade di condizioni aumentando la produttività e diminuendo i materiali richiesti34,35.
Un'area che può richiedere una risoluzione significativa dei problemi è l'ottimizzazione del movimentatore di liquidi acustico. L'erogazione acustica del trattamento di liquidi deve essere ottimizzata per ogni componente da trasferire e si consiglia vivamente di eseguire controlli per verificare la corretta distribuzione e riproducibilità prima di raccogliere i dati. Il tipo di piastra sorgente ideale e l'impostazione della classe del liquido dipenderanno dal liquido specifico da erogare e dai suoi componenti. Non è raccomandato l'uso di piastre rivestite di ammine per erogare il DNA, poiché il rivestimento amminico può interagire con il DNA. Va inoltre notato che la capacità di erogare concentrazioni più elevate di alcuni componenti può dipendere dal modello di manipolazione acustica dei liquidi. Un trasferimento di liquido di prova può essere effettuato erogando su una guarnizione della piastra di alluminio per visualizzare la corretta formazione di goccioline; Tuttavia, questo test fornisce informazioni limitate e le goccioline di impostazioni diverse possono apparire identiche. L'uso di un colorante solubile in acqua, come la tartrazina, può essere utilizzato per verificare con maggiore precisione che il volume corretto sia dispensato con una determinata impostazione o flusso di lavoro (vedere Risultati rappresentativi). La programmazione ottimale dei trasferimenti di liquidi può anche influenzare l'accuratezza e la coerenza dei dati generati; per trasferimenti >1 μL da un pozzo sorgente a un pozzo di destinazione, abbiamo scoperto che i trasferimenti sequenziali di ≤1 μL dovrebbero essere programmati per ridurre la variabilità sistematica well-to-well (Figura 4). Infine, i volumi di morte del pozzo di sorgente teorici ed effettivi possono variare notevolmente a seconda del tipo di piastra sorgente, dell'impostazione della classe del liquido e dei componenti del liquido specifico; L'utilizzo della funzione di rilevamento del gestore di liquidi acustici per valutare i volumi dei pozzi prima di eseguire un programma può aiutare a valutare con quanta precisione lo strumento è in grado di misurare un particolare liquido.
Le prestazioni di reazione del CFPS possono variare quando si confrontano i risultati tra diversi utenti, lotti di materiali, lettori di piastre e laboratori41. Per i casi in cui tali confronti sono necessari durante la prototipazione di circuiti genetici, si consiglia di includere reazioni di controllo interno con promotori costitutivi standard in ciascuna piastra di reazione per aiutare a normalizzare i risultati tra le configurazioni sperimentali. Anche il metodo di preparazione del DNA può contribuire in modo significativo all'attività della CFPS; Si raccomanda l'inclusione di una fase di precipitazione dell'etanolo. Inoltre, la composizione ottimale della reazione può variare in base al lotto di estratto34. Le concentrazioni ottimali di glutammato di magnesio e glutammato di potassio, in particolare, hanno dimostrato di variare per lotto42 o con la proteina promotore o reporter utilizzata24. Le concentrazioni di questi componenti devono essere ottimizzate mediante screening su diverse concentrazioni di ciascun componente per costrutto genetico e per preparazione di estratto cellulare per determinare le condizioni ottimali per l'espressione proteica. Infine, le migliori pratiche per prestazioni di reazione CFPS coerenti includono una miscelazione accurata, un pipettaggio accurato e coerenza nella preparazione di ciascun componente del reagente.
Oltre alla caratterizzazione delle singole parti, lo stesso metodo può essere utilizzato per vagliare combinazioni di parti che formano circuiti complessi, come i circuiti logici16 o gli oscillatori52,53. Questo metodo può essere applicato anche allo screening e all'ottimizzazione dei biosensori per applicazioni nella diagnostica epidemiologica 54,55,56,57 o nel rilevamento e quantificazione dei pericoli 3,58,59. L'applicazione di tecniche basate sull'intelligenza artificiale come l'apprendimento attivo34 può anche essere abbinata alla natura ad alto rendimento di questo metodo per guidare una rapida esplorazione di spazi di progettazione biologica complessi. In definitiva, prevediamo che questo approccio supporti tempi di sviluppo accelerati per nuovi disegni genetici in biologia sintetica.
Divulgazioni
RMM ha una partecipazione finanziaria in Tierra Biosciences, una società privata che utilizza tecnologie prive di cellule come quelle descritte in questo articolo per l'espressione e lo screening delle proteine.
Gli altri autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato reso possibile dal programma di ricerca applicata dell'Ufficio del Segretario della Difesa per l'avanzamento delle priorità scientifiche e tecnologiche. Ringraziamo Scott Walper (Naval Research Laboratory) per aver fornito lo stock di sfGFP utilizzato, e Zachary Sun e Abel Chiao (Tierra Biosciences) per le fruttuose discussioni relative alla prototipazione con sistemi cell-free e alla relativa risoluzione dei problemi di gestione acustica dei liquidi.
Materiali
| Name | Company | Catalog Number | Comments |
| 2x YT medium | Sigma-Aldrich | Y2377-250G | Alternative to making 2xYT media |
| Agar | Bacto | 214010 | For plating cells |
| Chromatography column (5 cm diameter) | BIO-RAD | 731-1550 | Used for protein purification. |
| Destination plate | Thermo Scientific Nunc plate | 142761 | For CFPS reactions |
| DMSO | Sigma-Aldrich | D2650 | For dialysis buffer |
| DpnI | NEB | R0176L | For digestion of plasmid templates |
| DTT | Roche | 20871723 | S30 Buffer B |
| E. coli BL21(DE3) Rosetta2 | Novagen | 70954 | Cell line used for production of lysate and purified proteins |
| Echo acoustic liquid handler | Labcyte | 525 | Acoustic liquid handler |
| French pressure cell | Thermo Spectronic | FA-078 | For lysing cells for CFPS |
| Imidazole | Sigma-Aldrich | 56750 | For buffers |
| Impermeable plastic sealable lid | Thermo | 232702 | Plate seal |
| IPTG | RPI | I56000-25.0 | Used for protein induction. |
| K-Glu | Sigma-Aldrich | g1501-500G | S30 Buffer B |
| Labcyte Echo source plate | Labcyte | PL-05525 | For use with Echo acoustic liquid handler |
| Mg-Glu | Sigma-Aldrich | 49605-250G | S30 Buffer B |
| NaCl | Sigma-Aldrich | S7653-250G | For buffers |
| NaHPO4 | Sigma-Aldrich | 71505 | For dialysis buffer |
| NaOH | Mallinckrodt Chemicals | 7708-10 | For making 2xYT media. Currently not produced by Mallinckrodt. Alternate: Sigma-Aldrich S0899 |
| Ni-NTA resin | Invitrogen | R901-15 | For production of purified proteins |
| PCR H2O | Ambion | AM9937 | PCR of linear templates |
| Plate Reader | BioTek | H10 | Plate reader used |
| Q5 PCR Master Mix | NEB | M0494S | PCR of linear templates |
| QIAquick Gel Extraction Kit | Qiagen | 28606 | PCR of linear templates |
| QIAquick PCR Purification Kit | Qiagen | 28004 | PCR of linear templates |
| QSonica Ultrasonic Processor | Qsonica | Q700 | Cell disruption during protein purification |
| RTS Amino Acid Sampler | biotechrabbit | BR1401801 | Updated supplier from Sun et al. |
| Tris | MP | 819623 | S30 Buffer B |
| Tris-Cl | Sigma-Aldrich | T5941 | For buffers |
| Tryptone | Fluka | T7293 | For making 2xYT media |
| Yeast Extract | Bacto | 212750 | For making 2xYT media |
Riferimenti
- Purnick, P. E. M., Weiss, R. The second wave of synthetic biology: from modules to systems. Nature Reviews. Molecular Cell Biology. 10 (6), 410-422 (2009).
- Saltepe, B., Kehribar, E. &. #. 3. 5. 0. ;., Su Yirmibeşoǧlu, S. S., Şafak Şeker, U. &. #. 2. 1. 4. ;. Cellular biosensors with engineered genetic circuits. ACS Sensors. 3 (1), 13-26 (2018).
- Bereza-Malcolm, L. T., Mann, G., Franks, A. E. Environmental sensing of heavy metals through whole cell microbial biosensors: A synthetic biology approach. ACS Synthetic Biology. 4 (5), 535-546 (2015).
- Mimee, M., et al. An ingestible bacterial-electronic system to monitor gastrointestinal health. Science. 360 (6391), 915-918 (2018).
- Isabella, V. M., et al. Development of a synthetic live bacterial therapeutic for the human metabolic disease phenylketonuria. Nature Biotechnology. 36 (9), 857-867 (2018).
- Healy, C. P., Deans, T. L. Genetic circuits to engineer tissues with alternative functions. Journal of Biological Engineering. 13, 39 (2019).
- Kaushik, A., Tiwari, S., Dev Jayant, R., Marty, A., Nair, M. Towards detection and diagnosis of Ebola virus disease at point-of-care. Biosensors and Bioelectronics. 75, 254-272 (2016).
- Jagadevan, S., et al. Recent developments in synthetic biology and metabolic engineering in microalgae towards biofuel production. Biotechnology for Biofuels. 11, 185 (2018).
- Keating, K. W., Young, E. M. Synthetic biology for bio-derived structural materials. Current Opinion in Chemical Engineering. 24, 107-114 (2019).
- Smanski, M. J., et al. Synthetic biology to access and expand nature's chemical diversity. Nature Reviews Microbiology. 14 (3), 135-149 (2016).
- Xiang, Y., Dalchau, N., Wang, B. Scaling up genetic circuit design for cellular computing: advances and prospects. Natural Computing. 17 (4), 833-853 (2018).
- Galdzicki, M., Rodriguez, C., Chandran, D., Sauro, H. M., Gennari, J. H. Standard biological parts knowledgebase. PLoS One. 6 (2), 17005 (2011).
- Kelwick, R., Bowater, L., Yeoman, K. H., Bowater, R. P. Promoting microbiology education through the iGEM synthetic biology competition. FEMS Microbiology Letters. 362 (16), (2015).
- Torella, J. P., et al. Unique nucleotide sequence-guided assembly of repetitive DNA parts for synthetic biology applications. Nature Protocols. 9 (9), 2075-2089 (2014).
- Halleran, A. D., Swaminathan, A., Murray, R. M. Single day construction of multigene circuits with 3G assembly. ACS Synthetic Biology. 7 (5), 1477-1480 (2018).
- Nielsen, A. K., et al. Genetic circuit design automation. Science. 352 (6281), (2016).
- Canton, B., Labno, A., Endy, D. Refinement and standardization of synthetic biological parts and devices. Nature Biotechnology. 26 (7), 787-793 (2008).
- Boehm, C. R., Bock, R. Recent advances and current challenges in synthetic biology of the plastid genetic system and metabolism. Plant Physiology. 179 (3), 794-802 (2019).
- Takahashi, M. K., et al. Characterizing and prototyping genetic networks with cell-free transcription-translation reactions. Methods. 86, 60-72 (2015).
- Chappell, J., Watters, K. E., Takahashi, M. K., Lucks, J. B. A renaissance in RNA synthetic biology: New mechanisms, applications and tools for the future. Current Opinion in Chemical Biology. 28, 47-56 (2015).
- Endy, D. Foundations for engineering biology. Nature. 438 (7067), 449-453 (2005).
- Silverman, A. D., Karim, A. S., Jewett, M. C. Cell-free gene expression: an expanded repertoire of applications. Nat. Reviews. Genetics. 21 (3), 151-170 (2020).
- Dopp, J. L., Rothstein, S. M., Mansell, T. J., Reuel, N. F. Rapid prototyping of proteins: Mail order gene fragments to assayable proteins within 24 hours. Biotechnology and Bioengineering. 116 (3), 667-676 (2019).
- Garamella, J., Marshall, R., Rustad, M., Noireaux, V. The all E. coli TX-TL toolbox 2.0: A platform for cell-free synthetic biology. ACS Synthetic Biology. 5 (4), 344-355 (2016).
- Kopniczky, M. B., et al. Cell-free protein synthesis as a prototyping platform for mammalian synthetic biology. ACS Synthetic Biology. 9 (1), 144-156 (2020).
- Kelwick, R., et al. Cell-free prototyping strategies for enhancing the sustainable production of polyhydroxyalkanoates bioplastics. Synthetic Biology. 3 (1), (2018).
- Karim, A. S., et al. In vitro prototyping and rapid optimization of biosynthetic enzymes for cell design. Nature Chemical Biology. 16 (8), 912-919 (2020).
- Takahashi, M. K., et al. Rapidly characterizing the fast dynamics of RNA genetic circuitry with cell-free Transcription-Translation (TX-TL) systems. ACS Synthetic Biology. 4 (5), 503-515 (2015).
- Chappell, J., Jensen, K., Freemont, P. S. Validation of an entirely in vitro approach for rapid prototyping of DNA regulatory elements for synthetic biology. Nucleic Acids Research. 41 (5), 3471-3481 (2013).
- Borkowski, O., et al. Cell-free prediction of protein expression costs for growing cells. Nature Communications. 9 (1), 1457 (2018).
- Dudley, Q. M., Karim, A. S., Nash, C. J., Jewett, M. C. In vitro prototyping of limonene biosynthesis using cell-free protein synthesis. Metabolic Engineering. 61, 251-260 (2020).
- McManus, J. B., Emanuel, P. A., Murray, R. M., Lux, M. W. A method for cost-effective and rapid characterization of engineered T7-based transcription factors by cell-free protein synthesis reveals insights into the regulation of T7 RNA polymerase-driven expression. Archives of Biochemistry and Biophysics. 674, 108045 (2019).
- Sun, Z. Z., Yeung, E., Hayes, C. A., Noireaux, V., Murray, R. M. Linear DNA for rapid prototyping of synthetic biological circuits in an escherichia coli based TX-TL cell-free system. ACS Synthetic Biology. 3 (6), 387-397 (2014).
- Borkowski, O., et al. Large scale active-learning-guided exploration for in vitro protein production optimization. Nature Communications. 11 (1), 1872 (2020).
- Caschera, F., et al. High-throughput optimization cycle of a cell-free ribosome assembly and protein synthesis system. ACS Synthetic Biology. 7 (12), 2841-2853 (2018).
- Iyer, S., Karig, D. K., Norred, S. E., Simpson, M. L., Doktycz, M. J. Multi-input regulation and logic with T7 promoters in cells and cell-free systems. PLoS One. 8 (10), 78442 (2013).
- Karig, D. K., Iyer, S., Simpson, M. L., Doktycz, M. J. Expression optimization and synthetic gene networks in cell-free systems. Nucleic Acids Research. 40 (8), 3763-3774 (2012).
- Jung, J. K., et al. Cell-free biosensors for rapid detection of water contaminants. Nature Biotechnology. 38 (12), 1451-1459 (2020).
- Bailey, J., Eggenstein, E., Lesnick, J. Miniaturization and rapid processing of TXTL reactions using acoustic liquid handling. Labcyte Technical Note. , 1-12 (2018).
- Marshall, R., Garamella, J., Noireaux, V., Pierson, A. High-throughput microliter-sized cell-free transcription-translation reactions for synthetic biology applications using the echo 550 liquid handler. Labcyte Application Note. , 1-6 (2018).
- Cole, S. D., et al. Quantification of interlaboratory cell-free protein synthesis variability. ACS Synthetic Biology. 8 (9), 2080-2091 (2019).
- Sun, Z. Z., et al. Protocols for implementing an Escherichia coli based TX-TL cell-free expression system for synthetic biology. Journal of Visualized Experiments: JoVE. (79), e50762 (2013).
- Dopp, J. L., Tamiev, D. D., Reuel, N. F. Cell-free supplement mixtures: Elucidating the history and biochemical utility of additives used to support in vitro protein synthesis in E. coli extract. Biotechnology Advances. 37 (1), 246-258 (2019).
- Cole, S. D., Miklos, A. E., Chiao, A. C., Sun, Z. Z., Lux, M. W. Methodologies for preparation of prokaryotic extracts for cell-free expression systems. Synthetic and Systems Biotechnology. 5 (4), 252-267 (2020).
- Sitaraman, K., et al. A novel cell-free protein synthesis system. Journal of Biotechnology. 110 (3), 257-263 (2004).
- Marshall, R., Maxwell, C. S., Collins, S. P., Beisel, C. L., Noireaux, V. Short DNA containing χ sites enhances DNA stability and gene expression in E. coli cell-free transcription-translation systems. Biotechnology and Bioengineering. 114 (9), 2137-2141 (2017).
- Yim, S. S., Johns, N. I., Noireaux, V., Wang, H. H. Protecting linear DNA templates in cell-free expression systems from diverse bacteria. ACS Synthetic Biology. 9 (10), 2851-2855 (2020).
- Zhu, B., et al. Increasing cell-free gene expression yields from linear templates in Escherichia coli and Vibrio natriegens extracts by using DNA-binding proteins. Biotechnology and Bioengineering. 117 (12), 3849-3857 (2020).
- Tuckey, C., Asahara, H., Zhou, Y., Chong, S. Protein synthesis using a reconstituted cell-free system. Current Protocols in Molecular Biology. 108, 1-22 (2014).
- Hoffman, R. A., Wang, L., Bigos, M., Nolan, J. P. NIST/ISAC standardization study: Variability in assignment of intensity values to fluorescence standard beads and in cross calibration of standard beads to hard dyed beads. Cytometry. Part A. 81 (9), 785-796 (2012).
- Karzbrun, E., Shin, J., Bar-Ziv, R. H., Noireaux, V. Coarse-grained dynamics of protein synthesis in a cell-free system. Physical Review Letters. 106 (4), 048104 (2011).
- Rossi, N. A., Dunlop, M. J. Making waves with synthetic oscillators. Cell Systems. 6 (4), 406-407 (2018).
- Niederholtmeyer, H., et al. Rapid cell-free forward engineering of novel genetic ring oscillators. eLife. 4, 09771 (2015).
- Chang, H., Voyvodic, P. L., Structurale, C. D. B. Microbially derived biosensors for diagnosis, monitoring and epidemiology. Microbial Biotechnology. 10 (5), 1031-1035 (2017).
- Pardee, K., et al. low-cost detection of Zika virus using programmable biomolecular components. Cell. 165 (5), 1255-1266 (2016).
- Pardee, K., et al. Portable, on-demand biomolecular manufacturing. Cell. 167 (1), 248-259 (2016).
- Pardee, K., et al. Paper-based synthetic gene networks. Cell. 159 (4), 940-954 (2014).
- Liu, X., Germaine, K. J., Ryan, D., Dowling, D. N. Whole-cell fluorescent biosensors for bioavailability and biodegradation of polychlorinated biphenyls. Sensors. 10 (2), 1377-1398 (2010).
- Gautam, P., Suniti, S., Amrita, K., Madathil, D., Nair, B. A Review on Recent Advances in Biosensors for Detection of Water Contamination. International Journal of Environmental Sciences. 2 (3), 1565-1574 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon