Method Article
In situ Visualización del crecimiento de axones y la dinámica de los conos de crecimiento en cultivos agudos de cortes cerebrales embrionarios ex vivo
En este artículo
Resumen
Este protocolo demuestra un método sencillo y robusto para estudiar el crecimiento del axón in situ y la dinámica del cono de crecimiento. Describe cómo preparar cortes cerebrales agudos fisiológicamente relevantes ex vivo y proporciona una tubería de análisis fácil de usar.
Resumen
Durante el desarrollo neuronal, los axones navegan por el entorno cortical para llegar a sus destinos finales y establecer conexiones sinápticas. Los conos de crecimiento -las estructuras sensoriales ubicadas en las puntas distales de los axones en desarrollo- ejecutan este proceso. El estudio de la estructura y la dinámica del cono de crecimiento es crucial para comprender el desarrollo axonal y las interacciones con el sistema nervioso central (SNC) circundante que le permiten formar circuitos neuronales. Esto es esencial cuando se idean métodos para reintegrar los axones en los circuitos neuronales después de una lesión en la investigación fundamental y en contextos preclínicos. Hasta ahora, la comprensión general de la dinámica de los conos de crecimiento se basa principalmente en estudios de neuronas cultivadas en dos dimensiones (2D). Aunque indudablemente fundamentales para el conocimiento actual de la dinámica estructural del cono de crecimiento y la respuesta a los estímulos, los estudios 2D tergiversan el entorno fisiológico tridimensional (3D) encontrado por los conos de crecimiento neuronal en el tejido intacto del SNC. Más recientemente, se emplearon geles de colágeno para superar algunas de estas limitaciones, lo que permitió la investigación del desarrollo neuronal en 3D. Sin embargo, tanto los entornos sintéticos 2D como los 3D carecen de señales de señalización dentro del tejido del SNC, que dirigen la extensión y la búsqueda de rutas de los axones en desarrollo. Este protocolo proporciona un método para estudiar axones y conos de crecimiento utilizando cortes cerebrales organotípicos, donde los axones en desarrollo encuentran señales físicas y químicas fisiológicamente relevantes. Al combinar electroporación afinada en el útero y ex útero para entregar escasamente reporteros fluorescentes junto con microscopía de superresolución, este protocolo presenta una tubería metodológica para la visualización de la dinámica del axón y el cono de crecimiento in situ. Además, se incluye una descripción detallada del conjunto de herramientas del análisis de datos de imágenes a largo plazo y de células vivas.
Introducción
Las neuronas son células altamente polarizadas que representan la unidad computacional básica en el sistema nervioso. Reciben y emiten información que se basa en la compartimentación de los sitios de entrada y salida: dendritas y axones, respectivamente1. Durante el desarrollo, los axones se extienden mientras navegan por un entorno complejo increíble para llegar a su destino. La navegación del axón está guiada por el cono de crecimiento, una estructura sensorial ubicada en la punta del axón en desarrollo. El cono de crecimiento es responsable de detectar señales ambientales y traducirlas en la reorganización espacial dinámica de su citoesqueleto 2,3. Las reacciones morfomecánicas resultantes instruyen al cono de crecimiento para que se extienda o se retraiga de la señal desencadenante, lo que lleva a maniobras específicas del axón.
La comprensión actual de la extensión del axón y la dinámica del cono de crecimiento se deriva de estudios que evalúan el crecimiento del axón sobre sustratos bidimensionales (2D) 2,4,5,6,7. Estos estudios pioneros identificaron una interacción sofisticada entre los conos de crecimiento y los sustratos de crecimiento y revelaron diferencias sorprendentes dependiendo de las características del sustrato, como la adhesividad y la rigidez 8,9. Liderados por estos conocimientos, se planteó la hipótesis de que las señales ambientales extracelulares dictaban el crecimiento del axón, con el citoesqueleto del cono de crecimiento ejecutando este crecimiento 2,10,11,12. En particular, las neuronas pueden extender axones en sustratos no adhesivos (por ejemplo, polilisina, poliornitina)13. Además, la rigidez del sustrato puede influir en la tasa de crecimiento del axón independientemente de los complejos adhesivos celulares8. Por lo tanto, el estudio de la dinámica de los conos de crecimiento en sustratos 2D por sí solo no puede modelar con precisión el equilibrio de fuerzas que surgen de la interacción de los conos de crecimiento axonal con entornos tridimensionales (3D) fisiológicamente relevantes, como los que se encuentran in vivo.
Para superar las limitaciones de los ensayos 2D, se ha estudiado el crecimiento del axón y la dinámica del cono de crecimiento en matrices 3D 8,9. Estas matrices plantean un contexto más fisiológico pero permiten estudiar los mecanismos intrínsecos celulares del crecimiento del axón. Permite el examen del cono de crecimiento de una sola célula en una variedad de condiciones y tratamientos farmacológicos9. En tales entornos 3D, los axones mostraron una dinámica citoesquelética distinta y crecieron más rápido que los observados en neuronas cultivadas en 2D9. Estos elegantes estudios demostraron la influencia de una dimensión extra en la reorganización del citoesqueleto del cono de crecimiento y, en consecuencia, en su comportamiento.
A pesar de las aparentes ventajas presentadas por las matrices 3D sobre las superficies 2D para apoyar el desarrollo neuronal nativo y el crecimiento de los axones, siguen siendo un andamio sintético simplificado que no puede reflejar la complejidad de la dinámica observada en el tejido del sistema nervioso central (SNC). Aquí, la administración de plásmidos reporteros por electroporación ex utero e in utero se combinó con cultivo de rebanadas organotípicas cerebrales e imágenes de superresolución en vivo in situ para analizar la dinámica del cono de crecimiento dentro de un contexto fisiológico. Esta metodología permite visualizar los axones en desarrollo mientras se experimenta la 3-dimensionalidad de los ambientes in vivo y la complejidad de su composición fisicoquímica. Por último, se describen procedimientos fáciles de usar para medir el crecimiento del axón y la dinámica del cono de crecimiento utilizando software comúnmente licenciado y disponible públicamente.
Protocolo
Los experimentos con animales deben cumplir con las regulaciones institucionales y federales pertinentes. En este protocolo se utilizaron ratones embrionarios C57BL/6JRj hembras embrionarias de día 15.5 y 12.5 (E15.5). Los experimentos se realizaron de acuerdo con la Ley de Bienestar Animal de la Agencia Ambiental Estatal de Renania del Norte-Westfalia (Landesamt für Natur, Umwelt und Verbraucherschutz (LANUV)).
1. Preparación de plásmidos para inyección
- Aislar el ADN utilizando el kit Maxiprep libre de endotoxinas de acuerdo con el protocolo del fabricante (ver Tabla de Materiales).
- Mezcle el ADN seleccionado a la concentración deseada (Tabla 1) y la solución Fast Green al 10% (consulte la Tabla de materiales) para visualizar la entrega de la mezcla de ADN en los ventrículos cerebrales.
NOTA: Se utilizaron plásmidos específicos para el etiquetado disperso de las neuronas corticales (Figura 1A), las estructuras filamentosas de actina (F-actina) en el cono de crecimiento (Figura 1B) y el doble marcado de los conos de crecimiento dentro de la misma corteza (Figura 1C). Todos los plásmidos (Tabla 1) utilizados en este protocolo han sido depositados en Addgene (ver Tabla de Materiales). - Prepare los capilares de vidrio utilizando un conjunto de extractores de electrodos capilares según el siguiente programa: presión: 500, calor: 800, tracción: 30 y velocidad: 40.
- Cargue 15 μL de mezcla DNA/Fast Green en cada capilar de vidrio utilizando puntas de pipeta de microcargador.
NOTA: Asegúrese de que no se formen burbujas. - Almacene los capilares llenos de ADN en un plato de 10 cm con un trozo de arcilla de modelado a través del diámetro del plato. Los capilares se pueden cargar y almacenar a 4 °C el día antes del experimento. Selle el extremo posterior del capilar con una película flexible para evitar el secado.
2. Preparación de soluciones
- Prepare la solución de sal tamponada de Hank suplementada con glucosa (HBSS-G).
- Agregue 0.5% de 20% de contenido de glucosa a una botella de 1x HBSS. Mezclar bien y conservar a 4 °C durante un máximo de 2 semanas. Para la extracción embrionaria, bubble HBSS-G solución con carbógeno (95% O2 y 5% CO2) utilizando piedra burbujeante poco antes de la recolección del embrión.
- Solución de medios slice
- Prepare un medio de rebanada fresco que contenga Neurobasal 1x, 5% de suero de caballo, 5% de suero fetal de terneros, suplemento de B27 1:50, suplemento de L-glutamina 1:400, penicilina-estreptomicina 1:200 y suplemento de Neuropan-2 1:100 (a pH = 7.3), en condiciones estériles (ver Tabla de Materiales).
- Preparar platos de 3 cm con 1 ml de media en rodajas cada uno. Coloque en la incubadora a 35 °C con un 5% de CO2 durante al menos 1 h antes del experimento para equilibrar el pH del medio a través del intercambio de gases.
NOTA: El equilibrio del pH de los medios es causado por la acidificación de los medios por el CO2 de la incubadora. Los medios de corte se pueden almacenar a 4 °C durante un máximo de 1 semana.
- Solución de agarosa de bajo punto de fusión (3%)
- Pesar la cantidad deseada de polvo de agarosa de bajo punto de fusión y disolver en un volumen apropiado de 1x HBSS-G en una botella de vidrio. Se necesitan aproximadamente 7 ml de solución de agarosa por cerebro.
- Coloque la botella en un microondas durante 2-3 minutos, con la tapa suelta y agite cada 10-20 s.
- Una vez que el polvo se haya disuelto por completo, coloque la botella en un baño de agua o en un baño de cuentas a 37 ° C al menos 1 h antes del experimento para permitir que la agarosa se enfríe.
NOTA: Se recomienda calentar la agarosa dos veces durante 15 minutos para asegurarse de que el polvo de agarosa se disuelva. Esto es crucial para la correcta adhesión de la agarosa al tejido cerebral. Se debe usar un termómetro para medir la temperatura de la solución de agarosa mientras se incrustan los cerebros, asegurándose de que esté entre 37-40 ° C. Los cerebros de diferentes animales envejecidos tienen una rigidez diferente. Se recomienda probar un rango de concentraciones de agarosa para encontrar homogeneidad entre el tejido y la agarosa. - Prepare solución salina tamponada con fosfato con 0.3% Triton X-100 (PBS-T).
- Prepare solución salina tamponada con fosfato con azida de sodio al 0,2% (PBS-NaN3).
NOTA: Las soluciones descritas en los pasos 2.4-2.5 se utilizan en la etapa posterior de inmunohistoquímica.
3. Preparación de la estación de cirugía
- Limpie la estación de cirugía con etanol al 70% -96% y coloque la base de operación en la superficie de la estación.
- Esterilice los instrumentos de cirugía enjuagando con etanol al 70% -96%, seguido de esterilización en seco en un esterilizador de cuentas calientes.
- Limpie los electrodos de las pinzas de platino (consulte la Tabla de materiales) con 70% -96% de etanol antes de conectarlos al generador de impulsos.
- Inserte un capilar de vidrio relleno de ADN / Fast Green en el soporte capilar. Inmediatamente antes de usar, rompa suavemente la punta capilar con tijeras finas y pruebe el flujo de la solución dentro de un tubo de microcentrífuga de 1,5 ml lleno de solución salina o agua precalentada.
NOTA: La Figura 2A, B muestra la configuración de la estación de cirugía y las herramientas utilizadas para la electroporación ex utero (EUE) y la electroporación in utero (IUE). - Para la IUE, calentar la solución salina a 37 °C en un baño de agua.
4. Extracción de embriones
- Coloque el ratón preñado en la cámara inductora de anestesia con 5% de isoflurano hasta que el ratón esté profundamente anestesiado. Confirmar la anestesia por la ausencia del reflejo de retirada del pedal.
- Transfiera el ratón a una capa inferior de operación y mantenga el isoflurano al 1,5%-2% a través de un cono nasal.
- Aplique ungüento en ambos ojos para evitar el secado corneal.
- Afeitar el abdomen del ratón y luego usar una gasa empapada en etanol al 70% -96% para eliminar el vello afeitado. Limpie el área con betadina.
- Usando tijeras quirúrgicas pequeñas estériles, haga una incisión de piel de 2 cm a lo largo de la línea media abdominal, seguida de una incisión muscular de 1,5 cm.
NOTA: El tamaño de la incisión depende del tamaño del embrión. De hecho, los embriones más grandes requerirán una incisión más grande para acomodar su extracción. - Corte un agujero en el medio de la gasa lo suficientemente ancho como para que quepa la incisión en la piel (~ 2 cm de diámetro), sumérjalo con solución salina tibia y colóquelo alrededor de la abertura abdominal.
- Saque ambos cuernos uterinos usando un bastoncillo de algodón empapado en solución salina tibia o usando fórceps, agarrando cuidadosamente los espacios entre los embriones para sacarlos. Coloque los embriones sobre una gasa húmeda (Figura 2C).
NOTA: Incluso un pequeño daño a los vasos sanguíneos y capilares alrededor de los cuernos uterinos probablemente resultará en sangrado profuso. Por lo tanto, evite el manejo directo de estas áreas vascularizadas en todo momento. - Abra el saco uterino y extraiga cada embrión (Figura 2D).
- Eutanasiar cada embrión inmediatamente después de la extracción a través de un corte diagonal descendente, asegurando la transección completa de la médula espinal (Figura 2E).
- Coloque los embriones en un plato de 10 cm que contenga HBSS-G sobre hielo.
NOTA: Se evita la decapitación del embrión para evitar la fuga de la mezcla de ADN / Fast Green del cerebro y facilitar el posicionamiento fácil del embrión en el soporte (ver electroporación ex utero , paso 5). - Sacrificar a la madre inmediatamente después de la extracción de embriones mediante la realización de la luxación cervical.
NOTA: Aquí, la madre es sacrificada bajo anestesia para evitar que sufra más después del procedimiento en cumplimiento con el protocolo aprobado por la Ley de Bienestar Animal de la Agencia Ambiental Estatal de Renania del Norte-Westfalia (LANUV).
5. Electroporación ex utero (EUE)
- Recoge un embrión y colócalo en el soporte.
NOTA: Se utiliza una punta de pipeta cortada de 1 ml unida al extremo de un raspador celular como soporte de embriones. Es esencial mantener los brazos de los embriones fuera de la punta durante el procedimiento para evitar que se deslicen hacia la punta (Figura 2F-I). El diámetro de la punta de la pipeta es fácilmente ajustable para acomodar embriones de varios tamaños. Corte una segunda punta en longitud donde el diámetro de la punta coincida con el tamaño del embrión y úsela como un inserto adaptador para el soporte mencionado anteriormente. - Inserte cuidadosamente el capilar de vidrio relleno de ADN / Fast Green a través del cráneo del embrión en el ventrículo lateral e inyecte 2-3 μL de mezcla de plásmidos de ADN (Figura 1A, B; Tabla 1) en cada ventrículo (Figura 2F).
NOTA: Utilice las suturas lambdoidales y sagitales como guía para la ubicación de la inyección de ADN. Las suturas lambdoidal y sagital son articulaciones fibrosas que conectan la placa ósea del cráneo. El primero une el hueso parietal con el hueso occipital, y el segundo se une a los dos huesos parietales. - Sostenga la cabeza del embrión entre los electrodos de pinza de platino en el ángulo apropiado para apuntar al área cerebral deseada (ángulo de 60 ° en este caso), con el cátodo mirando hacia el área donde se pretende la transferencia de ADN (Figura 2G-H).
- Aplicar cinco pulsos a 30 mV con un intervalo de 1 s y una duración de 50 ms utilizando un generador de pulsos de onda cuadrada.
NOTA: Tenga en cuenta que los cerebros en EUE experimentan un campo eléctrico más efectivo que los de IUE. Por lo tanto, a una concentración de ADN dada, eue da como resultado una mayor eficiencia de transferencia de ADN que IUE, y las concentraciones de ADN deben ajustarse en consecuencia. - Si se desea electroporación bilateral, repita los pasos 5.3-5.4 con el cátodo y el ánodo reflejando la posición anterior para apuntar a la corteza contralateral.
NOTA: Dado que ambos ventrículos fueron inyectados con ADN, las cortezas de ambos hemisferios fueron atacadas. - Coloque el embrión electroporado en un plato de 6 cm que contenga HBSS-G helado. Repita los pasos 5.1-5.6 para todos los embriones requeridos.
6. Electroporación in utero (IUE)
- Inyecte analgésico al ratón embarazada; 50 μL de buprenorfina (0,1 mg/kg) (ver Tabla de materiales) por vía subcutánea, 20 min antes del procedimiento.
- Realice los pasos 4.1-4.8 desde la sección de extracción de embriones.
NOTA: Evite dejar los embriones expuestos innecesariamente cubriéndolos con una gasa estéril empapada en solución salina tibia. - Usando las yemas de los dedos, gire suavemente el embrión dentro del útero hasta que se localicen las suturas lambdoidales y sagitales (Figura 2J). Inserte cuidadosamente el capilar de ADN / vidrio verde rápido a través de la pared uterina y el cráneo del embrión en el ventrículo lateral e inyecte 2-3 μL de mezcla de plásmidos de ADN (Figura 1A, C) en uno o ambos ventrículos según lo desee (Figura 2K-L).
NOTA: La presión excesiva de los dedos sobre los cuernos uterinos podría provocar el colapso del saco amniótico. - Sostenga la cabeza del embrión entre los electrodos de pinza de platino en el ángulo apropiado para apuntar al área cerebral deseada (ángulo de 60 ° en este caso), con el cátodo mirando hacia el área donde se pretende la transferencia de ADN. Evite apretar el útero ya que puede causar el colapso del saco amniótico (Figura 2M).
- Aplicar cinco pulsos a 35 mV con un intervalo de 600 ms y una duración de 50 ms utilizando un generador de pulsos de onda cuadrada.
- Si se inyectaron ambos ventrículos laterales, repita los pasos 6.5-6.6 con el cátodo y el ánodo reflejando la posición anterior para apuntar a la corteza contralateral.
- Repita los pasos 6.3-6.6 para todos los embriones requeridos.
- Una vez que todos los embriones requeridos hayan sido electroporados, use un bastoncillo de algodón empapado en solución salina para colocar los cuernos uterinos dentro de la cavidad abdominal suavemente.
NOTA: La adición de solución salina en la cavidad peritoneal ayudará a que los cuernos uterinos se deslicen de nuevo en su posición. - Suturar incisiones musculares y cutáneas utilizando material de sutura 5-0. Use clips de sutura para asegurar la herida y desinfecte la herida de sutura rociándola con betadina (Figura 2N-P).
- Inyecte al ratón 200 μL de glucosa al 5% por vía subcutánea.
- Inyecte al ratón con un antibiótico; 50 μL de enrofloxacina (5 mg/kg) por vía subcutánea (ver Tabla de Materiales).
- Coloque el ratón de nuevo en la jaula de recuperación y mantenga el calor utilizando una luz de calentamiento de infrarrojo lejano o una almohadilla térmica durante al menos 20 minutos después del procedimiento (Figura 2Q).
- Monitoree al ratón diariamente e inyecte meloxicam después del procedimiento para aliviar el dolor siguiendo las pautas institucionales y federales.
- Extraiga los embriones 2 días después del procedimiento (es decir, E17.5) después del paso 4.
7. Extracción cerebral e incrustación en agarosa
NOTA: Se recomienda llevar a cabo los siguientes pasos bajo un microscopio de disección para una mejor precisión. Evitar el daño al cerebro es fundamental para el éxito del procedimiento.
- Instale las herramientas de extracción en un espacio de trabajo estéril bajo una campana de disección (Figura 3A).
- Separe la cabeza de un embrión del resto del cuerpo usando tijeras de disección.
- Fije la cabeza como se muestra en la Figura 3C, y luego retire la piel y el cráneo cortando a lo largo de la línea media, comenzando desde la base de la cabeza hacia la nariz (Figura 3D).
- Pelar la piel y el cráneo lateralmente, haciendo un espacio lo suficientemente grande (~ 1 cm) para que el cerebro sea extirpado.
- Para extraer el cerebro, inserte la punta cerrada de las tijeras de disección estériles, comenzando desde debajo del bulbo olfatorio que se mueve hacia el tronco encefálico (Figura 3E).
- Corte el tronco encefálico y recorte cualquier trozo suelto de meninges alrededor del cerebro (Figura 3F).
NOTA: Las meninges sueltas a menudo hacen que las rodajas permanezcan adheridas al bloque de agarosa después del corte, lo que lleva a la separación del tejido de la agarosa durante la recolección de la rebanada. - Repita los pasos 7.1-7.6 para todos los embriones y mantenga los cerebros en hielo (idealmente no más de 30 minutos) hasta el paso de incrustación.
NOTA: Los siguientes pasos 7.7.1-7.7.4 se refieren a la extracción cerebral de cerebros E12.5.- Aísle la parte superior de la cabeza justo debajo del ojo, como se muestra en la Figura 3G.
- Corte la piel y el cráneo en la parte superior del tronco encefálico, siguiendo la línea discontinua como se muestra en la Figura 3H, sin extirpar el tronco encefálico.
- Haga una incisión piel-cráneo de 2 mm en la parte posterior de la cabeza, como se muestra en la Figura 3I (ver dibujos para mayor claridad).
NOTA: Esta incisión proporciona puntos de agarre iniciales para despegar las capas de piel y cráneo. Por lo general, se desprenden como una capa. El tamaño típico de la incisión es de 2 mm, correspondiente a la longitud del filo de corte de las tijeras de micro resorte utilizadas. - Comience a despegar las capas de piel y cráneo asegurando un lado de la incisión y tirando del otro con cuidado. Con el mismo cuidado, finalice despegando la base de la cabeza hasta liberar el cerebro (Figura 3J).
NOTA: Esto debe hacerse con mucho cuidado, observando que el cerebro no está siendo arrastrado a lo largo de las capas de tejido. Lados alternativos para eliminar el tejido que cubre el cerebro.
- Vierta la agarosa tibia (a 37-40 °C) en un plato de 3 cm.
- Recoja el cerebro con una cuchara perforada y elimine el exceso de líquido frotando la parte inferior de la cuchara contra el papel de seda seco. Coloque el cerebro en un plato de agarosa.
NOTA: Es esencial eliminar la mayor cantidad de líquido de alrededor del cerebro como sea posible para permitir una mejor adhesión de la agarosa al tejido. - Poner el plato con agarosa líquida sobre hielo. Usando una cuchara más pequeña, mezcle la agarosa durante 10 s para un enfriamiento uniforme. Maniobra el cerebro hasta el centro del plato. Coloque el cerebro horizontalmente en el plato con el lado dorsal hacia arriba, asegurándose de que esté completamente cubierto con agarosa desde todas las direcciones (Figura 3K).
NOTA: Los cerebros a menudo se hunden en el fondo del plato una vez colocados en agarosa; levante el cerebro con una cuchara pequeña hasta que se establezca un espacio de 1-2 mm debajo del cerebro. - Repita los pasos 7.8-7.10 para todos los cerebros.
- Una vez que la agarosa se haya polimerizado, agregue 500 μL de HBSS-G sobre el bloque de agarosa para evitar el secado. Luego, cubra el plato con hielo.
NOTA: Mantenga la muestra en hielo durante 5 minutos antes de seccionar para permitir que la temperatura del cerebro alcance los 4 ° C.
8. Cultivo organotípico de rebanadas
NOTA: Limpie el vibratomo y las superficies circundantes con etanol al 70% -96% para evitar la contaminación de las rodajas. La configuración de la estación de trabajo vibratome (consulte la Tabla de materiales) se muestra en la Figura 3B.
- Llene la bandeja tampón del vibratomo con HBSS-G frío y la bandeja exterior con hielo para mantener el HBSS-G frío durante todo el procedimiento.
- Suministre continuamente HBSS-G en la bandeja tampón con carbógeno utilizando una piedra burbujeante.
- Usando una cuchilla fresca, haga un corte grande (~ 2 x 2 cm) alrededor del cerebro y retire un bloque de agarosa que contiene el cerebro, con suficiente agarosa circundante para recortar la agarosa en un pequeño bloque rectangular.
NOTA: Este paso permite ajustar el ángulo del bloque para que el eje sagital del cerebro sea perpendicular a la placa vibratómica y el eje coronal se alinee paralelo a la cuchilla. Deje alrededor de 5 mm de agarosa en el lado dorsal del cerebro para facilitar el manejo de las rodajas. - Coloque una pequeña gota de superpegamento sin disolvente de adhesivo rápido en el centro del soporte de la muestra y extiéndala a un área que cubra la parte inferior del bloque de agarosa.
- Recoja suavemente el bloque de agarosa y seque la parte inferior frotando contra el papel de seda. Coloque el bloque en el área pegada del soporte de la muestra, con el lado rostral del cerebro hacia arriba. Coloque el soporte de la muestra en hielo y deje que el pegamento se seque durante 1 minuto.
- Una vez que el pegamento se haya secado, coloque el soporte de la muestra en la bandeja tampón.
- Cortar el cerebro en rodajas coronales en un ángulo de 15°.
NOTA: El grosor de las rodajas puede variar dependiendo de la aplicación. Aquí, los cerebros se cortaron en rodajas a un grosor de 150 μm. Ajuste la velocidad del vibratomo a 1.0-1.5 mm / s para recortar el exceso de agarosa en la parte superior y recortar los bulbos olfativos. Reduzca la velocidad de corte a 0,5 mm/s para recoger rodajas corticales para su análisis. La mayoría de los vibratomas se pueden pausar para recoger cada rebanada. Si se experimenta una calidad reducida de las rodajas o la separación del tejido de la agarosa, la reducción de la velocidad de corte o el reemplazo de la cuchilla del vibratomo pueden ayudar. - Usando espátulas limpias, recoja rebanadas de cerebro y colóquelas sobre la membrana de politetrafluoroetileno (PTFE), inmovilizadas en un plato con fondo de vidrio de 35 mm con parafina (hasta cinco rebanadas de cerebro / membrana) (Figura 3L-M).
NOTA: Fije la membrana de PTFE dentro de un plato con fondo de vidrio de 35 mm con cera. Esto estabilizará la membrana al agregar el medio de cultivo de la rebanada y también durante la toma de imágenes. - Usando una pipeta de 200 μL, retire el exceso de HBSS-G de alrededor de las rodajas en la membrana de PTFE, dejando las rodajas semisecas.
- Añadir 500 μL de purán (precalentado a 35 °C) directamente al espacio debajo de la membrana de PTFE.
NOTA: No se deben formar burbujas debajo de la membrana al agregar el medio. Esto dejará cortes enteros o parciales sin intercambio de medios. Reemplace 200 μL de medios cada 2 días en cultivo o después de cada sesión de imágenes. - Incubar las rodajas a 35 °C con un 5% de CO2.
9. Inmunohistoquímica
- Fije las rodajas con 1 ml de paraformaldehído al 4% (PFA) suplementadas con sacarosa al 4% por plato. Incubar en RT durante 30 min.
PRECAUCIÓN: Cuando manipule PFA, use una bata de laboratorio y guantes. Realice los pasos de fijación bajo una capucha química y elimine los desechos de PFA de manera adecuada. - Lave las rodajas dos veces con 300 μL PBS durante 5 min. Transfiera las rodajas a un plato de 24 pocillos.
NOTA: El experimento se puede pausar en esta etapa. Añadir PBS-NaN3 a las rodajas y conservar a 4 °C. NaN3 es un compuesto tóxico; cuando manipule soluciones con él, use una bata de laboratorio y guantes. Los pasos 9.3-9.10 deben realizarse en un agitador orbital. - Apague las rodajas con 300 μL de 0,1 M de glicina a 4 °C durante la noche.
- Lave la glicina con PBS a RT 3x durante 10 min.
- Permeabilizar las rodajas con 300 μL de PBS-T en RT durante 2 h.
- Bloquee usando suero de cabra al 10% en PBS-T en RT durante 2 h.
- Añadir 300 μL de anticuerpo primario (anticuerpo antivimentina a una dilución de 1:200; ver Tabla de Materiales) diluido en suero de cabra al 10% en solución de PBS-T a 4 °C durante la noche.
NOTA: En los pasos 9.8-9.12, las rebanadas estaban protegidas contra la luz para evitar la pérdida de fluorescencia. - Lavar el anticuerpo primario con PBS a RT 3x durante 20 min.
NOTA: PbS se utilizó en lugar de PBS-T para lavar Triton X-100. - Agregue 300 μL de anticuerpo secundario (ya sea Alexa Fluor 488 o 647 a una dilución de 1:400; consulte la Tabla de materiales) en PBS en RT durante 2 h.
NOTA: DAPI se agrega inmediatamente después de eliminar el anticuerpo secundario a una dilución de 1:10,000 durante 5 min. - Lave el anticuerpo secundario con PBS a RT 3x durante 20 min. Lavar con agua destilada 2x durante 1 min.
- Transfiera las rodajas a un portaobjetos de vidrio con un cepillo fino y luego séquelas a 30 °C durante 20 min.
- Monte las rodajas utilizando un medio de montaje acuoso. Mantenga las diapositivas en RT durante la noche para que los medios de montaje se curen.
10. Adquisición de imágenes
NOTA: Independientemente del enfoque de entrega de ADN (IUE o EUE), las rebanadas se analizaron en el mismo rango de edad de desarrollo (E17.5-E18.5). IUE permite que los progenitores neuronales se dividan y se desarrollen durante dos días más in vivo. EUE, por otro lado, permite el seguimiento de eventos de desarrollo tempranos.
- Encienda la incubadora de cámara y configúrela a 35 °C con un 5% de CO2, idealmente 4 h antes de la obtención de imágenes, para permitir que los componentes del microscopio se equilibren a 35 °C.
- Para obtener imágenes profundas de rodajas, use objetivos de inmersión en agua para reducir el desajuste en el índice de refracción entre el tejido y el objetivo.
NOTA: Aquí, se utilizó el modo de imagen de superresolución. La obtención de imágenes a través de la membrana de PTFE requiere un objetivo con una larga distancia de trabajo (~ 1 mm). Si no se dispone de un objetivo de larga distancia de trabajo, las rebanadas se pueden transferir a un plato con fondo de vidrio de 8 pocillos. Para transferir rebanadas, agregue 1 ml de medios de rebanada a la parte superior de la membrana y luego use una espátula para levantar una rebanada y transferirla a un pozo que contenga 200 μL de medio. Retire el exceso de medios con una punta de pipeta de 1 ml dejando las rodajas semisecas. - Para obtener imágenes del crecimiento del axón, localice una región de la corteza con densidad celular baja a media. Para obtener imágenes de la dinámica del cono de crecimiento, localice un cono de crecimiento en la zona intermedia o zona subventricular de la corteza.
- Defina un tamaño de pila z en el software de procesamiento de imágenes (consulte la Tabla de materiales). Para el crecimiento del axón en una pila z grande, establezca un tamaño de paso de 2 μm. Para los conos de crecimiento en una pila z más pequeña, establezca un tamaño de paso de 1 μm.
NOTA: Siempre tenga en cuenta el movimiento potencial del cono de crecimiento y el axón a través de los planos x, y y z. Los axones crecen a un ritmo mucho mayor en cultivos organotípicos que en cultivos in vitro . Aquí, una pila z de alrededor de 80 μm para obtener imágenes del crecimiento del axón fue suficiente. Para la dinámica del cono de crecimiento, una pila z de ~ 6 μm fue adecuada. - Para obtener imágenes del crecimiento axónico de las neuronas en un área más grande, defina una exploración de mosaicos.
- Utilice la potencia láser más baja posible para minimizar las posibilidades de blanquear los conos de crecimiento durante la adquisición.
- Para el crecimiento del axón por imágenes, adquiera lapsos de tiempo durante 2 h con un intervalo de 5 min. Para la dinámica del cono de crecimiento de imágenes, adquiera lapsos de tiempo durante 2-5 min con un intervalo de 2.5-3 s.
11. Análisis de datos
- Medir la velocidad de crecimiento del axón usando kymographs
- Abra el archivo de imagen en Fiji14 a través de archivo > Abrir y seleccione la imagen.
- Obtenga la proyección de máxima intensidad del lapso de tiempo a través de Image > Stacks > Z-Projection > Maximum Intensity Projection.
- Revisa el lapso de tiempo y localiza un axón en crecimiento.
- Una vez localizado, dibuja una línea a través del axón en crecimiento. Comience desde la punta del axón en el primer fotograma y siga el axón a través de todo el lapso de tiempo.
- Genera un kymograph usando el plugin KymoResliceWide.
- Establezca la escala del kymograph yendo a Propiedades de la > de la imagen. Establezca la distancia en μm en Pixel Width y establezca el tiempo en s o min en Pixel Height.
- Vaya a Analizar > medir.
NOTA: Se dará un ángulo relativo al eje x. - Calcule la velocidad de crecimiento del axón sustituyendo el ángulo en la siguiente ecuación: SIN(RADIANS(θ))/COS(RADIANS(θ)) en una hoja de cálculo.
- Mida el volumen del cono de crecimiento utilizando un software de análisis de imágenes (consulte la Tabla de materiales).
- Abra el archivo de imagen en el software de análisis de imágenes a través de Archivo > Abrir y seleccione el archivo de interés.
- Seleccione el asistente Agregar nuevas superficies .
NOTA: Aparecerá una sección en la esquina inferior izquierda con seis pasos para la edición manual. - En el paso 1 -en Configuración del algoritmo- seleccione Segmentar solo una región de interés. En el paso 2, recorte el marco para que quepa todo el cono de crecimiento en todos los marcos.
- Mantenga el umbral a Intensidad absoluta en el paso 3 y asegúrese de que toda la región del cono de crecimiento esté umbralizada en el paso 4.
- En el paso 5, seleccione Número de vóxeles lmg = 1 en Tipo de filtro.
NOTA: En el último paso, se pueden crear varios conjuntos de medición. Aquí, solo se creó una medición para el volumen. - Seleccione el botón Ejecutar para realizar todos los pasos de creación y finalizar el Asistente para agregar nuevas superficies.
- En la pestaña Estadísticas en la parte superior de la ventana del asistente, seleccione Valores específicos y volumen en la pestaña Detallado .
Resultados
Se muestran los resultados representativos obtenidos con el flujo de trabajo del método descrito. En la presente demostración se utilizaron ratones E15.5, aunque este protocolo es fácilmente adaptable a prácticamente todas las edades embrionarias que van desde E11 hasta finales de E17. En este protocolo, ya sea electroporación ex utero (EUE; Figura 2A, 2C-I) o electroporación in utero (IUE; Figuras 2B, C y 2J-Q) se utilizaron para administrar plásmidos en las neuronas progenitoras que recubren los ventrículos laterales. Estos progenitores son la fuente de futuras neuronas proyectantes corticales (CPN)15,16. Se prepararon mezclas de plásmidos para impulsar la expresión dispersa específica de neuronas de mNeonGreen (Figura 1A) dirigida a la membrana (Lyn)-mNeonGreen (Figura 1A) o mejorada por LifeAct (E) GFP (Figura 1B) para evaluar el comportamiento general y la dinámica de la actina en los conos de crecimiento, respectivamente. Además, se incluyó una mezcla de plásmidos destinada a etiquetar neuronas individuales con turbo(t)-RFP o zoanthus sp. (Zs) proteína fluorescente verde (ZsGreen) (Figura 1C). Esto facilita el monitoreo del comportamiento del cono de crecimiento de las neuronas vecinas independientes.
La disección cerebral a partir de embriones electroporados es un paso crucial que debe realizarse cuidadosamente para obtener rebanadas de alta calidad, preservando la estructura cerebral nativa. Los instrumentos de disección y el vibratomo se prepararon de antemano y se esterilizaron cuidadosamente con etanol (Figura 3A, B). A continuación, se diseccionaron cuidadosamente las cabezas de los embriones electroporados y se extrajeron los cerebros. Aquí, se muestra la disección representativa de cerebros de los embriones sometidos a EUE en E15 (Figura 3C-F) y E12.5 (Figura 3G-J). Los cerebros se encierran inmediatamente en una matriz de agarosa, se cortan en rodajas y se colocan en insertos de membrana de PTFE dentro de un plato de vidrio inferior para la incubación (Figura 3K-M).
El estado de salud de las rebanadas cerebrales es un punto importante para el control para garantizar resultados confiables. Diariamente se realizaba una inspección visual para detectar cualquier contaminación. Además, una vez finalizado el cultivo, las rebanadas cerebrales se fijan y se someten a inmunohistoquímica. Aquí,4′,6-diamidino-2-fenilindol (DAPI) se utilizó para controlar la organización celular general y la tinción de vimentina para revelar la organización glial; en particular, andamio de glía radial (RG). Por lo general, las rebanadas cerebrales cultivadas con éxito derivadas de IUE o EUE muestran una distribución celular normal según lo revelado por DAPI y una matriz algo organizada de RG con procesos de contacto pial orientados apicalmente17 (Figura 4A, B respectivamente). Ocasionalmente, se observan marcadas alteraciones en el andamiaje RG en cortes cerebrales cultivados, especialmente en los derivados de la electroporación EUE (Figura 4C). Las rebanadas cerebrales con andamio RG extremadamente desorganizado muestran una migración neuronal deteriorada y un crecimiento defectuoso del axón (no se muestra). Por lo tanto, controlar el andamio RG es un método fácil después del cultivo para clasificar los datos obtenidos de cortes cerebrales confiables.
Las rebanadas cerebrales derivadas de IUE o EUE con una mezcla de plásmidos que expresan Lyn-mNeonGreen dan como resultado un etiquetado de neuronas disperso similar. Como ejemplo se muestra un CPN piramidal representativo que expresa Lyn-mNeonGreen y el comportamiento dinámico de su cono de crecimiento (Figura 5A y Video Suplementario 1, arriba a la izquierda). Además, las neuronas se etiquetaron utilizando un plásmido que expresa una sonda de actina para analizar la dinámica de la actina de los conos de crecimiento axonal in situ (Figura 5B y Video complementario 1, abajo a la izquierda). También se realizaron experimentos in situ con un diseño de plásmido que expresa fluoróforos de doble Cre/Dre (Figura 1C y Video Complementario 1, derecha). Los fluoróforos tRFP o ZsGreen en este plásmido podrían activarse específica e individualmente por recombinasas Dre o Cre, respectivamente, en neuronas vecinas (Figura 5C). Esta línea experimental permite el análisis lado a lado de los conos de crecimiento de las neuronas de control con neuronas modificadas vecinas (cualquier pérdida o ganancia de función dada). Esto evita la variabilidad que surge del uso de diferentes cortes para probar las condiciones de control y experimentales.
Se analizaron los kymógrafos generados a partir de la película grabada, a partir de los cuales se pueden obtener fácilmente parámetros de crecimiento dinámico como la actividad protuberante a lo largo del tiempo y la longitud del crecimiento (Figura 6A). Tenga en cuenta que un simple ajuste en la resolución temporal del lapso de tiempo permite medir la velocidad de elongación del axón durante 2 h (Figura 6A). Además, la variación del volumen del cono de crecimiento a lo largo del tiempo, una medida de la actividad dinámica del cono de crecimiento general, se puede obtener fácilmente, en este caso con software con licencia (Figura 6B y Figura 6E, F). Esto se puede utilizar para evaluar la velocidad de la cinta de correr con actina y el equilibrio de filopodia / lamellipodia durante la actividad de exploración del cono de crecimiento.
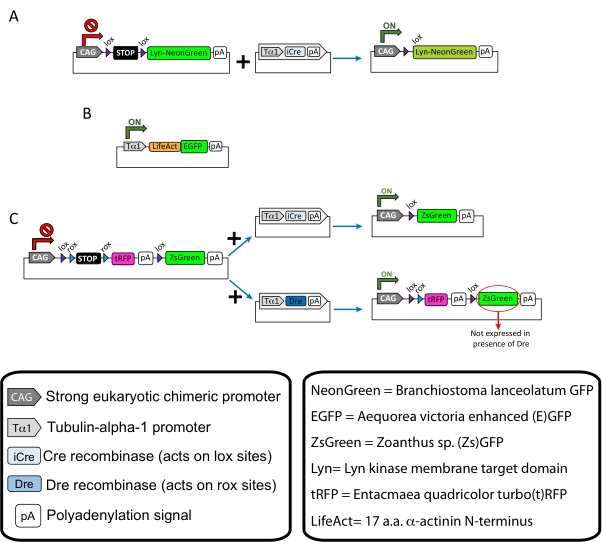
Figura 1: Esquemas de los plásmidos utilizados en el protocolo. (A) pCAG-lox-STOP-lox-Lyn-mNeonGreen. (B) p-Tub-alfa-1-LifeAct-GFP. (C) pCAG-lox-rox-STOP-rox-tRFP-pA-lox-ZsGreen-pA. La información relevante sobre los componentes del plásmido y el origen del fluoróforo se encuentra en las cajas. Haga clic aquí para ver una versión más grande de esta figura.
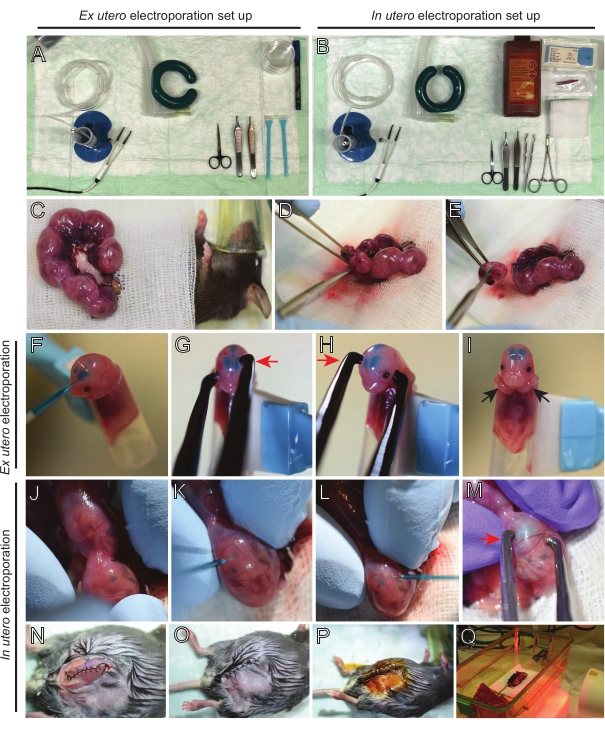
Figura 2: Flujo de trabajo de electroporación ex utero e in utero de ratones E15.5. (A) Instalación de una estación de cirugía para la electroporación ex utero. (B) Instalación de una estación de cirugía para la electroporación in utero. (C) Cuernos uterinos tirados fuera de la cavidad abdominal del ratón anestesiado. (D) Extracción de un embrión del saco uterino. (E) Sacrificio de embriones por transección completa de la médula espinal a través de una incisión diagonal; nótese que se evitó la decapitación. (F) Colocación del embrión en el soporte e inyectado con mezcla de ADN/Fast Green en el ventrículo lateral izquierdo. (G,H) Coloque la cabeza del embrión entre los electrodos de pinza de platino con el cátodo (flecha roja) sobre la corteza en un ángulo de 60°. (I) Colocación de los brazos del embrión (flechas negras) fuera del soporte para evitar el deslizamiento del embrión durante el procedimiento. (J) Rotación del embrión dentro del saco uterino para exponer la cabeza. (K,L) Inyección de la mezcla de ADN/Fast Green en los ventrículos laterales del embrión a través de la pared uterina. (M) Coloque la cabeza del embrión entre los electrodos de pinza de platino con un cátodo (flecha roja) sobre la corteza en un ángulo de 60 °. (N) Incisión muscular suturada a través de la sutura de bloqueo de carrera. (O) Incisión en la piel suturada a través de una sutura interrumpida. (P) Aseguramiento de la herida con clips quirúrgicos para heridas y desinfección con betadina. (Q) Colocación del ratón en la jaula de recuperación con luz de calentamiento infrarroja lejana. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Extracción de cerebros E15.5 y E12.5 y procedimiento de cultivo organotípico de rebanadas. (A) Herramientas utilizadas para el procedimiento de extracción cerebral. B) Instalación de una estación de cultivo organotípico. (C-F) Extracción de E15.5 cerebral. (G-J) Extracción de E12.5 cerebral. Las líneas punteadas resaltan la ubicación de las incisiones. Las flechas rojas señalan la dirección de tirar con fórceps. (K) Incrustar el cerebro en un plato de 3 cm que contiene 3% de agarosa de fusión baja, dejando un espacio de espaciamiento de agarosa de 1-2 mm debajo del cerebro. (L) Colección de rebanada de cerebro de 150 μm. (M) Colocación de rodajas cerebrales en insertos de membrana de PTFE inmovilizados en un plato de 35 mm utilizando película de parafina (flecha azul). La marca de estrella roja indica una colección dada de corte cerebral del vibratomo (L) y su transferencia a la membrana de PTFE (M). Haga clic aquí para ver una versión más grande de esta figura.
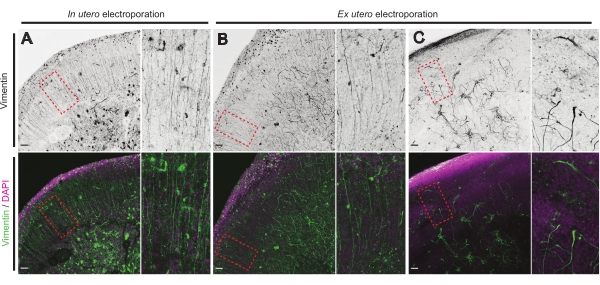
Figura 4: Estructura de células gliales radiales conservadas en rodajas organotípicas sanas. Imágenes confocales de cortes cerebrales E17.5 que revelan la matriz RG (vimentina; verde) y la organización celular general (DAPI; magenta) después de IUE (A) y EUE (B, C). Tenga en cuenta las fuertes perturbaciones en la matriz RG que ocasionalmente pueden resultar de EUE (C). Los aumentos corresponden a los marcos punteados rojos en la figura principal: barras de escala, 10 μm. Haga clic aquí para ver una versión más grande de esta figura.
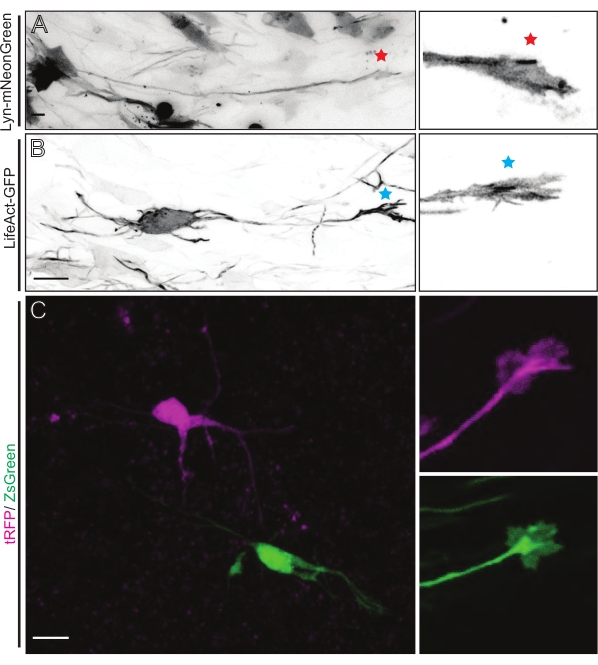
Figura 5: Visualización in situ de la dinámica del cono de crecimiento en rodajas organotípicas agudas. (A,B) Neuronas y sus correspondientes conos de crecimiento marcados con Lyn-mNeonGreen y LifeAct-GFP, respectivamente. Estrella roja que marca el cono de crecimiento de la neurona que expresa Lyn-mNeonGreen. Asterisco azul que marca el cono de crecimiento de la neurona que expresa LifeAct-GFP. (C) Neuronas vecinas marcadas con el sistema de plásmidos duales que contiene tRFP (magenta) y ZsGreen (verde) y sus correspondientes conos de crecimiento. Los conos de crecimiento fotografiados (derecha) estaban fuera del marco capturado (izquierda), obtenidos poco después de adquirir el lapso de tiempo del cono de crecimiento; barras de escala, 5 μm. Haga clic aquí para ver una versión más grande de esta figura.
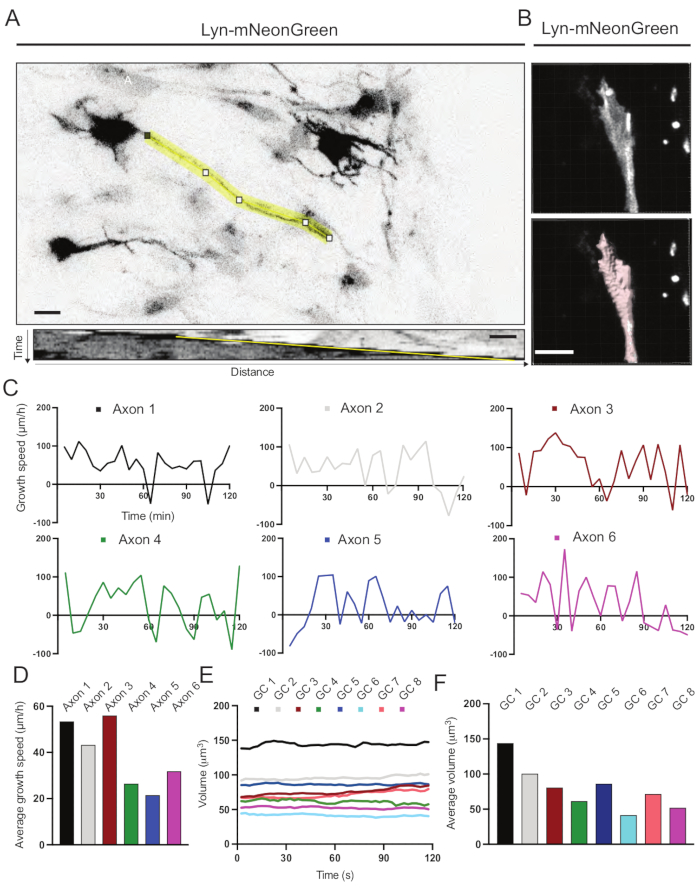
Figura 6: Análisis de la velocidad de crecimiento del axón y el volumen del cono de crecimiento. (A) Trazado de axones en una neurona que expresa Lyn-mNeonGreen (arriba) y su correspondiente kymograph (abajo) generado utilizando ImageJ. (B) Reconstrucción del video z-stack del cono de crecimiento utilizando el software de análisis de imágenes (arriba) y el mismo cono de crecimiento resaltado utilizando la herramienta de medición de superficies (abajo). (C) Gráficos que muestran los cambios en la velocidad de crecimiento a lo largo del tiempo para varios axones. (D) La velocidad media de crecimiento de los axones se cuantifica en (C). (E) Gráfico que muestra los cambios en el volumen del cono de crecimiento a lo largo del tiempo. F) El volumen medio de los conos de crecimiento se cuantifica en (E); barra de escala, 5 μm. Haga clic aquí para ver una versión más grande de esta figura.
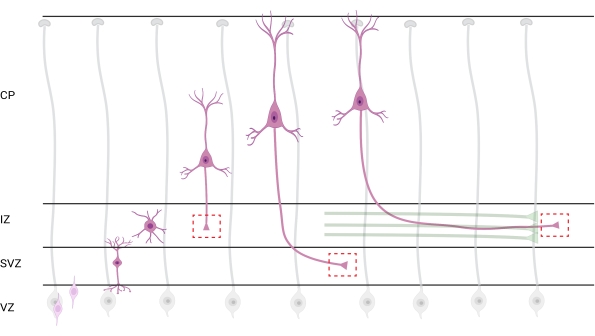
Figura 7: Migración radial y polarización neuronal de neuronas corticales piramidales. Diagrama que ilustra el desarrollo de neuronas corticales piramidales (rosa) que migran radialmente desde la zona ventricular germinal (VZ) hacia la superficie pia. Guiados por procesos de glía radial (gris), las neuronas polarizadas migratorias establecen un proceso líder, la dendrita futura, y el proceso de arrastre, el axón futuro, que continúan extendiéndose hacia abajo hacia la zona intermedia (IZ). Las cajas rojas discontinuas representan las áreas corticales donde se tomaron imágenes de los conos de crecimiento. Específicamente en la IZ, zona subventricular (SVZ), o unión de haces de axones (verde). La ilustración fue creada con una herramienta basada en la web, BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.
| Plásmido | Concentración (μg/μL) | Uso previsto |
| pCAG-lox-STOP-lox-Lyn-mNeonGreen | 0.25 | Etiquetado de la proteína dirigida a la membrana (Lyn) |
| + | + | |
| p-Tub-alfa-1-iCre | 0.08 | |
| p-Tub-alfa-1-LifeAct-GFP | 0.125 | Etiquetado de actina filamentosa (F-actina) en conos de crecimiento |
| pCAG-lox-rox-STOP-rox-tRFP-lox-Lyn-ZsGreen | 1 | Etiquetado independiente de dos poblaciones de neuronas vecinas |
| + | + | |
| p-Tub-alfa-1-iCre | 0.004 | |
| + | + | |
| p-Tub-alfa-1-Dre | 0.2 |
Tabla 1: Lista de plásmidos utilizados en el Protocolo. Nombre, concentración y uso previsto de cada plásmido utilizado.
Video complementario 1: Visualización in situ de la dinámica del cono de crecimiento en rodajas organotípicas agudas. Dinámica de los conos de crecimiento etiquetados con Lyn-mNeonGreen (arriba a la izquierda) y LifeAct-GFP (abajo a la izquierda). Los conos de crecimiento vecinos se etiquetan diferencialmente con el sistema de plásmidos duales que contiene tRFP (magenta; arriba a la derecha) y ZsGreen (verde; abajo a la derecha). Intervalo de imagen, 2,5 s. Barras de escala, 5 μm. Haga clic aquí para descargar este archivo.
Discusión
La forma en que los conos de crecimiento perciben y reaccionan a su entorno circundante para coordinar la extensión y la guía simultáneas del axón sigue siendo un tema de debate 3,18. Los estudios pioneros en sustratos 2D proporcionaron una visión de los mecanismos moleculares fundamentales que generan las fuerzas que impulsan la dinámica del cono de crecimiento durante la formación de axones, el crecimiento y la navegación 2,10,11,12,19. Más recientemente, estudios en matrices 3D revelaron cuánta influencia tiene una tercera dimensión en el comportamiento del cono de crecimiento y, en consecuencia, en el crecimiento del axón 8,9. Sin embargo, los intrincados mecanismos que instruyen la dinámica del cono de crecimiento in vivo aún no se han examinado a fondo.
La preparación de cultivos organotípicos de corte de cerebros IUE o EUE es ampliamente utilizada y bien documentada. Se ha convertido en un estándar de oro que permite a los científicos obtener información sobre el desarrollo y el comportamiento de las neuronas en el tejido cerebral vivo20,21. De hecho, esta técnica se ha utilizado con éxito en combinación con varias técnicas de imagen de alta resolución para visualizar procesos moleculares específicos y eventos morfológicos in situ. Dichos estudios incluyen, pero no se limitan a la formación y extensión de axones19,22, la migración neuronal cortical 19,22,23,24, la dinámica del centrosoma 25,26, la dinámica de microtúbulos27, así como la dinámica funcional de los compartimentos pre y postsinápticos28,29.
Este protocolo aborda una brecha en la neurobiología experimental, visualizando la dinámica del cono de crecimiento del desarrollo de neuronas corticales in situ, en cultivos de cortes cerebrales agudos ex vivo y las herramientas para analizar los datos obtenidos.
Se utilizaron cultivos agudos de corte cerebral para establecer este protocolo porque (1) con algo de práctica, son fáciles de generar; (2) presentar un sistema accesible para estudiar los conos de crecimiento incrustados en un entorno cuasi-totalmente fisiológico, pero lo suficientemente transparente como para permitir imágenes de células vivas de alta resolución; (3) se puede ampliar para su uso con una miríada de líneas de ratones transgénicos; (4) combinado con IUE o EUE, proporcionan un potencial prácticamente ilimitado para entregar herramientas moleculares para evaluar el rendimiento de los conos de crecimiento y los axones in vivo bajo regímenes de pérdida/ganancia de función, junto con reporteros fluorescentes y sondas de citoesqueleto.
Esta metodología se describió en el contexto tanto de la EUE como de la IUE. Aunque sigue siendo un método altamente confiable, eue resultó en una mayor incidencia de cortes cerebrales que muestran una red RG desorganizada en comparación con las obtenidas con IUE como método de administración (Figura 4C). Las alteraciones en la matriz RG afectan fuertemente la migración neuronal y el patrón de elongación del axón30,31. Estos son parámetros clave que predicen dónde encontrar axones para el análisis en un momento dado y el tipo de entorno en el que están navegando. Las rebanadas cerebrales con una red RG significativamente interrumpida generalmente tienen una estratificación de neuronas corticales deteriorada. Esto, a su vez, produce axones con trayectorias caóticas. Por lo tanto, se recomienda encarecidamente controlar la integridad estructural de la red RG. Curiosamente, la mala integridad estructural se correlaciona con el aumento de la edad del cerebro embrionario. De hecho, tales efectos en embriones E12.5-E13.5 más jóvenes generalmente no se observaron19.
El presente protocolo es exhaustivo y sencillo. Sin embargo, hay algunos pasos críticos en los que se debe tener especial cuidado y atención para obtener resultados óptimos. Estos se han señalado expresamente en el protocolo e incluyen (1) ajustar la cantidad de ADN utilizado en la electroporación para obtener un etiquetado escaso; (2) evitar daños durante la extracción de cerebros; (3) controlar la temperatura de la agarosa durante la envoltura cerebral; (4) solución de problemas del porcentaje ideal de agarosa para cerebros de una edad determinada; y (5) selección de fluoróforos, cuya experiencia sigue. Durante la optimización del protocolo, se probó el rendimiento de varios fluoróforos en imágenes in situ de células vivas. Para este protocolo se eligieron variantes monoméricas GFP EGFP y NeonGreen para la preparación de los plásmidos marcados con LifeAct y Lyn (Figura 5A, B). Además, la variante RFP mScarlet se probó y se encontró muy adecuada para esta configuración (datos no mostrados). También se probaron tRFP multimérica (dímero) y ZsGreen (tetrámero) (Figura 5C y Video complementario 1, derecha). Estos fluoróforos súper brillantes de plegamiento rápido se recomiendan cuando el método requiere una rápida generación de señal fluorescente después de la entrega de ADN.
Una práctica común en el uso de cultivos de rebanadas es utilizar rebanadas de diferentes cerebros para probar el control y las condiciones experimentales. Esto representa una fuente inherente de variabilidad no deseada. Aquí, se utilizó un sistema de expresión que permite la modificación independiente de las neuronas vecinas y la expresión de los reporteros para la identificación. Nótese que en esta demostración (Figura 5C), no hubo diferencias entre las neuronas que expresan ninguno de los fluoróforos. Sin embargo, como ejemplo, tal mezcla de plásmidos combinada con una línea de ratón transgénico que alberga un gen sensible a Cre etiquetará con neuronas tRFP (sensibles a Dre) que permanecieron como de tipo salvaje. En contraste, el ZsGreen (también sensible a Cre) etiquetará las neuronas recombinadas. Por lo tanto, los conos de crecimiento de los dos genotipos diferentes, y probablemente también los fenotipos, podrían estudiarse uno al lado del otro simultáneamente en la misma rebanada cerebral.
La localización de axones y conos de crecimiento para el análisis es una consideración importante. Las neuronas corticales se polarizan mientras migran radialmente desde la zona ventricular (VZ) hacia la placa cortical (CP). Durante este proceso, las neuronas forman un proceso líder (una dendrita futura) y un proceso de arrastre que se convertirá en el axón, uniéndose eventualmente a los axones pioneros en la zona intermedia (IZ), estableciendo tractos axónicos32. Por lo tanto, para capturar los conos de crecimiento axonal, se realizaron imágenes sobre fibras axonales en la IZ, incluidos los axones que salen de la CP y los axones generados tempranamente ya asociados con haces axonales; o eventualmente, en fibras que atraviesan la IZ y se extienden por debajo de ella (Figura 7).
Este protocolo hace que sea factible realizar imágenes de superresolución de neuronas dentro de cortes organotípicos. Históricamente, la dispersión de la luz era un problema importante que se enfrentaba al obtener imágenes de especímenes gruesos. En las últimas dos décadas, los amplios avances en tecnologías ópticas hicieron posible la obtención de imágenes de especímenes gruesos. Aquí, se utilizó un objetivo de larga distancia de trabajo para visualizar mejor las estructuras más pequeñas, como los conos de crecimiento. Inevitablemente, este protocolo no captura eventos más detallados como el flujo retrógrado de actina o la dinámica de microtúbulos. El objetivo de larga distancia de trabajo, que requiere una apertura numérica (NA) más baja, conserva la información de cortes gruesos. Sin embargo, también fue posible adaptar este protocolo para su uso con objetivos de menor distancia de trabajo. Esto requirió una transferencia suave de rebanadas a un plato con fondo de vidrio para preservar la integridad estructural. Sin embargo, el uso de este método resultó en una supervivencia más corta- ~ 15 h- debido a la pérdida de intercambio de gases (datos no mostrados). A diferencia de los cultivos 2D, los conos de crecimiento en 3D ocupan un volumen mayor y requieren compensación de movimiento-artefacto en el eje z. Para aumentar la capacidad de obtener imágenes de eventos detallados, se debe utilizar la tecnología confocal moderna. Por lo tanto, se recomienda utilizar un motor z-stack de escaneo rápido, como el z-Galvo disponible en microscopios confocales altamente sensibles33.
Cabe destacar que este protocolo presenta tres limitaciones principales. En primer lugar, a menudo es difícil controlar los niveles de expresión / número de células que expresan de cualquier plásmido dado in vivo. Esto introduce variabilidad entre todas las rodajas incluso cuando se mantiene la misma concentración de plásmidos. Por lo tanto, la selección de los elementos reguladores en los vectores de expresión utilizados debe estar predeterminada con cuidado. En segundo lugar, la obtención de imágenes de eventos detallados utilizando insertos de membrana actualmente no es factible. Esta segunda limitación podrá superarse con las actualizaciones metodológicas propuestas en el párrafo anterior. Por último, los conos de crecimiento son altamente fotosensibles y pueden ser rápidamente fotoblanqueados. Por lo tanto, las imágenes frecuentes de los conos de crecimiento, durante tan solo 5 minutos utilizando microscopios de escaneo láser, a menudo pueden colapsar los conos de crecimiento. En este sentido, los nuevos avances en los dispositivos generados por microscopía de láminas de luz se pueden adaptar para la obtención de imágenes a largo plazo de las rebanadas cerebrales34.
Se prevén protocolos similares para abrir nuevas vías de investigación, permitiendo una mejor comprensión de lo que se necesita para que un cono de crecimiento lea y reaccione hacia un entorno in vivo complejo y, lo que es más importante, para desentrañar la mecánica de esta sofisticada interacción.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Nos gustaría agradecer a Maria Eugenia Bernis por fotografiar los procedimientos. También agradecemos a Emily Burnside, Emily Handley, Thorben Pietralla, Max Schelski y Sina Stern por leer y discutir el manuscrito. Estamos agradecidos a nuestros destacados asistentes técnicos, Jessica Gonyer, Blanca Randel y Anh-Tuan Pham. Reconocemos el valioso apoyo de la instalación de microscopio de luz y la instalación de animales de DZNE. Este trabajo fue apoyado por Deutsche Forschungsgesellschaft (DFG), la Fundación Internacional para la Investigación en Paraplejia (IRP) y Wings for Life (a F.B). F.B. es miembro del clúster de excelencia ImmunoSensation2, los SFB 1089 y 1158, y ha recibido el Premio Roger De Spoelberch.
Materiales
| Name | Company | Catalog Number | Comments |
| Adson Forceps | Fine Science Tools | 11006-12 | |
| Alexa Fluor 488 | Invitrogen | A21202 | Goat Anti-Mouse |
| Alexa Fluor 647 | Invitrogen | A21236 | Goat Anti-Mouse |
| Anti-Vimentin antibody | sigma-Aldrich | V2258-.2ML | Monoclonal mouse, clone LN-6, ascites fluid |
| B27 supplement | ThermoFisher Scientific | 17504044 | |
| Betadine | B. Braun | 3864154 | |
| Biozym Sieve GP Agarose | Biozyme | 850080 | |
| Braunol, Sprühflasche | B. Braun | 3864073 | |
| Buprenorphine (Temgesic) | GEHE Pharma | 345928 | |
| DAPI | sigma-Aldrich | D9542 | |
| DMZ unevirsal electrode puller | Zeitz | NA | |
| Electric razor | Andes | NA | ProClip UltraEdge Super 2-Speed model |
| Enrofloxacin (Baytril) | Bayer | 3543238 | 2,5% (wt/vol) |
| Eppendorf microloader pipette tips | FischerScientific | 10289651 | |
| Fast Green FCF | Sigma-Aldrich | F7252-5G | Dye content ≥ 85 % |
| Fetal Bovine Serum | ThermoFisher Scientific | 10500064 | |
| Fiji 2.1.0 | NIH | NA | https://imagej.net/software/fiji/downloads |
| Fine Scissors | Fine Science Tools | 14058-09 | ToughCut/Straight/9cm |
| FluoroDish Cell Culture Dish | World Precision Instruments | FD5040-100 | |
| Fluoromount Aqueous Mounting Medium | sigma-Aldrich | F4680-25ML | |
| Glucose | MedPex | 3705391 | 5% |
| GlutaMAX Supplement | ThermoFisher Scientific | 35050061 | |
| Glycine | Sigma-Aldrich | G8898 | |
| HBSS | Life Technologies | 14025092 | calcium, magnesium, no phenol red |
| Horse serum | Pan-Biotech | P30-0711 | |
| Imaris 9.7.2 | Bitplane | NA | https://imaris.oxinst.com/products/imaris-for-neuroscientists |
| Isoflurane | Virbac | NA | |
| Isotonic saline solution | B. Braun | 8609261 | 0.90% |
| Leica VT1200 S vibratome | Leica | 14048142066 | |
| LSM 880 with Airyscan | Zeiss | NA | |
| Metacam | Venusberg Apotheke | 8890217 | 5 mg/ml |
| Mice | Janvier Labs | NA | C57BL/6JRj |
| Micro-Adson Forceps | Fine Science Tools | 11018-12 | |
| Micropipette Storage Jar | World Precision Instruments | E210 | 16.16.27 |
| Microsoft Excel | Microsoft | NA | https://www.microsoft.com/en-us/microsoft-365/p/excel/cfq7ttc0k7dx?activetab=pivot:overviewtab |
| Millicell Cell Culture Insert | EMD Millipore | PICM0RG50 | 30 mm, hydrophilic PTFE, 0.4 µm |
| Moria Perforated Spoons | Fine Science Tools | 10370-18 | |
| Moria Spoon | Fine Science Tools | 10321-08 | |
| Neurobasal Medium, minus phenol red | ThermoFisher Scientific | 12348017 | |
| Neuropan-2 supplement | Pan-Biotech | P07-11010 | |
| Normal goat serum | Abcam | ab138478 | |
| Olsen-Hegar Needle Holder with Scissors | Fine Science Tools | 12002-12 | |
| p-Tub-alpha-1-Dre | Addgene | 133925 | |
| p-Tub-alpha-1-iCre | Addgene | 133924 | |
| p-Tub-alpha-1-LifeAct-GFP | Addgene | 175437 | |
| Parafilm | VWR | 52858-000 | |
| Paraformaldehyde | sigma-Aldrich | P6148 | |
| PBS | Sigma-Aldrich | P3813-10PAK | |
| pCAG-lox-rox-STOP-rox-tRFP-lox-Lyn-ZsGreen | Addgene | 175438 | |
| pCAG-lox-STOP-lox-Lyn-mNeonGreen | Addgene | 175257 | |
| Penicillin-Streptomycin | ThermoFisher Scientific | 15140122 | |
| PicoNozzle Kit v2 | World Precision Instruments | 5430-ALL | |
| Platinum Tweezertrodes | Harvard Apparatus | 45-0487 | 1 mm / 3 mm |
| QIAGEN Maxi kit | QIAGEN | 12162 | |
| Reflex wound closure Clip | World Precision Instruments | 500344-10 | 7 mm |
| Sekundenkleber Pattex Mini Trio | Lyreco | 4722659 | |
| Square wave electroporation system ECM830 | Harvard Apparatus | W3 45-0052 | |
| Sterile gauze | Braun Askina | 9031216 | |
| Sterile lubricant eye ointment | Bayer Vital | PZN1578675 | |
| Sterile surgical gloves | Sempermed | 14C0451 | |
| Sucrose | Roth | 4621.2 | |
| Supramid 5-0 surgical silk sutures | B. Braun | NA | |
| Thin-wall glass capillaries | World Precision Instruments | TW100-4 | |
| Triton X-100 | Sigma-Aldrich | X100 | |
| Vannas spring scissors | Fine Science Tools | 15000-03 | |
| µ-Slide 8 Well Glass Bottom | Ibidi | 80827 |
Referencias
- Schelski, M., Bradke, F. Neuronal polarization: From spatiotemporal signaling to cytoskeletal dynamics. Molecular and Cellular Neurosciences. 84, 11-28 (2017).
- Lowery, L. A., Van Vactor, D. The trip of the tip: understanding the growth cone machinery. Nature Reviews: Molecular Cell Biology. 10 (5), 332-343 (2009).
- Stoeckli, E. T. Understanding axon guidance: are we nearly there yet. Development. 145 (10), (2018).
- Bradke, F., Dotti, C. G. The role of local actin instability in axon formation. Science. 283 (5409), 1931-1934 (1999).
- Neukirchen, D., Bradke, F. Cytoplasmic linker proteins regulate neuronal polarization through microtubule and growth cone dynamics. Journal of Neuroscience. 31 (4), 1528-1538 (2011).
- Witte, H., Bradke, F. The role of the cytoskeleton during neuronal polarization. Current Opinion in Neurobiology. 18 (5), 479-487 (2008).
- Witte, H., Neukirchen, D., Bradke, F. Microtubule stabilization specifies initial neuronal polarization. Journal of Cell Biology. 180 (3), 619-632 (2008).
- Nichol, R. H., Catlett, T. S., Onesto, M. M., Hollender, D., Gomez, T. M. Environmental elasticity regulates cell-type specific RHOA signaling and neuritogenesis of human neurons. Stem Cell Reports. 13 (6), 1006-1021 (2019).
- Santos, T. E., et al. Axon growth of CNS neurons in three dimensions is amoeboid and independent of adhesions. Cell Reports. 32 (3), 107907 (2020).
- Mitchison, T., Kirschner, M. Cytoskeletal dynamics and nerve growth. Neuron. 1 (9), 761-772 (1988).
- Lin, C. H., Thompson, C. A., Forscher, P. Cytoskeletal reorganization underlying growth cone motility. Current Opinion in Neurobiology. 4 (5), 640-647 (1994).
- Myers, J. P., Gomez, T. M. Focal adhesion kinase promotes integrin adhesion dynamics necessary for chemotropic turning of nerve growth cones. Journal of Neuroscience. 31 (38), 13585-13595 (2011).
- Turney, S. G., et al. Nerve growth factor stimulates axon outgrowth through negative regulation of growth cone actomyosin restraint of microtubule advance. Molecular Biology of the Cell. 27 (3), 500-517 (2016).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Noctor, S. C., Flint, A. C., Weissman, T. A., Dammerman, R. S., Kriegstein, A. R. Neurons derived from radial glial cells establish radial units in neocortex. Nature. 409 (6821), 714-720 (2001).
- Noctor, S. C., Martinez-Cerdeno, V., Ivic, L., Kriegstein, A. R. Cortical neurons arise in symmetric and asymmetric division zones and migrate through specific phases. Nature Neuroscience. 7 (2), 136-144 (2004).
- Ferent, J., Zaidi, D., Francis, F. Extracellular control of radial glia proliferation and scaffolding during cortical development and pathology. Frontiers in Cell and Developmental Biology. 8, 578341 (2020).
- Dent, E. W., Gupton, S. L., Gertler, F. B. The growth cone cytoskeleton in axon outgrowth and guidance. Cold Spring Harbor Perspectives in Biology. 3 (3), (2011).
- Dupraz, S., et al. RhoA controls axon extension independent of specification in the developing brain. Current Biology. 29 (22), 3874-3886 (2019).
- Azzarelli, R., Oleari, R., Lettieri, A., Andre, V., Cariboni, A. In vitro, ex vivo and in vivo techniques to study neuronal migration in the developing cerebral cortex. Brain Sciences. 7 (5), (2017).
- Humpel, C. Organotypic brain slice cultures: A review. Neuroscience. 305, 86-98 (2015).
- Namba, T., et al. Pioneering axons regulate neuronal polarization in the developing cerebral cortex. Neuron. 81 (4), 814-829 (2014).
- Shah, B., et al. Rap1 GTPases are master regulators of neural cell polarity in the developing neocortex. Cerebral Cortex. 27 (2), 1253-1269 (2017).
- Wiegreffe, C., Feldmann, S., Gaessler, S., Britsch, S. Time-lapse confocal imaging of migrating neurons in organotypic slice culture of embryonic mouse brain using in utero electroporation. Journal of Visualized Experiments: JoVE. (125), (2017).
- de Anda, F. C., Meletis, K., Ge, X., Rei, D., Tsai, L. H. Centrosome motility is essential for initial axon formation in the neocortex. Journal of Neuroscience. 30 (31), 10391-10406 (2010).
- Sakakibara, A., et al. Dynamics of centrosome translocation and microtubule organization in neocortical neurons during distinct modes of polarization. Cerebral Cortex. 24 (5), 1301-1310 (2014).
- Schatzle, P., Kapitein, L. C., Hoogenraad, C. C. Live imaging of microtubule dynamics in organotypic hippocampal slice cultures. Methods in Cell Biology. 131, 107-126 (2016).
- Qu, X., Kumar, A., Bartolini, F. Live imaging of microtubule dynamics at excitatory presynaptic boutons in primary hippocampal neurons and acute hippocampal slices. STAR Protocols. 2 (1), 100342 (2021).
- Tonnesen, J., Katona, G., Rozsa, B., Nagerl, U. V. Spine neck plasticity regulates compartmentalization of synapses. Nature Neuroscience. 17 (5), 678-685 (2014).
- Buchsbaum, I. Y., Cappello, S. Neuronal migration in the CNS during development and disease: insights from in vivo and in vitro models. Development. 146 (1), (2019).
- Rigby, M. J., Gomez, T. M., Puglielli, L. Glial cell-axonal growth cone interactions in neurodevelopment and regeneration. Frontiers in Neuroscience. 14, 203 (2020).
- Barnes, A. P., Polleux, F. Establishment of axon-dendrite polarity in developing neurons. Annual Review of Neuroscience. 32, 347-381 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados