Method Article
In situ Visualisation de la croissance des axones et de la dynamique des cônes de croissance dans des cultures de tranches cérébrales embryonnaires ex vivo aiguës
Dans cet article
Résumé
Ce protocole démontre une méthode simple et robuste pour étudier la croissance in situ des axones et la dynamique des cônes de croissance. Il décrit comment préparer ex vivo des tranches de cerveau aiguës physiologiquement pertinentes et fournit un pipeline d’analyse convivial.
Résumé
Au cours du développement neuronal, les axones naviguent dans l’environnement cortical pour atteindre leur destination finale et établir des connexions synaptiques. Les cônes de croissance - les structures sensorielles situées aux extrémités distales des axones en développement - exécutent ce processus. L’étude de la structure et de la dynamique du cône de croissance est cruciale pour comprendre le développement axonal et les interactions avec le système nerveux central (SNC) environnant qui lui permettent de former des circuits neuronaux. Ceci est essentiel lors de la conception de méthodes pour réintégrer les axones dans les circuits neuronaux à la suite d’une blessure dans la recherche fondamentale et les contextes précliniques. Jusqu’à présent, la compréhension générale de la dynamique des cônes de croissance est principalement fondée sur des études de neurones cultivés en deux dimensions (2D). Bien qu’elles soient sans aucun doute fondamentales pour les connaissances actuelles sur la dynamique structurelle des cônes de croissance et la réponse aux stimuli, les études 2D déforment l’environnement physiologique tridimensionnel (3D) rencontré par les cônes de croissance neuronale dans les tissus intacts du SNC. Plus récemment, des gels de collagène ont été utilisés pour surmonter certaines de ces limitations, permettant l’étude du développement neuronal en 3D. Cependant, les environnements synthétiques 2D et 3D manquent d’indices de signalisation dans le tissu du SNC, qui dirigent l’extension et la recherche d’axones en développement. Ce protocole fournit une méthode pour étudier les axones et les cônes de croissance à l’aide de tranches de cerveau organotypiques, où les axones en développement rencontrent des indices physiques et chimiques physiologiquement pertinents. En combinant l’électroporation in utero et ex utero affinée pour fournir des rapporteurs fluorescents à faible résolution avec la microscopie à super-résolution, ce protocole présente un pipeline méthodologique pour la visualisation de la dynamique des axones et des cônes de croissance in situ. En outre, une description détaillée de l’analyse des données d’imagerie à long terme et des cellules vivantes est incluse.
Introduction
Les neurones sont des cellules hautement polarisées qui représentent l’unité de calcul de base du système nerveux. Ils reçoivent et émettent des informations qui reposent sur le cloisonnement des sites d’entrée et de sortie : dendrites et axones, respectivement1. Pendant le développement, les axones s’étendent tout en naviguant dans un environnement incroyablement complexe pour atteindre leur destination. La navigation axonale est guidée par le cône de croissance, une structure sensorielle située à l’extrémité de l’axone en développement. Le cône de croissance est responsable de la détection des signaux environnementaux et de leur traduction en réorganisation spatiale dynamique de son cytosquelette 2,3. Les réactions morpho-mécaniques qui en résultent ordonnent au cône de croissance de s’étendre ou de se rétracter du signal de déclenchement, conduisant à des manœuvres axonales spécifiques.
La compréhension actuelle de l’extension des axones et de la dynamique des cônes de croissance découle d’études évaluant la croissance des axones sur des substrats bidimensionnels (2D) 2,4,5,6,7. Ces études pionnières ont identifié une interaction sophistiquée entre les cônes de croissance et les substrats de croissance et ont révélé des différences frappantes en fonction des caractéristiques du substrat telles que l’adhésivité et la rigidité 8,9. Grâce à ces connaissances, des indices environnementaux extracellulaires ont été supposés dicter la croissance des axones, le cytosquelette du cône de croissance exécutant cette croissance 2,10,11,12. Notamment, les neurones peuvent étendre les axones dans des substrats non adhésifs (p. ex. poly-lysine, poly-ornithine)13. De plus, la rigidité du substrat peut influencer le taux de croissance des axones indépendamment des complexesadhésifs cellulaires 8. Par conséquent, l’étude de la dynamique des cônes de croissance dans les substrats 2D ne peut pas à elle seule modéliser avec précision l’équilibre des forces résultant de l’interaction des cônes de croissance axonaux avec des environnements tridimensionnels (3D) physiologiquement pertinents, tels que ceux trouvés in vivo.
Pour surmonter les limites des essais 2D, la croissance des axones et la dynamique des cônes de croissance ont été étudiées dans des matrices 3D 8,9. Ces matrices posent un contexte plus physiologique mais permettent d’étudier les mécanismes cellulaires intrinsèques de la croissance axonale. Il permet l’examen du cône de croissance de manière unicellulaire dans une variété de conditions et de traitements pharmacologiques9. Dans de tels environnements 3D, les axones présentaient une dynamique cytosquelettique distincte et se développaient plus rapidement que ceux observés dans les neurones cultivés en 2D9. Ces études élégantes ont démontré l’influence d’une dimension supplémentaire sur la réorganisation du cytosquelette du cône de croissance et, par conséquent, sur son comportement.
Malgré les avantages apparents présentés par les matrices 3D par rapport aux surfaces 2D pour soutenir le développement neuronal natif et la croissance des axones, elles restent un échafaudage synthétique simplifié qui ne peut pas refléter la complexité de la dynamique observée dans les tissus du système nerveux central (SNC). Ici, l’administration de plasmides rapporteurs par électroporation ex utero et in utero a été combinée à une culture de tranches organotypiques cérébrales et à une imagerie in situ à super-résolution en direct pour analyser la dynamique des cônes de croissance dans un contexte physiologique. Cette méthodologie permet de visualiser les axones en développement tout en expérimentant la 3-dimensionnalité des environnements in vivo et la complexité de sa composition physico-chimique. Enfin, des procédures conviviales pour mesurer la croissance des axones et la dynamique des cônes de croissance à l’aide de logiciels sous licence courante et accessibles au public sont décrites.
Protocole
L’expérimentation animale doit être conforme aux réglementations institutionnelles et fédérales pertinentes. Les femelles femelles gravides C57BL/6JRj du jour embryonnaire 15,5 et 12,5 (E15.5 et E12.5) ont été utilisées dans ce protocole. Les expériences ont été réalisées conformément à la loi sur le bien-être animal de l’Agence nationale de l’environnement de Rhénanie-du-Nord-Westphalie (Landesamt für Natur, Umwelt und Verbraucherschutz (LANUV)).
1. Préparation des plasmides pour injection
- Isolez l’ADN à l’aide du kit Maxiprep sans endotoxine selon le protocole du fabricant (voir tableau des matériaux).
- Mélanger l’ADN sélectionné à la concentration souhaitée (tableau 1) et la solution Fast Green à 10 % (voir tableau des matériaux) pour visualiser l’administration du mélange d’ADN dans les ventricules cérébraux.
REMARQUE : Des plasmides spécifiques ont été utilisés pour le marquage clairsemé des neurones corticaux (Figure 1A), les structures filamenteuses de l’actine (F-actine) dans le cône de croissance (Figure 1B) et le double marquage des cônes de croissance dans le même cortex (Figure 1C). Tous les plasmides (tableau 1) utilisés dans ce protocole ont été déposés dans Addgene (voir tableau des matériaux). - Préparez les capillaires en verre à l’aide d’un ensemble d’électrodes capillaires selon le programme suivant: pression: 500, chaleur: 800, traction: 30 et vitesse: 40.
- Chargez 15 μL de mélange ADN/Fast Green dans chaque capillaire en verre à l’aide d’embouts de pipette de micro-chargeur.
REMARQUE: Assurez-vous qu’aucune bulle ne se forme. - Stockez les capillaires remplis d’ADN dans un plat de 10 cm avec un morceau de pâte à modeler sur le diamètre du plat. Les capillaires peuvent être chargés et stockés à 4 °C la veille de l’expérience. Scellez l’extrémité arrière du capillaire avec un film flexible pour éviter le séchage.
2. Préparation des solutions
- Préparez la solution de sel tamponnée de Hank, complétée par du glucose (HBSS-G).
- Ajouter 0,5% de 20% de glucose dans une bouteille de 1x HBSS. Bien mélanger et conserver à 4 °C jusqu’à 2 semaines. Pour l’extraction de l’embryon, la solution HBSS-G à bulles avec du carbogène (95% O2 et 5% CO2) en utilisant de la pierre bouillonnante peu de temps avant la collecte de l’embryon.
- Solution de média de tranche
- Préparer des tranches fraîches contenant du sérum neurobasal 1x, 5 % de sérum de cheval, 5 % de sérum de veau fœtal, supplément de B27 1:50, supplément de L-glutamine 1:400, pénicilline-streptomycine 1:200 et supplément de Neuropan-2 1:100 (à pH = 7,3), dans des conditions stériles (voir tableau des matériaux).
- Préparez des plats de 3 cm avec 1 mL de tranches chacune. Placer dans l’incubateur à 35 °C avec 5% de CO2 pendant au moins 1 h avant l’expérience pour équilibrer le pH du milieu par échange gazeux.
REMARQUE: L’équilibrage du pH des milieux est causé par l’acidification des milieux par le CO2 de l’incubateur. Les tranches peuvent être conservées à 4 °C jusqu’à 1 semaine.
- Solution d’agarose à faible point de fusion (3%)
- Peser la quantité souhaitée de poudre d’agarose à faible point de fusion et dissoudre dans un volume approprié de 1x HBSS-G dans une bouteille en verre. Environ 7 mL de solution d’agarose par cerveau sont nécessaires.
- Placez la bouteille dans un micro-ondes pendant 2-3 min, avec le bouchon lâchement placé, et secouez toutes les 10-20 s.
- Une fois la poudre complètement dissoute, placez la bouteille dans un bain-marie ou un bain de perles réglé à 37 °C au moins 1 h avant l’expérience pour permettre à l’agarose de refroidir.
REMARQUE: Il est recommandé de chauffer l’agarose deux fois pendant 15 minutes pour s’assurer que la poudre d’agarose est dissoute. Ceci est crucial pour la bonne adhésion de l’agarose au tissu cérébral. Un thermomètre doit être utilisé pour mesurer la température de la solution d’agarose tout en intégrant le cerveau, en s’assurant qu’elle se situe entre 37 et 40 ° C. Les cerveaux de différents animaux âgés ont une rigidité différente. Il est recommandé de tester une gamme de concentrations d’agarose pour trouver l’homogénéité entre les tissus et l’agarose. - Préparer une solution saline tamponnée au phosphate avec 0,3 % de triton X-100 (PBS-T).
- Préparer une solution saline tamponnée au phosphate avec de l’azoture de sodium à 0,2 % (PBS-NaN3).
REMARQUE: Les solutions décrites aux étapes 2.4 à 2.5 sont destinées à être utilisées dans l’étape ultérieure d’immunohistochimie.
3. Préparation du poste de chirurgie
- Nettoyez la station chirurgicale avec de l’éthanol à 70% à 96% et placez la sous-couche de fonctionnement sur la surface de la station.
- Stériliser les instruments chirurgicaux en rinçant avec de l’éthanol à 70% à 96%, suivi d’une stérilisation sèche dans un stérilisateur à billes chaudes.
- Nettoyez les électrodes de la pince à épiler en platine (voir tableau des matériaux) avec de l’éthanol à 70 % à 96 % avant de les connecter au générateur d’impulsions.
- Insérez un capillaire en verre rempli d’ADN / Fast Green dans le support capillaire. Immédiatement avant utilisation, casser délicatement l’extrémité capillaire à l’aide de ciseaux fins et tester le débit de la solution à l’intérieur d’un tube de microcentrifugation de 1,5 mL rempli d’eau saline ou d’eau préchauffée.
REMARQUE: La figure 2A, B montre la configuration de la station chirurgicale et les outils utilisés pour l’électroporation ex utero (EUE) et l’électroporation in utero (IUE). - Pour l’IUE, réchauffer la solution saline à 37 °C au bain-marie.
4. Extraction d’embryons
- Placez la souris enceinte dans la chambre inductrice d’anesthésie avec 5% d’isoflurane jusqu’à ce que la souris soit profondément anesthésiée. Confirmer l’anesthésie par l’absence de réflexe de retrait de la pédale.
- Transférer la souris dans une sous-couche d’opération et maintenir l’isoflurane à 1,5% -2% à travers un cône de nez.
- Appliquez une pommade sur les deux yeux pour éviter le dessèchement de la cornée.
- Rasez l’abdomen de la souris, puis utilisez de la gaze imbibée d’éthanol à 70% à 96% pour enlever les poils rasés. Nettoyez la zone avec de la bétadine.
- À l’aide de petits ciseaux chirurgicaux stériles, faites une incision cutanée de 2 cm le long de la ligne médiane abdominale, suivie d’une incision musculaire de 1,5 cm.
REMARQUE: La taille de l’incision dépend de la taille de l’embryon. En effet, les embryons plus gros nécessiteront une incision plus grande pour permettre leur extraction. - Coupez un trou au milieu de la gaze suffisamment large pour s’adapter à l’incision cutanée (~ 2 cm de diamètre), trempez-le avec une solution saline chaude et placez-le autour de l’ouverture abdominale.
- Retirez les deux cornes utérines à l’aide d’un coton-tige imbibé d’une solution saline chaude ou à l’aide d’une pince, en saisissant soigneusement les espaces entre les embryons pour les retirer. Placer les embryons sur de la gaze humide (Figure 2C).
REMARQUE: Même un petit dommage aux vaisseaux sanguins et aux capillaires autour des cornes utérines entraînera probablement des saignements abondants. Par conséquent, évitez la manipulation directe de ces zones vascularisées à tout moment. - Ouvrir le sac utérin et retirer chaque embryon (Figure 2D).
- Euthanasier chaque embryon immédiatement après l’extraction via une coupe diagonale descendante, assurant une transsection complète de la moelle épinière (Figure 2E).
- Placez les embryons dans un plat de 10 cm contenant hbSS-G sur de la glace.
REMARQUE: La décapitation de l’embryon est évitée pour éviter les fuites de mélange ADN/ Fast Green du cerveau et faciliter le positionnement facile de l’embryon dans le support (voir électroporation ex utero , étape 5). - Sacrifier la mère immédiatement après l’extraction des embryons en effectuant une luxation cervicale.
REMARQUE: Ici, la mère est euthanasiée sous anesthésie pour l’épargner de toute autre douleur ou souffrance post-procédure conformément au protocole approuvé par la loi sur le bien-être animal de l’Agence environnementale de Rhénanie-du-Nord-Westphalie (LANUV).
5. Électroporation ex utero (EUE)
- Prenez un embryon et placez-le dans le support.
REMARQUE: Une pointe de pipette coupée de 1 mL attachée à l’extrémité d’un grattoir à cellules est utilisée comme support d’embryon. Il est essentiel de garder les bras des embryons à l’extérieur de la pointe pendant la procédure pour éviter qu’ils ne glissent dans la pointe (Figure 2F-I). Le diamètre de la pointe de la pipette est facilement réglable pour accueillir des embryons de différentes tailles. Coupez une deuxième pointe à la longueur où le diamètre de la pointe correspond à la taille de l’embryon et utilisez-la comme insert adaptateur au support mentionné ci-dessus. - Insérez soigneusement l’ADN/ capillaire en verre rempli de fast green à travers le crâne de l’embryon dans le ventricule latéral et injectez 2 à 3 μL de mélange de plasmides d’ADN (Figure 1A, B; Tableau 1) dans chaque ventricule (figure 2F).
REMARQUE: Utilisez les sutures lambdoïdales et sagittales comme guide pour l’emplacement de l’injection d’ADN. Les sutures lambdoïdales et sagittales sont des articulations fibreuses reliant la plaque osseuse du crâne. Le premier joint l’os pariétal à l’os occipital, et le second joint les deux os pariétaux. - Maintenez la tête de l’embryon entre les électrodes de la pince à platine à l’angle approprié pour cibler la zone cérébrale souhaitée (angle de 60 ° dans ce cas), la cathode faisant face à la zone où le transfert d’ADN est prévu (Figure 2G-H).
- Appliquer cinq impulsions à 30 mV avec un intervalle de 1 s et une durée de 50 ms à l’aide d’un générateur d’impulsions à ondes carrées.
REMARQUE: Gardez à l’esprit que les cerveaux de l’EUE connaissent un champ électrique plus efficace que ceux de l’IUE. Par conséquent, à une concentration d’ADN donnée, l’EUE entraîne une plus grande efficacité du transfert d’ADN que l’IUE, et les concentrations d’ADN doivent être ajustées en conséquence. - Si une électroporation bilatérale est souhaitée, répétez les étapes 5.3-5.4 avec la cathode et l’anode reflétant la position précédente pour cibler le cortex controlatéral.
REMARQUE: Étant donné que les deux ventricules ont été injectés avec de l’ADN, les cortex des deux hémisphères ont été ciblés. - Placez l’embryon électroporé dans un plat de 6 cm contenant du HBSS-G glacé. Répétez les étapes 5.1 à 5.6 pour tous les embryons requis.
6. Électroporation in utero (IUE)
- Injecter à la souris enceinte un analgésique; 50 μL de buprénorphine (0,1 mg/kg) (voir tableau des matériaux) par voie sous-cutanée, 20 min avant l’intervention.
- Effectuez les étapes 4.1 à 4.8 de la section d’extraction d’embryons.
REMARQUE: Évitez de laisser les embryons exposés inutilement en les recouvrant de gaze stérile imbibée de solution saline chaude. - Du bout des doigts, faire pivoter doucement l’embryon à l’intérieur de l’utérus jusqu’à ce que les sutures lambdoïdales et sagittales soient localisées (Figure 2J). Insérez soigneusement l’ADN/capillaire en verre Fast Green à travers la paroi utérine et le crâne de l’embryon dans le ventricule latéral et injectez 2 à 3 μL de mélange de plasmides d’ADN (Figure 1A,C) dans l’un ou les deux ventricules comme vous le souhaitez (Figure 2K-L).
REMARQUE: Une pression excessive des doigts sur les cornes utérines pourrait entraîner un effondrement amniotique du sac. - Maintenez la tête de l’embryon entre les électrodes de la pince à platine à l’angle approprié pour cibler la zone cérébrale souhaitée (angle de 60 ° dans ce cas), la cathode faisant face à la zone où le transfert d’ADN est prévu. Évitez de presser l’utérus car cela pourrait provoquer l’effondrement du sac amniotique (Figure 2M).
- Appliquer cinq impulsions à 35 mV avec un intervalle de 600 ms et une durée de 50 ms à l’aide d’un générateur d’impulsions à ondes carrées.
- Si les deux ventricules latéraux ont été injectés, répétez les étapes 6.5-6.6 avec la cathode et l’anode reflétant la position précédente pour cibler le cortex controlatéral.
- Répétez les étapes 6.3 à 6.6 pour tous les embryons requis.
- Une fois que tous les embryons requis ont été électroporés, utilisez un coton-tige imbibé de solution saline pour replacer doucement les cornes utérines à l’intérieur de la cavité abdominale.
REMARQUE: L’ajout d’une solution saline dans la cavité péritonéale aidera les cornes utérines à se remettre en position. - Suture des incisions musculaires et cutanées à l’aide d’un matériau de suture 5-0. Utilisez des clips de suture pour fixer la plaie et désinfectez la plaie de suture en la pulvérisant avec de la bétadine (Figure 2N-P).
- Injecter à la souris 200 μL de glucose à 5% par voie sous-cutanée.
- Injecter un antibiotique à la souris; 50 μL d’enrofloxacine (5 mg/kg) par voie sous-cutanée (voir tableau des matériaux).
- Replacez la souris dans la cage de récupération et maintenez la chaleur à l’aide d’une lumière chauffante infrarouge lointaine ou d’un coussin chauffant pendant au moins 20 minutes après la procédure (Figure 2Q).
- Surveillez la souris quotidiennement et injectez du méloxicam après la procédure pour soulager la douleur en suivant les directives institutionnelles et fédérales.
- Extrayez les embryons 2 jours après la procédure (c.-à-d. E17.5) après l’étape 4.
7. Extraction cérébrale et incorporation dans l’agarose
REMARQUE: Il est recommandé d’effectuer les étapes suivantes sous un microscope à dissection pour une meilleure précision. Éviter les dommages au cerveau est essentiel pour le succès de la procédure.
- Installez les outils d’extraction dans un espace de travail stérile sous une hotte de dissection (Figure 3A).
- Séparez la tête d’un embryon du reste du corps à l’aide de ciseaux à dissection.
- Fixez la tête comme illustré à la figure 3C, puis retirez la peau et le crâne en coupant le long de la ligne médiane, en partant de la base de la tête vers le nez (figure 3D).
- Pelez la peau et le crâne latéralement, en faisant un espace assez grand (~ 1 cm) pour que le cerveau soit excisé.
- Pour retirer le cerveau, insérez la pointe fermée des ciseaux de dissection stériles, en partant de sous le bulbe olfactif se déplaçant vers le tronc cérébral (Figure 3E).
- Coupez le tronc cérébral et coupez tous les morceaux lâches de méninges autour du cerveau (Figure 3F).
REMARQUE: Les méninges lâches font souvent que les tranches restent attachées au bloc d’agarose après la coupe, ce qui entraîne le détachement des tissus de l’agarose lors de la collecte des tranches. - Répétez les étapes 7.1 à 7.6 pour tous les embryons et gardez le cerveau sur la glace (idéalement pas plus de 30 minutes) jusqu’à l’étape d’intégration.
REMARQUE: Les étapes suivantes 7.7.1-7.7.4 se réfèrent à l’extraction cérébrale des cerveaux E12.5.- Isolez le haut de la tête juste en dessous de l’œil, comme le montre la figure 3G.
- Coupez la peau et le crâne sur le dessus du tronc cérébral, en suivant la ligne pointillée comme le montre la figure 3H, sans retirer le tronc cérébral.
- Faites une incision peau-crâne de 2 mm à l’arrière de la tête, comme le montre la figure 3I (voir les dessins pour plus de clarté).
REMARQUE: Cette incision fournit des points de préhension initiaux pour décoller les couches de peau et de crâne. En règle générale, ils se détachent comme une seule couche. La taille typique de l’incision est de 2 mm, correspondant à la longueur du bord de coupe des ciseaux à micro-ressort utilisés. - Commencez à décoller les couches de la peau et du crâne en fixant un côté de l’incision et en tirant l’autre avec précaution. Avec le même soin, finaliser en décollant la base de la tête jusqu’à libérer le cerveau (Figure 3J).
REMARQUE: Cela doit être fait avec beaucoup de soin, en observant que le cerveau n’est pas tiré le long des couches tissulaires. Alternez les côtés pour enlever le tissu recouvrant le cerveau.
- Verser l’agarose tiède (à 37-40 °C) dans un plat de 3 cm.
- Ramassez le cerveau à l’aide d’une cuillère perforée et éliminez l’excès de liquide en tamponnant le fond de la cuillère contre du papier de soie sec. Placez le cerveau dans un plat d’agarose.
REMARQUE: Il est essentiel d’éliminer autant de liquide que possible autour du cerveau pour permettre une meilleure adhésion de l’agarose aux tissus. - Mettez le plat avec de l’agarose liquide sur de la glace. À l’aide d’une cuillère plus petite, mélanger l’agarose pendant 10 s pour un refroidissement uniforme. Manœuvrez le cerveau au milieu du plat. Placez le cerveau horizontalement dans le plat avec la face dorsale vers le haut, en vous assurant qu’il est complètement recouvert d’agarose de toutes les directions (Figure 3K).
REMARQUE: Les cerveaux coulent souvent au fond du plat une fois placés dans l’agarose; soulevez le cerveau à l’aide d’une petite cuillère jusqu’à ce qu’un espace de 1 à 2 mm sous le cerveau soit établi. - Répétez les étapes 7.8 à 7.10 pour tous les cerveaux.
- Une fois l’agarose polymérisée, ajoutez 500 μL de HBSS-G sur le bloc d’agarose pour éviter le dessèchement. Ensuite, couvrez le plat de glace.
REMARQUE: Gardez l’échantillon sur la glace pendant 5 minutes avant le sectionnement pour permettre à la température du cerveau d’atteindre 4 ° C.
8. Culture organotypique en tranches
REMARQUE: Nettoyez le vibratome et les surfaces environnantes avec de l’éthanol à 70% à 96% pour éviter la contamination des tranches. La configuration du poste de travail vibratome (voir Tableau des matériaux) est illustrée à la figure 3B.
- Remplissez le plateau tampon vibratome avec du HBSS-G froid et le plateau extérieur avec de la glace pour maintenir le HBSS-G froid tout au long de la procédure.
- Alimentez en continu HBSS-G dans le bac tampon avec du carbogène à l’aide d’une pierre bouillonnante.
- À l’aide d’une lame fraîche, faites une grande coupe (~ 2 x 2 cm) autour du cerveau et retirez un bloc d’agarose contenant le cerveau, avec suffisamment d’agarose environnante pour couper l’agarose en un petit bloc rectangulaire.
REMARQUE: Cette étape permet d’ajuster l’angle du bloc de sorte que l’axe sagittal du cerveau soit perpendiculaire à la plaque vibratomique et que l’axe coronal s’aligne parallèlement à la lame. Laissez environ 5 mm d’agarose sur la face dorsale du cerveau pour une manipulation facile des tranches. - Placez une petite goutte de supercolle sans solvant à adhésif rapide au milieu du porte-échantillon et étalez-la dans une zone qui couvrira le fond du bloc d’agarose.
- Ramassez doucement le bloc d’agarose et séchez le fond en tamponnant contre du papier de soie. Placez le bloc sur la zone collée du porte-spécimen, avec le côté rostral du cerveau vers le haut. Mettez le porte-spécimen sur de la glace et laissez la colle sécher pendant 1 min.
- Une fois la colle séchée, placez le porte-échantillon dans le bac tampon.
- Coupez le cerveau en tranches coronales à un angle de 15°.
REMARQUE: L’épaisseur des tranches peut varier en fonction de l’application. Ici, les cerveaux ont été tranchés à une épaisseur de 150 μm. Réglez la vitesse du vibratome à 1,0-1,5 mm / s pour couper l’excès d’agarose sur le dessus et couper les bulbes olfactifs. Réduisez la vitesse de coupe à 0,5 mm/s pour la collecte des tranches corticales à des fins d’analyse. La plupart des vibratomes peuvent être mis en pause pour recueillir chaque tranche. Si la qualité réduite des tranches ou le détachement des tissus de l’agarose sont ressentis, la réduction de la vitesse de coupe ou le remplacement de la lame du vibratome peut aider. - À l’aide de spatules propres, collectez des tranches de cerveau et placez-les sur une membrane de polytétrafluoroéthylène (PTFE), immobilisée dans un plat à fond de verre de 35 mm à l’aide de paraffine (jusqu’à cinq tranches de cerveau / membrane) (Figure 3L-M).
REMARQUE: Fixez la membrane en PTFE à l’intérieur d’un plat à fond de verre de 35 mm à l’aide de cire. Cela stabilisera la membrane lors de l’ajout du milieu de culture en tranches et également pendant l’imagerie. - À l’aide d’une pipette de 200 μL, retirer l’excès de HBSS-G autour des tranches de la membrane en PTFE, en laissant les tranches semi-sèches.
- Ajouter 500 μL de milieu de tranche (préchauffé à 35 °C) directement dans l’espace sous la membrane en PTFE.
REMARQUE: Aucune bulle ne doit se former sous la membrane lors de l’ajout du média. Cela laissera des tranches entières ou partielles sans échange de médias. Remplacez 200 μL de média tous les 2 jours en culture ou après chaque séance d’imagerie. - Incuber les tranches à 35 °C avec 5 % de CO2.
9. Immunohistochimie
- Fixer les tranches avec 1 mL de paraformaldéhyde (PFA) à 4 % complété par 4 % de saccharose par plat. Incuber à RT pendant 30 min.
ATTENTION : Lorsque vous manipulez du PFA, portez un manteau de laboratoire et des gants. Effectuez des étapes de fixation sous une hotte chimique et éliminez les déchets PFA de manière appropriée. - Lavez les tranches deux fois avec 300 μL de PBS pendant 5 min. Transférer les tranches dans une assiette de 24 puits.
REMARQUE: L’expérience peut être suspendue à ce stade. Ajouter PBS-NaN3 aux tranches et conserver à 4 °C. NaN3 est un composé toxique; lorsque vous manipulez des solutions avec elle, portez un manteau de laboratoire et des gants. Les étapes 9.3 à 9.10 doivent être effectuées dans un agitateur orbital. - Tremper les tranches avec 300 μL de glycine 0,1 M à 4 °C pendant la nuit.
- Laver la glycine avec PBS à RT 3x pendant 10 min.
- Perméabiliser les tranches avec 300 μL de PBS-T à RT pendant 2 h.
- Bloquer en utilisant 10% de sérum de chèvre dans PBS-T à RT pendant 2 h.
- Ajouter 300 μL d’anticorps primaire (anticorps anti-vimentine à une dilution de 1:200; voir tableau des matières) dilué dans du sérum de chèvre à 10 % dans une solution de PBS-T à 4 °C pendant la nuit.
REMARQUE: Aux étapes 9.8 à 9.12, les tranches étaient protégées contre la lumière pour éviter la perte de fluorescence. - Laver l’anticorps primaire avec PBS à RT 3x pendant 20 min.
REMARQUE: PBS a été utilisé à la place de PBS-T pour laver Triton X-100. - Ajouter 300 μL d’anticorps secondaire (Alexa Fluor 488 ou 647 à une dilution de 1:400; voir tableau des matériaux) dans PBS à RT pendant 2 h.
REMARQUE: DAPI est ajouté immédiatement après avoir retiré l’anticorps secondaire à une dilution de 1:10 000 pendant 5 min. - Laver l’anticorps secondaire avec PBS à RT 3x pendant 20 min. Laver à l’eau distillée 2x pendant 1 min.
- Transférer les tranches sur une lame de verre à l’aide d’une brosse fine, puis sécher à 30 °C pendant 20 min.
- Montez les tranches à l’aide d’un support de montage aqueux. Gardez les diapositives à RT pendant la nuit pour que les supports de montage puissent être conservés.
10. Acquisition d’imagerie
REMARQUE: Quelle que soit l’approche d’administration de l’ADN (IUE ou EUE), les tranches ont été analysées dans la même tranche d’âge de développement (E17.5-E18.5). L’IUE permet aux progéniteurs neuronaux de se diviser et de se développer pendant deux jours supplémentaires in vivo. L’EUE, d’autre part, permet de suivre les événements de développement précoces.
- Allumez l’incubateur à chambre et réglez-le à 35 °C avec 5 % de CO2 – idéalement 4 h avant l’imagerie – pour permettre aux composants du microscope de s’équilibrer à 35 °C.
- Pour l’imagerie profonde des tranches, utilisez des objectifs d’immersion dans l’eau pour réduire l’inadéquation de l’indice de réfraction entre le tissu et l’objectif.
REMARQUE: Ici, le mode d’imagerie super-résolution a été utilisé. L’imagerie à travers la membrane PTFE nécessite un objectif avec une longue distance de travail (~ 1 mm). Si un objectif à longue distance de travail n’est pas disponible, les tranches peuvent être transférées dans un plat à fond de verre à 8 puits. Pour transférer les tranches, ajoutez 1 mL de milieu de tranche au sommet de la membrane, puis utilisez une spatule pour soulever une tranche et transférez-la dans un puits contenant 200 μL de milieu. Enlever l’excès de média à l’aide d’une pointe de pipette de 1 mL en laissant les tranches semi-sèches. - Pour l’imagerie de la croissance des axones, localisez une région du cortex avec une densité cellulaire faible à moyenne. Pour l’imagerie de la dynamique du cône de croissance, localisez un cône de croissance dans la zone intermédiaire ou la zone sous-ventriculaire du cortex.
- Définissez une taille de pile z dans le logiciel de traitement d’image (voir Tableau des matériaux). Pour la croissance des axones dans une grande pile z, définissez une taille d’étape de 2 μm. Pour les cônes de croissance dans une pile z plus petite, définissez une taille de pas de 1 μm.
REMARQUE: Tenez toujours compte du mouvement potentiel du cône de croissance et de l’axone à travers les plans x, y et z. Les axones se développent à un rythme beaucoup plus élevé dans les cultures organotypiques que dans les cultures in vitro . Ici, une pile z d’environ 80 μm pour imager la croissance des axones était suffisante. Pour la dynamique des cônes de croissance, une pile z d’environ 6 μm était adéquate. - Pour l’imagerie de la croissance axonale des neurones dans une plus grande zone, définissez un balayage de tuiles.
- Utilisez la puissance laser la plus faible possible pour minimiser les risques de blanchiment des cônes de croissance lors de l’acquisition.
- Pour l’imagerie de la croissance des axones, acquérir des time-lapses pendant 2 h avec un intervalle de 5 min. Pour l’imagerie de la dynamique des cônes de croissance, acquérez des time-lapses pendant 2-5 min avec un intervalle de 2,5-3 s.
11. Analyse des données
- Mesurer la vitesse de croissance des axones à l’aide de kymographes
- Ouvrez le fichier image dans Fiji14 à l'> Ouvrir et sélectionnez l’image.
- Obtenez la projection d’intensité maximale du time-lapse via image > Stacks > Z-Projection > Maximum Intensity Projection.
- Passez par le time-lapse et localisez un axone en croissance.
- Une fois localisé, tracez une ligne à travers l’axone en croissance. Commencez à partir de la pointe de l’axone dans la première image et suivez l’axone tout au long du time-lapse.
- Générez un kymographe à l’aide du plugin KymoResliceWide.
- Définissez l’échelle du kymographe en accédant à Propriétés de l’image >. Définissez la distance en μm en Largeur de pixel et réglez l’heure en s ou min en Hauteur de pixel.
- Accédez à Analyser > mesure.
REMARQUE: Un angle par rapport à l’axe des x sera donné. - Calculez la vitesse de croissance des axones en substituant l’angle dans l’équation suivante : SIN(RADIANS(θ))/COS(RADIANS(θ)) dans une feuille de calcul.
- Mesurez le volume du cône de croissance à l’aide d’un logiciel d’analyse d’images (voir Tableau des matériaux).
- Ouvrez le fichier image dans le logiciel d’analyse d’images via Fichier > Ouvrir et sélectionnez le fichier d’intérêt.
- Sélectionnez l’Assistant Ajout de nouvelles surfaces .
REMARQUE: Une section dans le coin inférieur gauche apparaîtra avec six étapes pour l’édition manuelle. - À l’étape 1, sous Paramètres de l’algorithme, sélectionnez Segmenter uniquement une région d’intérêt. À l’étape 2, recadrez le cadre pour l’adapter à l’ensemble du cône de croissance dans tous les cadres.
- Maintenez le seuil à l’intensité absolue à l’étape 3 et assurez-vous que toute la région du cône de croissance est seuillée à l’étape 4.
- À l’étape 5, sélectionnez Nombre de voxels lmg = 1 sous Type de filtre.
REMARQUE : Dans la dernière étape, plusieurs ensembles de mesures peuvent être créés. Ici, une seule mesure a été créée pour le volume. - Sélectionnez le bouton Exécuter pour effectuer toutes les étapes de création et terminer l’Assistant Ajout de nouvelles surfaces.
- Dans l’onglet Statistiques en haut de la fenêtre de l’Assistant, sélectionnez Valeurs spécifiques et volume sous l’onglet Détaillé .
Résultats
Les résultats représentatifs obtenus avec le flux de travail de la méthode décrite sont affichés. Les souris E15.5 ont été utilisées dans la présente démonstration, bien que ce protocole soit facilement adaptable à pratiquement tous les âges embryonnaires allant de E11 à la fin de E17. Dans ce protocole, soit l’électroporation ex utero (EUE; Figure 2A, 2C-I) ou électroporation in utero (IUE; Figure 2B, C et 2J-Q) ont été utilisés pour délivrer des plasmides dans les neurones progéniteurs tapissant les ventricules latéraux. Ces progéniteurs sont la source des futurs neurones projetés corticaux (CPN)15,16. Des mélanges de plasmides ont été préparés pour conduire une expression spécifique aux neurones clairsemés de Lyn)-mNeonGreen ciblés par la membrane (Figure 1A) ou de LifeAct-enhanced (E)GFP (Figure 1B) pour évaluer le comportement global et la dynamique de l’actine dans les cônes de croissance, respectivement. De plus, un mélange de plasmides visait à marquer des neurones individuels avec une protéine fluorescente verte turbo(t)-RFP ou zoanthus sp. (Zs) (ZsGreen) (Figure 1C) a été inclus. Cela facilite la surveillance du comportement du cône de croissance des neurones voisins indépendants.
La dissection cérébrale à partir d’embryons électroporés est une étape cruciale qui doit être soigneusement effectuée pour obtenir des tranches de haute qualité, préservant la structure cérébrale native. Les instruments de dissection et le vibratome ont été préparés à l’avance et soigneusement stérilisés à l’éthanol (Figure 3A,B). Ensuite, les têtes des embryons électroporés ont été soigneusement disséquées et les cerveaux ont été extraits. Ici, la dissection représentative des cerveaux des embryons soumis à l’EUE à E15 (Figure 3C-F) et E12.5 (Figure 3G-J) est montrée. Les cerveaux sont immédiatement enfermés dans une matrice d’agarose, tranchés et placés sur des inserts de membrane en PTFE dans une boîte en verre de fond pour l’incubation (Figure 3K-M).
L’état de santé des tranches de cerveau est un point important pour le contrôle afin d’assurer des résultats fiables. Une inspection visuelle de toute contamination a été effectuée quotidiennement. De plus, une fois la culture finalisée, les tranches de cerveau sont fixées et soumises à l’immunohistochimie. Ici, le 4′,6-diamidino-2-phénylindole (DAPI) a été utilisé pour contrôler l’organisation cellulaire globale et la coloration de la vimentine pour révéler l’organisation gliale; en particulier, l’échafaudage de la glie radiale (RG). En règle générale, les tranches de cerveau cultivées avec succès dérivées de l’IUE ou de l’EUE montrent une distribution cellulaire normale révélée par DAPI et un ensemble quelque peu organisé de RG avec des processus de contact pial orientés vers l’apique17 (Figure 4A, B respectivement). Parfois, des perturbations marquées de l’échafaudage RG dans les tranches de cerveau cultivées sont observées, en particulier dans celles dérivées de l’électroporation EUE (Figure 4C). Les tranches de cerveau avec un échafaudage RG extrêmement désorganisé montrent une migration neuronale altérée et une croissance axonale défectueuse (non montrée). Par conséquent, le contrôle de l’échafaudage RG est une méthode de post-culture facile pour trier les données obtenues à partir de tranches de cerveau fiables.
Les tranches de cerveau dérivées de l’IUE ou de l’EUE avec un mélange de plasmides exprimant Lyn-mNeonGreen entraînent un marquage similaire des neurones clairsemés. Un CPN pyramidal représentatif exprimant Lyn-mNeonGreen et le comportement dynamique de son cône de croissance est illustré à titre d’exemple (Figure 5A et Vidéo supplémentaire 1, en haut à gauche). De plus, les neurones ont été marqués à l’aide d’un plasmide exprimant une sonde d’actine pour analyser la dynamique de l’actine des cônes de croissance axonale in situ (Figure 5B et vidéo supplémentaire 1, en bas à gauche). Des expériences in situ ont également été réalisées avec un modèle de plasmide exprimant le fluorophore à double Cre/Dre (Figure 1C et Vidéo supplémentaire 1, à droite). Les fluorophores tRFP ou ZsGreen contenus dans ce plasmide pourraient être activés spécifiquement et individuellement par les recombinases Dre ou Cre, respectivement, dans les neurones voisins (Figure 5C). Cette gamme expérimentale permet une analyse côte à côte des cônes de croissance des neurones témoins avec des neurones modifiés voisins (toute perte ou gain de fonction donné). Cela permet de contourner la variabilité résultant de l’utilisation de différentes tranches pour tester le contrôle et les conditions expérimentales.
Les kymographes générés à partir du film enregistré ont été analysés, à partir desquels des paramètres de croissance dynamique tels que l’activité saillante dans le temps et la durée de croissance peuvent être facilement obtenus (Figure 6A). Notez qu’un simple réglage de la résolution temporelle du time-lapse permet de mesurer la vitesse d’allongement de l’axone pendant 2 h (Figure 6A). En outre, la variation du volume du cône de croissance au fil du temps - une mesure de l’activité dynamique générale du cône de croissance - peut être facilement obtenue, dans ce cas avec un logiciel sous licence (Figure 6B et Figure 6E,F). Cela peut être utilisé pour évaluer la vitesse du tapis roulant à l’actine et l’équilibre des filopodia/lamellipodies pendant l’activité d’exploration du cône de croissance.
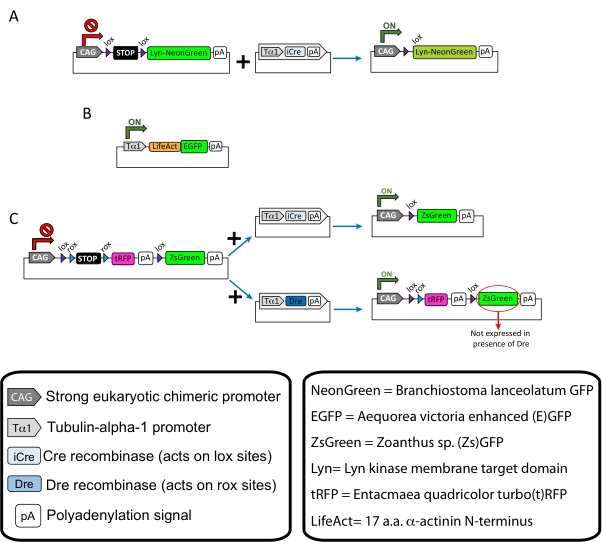
Figure 1 : Schémas des plasmides utilisés dans le protocole. (A) pCAG-lox-STOP-lox-Lyn-mNeonGreen. (B) p-Tub-alpha-1-LifeAct-GFP. (C) pCAG-lox-rox-STOP-rox-tRFP-pA-lox-ZsGreen-pA. Des informations pertinentes concernant les composants plasmidiques et l’origine du fluorophore se trouvent dans les boîtes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
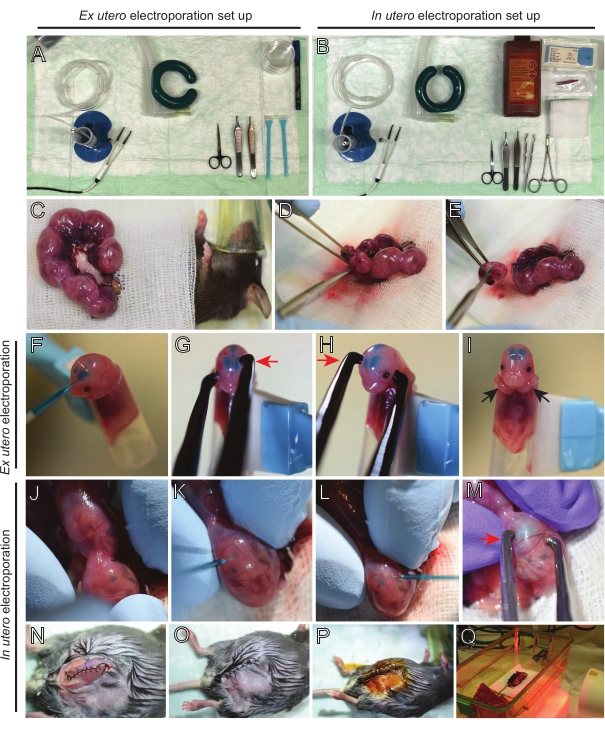
Figure 2 : Flux de travail de l’électroporation ex utero et in utero de souris E15.5. (A) Mise en place d’un poste de chirurgie pour l’électroporation ex utero. (B) Mise en place d’un poste de chirurgie pour l’électroporation in utero. (C) Cornes utérines tirées à l’extérieur de la cavité abdominale de la souris anesthésiée. D) Extraction d’un embryon du sac utérin. E) Sacrifice d’embryons par transsection complète de la moelle épinière par incision diagonale; notez que la décapitation a été évitée. (F) Placement de l’embryon dans le support et injection avec un mélange ADN/Fast Green dans le ventricule latéral gauche. (G,H) Positionnez la tête de l’embryon entre les électrodes de la pince à platine avec la cathode (flèche rouge) sur le cortex à un angle de 60°. (I) Placement des bras de l’embryon (flèches noires) à l’extérieur du support pour éviter le glissement de l’embryon pendant la procédure. (J) Rotation de l’embryon à l’intérieur du sac utérin pour exposer la tête. (K,L) Injection du mélange ADN/Fast Green dans les ventricules latéraux de l’embryon à travers la paroi utérine. (M) Positionner la tête de l’embryon entre les électrodes de la pince à épiler en platine avec une cathode (flèche rouge) sur le cortex à un angle de 60°. (N) Incision musculaire suturée via une suture de verrouillage de course. (O) Incision cutanée suturée par une suture interrompue. (P) Fixation de la plaie à l’aide de clips chirurgicaux et désinfection à l’aide de bétadine. (Q) Placement de la souris dans la cage de récupération avec une lumière chauffante infrarouge lointaine. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Extraction des cerveaux E15.5 et E12.5 et procédure de culture de tranches organotypiques. (A) Outils utilisés pour la procédure d’extraction cérébrale. (B) Mise en place d’une station de culture organotypique. (C-F) Extraction du cerveau E15.5. (G-J) Extraction du cerveau E12.5. Les lignes pointillées mettent en évidence l’emplacement des incisions. Des flèches rouges indiquent la direction de la traction par pince. (K) Intégrer le cerveau dans un plat de 3 cm contenant 3% d’agarose à faible teneur en fusion, laissant un espacement d’espacement des agaroses de 1 à 2 mm sous le cerveau. (L) Collecte d’une tranche de cerveau de 150 μm. (M) Placement de tranches de cerveau sur des inserts membranaires en PTFE immobilisés dans un plat de 35 mm à l’aide d’un film de paraffine (flèche bleue). Le marquage en étoile rouge indique une collection donnée de tranches de cerveau à partir du vibratome (L) et son transfert à la membrane PTFE (M). Veuillez cliquer ici pour voir une version agrandie de cette figure.
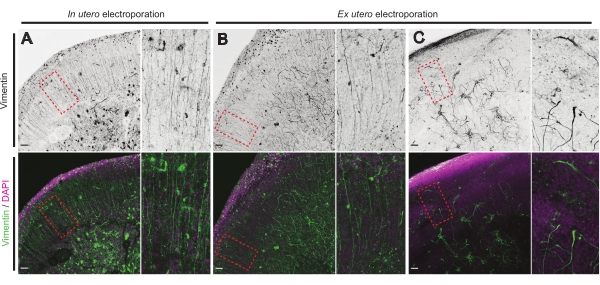
Figure 4 : Structure cellulaire gliale radiale conservée dans des tranches organotypiques saines. Images confocales de tranches cérébrales E17.5 révélant un réseau RG (vimentine; vert) et une organisation cellulaire globale (DAPI; magenta) après IUE (A) et EUE (B, C). Notez les fortes perturbations dans le réseau RG qui peuvent parfois résulter de l’EUE (C). Les grossissements correspondent aux cadres pointillés rouges de la figure principale : barres d’échelle, 10 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
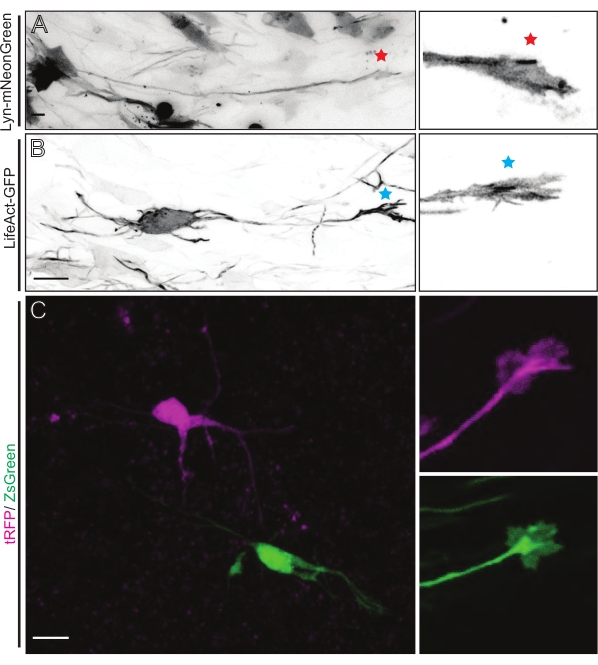
Figure 5 : Visualisation in situ de la dynamique du cône de croissance dans les tranches organotypiques aiguës. (A,B) Neurones et leurs cônes de croissance correspondants marqués respectivement avec Lyn-mNeonGreen et LifeAct-GFP. Étoile rouge marquant le cône de croissance du neurone exprimant Lyn-mNeonGreen. Astérisque bleu marquant le cône de croissance du neurone exprimant LifeAct-GFP. (C) Neurones voisins marqués avec le système plasmidique double contenant tRFP (magenta) et ZsGreen (vert) et leurs cônes de croissance correspondants. Les cônes de croissance imagés (à droite) étaient en dehors du cadre capturé (à gauche), obtenus peu de temps après l’acquisition du time-lapse du cône de croissance; barres d’échelle, 5 μm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
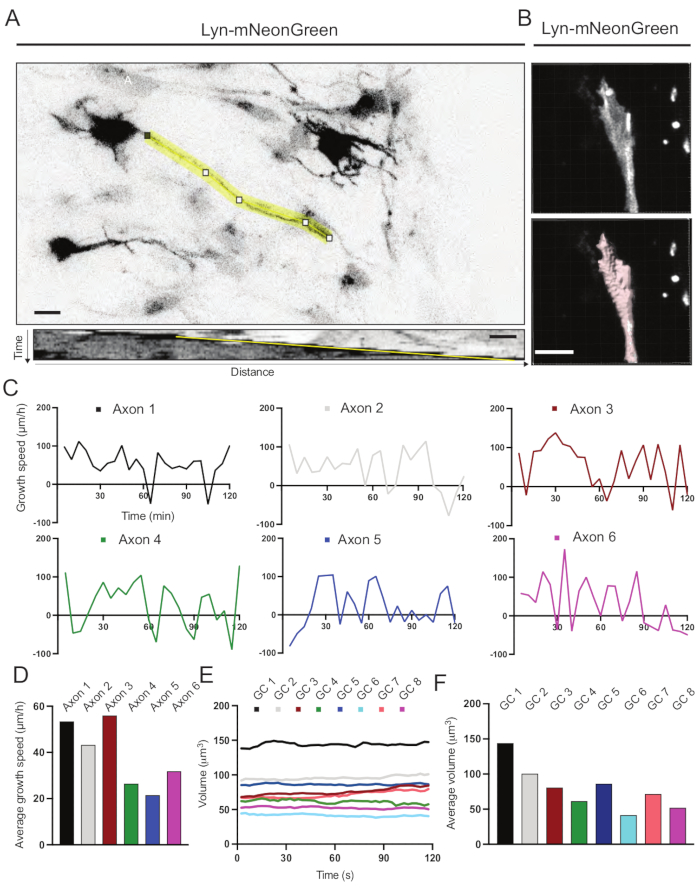
Figure 6 : Analyse de la vitesse de croissance des axones et du volume du cône de croissance. (A) Traçage des axones sur un neurone exprimant Lyn-mNeonGreen (en haut) et son kymographe correspondant (ci-dessous) généré à l’aide d’ImageJ. (B) Reconstruction de la vidéo z-stack du cône de croissance à l’aide du logiciel d’analyse d’images (en haut) et du même cône de croissance mis en évidence à l’aide de l’outil de mesure des surfaces (ci-dessous). (C) Graphiques montrant les changements de vitesse de croissance au fil du temps pour plusieurs axones. (D) La vitesse de croissance moyenne des axones est quantifiée en (C). (E) Graphique montrant les variations du volume du cône de croissance au fil du temps. F) Le volume moyen des cônes de croissance est quantifié en (E); barre d’échelle, 5 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
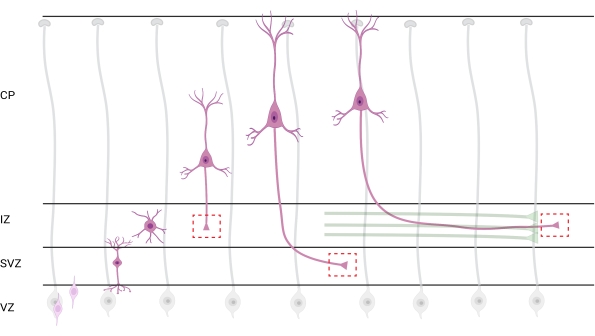
Figure 7 : Migration radiale et polarisation neuronale des neurones corticaux pyramidaux. Diagramme illustrant le développement de neurones corticaux pyramidaux (rose) migrant radialement de la zone ventriculaire germinale (VZ) vers la surface pia. Guidés par des processus de glie radiale (gris), les neurones polarisés en migration établissent un processus principal, la dendrite future, et le processus de suivi, l’axone futur, qui continuent de s’étendre vers le bas vers la zone intermédiaire (IZ). Les boîtes rouges en pointillés représentent les zones corticales où les cônes de croissance ont été imagés. Plus précisément dans l’IZ, zone sous-ventriculaire (SVZ), ou jonction de faisceaux d’axones (vert). L’illustration a été créée à l’aide d’un outil Web, BioRender.com. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Plasmide | Concentration (μg/μL) | Utilisation prévue |
| pCAG-lox-STOP-lox-Lyn-mNeonGreen | 0.25 | Marquage de la protéine membranaire ciblée (Lyn) |
| + | + | |
| p-Tub-alpha-1-iCre | 0.08 | |
| p-Tub-alpha-1-LifeAct-GFP | 0.125 | Marquage de l’actine filamenteuse (F-actine) dans les cônes de croissance |
| pCAG-lox-rox-STOP-rox-tRFP-lox-Lyn-ZsGreen | 1 | Marquage indépendant de deux populations de neurones voisins |
| + | + | |
| p-Tub-alpha-1-iCre | 0.004 | |
| + | + | |
| p-Tub-alpha-1-Dre | 0.2 |
Tableau 1 : Liste des plasmides utilisés dans le Protocole. Nom, concentration et utilisation prévue de chaque plasmide utilisé.
Vidéo supplémentaire 1 : Visualisation in situ de la dynamique du cône de croissance dans les tranches organotypiques aiguës. Dynamique des cônes de croissance étiquetés avec Lyn-mNeonGreen (en haut à gauche) et LifeAct-GFP (en bas à gauche). Les cônes de croissance voisins sont marqués différentiellement avec le système plasmidique double contenant tRFP (magenta; en haut à droite) et ZsGreen (vert; en bas à droite). Intervalle d’imagerie, 2,5 s. Barres d’échelle, 5 μm. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
La façon dont les cônes de croissance détectent et réagissent à leur environnement environnant pour coordonner l’extension et le guidage simultanés des axones est encore un sujet de débat 3,18. Des études pionnières sur des substrats 2D ont donné un aperçu des mécanismes moléculaires fondamentaux générant les forces qui stimulent la dynamique des cônes de croissance pendant la formation des axones, l’excroissance et la navigation 2,10,11,12,19. Plus récemment, des études dans des matrices 3D ont révélé l’influence d’une troisième dimension sur le comportement du cône de croissance et par conséquent sur la croissance desaxones 8,9. Néanmoins, les mécanismes complexes instruisant la dynamique des cônes de croissance in vivo restent à examiner en profondeur.
La préparation de cultures de tranches organotypiques à partir de cerveaux IUE ou EUE est largement utilisée et bien documentée. C’est devenu une norme d’or permettant aux scientifiques d’acquérir des connaissances sur le développement et le comportement des neurones dans le tissu cérébral vivant20,21. En effet, cette technique a été utilisée avec succès en combinaison avec diverses techniques d’imagerie à haute résolution pour visualiser des processus moléculaires spécifiques et des événements morphologiques in situ. Ces études incluent, sans toutefois s’y limiter, la formation et l’extension des axones19,22, la migration neuronale corticale 19,22,23,24, la dynamique des centrosomes 25,26, la dynamique des microtubules27, ainsi que la dynamique fonctionnelle des compartiments pré-et postsynaptiques 28,29.
Ce protocole comble une lacune dans la neurobiologie expérimentale, en visualisant la dynamique des cônes de croissance des neurones corticaux en développement in situ, dans les cultures de tranches cérébrales aiguës ex vivo , et les outils pour analyser les données obtenues.
Des cultures de tranches cérébrales aiguës ont été utilisées pour établir ce protocole parce qu’elles (1) avec un peu de pratique, sont faciles à générer; (2) présenter un système accessible pour étudier les cônes de croissance intégrés dans un environnement quasi entièrement physiologique, mais suffisamment transparent pour permettre l’imagerie de cellules vivantes à haute résolution; (3) peut être élargi pour son utilisation avec une myriade de lignées de souris transgéniques; (4) combinés à l’IUE ou à l’EUE, offrent un potentiel pratiquement illimité pour fournir des outils moléculaires permettant d’évaluer les performances des cônes de croissance et des axones in vivo sous des régimes de perte/gain de fonction, ainsi que des rapporteurs fluorescents et des sondes de cytosquelette.
Cette méthodologie a été décrite dans le contexte de l’EUE et de l’IUE. Bien qu’il s’agisse toujours d’une méthode très fiable, l’EUE a entraîné une augmentation de l’incidence des tranches de cerveau montrant un réseau RG désorganisé par rapport à celles obtenues avec l’IUE comme méthode d’administration (Figure 4C). Les perturbations dans le réseau RG affectent fortement la migration neuronale et le modèle d’allongement axonal30,31. Ce sont des paramètres clés qui prédisent où trouver des axones à analyser à un moment donné et le type d’environnement dans lequel ils naviguent. Les tranches de cerveau avec un réseau RG significativement perturbé ont généralement une stratification altérée des neurones corticaux. Ceci, à son tour, produit des axones avec des trajectoires chaotiques. Par conséquent, il est fortement recommandé de contrôler l’intégrité structurelle du réseau RG. Fait intéressant, une mauvaise intégrité structurelle est en corrélation avec l’augmentation de l’âge du cerveau embryonnaire. En effet, de tels effets chez les embryons E12.5-E13.5 plus jeunes n’ont généralement pas été observés19.
Le présent protocole est complet et simple. Néanmoins, il y a quelques étapes critiques où un soin et une attention particuliers doivent être pris pour obtenir des résultats optimaux. Ceux-ci ont été expressément notés dans le protocole et comprennent (1) l’ajustement de la quantité d’ADN utilisée dans l’électroporation pour obtenir un marquage clairsemé; (2) éviter les dommages lors de l’extraction des cerveaux; (3) contrôler la température de l’agarose pendant le tubage du cerveau; (4) dépannage du pourcentage idéal d’agarose pour les cerveaux d’un âge donné; et (5) la sélection des fluorophores, dont l’expérience suit. Au cours de l’optimisation du protocole, les performances de plusieurs fluorophores en imagerie in situ à cellules vivantes ont été testées. Les variantes monomères de GFP EGFP et NeonGreen pour la préparation des plasmides marqués LifeAct et Lyn ont été choisies pour ce protocole (Figure 5A,B). De plus, la variante d’appel d’offres mScarlet a été testée et jugée très appropriée pour cette configuration (données non présentées). Le tRFP multimérique (dimère) et le ZsGreen (tétramère) (figure 5C et vidéo supplémentaire 1, à droite) ont également été testés. Ces fluorophores ultra-brillants à pliage rapide sont recommandés lorsque la méthode nécessite une génération rapide de signaux fluorescents après la livraison de l’ADN.
Une pratique courante dans l’utilisation de cultures en tranches consiste à utiliser des tranches de différents cerveaux pour tester le contrôle et les conditions expérimentales. Cela représente une source inhérente de variabilité indésirable. Ici, un système d’expression qui permet une modification indépendante des neurones voisins et de l’expression des rapporteurs pour l’identification a été utilisé. Notez que dans cette démonstration (Figure 5C), il n’y avait aucune différence entre les neurones exprimant l’un ou l’autre des fluorophores. Cependant, à titre d’exemple, un tel mélange de plasmides combiné à une lignée de souris transgénique abritant un gène sensible à la Cre marquera avec des neurones tRFP (sensibles à la Dre) qui sont restés de type sauvage. En revanche, le ZsGreen (également sensible à la Cre) marquera les neurones recombinés. Par conséquent, les cônes de croissance des deux génotypes différents, et probablement aussi les phénotypes, pourraient être étudiés côte à côte simultanément dans la même tranche de cerveau.
La localisation des axones et des cônes de croissance pour l’analyse est une considération importante. Les neurones corticaux se polarisent lors de la migration radiale de la zone ventriculaire (VZ) vers la plaque corticale (CP). Au cours de ce processus, les neurones forment un processus principal (une future dendrite) et un processus de suivi qui deviendra l’axone, rejoignant éventuellement les axones pionniers dans la zone intermédiaire (IZ), établissant les voies axonales32. Par conséquent, pour capturer les cônes de croissance axonale, l’imagerie a été effectuée sur les fibres axonales dans l’IZ, y compris les axones sortant de la CP et les axones générés tôt déjà associés aux faisceaux axonal; ou éventuellement, dans des fibres qui traversent l’IZ et s’étendent en dessous (Figure 7).
Ce protocole permet d’effectuer une imagerie à super-résolution des neurones dans des tranches organotypiques. Historiquement, la diffusion de la lumière était un problème important rencontré lors de l’imagerie de spécimens épais. Au cours des deux dernières décennies, les progrès considérables des technologies optiques ont rendu possible l’imagerie de spécimens épais. Ici, un objectif à longue distance de travail a été utilisé pour mieux visualiser les structures plus petites, telles que les cônes de croissance. Inévitablement, ce protocole ne capture pas des événements plus détaillés tels que l’écoulement rétrograde de l’actine ou la dynamique des microtubules. L’objectif à distance de travail à long terme, qui nécessite une ouverture numérique (NA) plus faible, préserve les informations des tranches épaisses. Cependant, il a également été possible d’adapter ce protocole pour l’utiliser avec des objectifs de distance de travail plus courte. Cela nécessitait un transfert en douceur des tranches dans un plat à fond de verre pour préserver l’intégrité structurelle. Cependant, l’utilisation de cette méthode a entraîné une survie plus courte - ~ 15 h - en raison de la perte d’échange de gaz (données non montrées). Contrairement aux cultures 2D, les cônes de croissance en 3D occupent un volume plus important et nécessitent une compensation mouvement-artefact dans l’axe z. Pour augmenter la capacité d’image d’événements détaillés, la technologie confocale moderne doit être utilisée. Par conséquent, il est recommandé d’utiliser un moteur z-stack à balayage rapide, tel que le z-Galvo disponible sur les microscopes confocaux très sensibles33.
Il convient de noter que ce protocole présente trois limites principales. Premièrement, il est souvent difficile de contrôler les niveaux d’expression / nombre de cellules exprimant un plasmide donné in vivo. Cela introduit une variabilité entre toutes les tranches, même en maintenant la même concentration de plasmides. Par conséquent, le choix des éléments régulateurs dans les vecteurs d’expression utilisés doit être prédéterminé avec soin. Deuxièmement, il n’est actuellement pas possible d’imager des événements détaillés à l’aide d’inserts membranaires. Cette deuxième limite peut être surmontée avec les mises à jour méthodologiques proposées au paragraphe précédent. Enfin, les cônes de croissance sont très photosensibles et peuvent rapidement devenir photoblanchis. Par conséquent, l’imagerie fréquente des cônes de croissance, pendant aussi peu que 5 minutes à l’aide de microscopes à balayage laser, peut souvent faire s’effondrer les cônes de croissance. À cet égard, les nouvelles avancées dans les dispositifs générés par microscopie à feuille de lumière peuvent être adaptées à l’imagerie à long terme des tranches de cerveau34.
Des protocoles similaires sont envisagés pour ouvrir de nouvelles voies de recherche, permettant une meilleure compréhension de ce qu’il faut pour qu’un cône de croissance lise et réagisse à un environnement in vivo complexe, et plus important encore, pour démêler la mécanique de cette interaction sophistiquée.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous tenons à remercier Maria Eugenia Bernis d’avoir photographié les procédures. Nous remercions également Emily Burnside, Emily Handley, Thorben Pietralla, Max Schelski et Sina Stern d’avoir lu et discuté du manuscrit. Nous sommes reconnaissants à nos assistants techniques exceptionnels, Jessica Gonyer, Blanca Randel et Anh-Tuan Pham. Nous reconnaissons le soutien précieux de l’installation de microscope optique et de l’installation pour animaux de la DZNE. Ce travail a été soutenu par la Deutsche Forschungsgesellschaft (DFG), la Fondation internationale pour la recherche sur la paraplégie (IRP) et Wings for Life (à F.B). F.B. est membre du pôle d’excellence ImmunoSensation2, des SFB 1089 et 1158, et est récipiendaire du prix Roger De Spoelberch.
matériels
| Name | Company | Catalog Number | Comments |
| Adson Forceps | Fine Science Tools | 11006-12 | |
| Alexa Fluor 488 | Invitrogen | A21202 | Goat Anti-Mouse |
| Alexa Fluor 647 | Invitrogen | A21236 | Goat Anti-Mouse |
| Anti-Vimentin antibody | sigma-Aldrich | V2258-.2ML | Monoclonal mouse, clone LN-6, ascites fluid |
| B27 supplement | ThermoFisher Scientific | 17504044 | |
| Betadine | B. Braun | 3864154 | |
| Biozym Sieve GP Agarose | Biozyme | 850080 | |
| Braunol, Sprühflasche | B. Braun | 3864073 | |
| Buprenorphine (Temgesic) | GEHE Pharma | 345928 | |
| DAPI | sigma-Aldrich | D9542 | |
| DMZ unevirsal electrode puller | Zeitz | NA | |
| Electric razor | Andes | NA | ProClip UltraEdge Super 2-Speed model |
| Enrofloxacin (Baytril) | Bayer | 3543238 | 2,5% (wt/vol) |
| Eppendorf microloader pipette tips | FischerScientific | 10289651 | |
| Fast Green FCF | Sigma-Aldrich | F7252-5G | Dye content ≥ 85 % |
| Fetal Bovine Serum | ThermoFisher Scientific | 10500064 | |
| Fiji 2.1.0 | NIH | NA | https://imagej.net/software/fiji/downloads |
| Fine Scissors | Fine Science Tools | 14058-09 | ToughCut/Straight/9cm |
| FluoroDish Cell Culture Dish | World Precision Instruments | FD5040-100 | |
| Fluoromount Aqueous Mounting Medium | sigma-Aldrich | F4680-25ML | |
| Glucose | MedPex | 3705391 | 5% |
| GlutaMAX Supplement | ThermoFisher Scientific | 35050061 | |
| Glycine | Sigma-Aldrich | G8898 | |
| HBSS | Life Technologies | 14025092 | calcium, magnesium, no phenol red |
| Horse serum | Pan-Biotech | P30-0711 | |
| Imaris 9.7.2 | Bitplane | NA | https://imaris.oxinst.com/products/imaris-for-neuroscientists |
| Isoflurane | Virbac | NA | |
| Isotonic saline solution | B. Braun | 8609261 | 0.90% |
| Leica VT1200 S vibratome | Leica | 14048142066 | |
| LSM 880 with Airyscan | Zeiss | NA | |
| Metacam | Venusberg Apotheke | 8890217 | 5 mg/ml |
| Mice | Janvier Labs | NA | C57BL/6JRj |
| Micro-Adson Forceps | Fine Science Tools | 11018-12 | |
| Micropipette Storage Jar | World Precision Instruments | E210 | 16.16.27 |
| Microsoft Excel | Microsoft | NA | https://www.microsoft.com/en-us/microsoft-365/p/excel/cfq7ttc0k7dx?activetab=pivot:overviewtab |
| Millicell Cell Culture Insert | EMD Millipore | PICM0RG50 | 30 mm, hydrophilic PTFE, 0.4 µm |
| Moria Perforated Spoons | Fine Science Tools | 10370-18 | |
| Moria Spoon | Fine Science Tools | 10321-08 | |
| Neurobasal Medium, minus phenol red | ThermoFisher Scientific | 12348017 | |
| Neuropan-2 supplement | Pan-Biotech | P07-11010 | |
| Normal goat serum | Abcam | ab138478 | |
| Olsen-Hegar Needle Holder with Scissors | Fine Science Tools | 12002-12 | |
| p-Tub-alpha-1-Dre | Addgene | 133925 | |
| p-Tub-alpha-1-iCre | Addgene | 133924 | |
| p-Tub-alpha-1-LifeAct-GFP | Addgene | 175437 | |
| Parafilm | VWR | 52858-000 | |
| Paraformaldehyde | sigma-Aldrich | P6148 | |
| PBS | Sigma-Aldrich | P3813-10PAK | |
| pCAG-lox-rox-STOP-rox-tRFP-lox-Lyn-ZsGreen | Addgene | 175438 | |
| pCAG-lox-STOP-lox-Lyn-mNeonGreen | Addgene | 175257 | |
| Penicillin-Streptomycin | ThermoFisher Scientific | 15140122 | |
| PicoNozzle Kit v2 | World Precision Instruments | 5430-ALL | |
| Platinum Tweezertrodes | Harvard Apparatus | 45-0487 | 1 mm / 3 mm |
| QIAGEN Maxi kit | QIAGEN | 12162 | |
| Reflex wound closure Clip | World Precision Instruments | 500344-10 | 7 mm |
| Sekundenkleber Pattex Mini Trio | Lyreco | 4722659 | |
| Square wave electroporation system ECM830 | Harvard Apparatus | W3 45-0052 | |
| Sterile gauze | Braun Askina | 9031216 | |
| Sterile lubricant eye ointment | Bayer Vital | PZN1578675 | |
| Sterile surgical gloves | Sempermed | 14C0451 | |
| Sucrose | Roth | 4621.2 | |
| Supramid 5-0 surgical silk sutures | B. Braun | NA | |
| Thin-wall glass capillaries | World Precision Instruments | TW100-4 | |
| Triton X-100 | Sigma-Aldrich | X100 | |
| Vannas spring scissors | Fine Science Tools | 15000-03 | |
| µ-Slide 8 Well Glass Bottom | Ibidi | 80827 |
Références
- Schelski, M., Bradke, F. Neuronal polarization: From spatiotemporal signaling to cytoskeletal dynamics. Molecular and Cellular Neurosciences. 84, 11-28 (2017).
- Lowery, L. A., Van Vactor, D. The trip of the tip: understanding the growth cone machinery. Nature Reviews: Molecular Cell Biology. 10 (5), 332-343 (2009).
- Stoeckli, E. T. Understanding axon guidance: are we nearly there yet. Development. 145 (10), (2018).
- Bradke, F., Dotti, C. G. The role of local actin instability in axon formation. Science. 283 (5409), 1931-1934 (1999).
- Neukirchen, D., Bradke, F. Cytoplasmic linker proteins regulate neuronal polarization through microtubule and growth cone dynamics. Journal of Neuroscience. 31 (4), 1528-1538 (2011).
- Witte, H., Bradke, F. The role of the cytoskeleton during neuronal polarization. Current Opinion in Neurobiology. 18 (5), 479-487 (2008).
- Witte, H., Neukirchen, D., Bradke, F. Microtubule stabilization specifies initial neuronal polarization. Journal of Cell Biology. 180 (3), 619-632 (2008).
- Nichol, R. H., Catlett, T. S., Onesto, M. M., Hollender, D., Gomez, T. M. Environmental elasticity regulates cell-type specific RHOA signaling and neuritogenesis of human neurons. Stem Cell Reports. 13 (6), 1006-1021 (2019).
- Santos, T. E., et al. Axon growth of CNS neurons in three dimensions is amoeboid and independent of adhesions. Cell Reports. 32 (3), 107907(2020).
- Mitchison, T., Kirschner, M. Cytoskeletal dynamics and nerve growth. Neuron. 1 (9), 761-772 (1988).
- Lin, C. H., Thompson, C. A., Forscher, P. Cytoskeletal reorganization underlying growth cone motility. Current Opinion in Neurobiology. 4 (5), 640-647 (1994).
- Myers, J. P., Gomez, T. M. Focal adhesion kinase promotes integrin adhesion dynamics necessary for chemotropic turning of nerve growth cones. Journal of Neuroscience. 31 (38), 13585-13595 (2011).
- Turney, S. G., et al. Nerve growth factor stimulates axon outgrowth through negative regulation of growth cone actomyosin restraint of microtubule advance. Molecular Biology of the Cell. 27 (3), 500-517 (2016).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Noctor, S. C., Flint, A. C., Weissman, T. A., Dammerman, R. S., Kriegstein, A. R. Neurons derived from radial glial cells establish radial units in neocortex. Nature. 409 (6821), 714-720 (2001).
- Noctor, S. C., Martinez-Cerdeno, V., Ivic, L., Kriegstein, A. R. Cortical neurons arise in symmetric and asymmetric division zones and migrate through specific phases. Nature Neuroscience. 7 (2), 136-144 (2004).
- Ferent, J., Zaidi, D., Francis, F. Extracellular control of radial glia proliferation and scaffolding during cortical development and pathology. Frontiers in Cell and Developmental Biology. 8, 578341(2020).
- Dent, E. W., Gupton, S. L., Gertler, F. B. The growth cone cytoskeleton in axon outgrowth and guidance. Cold Spring Harbor Perspectives in Biology. 3 (3), (2011).
- Dupraz, S., et al. RhoA controls axon extension independent of specification in the developing brain. Current Biology. 29 (22), 3874-3886 (2019).
- Azzarelli, R., Oleari, R., Lettieri, A., Andre, V., Cariboni, A. In vitro, ex vivo and in vivo techniques to study neuronal migration in the developing cerebral cortex. Brain Sciences. 7 (5), (2017).
- Humpel, C. Organotypic brain slice cultures: A review. Neuroscience. 305, 86-98 (2015).
- Namba, T., et al. Pioneering axons regulate neuronal polarization in the developing cerebral cortex. Neuron. 81 (4), 814-829 (2014).
- Shah, B., et al. Rap1 GTPases are master regulators of neural cell polarity in the developing neocortex. Cerebral Cortex. 27 (2), 1253-1269 (2017).
- Wiegreffe, C., Feldmann, S., Gaessler, S., Britsch, S. Time-lapse confocal imaging of migrating neurons in organotypic slice culture of embryonic mouse brain using in utero electroporation. Journal of Visualized Experiments: JoVE. (125), (2017).
- de Anda, F. C., Meletis, K., Ge, X., Rei, D., Tsai, L. H. Centrosome motility is essential for initial axon formation in the neocortex. Journal of Neuroscience. 30 (31), 10391-10406 (2010).
- Sakakibara, A., et al. Dynamics of centrosome translocation and microtubule organization in neocortical neurons during distinct modes of polarization. Cerebral Cortex. 24 (5), 1301-1310 (2014).
- Schatzle, P., Kapitein, L. C., Hoogenraad, C. C. Live imaging of microtubule dynamics in organotypic hippocampal slice cultures. Methods in Cell Biology. 131, 107-126 (2016).
- Qu, X., Kumar, A., Bartolini, F. Live imaging of microtubule dynamics at excitatory presynaptic boutons in primary hippocampal neurons and acute hippocampal slices. STAR Protocols. 2 (1), 100342(2021).
- Tonnesen, J., Katona, G., Rozsa, B., Nagerl, U. V. Spine neck plasticity regulates compartmentalization of synapses. Nature Neuroscience. 17 (5), 678-685 (2014).
- Buchsbaum, I. Y., Cappello, S. Neuronal migration in the CNS during development and disease: insights from in vivo and in vitro models. Development. 146 (1), (2019).
- Rigby, M. J., Gomez, T. M., Puglielli, L. Glial cell-axonal growth cone interactions in neurodevelopment and regeneration. Frontiers in Neuroscience. 14, 203(2020).
- Barnes, A. P., Polleux, F. Establishment of axon-dendrite polarity in developing neurons. Annual Review of Neuroscience. 32, 347-381 (2009).
- Multiphoton Microscope Leica TCS SP8 MP. Microsystems. , Available from: http://www.leica-microsystems.com/products/confocal-microscopes/p/leica-tcs-sp8-sted-one/ (2021).
- ZEISS Lattice Lightsheet 7. Zeiss. , Available from: http://www.zeiss.com/microscopy/int/products/imaging-systems/lattice-lightsheet-7.html (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon