Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Imágenes en vivo del estado redox de glutatión mitocondrial en neuronas primarias utilizando un indicador ratiométrico
En este artículo
Resumen
Este artículo describe un protocolo para determinar las diferencias en el estado redox basal y las respuestas redox a las perturbaciones agudas en las neuronas primarias del hipocampo y cortical utilizando microscopía viva confocal. El protocolo se puede aplicar a otros tipos de células y microscopios con modificaciones mínimas.
Resumen
La homeostasis redox mitocondrial es importante para la viabilidad y función neuronal. Aunque las mitocondrias contienen varios sistemas redox, el muy abundante glutatión tampón redox tiol-disulfuro se considera un jugador central en las defensas antioxidantes. Por lo tanto, la medición del potencial redox de glutatión mitocondrial proporciona información útil sobre el estado redox mitocondrial y el estrés oxidativo. Glutaredoxin1-roGFP2 (Grx1-roGFP2) es un indicador ratiométrico basado en proteína fluorescente verde (GFP) codificado genéticamente del potencial redox de glutatión que tiene dos picos de excitación sensibles al estado redox a 400 nm y 490 nm con un solo pico de emisión a 510 nm. Este artículo describe cómo realizar una microscopía viva confocal de Grx1-roGFP2 dirigida a mitocondrias en neuronas primarias del hipocampo y cortical. Describe cómo evaluar el potencial redox de glutatión mitocondrial en estado estacionario (por ejemplo, para comparar estados de enfermedad o tratamientos a largo plazo) y cómo medir los cambios redox en los tratamientos agudos (utilizando el fármaco excitotóxico N-metil-D-aspartato (NMDA) como ejemplo). Además, el artículo presenta imágenes conjuntas de Grx1-roGFP2 y el indicador de potencial de membrana mitocondrial, tetrametilrodamina, éster etílico (TMRE), para demostrar cómo Grx1-roGPF2 se puede multiplexar con indicadores adicionales para análisis multiparamétricos. Este protocolo proporciona una descripción detallada de cómo (i) optimizar la configuración del microscopio de barrido láser confocal, (ii) aplicar medicamentos para la estimulación seguido de la calibración del sensor con diamida y ditiotreitol, y (iii) analizar datos con ImageJ / FIJI.
Introducción
Varias enzimas mitocondriales importantes y moléculas de señalización están sujetas a la regulación tiol redox1. Además, las mitocondrias son una fuente celular importante de especies reactivas de oxígeno y son selectivamente vulnerables al daño oxidativo2. En consecuencia, el potencial redox mitocondrial afecta directamente a la bioenergética, la señalización celular, la función mitocondrial y, en última instancia, la viabilidad celular3,4. La matriz mitocondrial contiene altas cantidades (1-15 mM) del tampón tiol-disulfuro redox glutatión (GSH) para mantener la homeostasis redox y montar defensas antioxidantes5,6. El GSH se puede unir covalentemente a las proteínas diana (S-glutationilación) para controlar su estado y actividad redox y es utilizado por una variedad de enzimas desintoxicantes que reducen las proteínas oxidadas. Por lo tanto, el potencial redox de glutatión mitocondrial es un parámetro altamente informativo cuando se estudia la función mitocondrial y la fisiopatología.
roGFP2 es una variante de GFP que se ha hecho sensible a la redox mediante la adición de dos cisteínas expuestas a la superficie que forman un par artificial de ditiol-disulfuro7,8. Tiene un solo pico de emisión a ~510 nm y dos picos de excitación a ~400 nm y 490 nm. Es importante destacar que las amplitudes relativas de los dos picos de excitación dependen del estado redox de roGFP2 (Figura 1), lo que convierte a esta proteína en un sensor ratiométrico. En el sensor Grx1-roGFP2, la glutaredoxina-1 humana (Grx1) se ha fusionado con el extremo N de roGFP29,10. La unión covalente de la enzima Grx1 a roGFP2 ofrece dos mejoras importantes del sensor: hace que la respuesta del sensor sea específica para el par redox de glutatión GSH / GSSG (Figura 1), y acelera el equilibrio entre GSSG y roGFP2 en un factor de al menos 100,0009. Por lo tanto, Grx1-roGFP2 permite obtener imágenes específicas y dinámicas del potencial redox del glutatión celular.
Las imágenes Grx1-roGFP2 se pueden realizar en una amplia gama de microscopios, incluidos los microscopios de fluorescencia de campo amplio, los microscopios confocales de disco giratorio y los microscopios confocales de barrido láser. La expresión del sensor en las neuronas primarias se puede lograr mediante varios métodos que incluyen lipofección11, coprecipitación ADN/calcio-fosfato12, transferencia de genes mediada por virus o uso de animales transgénicos como fuente celular (Figura 2). Para los experimentos de este artículo se utilizaron virus adenoasociados recombinantes pseudotipados (rAAV) que contienen una proporción de 1:1 de proteínas de la cápside AAV1 y AAV2 13,14. Con este vector, la máxima expresión del sensor se alcanza típicamente 4-5 días después de la infección y se mantiene estable durante al menos dos semanas. Hemos utilizado con éxito Grx1-roGFP2 en neuronas primarias del hipocampo y cortical de ratones y ratas.
En este artículo, la expresión mediada por rAAV de Grx1-roGFP2 dirigida a mitocondrias en neuronas primarias del hipocampo y cortical de rata se utiliza para evaluar el estado redox de glutatión mitocondrial basal y su perturbación aguda. Se proporciona un protocolo para imágenes confocales en vivo con instrucciones detalladas sobre cómo (i) optimizar la configuración del microscopio confocal de escaneo láser, (ii) ejecutar un experimento de imágenes en vivo y (iii) analizar datos con FIJI.
Protocolo
Todos los experimentos con animales se ajustaron a las directrices nacionales e institucionales, incluida la Directiva 2010/63/UE del Consejo del Parlamento Europeo, y contaron con la aprobación ética completa del Ministerio del Interior (Oficina de Bienestar Animal de la Universidad de Heidelberg y Regierungspraesidium Karlsruhe, licencias T14/21 y T13/21). Las neuronas primarias del hipocampo y cortical se prepararon a partir de cachorros de ratón o rata recién nacidos de acuerdo con los procedimientos estándar y se mantuvieron durante 12-14 días como se describió anteriormente13.
1. Preparación de soluciones
- Soluciones de stock para búfer de imágenes
- Preparar cada solución madre según la Tabla 1 y mantenerlas a 4 °C. Para el almacenamiento a largo plazo (>3 meses), mantenga las alícuotas a -20 °C.
| Componente | MW | Concentración (M) | Importe (g) | Volumen (ml) |
| NaCl | 58.44 | 5 | 14.61 | 50 |
| Kcl | 74.55 | 3 | 1.12 | 5 |
| MgCl2· 6H2O | 203.3 | 1.9 | 2 | 5 |
| CaCl2·2H2O | 147.01 | 1 | 1.47 | 10 |
| Glicina | 75.07 | 0.1 | 0.375 | 50 |
| Sacarosa | 342.3 | 1.5 | 25.67 | 50 |
| Piruvato de sodio | 110.04 | 0.1 | 0.55 | 50 |
| HEPES | 238.3 | 1 | 11.9 | 50 |
| Glucosa | 180.15 | 2.5 | 45 | 100 |
Tabla 1: Soluciones de stock para el búfer de imágenes.
- Soluciones de stock de medicamentos y colorantes
- Disolver diamida (DA; utilizada para la calibración de una relación máxima de 405:488) en agua para obtener una solución madre de 0,5 M (por ejemplo, 1 g en 11,615 ml de agua). Alícuota y conservar a -20 °C.
- Disolver el ditiotreitol (TDT; utilizado para la calibración de una relación mínima de 405:488) en agua para obtener una solución madre de 1 M (por ejemplo, 5 g en 32.425 ml de agua). Alícuota y conservar a -20 °C durante un máximo de 3 meses.
- Disolver N-metil-D-aspartato (NMDA; utilizado para inducir excitotoxicidad y oxidación mitocondrial) en agua para obtener una solución madre de 10 mM (por ejemplo, 25 mg en 16.991 mL de agua). Conservar las alícuotas a -20 °C. Para el almacenamiento a largo plazo (>6 meses), mantenga las alícuotas a -80 °C.
- Perclorato de éster etílico de tetrametilrodamina (TMRE; un indicador de molécula pequeña del potencial de membrana mitocondrial)
- Disolver TMRE en polvo en metanol para obtener un stock de 20 mM (por ejemplo, 25 mg en 2.427 mL de metanol).
- Diluir el stock de 20 mM 1:1.000 en metanol para obtener un stock de 20 μM.
- Alícuota las soluciones madre de 20 mM y 20 μM, sella con parafilm y almacena protegida de la luz a -20 °C.
NOTA: Ambas soluciones de stock son estables durante varios años. Utilice la solución madre de 1.000x (20 μM) para experimentos.
- Búfer de imágenes
- Prepare 100 ml de tampón de imágenes agregando todos los componentes de la Tabla 2 a 80 ml de agua estéril en un cilindro de medición. Llevar el volumen hasta 100 ml con agua estéril. Mezclar agitando cuidadosamente el cilindro de medición hasta que la solución parezca homogénea.
NOTA: Se recomienda utilizar un osmómetro para comprobar la osmolaridad del búfer. Debe estar lo más cerca posible del medio de crecimiento de las células. Aquí, esto es 315 mOsmol / L. Aumente o disminuya la concentración de sacarosa según sea necesario para que coincida con la osmolaridad del tampón de imágenes y el medio de crecimiento. - Ajuste el pH a 7.4. Hacer alícuotas y mantenerlas a 4 °C durante un máximo de dos semanas. Para el almacenamiento a largo plazo, mantenga las alícuotas a -20 °C. Deje que el búfer de imágenes alcance la temperatura ambiente antes de su uso.
- Prepare 100 ml de tampón de imágenes agregando todos los componentes de la Tabla 2 a 80 ml de agua estéril en un cilindro de medición. Llevar el volumen hasta 100 ml con agua estéril. Mezclar agitando cuidadosamente el cilindro de medición hasta que la solución parezca homogénea.
| Componente | Solución de stock (M) | Concentración final (mM) | Volumen (ml) |
| NaCl | 5 | 114 | 2.3 |
| Kcl | 3 | 5.29 | 0.176 |
| MgCl2 | 1.9 | 1 | 0.053 |
| CaCl2 | 1 | 2 | 0.2 |
| Glicina | 0.1 | 0.005 | 0.005 |
| Sacarosa | 1.5 | 52 | 3.5 |
| Piruvato de sodio | 0.1 | 0.5 | 0.5 |
| HEPES | 1 | 10 | 1 |
| Glucosa | 2.5 | 5 | 0.2 |
Tabla 2: Composición del búfer de imágenes. Los volúmenes indicados se utilizan para la preparación de 100 ml de tampón de imagen.
- Soluciones para estimulación y calibración
NOTA: Siempre prepare soluciones de estimulación frescas agregando soluciones de stock de medicamentos indicados al búfer de imágenes justo antes del experimento. Las soluciones para la estimulación y la calibración se añadirán a la cámara de imágenes secuencialmente durante un experimento (ver secciones 3-5). Dependiendo del tipo de experimento, se requieren diferentes soluciones para alcanzar la misma concentración final en el volumen final respectivo en la cámara de imágenes.- Preparar 3x solución NMDA (90 μM; concentración final en la cámara: 30 μM) añadiendo 63 μL de un stock NMDA de 10 mM a 6.937 mL de tampón de imagen. Añadir 500 μL de la solución resultante a la cámara (volumen final: 1,5 mL).
- Prepare 2x solución de DA para los pasos 3 y 4 (1 mM; concentración final en la cámara: 0,5 mM) agregando 14 μL de un stock de DA de 0,5 M a 6,986 mL de tampón de imagen. Añadir 1 mL a la cámara (volumen final: 2 mL).
- Prepare la solución 4x DA para el paso 5 (2 mM; concentración final en la cámara: 0.5 mM) agregando 28 μL de un stock de DA de 0.5 M a 6.972 mL de tampón de imagen. Añadir 500 μL a la cámara (volumen final: 2 mL).
- Preparar 1x solución de TDT (5 mM; concentración final en la cámara: 5 mM) añadiendo 45 μL de material de TDT de 1 M a 8955 μL de tampón de imagen. Agregue 1 ml de esta solución a la cámara después de aspirar el tampón de imagen (volumen final: 1 ml).
2. Carga de células con TMRE
NOTA: En este protocolo, TMRE se utiliza en modo no quench15 a una concentración final de 20 nM. En general, se debe utilizar la concentración más baja posible de TMRE que aún proporcione suficiente intensidad de señal en el microscopio de elección. Debido a la evaporación desigual, el volumen de medio en diferentes pozos puede diferir en cultivos primarios a largo plazo. Para garantizar una concentración constante de TMRE en todos los pozos, no agregue TMRE directamente a los pozos. En su lugar, reemplace el medio en cada pozo con la misma cantidad de medio que contiene TMRE. El siguiente protocolo está diseñado para neuronas primarias en placas de 24 pocillos que contienen ~ 1 ml de medio por pozo.
- Trabajando en una campana de flujo laminar de cultivo de tejidos, recolecte 500 μL de medio de cada pozo en un solo tubo cónico.
- Por pozo, agregue 0,5 μL de 20 μM de TMRE en el tubo cónico (por ejemplo, 12 μL para 24 pocillos).
- Aspire cuidadosamente el medio restante del primer pozo y reemplácelo con 500 μL de medio que contenga TMRE. Continuar, pozo a pozo, con los pozos restantes.
NOTA: Tenga cuidado de no dejar que las células se sequen y no molestar a las células. - Devuelva las células a la incubadora y espere al menos 60 minutos para el equilibrio del tinte.
NOTA: El tiempo de carga se puede extender a varias horas sin efectos adversos. - Para garantizar concentraciones consistentes de TMRE y equilibrio a lo largo del experimento de imágenes, asegúrese de incluir una concentración final de 20 nM TMRE en el tampón de imágenes y todas las soluciones de estimulación.
3. Optimización de la configuración del microscopio confocal de barrido
NOTA: Este paso tiene como objetivo encontrar el mejor compromiso entre la calidad de imagen y la viabilidad celular durante las imágenes en vivo. En esta sección se describe la optimización de la configuración de las imágenes roGFP. Si se realizan imágenes multiparamétricas, se debe realizar una optimización similar, incluida la verificación de una línea de base estable sin signos de blanqueamiento o fototoxicidad, para los indicadores adicionales.
- Inicie el microscopio confocal y la configuración estándar de carga para imágenes GFP (excitación de 488 nm, emisión de 505 a 550 nm).
- Ajuste el detector a 12 bits o 16 bits.
NOTA: Por lo general, 8 bits no son suficientes para obtener imágenes cuantitativas. - Active el modo de escaneo secuencial y agregue una segunda secuencia/pista (excitación de 405 nm, emisión de 505 a 550 nm).
- Para ambos canales, seleccione una tabla de búsqueda pseudocolor que indique píxeles sobreexpuestos y subexpuestos (por ejemplo, GLOW OU).
- Seleccione un objetivo que sea adecuado para el objeto de interés.
NOTA: 10x-40x son adecuados para el análisis de una sola célula, 63x-100x son adecuados para el análisis de una sola mitocondria. - Monte un cobertor con células en la cámara de imágenes, agregue 1 ml de tampón de imágenes y coloque la cámara en el microscopio.
- Use el ocular y la luz transmitida para enfocar las células.
NOTA: No utilice luz de epifluorescencia para localizar y enfocar las células. Incluso a baja potencia, esto afectará negativamente a las células. - Graba imágenes con diferentes formatos de píxeles. En función de estas imágenes, seleccione el número de píxeles más bajo que proporcione una resolución aceptable de la estructura de interés.
NOTA: Por lo general, los píxeles de 512 x 512 funcionan bien para imágenes de una sola célula con objetivos de 20x y 40x, y los píxeles de 1024 x 1024 o 2048 x 2048 suelen funcionar bien para imágenes de una sola mitocondria con un objetivo de 63x. - Graba imágenes con diferentes tamaños de agujeros de alfiler. En función de estas imágenes, seleccione el tamaño de agujero de alfiler más grande que proporcione una resolución aceptable de la estructura de interés.
NOTA: Por lo general, 3-7 unidades aireadas funcionan bien. - Graba imágenes con diferentes intensidades láser.
- Ajuste la ganancia y el umbral del detector en consecuencia. En función de estas imágenes, seleccione la intensidad láser más baja que proporcione una intensidad de señal aceptable y una relación señal-fondo.
- Para determinar la relación señal-fondo, mida la intensidad de la señal en una región de interés (ROI) que contenga células o mitocondrias (ROI1) y en un ROI sin células o mitocondrias (ROI2). Luego, divida la intensidad de ROI1 por la intensidad de ROI2.
NOTA: Apunte a una relación señal-fondo de >3 e intensidades de señal de ROI individuales de 200-1,000 para excitación de 405 nm con potencia láser de 1-3% e intensidades de ROI individuales de 300-1,500 para excitación de 488 nm con potencia láser del 1%.
- Para determinar la relación señal-fondo, mida la intensidad de la señal en una región de interés (ROI) que contenga células o mitocondrias (ROI1) y en un ROI sin células o mitocondrias (ROI2). Luego, divida la intensidad de ROI1 por la intensidad de ROI2.
- Ajuste la ganancia y el umbral del detector en consecuencia. En función de estas imágenes, seleccione la intensidad láser más baja que proporcione una intensidad de señal aceptable y una relación señal-fondo.
- Grabe imágenes con diferentes velocidades de escaneo y número de promedios de fotogramas. Grabe 4-5 imágenes para cada combinación de configuraciones. En función de estas series de imágenes, seleccione la velocidad más alta y la configuración media más baja que proporcionen un ruido de imagen aceptable y una variabilidad de imagen a imagen.
NOTA: Una velocidad de escaneo de 600 Hz y 1-2 cuadros para promediar funcionan bien en la mayoría de los casos. - Con un nuevo coverslip, grabe una serie de lapso de tiempo con la configuración optimizada.
NOTA: La duración y el intervalo de imagen de la serie deben parecerse a los de los experimentos planificados. - Al final de la serie de lapso de tiempo, agregue 1 ml de solución 2x DA a la cámara de grabación. Imagen para 2 min adicionales.
- Aspire el tampón de imágenes con una bomba peristáltica o una pipeta de mano. Añadir 1 ml de solución de TDT 1x. Imagen para 5 min adicionales.
- Analizar el experimento de lapso de tiempo (ver sección 5).
- Verifique que ninguno de los dos canales se sobreexponga o subexponga durante el tratamiento de DA y TDT con la configuración optimizada.
- Asegúrese de que ninguno de los dos canales muestre un blanqueamiento considerable durante la grabación de lapso de tiempo; aspirar a una pérdida de intensidad del <2% entre la primera y la última imagen.
- Compruebe que la relación 405:488 no cambie considerablemente durante la toma de imágenes.
- Repita todo el procedimiento de manera iterativa, utilizando varias cubiertas, hasta que se hayan definido los ajustes que proporcionan resultados aceptables de manera consistente.
4. Evaluación del estado redox basal
- Inicie el microscopio y cargue los ajustes optimizados de la sección 3.
- Establezca el promedio de fotogramas en 3-5.
- Monte un cobertor con células en la cámara de imágenes, agregue 1 ml de tampón de imágenes y coloque la cámara en el microscopio.
- Use el ocular y la luz transmitida para enfocar las células.
NOTA: No utilice luz de epifluorescencia para localizar y enfocar las células. Incluso a baja potencia, esto afectará negativamente a las células. - Cambie al modo de escaneo y use el canal de 488 nm en vista en vivo para enfocar y localizar celdas para obtener imágenes.
- Utilice la función multipunto para seleccionar de 3 a 5 campos de visión en el escueto.
- Grabar una imagen de línea base.
- Agregue 1 ml de solución 2x DA a la cámara.
- Después de 1, 2 y 3 minutos, use la vista en vivo para confirmar / ajustar el enfoque y luego grabar una imagen.
NOTA: Las células generalmente se oxidan completamente después de 2 min. - Reemplace el búfer en la cámara de imágenes con 1 ml de solución de TDT 1x.
- Después de 3 y 5 minutos, use la vista en vivo para confirmar / ajustar el enfoque y luego grabar una imagen.
NOTA: Las celdas generalmente se reducen por completo después de 4-5 min.
5. Imágenes en vivo de tratamientos agudos
NOTA: El protocolo a continuación describe imágenes de la respuesta redox mitocondrial al tratamiento con NMDA. Es posible que sea necesario ajustar los intervalos de imagen y la duración del experimento para otros tratamientos.
- Inicie el microscopio y cargue los ajustes optimizados de la sección 3.
- Establezca el intervalo de lapso de tiempo en 30 s y la duración en 25 min.
- Monte un cobertor con células en la cámara de imágenes, agregue 1 ml de tampón de imágenes y coloque la cámara en el microscopio.
NOTA: Para evitar la deriva del enfoque térmico, deje las células en la etapa del microscopio durante 10-15 minutos antes de comenzar las imágenes de lapso de tiempo. - Use el ocular y la luz transmitida para enfocar las células.
NOTA: No utilice luz de epifluorescencia para localizar y enfocar las células. Incluso a baja potencia, esto afectará negativamente a las células. - Cambie al modo de escaneo y utilice el canal de 488 nm en vista real para enfocar y localizar celdas para obtener imágenes.
- Opcional: Para aumentar el número de celdas grabadas por ejecución, utilice la función multipunto para obtener imágenes de 2-3 campos de visión por recubierto.
- Inicie la adquisición de lapso de tiempo y grabe 5 imágenes como grabación de línea de base de 2 minutos.
- Agregue 500 μL de solución 3x NMDA a la cámara (concentración final 30 μM) y registre 20 imágenes adicionales como una respuesta NMDA de 10 minutos.
NOTA: Las neuronas son muy sensibles a los cambios en la osmolaridad. Por lo tanto, asegúrese de minimizar la evaporación del búfer de imágenes. Para tratamientos más largos, la cámara de imágenes debe cubrirse con una tapa. - Agregue 500 μL de solución 4x DA a la cámara y grabe 6 imágenes más (calibración máxima de 3 minutos).
- Aspire el tampón de la cámara de imagen y reemplácelo con 1 ml de solución de TDT 1x. Graba 10 imágenes más (calibración mínima de 5 min).
- Finalice la grabación y guarde la serie de imágenes.
6. Análisis de datos
- Importación de datos y preprocesamiento de imágenes en FIJI
- Utilice el Importador de bioformatos para abrir un grupo de imágenes del paso 4 o un archivo de imagen del paso 5. Haga clic en plugins | | de bioformatos Importador de Bioformatos. En el cuadro de diálogo, utilice Ver pila con: Hyperstack, establezca Modo de color: predeterminado, seleccione Escala automática y no se divida en ventanas separadas.
NOTA: Autoscale optimiza la visualización de los datos en la pantalla del ordenador. No cambia las intensidades de píxeles. - Si se abrieron imágenes individuales del paso 4, haga clic en Imagen | Pilas | Herramientas | Concatenar para fusionarlos en una pila de una sola imagen.
- Si hay XY-drift durante la serie de imágenes, haga clic en Plugins | StackReg para registrar las imágenes. En el cuadro de diálogo, seleccione Cuerpo rígido o Traducción.
- Cambie el formato de la imagen a 32 bits haciendo clic en imagen | Tipo | 32 bits.
- Divida los canales de color en ventanas separadas haciendo clic en Imagen | | de color Canales divididos.
- Seleccione el canal 1 (405 nm) y ajuste el umbral para seleccionar las mitocondrias para el análisis haciendo clic en Imagen | Ajustar | Umbral. En el cuadro de diálogo, seleccione Predeterminado, Rojo, Fondo oscuro e Histograma de pila y espere a que los píxeles seleccionados aparezcan en rojo. Haga clic en Aplicar. Seleccione Establecer píxeles de fondo en NaN y Procesar todas las imágenes.
NOTA: Para evitar posibles sesgos del observador, se debe utilizar la determinación automatizada del umbral. FIJI ofrece varios métodos automatizados (como Predeterminado, Huang, Intermodes, Otsu) que se pueden seleccionar desde un menú desplegable en el cuadro de diálogo umbral. Normalmente, el método Default da un buen resultado. Se recomienda comparar varios métodos durante el primer análisis para encontrar el mejor método de umbral para el conjunto dado de imágenes. Una vez que se ha elegido un método, debe aplicarse a todas las imágenes. - Repita el paso 6.1.6 para el canal 2 (488 nm).
- Cree una imagen de proporción para visualizar la relación de 405:488 nm haciendo clic en Procesar | Calculadora de imágenes. En el cuadro de diálogo, seleccione Imagen 1: canal 1, Operación: Dividir, Imagen 2: canal 2, Crear nueva ventana, Procesar todas las imágenes.
- Cambie la tabla de búsqueda de la imagen de relación a pseudocolor. Por ejemplo, para cambiar a Fire, haga clic en imagen | Tablas de búsqueda | Incendio.
- Utilice el Importador de bioformatos para abrir un grupo de imágenes del paso 4 o un archivo de imagen del paso 5. Haga clic en plugins | | de bioformatos Importador de Bioformatos. En el cuadro de diálogo, utilice Ver pila con: Hyperstack, establezca Modo de color: predeterminado, seleccione Escala automática y no se divida en ventanas separadas.
- Análisis de imágenes
- En la imagen de la proporción, dibuje ROI alrededor de células individuales o mitocondrias. Después de dibujar cada ROI, agréguelo al Administrador de ROI. Analizar | Herramientas | | de ROI Manager Añadir. (método abreviado de teclado: 'T') Seleccione Mostrar todo.
NOTA: Dado que los píxeles de fondo se han establecido en 'no es un número' (NaN) en los pasos 6.1.6 y 6.1.7, no afectarán al resultado de la medición. Por lo tanto, es aceptable incluir algunos píxeles de fondo en el ROI. - Mida las proporciones 405:488 de celdas individuales haciendo clic en ROI Manager | ctrl + A para seleccionar todos los ROI | Más | Multimedida. En el cuadro de diálogo, seleccione Medir todos los sectores y Una fila por sector.
- Exporte las mediciones al software de hoja de cálculo.
- Seleccione la imagen de 405 nm. Mida las intensidades de todos los ROI como en el paso 6.2.2. utilizando los ROI que se almacenan en el gestor de ROI.
- Exporte las mediciones al software de hoja de cálculo.
- Seleccione la imagen de 488 nm. Mida las intensidades de todos los ROI como en el paso 6.2.2. utilizando los ROI que se almacenan en el gestor de ROI.
- Exporte las mediciones al software de hoja de cálculo.
- Guarde los ROI para futuras referencias haciendo clic en ROI Manager | ctrl + A para seleccionar todos los ROI | Más | Guardar.
- Recomendado: Generar gráficos de intensidad frente a tiempo de las trazas de 405 y 488 nm. Verifique que no haya un blanqueamiento marcado en ninguno de los canales (la intensidad de la señal al final de la serie de imágenes debe ser ≥98% de la primera imagen) y que las dos trazas se muevan en direcciones opuestas durante las respuestas del sensor (por ejemplo, la traza de 405 nm debe aumentar durante la oxidación, mientras que la traza de 488 nm debe disminuir).
- En la imagen de la proporción, dibuje ROI alrededor de células individuales o mitocondrias. Después de dibujar cada ROI, agréguelo al Administrador de ROI. Analizar | Herramientas | | de ROI Manager Añadir. (método abreviado de teclado: 'T') Seleccione Mostrar todo.
- Normalización de datos
- Para cada ROI de la imagen de relación, determine el valor máximo durante el tratamiento con DA (Rmax) y el valor mínimo durante el tratamiento de TDT (Rmin).
- Calcule la relación normalizada de la siguiente manera:
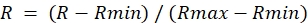
NOTA: Esto establecerá la relación máxima en 1.0 y la relación mínima en 0.
- Análisis de la morfología mitocondrial
- Para obtener mediciones de la morfología mitocondrial en paralelo a las intensidades roGFP en el paso 6.2.6, vaya a Analizar | Establezca Medidas y compruebe Descriptores de forma y Elipse de ajuste.
NOTA: Además de la intensidad media, las mediciones en la ventana de resultados incluirán la longitud del eje mayor (Mayor), la longitud del eje menor (Menor), la relación de aspecto (AR; eje mayor dividido por eje menor; las mitocondrias redondas tienen un AR ~ 1, las mitocondrias alargadas tienen una AR mayor), así como las mediciones de circularidad (Circ.) y redondez (Redondo).
- Para obtener mediciones de la morfología mitocondrial en paralelo a las intensidades roGFP en el paso 6.2.6, vaya a Analizar | Establezca Medidas y compruebe Descriptores de forma y Elipse de ajuste.
Resultados
Cuantificación de las diferencias en el estado redox mitocondrial en estado estacionario después de la retirada del factor de crecimiento
Para demostrar la cuantificación de las diferencias de estado estacionario en el estado redox mitocondrial, las neuronas primarias cultivadas en medio estándar se compararon con las neuronas cultivadas sin factores de crecimiento durante 48 horas antes de la obtención de imágenes. La abstinencia del factor de crecimiento resulta en la muerte celular neuronal a...
Discusión
Las mediciones cuantitativas y dinámicas del estado redox mitocondrial proporcionan información importante sobre la fisiología mitocondrial y celular. Hay varias sondas químicas fluorógenas disponibles que detectan especies reactivas de oxígeno, "estrés redox" o "estrés oxidativo". Sin embargo, estos últimos términos no están bien definidos y a menudo carecen de especificidad9,17,18. En comparación con los colorantes...
Divulgaciones
Los autores declaran que no tienen conflicto de intereses.
Agradecimientos
Este trabajo fue apoyado por la Deutsche Forschungsgemeinschaft (BA 3679/5-1; PARA 2289: BA 3679/4-2). A.K. cuenta con el apoyo de una beca ERASMUS+. Agradecemos a Iris Bünzli-Ehret, Rita Rosner y Andrea Schlicksupp por la preparación de las neuronas primarias. Agradecemos al Dr. Tobias Dick por proporcionar pLPCX-mito-Grx1-roGFP2. Los experimentos mostrados en la Figura 4 se realizaron en el Nikon Imaging Center de la Universidad de Heidelberg. La figura 2 se preparó con BioRender.com.
Materiales
| Name | Company | Catalog Number | Comments |
| reagents | |||
| Calcium chloride (CaCl2·2H2O) | Sigma-Aldrich | C3306 | |
| Diamide (DA) | Sigma-Aldrich | D3648 | |
| Dithiothreitol (DTT) | Carl Roth GmbH | 6908.1 | |
| Glucose (2.5 M stock solution) | Sigma-Aldrich | G8769 | |
| Glucose | Sigma-Aldrich | G7528 | |
| Glycine | neoFroxx GmbH | LC-4522.2 | |
| HEPES (1 M stock solution) | Sigma-Aldrich | 15630-080 | |
| HEPES | Sigma-Aldrich | H4034 | |
| Magnesium chloride (MgCl2·6H2O) | Sigma-Aldrich | 442611-M | |
| N-methyl-D-aspartate (NMDA) | Sigma-Aldrich | M3262 | |
| Potassium chloride (KCl) | Sigma-Aldrich | P3911 | |
| Sodium chloride (NaCl) | neoFroxx GmbH | LC-5932.1 | |
| Sodium pyruvate (0.1 M stock solution) | Sigma-Aldrich | S8636 | |
| Sodium pyruvate | Sigma-Aldrich | P8574 | |
| Sucrose | Carl Roth GmbH | 4621.1 | |
| Tetramethylrhodamine ethyl ester perchlorate (TMRE) | Sigma-Aldrich | 87917 | |
| equipment | |||
| imaging chamber | Life Imaging Services (Basel, Switzerland) | 10920 | Ludin Chamber Type 3 for Ø12mm coverslips |
| laser scanning confocal microscope, microscope | Leica | DMI6000 | |
| laser scanning confocal microscope, scanning unit | Leica | SP8 | |
| peristaltic pump | VWR | PP1080 181-4001 | |
| spinning disc confocal microscope, camera | Hamamatsu | C9100-02 EMCCD | |
| spinning disc confocal microscope, incubationsystem | TokaiHit | INU-ZILCF-F1 | |
| spinning disc confocal microscope, microscope | Nikon | Ti microscope | |
| spinning disc confocal microscope, scanning unit | Yokagawa | CSU-X1 | |
| software | |||
| FIJI | https://fiji.sc | ||
| StackReg plugin | https://github.com/fiji-BIG/StackReg/blob/master/src/main/java/StackReg_.java | ||
| TurboReg plugin | https://github.com/fiji-BIG/TurboReg/blob/master/src/main/java/TurboReg_.java |
Referencias
- Roede, J. R., Go, Y. M., Jones, D. P. Redox equivalents and mitochondrial bioenergetics. Methods in Molecular Biology. 810, 249-280 (2012).
- Turrens, J. F. Mitochondrial formation of reactive oxygen species. Journal of Physiology. 552, 335-344 (2003).
- Lin, M. T., Beal, M. F. Mitochondrial dysfunction and oxidative stress in neurodegenerative diseases. Nature. 443 (7113), 787-795 (2006).
- Manfredi, G., Beal, M. F. The role of mitochondria in the pathogenesis of neurodegenerative diseases. Brain Pathology. 10 (3), 462-472 (2000).
- Mari, M., Morales, A., Colell, A., Garcia-Ruiz, C., Fernandez-Checa, J. C. Mitochondrial glutathione, a key survival antioxidant. Antioxidants & Redox Signaling. 11 (11), 2685-2700 (2009).
- Murphy, M. P. Mitochondrial thiols in antioxidant protection and redox signaling: distinct roles for glutathionylation and other thiol modifications. Antioxidants & Redox Signaling. 16 (6), 476-495 (2012).
- Dooley, C. T., et al. Imaging dynamic redox changes in mammalian cells with green fluorescent protein indicators. Journal of Biological Chemistry. 279 (21), 22284-22293 (2004).
- Hanson, G. T., et al. Investigating mitochondrial redox potential with redox-sensitive green fluorescent protein indicators. Journal of Biological Chemistry. 279 (13), 13044-13053 (2004).
- Gutscher, M., et al. Real-time imaging of the intracellular glutathione redox potential. Nature Methods. 5 (6), 553-559 (2008).
- Morgan, B., Sobotta, M. C., Dick, T. P. Measuring E(GSH) and H2O2 with roGFP2-based redox probes. Free Radical Biology & Medicine. 51 (11), 1943-1951 (2011).
- Marwick, K. F. M., Hardingham, G. E. Transfection in primary cultured neuronal cells. Methods in Molecular Biology. 1677, 137-144 (2017).
- Kohrmann, M., et al. convenient, and effective method to transiently transfect primary hippocampal neurons. Journal of Neuroscience Research. 58 (6), 831-835 (1999).
- Depp, C., Bas-Orth, C., Schroeder, L., Hellwig, A., Bading, H. Synaptic activity protects neurons against calcium-mediated oxidation and contraction of mitochondria during excitotoxicity. Antioxidants & Redox Signaling. 29 (12), 1109-1124 (2018).
- Hauck, B., Chen, L., Xiao, W. Generation and characterization of chimeric recombinant AAV vectors. Molecular Therapy. 7 (3), 419-425 (2003).
- Brand, M. D., Nicholls, D. G. Assessing mitochondrial dysfunction in cells. Biochemical Journal. 435 (2), 297-312 (2011).
- Zhang, S. J., et al. Nuclear calcium signaling controls expression of a large gene pool: identification of a gene program for acquired neuroprotection induced by synaptic activity. PLoS Genetics. 5 (8), 1000604 (2009).
- Winterbourn, C. C. The challenges of using fluorescent probes to detect and quantify specific reactive oxygen species in living cells. Biochimica et Biophysica Acta. 1840 (2), 730-738 (2014).
- Sies, H. Oxidative stress: a concept in redox biology and medicine. Redox Biology. 4, 180-183 (2015).
- Lukyanov, K. A., Belousov, V. V. Genetically encoded fluorescent redox sensors. Biochimica et Biophysica Acta. 1840 (2), 745-756 (2014).
- Nietzel, T., et al. Redox-mediated kick-start of mitochondrial energy metabolism drives resource-efficient seed germination. Proceedings of the National Academy of Sciences of the United States of America. 117 (1), 741-751 (2020).
- Albrecht, S. C., et al. Redesign of genetically encoded biosensors for monitoring mitochondrial redox status in a broad range of model eukaryotes. Journal of Biomolecular Screening. 19 (3), 379-386 (2014).
- Albrecht, S. C., Barata, A. G., Grosshans, J., Teleman, A. A., Dick, T. P. In vivo mapping of hydrogen peroxide and oxidized glutathione reveals chemical and regional specificity of redox homeostasis. Cell Metabolism. 14 (6), 819-829 (2011).
- Breckwoldt, M. O., et al. Multiparametric optical analysis of mitochondrial redox signals during neuronal physiology and pathology in vivo. Nature Medicine. 20 (5), 555-560 (2014).
- Ricke, K. M., et al. Mitochondrial dysfunction combined with high calcium load leads to impaired antioxidant defense underlying the selective loss of nigral dopaminergic neurons. Journal of Neuroscience. 40 (9), 1975-1986 (2020).
- Bjornberg, O., Ostergaard, H., Winther, J. R. Mechanistic insight provided by glutaredoxin within a fusion to redox-sensitive yellow fluorescent protein. Biochemistry. 45 (7), 2362-2371 (2006).
- Shokhina, A. G., et al. Red fluorescent redox-sensitive biosensor Grx1-roCherry. Redox Biology. 21, 101071 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados