Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Imagerie en direct de l’état redox du glutathion mitochondrial dans les neurones primaires à l’aide d’un indicateur ratiométrique
Dans cet article
Résumé
Cet article décrit un protocole pour déterminer les différences dans l’état redox basal et les réponses redox aux perturbations aiguës dans les neurones primaires de l’hippocampe et cortical en utilisant la microscopie vivante confocale. Le protocole peut être appliqué à d’autres types de cellules et de microscopes avec un minimum de modifications.
Résumé
L’homéostasie redox mitochondriale est importante pour la viabilité et la fonction neuronales. Bien que les mitochondries contiennent plusieurs systèmes redox, le glutathion tampon redox thiol-disulfure très abondant est considéré comme un acteur central dans les défenses antioxydantes. Par conséquent, la mesure du potentiel redox du glutathion mitochondrial fournit des informations utiles sur le statut redox mitochondrial et le stress oxydatif. Glutaredoxin1-roGFP2 (Grx1-roGFP2) est un indicateur ratiométrique à base de protéine fluorescente verte (GFP) génétiquement codé du potentiel redox du glutathion qui a deux pics d’excitation sensibles à l’état redox à 400 nm et 490 nm avec un seul pic d’émission à 510 nm. Cet article explique comment effectuer une microscopie confocale vivante de Grx1-roGFP2 ciblant les mitochondries dans les neurones primaires de l’hippocampe et du cortical. Il décrit comment évaluer le potentiel redox du glutathion mitochondrial à l’état d’équilibre (par exemple, pour comparer les états pathologiques ou les traitements à long terme) et comment mesurer les changements redox lors des traitements aigus (en utilisant le médicament excitotoxique N-méthyl-D-aspartate (NMDA) à titre d’exemple). En outre, l’article présente la co-imagerie de Grx1-roGFP2 et de l’indicateur de potentiel de la membrane mitochondriale, la tétraméthylrhodamine, ester éthylique (TMRE), pour démontrer comment Grx1-roGPF2 peut être multiplexé avec des indicateurs supplémentaires pour les analyses multiparamétriques. Ce protocole fournit une description détaillée de la façon de (i) optimiser les réglages du microscope confocal à balayage laser, (ii) appliquer des médicaments pour la stimulation suivis d’un étalonnage du capteur avec du diamide et du dithiothréitol, et (iii) analyser les données avec ImageJ / FIJI.
Introduction
Plusieurs enzymes mitochondriales et molécules de signalisation importantes sont soumises à la régulation redox thiol1. De plus, les mitochondries sont une source cellulaire majeure d’espèces réactives de l’oxygène et sont sélectivement vulnérables aux dommages oxydatifs2. En conséquence, le potentiel redox mitochondrial affecte directement la bioénergétique, la signalisation cellulaire, la fonction mitochondriale et, en fin de compte, la viabilité cellulaire3,4. La matrice mitochondriale contient de grandes quantités (1-15 mM) de glutathion tampon redox thiol-disulfure (GSH) pour maintenir l’homéostasie redox et monter des défenses antioxydantes5,6. Le GSH peut être attaché de manière covalente aux protéines cibles (S-glutathionylation) pour contrôler leur statut et leur activité redox et est utilisé par une gamme d’enzymes détoxifiantes qui réduisent les protéines oxydées. Par conséquent, le potentiel redox du glutathion mitochondrial est un paramètre très informatif lors de l’étude de la fonction mitochondriale et de la physiopathologie.
roGFP2 est une variante de GFP qui a été rendue redox-sensible par l’ajout de deux cystéines exposées à la surface qui forment une paire artificielle dithiol-disulfure7,8. Il a un seul pic d’émission à ~510 nm et deux pics d’excitation à ~400 nm et 490 nm. Il est important de noter que les amplitudes relatives des deux pics d’excitation dépendent de l’état redox de roGFP2 (Figure 1), ce qui fait de cette protéine un capteur ratiométrique. Dans le capteur Grx1-roGFP2, la glutarédoxine-1 humaine (Grx1) a été fusionnée à la terminaison N de roGFP29,10. La fixation covalente de l’enzyme Grx1 à roGFP2 offre deux améliorations majeures du capteur : elle rend la réponse du capteur spécifique pour la paire redox GSH/GSSG glutathion (Figure 1), et elle accélère l’équilibrage entre GSSG et roGFP2 d’un facteur d’au moins 100 0009. Par conséquent, Grx1-roGFP2 permet une imagerie spécifique et dynamique du potentiel redox cellulaire du glutathion.
L’imagerie Grx1-roGFP2 peut être réalisée sur une large gamme de microscopes, y compris les microscopes à fluorescence à grand champ, les microscopes confocaux à disque rotatif et les microscopes confocaux à balayage laser. L’expression du capteur dans les neurones primaires peut être obtenue par diverses méthodes, notamment la lipofection11, la coprécipitation ADN/calcium-phosphate12, le transfert de gènes médié par le virus ou l’utilisation d’animaux transgéniques comme source cellulaire (Figure 2). Des virus adéno-associés recombinants pseudotypés (rAAV) contenant un rapport de 1:1 de protéines de capside AAV1 et AAV2 13,14 ont été utilisés pour les expériences de cet article. Avec ce vecteur, l’expression maximale du capteur est généralement atteinte 4 à 5 jours après l’infection et reste stable pendant au moins deux semaines. Nous avons utilisé avec succès Grx1-roGFP2 dans les neurones primaires de l’hippocampe et du cortical de souris et de rats.
Dans cet article, l’expression médiée par le rAAV de Grx1-roGFP2 ciblé par les mitochondries dans les neurones hippocampiques et corticaux primaires du rat est utilisée pour évaluer l’état redox du glutathion mitochondrial basal et sa perturbation aiguë. Un protocole est fourni pour l’imagerie confocale en direct avec des instructions détaillées sur la façon (i) d’optimiser les paramètres du microscope confocal à balayage laser, (ii) d’exécuter une expérience d’imagerie en direct et (iii) d’analyser les données avec FIJI.
Protocole
Toutes les expériences sur les animaux étaient conformes aux lignes directrices nationales et institutionnelles, y compris la directive 2010/63/UE du Conseil du Parlement européen, et avaient l’approbation éthique complète du ministère de l’Intérieur (Office du bien-être animal de l’Université de Heidelberg et Regierungspraesidium Karlsruhe, licences T14/21 et T13/21). Les neurones primaires de l’hippocampe et du cortical ont été préparés à partir de nouveau-nés de souris ou de ratons selon les procédures standard et ont été maintenus pendant 12 à 14 jours, comme décrit précédemment13.
1. Préparation des solutions
- Solutions de stock pour tampon d’imagerie
- Préparer chaque solution mère conformément au tableau 1 et les conserver à 4 °C. Pour le stockage à long terme (>3 mois), conserver les aliquotes à -20 °C.
| Composant | MW | Concentration (M) | Montant (g) | Volume (mL) |
| NaCl | 58.44 | 5 | 14.61 | 50 |
| Kcl | 74.55 | 3 | 1.12 | 5 |
| MgCl2· 6H2O | 203.3 | 1.9 | 2 | 5 |
| CaCl2·2H2O | 147.01 | 1 | 1.47 | 10 |
| Glycine | 75.07 | 0.1 | 0.375 | 50 |
| Saccharose | 342.3 | 1.5 | 25.67 | 50 |
| Pyruvate de sodium | 110.04 | 0.1 | 0.55 | 50 |
| HEPES | 238.3 | 1 | 11.9 | 50 |
| Glucose | 180.15 | 2.5 | 45 | 100 |
Tableau 1 : Solutions de base pour tampon d’imagerie.
- Solutions mères de médicaments et de colorants
- Dissoudre le diamide (DA; utilisé pour l’étalonnage du rapport maximal 405:488) dans l’eau pour obtenir une solution mère de 0,5 M (par exemple, 1 g dans 11,615 mL d’eau). Aliquote et conserver à -20 °C.
- Dissoudre le dithiothréitol (TNT; utilisé pour l’étalonnage du rapport minimal 405:488) dans l’eau pour obtenir une solution mère de 1 M (par exemple, 5 g dans 32,425 mL d’eau). Aliquote et conserver à -20 °C pendant un maximum de 3 mois.
- Dissoudre le N-méthyl-D-aspartate (NMDA; utilisé pour induire l’excitotoxicité et l’oxydation mitochondriale) dans l’eau pour obtenir une solution mère de 10 mM (par exemple, 25 mg dans 16,991 mL d’eau). Conserver les aliquotes à -20 °C. Pour le stockage à long terme (>6 mois), maintenir les aliquotes à -80 °C.
- Perchlorate d’ester éthylique de tétraméthylrhodamine (TMRE; indicateur à petite molécule du potentiel de la membrane mitochondriale)
- Dissoudre la poudre de TMRE dans du méthanol pour obtenir un stock de 20 mM (p. ex., 25 mg dans 2,427 mL de méthanol).
- Diluer le stock de 20 mM 1:1 000 dans du méthanol pour obtenir un stock de 20 μM.
- Aliquotez les solutions mères de 20 mM et 20 μM, scellez avec un parafilm et stockez-les à l’abri de la lumière à -20 °C.
REMARQUE: Les deux solutions d’origine sont stables depuis plusieurs années. Utilisez la solution mère 1 000x (20 μM) pour les expériences.
- Tampon d’imagerie
- Préparer 100 mL de tampon d’imagerie en ajoutant tous les composants du tableau 2 à 80 mL d’eau stérile dans un cylindre de mesure. Porter le volume jusqu’à 100 mL avec de l’eau stérile. Mélanger en agitant soigneusement le cylindre de mesure jusqu’à ce que la solution paraisse homogène.
REMARQUE: Il est recommandé d’utiliser un osmomètre pour vérifier l’osmolarité du tampon. Il doit être aussi proche que possible du milieu de croissance des cellules. Ici, il s’agit de 315 mOsmol / L. Augmenter ou diminuer la concentration de saccharose au besoin pour correspondre à l’osmolarité du tampon d’imagerie et du milieu de croissance. - Ajustez le pH à 7,4. Préparez des aliquotes et conservez-les à 4 °C jusqu’à deux semaines. Pour un stockage à long terme, conserver les aliquotes à -20 °C. Laissez le tampon d’imagerie atteindre la température ambiante avant utilisation.
- Préparer 100 mL de tampon d’imagerie en ajoutant tous les composants du tableau 2 à 80 mL d’eau stérile dans un cylindre de mesure. Porter le volume jusqu’à 100 mL avec de l’eau stérile. Mélanger en agitant soigneusement le cylindre de mesure jusqu’à ce que la solution paraisse homogène.
| Composant | Solution mère (M) | Concentration finale (mM) | Volume (mL) |
| NaCl | 5 | 114 | 2.3 |
| Kcl | 3 | 5.29 | 0.176 |
| MgCl2 | 1.9 | 1 | 0.053 |
| CaCl2 | 1 | 2 | 0.2 |
| Glycine | 0.1 | 0.005 | 0.005 |
| Saccharose | 1.5 | 52 | 3.5 |
| Pyruvate de sodium | 0.1 | 0.5 | 0.5 |
| HEPES | 1 | 10 | 1 |
| Glucose | 2.5 | 5 | 0.2 |
Tableau 2 : Composition du tampon d’imagerie. Les volumes indiqués sont utilisés pour la préparation de 100 mL de tampon d’imagerie.
- Solutions pour la stimulation et l’étalonnage
REMARQUE: Préparez toujours de nouvelles solutions de stimulation en ajoutant des solutions mères de médicaments indiqués au tampon d’imagerie juste avant l’expérience. Des solutions de stimulation et d’étalonnage seront ajoutées séquentiellement à la chambre d’imagerie au cours d’une expérience (voir rubriques 3-5). Selon le type d’expérience, différentes solutions sont nécessaires pour atteindre la même concentration finale dans le volume final respectif dans la chambre d’imagerie.- Préparer 3x solution de NMDA (90 μM; concentration finale dans la chambre: 30 μM) en ajoutant 63 μL d’un support NMDA de 10 mM à 6,937 mL de tampon d’imagerie. Ajouter 500 μL de la solution obtenue à la chambre (volume final : 1,5 mL).
- Préparer 2x solution da pour les étapes 3 et 4 (1 mM; concentration finale dans la chambre: 0,5 mM) en ajoutant 14 μL d’une souche DA de 0,5 M à 6,986 mL de tampon d’imagerie. Ajouter 1 mL à la chambre (volume final : 2 mL).
- Préparer 4x solution da pour l’étape 5 (2 mM; concentration finale dans la chambre: 0,5 mM) en ajoutant 28 μL d’une souche DA de 0,5 M à 6,972 mL de tampon d’imagerie. Ajouter 500 μL à la chambre (volume final : 2 mL).
- Préparer 1x solution TNT (5 mM; concentration finale dans la chambre: 5 mM) en ajoutant 45 μL de 1 M de papier TNT à 8955 μL de tampon d’imagerie. Ajouter 1 mL de cette solution à la chambre après aspiration du tampon d’imagerie (volume final : 1 mL).
2. Chargement des cellules avec TMRE
REMARQUE: Dans ce protocole, TMRE est utilisé en mode non trempé15 à une concentration finale de 20 nM. En général, la concentration la plus faible possible de TMRE qui fournit encore une intensité de signal suffisante sur le microscope de choix doit être utilisée. En raison de l’évaporation inégale, le volume de milieu dans différents puits peut différer dans les cultures primaires à long terme. Pour assurer une concentration constante de TMRE dans tous les puits, n’ajoutez pas de TMRE directement aux puits. Au lieu de cela, remplacez le milieu dans chaque puits par la même quantité de milieu contenant du TMRE. Le protocole ci-dessous est conçu pour les neurones primaires dans des plaques de 24 puits contenant environ 1 mL de milieu par puits.
- En travaillant dans une hotte à écoulement laminaire de culture tissulaire, collectez 500 μL de milieu de chaque puits dans un seul tube conique.
- Par puits, ajouter 0,5 μL de stock TMRE de 20 μM dans le tube conique (p. ex., 12 μL pour 24 puits).
- Aspirer soigneusement le milieu restant du premier puits et le remplacer par 500 μL de milieu contenant du TMRE. Continuez, puits par puits, avec les puits restants.
REMARQUE: Veillez à ne pas laisser les cellules sécher et à ne pas déranger les cellules. - Retournez les cellules dans l’incubateur et attendez au moins 60 minutes pour l’équilibrage du colorant.
REMARQUE: Le temps de chargement peut être prolongé à plusieurs heures sans effets indésirables. - Pour assurer des concentrations et un équilibre constants de TMRE tout au long de l’expérience d’imagerie, assurez-vous d’inclure une concentration finale de 20 nM TMRE dans le tampon d’imagerie et toutes les solutions de stimulation.
3. Optimisation des paramètres du microscope confocal à balayage
REMARQUE: Cette étape vise à trouver le meilleur compromis entre la qualité d’image et la viabilité des cellules lors de l’imagerie en direct. Cette section décrit l’optimisation des paramètres pour l’imagerie roGFP. Si l’imagerie multiparamétrique est effectuée, une optimisation similaire, y compris la vérification d’une base de référence stable sans signes de blanchiment ou de phototoxicité, doit être effectuée pour les indicateurs supplémentaires.
- Démarrez le microscope confocal et chargez les paramètres standard pour l’imagerie GFP (excitation de 488 nm, émission de 505 à 550 nm).
- Réglez le détecteur sur 12 bits ou 16 bits.
REMARQUE: Habituellement, 8 bits ne sont pas suffisants pour l’imagerie quantitative. - Activez le mode de balayage séquentiel et ajoutez une deuxième séquence/piste (excitation de 405 nm, émission de 505 à 550 nm).
- Pour les deux couches, sélectionnez une table de choix pseudocolore qui indique les pixels surexposés et sous-exposés (par exemple, GLOW OU).
- Sélectionnez un objectif adapté à l’objet d’intérêt.
REMARQUE: 10x-40x conviennent à l’analyse à cellule unique, 63x-100x conviennent à l’analyse de mitochondrie unique. - Montez un couvercle avec des cellules dans la chambre d’imagerie, ajoutez 1 mL de tampon d’imagerie et placez la chambre sur le microscope.
- Utilisez l’oculaire et la lumière transmise pour focaliser les cellules.
REMARQUE: N’utilisez pas de lumière d’épifluorescence pour localiser et focaliser les cellules. Même à faible puissance, cela affectera négativement les cellules. - Enregistrez des images avec différents formats de pixels. Sur la base de ces images, sélectionnez le nombre de pixels le plus bas qui donne une résolution acceptable de la structure d’intérêt.
REMARQUE: En règle générale, 512 x 512 pixels fonctionnent bien pour l’imagerie unicellulaire avec des objectifs 20x et 40x, et 1024 x 1024 ou 2048 x2048 pixels fonctionnent généralement bien pour l’imagerie à mitochondrie unique avec un objectif 63x. - Enregistrez des images avec différentes tailles de sténopés. Sur la base de ces images, sélectionnez la plus grande taille de sténopé qui donne une résolution acceptable de la structure d’intérêt.
REMARQUE: En règle générale, 3 à 7 unités aérées fonctionnent bien. - Enregistrez des images avec différentes intensités laser.
- Ajustez le gain et le seuil du détecteur en conséquence. Sur la base de ces images, sélectionnez l’intensité laser la plus faible qui donne une intensité de signal acceptable et un rapport signal/arrière-plan.
- Pour déterminer le rapport signal/arrière-plan, mesurez l’intensité du signal dans une région d’intérêt (ROI) qui contient des cellules ou des mitochondries (ROI1) et dans un ROI sans cellules ni mitochondries (ROI2). Ensuite, divisez l’intensité du ROI1 par l’intensité du ROI2.
REMARQUE: Visez un rapport signal/arrière-plan de >3 et des intensités de signal de 200 à 1 000 pour une excitation de 405 nm avec une puissance laser de 1 à 3% et des intensités de ROI individuels de 300 à 1 500 pour une excitation de 488 nm avec une puissance laser de 1%.
- Pour déterminer le rapport signal/arrière-plan, mesurez l’intensité du signal dans une région d’intérêt (ROI) qui contient des cellules ou des mitochondries (ROI1) et dans un ROI sans cellules ni mitochondries (ROI2). Ensuite, divisez l’intensité du ROI1 par l’intensité du ROI2.
- Ajustez le gain et le seuil du détecteur en conséquence. Sur la base de ces images, sélectionnez l’intensité laser la plus faible qui donne une intensité de signal acceptable et un rapport signal/arrière-plan.
- Enregistrez des images avec différentes vitesses de numérisation et nombre de moyennes d’images. Enregistrez 4 à 5 images pour chaque combinaison de paramètres. Sur la base de ces séries d’images, sélectionnez la vitesse la plus élevée et les paramètres moyens les plus bas qui donnent un bruit d’image acceptable et une variabilité d’image à image.
REMARQUE: Une vitesse de numérisation de 600 Hz et 1-2 images pour la moyenne fonctionne bien dans la plupart des cas. - À l’aide d’un nouveau couvercle, enregistrez une série time-lapse avec les paramètres optimisés.
REMARQUE: La durée et l’intervalle d’image de la série doivent ressembler à ceux des expériences prévues. - À la fin de la série time-lapse, ajoutez 1 mL de solution 2x DA à la chambre d’enregistrement. Image pour 2 min supplémentaires.
- Aspirer le tampon d’imagerie à l’aide d’une pompe péristaltique ou d’une pipette portative. Ajouter 1 mL de solution 1x TNT. Image pour 5 min supplémentaires.
- Analysez l’expérience time-lapse (voir rubrique 5).
- Vérifiez qu’aucun des deux canaux n’est surexposé ou sous-exposé pendant le traitement DA et DTT avec les paramètres optimisés.
- S’assurer qu’aucun des deux canaux ne présente un blanchiment considérable pendant l’enregistrement en accéléré; viser une perte d’intensité de <2 % entre la première et la dernière image.
- Vérifiez que le rapport 405:488 ne change pas considérablement pendant l’imagerie.
- Répétez l’ensemble de la procédure de manière itérative, en utilisant plusieurs couvercles, jusqu’à ce que des paramètres qui fournissent systématiquement des résultats acceptables aient été définis.
4. Évaluation du statut redox basal
- Démarrez le microscope et chargez les paramètres optimisés de la section 3.
- Définissez la moyenne de l’image sur 3-5.
- Montez un couvercle avec des cellules dans la chambre d’imagerie, ajoutez 1 mL de tampon d’imagerie et placez la chambre sur le microscope.
- Utilisez l’oculaire et la lumière transmise pour focaliser les cellules.
REMARQUE: N’utilisez pas de lumière d’épifluorescence pour localiser et focaliser les cellules. Même à faible puissance, cela affectera négativement les cellules. - Passez en mode de numérisation et utilisez le canal 488 nm en mode en direct pour faire la mise au point et localiser les cellules pour l’imagerie.
- Utilisez la fonction multipoint pour sélectionner 3 à 5 champs de vision sur le couvercle.
- Enregistrez une image de référence.
- Ajouter 1 mL de solution 2x DA à la chambre.
- Après 1, 2 et 3 minutes, utilisez la vue en direct pour confirmer/ajuster la mise au point, puis enregistrez une image.
REMARQUE: Les cellules sont généralement complètement oxydées après 2 min. - Remplacez le tampon dans la chambre d’imagerie par 1 mL de solution 1x DTT.
- Après 3 et 5 minutes, utilisez la vue en direct pour confirmer/ajuster la mise au point, puis enregistrez une image.
REMARQUE: Les cellules sont généralement complètement réduites après 4-5 min.
5. Imagerie en direct des traitements aigus
REMARQUE: Le protocole ci-dessous décrit l’imagerie de la réponse redox mitochondriale au traitement NMDA. Les intervalles d’image et la durée de l’expérience peuvent devoir être ajustés pour d’autres traitements.
- Démarrez le microscope et chargez les paramètres optimisés de la section 3.
- Réglez l’intervalle de temps sur 30 s et la durée sur 25 min.
- Montez un couvercle avec des cellules dans la chambre d’imagerie, ajoutez 1 mL de tampon d’imagerie et placez la chambre sur le microscope.
REMARQUE: Pour éviter la dérive de mise au point thermique, laissez les cellules sur la scène du microscope pendant 10-15 minutes avant de commencer l’imagerie time-lapse. - Utilisez l’oculaire et la lumière transmise pour focaliser les cellules.
REMARQUE: N’utilisez pas de lumière d’épifluorescence pour localiser et focaliser les cellules. Même à faible puissance, cela affectera négativement les cellules. - Passez en mode de numérisation et utilisez le canal 488 nm en mode en direct pour faire la mise au point et localiser les cellules pour l’imagerie.
- Facultatif : Pour augmenter le nombre de cellules enregistrées par exécution, utilisez la fonction multipoint pour imager 2 à 3 champs de vision par glissement de couverture.
- Démarrez l’acquisition time-lapse et enregistrez 5 images en tant qu’enregistrement de base de 2 minutes.
- Ajouter 500 μL de solution 3x NMDA à la chambre (concentration finale 30 μM) et enregistrer 20 images supplémentaires en tant que réponse NMDA de 10 minutes.
REMARQUE: Les neurones sont très sensibles aux changements d’osmolarité. Par conséquent, assurez-vous de minimiser l’évaporation du tampon d’imagerie. Pour les traitements plus longs, la chambre d’imagerie doit être recouverte d’un couvercle. - Ajoutez 500 μL de solution 4x DA à la chambre et enregistrez 6 images supplémentaires (étalonnage maximum de 3 min).
- Aspirer le tampon de la chambre d’imagerie et le remplacer par 1 mL de solution 1x TNT. Enregistrez 10 images supplémentaires (étalonnage minimum de 5 min).
- Terminez l’enregistrement et enregistrez la série d’images.
6. Analyse des données
- Importation de données et prétraitement d’images dans FIJI
- Utilisez l’importateur de bioformats pour ouvrir un groupe d’images à partir de l’étape 4 ou un fichier image à partir de l’étape 5. Cliquez sur Plugins | Bio-Formats | Importateur de bioformats. Dans la boîte de dialogue, utilisez Afficher la pile avec : Hyperstack, définissez le mode Couleur : par défaut, sélectionnez Mise à l’échelle automatique et ne vous divisez pas en fenêtres distinctes.
REMARQUE: La mise à l’échelle automatique optimise l’affichage des données sur l’écran de l’ordinateur. Il ne change pas l’intensité des pixels. - Si des images individuelles de l’étape 4 ont été ouvertes, cliquez sur Image | Piles | Outils | Concaténez pour les fusionner en une pile d’images uniques.
- S’il y a XY-drift pendant la série d’images, cliquez sur Plugins | StackReg pour enregistrer les images. Dans la boîte de dialogue, sélectionnez Corps rigide ou Traduction.
- Changez le format d’image en 32 bits en cliquant sur Image | Type | 32 bits.
- Divisez les couches de couleur en fenêtres séparées en cliquant sur Image | | de couleur Fractionner les canaux.
- Sélectionnez le canal 1 (405 nm) et ajustez le seuil pour sélectionner les mitochondries à analyser en cliquant sur Image | Ajuster | Seuil. Dans la boîte de dialogue, sélectionnez Par défaut, Rouge, Arrière-plan sombre et Histogramme pile et attendez que les pixels sélectionnés apparaissent en rouge. Cliquez sur Appliquer. Sélectionnez Définir les pixels d’arrière-plan sur NaN et Traiter toutes les images.
REMARQUE : Pour éviter tout biais potentiel de l’observateur, il convient d’utiliser la détermination automatisée des seuils. FIJI propose plusieurs méthodes automatisées (telles que Default, Huang, Intermodes, Otsu) qui peuvent être sélectionnées dans un menu déroulant dans la boîte de dialogue de seuil. En règle générale, la méthode Default donne un bon résultat. Il est recommandé de comparer plusieurs méthodes lors de la première analyse pour trouver la meilleure méthode de seuil pour l’ensemble d’images donné. Une fois qu’une méthode a été choisie, elle doit être appliquée à toutes les images. - Répétez l’étape 6.1.6 pour le canal 2 (488 nm).
- Créez une image de ratio pour visualiser le rapport 405:488 nm en cliquant sur Traiter | Calculatrice d’images. Dans la boîte de dialogue, sélectionnez Image 1: canal 1, Opération: Diviser, Image 2: canal 2, Créer une nouvelle fenêtre, Traiter toutes les images.
- Remplacez la table de choix de l’image de ratio en pseudocolore. Par exemple, pour passer à Fire, cliquez sur Image | Tables de choix | Incendie.
- Utilisez l’importateur de bioformats pour ouvrir un groupe d’images à partir de l’étape 4 ou un fichier image à partir de l’étape 5. Cliquez sur Plugins | Bio-Formats | Importateur de bioformats. Dans la boîte de dialogue, utilisez Afficher la pile avec : Hyperstack, définissez le mode Couleur : par défaut, sélectionnez Mise à l’échelle automatique et ne vous divisez pas en fenêtres distinctes.
- Analyse d’images
- Sur l’image de ratio, dessinez des ROI autour de cellules individuelles ou de mitochondries. Après avoir dessiné chaque roi, ajoutez-le au gestionnaire de retour sur investissement. Analyser | Outils | | roi Manager Ajouter. (raccourci clavier : 'T') Sélectionnez Afficher tout.
REMARQUE : Étant donné que les pixels d’arrière-plan ont été définis sur « pas un nombre » (NaN) aux étapes 6.1.6 et 6.1.7, ils n’affecteront pas le résultat de la mesure. Par conséquent, il est acceptable d’inclure des pixels d’arrière-plan dans le retour sur investissement. - Mesurez les ratios 405:488 des cellules individuelles en cliquant sur ROI Manager | ctrl+A pour sélectionner tous les ROI | Plus d'| Multi Mesure. Dans la boîte de dialogue, sélectionnez Mesurer toutes les tranches et Une ligne par tranche.
- Exportez les mesures vers un tableur.
- Sélectionnez l’image 405 nm. Mesurer l’intensité de tous les ROI comme à l’étape 6.2.2. en utilisant les rois de retour sur investissement stockés dans le gestionnaire de retour sur investissement.
- Exportez les mesures vers un tableur.
- Sélectionnez l’image 488 nm. Mesurer l’intensité de tous les retours sur investissement comme à l’étape 6.2.2. en utilisant les rois de retour sur investissement stockés dans le gestionnaire de retour sur investissement.
- Exportez les mesures vers un tableur.
- Enregistrez les retours sur investissement pour référence future en cliquant sur ROI Manager | ctrl + A pour sélectionner tous les retours sur investissement | Plus d'| Enregistrer.
- Recommandé : Générer des diagrammes d’intensité vs temps des traces de 405 et 488 nm. Vérifiez qu’il n’y a pas de blanchiment marqué dans l’un ou l’autre des canaux (l’intensité du signal à la fin de la série d’images doit être ≥98 % de la première image) et que les deux traces se déplacent dans des directions opposées pendant les réponses du capteur (par exemple, la trace de 405 nm doit augmenter pendant l’oxydation tandis que la trace de 488 nm doit diminuer).
- Sur l’image de ratio, dessinez des ROI autour de cellules individuelles ou de mitochondries. Après avoir dessiné chaque roi, ajoutez-le au gestionnaire de retour sur investissement. Analyser | Outils | | roi Manager Ajouter. (raccourci clavier : 'T') Sélectionnez Afficher tout.
- Normalisation des données
- Pour chaque retour sur investissement de l’image de ratio, déterminez la valeur maximale pendant le traitement DA (Rmax) et la valeur minimale pendant le traitement DTT (Rmin).
- Calculez le rapport normalisé comme suit :
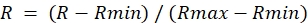
REMARQUE: Cela définira le rapport maximum à 1,0 et le rapport minimum à 0.
- Analyse de la morphologie mitochondriale
- Pour obtenir des mesures de la morphologie mitochondriale en parallèle des intensités roGFP à l’étape 6.2.6, allez à Analyser | Définissez Mesures et vérifiez les descripteurs de forme et ajuster les ellipses.
REMARQUE: En plus de l’intensité moyenne, les mesures dans la fenêtre de résultats incluront la longueur de l’axe principal (Majeur), la longueur de l’axe mineur (Mineur), le rapport d’aspect (AR; axe majeur divisé par axe mineur; les mitochondries rondes ont un AR ~ 1, les mitochondries allongées ont un AR plus grand), ainsi que des mesures de circularité (Circ.) et de rondeur (Ronde).
- Pour obtenir des mesures de la morphologie mitochondriale en parallèle des intensités roGFP à l’étape 6.2.6, allez à Analyser | Définissez Mesures et vérifiez les descripteurs de forme et ajuster les ellipses.
Résultats
Quantification des différences dans l’état redox mitochondrial à l’état d’équilibre après le retrait du facteur de croissance
Pour démontrer la quantification des différences à l’état d’équilibre dans l’état redox mitochondrial, les neurones primaires cultivés en milieu standard ont été comparés à des neurones cultivés sans facteurs de croissance pendant 48 h avant l’imagerie. Le retrait du facteur de croissance entraîne la mort des cellules neuronales apoptotiques apr...
Discussion
Les mesures quantitatives et dynamiques de l’état redox mitochondrial fournissent des informations importantes sur la physiologie mitochondriale et cellulaire. Plusieurs sondes chimiques fluorogéniques sont disponibles pour détecter les espèces réactives de l’oxygène, le « stress redox » ou le « stress oxydatif ». Cependant, ces derniers termes ne sont pas bien définis et manquent souvent de spécificité9,17,18.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont pas de conflit d’intérêts.
Remerciements
Ce travail a été soutenu par la Deutsche Forschungsgemeinschaft (BA 3679/5-1; POUR 2289: BA 3679/4-2). A.K. est soutenu par une bourse ERASMUS+. Nous remercions Iris Bünzli-Ehret, Rita Rosner et Andrea Schlicksupp pour la préparation des neurones primaires. Nous remercions le Dr Tobias Dick d’avoir fourni pLPCX-mito-Grx1-roGFP2. Les expériences illustrées à la figure 4 ont été réalisées au Nikon Imaging Center de l’Université de Heidelberg. La figure 2 a été préparée avec BioRender.com.
matériels
| Name | Company | Catalog Number | Comments |
| reagents | |||
| Calcium chloride (CaCl2·2H2O) | Sigma-Aldrich | C3306 | |
| Diamide (DA) | Sigma-Aldrich | D3648 | |
| Dithiothreitol (DTT) | Carl Roth GmbH | 6908.1 | |
| Glucose (2.5 M stock solution) | Sigma-Aldrich | G8769 | |
| Glucose | Sigma-Aldrich | G7528 | |
| Glycine | neoFroxx GmbH | LC-4522.2 | |
| HEPES (1 M stock solution) | Sigma-Aldrich | 15630-080 | |
| HEPES | Sigma-Aldrich | H4034 | |
| Magnesium chloride (MgCl2·6H2O) | Sigma-Aldrich | 442611-M | |
| N-methyl-D-aspartate (NMDA) | Sigma-Aldrich | M3262 | |
| Potassium chloride (KCl) | Sigma-Aldrich | P3911 | |
| Sodium chloride (NaCl) | neoFroxx GmbH | LC-5932.1 | |
| Sodium pyruvate (0.1 M stock solution) | Sigma-Aldrich | S8636 | |
| Sodium pyruvate | Sigma-Aldrich | P8574 | |
| Sucrose | Carl Roth GmbH | 4621.1 | |
| Tetramethylrhodamine ethyl ester perchlorate (TMRE) | Sigma-Aldrich | 87917 | |
| equipment | |||
| imaging chamber | Life Imaging Services (Basel, Switzerland) | 10920 | Ludin Chamber Type 3 for Ø12mm coverslips |
| laser scanning confocal microscope, microscope | Leica | DMI6000 | |
| laser scanning confocal microscope, scanning unit | Leica | SP8 | |
| peristaltic pump | VWR | PP1080 181-4001 | |
| spinning disc confocal microscope, camera | Hamamatsu | C9100-02 EMCCD | |
| spinning disc confocal microscope, incubationsystem | TokaiHit | INU-ZILCF-F1 | |
| spinning disc confocal microscope, microscope | Nikon | Ti microscope | |
| spinning disc confocal microscope, scanning unit | Yokagawa | CSU-X1 | |
| software | |||
| FIJI | https://fiji.sc | ||
| StackReg plugin | https://github.com/fiji-BIG/StackReg/blob/master/src/main/java/StackReg_.java | ||
| TurboReg plugin | https://github.com/fiji-BIG/TurboReg/blob/master/src/main/java/TurboReg_.java |
Références
- Roede, J. R., Go, Y. M., Jones, D. P. Redox equivalents and mitochondrial bioenergetics. Methods in Molecular Biology. 810, 249-280 (2012).
- Turrens, J. F. Mitochondrial formation of reactive oxygen species. Journal of Physiology. 552, 335-344 (2003).
- Lin, M. T., Beal, M. F. Mitochondrial dysfunction and oxidative stress in neurodegenerative diseases. Nature. 443 (7113), 787-795 (2006).
- Manfredi, G., Beal, M. F. The role of mitochondria in the pathogenesis of neurodegenerative diseases. Brain Pathology. 10 (3), 462-472 (2000).
- Mari, M., Morales, A., Colell, A., Garcia-Ruiz, C., Fernandez-Checa, J. C. Mitochondrial glutathione, a key survival antioxidant. Antioxidants & Redox Signaling. 11 (11), 2685-2700 (2009).
- Murphy, M. P. Mitochondrial thiols in antioxidant protection and redox signaling: distinct roles for glutathionylation and other thiol modifications. Antioxidants & Redox Signaling. 16 (6), 476-495 (2012).
- Dooley, C. T., et al. Imaging dynamic redox changes in mammalian cells with green fluorescent protein indicators. Journal of Biological Chemistry. 279 (21), 22284-22293 (2004).
- Hanson, G. T., et al. Investigating mitochondrial redox potential with redox-sensitive green fluorescent protein indicators. Journal of Biological Chemistry. 279 (13), 13044-13053 (2004).
- Gutscher, M., et al. Real-time imaging of the intracellular glutathione redox potential. Nature Methods. 5 (6), 553-559 (2008).
- Morgan, B., Sobotta, M. C., Dick, T. P. Measuring E(GSH) and H2O2 with roGFP2-based redox probes. Free Radical Biology & Medicine. 51 (11), 1943-1951 (2011).
- Marwick, K. F. M., Hardingham, G. E. Transfection in primary cultured neuronal cells. Methods in Molecular Biology. 1677, 137-144 (2017).
- Kohrmann, M., et al. convenient, and effective method to transiently transfect primary hippocampal neurons. Journal of Neuroscience Research. 58 (6), 831-835 (1999).
- Depp, C., Bas-Orth, C., Schroeder, L., Hellwig, A., Bading, H. Synaptic activity protects neurons against calcium-mediated oxidation and contraction of mitochondria during excitotoxicity. Antioxidants & Redox Signaling. 29 (12), 1109-1124 (2018).
- Hauck, B., Chen, L., Xiao, W. Generation and characterization of chimeric recombinant AAV vectors. Molecular Therapy. 7 (3), 419-425 (2003).
- Brand, M. D., Nicholls, D. G. Assessing mitochondrial dysfunction in cells. Biochemical Journal. 435 (2), 297-312 (2011).
- Zhang, S. J., et al. Nuclear calcium signaling controls expression of a large gene pool: identification of a gene program for acquired neuroprotection induced by synaptic activity. PLoS Genetics. 5 (8), 1000604 (2009).
- Winterbourn, C. C. The challenges of using fluorescent probes to detect and quantify specific reactive oxygen species in living cells. Biochimica et Biophysica Acta. 1840 (2), 730-738 (2014).
- Sies, H. Oxidative stress: a concept in redox biology and medicine. Redox Biology. 4, 180-183 (2015).
- Lukyanov, K. A., Belousov, V. V. Genetically encoded fluorescent redox sensors. Biochimica et Biophysica Acta. 1840 (2), 745-756 (2014).
- Nietzel, T., et al. Redox-mediated kick-start of mitochondrial energy metabolism drives resource-efficient seed germination. Proceedings of the National Academy of Sciences of the United States of America. 117 (1), 741-751 (2020).
- Albrecht, S. C., et al. Redesign of genetically encoded biosensors for monitoring mitochondrial redox status in a broad range of model eukaryotes. Journal of Biomolecular Screening. 19 (3), 379-386 (2014).
- Albrecht, S. C., Barata, A. G., Grosshans, J., Teleman, A. A., Dick, T. P. In vivo mapping of hydrogen peroxide and oxidized glutathione reveals chemical and regional specificity of redox homeostasis. Cell Metabolism. 14 (6), 819-829 (2011).
- Breckwoldt, M. O., et al. Multiparametric optical analysis of mitochondrial redox signals during neuronal physiology and pathology in vivo. Nature Medicine. 20 (5), 555-560 (2014).
- Ricke, K. M., et al. Mitochondrial dysfunction combined with high calcium load leads to impaired antioxidant defense underlying the selective loss of nigral dopaminergic neurons. Journal of Neuroscience. 40 (9), 1975-1986 (2020).
- Bjornberg, O., Ostergaard, H., Winther, J. R. Mechanistic insight provided by glutaredoxin within a fusion to redox-sensitive yellow fluorescent protein. Biochemistry. 45 (7), 2362-2371 (2006).
- Shokhina, A. G., et al. Red fluorescent redox-sensitive biosensor Grx1-roCherry. Redox Biology. 21, 101071 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon