È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Imaging dal vivo dello stato redox del glutatione mitocondriale nei neuroni primari utilizzando un indicatore raziometrico
In questo articolo
Riepilogo
Questo articolo descrive un protocollo per determinare le differenze nello stato redox basale e le risposte redox alle perturbazioni acute nei neuroni primari dell'ippocampo e corticale utilizzando la microscopia confocale dal vivo. Il protocollo può essere applicato ad altri tipi di cellule e microscopi con modifiche minime.
Abstract
L'omeostasi redox mitocondriale è importante per la vitalità e la funzione neuronale. Sebbene i mitocondri contengano diversi sistemi redox, il glutatione tampone redox tiolo-disolfuro altamente abbondante è considerato un attore centrale nelle difese antiossidanti. Pertanto, la misurazione del potenziale redox del glutatione mitocondriale fornisce informazioni utili sullo stato redox mitocondriale e sullo stress ossidativo. La glutaredossina1-roGFP2 (Grx1-roGFP2) è un indicatore raziometrico basato su proteine fluorescenti verdi (GFP) geneticamente codificate del potenziale redox del glutatione che ha due picchi di eccitazione sensibili allo stato redox a 400 nm e 490 nm con un singolo picco di emissione a 510 nm. Questo articolo descrive come eseguire la microscopia confocale dal vivo di Grx1-roGFP2 mirato ai mitocondri nei neuroni primari dell'ippocampo e corticale. Descrive come valutare il potenziale redox del glutatione mitocondriale allo stato stazionario (ad esempio, per confrontare stati patologici o trattamenti a lungo termine) e come misurare i cambiamenti redox sui trattamenti acuti (usando il farmaco eccitotossico N-metil-D-aspartato (NMDA) come esempio). Inoltre, l'articolo presenta la co-imaging di Grx1-roGFP2 e l'indicatore del potenziale di membrana mitocondriale, tetrametilrodramina, estere etilico (TMRE), per dimostrare come Grx1-roGPF2 può essere multiplexato con indicatori aggiuntivi per analisi multiparametriche. Questo protocollo fornisce una descrizione dettagliata di come (i) ottimizzare le impostazioni del microscopio a scansione laser confocale, (ii) applicare farmaci per la stimolazione seguiti dalla calibrazione del sensore con diammide e ditiotreitolo e (iii) analizzare i dati con ImageJ / FIJI.
Introduzione
Diversi importanti enzimi mitocondriali e molecole di segnalazione sono soggetti alla regolazione redox del tiolo1. Inoltre, i mitocondri sono una delle principali fonti cellulari di specie reattive dell'ossigeno e sono selettivamente vulnerabili al danno ossidativo2. Di conseguenza, il potenziale redox mitocondriale influenza direttamente la bioenergetica, la segnalazione cellulare, la funzione mitocondriale e, infine, la vitalità cellulare3,4. La matrice mitocondriale contiene elevate quantità (1-15 mM) del glutatione tampone redox tiolo-disolfuro (GSH) per mantenere l'omeostasi redox e montare difese antiossidanti5,6. Il GSH può essere attaccato covalentemente alle proteine bersaglio (S-glutationilazione) per controllarne lo stato redox e l'attività ed è utilizzato da una serie di enzimi disintossicanti che riducono le proteine ossidate. Pertanto, il potenziale redox del glutatione mitocondriale è un parametro altamente informativo quando si studia la funzione mitocondriale e la fisiopatologia.
roGFP2 è una variante della GFP che è stata resa redox-sensibile dall'aggiunta di due ciste esposte in superficie che formano una coppia artificiale ditiolo-disolfuro7,8. Ha un singolo picco di emissione a ~ 510 nm e due picchi di eccitazione a ~ 400 nm e 490 nm. È importante sottolineare che le ampiezze relative dei due picchi di eccitazione dipendono dallo stato redox di roGFP2 (Figura 1), rendendo questa proteina un sensore raziometrico. Nel sensore Grx1-roGFP2, la glutaredossina-1 umana (Grx1) è stata fusa al N-terminus di roGFP29,10. L'attacco covalente dell'enzima Grx1 a roGFP2 offre due importanti miglioramenti del sensore: rende la risposta del sensore specifica per la coppia GSH/GSSG glutatione redox (Figura 1) e accelera l'equilibrio tra GSSG e roGFP2 di un fattore di almeno 100.0009. Pertanto, Grx1-roGFP2 consente l'imaging specifico e dinamico del potenziale redox del glutatione cellulare.
L'imaging Grx1-roGFP2 può essere eseguito su una vasta gamma di microscopi, tra cui microscopi a fluorescenza a campo largo, microscopi confocali a disco rotante e microscopi confocali a scansione laser. L'espressione del sensore nei neuroni primari può essere ottenuta con vari metodi che includono la lipofezione11, la coprecipitazione DNA/calcio-fosfato12, il trasferimento genico mediato da virus o l'uso di animali transgenici come fonte cellulare (Figura 2). Per gli esperimenti in questo articolo sono stati utilizzati virus adeno-associati ricombinanti pseudotipizzati (rAAV) contenenti un rapporto 1:1 delle proteine del capside AAV1 e AAV2 13,14. Con questo vettore, l'espressione massima del sensore viene in genere raggiunta 4-5 giorni dopo l'infezione e rimane stabile per almeno due settimane. Abbiamo utilizzato con successo Grx1-roGFP2 nei neuroni primari dell'ippocampo e corticale di topi e ratti.
In questo articolo, l'espressione mediata da rAAV di Grx1-roGFP2 mirato ai mitocondri nei neuroni primari dell'ippocampo e corticale del ratto viene utilizzata per valutare lo stato redox del glutatione mitocondriale basale e la sua perturbazione acuta. Viene fornito un protocollo per l'imaging confocale dal vivo con istruzioni dettagliate su come (i) ottimizzare le impostazioni del microscopio confocale a scansione laser, (ii) eseguire un esperimento di imaging dal vivo e (iii) analizzare i dati con FIJI.
Protocollo
Tutti gli esperimenti sugli animali erano conformi alle linee guida nazionali e istituzionali, compresa la direttiva 2010/63/UE del Parlamento europeo del Consiglio, e avevano la piena approvazione etica del Ministero degli Interni (Ufficio per il benessere degli animali dell'Università di Heidelberg e Regierungspraesidium Karlsruhe, licenze T14/21 e T13/21). I neuroni primari dell'ippocampo e corticale sono stati preparati da cuccioli di topo o ratto appena nati secondo procedure standard e sono stati mantenuti per 12-14 giorni come descritto in precedenza13.
1. Preparazione delle soluzioni
- Soluzioni stock per il buffer di imaging
- Preparare ogni soluzione madre secondo la Tabella 1 e mantenerla a 4 °C. Per la conservazione a lungo termine (>3 mesi), mantenere le aliquote a -20 °C.
| Componente | MW | Concentrazione (M) | Importo (g) | Volume (mL) |
| NaCl | 58.44 | 5 | 14.61 | 50 |
| Kcl | 74.55 | 3 | 1.12 | 5 |
| MgCl2· 6H2O | 203.3 | 1.9 | 2 | 5 |
| CaCl2·2H2O | 147.01 | 1 | 1.47 | 10 |
| Glicina | 75.07 | 0.1 | 0.375 | 50 |
| Saccarosio | 342.3 | 1.5 | 25.67 | 50 |
| Piruvato di sodio | 110.04 | 0.1 | 0.55 | 50 |
| HEPES · | 238.3 | 1 | 11.9 | 50 |
| Glucosio | 180.15 | 2.5 | 45 | 100 |
Tabella 1: Soluzioni stock per il buffer di imaging.
- Soluzioni stock di farmaci e coloranti
- Sciogliere la diammide (DA; utilizzata per la calibrazione del rapporto massimo 405:488) in acqua per ottenere una soluzione madre da 0,5 M (ad esempio, 1 g in 11,615 mL di acqua). Aliquota e conservare a -20 °C.
- Sciogliere il ditiotreitolo (DTT; utilizzato per la calibrazione del rapporto minimo 405:488) in acqua per ottenere una soluzione madre da 1 M (ad esempio, 5 g in 32.425 ml di acqua). Aliquota e conservare a -20 °C per un massimo di 3 mesi.
- Sciogliere N-metil-D-aspartato (NMDA; usato per indurre eccitotossicità e ossidazione mitocondriale) in acqua per ottenere una soluzione madre da 10 mM (ad esempio, 25 mg in 16.991 mL di acqua). Conservare le aliquote a -20 °C. Per la conservazione a lungo termine (>6 mesi), mantenere le aliquote a -80 °C.
- Tetrametilrodramina estere etilico perclorato (TMRE; un indicatore a piccola molecola del potenziale di membrana mitocondriale)
- Sciogliere la polvere TMRE in metanolo per ottenere una scorta di 20 mM (ad esempio, 25 mg in 2.427 ml di metanolo).
- Diluire lo stock di 20 mM 1:1.000 in metanolo per ottenere uno stock da 20 μM.
- Aliquotare le soluzioni stock da 20 mM e 20 μM, sigillare con parafilm e conservare al riparo dalla luce a -20 °C.
NOTA: Entrambe le soluzioni stock sono stabili per diversi anni. Utilizzare la soluzione madre 1.000x (20 μM) per gli esperimenti.
- Buffer di imaging
- Preparare 100 mL di tampone di imaging aggiungendo tutti i componenti dalla Tabella 2 a 80 mL di acqua sterile in un cilindro di misurazione. Portare il volume fino a 100 ml con acqua sterile. Mescolare agitando accuratamente il cilindro di misurazione fino a quando la soluzione appare omogenea.
NOTA: si consiglia di utilizzare un osmometro per verificare l'osmolarità del buffer. Dovrebbe essere il più vicino possibile al mezzo di crescita delle cellule. Qui, questo è 315 mOsmol / L. Aumentare o diminuire la concentrazione di saccarosio secondo necessità per abbinare l'osmolarità del tampone di imaging e del mezzo di crescita. - Regolare il pH a 7,4. Fare aliquote e tenerle a 4 °C per un massimo di due settimane. Per la conservazione a lungo termine, mantenere le aliquote a -20 °C. Lasciare che il buffer di imaging raggiunga la temperatura ambiente prima dell'uso.
- Preparare 100 mL di tampone di imaging aggiungendo tutti i componenti dalla Tabella 2 a 80 mL di acqua sterile in un cilindro di misurazione. Portare il volume fino a 100 ml con acqua sterile. Mescolare agitando accuratamente il cilindro di misurazione fino a quando la soluzione appare omogenea.
| Componente | Soluzione madre (M) | Concentrazione finale (mM) | Volume (mL) |
| NaCl | 5 | 114 | 2.3 |
| Kcl | 3 | 5.29 | 0.176 |
| MgCl2 | 1.9 | 1 | 0.053 |
| CaCl2 · | 1 | 2 | 0.2 |
| Glicina | 0.1 | 0.005 | 0.005 |
| Saccarosio | 1.5 | 52 | 3.5 |
| Piruvato di sodio | 0.1 | 0.5 | 0.5 |
| HEPES · | 1 | 10 | 1 |
| Glucosio | 2.5 | 5 | 0.2 |
Tabella 2: Composizione del buffer di imaging. I volumi indicati vengono utilizzati per la preparazione di 100 mL di tampone di imaging.
- Soluzioni per la stimolazione e la calibrazione
NOTA: Preparare sempre nuove soluzioni di stimolazione aggiungendo soluzioni di riserva di farmaci indicati al tampone di imaging appena prima dell'esperimento. Le soluzioni per la stimolazione e la calibrazione saranno aggiunte alla camera di imaging in sequenza durante un esperimento (vedere paragrafi 3-5). A seconda del tipo di esperimento, sono necessarie soluzioni diverse per raggiungere la stessa concentrazione finale nel rispettivo volume finale nella camera di imaging.- Preparare 3x soluzione NMDA (90 μM; concentrazione finale nella camera: 30 μM) aggiungendo 63 μL di uno stock NMDA da 10 mM a 6.937 mL di tampone di imaging. Aggiungere 500 μL della soluzione risultante alla camera (volume finale: 1,5 ml).
- Preparare 2x soluzione DA per le fasi 3 e 4 (1 mM; concentrazione finale nella camera: 0,5 mM) aggiungendo 14 μL di uno stock DA da 0,5 M a 6,986 mL di buffer di imaging. Aggiungere 1 mL alla camera (volume finale: 2 mL).
- Preparare 4x soluzione DA per la fase 5 (2 mM; concentrazione finale nella camera: 0,5 mM) aggiungendo 28 μL di uno stock DA da 0,5 M a 6,972 mL di buffer di imaging. Aggiungere 500 μL alla camera (volume finale: 2 mL).
- Preparare 1 soluzione DTT (5 mM; concentrazione finale nella camera: 5 mM) aggiungendo 45 μL di 1 M di stock DTT a 8955 μL di tampone di imaging. Aggiungere 1 mL di questa soluzione alla camera dopo aver aspirato il tampone di imaging (volume finale: 1 mL).
2. Caricamento delle celle con TMRE
NOTA: In questo protocollo, TMRE viene utilizzato in modalità non quench15 ad una concentrazione finale di 20 nM. In generale, deve essere utilizzata la concentrazione più bassa possibile di TMRE che fornisce ancora un'intensità del segnale sufficiente sul microscopio di scelta. A causa dell'evaporazione irregolare, il volume del mezzo in diversi pozzi può differire nelle colture primarie a lungo termine. Per garantire una concentrazione costante di TMRE in tutti i pozzetti, non aggiungere TMRE direttamente ai pozzetti. Invece, sostituire il mezzo in ogni pozzetto con la stessa quantità di mezzo contenente TMRE. Il protocollo seguente è progettato per i neuroni primari in piastre a 24 pozzetti contenenti ~ 1 mL di mezzo per pozzetto.
- Lavorando in una cappa a flusso laminare per colture tissutali, raccogliere 500 μL di terreno da ciascun pozzo in un singolo tubo conico.
- Per pozzetto, aggiungere 0,5 μL di 20 μM TMRE stock nel tubo conico (ad esempio, 12 μL per 24 pozzetti).
- Aspirare accuratamente il mezzo rimanente dal primo pozzo e sostituirlo con 500 μL di mezzo contenente TMRE. Continuate, bene per bene, con i restanti pozzi.
NOTA: Fare attenzione a non lasciare asciugare le cellule e a non disturbare le cellule. - Riportare le cellule all'incubatrice e attendere almeno 60 minuti per l'equilibrio del colorante.
NOTA: il tempo di caricamento può essere esteso a diverse ore senza effetti negativi. - Per garantire concentrazioni TMRE coerenti ed equilibrio durante l'esperimento di imaging, assicurarsi di includere una concentrazione finale di 20 nM TMRE nel tampone di imaging e in tutte le soluzioni di stimolazione.
3. Ottimizzazione delle impostazioni del microscopio confocale a scansione
NOTA: questo passaggio mira a trovare il miglior compromesso tra qualità dell'immagine e vitalità cellulare durante l'imaging dal vivo. In questa sezione viene descritta l'ottimizzazione delle impostazioni per l'imaging roGFP. Se viene eseguita l'imaging multiparametrico, è necessario eseguire un'ottimizzazione simile, incluso il controllo di una linea di base stabile senza segni di sbiancamento o fototossicità, per gli indicatori aggiuntivi.
- Avviare il microscopio confocale e caricare le impostazioni standard per l'imaging GFP (eccitazione a 488 nm, emissione 505 - 550 nm).
- Impostare il rilevatore su 12 bit o 16 bit.
NOTA: di solito, 8 bit non sono sufficienti per l'imaging quantitativo. - Attivare la modalità di scansione sequenziale e aggiungere la seconda sequenza/traccia (eccitazione 405 nm, emissione 505 - 550 nm).
- Per entrambi i canali, selezionare una tabella di ricerca pseudocolore che indichi i pixel sovraesposti e sottoesposti (ad esempio, GLOW OU).
- Selezionare un obiettivo adatto all'oggetto di interesse.
NOTA: 10x-40x sono adatti per l'analisi a singola cellula, 63x-100x sono adatti per l'analisi a mitocondrio singolo. - Montare un coverslip con le cellule nella camera di imaging, aggiungere 1 mL di buffer di imaging e posizionare la camera sul microscopio.
- Utilizzare l'oculare e la luce trasmessa per mettere a fuoco le cellule.
NOTA: non utilizzare la luce di epifluorescenza per localizzare e mettere a fuoco le cellule. Anche a bassa potenza, questo influenzerà negativamente le cellule. - Registra immagini con diversi formati di pixel. Sulla base di queste immagini, selezionare il numero di pixel più basso che fornisce una risoluzione accettabile della struttura di interesse.
NOTA: in genere, 512 x 512 pixel funzionano bene per l'imaging a cella singola con obiettivi 20x e 40x e 1024 x 1024 o 2048 x2048 pixel in genere funzionano bene per l'imaging a mitocondrio singolo con un obiettivo 63x. - Registra immagini con fori stenopeici diversi. Sulla base di queste immagini, selezionare la dimensione del foro stenopeico più grande che fornisce una risoluzione accettabile della struttura di interesse.
NOTA: in genere, 3-7 unità ariose funzionano bene. - Registra immagini con diverse intensità laser.
- Regolare di conseguenza il guadagno e la soglia del rilevatore. Sulla base di queste immagini, selezionare l'intensità laser più bassa che fornisce un'intensità del segnale accettabile e un rapporto segnale-sfondo.
- Per determinare il rapporto segnale-sfondo, misurare l'intensità del segnale in una regione di interesse (ROI) che contiene cellule o mitocondri (ROI1) e in un ROI senza cellule o mitocondri (ROI2). Quindi, dividi l'intensità del ROI1 per l'intensità del ROI2.
NOTA: Mirare a un rapporto segnale-sfondo di >3 e intensità del segnale dei singoli ROI di 200-1.000 per eccitazione a 405 nm con potenza laser dell'1-3% e intensità dei singoli ROI di 300-1.500 per eccitazione a 488 nm con potenza laser dell'1%.
- Per determinare il rapporto segnale-sfondo, misurare l'intensità del segnale in una regione di interesse (ROI) che contiene cellule o mitocondri (ROI1) e in un ROI senza cellule o mitocondri (ROI2). Quindi, dividi l'intensità del ROI1 per l'intensità del ROI2.
- Regolare di conseguenza il guadagno e la soglia del rilevatore. Sulla base di queste immagini, selezionare l'intensità laser più bassa che fornisce un'intensità del segnale accettabile e un rapporto segnale-sfondo.
- Registra immagini con diverse velocità di scansione e numero di medie dei fotogrammi. Registra 4-5 immagini per ogni combinazione di impostazioni. In base a queste serie di immagini, selezionare la velocità più alta e le impostazioni medie più basse che forniscano un rumore dell'immagine accettabile e una variabilità da immagine a immagine.
NOTA: una velocità di scansione di 600 Hz e 1-2 fotogrammi per la media funzionano bene nella maggior parte dei casi. - Utilizzando un nuovo coverslip, registra una serie time-lapse con le impostazioni ottimizzate.
NOTA: la durata e l'intervallo di immagine della serie devono essere simili a quelli degli esperimenti pianificati. - Alla fine della serie time-lapse, aggiungere 1 mL di soluzione 2x DA alla camera di registrazione. Immagine per ulteriori 2 min.
- Aspirare il buffer di imaging utilizzando una pompa peristaltica o una pipetta portatile. Aggiungere 1 mL di 1 soluzione DTT. Immagine per ulteriori 5 min.
- Analizzare l'esperimento time-lapse (vedere la sezione 5).
- Verificare che nessuno dei due canali venga sovra o sottoesposto durante il trattamento DA e DTT con le impostazioni ottimizzate.
- Assicurarsi che nessuno dei due canali mostri un notevole sbiancamento durante la registrazione time-lapse; mirare a <2% di perdita di intensità tra la prima e l'ultima immagine.
- Verificare che il rapporto 405:488 non cambi considerevolmente durante l'imaging.
- Ripetere l'intera procedura in modo iterativo, utilizzando diversi coverslip, fino a quando non sono state definite impostazioni che forniscono costantemente risultati accettabili.
4. Valutazione dello stato redox basale
- Avviare il microscopio e caricare le impostazioni ottimizzate dalla sezione 3.
- Imposta la media dei fotogrammi su 3-5.
- Montare un coverslip con le cellule nella camera di imaging, aggiungere 1 mL di buffer di imaging e posizionare la camera sul microscopio.
- Utilizzare l'oculare e la luce trasmessa per mettere a fuoco le cellule.
NOTA: non utilizzare la luce di epifluorescenza per localizzare e mettere a fuoco le cellule. Anche a bassa potenza, questo influenzerà negativamente le cellule. - Passare alla modalità di scansione e utilizzare il canale a 488 nm in live view per mettere a fuoco e individuare le celle per l'imaging.
- Utilizzare la funzione multipunto per selezionare 3-5 campi visivi sulla copertina.
- Registrare un'immagine di base.
- Aggiungere 1 mL di 2x soluzione DA alla camera.
- Dopo 1, 2 e 3 minuti, utilizzare la vista dal vivo per confermare/regolare la messa a fuoco e quindi registrare un'immagine.
NOTA: le cellule sono in genere completamente ossidate dopo 2 minuti. - Sostituire il buffer nella camera di imaging con 1 mL di 1 soluzione DTT.
- Dopo 3 e 5 minuti, utilizzare la vista dal vivo per confermare / regolare la messa a fuoco e quindi registrare un'immagine.
NOTA: le celle sono in genere completamente ridotte dopo 4-5 minuti.
5. Imaging dal vivo di trattamenti acuti
NOTA: Il protocollo seguente descrive l'imaging della risposta redox mitocondriale al trattamento con NMDA. Gli intervalli di immagine e la durata dell'esperimento potrebbero dover essere regolati per altri trattamenti.
- Avviare il microscopio e caricare le impostazioni ottimizzate dalla sezione 3.
- Impostare l'intervallo di time-lapse su 30 s e la durata su 25 min.
- Montare un coverslip con le cellule nella camera di imaging, aggiungere 1 mL di buffer di imaging e posizionare la camera sul microscopio.
NOTA: per evitare la deriva della messa a fuoco termica, lasciare le cellule sullo stadio del microscopio per 10-15 minuti prima di iniziare l'imaging time-lapse. - Utilizzare l'oculare e la luce trasmessa per mettere a fuoco le cellule.
NOTA: non utilizzare la luce di epifluorescenza per localizzare e mettere a fuoco le cellule. Anche a bassa potenza, questo influenzerà negativamente le cellule. - Passare alla modalità di scansione e utilizzare il canale a 488 nm in live view per mettere a fuoco e individuare le celle per l'imaging.
- Facoltativo: per aumentare il numero di celle registrate per esecuzione, utilizzare la funzione multipunto per visualizzare 2-3 campi di visualizzazione per copertina.
- Avvia l'acquisizione time-lapse e registra 5 immagini come registrazione di base di 2 minuti.
- Aggiungere 500 μL di 3x soluzione NMDA alla camera (concentrazione finale 30 μM) e registrare ulteriori 20 immagini come risposta NMDA di 10 minuti.
NOTA: I neuroni sono molto sensibili ai cambiamenti nell'osmolarità. Pertanto, assicurarsi di ridurre al minimo l'evaporazione del buffer di imaging. Per trattamenti più lunghi, la camera di imaging deve essere coperta con un coperchio. - Aggiungere 500 μL di soluzione 4x DA alla camera e registrare altre 6 immagini (calibrazione massima di 3 minuti).
- Aspirare il buffer dalla camera di imaging e sostituirlo con 1 mL di 1 soluzione DTT. Registra altre 10 immagini (calibrazione minima di 5 minuti).
- Terminare la registrazione e salvare la serie di immagini.
6. Analisi dei dati
- Importazione di dati e pre-elaborazione di immagini in FIJI
- Utilizzare Bio-Formats Importer per aprire un gruppo di immagini dal passaggio 4 o un file di immagine dal passaggio 5. Clicca su Plugin | Bio-Formati | Importatore di bioformati. Nella finestra di dialogo, utilizzare Visualizza stack con: Hyperstack, impostare Modalità colore: predefinita, selezionare Ridimensiona automaticamente e non dividere in finestre separate.
NOTA: la scalabilità automatica ottimizza la visualizzazione dei dati sullo schermo del computer. Non modifica l'intensità dei pixel. - Se sono state aperte singole immagini del passaggio 4, fare clic su Immagine | Stack | Strumenti | Concatena per unirli in una pila di immagini singole.
- Se c'è XY-drift durante la serie di immagini, fai clic su Plugin | StackReg per registrare le immagini. Nella finestra di dialogo, selezionate Corpo rigido (Rigid Body ) o Traslazione (Translation).
- Cambia il formato dell'immagine a 32 bit facendo clic su Immagine | Tipo | 32 bit.
- Dividi i canali di colore in finestre separate facendo clic su Immagine | | colore Canali divisi.
- Selezionare il canale 1 (405 nm) e regolare la soglia per selezionare i mitocondri da analizzare facendo clic su Immagine | Regolare | Soglia. Nella finestra di dialogo, selezionate Predefinito, Rosso, Sfondo scuro e Istogramma impila e attendete che i pixel selezionati appaiano rossi. Fare clic su Applica. Selezionare Imposta pixel di sfondo su NaN ed Elabora tutte le immagini.
NOTA: per evitare potenziali distorsioni dell'osservatore, è necessario utilizzare la determinazione automatica della soglia. FIJI offre diversi metodi automatizzati (come Default, Huang, Intermodes, Otsu) che possono essere selezionati da un menu a discesa nella finestra di dialogo soglia. In genere, il metodo Default fornisce un buon risultato. Si consiglia di confrontare diversi metodi durante la prima analisi per trovare il miglior metodo di soglia per il set di immagini specificato. Una volta scelto un metodo, deve essere applicato a tutte le immagini. - Ripetere il passaggio 6.1.6 per il canale 2 (488 nm).
- Creare un'immagine del rapporto per visualizzare il rapporto 405:488 nm facendo clic su Elabora | Calcolatore di immagini. Nella finestra di dialogo, selezionare Immagine 1: canale 1, Operazione: Dividi, Immagine 2: canale 2, Crea nuova finestra, Elabora tutte le immagini.
- Modificare la tabella di ricerca dell'immagine del rapporto con lo pseudocolore. Ad esempio, per passare a Fire, fai clic su Immagine | | delle tabelle di ricerca Fuoco.
- Utilizzare Bio-Formats Importer per aprire un gruppo di immagini dal passaggio 4 o un file di immagine dal passaggio 5. Clicca su Plugin | Bio-Formati | Importatore di bioformati. Nella finestra di dialogo, utilizzare Visualizza stack con: Hyperstack, impostare Modalità colore: predefinita, selezionare Ridimensiona automaticamente e non dividere in finestre separate.
- Analisi delle immagini
- Nell'immagine del rapporto, disegna ROI attorno a singole cellule o mitocondri. Dopo aver disegnato ogni ROI, aggiungilo al ROI Manager. Analizza | Strumenti | ROI Manager | Aggiungi. (scorciatoia da tastiera: 'T') Seleziona Mostra tutto.
NOTA: poiché i pixel di sfondo sono stati impostati su 'not a number' (NaN) nei passaggi 6.1.6 e 6.1.7, non influiranno sul risultato della misurazione. Pertanto, è accettabile includere alcuni pixel di sfondo nel ROI. - Misura i rapporti 405:488 delle singole celle facendo clic su ROI Manager | ctrl+A per selezionare tutti i ROI | Per saperne di più | Multi misura. Nella finestra di dialogo, selezionate Misura tutte le sezioni e Una riga per sezione.
- Esporta le misurazioni in un software per fogli di calcolo.
- Selezionare l'immagine a 405 nm. Misurare le intensità di tutti i ROI come nel passaggio 6.2.2. utilizzando i ROI memorizzati nel ROI manager.
- Esporta le misurazioni in un software per fogli di calcolo.
- Selezionare l'immagine a 488 nm. Misurare le intensità di tutti i ROI come nel passaggio 6.2.2. utilizzando i ROI memorizzati nel ROI manager.
- Esporta le misurazioni in un software per fogli di calcolo.
- Salva i ROI per riferimento futuro facendo clic su ROI Manager | ctrl + A per selezionare tutti i ROI | Per saperne di più | Salva.
- Consigliato: Genera grafici di intensità rispetto a quelli temporali delle tracce da 405 e 488 nm. Verificare che non vi sia uno sbiancamento marcato in nessuno dei canali (l'intensità del segnale alla fine della serie di immagini deve essere ≥98% della prima immagine) e che le due tracce si muovano in direzioni opposte durante le risposte del sensore (ad esempio, la traccia di 405 nm dovrebbe aumentare durante l'ossidazione mentre la traccia di 488 nm dovrebbe diminuire).
- Nell'immagine del rapporto, disegna ROI attorno a singole cellule o mitocondri. Dopo aver disegnato ogni ROI, aggiungilo al ROI Manager. Analizza | Strumenti | ROI Manager | Aggiungi. (scorciatoia da tastiera: 'T') Seleziona Mostra tutto.
- Normalizzazione dei dati
- Per ogni ROI dall'immagine del rapporto, determinare il valore massimo durante il trattamento DA (Rmax) e il valore minimo durante il trattamento DTT (Rmin).
- Calcola il rapporto normalizzato come segue:
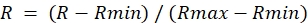
NOTA: questo imposterà il rapporto massimo a 1,0 e il rapporto minimo a 0.
- Analisi della morfologia mitocondriale
- Per ottenere misurazioni della morfologia mitocondriale in parallelo alle intensità roGFP nel passaggio 6.2.6, andare su Analizza | Impostate Misure e selezionate Descrittori forma e Adatta ellisse.
NOTA: Oltre all'intensità media, le misurazioni nella finestra dei risultati includeranno la lunghezza dell'asse maggiore (Maggiore), la lunghezza dell'asse minore (Minore), le proporzioni (AR; asse maggiore diviso per asse minore; i mitocondri rotondi hanno un AR ~ 1, i mitocondri allungati hanno un AR maggiore), nonché le misure di circolarità (Circ.) e rotondità (Round).
- Per ottenere misurazioni della morfologia mitocondriale in parallelo alle intensità roGFP nel passaggio 6.2.6, andare su Analizza | Impostate Misure e selezionate Descrittori forma e Adatta ellisse.
Risultati
Quantificazione delle differenze nello stato redox mitocondriale allo stato stazionario dopo la sospensione del fattore di crescita
Per dimostrare la quantificazione delle differenze allo stato stazionario nello stato redox mitocondriale, i neuroni primari cresciuti in mezzo standard sono stati confrontati con i neuroni coltivati senza fattori di crescita per 48 ore prima dell'imaging. L'astinenza del fattore di crescita provoca la morte delle cellule neuronali apoptotiche dopo 72 h1...
Discussione
Le misurazioni quantitative e dinamiche dello stato redox mitocondriale forniscono importanti informazioni sulla fisiologia mitocondriale e cellulare. Sono disponibili diverse sonde chimiche fluorogeniche che rilevano specie reattive dell'ossigeno, "stress redox" o "stress ossidativo". Tuttavia, questi ultimi termini non sono ben definiti e spesso mancano di specificità9,17,18. Rispetto ai coloranti chimici, Grx1-roGFP2 offre d...
Divulgazioni
Gli autori dichiarano di non avere alcun conflitto di interessi.
Riconoscimenti
Questo lavoro è stato sostenuto dalla Deutsche Forschungsgemeinschaft (BA 3679/5-1; PER 2289: BA 3679/4-2). A.K. è supportato da una borsa di studio ERASMUS+. Ringraziamo Iris Bünzli-Ehret, Rita Rosner e Andrea Schlicksupp per la preparazione dei neuroni primari. Ringraziamo il Dr. Tobias Dick per aver fornito pLPCX-mito-Grx1-roGFP2. Gli esperimenti mostrati nella Figura 4 sono stati eseguiti presso il Nikon Imaging Center, Università di Heidelberg. La figura 2 è stata preparata con BioRender.com.
Materiali
| Name | Company | Catalog Number | Comments |
| reagents | |||
| Calcium chloride (CaCl2·2H2O) | Sigma-Aldrich | C3306 | |
| Diamide (DA) | Sigma-Aldrich | D3648 | |
| Dithiothreitol (DTT) | Carl Roth GmbH | 6908.1 | |
| Glucose (2.5 M stock solution) | Sigma-Aldrich | G8769 | |
| Glucose | Sigma-Aldrich | G7528 | |
| Glycine | neoFroxx GmbH | LC-4522.2 | |
| HEPES (1 M stock solution) | Sigma-Aldrich | 15630-080 | |
| HEPES | Sigma-Aldrich | H4034 | |
| Magnesium chloride (MgCl2·6H2O) | Sigma-Aldrich | 442611-M | |
| N-methyl-D-aspartate (NMDA) | Sigma-Aldrich | M3262 | |
| Potassium chloride (KCl) | Sigma-Aldrich | P3911 | |
| Sodium chloride (NaCl) | neoFroxx GmbH | LC-5932.1 | |
| Sodium pyruvate (0.1 M stock solution) | Sigma-Aldrich | S8636 | |
| Sodium pyruvate | Sigma-Aldrich | P8574 | |
| Sucrose | Carl Roth GmbH | 4621.1 | |
| Tetramethylrhodamine ethyl ester perchlorate (TMRE) | Sigma-Aldrich | 87917 | |
| equipment | |||
| imaging chamber | Life Imaging Services (Basel, Switzerland) | 10920 | Ludin Chamber Type 3 for Ø12mm coverslips |
| laser scanning confocal microscope, microscope | Leica | DMI6000 | |
| laser scanning confocal microscope, scanning unit | Leica | SP8 | |
| peristaltic pump | VWR | PP1080 181-4001 | |
| spinning disc confocal microscope, camera | Hamamatsu | C9100-02 EMCCD | |
| spinning disc confocal microscope, incubationsystem | TokaiHit | INU-ZILCF-F1 | |
| spinning disc confocal microscope, microscope | Nikon | Ti microscope | |
| spinning disc confocal microscope, scanning unit | Yokagawa | CSU-X1 | |
| software | |||
| FIJI | https://fiji.sc | ||
| StackReg plugin | https://github.com/fiji-BIG/StackReg/blob/master/src/main/java/StackReg_.java | ||
| TurboReg plugin | https://github.com/fiji-BIG/TurboReg/blob/master/src/main/java/TurboReg_.java |
Riferimenti
- Roede, J. R., Go, Y. M., Jones, D. P. Redox equivalents and mitochondrial bioenergetics. Methods in Molecular Biology. 810, 249-280 (2012).
- Turrens, J. F. Mitochondrial formation of reactive oxygen species. Journal of Physiology. 552, 335-344 (2003).
- Lin, M. T., Beal, M. F. Mitochondrial dysfunction and oxidative stress in neurodegenerative diseases. Nature. 443 (7113), 787-795 (2006).
- Manfredi, G., Beal, M. F. The role of mitochondria in the pathogenesis of neurodegenerative diseases. Brain Pathology. 10 (3), 462-472 (2000).
- Mari, M., Morales, A., Colell, A., Garcia-Ruiz, C., Fernandez-Checa, J. C. Mitochondrial glutathione, a key survival antioxidant. Antioxidants & Redox Signaling. 11 (11), 2685-2700 (2009).
- Murphy, M. P. Mitochondrial thiols in antioxidant protection and redox signaling: distinct roles for glutathionylation and other thiol modifications. Antioxidants & Redox Signaling. 16 (6), 476-495 (2012).
- Dooley, C. T., et al. Imaging dynamic redox changes in mammalian cells with green fluorescent protein indicators. Journal of Biological Chemistry. 279 (21), 22284-22293 (2004).
- Hanson, G. T., et al. Investigating mitochondrial redox potential with redox-sensitive green fluorescent protein indicators. Journal of Biological Chemistry. 279 (13), 13044-13053 (2004).
- Gutscher, M., et al. Real-time imaging of the intracellular glutathione redox potential. Nature Methods. 5 (6), 553-559 (2008).
- Morgan, B., Sobotta, M. C., Dick, T. P. Measuring E(GSH) and H2O2 with roGFP2-based redox probes. Free Radical Biology & Medicine. 51 (11), 1943-1951 (2011).
- Marwick, K. F. M., Hardingham, G. E. Transfection in primary cultured neuronal cells. Methods in Molecular Biology. 1677, 137-144 (2017).
- Kohrmann, M., et al. convenient, and effective method to transiently transfect primary hippocampal neurons. Journal of Neuroscience Research. 58 (6), 831-835 (1999).
- Depp, C., Bas-Orth, C., Schroeder, L., Hellwig, A., Bading, H. Synaptic activity protects neurons against calcium-mediated oxidation and contraction of mitochondria during excitotoxicity. Antioxidants & Redox Signaling. 29 (12), 1109-1124 (2018).
- Hauck, B., Chen, L., Xiao, W. Generation and characterization of chimeric recombinant AAV vectors. Molecular Therapy. 7 (3), 419-425 (2003).
- Brand, M. D., Nicholls, D. G. Assessing mitochondrial dysfunction in cells. Biochemical Journal. 435 (2), 297-312 (2011).
- Zhang, S. J., et al. Nuclear calcium signaling controls expression of a large gene pool: identification of a gene program for acquired neuroprotection induced by synaptic activity. PLoS Genetics. 5 (8), 1000604 (2009).
- Winterbourn, C. C. The challenges of using fluorescent probes to detect and quantify specific reactive oxygen species in living cells. Biochimica et Biophysica Acta. 1840 (2), 730-738 (2014).
- Sies, H. Oxidative stress: a concept in redox biology and medicine. Redox Biology. 4, 180-183 (2015).
- Lukyanov, K. A., Belousov, V. V. Genetically encoded fluorescent redox sensors. Biochimica et Biophysica Acta. 1840 (2), 745-756 (2014).
- Nietzel, T., et al. Redox-mediated kick-start of mitochondrial energy metabolism drives resource-efficient seed germination. Proceedings of the National Academy of Sciences of the United States of America. 117 (1), 741-751 (2020).
- Albrecht, S. C., et al. Redesign of genetically encoded biosensors for monitoring mitochondrial redox status in a broad range of model eukaryotes. Journal of Biomolecular Screening. 19 (3), 379-386 (2014).
- Albrecht, S. C., Barata, A. G., Grosshans, J., Teleman, A. A., Dick, T. P. In vivo mapping of hydrogen peroxide and oxidized glutathione reveals chemical and regional specificity of redox homeostasis. Cell Metabolism. 14 (6), 819-829 (2011).
- Breckwoldt, M. O., et al. Multiparametric optical analysis of mitochondrial redox signals during neuronal physiology and pathology in vivo. Nature Medicine. 20 (5), 555-560 (2014).
- Ricke, K. M., et al. Mitochondrial dysfunction combined with high calcium load leads to impaired antioxidant defense underlying the selective loss of nigral dopaminergic neurons. Journal of Neuroscience. 40 (9), 1975-1986 (2020).
- Bjornberg, O., Ostergaard, H., Winther, J. R. Mechanistic insight provided by glutaredoxin within a fusion to redox-sensitive yellow fluorescent protein. Biochemistry. 45 (7), 2362-2371 (2006).
- Shokhina, A. G., et al. Red fluorescent redox-sensitive biosensor Grx1-roCherry. Redox Biology. 21, 101071 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon