Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Caracterización tridimensional de sitios de contacto interorgánicos en hepatocitos mediante microscopía electrónica de sección serie
En este artículo
Resumen
Un protocolo simple y completo para adquirir detalles tridimensionales de los sitios de contacto de membrana entre orgánulos en hepatocitos del hígado o células en otros tejidos.
Resumen
La microscopía electrónica de transmisión se ha considerado durante mucho tiempo como el estándar de oro para la visualización de la ultraestructura celular. Sin embargo, el análisis a menudo se limita a dos dimensiones, lo que dificulta la capacidad de describir completamente la ultraestructura tridimensional (3D) y la relación funcional entre los orgánulos. La microscopía electrónica de volumen (vEM) describe una colección de técnicas que permiten el interrogatorio de la ultraestructura celular en 3D a resoluciones de mesoescala, microescala y nanoescala.
Este protocolo proporciona un método accesible y robusto para adquirir datos vEM utilizando la transmisión de sección serie EM (TEM) y cubre los aspectos técnicos del procesamiento de muestras hasta la reconstrucción digital en 3D en un flujo de trabajo único y sencillo. Para demostrar la utilidad de esta técnica, se presenta la relación ultraestructural 3D entre el retículo endoplásmico y las mitocondrias y sus sitios de contacto en los hepatocitos hepáticos. Los contactos interorgánicos desempeñan funciones vitales en la transferencia de iones, lípidos, nutrientes y otras moléculas pequeñas entre orgánulos. Sin embargo, a pesar de su descubrimiento inicial en los hepatocitos, todavía hay mucho que aprender sobre sus características físicas, dinámicas y funciones.
Los contactos interorgánicos pueden mostrar una variedad de morfologías, que varían en la proximidad de los dos orgánulos entre sí (típicamente ~ 10-30 nm) y la extensión del sitio de contacto (desde contactos punteados hasta contactos cisternales 3D más grandes). El examen de contactos cercanos requiere imágenes de alta resolución, y la TEM de sección seriada es muy adecuada para visualizar la ultraestructural 3D de los contactos interorgánicos durante la diferenciación de hepatocitos, así como las alteraciones en la arquitectura de los hepatocitos asociadas con enfermedades metabólicas.
Introducción
Desde su invención en la década de 1930, los microscopios electrónicos han permitido a los investigadores visualizar los componentes estructurales de las células y los tejidos 1,2. La mayoría de las investigaciones han proporcionado información 2D, ya que la construcción de modelos 3D requiere una minuciosa colección de secciones en serie, fotografía manual, procesamiento negativo, rastreo manual y la creación y ensamblaje de modelos 3D a partir de láminas de vidrio, plástico o espuma depoliestireno 3,4. Casi 70 años después, ha habido avances considerables en numerosos aspectos del proceso, desde el rendimiento del microscopio, la recolección de secciones en serie, la imagen digital automatizada, el software y el hardware sofisticados para la reconstrucción, visualización y análisis 3D hasta los enfoques alternativos para lo que ahora se denomina colectivamente EM de volumen (vEM). En general, se considera que estas técnicas de vEM proporcionan información ultraestructural 3D a resoluciones nanométricas a través de escalas de micras y abarcan la microscopía electrónica de transmisión (TEM) y las nuevas técnicas de microscopía electrónica de barrido (SEM); ver reseñas 5,6,7,8.
Por ejemplo, el haz de iones enfocado SEM (FIB-SEM) utiliza un haz de iones enfocado dentro de un SEM para fresar la superficie del bloque entre escaneos secuenciales de imágenes SEM de la superficie del bloque, lo que permite el fresado / imagen automatizado repetido de una muestra y la construcción de un conjunto de datos 3D para la reconstrucción 9,10. Por el contrario, el SEM de cara de bloque serie (SBF-SEM) utiliza un ultramicrotomo dentro del SEM para eliminar el material de la cara del bloque antes de obtener imágenes11,12, mientras que la tomografía de matriz es un proceso no destructivo que requiere la recopilación de secciones en serie, en hojas de cubierta, obleas o cinta, antes de configurar un flujo de trabajo automatizado de imágenes de la región de interés en secciones secuenciales en el SEM para generar el conjunto de datos 3D13 . Similar a la tomografía de matriz, la sección seriada TEM (ssTEM) requiere que las secciones físicas se recopilen antes de la toma de imágenes; sin embargo, estas secciones se recogen en cuadrículas TEM y se visualizan en un TEM 14,15,16. ssTEM se puede ampliar realizando tomografía de inclinación 17,18,19. La tomografía de inclinación en serie proporciona la mejor resolución en x, y y z, y aunque se ha utilizado para reconstruir células enteras20, es razonablemente desafiante. Este protocolo se centra en los aspectos prácticos de ssTEM como la técnica de vEM más accesible disponible para muchos laboratorios de EM que actualmente no tienen acceso a seccionamiento especializado o instrumentos de vEM, pero se beneficiarían de la generación de datos de vEM 3D.
La ultramicrotomía en serie para la reconstrucción 3D se ha considerado previamente un desafío. Era difícil cortar cintas rectas de espesor de sección uniforme, poder organizar y recoger cintas del tamaño correcto, en el orden correcto, en rejillas con suficiente soporte, pero sin barras de rejilla que oscurecieran las regiones de interés, y lo más importante, sin perder secciones, ya que una serie incompleta puede evitar la reconstrucción 3D completa21. Sin embargo, las mejoras en los ultramicrotomías comerciales, los cuchillos de corte y recorte de diamantes22,23, las películas de soporte de electrones en rejillas21,24 y los adhesivos para ayudar a la adhesión de la sección y la preservación de la cinta13,21 son solo algunos de los avances incrementales a lo largo de los años que han hecho que la técnica sea más rutinaria en muchos laboratorios. Una vez que se han recopilado las secciones en serie, las imágenes en serie en TEM son sencillas y pueden proporcionar imágenes EM con tamaños de px del subnanómetro en x e y, lo que permite el interrogatorio de alta resolución de las estructuras subcelulares, un requisito potencial para muchas preguntas de investigación. El estudio de caso aquí presentado demuestra el uso de ssTEM y reconstrucción 3D en el estudio de los contactos endoplásmicos retículo (RE)-orgánulo en hepatocitos hepáticos, donde se observaron por primera vez contactos ER-orgánulos25,26.
Si bien es contiguo con la envoltura nuclear, el RE también hace contactos cercanos con muchos otros orgánulos celulares, incluidos los lisosomas, las mitocondrias, las gotas de lípidos y la membrana plasmática27. Los contactos ER-orgánulos se han implicado en el metabolismo lipídico28, la señalización de fosfoinositida y calcio29, la regulación de la autofagia y la respuesta al estrés30,31. Los contactos ER-orgánulos y otros contactos interorgánicos son estructuras altamente dinámicas que responden a las necesidades metabólicas celulares y las señales extracelulares. Se ha demostrado que varían morfológicamente en su tamaño y forma y las distancias entre las membranas de orgánulos32,33. Se cree que es probable que estas diferencias ultraestructurales reflejen sus diferentes composiciones de proteínas/lípidos y su función34,35. Sin embargo, sigue siendo una tarea difícil definir los contactos interorgánicos y analizarlos36. Por lo tanto, se requiere un protocolo confiable pero simple para examinar y caracterizar los contactos interorgánicos para futuras investigaciones.
Como los contactos ER-orgánulo pueden variar de 10 a 30 nm en la separación de membrana a membrana, el estándar de oro para la identificación ha sido históricamente TEM. Tem de sección delgada ha revelado localización específica de subdominios para proteínas ER residentes en distintos contactos de membrana37. Tradicionalmente, esto ha revelado contactos de orgánulos ER con resolución nm, pero a menudo solo presentaba una vista 2D de estas interacciones. Sin embargo, los enfoques vEM revelan la presentación ultraestructural y el contexto de estos sitios de contacto en 3D, lo que permite la reconstrucción completa de los contactos y una clasificación más precisa de los contactos (punto vs. tubular vs. cisternal-tipo) y cuantificación38,39. Además de ser el primer tipo celular donde se observaron contactos ER-orgánulo25,26, los hepatocitos tienen un extenso sistema de otros contactos interorgánicos que cumplen funciones vitales en su arquitectura y fisiología28,40. Sin embargo, todavía falta una caracterización morfológica completa del orgánulo ER y otros contactos interorgánicos en los hepatocitos. En consecuencia, la forma en que los contactos interorgánicos se forman y remodelan durante la regeneración y la reparación es de particular relevancia para la biología de los hepatocitos y la función hepática.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Todos los animales fueron alojados de acuerdo con las directrices del Ministerio del Interior del Reino Unido, y la recolección de tejidos se llevó a cabo de acuerdo con la Ley de Animales (Procedimientos Científicos) del Reino Unido de 1986.
1. Fijación y preparación de la muestra
- Diseccionar el tejido hepático en trozos de tamaño apropiado, aproximadamente 8 mm x 8 mm x 3 mm, y colocar las piezas en solución salina tamponada con fosfato tibio (PBS, 37 °C).
- Inyecte fijativo a temperatura ambiente (20-25 °C) (glutaraldehído al 1%, sacarosa al 1%, cacodilato de sodio 0,1 M) en las piezas hepáticas y transfiéralas de PBS a fijador durante un máximo de 20 minutos a temperatura ambiente. Mantenga siempre el tejido sumergido en soluciones para evitar que se seque.
NOTA: Los aldehídos son irritantes que son corrosivos y potencialmente cancerígenos. El cacodilato de sodio es tóxico si se ingiere o inhala. Todos los fijadores deben manipularse mientras se usa el equipo de protección personal adecuado, y el experimento debe realizarse en una campana extractora de humos. Una buena fijación dará como resultado un tejido más firme. - Configure el microtomo vibratorio con una cuchilla, un baño de hielo y una bandeja tampón fría llena de PBS. Monte la primera pieza de tejido hepático fijo en un soporte de muestra con pegamento de cianoacrilato y transfiera el bloque al microtomo vibratorio.
- Siguiendo las recomendaciones del fabricante, acérquese al tejido y corte el hígado fijo en rodajas de 100 μm de espesor.
- Recoge las rodajas con una espátula o un pincel natural para el cabello y transfiérelas a un plato de 12 o 24 pocillos que contenga una solución helada (1,5% de glutaraldehído, 0,1 M de cacodilato de sodio) sobre hielo. Deje las rodajas en hielo hasta que todas las muestras hayan sido cortadas y estén listas para ser procesadas aún más.
- Seleccione las rebanadas que contienen las regiones de interés para su posterior procesamiento y lávelas con una agitación suave. Realice tres lavados de 5 minutos con cacodilato de sodio a temperatura ambiente de 0,1 M en un plato de 12 o 24 pocillos, asegurando que las rodajas tengan suficiente amortiguador para moverse libremente.
NOTA: En general, las regiones de interés se seleccionan en función de las características anatómicas relacionadas con la cuestión biológica del estudio y se guían por regiones del tejido que probablemente estén presentes en toda la serie, por ejemplo, no en el borde de la sección, y que están bien conservadas. - En una campana extractora, reemplace el cacodilato de sodio 0.1 M con tetróxido de osmio al 1% recién preparado / ferricianuro de potasio al 1.5%. Coloque el plato de 12 o 24 pocillos en un recipiente sellado y transfiera el recipiente a un refrigerador de productos químicos peligrosos durante 1 h.
NOTA: El osmio es extremadamente peligroso en caso de ingestión, inhalación y contacto con la piel. La ferricianuro de potasio es un irritante y es perjudicial por inhalación y contacto con la piel. Siempre manipule con el equipo de protección personal adecuado y realice el experimento en una campana extractora de humos. - En una campana extractora, retire el tetróxido de osmio/ferricianuro de potasio a una botella de residuos de osmio dedicada y lave las muestras durante 5 minutos con 0,1 M de cacodilato de sodio tres veces. Deje las muestras en un recipiente sellado durante la noche a 4 °C.
NOTA: Posible punto de pausa. Las muestras se pueden almacenar en 0,1 M de cacodilato de sodio en un recipiente sellado a 4 °C durante semanas con poco detrimento de la conservación. Asegúrese de que haya suficiente amortiguador para evitar el secado. - Incubar las muestras con ácido tánico al 1% recién preparado en 0,05 M de cacodilato de sodio durante 45 min en la oscuridad a temperatura ambiente.
NOTA: El ácido tánico es un irritante y puede causar daño ocular. Use el equipo de protección personal adecuado y realice el experimento en una campana extractora de humos. - Realice tres lavados de 5 minutos con ddH2O antes de la deshidratación y la incrustación.
2. Deshidratación de la muestra, incrustación y montaje de resina Epon
- Prepare la resina Epon de acuerdo con la siguiente proporción por peso (consulte el paso 2.2). Tara una balanza con un vaso de precipitados de plástico desechable de 100 ml que contiene una barra de agitación. Corte los extremos de 5 pipetas Pasteur de plástico desechables y úselas para transferir componentes de resina viscosa al vaso de precipitados.
- Agregue secuencialmente 19.2 g de resina-812, 7.6 g de DDSA, 13.2 g de MNA y 0.8 g de acelerador DMP-30 en el vaso de precipitados. Usando la quinta pipeta Pasteur de plástico limpio, mezcle bien los componentes de resina a mano.
NOTA: Evite introducir burbujas, pero asegúrese de mezclar lo suficiente la resina inferior con la parte superior para lograr un cambio de color y una mezcla áspera de las capas de componentes. Todos los componentes de resina son irritantes y son dañinos por inhalación y contacto con la piel. DMP-30 es corrosivo y puede causar corrosión en la piel. Use el equipo de protección personal adecuado. - Coloque el vaso de precipitados en un agitador magnético y deje remover suavemente, mezclando periódicamente la resina manualmente.
- Lavar las muestras con agitación suave durante 5 min con etanol al 70%; repetir una vez.
- Lavar las muestras con agitación suave durante 5 min con etanol al 90%; repetir una vez.
- Lavar las muestras con agitación suave durante 5 min con etanol al 100%; repetir una vez.
- Mientras las muestras están en lavados de etanol al 100% en una campana extractora de humos, prepare una mezcla de óxido de propileno (PO): Epon 50:50 (v/v) en un vial de vidrio con una tapa de plástico resistente al óxido de propileno (PO). Enganche con cuidado pero de forma segura la tapa del vial de vidrio y, mientras mantiene la tapa y el vial, agite o vórtice para mezclar.
NOTA: El óxido de propileno es un irritante inflamable y agudamente tóxico que disuelve algunos plásticos. Use el equipo de protección personal adecuado y realice el experimento en una campana extractora de humos. - Después del paso 2.6, incube las muestras con PO:Epon durante 1 h en un recipiente resistente a PO (por ejemplo, bandejas de aluminio o viales de vidrio), con balanceo/ agitación suave en la campana de humos.
- En la campana extractora, transfiera las muestras al 100% Epon. Incubar durante 2 h a temperatura ambiente en la campana extractora de humos con balanceo/rotación/agitación. Transfiera la mezcla PO:Epon a una botella de residuos epon de vidrio dedicada.
- Repita el paso 2.9 una vez.
- Monte las muestras para incrustarlas. Dependiendo del tamaño de las rebanadas y la región de interés, monte directamente las rodajas en trozos de resina prepolimerizados o incrustelos para su disección y vuelva a incrustarlos en una fecha posterior.
NOTA: Para la incrustación plana, se puede usar una "diapositiva de fundición" para incrustar muchas rebanadas a la vez. La resina sobrante se puede usar para llenar cápsulas de haz y hornearse para hacer trozos prepolimerizados o congelados para su uso posterior. - Una vez montadas y cubiertas con suficiente resina para llenar la cavidad "fundir un portaobjetos", hornee las muestras durante la noche en un horno de 60 °C.
NOTA: Posible punto de pausa. Las muestras se pueden almacenar a temperatura ambiente durante años. - Para volver a incrustar, identifique la región de interés en las rodajas planas de tejido incrustado. Con una sierra de joyero, recorte la pieza de tejido de tamaño apropiado (1 mm2 a 4 mm2) y vuelva a incrustar con resina preparada, como en el paso 2.2, en la parte superior de un bloque prepolimerizado y hornee durante la noche en un horno de 60 ° C.
NOTA: Alternativamente, la pieza de tejido se puede pegar a un talón o pasador con resina epoxi de dos partes. Dejar reposar durante la noche. Posible punto de pausa.
3. Recorte y seccionamiento en serie de muestras incrustadas
NOTA: El seccionamiento es una habilidad aprendida; los usuarios deben ser competentes en la seccionamiento ultrafino antes de intentar el seccionamiento en serie. Como los controles exactos del microtomo varían entre los fabricantes, siga las instrucciones y pautas del fabricante.
- Con la muestra bloqueada en el adaptador de recorte, use una cuchilla de afeitar para recortar cuidadosamente el tejido incrustado de resina para cumplir con los siguientes criterios (consulte la Figura 1A, B):
- Asegúrese de que haya una superficie plana y superior que exponga el tejido alrededor de la región de interés.
- Asegúrese de una forma trapezoidal con los bordes superior e inferior limpios y paralelos.
- Asegurar dimensiones globales de 200-500 μm en x, 100-500 μm en y.
- Asegúrese de una cara de bloque asimétrica, por ejemplo, esquinas laterales derechas de ~90 °, esquina superior izquierda obtusa y esquina inferior izquierda aguda.
NOTA: Una criofenife de recorte puede ser una herramienta alternativa para una cuchilla de afeitar. Las otras recomendaciones son para la conveniencia del usuario para ordenar secciones al crear imágenes. Opcional: Si las secciones no forman cintas estables, se puede aplicar un cemento de contacto al borde delantero de la cara del bloque para ayudar a la formación de la cinta. Las cuchillas de afeitar son afiladas; tenga cuidado de sostener la hoja de afeitar de tal manera que es poco probable que los deslizamientos accidentales resulten en daños personales.
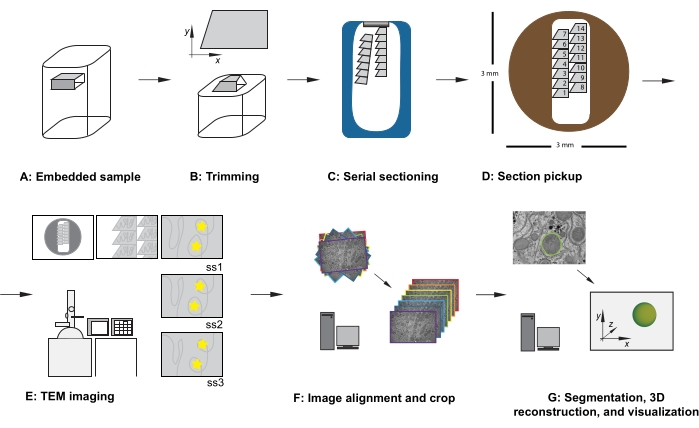
Figura 1: Flujo de trabajo TEM de sección serie. (A) Diagrama de la muestra en el bloque de resina. (B) Bloque de recorte para generar una forma trapezoidal con bordes adecuados para la sección en serie y la cara del bloque asimétrico para garantizar una orientación conocida. (C) Diagrama que muestra cintas de secciones en serie, flotando en la superficie del agua en el bote de cuchillo de diamante. (D) Diagrama que muestra la organización de la sección y la cinta, dictando el orden de las secciones, en una cuadrícula de ranura TEM de 3 mm de diámetro. (E) Imágenes y navegación TEM. Mostrar el orden de la cinta y la sección y usar "pegatinas de estrellas amarillas" en el monitor para hacer referencia a la pantalla para garantizar la reimagen de la misma región de interés en las secciones posteriores. (F) Alineación y recorte de imágenes. (G) Segmentación, reconstrucción 3D y visualización. Abreviatura: TEM = microscopía electrónica de transmisión. Haga clic aquí para ver una versión más grande de esta figura.
- Una vez recortado, transfiera el arco de la muestra, junto con el mandril y la muestra, al brazo de la muestra del microtomo, posicionando el arco de la muestra de modo que el rango de viaje del arco se extienda de arriba a abajo; asegure el arco de la muestra en su lugar.
- Coloque y bloquee el cuchillo de diamante en el soporte del cuchillo, asegurándose de que el ángulo de corte se ajuste adecuadamente al cuchillo. Bloquee el soporte del cuchillo en el escenario de forma segura.
- Con la iluminación del escenario encendida, use el avance del cuchillo mientras verifica constantemente la relación entre la cara del bloque y el filo del cuchillo. Avance cautelosamente el cuchillo hacia la muestra, ajustando continuamente el ángulo lateral del cuchillo, la inclinación de la muestra y la rotación de la muestra ajustando las perillas relevantes hasta que el bloque esté alineado con el filo del cuchillo.
- Apague la iluminación ascendente del escenario; encender el downlighting del escenario; coloque la parte superior e inferior de la ventana de corte del brazo de la muestra; y deje el espécimen justo debajo del filo de la navaja.
- Llene el bote del cuchillo con ddH2O limpio y asegúrese de que la superficie del agua esté nivelada con el filo del cuchillo y ligeramente cóncava.
- Opcional: Sumerja una pestaña en el Triton X-100 al 0.1% y luego en el agua del bote del cuchillo para reducir la tensión superficial del agua para ayudar al cloroformo y la recolección de cinta.
- Prepare la estación de trabajo con pestañas (pestaña pegada a un palo de cóctel), rejillas de ranura recubiertas de formvar, pinzas cruzadas etiquetadas, cloroformo, solución Triton X-100 al 0,1%, agua destilada, papel de filtro y caja de rejilla con notas de caja de rejilla.
- Ajuste la velocidad de corte a 1 mm/s y el espesor de corte inicial a 100 nm y comience el ciclo de corte.
- Después de cortar la primera sección, cambie los parámetros de corte a una velocidad de corte de 0,8 mm / s y el espesor de corte a 70 nm, y continúe cortando, permitiendo que las secciones formen una cinta que se mueve por la superficie del bote de cuchillo lleno de agua (Figura 1C).
NOTA: Es importante tener en cuenta el color de las secciones que se producen, ya que a menudo es una guía más precisa para el grosor de las secciones de resina. Las secciones de plata suelen tener un grosor de alrededor de 70 nm, mientras que las secciones grises son más delgadas y las secciones de oro son más gruesas. - Permita que el microtomo continúe cortando secciones y que la cinta se alargue.
NOTA: Es importante evitar grandes vibraciones y perturbaciones físicas en la habitación. Las corrientes de aire pueden hacer que las secciones se muevan en la superficie del agua en el bote de cuchillos, y las vibraciones físicas pueden hacer que el microtomo corte de manera desigual. - Una vez que se hayan recogido suficientes secciones y antes de que la cinta llegue al final del bote, detenga el corte (justo después de que la muestra haya pasado el filo del cuchillo).
NOTA: El número de secciones necesarias depende del tamaño de la cara del bloque y del tamaño del conjunto de datos que se recopilará. Por lo tanto, es útil ser consciente de la relación entre el tamaño del bloque y la rejilla de ranura a medida que se desprenden las secciones cortadas. - Usando una pestaña en cada mano, rompa suavemente la cinta en cintas más pequeñas que puedan caber en la longitud de la rejilla de la ranura, teniendo cuidado de tomar nota de sus posiciones relativas desde dentro de la muestra.
NOTA: Si su ancho combinado encaja, varias cintas se pueden colocar suavemente una al lado de la otra y recogerse juntas en una sola cuadrícula de ranura. Si recoge varias cintas en una sola cuadrícula de ranura, preste atención al orden y la posición relativa de las cintas. Por ejemplo, coloque siempre las cintas más adentro de la muestra a la derecha de una cinta que ya está en la muestra (Figura 1D). - Opcional: Usando una varilla aplicadora de vidrio, coloque una gota de cloroformo sobre las secciones para aplanarlas.
NOTA: El cloroformo es tóxico e irritante. No deje que el cloroformo toque la superficie o secciones del agua. Si lo hace accidentalmente, el agua debe eliminarse y lavarse el cuchillo antes de volver a la sección. El cloroformo puede dañar las secciones y degradar el pegamento que asegura el diamante en el bote del cuchillo. - Usando las primeras pinzas numeradas, tome la primera rejilla de ranura vacía (en el lado derecho de la ranura, del lado formvar hacia abajo), sumerja suavemente en el Tritón X-100 y luego dos veces en el agua destilada antes de eliminar el exceso de agua del borde de las pinzas con un trozo de papel de filtro.
- Con una pestaña en una mano y los fórceps en la otra, baje suavemente aproximadamente 2/3 de la rejilla de ranura recubierta de formvar en el agua del bote de cuchillo (lejos de las secciones), de modo que el lado de formvar esté hacia abajo, y el borde largo de la ranura a la derecha esté en la superficie del agua y paralelo al borde del agua.
- Mueva suavemente la rejilla en el agua hacia las cintas para que en el golpe de retorno, las secciones se desvíen hacia la rejilla. Continúe haciendo esto en ondulaciones cada vez más pequeñas hasta que el borde derecho de la cinta se alinee con el borde derecho de la ranura. Luego, con el último waft, suba suavemente la rejilla para recoger las secciones en la rejilla de la ranura.
- Deje secar la rejilla en los fórceps antes de guardarla en la caja de rejilla, debidamente anotada en la hoja de referencia de la caja de rejilla.
- Repita el paso 3.16 hasta que se recopilen todas las cintas, lo que garantiza que se mantiene el orden de las cintas.
- Si se requieren más secciones, retraiga el cuchillo 150 nm más o menos, verifique el nivel de agua en el bote y agregue más si es necesario. Comience el proceso de corte nuevamente, siguiendo los pasos 3.11-3.18.
- Una vez que se hayan recogido todas las secciones, asegúrese de que el filo del cuchillo esté libre de escombros de sección, retire el cuchillo de la cara del bloque y retire y limpie el cuchillo.
4. Tinción de la rejilla
- Una vez secas, tiñe las secciones con el citrato de plomo de Reynolds, ya sea en parafilm en el banco o en una placa de Petri. Coloque varios gránulos de hidróxido de sodio debajo de la tapa de una placa de Petri para proporcionar un ambiente libre de dióxido de carbono. Luego, con cuidado, lejos de los gránulos, pipetee gotas de 40 μL de citrato de plomo de Reynolds en la parapelícula, una para cada rejilla.
NOTA: No manche demasiadas rejillas a la vez; por ejemplo, el máximo debe ser 6. Trate de no respirar directamente sobre el plato que mancha. El dióxido de carbono puede reaccionar con el citrato de plomo y causar precipitados no deseados en las rejillas. - Invierta cada rejilla (sección lateral hacia abajo) en la gota de citrato de plomo y déjela protegida por la tapa de la placa de Petri durante 7 a 10 minutos. Mientras las rejillas se tiñen, prepare una segunda pieza más grande de parafilm en el banco con cinco gotas de agua destilada de 300 μL para cada rejilla.
- Al final de la incubación del citrato de plomo, transfiera cada rejilla a una gota de agua destilada para lavar durante 1 minuto sin respirar directamente en las rejillas.
- Repita el paso 4.3 un total de cinco veces.
- Usando pinzas cruzadas numeradas, levante la primera rejilla, toque el borde de la rejilla para filtrar el papel para absorber la mayor parte del agua y deje secar en las pinzas (durante al menos 20 minutos). Repita para cada cuadrícula.
5. Adquisición de imágenes por TEM
NOTA: Como los controles TEM exactos varían entre los fabricantes, siga las instrucciones y directrices del fabricante. Los siguientes pasos deben ser realizados por usuarios que ya son competentes en el uso de TEM.
- Antes de tomar imágenes, realice las comprobaciones habituales, por ejemplo, alineación del haz, referencias de ganancia y eucentricidad de la muestra.
- Cargue cuidadosamente la primera rejilla de secciones en serie en el soporte de la muestra, teniendo cuidado de alinear la ranura (y por lo tanto las secciones) con el eje vertical de la etapa del microscopio.
NOTA: Esta precisión no es esencial, pero ahorra tiempo durante las etapas de adquisición y manejo de datos futuras. Al insertar la cuadrícula (sección lado abajo o sección lado arriba), tenga cuidado de obtener imágenes de todas las cuadrículas en la misma orientación. - Con un aumento bajo, observe el orden, la ubicación y la posición de las secciones en serie (Figura 1E). Desplácese hasta la sección central de la serie en la cuadrícula.
NOTA: Dependiendo del objetivo exacto de la investigación, los enfoques para la obtención de imágenes pueden variar; sin embargo, el siguiente es un punto de partida útil. La forma de las secciones y la relación de las cintas (como se recoge en el paso 3.14) dictan qué sección fue la primera y qué sección fue la última en la cuadrícula. - Examine la muestra e identifique una región de interés. Observe la muestra con el aumento deseado y considere la posibilidad de recopilar la serie con un aumento ligeramente inferior, ya que las secciones a menudo no están perfectamente alineadas y es posible que las imágenes deban recortarse más tarde.
- Tome imágenes de referencia con aumentos más bajos para apreciar el contexto de la región de interés, su ubicación aproximada en diferentes aumentos en relación con los límites de la sección y las características de referencia dentro de la muestra. Utilícelos para firmar la región de interés en otras secciones.
- Opcional: para referenciar la pantalla, use masilla adhesiva reutilizable, pegatinas o un trozo de papel de retroproyector (OHP), pegado a la pantalla, para colocar marcadores temporales en la pantalla para permitir la reimagen rutinaria de las mismas características de la región de interés en el centro de la imagen, en todo el conjunto de datos (consulte estrellas amarillas en la Figura 1E).
- Con las imágenes de referencia, navegue hasta la región de interés en la primera sección de la cuadrícula y adquiera una imagen con el aumento deseado.
NOTA: Al guardar imágenes, anote el primer nombre de archivo de la primera imagen de la serie y utilice la nomenclatura de nomenclatura secuencial para que todos los nombres de imagen sigan el orden secuencial de las secciones serie. - Desplácese hasta la siguiente sección y repita el paso 5.7 hasta que se hayan creado imágenes de todas las secciones para esa región de interés.
6. Exportación de imágenes y registro de alineación de secciones en serie
- Exporte los archivos de imagen que pertenecen a la misma pila en una sola carpeta. Asegúrese de que la carpeta esté ordenada por nombre de archivo.
NOTA: Lo ideal es que las imágenes tengan el mismo nombre de raíz y sigan el orden en que se han adquirido. - Abra Fiji y haga clic en archivo | Importar | Secuencia de imágenes.
- Haga clic en la primera imagen de la carpeta y haga clic en Abrir. Espere a que aparezca una ventana emergente de Opciones de secuencia (Figura 2A).
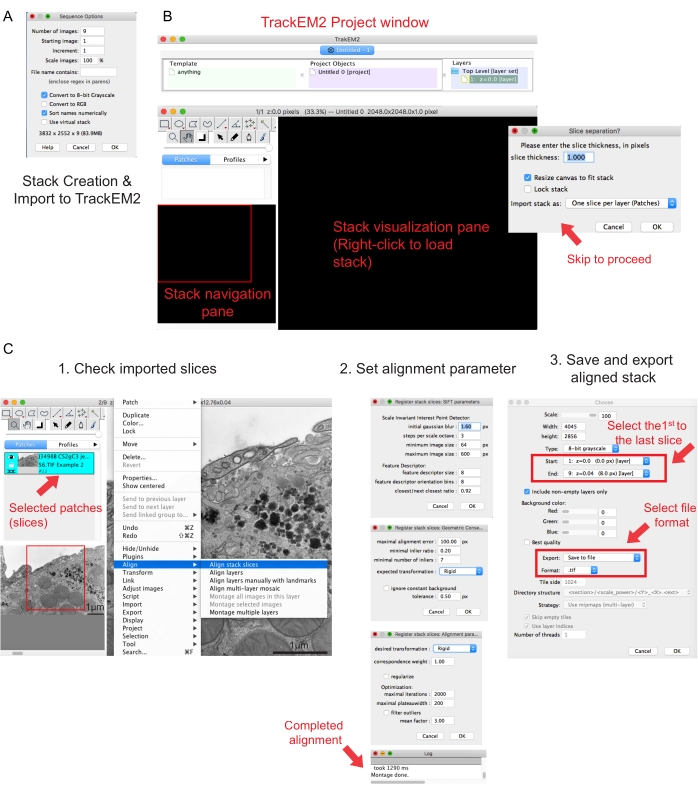
Figura 2: Creación de una pila serie y alineación de sección serie utilizando Fiji. (A) Captura de pantalla que muestra las opciones de secuencia al cargar las imágenes para hacer una pila serie. (B) Captura de pantalla del plugin TrackEM2 y las ventanas clave del plugin. Pulse OK en la separación de sectores para continuar con la alineación. (C) Captura de pantalla después de cargar correctamente la pila serie en el panel de visualización. Tres ventanas secuenciales de parámetros de alineación aparecerán una vez que se seleccione Alinear segmentos de pila . Exporte la pila alineada una vez completada la alineación. Haga clic aquí para ver una versión más grande de esta figura.
- Haga clic en Ordenar nombres numéricamente | Convertir a opción de 8 bits . Pulse OK.
NOTA: La conversión a 8 bits ayuda a la importación de los datos en Amira y reduce el tamaño del archivo, lo que permite velocidades de procesamiento más rápidas en pasos posteriores. - Compruebe la integridad, la secuencia y la ampliación de la pila creada. Guarde la pila creada como un archivo .tif.
NOTA: Las imágenes deberían haberse adquirido con el mismo aumento. - Ejecute el plugin TrakEM241. Ir a archivo | Nuevas | TrackEM2 (en blanco).
NOTA: El plugin pedirá al usuario que guarde los archivos TrackEM2. Si es necesario, guarde los archivos TrackEM2 en la carpeta de imágenes. Deben aparecer tres ventanas: una ventana de proyecto, una ventana de navegación de pila (izquierda) y un panel de visualización de pila (Figura 2B). - Haga clic con el botón secundario en el panel de visualización negro. Haga clic en Importar | Importe la pila y seleccione la pila creada anteriormente.
- Haga clic en Aceptar para cargar la pila en la ventana de navegación de la pila.
NOTA: Aparecerá una ventana de separación de sectores para solicitar la relación de píxeles y dimensiones. Para solo la alineación de la pila, haga clic en Aceptar para omitir este paso. - Utilice el control deslizante para comprobar todos los sectores de la pila. Busque el segmento cargado, que aparecerá como un parche en el plan de navegación. Seleccione los parches que se incluirán en la siguiente alineación.
NOTA: Los parches seleccionados se volverán azules. - Coloque el ratón sobre el panel de visualización. Haga clic con el botón derecho en la imagen, seleccione Alinear | Alinear sectores de pila (Figura 2C-1).
- Especifique los parámetros de alineación a través de un conjunto de tres ventanas secuenciales.
NOTA: Para la mayoría de los datos, comience con una alineación rígida (permite la rotación y la traslación, pero no la transformación) y mantenga otros parámetros como predeterminados (Figura 2C-2). - Permita que la alineación se ejecute hasta que el registro de lectura diga Montaje realizado.
NOTA: El tiempo de ejecución depende del número de vóxeles y la velocidad del equipo. - Compruebe la pila alineada en el panel de visualización. Presione las teclas Alt y - (en PC) o las teclas Ctrl y - (en Mac) para obtener una vista de alejamiento de la pila alineada.
- Si está satisfecho con la pila alineada, haga clic con el botón secundario en Exportar | Crear una imagen plana para guardar la pila alineada.
- Seleccione la primera imagen como inicio de la pila y la última imagen como final de la pila, haga clic en Aceptar (Figura 2C-3). Guarde la pila alineada como .tif.
NOTA: Para reducir el tamaño del archivo, recorte los datos para que solo contengan la región de interés necesaria. - Si es necesario, ejecute una alineación afín en la pila alineada. Abra la pila alineada en Fiji, seleccione Plugin | | de registro StackReg.
- Elija la opción afín y pulse OK. Espere hasta que se complete el programa.
- Guarde la pila alineada afín con un nombre de archivo diferente.
7. Segmentación y reconstrucción 3D
- Abierto Amira42. Haga clic en | de archivos Datos abiertos para cargar la pila alineada.
- Especifique las medidas del vóxel en la nueva ventana emergente (Figura 3A).
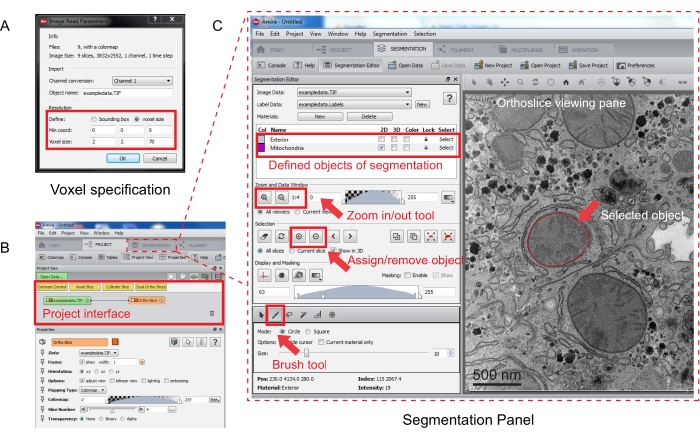
Figura 3: Segmentación de la pila serie usando Amira. (A) La ventana emergente de definición de Voxel antes de cargar una pila alineada. (B) Captura de pantalla de la interfaz del proyecto después de la importación de una pila. Seleccione la ficha Segmentación para iniciar el seguimiento de objetos en el panel Editor de segmentación. (C) Características clave de la pestaña de segmentación. Defina los objetos para la segmentación en la sección Editor de segmentación de la ficha Segmentación. Utilice la función de zoom para ayudar a la identificación de objetos. Seleccione la herramienta Pincel y trace el límite del objeto. Haga clic en el símbolo + en Selección para asignar el seguimiento. Un objeto asignado parecerá tener un límite rojo en el panel de visualización de ortoslice. Haga clic aquí para ver una versión más grande de esta figura.
NOTA: Aparecerá un nodo de pila de imágenes en la interfaz del proyecto y una ortoslice aparecerá en el panel de visualización de la derecha (Figura 3B).
- Para iniciar la segmentación, seleccione la ficha Segmentación (Figura 3B).
NOTA: Se recomienda guardar el progreso de la segmentación antes y durante la segmentación. Ir a | de modelos Guarde el modelo como cualquier archivo .am que se adapte. - Haga clic en Nuevo en el panel del editor de segmentación para definir nuevos objetos en la lista de materiales. Haga clic con el botón secundario para cambiar el color del objeto y haga doble clic para cambiar el nombre del objeto.
- Para la segmentación manual, elija la herramienta de segmentación debajo de la lista de materiales. Seleccione la herramienta Pincel predeterminada para resaltar los píxeles (Figura 3C).
NOTA: Como alternativa, utilice la herramienta pincel para trazar el contorno del objeto y presione Mayús + F para rellenar el objeto. - Para convertir la herramienta de pincel en un borrador, presione continuamente Ctrl mientras selecciona píxeles para su corrección. Anota cada rebanada de la pila.
- Una vez confirmada, asigne la selección a una etiqueta haciendo clic en el signo + . Haga clic en el signo - para eliminar la selección.
- Vuelva a la interfaz del proyecto una vez completada la segmentación. Busque un nodo con una extensión ".label" conectada a la pila de imágenes.
- Haga clic con el botón secundario en la extensión ".label" y seleccione Generar | Aplicar para crear un archivo .surf.
- Para representar el modelo 3D de un objeto segmentado, haga clic con el botón secundario en el archivo .surf y seleccione Vista de superficie para generar un modelo 3D en el panel de visualización.
- Guarde el modelo 3D para su visualización o análisis cuantitativos adicionales.
Access restricted. Please log in or start a trial to view this content.
Resultados
Para esta técnica, las regiones de interés se seleccionan en función del objetivo de la investigación biológica y se identifican antes del recorte y seccionamiento del tejido incrustado. Del mismo modo, el tamaño de la cara del bloque puede ser dictado por la pregunta de investigación; en este caso, la muestra se recortó para dejar una cara de bloque de aproximadamente 0,3 mm x 0,15 mm (Figura 4A). Esto permitió dos rejillas de 9 secciones en serie por rejilla, proporcionando 18 sec...
Access restricted. Please log in or start a trial to view this content.
Discusión
En este protocolo se describe una técnica vEM accesible para visualizar la estructura de orgánulos y las interacciones en 3D. La morfología de los contactos interorgánicos en los hepatocitos se presenta como un estudio de caso aquí. Sin embargo, este enfoque también se ha aplicado para investigar una variedad de otras muestras y áreas de investigación, incluidas las interacciones célula-endotelial de Schwann en nervios periféricos45, la biogénesis corporal de Weibel Palade en células e...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Agradecemos a Joanna Hanley, Rebecca Fiadeiro y Ania Straatman-Iwanowska por su asistencia técnica experta. También agradecemos a los miembros del laboratorio Stefan e Ian J. White por sus útiles discusiones. J.J.B. cuenta con el apoyo de la financiación de MRC para el Laboratorio MRC de Biología Celular Molecular en UCL, el código de premio MC_U12266B. C.J.S. cuenta con el apoyo de la financiación de MRC para la Unidad Universitaria del Laboratorio de Biología Celular Molecular de MRC en UCL, código de premio MC_UU_00012/6. P.G. está financiado por el Consejo Europeo de Investigación, código de subvención ERC-2013-StG-337057.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.22 µm syringe filter | Sarstedt | 83.1826.001 | |
| Aluminum trays | Agar Scientific | AGG3912 | |
| Amira v6 | ThermoFisher | https://www.thermofisher.com | |
| Chloroform | Fisher | C/4960/PB08 | |
| DDSA/Dodecenyl Succinic Anhydride | TAAB | T027 | Epon ingredient |
| Diamond knife | DiaTOME | ultra 45° | |
| DMP-30/2,4,6-tri (Dimethylaminomethyl) phenol | TAAB | D032 | Epon ingredient |
| Dumont Tweezers N5 | Agar Scientific | AGT5293 | |
| Fiji | https://imagej.net/ | ||
| Fiji TrakEM2 plugin | https://imagej.net/ | ||
| Formaldehyde 36% solution | TAAB | F003 | |
| Formvar coated slot grid | Homemade | Alternative: EMS diasum (FF2010-Cu) | |
| Glass bottle with applicator rod | Medisca | 6258 | |
| Glass vials | Fisher Scientific | 15364769 | |
| Gluteraldehyde 25% solution | TAAB | G011 | |
| MNA/Methyl Nadic Anhydride | TAAB | M011 | Epon ingredient |
| Osmium Tetroxide 2% solution | TAAB | O005 | |
| Potassium Ferricyanide | Sigma-Aldrich | P-8131 | |
| Propylene oxide | Fisher Scientific | E/0050/PB08 | |
| Reuseable adhesive | Blue Tack | ||
| Reynolds Lead Citrate | TAAB | L037 | Section stain |
| Sodium Cacodylate | Sigma-Aldrich | C-0250 | to make 0.1 M Caco buffer |
| Super Glue | RS Components | 918-6872 | Cyanoacrylate glue, Step 1.3 |
| TAAB 812 Resin | TAAB | T023 | Epon ingredient |
| Tannic acid | TAAB | T046 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Two part Epoxy Resin | RS Components | 132-605 | Alternative: Step 2.13 |
| Ultramicrotome | Leica | UC7 | |
| Vibrating microtome | Leica | 100 µm thick slices, 0.16 mm/s cutting at 1 mm amplitude . | |
| Weldwood Original Contact cement | DAP | 107 | Contact adhesive: Step 3.1.4 |
Referencias
- Knoll, M., Ruska, E. Das elektronenmikroskop. Zeitschrift für Physik. 78 (5), 318-339 (1932).
- von Ardenne, M. Daselektronen-rastermikroskop. Zeitschrift für Physik. 109 (9), 553-572 (1938).
- Bang, B. H., Bang, F. B. Graphic reconstruction of the third dimension from serial electron microphotographs. Journal of Ultrastructure Research. 1 (2), 138-139 (1957).
- Birch-Andersen, A. Reconstruction of the nuclear sites of Salmonella typhimurium from electron micrographs of serial sections. Journal of General Microbiology. 13 (2), 327-329 (1955).
- Denk, W., Horstmann, H. Serial block-face scanning electron microscopy to reconstruct three-dimensional tissue nanostructure. PLoS Biology. 2 (11), 329(2004).
- Peddie, C. J., Collinson, L. M. Exploring the third dimension: volume electron microscopy comes of age. Micron. 61, 9-19 (2014).
- Titze, B., Genoud, C. Volume scanning electron microscopy for imaging biological ultrastructure. Biology of the Cell. 108 (11), 307-323 (2016).
- Kornfeld, J., Denk, W. Progress and remaining challenges in high-throughput volume electron microscopy. Current Opinion in Neurobiology. 50, 261-267 (2018).
- Heymann, J. A., et al. Site-specific 3D imaging of cells and tissues with a dual beam microscope. Journal of Structural Biology. 155 (1), 63-73 (2006).
- Knott, G., Marchman, H., Wall, D., Lich, B. Serial section scanning electron microscopy of adult brain tissue using focused ion beam milling. Journal of Neuroscience. 28 (12), 2959-2964 (2008).
- Leighton, S. B. SEM images of block faces, cut by a miniature microtome within the SEM - a technical note. Scanning Electron Microscopy. , 73-76 (1981).
- Martone, M. E., Deerinck, T. J., Yamada, N., Bushong, E., Ellisman, M. H. Correlated 3D light and electron microscopy: use of high voltage electron microscopy and electron tomography for imaging large biological structures. Journal of Histotechnology. 23 (3), 261-270 (2000).
- Micheva, K. D., Smith, S. J. Array tomography: a new tool for imaging the molecular architecture and ultrastructure of neural circuits. Neuron. 55 (1), 25-36 (2007).
- Sjostrand, F. S. Ultrastructure of retinal rod synapses of the guinea pig eye as revealed by three-dimensional reconstructions from serial sections. Journal of Ultrastructure Research. 2 (1), 122-170 (1958).
- Ware, R. W. Three-dimensional reconstruction from serial sections. International Review of Cytology. 40, 325(1975).
- Stevens, J. K., Davis, T. L., Friedman, N., Sterling, P. A systematic approach to reconstructing microcircuitry by electron microscopy of serial sections. Cognitive Brain Research. 2 (3), 265-293 (1980).
- Hoppe, W. Three-dimensional electron microscopy. Annual Review of Biophysics. 10, 563-592 (1981).
- Frank, J. Electron tomography: methods for three-dimensional visualization of structures in the cell. , Springer. New York, NY. (2008).
- Baumeister, W. Electron tomography: towards visualizing the molecular organization of the cytoplasm. Current Opinion in Structural Biology. 12 (5), 679-684 (2002).
- Hoog, J. L., Schwartz, C., Noon, A. T., O'Toole, E. T. Organization of interphase microtubules in fission yeast analyzed by electron tomography. Developmental Cell. 12 (3), 349-361 (2007).
- Harris, K. M., Perry, E., Bourne, J., Feinberg, M., Ostroff, L., Hurlburt, J. Uniform serial sectioning for transmission electron microscopy. Journal of Neuroscience. 26 (47), 12101-12103 (2006).
- Jesior, J. C. Use of low-angle diamond knives leads to improved ultrastructural preservation of ultrathin sections. Scanning Microscopy Supplement. 3, 147-152 (1989).
- Studer, D., Gnaegi, H. Minimal compression of ultrathin sections with use of an oscillating diamond knife. Journal of Microscopy. 197, 94-100 (2000).
- Gay, H., Anderson, T. F. Serial sections for electron microscopy. Science. 120 (3130), 1071-1073 (1954).
- Bernhard, W., Rouiller, C. Close topographical relationship between mitochondria and ergastoplasm of liver cells in a definite phase of cellular activity. The Journal of Biophysical and Biochemical Cytology. 2, 73-78 (1956).
- Palade, G. E. An electron microscope study of the mitochondrial structure. The Journal of Histochemistry & Cytochemistry. 1 (4), 188-211 (1953).
- Wu, H., Carvalho, P., Voeltz, G. K. Here, there, and everywhere: The importance of ER membrane contact sites. Science. 361 (6401), (2018).
- Vance, J. E. Inter-organelle membrane contact sites: implications for lipid metabolism. Biology Direct. 15 (1), 24(2020).
- Stefan, C. J. Endoplasmic reticulum-plasma membrane contacts: Principals of phosphoinositide and calcium signaling. Current Opinion in Cell Biology. 63, 125-134 (2020).
- Zaman, M. F., Nenadic, A., Radojicic, A., Rosado, A., Beh, C. T. Sticking with it: ER-PM membrane contact sites as a coordinating nexus for regulating lipids and proteins at the cell cortex. Frontiers in Cell and Developmental Biology. 8, 675(2020).
- van Vliet, A. R., Sassano, M. L., Agostinis, P. The unfolded protein response and membrane contact sites: tethering as a matter of life and death. Contact. 1, 1-15 (2018).
- Cohen, S., Valm, A. M., Lippincott-Schwartz, J. Interacting organelles. Current Opinion in Cell Biology. 53, 84-91 (2018).
- Hariri, H., et al. Lipid droplet biogenesis is spatially coordinated at ER-vacuole contacts under nutritional stress. EMBO Reports. 19 (1), 57-72 (2018).
- Stefan, C. J., Trimble, W. S., Grinstein, S., Drin, G. Membrane dynamics and organelle biogenesis-lipid pipelines and vesicular carriers. BMC Biology. 15 (1), 102(2017).
- Eisenberg-Bord, M., Shai, N., Schuldiner, M., Bohnert, M. A tether is a tether is a tether: tethering at membrane contact sites. Developmental Cell. 39 (4), 395-409 (2016).
- Scorrano, L., De Matteis, M. A., Emr, S., Giordano, F. Coming together to define membrane contact sites. Nature Communications. 10 (1), 1287(2019).
- Lak, B., Li, S., Belevich, I., Sree, S. Specific subdomain localization of ER resident proteins and membrane contact sites resolved by electron microscopy. European Journal of Cell Biology. 100 (7), 151180(2021).
- Collado, J., Kalemanov, M., Campelo, F., Bourgoint, C. Tricalbin-mediated contact sites control ER curvature to maintain plasma membrane integrity. Developmental Cell. 51 (4), 476-487 (2019).
- West, M., Zurek, N., Hoenger, A., Voeltz, G. K. A 3D analysis of yeast ER structure reveals how ER domains are organized by membrane curvature. Journal of Cell Biology. 193 (2), 333-346 (2011).
- Ilacqua, N., Anastasia, I., Raimondi, A., Lemieux, P. A three-organelle complex made by wrappER contacts with peroxisomes and mitochondria responds to liver lipid flux changes. Journal of Cell Science. 135 (5), 259091(2022).
- Cardona, A., Saalfeld, S., Schindelin, J., Arganda-Carreras, I. TrakEM2 software for neural circuit reconstruction. PLoS One. 7 (6), 38011(2012).
- Stalling, D., Westerhoff, M., Hege, H. -C. Amira: A highly interactive system for visual data analysis. The Visualization Handbook. 38, 749-767 (2005).
- Hsieh, T. S., Chen, Y. J., Chang, C. L., Lee, W. R., Liou, J. Cortical actin contributes to spatial organization of ER-PM junctions. Molecular Biology of the Cell. 28 (23), 3171-3180 (2017).
- Anastasia, I., Ilacqua, N., Raimondi, A., Lemieux, P. Mitochondria-rough-ER contacts in the liver regulate systemic lipid homeostasis. Cell Reports. 34 (11), 108873(2021).
- Cattin, A. L., Burden, J. J., Van Emmenis, L., Mackenzie, F. E. Macrophage-Induced Blood Vessels Guide Schwann Cell-Mediated Regeneration of Peripheral Nerves. Cell. 162 (5), 1127-1139 (2015).
- Lopes-da-Silva, M., et al. A GBF1-dependent mechanism for environmentally responsive regulation of ER-Golgi transport. Developmental Cell. 49 (5), 786-801 (2019).
- Banushi, B., Forneris, F., Straatman-Iwanowska, A., Strange, A. Regulation of post-Golgi LH3 trafficking is essential for collagen homeostasis. Nature Communications. 7, 12111(2016).
- Rey, S. A., et al. Ultrastructural and functional fate of recycled vesicles in hippocampal synapses. Nature Communications. 6, 8043(2015).
- Belicova, L., Repnik, U., Delpierre, J., Gralinska, E. Anisotropic expansion of hepatocyte lumina enforced by apical bulkheads. Journal of Cell Biology. 220 (10), 202303003(2021).
- Kizilyaprak, C., Daraspe, J., Humbel, B. M. Focused ion beam scanning electron microscopy in biology. Journal of Microscopy. 254 (3), 109-114 (2014).
- Xu, C. S., Hayworth, K. J., Lu, Z., Grob, P. Enhanced FIB-SEM systems for large-volume 3D imaging. Elife. 6, 1-36 (2017).
- Parlakgül, G., Arruda, A. P., Cagampan, E., Pang, S. High resolution 3D imaging of liver reveals a central role for subcellular architectural organization in metabolism. bioRxiv. , (2020).
- Guerin, C. J., Kremer, A., Borghgraef, P., Lippens, S. Targeted studies using serial block face and focused ion beam scan electron microscopy. The Journal of Visualized Experiments: JoVE. (150), e59480(2019).
- Kremer, A., et al. A workflow for 3D-CLEM investigating liver tissue. Journal of Microscopy. 281 (3), 231-242 (2021).
- Hayat, M. Principles and techniques of electron microscopy: biological applications. , Cambridge University Press. (2000).
- Wisse, E., Braet, F., Duimel, H., Vreuls, C. Fixation methods for electron microscopy of human and other liver. World Journal of Gastroenterology. 16 (23), 2851-2866 (2010).
- Hanley, J., Dhar, D. K., Mazzacuva, F., Fiadeiro, R. Vps33b is crucial for structural and functional hepatocyte polarity. Journal of Hepatology. 66 (5), 1001-1011 (2017).
- Deerinck, T. J., Bushong, E. A., Thor, A., Ellisman, M. H. NCMIR methods for 3D EM: a new protocol for preparation of biological specimens for serial block face scanning electron microscopy. Microscopy. 1, 6-8 (2010).
- Miranda, K., Girard-Dias, W., Attias, M., de Souza, W., Ramos, I. Three dimensional reconstruction by electron microscopy in the life sciences: An introduction for cell and tissue biologists. Molecular Reproduction and Development. 82 (7-8), 530-547 (2015).
- Yamaguchi, M., Chibana, H. A method for obtaining serial ultrathin sections of microorganisms in transmission electron microscopy. The Journal of Visualized Experiments: JoVE. (131), e56235(2018).
- Hall, D. H., Hartwieg, E., Nguyen, K. C. Modern electron microscopy methods for C. elegans. Methods in Cell Biology. 107, 93-149 (2012).
- Hagler, H. K. Ultramicrotomy for biological electron microscopy. Methods in Molecular Biology. 369, 67-96 (2007).
- Arganda-Carreras, I., et al. Consistent and elastic registration of histological sections using vector-spline regularization. Computer vision approaches to medical image analysis, CVAMIA 2006, Lecture Notes in Computer Science. Beichel, R. R., Sonka, M., et al. 4241, Springer Berlin Heidelberg. Berlin, Heidelberg. 85-95 (2006).
- Belevich, I., Joensuu, M., Kumar, D., Vihinen, H., Jokitalo, E. Microscopy image browser: a platform for segmentation and analysis of multidimensional datasets. PLoS Biology. 14 (1), 1002340(2016).
- Fiala, J. C. Reconstruct: a free editor for serial section microscopy. Journal of Microscopy. 218, 52-61 (2005).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD). Journal of Structural Biology. 116 (1), 71-76 (1996).
- Iudin, A., Korir, P. K., Salavert-Torres, J., Kleywegt, G. J., Patwardhan, A. EMPIAR: a public archive for raw electron microscopy image data. Nature Methods. 13 (5), 387-388 (2016).
- Xu, C. S., Pang, S., Shtengel, G., Muller, A. An open-access volume electron microscopy atlas of whole cells and tissues. Nature. 599 (7883), 147-151 (2021).
- Karabag, C., et al. Semantic segmentation of HeLa cells: An objective comparison between one traditional algorithm and four deep-learning architectures. PLoS One. 15 (10), 0230605(2020).
- Heinrich, L., Bennett, D., Ackerman, D., Park, W. Whole-cell organelle segmentation in volume electron microscopy. Nature. 599 (7883), 141-146 (2021).
- Kim, J. S., Greene, M. J., Zlateski, A., Lee, K. Space-time wiring specificity supports direction selectivity in the retina. Nature. 509 (7500), 331-336 (2014).
- Spiers, H., Songhurst, H., Nightingale, L., de Folter, J. Deep learning for automatic segmentation of the nuclear envelope in electron microscopy data, trained with volunteer segmentations. Traffic. 22 (7), 240-253 (2021).
- Hasan, N. M., Gupta, A., Polishchuk, E., Yu, C. H. Molecular events initiating exit of a copper-transporting ATPase ATP7B from the trans-Golgi network. The Journal of Biological Chemistry. 287 (43), 36041-36050 (2012).
- Stoeck, I. K., Lee, J. Y., Tabata, K., Romero-Brey, I. Hepatitis C virus replication depends on endosomal cholesterol homeostasis. The Journal of Virology. 92 (1), 01196(2018).
- Ma, X., Qian, H., Chen, A., Ni, H. M., Ding, W. X. Perspectives on mitochondria-ER and mitochondria-lipid droplet contact in hepatocytes and hepatic lipid metabolism. Cells. 10 (9), 2273(2021).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados