JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
직렬 섹션 전자 현미경을 사용하여 간세포에서 소기관 간 접촉 부위의 입체 특성화
요약
간세포의 소기관 또는 다른 조직의 세포 사이의 막 접촉 부위의 입체 세부 사항을 획득하는 간단하고 포괄적 인 프로토콜.
초록
투과 전자 현미경 검사는 오랫동안 세포 초 구조의 시각화를위한 황금 표준으로 간주되어 왔습니다. 그러나 분석은 종종 두 차원으로 제한되어 3차원(3D) 초구조와 소기관 간의 기능적 관계를 완전히 설명하는 능력을 방해합니다. 부피 전자 현미경 (vEM)은 메조 스케일, 마이크로 스케일 및 나노 스케일 해상도에서 3D로 세포 초 구조의 심문을 가능하게하는 기술 모음을 설명합니다.
이 프로토콜은 TEM(직렬 섹션 전송 EM)을 사용하여 vEM 데이터를 획득할 수 있는 액세스 가능하고 강력한 방법을 제공하며 샘플 처리부터 디지털 3D 재구성에 이르는 기술적 측면을 하나의 간단한 워크플로우에서 다룹니다. 이 기술의 유용성을 입증하기 위해, 소포체와 미토콘드리아와 간 간세포에서의 접촉 부위 사이의 3D 초구조적 관계가 제시된다. 소기관 간 접촉은 소기관 사이의 이온, 지질, 영양소 및 기타 소분자의 전달에 중요한 역할을합니다. 그러나 간세포에서의 초기 발견에도 불구하고 물리적 특징, 역학 및 기능에 대해 배울 것이 여전히 많습니다.
소기관 간 접촉은 두 소기관의 서로 근접성 (일반적으로 ~ 10-30nm)과 접촉 부위의 범위 (펑크 접촉에서 더 큰 3D cisternal-like 접촉에 이르기까지)에 따라 다양한 형태를 표시 할 수 있습니다. 밀접한 접촉을 검사하려면 고해상도 이미징이 필요하며 직렬 섹션 TEM은 간세포 분화 중 소기관 간 접촉의 3D 초구조뿐만 아니라 대사성 질환과 관련된 간세포 구조의 변화를 시각화하는 데 매우 적합합니다.
서문
1930 년대에 발명 된 이래로 전자 현미경을 통해 연구자들은 세포와 조직의 구조적 구성 요소 1,2를 시각화 할 수있었습니다. 대부분의 조사는 3D 모델을 구축하기 위해 힘든 직렬 섹션 수집, 수동 사진, 네거티브 처리, 수동 추적 및 유리, 플라스틱 또는 스티로폼 3,4 시트에서 3D 모델의 생성 및 조립이 필요하기 때문에 2D 정보를 제공했습니다. 거의 70년이 지난 지금, 현미경 성능, 직렬 섹션 수집, 자동화된 디지털 이미징, 3D 재구성, 시각화 및 분석을 위한 정교한 소프트웨어 및 하드웨어에서부터 현재 총칭되는 볼륨 EM(vEM)에 대한 대안적인 접근법에 이르기까지 프로세스의 다양한 측면에서 상당한 발전이 있었습니다. 이러한 vEM 기술은 일반적으로 미크론 스케일에 걸쳐 나노미터 해상도에서 3D 초구조 정보를 제공하는 것으로 간주되며 투과 전자 현미경 (TEM) 및 새로운 주사 전자 현미경 (SEM) 기술을 포함합니다. 리뷰 5,6,7,8 참조.
예를 들어, 집속 이온 빔 SEM (FIB-SEM)은 SEM 내부에 집중된 이온 빔을 사용하여 블록 표면의 순차적 SEM 이미징 스캔 사이에서 블록 표면을 밀링하여 샘플의 반복적인 자동 밀링 / 이미징을 허용하고재구성 9,10을위한 3D 데이터 세트를 구축 할 수 있습니다. 대조적으로, 직렬 블록 페이스 SEM(SBF-SEM)은 이미징(11,12) 이전에 블록 페이스로부터 재료를 제거하기 위해 SEM 내부의 초미세톰을 사용하는 반면, 어레이 단층 촬영은 3D 데이터 세트(13)를 생성하기 위해 SEM의 순차적 섹션에 관심 영역을 이미징하는 자동화된 워크플로우를 설정하기 전에 커버슬립, 웨이퍼 또는 테이프 상으로 직렬 섹션을 수집해야 하는 비파괴 프로세스이다. . 어레이 단층 촬영과 마찬가지로 직렬 섹션 TEM (ssTEM)은 이미징 전에 물리적 섹션을 수집해야합니다. 그러나 이러한 섹션은 TEM 그리드에서 수집되고 TEM14,15,16으로 이미지화됩니다. ssTEM은 틸트 단층 촬영17,18,19를 수행함으로써 확장될 수 있다. 직렬 틸트 단층 촬영은 x, y 및 z에서 최상의 해상도를 제공하며, 전체 셀(20)을 재구성하는 데 사용되었지만, 이는 합리적으로 도전적이다. 이 프로토콜은 현재 특수 단면화 또는 vEM 계측기에 액세스할 수 없지만 3D vEM 데이터 생성의 이점을 누릴 수 있는 많은 EM 실험실에서 사용할 수 있는 가장 접근하기 쉬운 vEM 기술로서 ssTEM의 실용적인 측면에 초점을 맞춥니다.
3D 재구성을위한 직렬 초미세 절제술은 이전에 도전적인 것으로 간주되었습니다. 짝수 단면 두께의 직선 리본을 절단하는 것이 어려웠고, 정확한 순서로, 정확한 크기의 리본을 충분한 지지를 받는 격자 상에 배열하고 픽업할 수 있어야 했지만, 관심 영역을 가리는 격자 막대가 없고, 가장 중요한 것은, 단면을 잃지 않고, 불완전한 시리즈가 완전한 3D 재구성(21)을 방지할 수 있기 때문이다. 그러나 상업용 초미세 토메, 다이아몬드 절단 및 트리밍 나이프 22,23, 그리드21,24의 전자 광택 지지 필름, 섹션 접착 및 리본 보존 보조 접착제 13,21에 대한 개선은 수년 동안 많은 실험실에서 기술을 더욱 일상적으로 만든 점진적 발전 중 일부에 지나지 않습니다. 직렬 섹션이 수집되면 TEM의 직렬 이미징이 간단하며 x 및 y의 서브나노미터 px 크기의 EM 이미지를 제공할 수 있으므로 많은 연구 질문에 대한 잠재적 요구 사항인 하위 세포 구조의 고해상도 심문이 가능합니다. 여기에 제시된 사례 연구는 ER - 소기관 접촉이 처음 관찰 된 간 간세포에서 소포체 (ER) - 소기관 접촉 연구에서 ssTEM 및 3D 재구성의 사용을 보여줍니다25,26.
핵 외피와 인접하는 동안, ER은 또한 리소좀, 미토콘드리아, 지질 방울, 및 원형질막(27)을 포함하는 수많은 다른 세포 소기관과 밀접한 접촉을 한다. ER-소기관 접촉은 지질 대사28, 포스포이노시타이드 및 칼슘 신호전달(29), 자가포식 조절, 및 스트레스 반응30,31에 연루되어 있다. ER-소기관 접촉 및 기타 소기관 간 접촉은 세포 대사 요구와 세포외 단서에 반응하는 매우 역동적인 구조이다. 그들은 크기와 모양, 그리고 소기관막 사이의 거리(32,33)에서 형태학적으로 변하는 것으로 나타났다. 이러한 초구조적 차이는 그들의 상이한 단백질/지질 조성 및 기능(34,35)을 반영할 가능성이 높다고 생각된다. 그러나, 소기관 간 접촉을 정의하고 이들을 분석하는 것은 여전히 어려운 과제이다(36). 따라서 추가 조사를 위해 소기관 간 접촉을 검사하고 특성화하기위한 신뢰할 수 있지만 간단한 프로토콜이 필요합니다.
ER-소기관 접촉은 멤브레인 대 멤브레인 분리에서 10 내지 30 nm의 범위를 가질 수 있기 때문에, 식별을 위한 황금 표준은 역사적으로 TEM이었다. 얇은 절편 TEM은 별개의 막 접촉부(37)에서 상주 ER 단백질에 대한 특이적 하위도메인 국재화를 밝혀냈다. 전통적으로 이것은 nm 분해능을 가진 ER - 소기관 접촉을 밝혀 냈지만 종종 이러한 상호 작용에 대한 2D 뷰 만 제시했습니다. 그러나 vEM 접근법은 이러한 접촉 부위의 초구조적 표현 및 컨텍스트를 3D로 밝혀 접점을 완전히 재구성하고 접점의 더 정확한 분류 (점 대 관형 대 수흉골 유사) 및 정량화38,39를 가능하게합니다. ER-소기관 접촉이 관찰된 최초의 세포 유형인 것 외에도(25,26), 간세포는 그들의 건축과 생리학 28,40에서 중요한 역할을 하는 다른 소기관 간 접촉의 광범위한 시스템을 가지고 있다. 그러나, 간세포에서의 ER-소기관 및 다른 소기관 간 접촉의 철저한 형태학적 특성화는 여전히 부족하다. 따라서, 재생 및 복구 동안 소기관 간 접촉이 형성되고 리모델링되는 방식은 간세포 생물학 및 간 기능과 특히 관련이 있습니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
모든 동물은 영국 내무부 지침에 따라 수용되었으며, 조직 수확은 영국 동물 (과학 절차) 법 1986에 따라 수행되었습니다.
1. 시편 고정 및 준비
- 간 조직을 적절한 크기의 조각, 대략 8 mm x 8 mm x 3 mm로 해부하고, 그 조각을 따뜻한 인산염 완충 식염수 (PBS, 37°C)에 넣는다.
- 실온 (20-25 °C) 고정제 (1 % 수크로오스 중 1.5 % 글루타르 알데히드, 0.1 M 카코 딜 레이트 나트륨)를 간 조각에 주입하고 PBS에서 실온에서 최대 20 분 동안 고정제로 옮깁니다. 건조를 피하기 위해 항상 조직을 용액에 담그십시오.
참고: 알데히드는 부식성이 있고 발암성 가능성이 있는 자극제입니다. 카코딜레이트 나트륨은 삼키거나 흡입하면 독성이 있습니다. 모든 고정 장치는 적절한 개인 보호 장비를 착용하는 동안 처리되어야하며 실험은 흄 후드에서 수행되어야합니다. 좋은 고정은 더 단단한 조직을 초래할 것입니다. - 진동하는 마이크로톰을 블레이드, 얼음 욕조 및 차가운 PBS로 채워진 버퍼 트레이로 설정합니다. 고정 된 간 조직의 첫 번째 조각을 시아 노 아크릴레이트 접착제로 표본 홀더에 장착하고 블록을 진동하는 마이크로 톰으로 옮깁니다.
- 제조업체의 권장 사항에 따라 조직에 접근하여 고정 된 간을 100 μm 두께의 조각으로 슬라이스하십시오.
- 주걱 또는 천연 헤어 페인트 브러시를 사용하여 조각을 수집하고 얼음 위에 얼음 차가운 픽스 (1.5 % 글루타르 알데히드, 0.1M 카코 딜레이트 나트륨)가 들어있는 12 또는 24 웰 접시에 옮깁니다. 모든 샘플이 슬라이스되고 더 이상 처리 될 준비가 될 때까지 슬라이스를 얼음 위에 두십시오.
- 추가 처리를 위해 관심 영역이 포함 된 조각을 선택하고 부드러운 교반으로 씻으십시오. 12 또는 24 웰 접시에서 실온 0.1 M 소듐 카코딜레이트로 3, 5 분 동안 세척을 수행하여 슬라이스가 자유롭게 움직일 수있는 충분한 버퍼를 갖도록 하십시오.
참고: 일반적으로, 관심 영역은 연구의 생물학적 문제와 관련된 해부학적 특징에 기초하여 선택되고, 전체 시리즈에 존재할 가능성이 있는 조직의 영역, 예를 들어, 섹션의 가장자리에 있지 않고, 잘 보존되어 있는 조직에 의해 안내된다. - 흄 후드에서 0.1M 소듐 카코딜레이트를 갓 준비한 1% 오스뮴 테트록사이드/1.5% 칼륨 페리시아나이드로 교체하십시오. 12 웰 또는 24 웰 접시를 밀폐 된 용기에 넣고 용기를 1 시간 동안 유해 화학 물질 냉장고로 옮깁니다.
참고: 오스뮴은 섭취, 흡입 및 피부 접촉의 경우 매우 위험합니다. 칼륨 페리시아나이드는 자극제이며 흡입과 피부 접촉에 의해 해롭다. 항상 적절한 개인 보호 장비를 사용하여 취급하고 흄 후드에서 실험을 수행하십시오. - 흄 후드에서 오스뮴 테트록사이드/칼륨 페리시아나이드를 전용 오스뮴 폐기물 병에 넣고 0.1M 카코딜레이트 나트륨으로 5분 동안 샘플을 세 번 세척합니다. 샘플을 4°C에서 하룻밤 동안 밀봉된 용기에 보관한다.
참고: 잠재적 일시 중지 지점입니다. 샘플은 보존에 거의 해가 없는 몇 주 동안 4°C에서 밀봉된 용기 내의 0.1 M 소듐 카코딜레이트에 저장될 수 있다. 건조를 방지하기에 충분한 완충액이 있는지 확인하십시오. - 샘플을 0.05 M 소듐 카코딜레이트 중의 갓 제조된 1% 탄닌산으로 실온에서 어둠 속에서 45분 동안 인큐베이션한다.
참고: 탄닌산은 자극제이며 눈 손상을 일으킬 수 있습니다. 적절한 개인 보호 장비를 착용하고 흄 후드에서 실험을 수행하십시오. - 탈수 및 임베딩 전에ddH2O로 3 5 분 세척을 수행하십시오.
2. 샘플 탈수, Epon 수지 임베딩 및 장착
- 하기 중량비에 따라 에폰 수지를 준비한다(단계 2.2 참조). 교반 막대가 들어있는 100 mL 일회용 플라스틱 비커와 균형을 맞춥니다. 5 일회용 플라스틱 파스퇴르 피펫에서 끝을 자르고 점성 수지 성분을 비이커로 옮기는 데 사용하십시오.
- 19.2 g의 수지-812, 7.6 g의 DDSA, 13.2 g의 MNA 및 0.8 g의 DMP-30 가속기를 비이커에 순차적으로 첨가한다. 다섯 번째 깨끗한 플라스틱 파스퇴르 피펫을 사용하여 수지 성분을 손으로 철저히 섞으십시오.
참고: 거품 도입은 피하되 하단 수지와 상단을 충분히 혼합하여 색상 변화와 부품 층의 거친 혼합을 달성하십시오. 모든 수지 성분은 자극제이며 흡입 및 피부 접촉에 의해 해롭다. DMP-30은 부식성이 있으며 피부 부식을 일으킬 수 있습니다. 적절한 개인 보호 장비를 착용하십시오. - 비커를 마그네틱 교반기에 놓고 부드럽게 저어주고 주기적으로 수지를 수동으로 혼합합니다.
- 샘플을 70% 에탄올로 5분 동안 완만하게 교반하면서 세척하고; 한 번 반복하십시오.
- 샘플을 90% 에탄올로 5분 동안 완만하게 교반하면서 세척하고; 한 번 반복하십시오.
- 샘플을 100% 에탄올로 5분 동안 완만한 교반으로 세척하고; 한 번 반복하십시오.
- 샘플이 흄 후드에서 100 % 에탄올 세척을하는 동안 프로필렌 옥사이드 (PO) 내성 플라스틱 뚜껑이있는 유리 바이알에 50:50 (v / v) 프로필렌 옥사이드 (PO) : 에폰 혼합물을 준비하십시오. 유리 바이알 뚜껑을 조심스럽게 그러나 단단히 클립하고 뚜껑과 바이알을 모두 잡고 흔들거나 와류하여 혼합하십시오.
참고: 프로필렌 옥사이드는 일부 플라스틱을 용해시키는 매우 독성이 강하고 가연성 자극제입니다. 적절한 개인 보호 장비를 착용하고 흄 후드에서 실험을 수행하십시오. - 단계 2.6 후에, PO:Epon으로 샘플을 PO 내성 용기 (예를 들어, 알루미늄 트레이 또는 유리 바이알)에서 1 시간 동안 인큐베이션하고, 흄 후드에서 부드럽게 흔들거나 교반시킨다.
- 흄 후드에서 샘플을 100 % Epon으로 옮깁니다. 흄 후드에서 실온에서 2 시간 동안 흔들림 / 회전 / 교반으로 배양하십시오. PO:Epon 혼합물을 전용 유리 Epon 폐기물 병으로 옮깁니다.
- 2.9단계를 한 번 반복합니다.
- 임베딩을 위해 샘플을 마운트합니다. 슬라이스의 크기와 관심 영역에 따라 슬라이스를 사전 중합 된 수지 스터브에 직접 장착하거나 해부를 위해 플랫 임베드하고 나중에 다시 임베드하십시오.
참고: 플랫 임베딩의 경우 "캐스트 a 슬라이드"를 사용하여 한 번에 여러 슬라이스를 임베드할 수 있습니다. 남은 수지는 빔 캡슐을 채우고 미리 중합 된 스텁을 만들기 위해 구워 지거나 나중에 사용하기 위해 냉동 할 수 있습니다. - 일단 장착되고 "캐스트-a 슬라이드" 공동을 채우기에 충분한 수지에 덮이면, 샘플을 60°C 오븐에서 밤새 굽는다.
참고: 잠재적 일시 중지 지점입니다. 샘플은 수년간 실온에서 보관할 수 있습니다. - 재임베딩을 위해, 편평한 포매된 조직 조각에서 관심 영역을 확인한다. 보석상 톱을 사용하여, 적절한 크기(1mm2 내지 4mm2)의 조직 조각을 잘라내고, 단계 2.2에서와 같이 제조된 수지를 사용하여 재임포하고, 예비중합된 블록의 상부에 60°C 오븐에서 밤새 베이킹한다.
참고: 또는 티슈 피스를 두 부분으로 구성된 에폭시 수지로 스텁 또는 핀에 접착할 수 있습니다. 하룻밤 사이에 출발하십시오. 잠재적 일시 중지 지점입니다.
3. 임베디드 샘플의 트리밍 및 직렬 단면화
참고 : 단면화는 배운 기술입니다. 사용자는 직렬 단면화를 시도하기 전에 초박형 단면화에 능숙해야합니다. 정확한 마이크로톰 제어는 제조업체마다 다르므로 제조업체의 지침과 지침을 따르십시오.
- 샘플을 트리밍 어댑터에 고정한 상태에서 면도날을 사용하여 다음 기준을 충족하도록 수지 매립 조직을 조심스럽게 트리밍합니다(그림 1A,B 참조).
- 관심 영역 주위에 조직을 노출시키는 평평한 상부 표면이 있는지 확인하십시오.
- 위쪽과 아래쪽 가장자리가 깨끗하고 평행한 사다리꼴 모양을 확인하십시오.
- x에서 200-500 μm, y에서 100-500 μm의 전체 치수를 보장하십시오.
- 비대칭 블록 면(예: ~90°의 오른쪽 모서리 , 왼쪽 위 모서리 무딘 및 왼쪽 아래쪽 모서리 급성)을 확인합니다.
참고 : 트리밍 냉동 나이프는 면도날의 대체 도구가 될 수 있습니다. 다른 권장 사항은 이미징 할 때 섹션을 주문하기위한 사용자 편의를위한 것입니다. 선택 사항 : 섹션이 안정적인 리본을 형성하지 못하면 리본 형성을 돕기 위해 블록 면의 앞쪽 가장자리에 접촉 시멘트를 적용 할 수 있습니다. 면도날은 날카롭다. 우발적 인 미끄러짐이 개인적인 해를 입지 않도록 면도날을 잡으십시오.
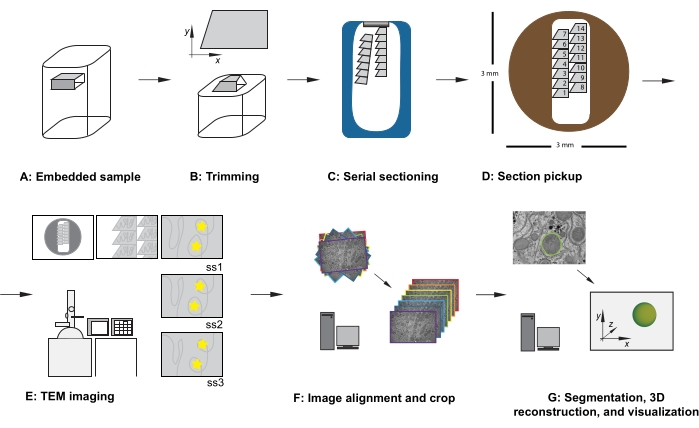
그림 1: 직렬 단면 TEM 워크플로우 . (A) 수지 블록 내의 시편의 다이어그램. (B) 트림 블록은 알려진 방향을 보장하기 위해 직렬 단면화 및 비대칭 블록 면에 적합한 가장자리가있는 사다리꼴 모양을 생성합니다. (C) 다이아몬드 나이프 보트의 물 표면에 떠있는 직렬 섹션의 리본을 보여주는 다이어그램. (D) 직경 3mm TEM 슬롯 그리드에서 단면의 순서를 지시하는 섹션 및 리본 구성을 보여주는 다이어그램. (E) TEM 이미징 및 네비게이션. 리본 및 섹션 순서를 표시하고 모니터에서 "노란색 별 스티커"를 사용하여 화면 참조에 사용하여 후속 섹션에서 동일한 관심 영역을 다시 이미징합니다. (F) 이미지 정렬 및 자르기. (G) 세분화, 3D 재구성 및 시각화. 약어 : TEM = 투과 전자 현미경. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 일단 트리밍되면, 척 및 샘플과 함께 시편 아크를 마이크로톰의 시편 암으로 옮기고 아크의 이동 범위가 위에서 아래로 흐르도록 시편 아크를 배치합니다. 시편 아크를 제자리에 고정하십시오.
- 다이아몬드 나이프를 나이프 홀더에 넣고 잠그고 절단 각도가 칼에 적절하게 설정되었는지 확인하십시오. 나이프 홀더를 스테이지에 안전하게 잠급니다.
- 무대가 위로 켜진 상태에서 칼 전진을 사용하여 블록 면과 칼 가장자리 사이의 관계를 지속적으로 확인합니다. 조심스럽게 칼을 시편 쪽으로 전진시키고, 블록이 칼의 가장자리에 정렬될 때까지 관련 노브를 조정하여 칼의 측면 각도, 시편 기울기 및 시편 회전을 지속적으로 조정합니다.
- 무대 업라이트를 끄십시오. 무대 다운 라이팅을 켭니다. 시편 팔의 절단 창의 상단과 하단을 설정; 표본을 칼 가장자리 바로 아래에 두십시오.
- 칼 보트를 깨끗한 ddH2O로 채우고 물 표면이 칼의 가장자리와 수평이고 약간 오목한지 확인하십시오.
- 선택 사항: 속눈썹을 0.1% Triton X-100에 담근 다음 나이프 보트 물에 담그면 물의 표면 장력을 줄여 클로로포밍 및 리본 픽업을 돕습니다.
- 속눈썹(칵테일 스틱에 붙인 속눈썹), 포름바 코팅 슬롯 그리드, 크로스오버 포셉, 클로로포름, 0.1% Triton X-100 용액, 증류수, 여과지 및 그리드 박스 노트가 있는 그리드 박스로 워크스테이션을 준비합니다.
- 절삭 속도를 1mm/s로 설정하고 초기 절삭 두께를 100nm로 설정하고 절삭 사이클을 시작합니다.
- 첫 번째 단면을 절단한 후 절삭 매개변수를 0.8mm/s의 절삭 속도로, 절단 두께를 70nm로 변경하고 절단을 계속하여 단면이 물이 채워진 나이프 보트의 표면 아래로 이동하는 리본을 형성할 수 있도록 합니다(그림 1C).
참고: 이는 종종 수지 섹션의 두께에 대한 더 정확한 가이드이기 때문에 생성되는 섹션의 색상을 인식하는 것이 중요합니다. 은 섹션은 일반적으로 약 70nm 두께이며 회색 섹션은 더 얇고 금 섹션은 더 두껍습니다. - 마이크로톰이 섹션을 계속 자르고 리본이 길어지도록 허용하십시오.
참고 : 실내에서 큰 진동과 신체적 장애를 피하는 것이 중요합니다. 초안으로 인해 섹션이 나이프 보트의 물 표면에서 움직일 수 있으며 물리적 진동으로 인해 미세 톰이 고르지 않게 절단 될 수 있습니다. - 충분한 섹션이 수집되고 리본이 보트 끝에 도달하기 전에 절단을 중지하십시오 (샘플이 나이프의 가장자리를 통과 한 직후).
참고: 필요한 섹션 수는 블록 면의 크기와 수집할 데이터 세트의 크기에 따라 다릅니다. 따라서, 컷 섹션이 벗겨질 때 블록의 크기와 슬롯 그리드 사이의 관계를 인식하는 것이 유용하다. - 각 손의 속눈썹을 사용하여 리본을 슬롯 그리드의 길이에 맞을 수 있는 작은 리본으로 부드럽게 분할하여 샘플 내에서 상대적인 위치를 기록해 둡니다.
참고: 결합된 너비가 맞으면 여러 리본을 서로 옆에 부드럽게 배치하고 단일 슬롯 그리드에서 함께 선택할 수 있습니다. 단일 슬롯 격자에서 여러 리본을 선택하는 경우 리본의 순서와 상대적 위치에주의하십시오. 예를 들어, 리본을 항상 샘플에 이미 있는 리본의 오른쪽에 있는 샘플에 더 배치합니다(그림 1D). - 선택 사항: 유리 어플리케이터 로드를 사용하여 클로로포름 한 방울을 섹션 위로 가져가 평평하게 만듭니다.
참고: 클로로포름은 독성이 있으며 자극성이 있습니다. 클로로포름이 물 표면이나 부분에 닿지 않도록하십시오. 실수로 물을 제거하고 칼을 씻어 낸 후 절개로 돌아 가야합니다. 클로로포름은 섹션을 손상시키고 다이아몬드를 나이프 보트에 고정시키는 접착제를 저하시킬 수 있습니다. - 첫 번째 번호가 매겨진 포셉을 사용하여 첫 번째 빈 슬롯 그리드 (슬롯의 오른쪽, formvar 측면 아래)를 집어 들고 Triton X-100에 부드럽게 담근 다음 여과지 조각을 사용하여 포셉 가장자리에서 과도한 물을 제거하기 전에 증류수에 두 번 담그십시오.
- 한 손에는 속눈썹을, 다른 손에는 포셉을 사용하여 폼바 코팅 슬롯 그리드의 약 2/3번째 를 칼 보트의 물 (섹션에서 멀리 떨어져)으로 부드럽게 낮추어 formvar 측면이 아래를 향하게하고 슬롯의 오른쪽 긴 가장자리가 물 표면에 있고 물 가장자리와 평행합니다.
- 물 속의 그리드를 리본 쪽으로 부드럽게 흔들어서 복귀 스트로크 시 섹션이 그리드 쪽으로 드리프트되도록 합니다. 리본의 오른쪽 가장자리가 슬롯의 오른쪽 가장자리와 정렬될 때까지 더 작고 작은 웨이트에서 이 작업을 계속합니다. 그런 다음 마지막 waft를 사용하여 그리드를 부드럽게 위로 가져 와서 섹션을 슬롯 그리드로 가져옵니다.
- 그리드 상자에 저장하기 전에 포셉의 그리드를 건조시켜 두고, 그리드 상자 참조 시트에 적절하게 주석을 달으십시오.
- 모든 리본이 수집될 때까지 3.16단계를 반복하여 리본의 순서가 유지되도록 합니다.
- 추가 섹션이 필요한 경우 나이프를 150nm 정도 후퇴시키고 보트의 수위를 확인한 다음 필요한 경우 더 추가하십시오. 3.11-3.18 단계에 따라 절단 공정을 다시 시작하십시오.
- 모든 섹션이 수집되면 나이프 가장자리에 섹션 파편이 없는지 확인하고 칼을 블록 면에서 떼어 내고 칼을 제거하고 청소하십시오.
4. 그리드 염색
- 일단 건조되면, 레이놀즈의 납 구연산염으로 섹션을 벤치의 파라 필름 또는 페트리 접시에 얼룩지게하십시오. 이산화탄소가없는 환경을 제공하기 위해 페트리 접시 뚜껑 아래에 수산화 나트륨 몇 알갱이를 놓습니다. 그런 다음, 조심스럽게 펠릿을 떼어내고, 피펫 40 μL의 레이놀즈의 납 시트레이트를 파라필름 상에 떨어뜨린다.
참고: 한 번에 너무 많은 격자를 얼룩지게 하지 마십시오. 예를 들어, 최대값은 6이어야 합니다. 얼룩진 접시에 직접 숨을 쉬지 마십시오. 이산화탄소는 납 시트레이트와 반응하여 그리드에 원치 않는 침전물을 일으킬 수 있습니다. - 각 그리드 (단면 아래쪽)를 리드 시트레이트 드롭에 뒤집어 놓고 7 ~ 10 분 동안 페트리 접시 뚜껑에 의해 보호하십시오. 그리드가 염색되는 동안, 각 그리드에 대해 다섯 개의 300 μL 증류수 방울로 벤치에 더 큰 두 번째 파라필름 조각을 준비하십시오.
- 납 시트레이트 인큐베이션이 끝나면 각 그리드를 증류수 방울로 옮겨 그리드에서 직접 호흡하지 않고 1 분 동안 세척하십시오.
- 4.3단계를 총 다섯 번 반복합니다.
- 번호가 매겨진 크로스 오버 포셉을 사용하여 첫 번째 그리드를 집어 들고 그리드의 가장자리를 터치하여 용지를 필터링하여 대부분의 물을 윅 아웃하고 포셉에서 건조시킵니다 (최소 20 분 동안). 각 격자선에 대해 이 단계를 반복합니다.
5. TEM에 의한 이미징 획득
참고: 정확한 TEM 제어는 제조업체마다 다르므로 제조업체의 지침과 지침을 따르십시오. 다음 단계는 이미 TEM 사용에 능숙한 사용자가 수행해야 합니다.
- 이미징 전에, 일반적인 검사, 예를 들어, 빔 정렬, 이득 기준, 샘플 공심성을 수행한다.
- 직렬 섹션의 첫 번째 그리드를 시편 홀더에 조심스럽게 적재하여 슬롯 (따라서 섹션)을 현미경 단계의 수직 축에 정렬하도록주의하십시오.
참고: 이 정확도는 반드시 필요한 것은 아니지만 수집 및 향후 데이터 처리 단계에서 시간을 절약할 수 있습니다. 격자선을 삽입할 때(단면을 아래쪽으로 또는 단면을 위쪽으로 향하게) 모든 격자를 동일한 방향으로 이미지화해야 합니다. - 낮은 배율에서 직렬 단면의 순서, 위치 및 위치를 관찰합니다(그림 1E). 그리드에서 계열의 중간 섹션으로 이동합니다.
참고 : 정확한 연구 목표에 따라 이미징에 대한 접근 방식이 다를 수 있습니다. 그러나 다음은 유용한 출발점입니다. 단면의 모양과 리본의 관계(3.14단계에서 선택한 대로)는 그리드에서 어떤 섹션이 첫 번째이고 어떤 섹션이 마지막이었는지를 지정합니다. - 샘플을 탐색하고 관심 영역을 식별합니다. 원하는 배율로 샘플을 관찰하고 단면이 완벽하게 정렬되지 않는 경우가 많으며 나중에 이미지를 잘라야 할 수 있으므로 약간 낮은 배율로 시리즈를 수집하는 것이 좋습니다.
- 더 낮은 배율로 참조 이미지를 촬영하여 관심 영역의 컨텍스트, 섹션 경계와 관련된 다양한 배율에서의 대략적인 위치 및 샘플 내의 랜드마크 특징을 파악합니다. 이를 사용하여 다른 섹션에 관심 영역을 서명-게시합니다.
- 옵션: 화면 참조의 경우 재사용 가능한 접착제 퍼티, 스티커 또는 화면에 테이프로 붙인 오버헤드 프로젝터(OHP) 용지를 사용하여 화면에 임시 마커를 배치하여 데이터 세트 전체에서 이미지 중앙에 있는 관심 영역의 동일한 기능을 일상적으로 재이미징할 수 있도록 합니다( 그림 1E의 노란색 별 참조).
- 참조 이미지를 사용하여 그리드의 첫 번째 섹션에 있는 관심 영역으로 이동하고 원하는 배율로 이미지를 획득합니다.
참고: 이미지를 저장할 때 시리즈의 첫 번째 이미지의 첫 번째 파일 이름을 기록하고 모든 이미지 이름이 직렬 섹션의 순차적 순서를 따르도록 순차적 명명 명명법을 사용합니다. - 다음 섹션으로 이동하여 해당 관심 영역에 대해 모든 섹션이 이미지화될 때까지 5.7단계를 반복합니다.
6. 이미지 내보내기 및 직렬 섹션 정렬 등록
- 동일한 스택에 속하는 이미지 파일을 단일 폴더로 내보냅니다. 폴더가 파일 이름별로 정렬되어 있는지 확인합니다.
참고: 이미지는 이상적으로 동일한 루트 이름을 가져야 하며 획득된 순서를 따라야 합니다. - 피지를 열고 파일 |를 클릭하십시오. 가져오기 | 이미지 시퀀스.
- 폴더의 첫 번째 이미지를 클릭하고 열기를 클릭합니다. 시퀀스 옵션 의 팝업 창이 나타날 때까지 기다립니다(그림 2A).
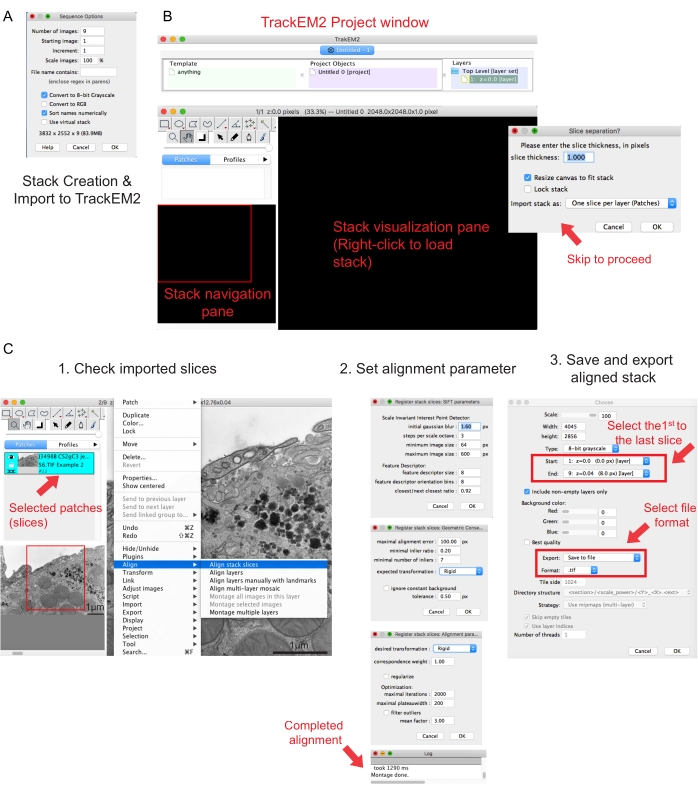
그림 2: 피지를 사용하여 직렬 스택 및 직렬 단면 정렬 만들기. (A) 직렬 스택을 만들기 위해 이미지를 로드할 때 시퀀스 옵션을 보여 주는 스크린샷입니다. (B) TrackEM2 플러그인의 스크린 샷과 플러그인의 주요 창. 슬라이스 분리에서 OK 를 눌러 정렬을 진행합니다. (C) 직렬 스택을 시각화 창에 성공적으로 로드한 후의 스크린샷입니다. 정렬 매개 변수의 세 개의 순차적 창이 나타나며 스택 슬라이스 정렬이 선택되면 나타납니다. 정렬이 완료되면 정렬된 스택을 내보냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 숫자로 | 이름 정렬을 클릭합 8비트 옵션으로 변환합니다. OK를 누릅니다.
참고: 8비트로 변환하면 데이터를 Amira로 가져오고 파일 크기를 줄여 이후 단계에서 처리 속도를 높일 수 있습니다. - 생성된 스택의 완전성, 시퀀스 및 배율을 확인합니다. 생성 된 스택을 .tif 파일로 저장하십시오.
참고: 이미지는 동일한 배율로 획득해야 합니다. - TrakEM2 플러그인41을 실행합니다. 파일 |로 이동 새로운 | 트랙EM2 (공백).
참고 : 플러그인은 사용자에게 TrackEM2 파일을 저장하도록 요청합니다. 필요한 경우 TrackEM2 파일을 이미지 폴더에 저장합니다. 프로젝트 창, 스택 탐색(왼쪽) 창, 스택 시각화 창(그림 2B)의 세 개의 창이 나타납니다. - 검정색 시각화 창을 마우스 오른쪽 단추로 클릭합니다. | 가져오기를 클릭합니다. 스택을 가져오 고 이전에 만든 스택을 선택합니다.
- 확인을 클릭하여 스택을 스택 탐색 창에 로드합니다.
참고: 슬라이스 분리 창이 나타나 픽셀과 치수 관계를 요청합니다. 스택 정렬만 수행하려면 확인을 클릭하여 이 단계를 건너뜁니다. - 슬라이더를 사용하여 스택의 모든 슬라이스를 확인합니다. 탐색 계획에 패치로 표시될 로드된 슬라이스를 찾습니다. 다음 정렬에 포함될 패치를 선택합니다.
참고: 선택한 패치는 파란색으로 바뀝니다. - 보기 창 위로 마우스를 가져갑니다. 이미지를 마우스 오른쪽 단추로 클릭하고 정렬을 선택합| 스택 슬라이스를 정렬합니다(그림 2C-1).
- 세 개의 순차적 창 집합을 통해 정렬 매개변수를 지정합니다.
참고: 대부분의 데이터의 경우 엄격한 정렬(회전 및 변환은 허용되지만 변환은 허용하지 않음)으로 시작하고 다른 매개 변수는 기본값으로 유지합니다(그림 2C-2). - 판독 로그에 Montage 완료가 표시될 때까지 정렬이 실행되도록 허용합니다.
참고: 런타임은 복셀 수와 컴퓨터 속도에 따라 다릅니다. - 보기 창에서 정렬된 스택을 확인합니다. Alt 및 - 키(PC에서) 또는 Ctrl 및 - 키(Mac)를 눌러 정렬된 스택을 축소합니다.
- 정렬된 스택이 만족스러우면 내보내기를 마우스 오른쪽 단추로 클릭 | 플랫 이미지를 만들어 정렬된 스택을 저장합니다.
- 첫 번째 이미지를 스택의 시작으로, 마지막 이미지를 스택의 끝으로 선택하고 확인을 클릭합니다(그림 2C-3). 정렬된 스택을 .tif로 저장합니다.
참고: 파일 크기를 줄이려면 필요한 관심 영역만 포함하도록 데이터를 자릅니다. - 필요한 경우 정렬된 스택에서 affine 정렬을 실행합니다. 피지에서 정렬 된 스택을 열고 플러그인을 선택하십| 등록 | 스택 레지.
- 아파인 옵션을 선택하고 OK를 누릅니다. 프로그램이 완료 될 때까지 기다리십시오.
- 아파인 정렬 스택을 다른 파일 이름으로 저장합니다.
7. 세분화 및 3D 재구성
- 아미라42를 엽니다. 파일 |를 클릭합니다. 데이터를 열어 정렬된 스택을 로드합니다.
- 새 팝업 창에서 복셀 측정값을 지정합니다(그림 3A).
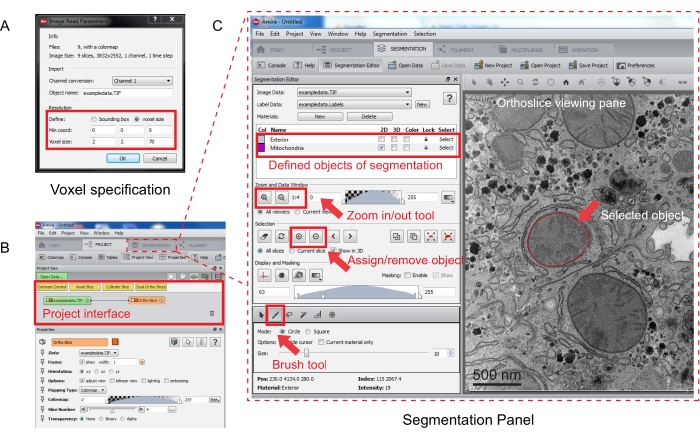
그림 3: Amira를 사용한 직렬 스택 세분화. (A) 정렬된 스택을 로드하기 전의 Voxel 정의 팝업 창. (B) 스택 가져오기 후 프로젝트 인터페이스의 스크린샷. 세그멘테이션 탭을 선택하여 세그멘 테이션 편집기 패널에서 객체 추적을 시작합니다. (C) 세그멘테이션 탭의 주요 기능. 세그멘테이션 탭의 세그멘테이션 편집기 섹션에서 세그멘테이션을 위한 객체를 정의합니다. 확대/축소 기능을 사용하여 객체 식별을 지원합니다. [브러쉬] 도구를 선택하고 객체의 경계를 추적합니다. 선택 아래에서 + 기호를 클릭하여 추적을 할당합니다. 지정된 객체는 직교 슬라이스 보기 창에 빨간색 경계가 있는 것처럼 보입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
참고: 이미지 스택 노드가 프로젝트 인터페이스에 나타나고 오른쪽의 보기 창에 직교 슬라이스가 나타납니다(그림 3B).
- 세그멘테이션을 시작하려면 세 그멘테이션 탭을 선택합니다(그림 3B).
참고: 세그멘테이션 전과 세그멘테이션 중에 세그멘테이션 진행률을 저장하는 것이 좋습니다. 모델 |로 이동 모델을 적합한 .am 파일로 저장합니다. - 세그멘테이션 편집기 패널에서 새로 만들기 를 클릭하여 재료 목록에서 새 객체를 정의합니다. 마우스 오른쪽 단추를 클릭하여 개체의 색상을 변경하고 두 번 클릭하여 개체의 이름을 바꿉니다.
- 수동 세분화의 경우 재료 목록 아래에 있는 세분화 도구를 선택합니다. 기본 [브러쉬] 도구를 선택하여 픽셀을 강조 표시합니다(그림 3C).
참고: 또는 브러시 도구를 사용하여 개체의 윤곽선을 추적하고 Shift + F 를 눌러 개체를 채웁니다. - 브러시 도구를 지우개로 변환하려면 수정할 픽셀을 선택하는 동안 Ctrl 키를 계속 누릅니다. 스택의 모든 슬라이스에 주석을 추가합니다.
- 확인되면 + 기호를 클릭하여 선택 항목을 레이블에 할당합니다. - 기호를 클릭하여 선택 항목을 제거합니다.
- 세분화가 완료되면 프로젝트 인터페이스로 돌아갑니다. 이미지 스택에 연결된 ".label" 확장자를 가진 노드를 찾습니다.
- ".label" 확장을 마우스 오른쪽 단추로 클릭하고 서피스 | 생성을 선택합니다. .surf 파일을 만들려면 적용합니다.
- 세그먼트화된 객체의 3D 모델을 렌더링하려면 .surf 파일을 마우스 오른쪽 단추로 클릭하고 서피스 뷰 를 선택하여 보기 창에서 3D 모델을 생성합니다.
- 시각화 또는 추가 정량적 분석을 위해 3D 모델을 저장합니다.
Access restricted. Please log in or start a trial to view this content.
결과
이 기술의 경우, 관심 영역은 생물학적 연구 목표에 따라 선택되고 포매된 조직의 트리밍 및 절편화 전에 확인된다. 유사하게, 블록 얼굴의 크기는 연구 질문에 의해 결정될 수 있다; 이 경우, 샘플은 대략 0.3 mm x 0.15 mm의 블록 면을 남기도록 트리밍되었다(도 4A). 이를 통해 그리드 당 9 개의 직렬 섹션으로 구성된 두 개의 그리드를 허용하여 18 개의 직렬 섹션을 제공하고 약 62 ...
Access restricted. Please log in or start a trial to view this content.
토론
소기관 구조 및 상호작용을 3D로 시각화하기 위한 접근 가능한 vEM 기술이 이 프로토콜에 설명되어 있습니다. 간세포에서 소기관 간 접촉의 형태학은 여기에서 사례 연구로 제시됩니다. 그러나, 이러한 접근법은 또한 말초 신경에서의 슈완 세포-내피 상호작용(45), 내피 세포에서의 바이벨 팔레이드 바디 생물발생(46), 신장 세포에서의 화물 분비(47), 및 해마 뉴런<...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 공개 할 이해 상충이 없습니다.
감사의 말
전문적인 기술 지원을 위해 Joanna Hanley, Rebecca Fiadeiro 및 Ania Straatman-Iwanowska에게 감사드립니다. 또한 Stefan 연구실 멤버들과 Ian J. White에게 도움이 되는 토론에 감사드립니다. J.J.B.는 UCL의 MRC 분자 세포 생물학 연구소에 MRC 기금을 지원하여 코드 MC_U12266B을 수여합니다. C.J.S.는 UCL의 분자 세포 생물학 대학 단위의 MRC 실험실에 MRC 기금으로 지원되며, 코드 MC_UU_00012/6을 수여합니다. P.G.는 유럽 연구위원회 (European Research Council)의 보조금 코드 ERC-2013-StG-337057이 자금을 지원합니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| 0.22 µm syringe filter | Sarstedt | 83.1826.001 | |
| Aluminum trays | Agar Scientific | AGG3912 | |
| Amira v6 | ThermoFisher | https://www.thermofisher.com | |
| Chloroform | Fisher | C/4960/PB08 | |
| DDSA/Dodecenyl Succinic Anhydride | TAAB | T027 | Epon ingredient |
| Diamond knife | DiaTOME | ultra 45° | |
| DMP-30/2,4,6-tri (Dimethylaminomethyl) phenol | TAAB | D032 | Epon ingredient |
| Dumont Tweezers N5 | Agar Scientific | AGT5293 | |
| Fiji | https://imagej.net/ | ||
| Fiji TrakEM2 plugin | https://imagej.net/ | ||
| Formaldehyde 36% solution | TAAB | F003 | |
| Formvar coated slot grid | Homemade | Alternative: EMS diasum (FF2010-Cu) | |
| Glass bottle with applicator rod | Medisca | 6258 | |
| Glass vials | Fisher Scientific | 15364769 | |
| Gluteraldehyde 25% solution | TAAB | G011 | |
| MNA/Methyl Nadic Anhydride | TAAB | M011 | Epon ingredient |
| Osmium Tetroxide 2% solution | TAAB | O005 | |
| Potassium Ferricyanide | Sigma-Aldrich | P-8131 | |
| Propylene oxide | Fisher Scientific | E/0050/PB08 | |
| Reuseable adhesive | Blue Tack | ||
| Reynolds Lead Citrate | TAAB | L037 | Section stain |
| Sodium Cacodylate | Sigma-Aldrich | C-0250 | to make 0.1 M Caco buffer |
| Super Glue | RS Components | 918-6872 | Cyanoacrylate glue, Step 1.3 |
| TAAB 812 Resin | TAAB | T023 | Epon ingredient |
| Tannic acid | TAAB | T046 | |
| Triton X-100 | Sigma-Aldrich | T9284 | |
| Two part Epoxy Resin | RS Components | 132-605 | Alternative: Step 2.13 |
| Ultramicrotome | Leica | UC7 | |
| Vibrating microtome | Leica | 100 µm thick slices, 0.16 mm/s cutting at 1 mm amplitude . | |
| Weldwood Original Contact cement | DAP | 107 | Contact adhesive: Step 3.1.4 |
참고문헌
- Knoll, M., Ruska, E. Das elektronenmikroskop. Zeitschrift für Physik. 78 (5), 318-339 (1932).
- von Ardenne, M. Daselektronen-rastermikroskop. Zeitschrift für Physik. 109 (9), 553-572 (1938).
- Bang, B. H., Bang, F. B. Graphic reconstruction of the third dimension from serial electron microphotographs. Journal of Ultrastructure Research. 1 (2), 138-139 (1957).
- Birch-Andersen, A. Reconstruction of the nuclear sites of Salmonella typhimurium from electron micrographs of serial sections. Journal of General Microbiology. 13 (2), 327-329 (1955).
- Denk, W., Horstmann, H. Serial block-face scanning electron microscopy to reconstruct three-dimensional tissue nanostructure. PLoS Biology. 2 (11), 329(2004).
- Peddie, C. J., Collinson, L. M. Exploring the third dimension: volume electron microscopy comes of age. Micron. 61, 9-19 (2014).
- Titze, B., Genoud, C. Volume scanning electron microscopy for imaging biological ultrastructure. Biology of the Cell. 108 (11), 307-323 (2016).
- Kornfeld, J., Denk, W. Progress and remaining challenges in high-throughput volume electron microscopy. Current Opinion in Neurobiology. 50, 261-267 (2018).
- Heymann, J. A., et al. Site-specific 3D imaging of cells and tissues with a dual beam microscope. Journal of Structural Biology. 155 (1), 63-73 (2006).
- Knott, G., Marchman, H., Wall, D., Lich, B. Serial section scanning electron microscopy of adult brain tissue using focused ion beam milling. Journal of Neuroscience. 28 (12), 2959-2964 (2008).
- Leighton, S. B. SEM images of block faces, cut by a miniature microtome within the SEM - a technical note. Scanning Electron Microscopy. , 73-76 (1981).
- Martone, M. E., Deerinck, T. J., Yamada, N., Bushong, E., Ellisman, M. H. Correlated 3D light and electron microscopy: use of high voltage electron microscopy and electron tomography for imaging large biological structures. Journal of Histotechnology. 23 (3), 261-270 (2000).
- Micheva, K. D., Smith, S. J. Array tomography: a new tool for imaging the molecular architecture and ultrastructure of neural circuits. Neuron. 55 (1), 25-36 (2007).
- Sjostrand, F. S. Ultrastructure of retinal rod synapses of the guinea pig eye as revealed by three-dimensional reconstructions from serial sections. Journal of Ultrastructure Research. 2 (1), 122-170 (1958).
- Ware, R. W. Three-dimensional reconstruction from serial sections. International Review of Cytology. 40, 325(1975).
- Stevens, J. K., Davis, T. L., Friedman, N., Sterling, P. A systematic approach to reconstructing microcircuitry by electron microscopy of serial sections. Cognitive Brain Research. 2 (3), 265-293 (1980).
- Hoppe, W. Three-dimensional electron microscopy. Annual Review of Biophysics. 10, 563-592 (1981).
- Frank, J. Electron tomography: methods for three-dimensional visualization of structures in the cell. , Springer. New York, NY. (2008).
- Baumeister, W. Electron tomography: towards visualizing the molecular organization of the cytoplasm. Current Opinion in Structural Biology. 12 (5), 679-684 (2002).
- Hoog, J. L., Schwartz, C., Noon, A. T., O'Toole, E. T. Organization of interphase microtubules in fission yeast analyzed by electron tomography. Developmental Cell. 12 (3), 349-361 (2007).
- Harris, K. M., Perry, E., Bourne, J., Feinberg, M., Ostroff, L., Hurlburt, J. Uniform serial sectioning for transmission electron microscopy. Journal of Neuroscience. 26 (47), 12101-12103 (2006).
- Jesior, J. C. Use of low-angle diamond knives leads to improved ultrastructural preservation of ultrathin sections. Scanning Microscopy Supplement. 3, 147-152 (1989).
- Studer, D., Gnaegi, H. Minimal compression of ultrathin sections with use of an oscillating diamond knife. Journal of Microscopy. 197, 94-100 (2000).
- Gay, H., Anderson, T. F. Serial sections for electron microscopy. Science. 120 (3130), 1071-1073 (1954).
- Bernhard, W., Rouiller, C. Close topographical relationship between mitochondria and ergastoplasm of liver cells in a definite phase of cellular activity. The Journal of Biophysical and Biochemical Cytology. 2, 73-78 (1956).
- Palade, G. E. An electron microscope study of the mitochondrial structure. The Journal of Histochemistry & Cytochemistry. 1 (4), 188-211 (1953).
- Wu, H., Carvalho, P., Voeltz, G. K. Here, there, and everywhere: The importance of ER membrane contact sites. Science. 361 (6401), (2018).
- Vance, J. E. Inter-organelle membrane contact sites: implications for lipid metabolism. Biology Direct. 15 (1), 24(2020).
- Stefan, C. J. Endoplasmic reticulum-plasma membrane contacts: Principals of phosphoinositide and calcium signaling. Current Opinion in Cell Biology. 63, 125-134 (2020).
- Zaman, M. F., Nenadic, A., Radojicic, A., Rosado, A., Beh, C. T. Sticking with it: ER-PM membrane contact sites as a coordinating nexus for regulating lipids and proteins at the cell cortex. Frontiers in Cell and Developmental Biology. 8, 675(2020).
- van Vliet, A. R., Sassano, M. L., Agostinis, P. The unfolded protein response and membrane contact sites: tethering as a matter of life and death. Contact. 1, 1-15 (2018).
- Cohen, S., Valm, A. M., Lippincott-Schwartz, J. Interacting organelles. Current Opinion in Cell Biology. 53, 84-91 (2018).
- Hariri, H., et al. Lipid droplet biogenesis is spatially coordinated at ER-vacuole contacts under nutritional stress. EMBO Reports. 19 (1), 57-72 (2018).
- Stefan, C. J., Trimble, W. S., Grinstein, S., Drin, G. Membrane dynamics and organelle biogenesis-lipid pipelines and vesicular carriers. BMC Biology. 15 (1), 102(2017).
- Eisenberg-Bord, M., Shai, N., Schuldiner, M., Bohnert, M. A tether is a tether is a tether: tethering at membrane contact sites. Developmental Cell. 39 (4), 395-409 (2016).
- Scorrano, L., De Matteis, M. A., Emr, S., Giordano, F. Coming together to define membrane contact sites. Nature Communications. 10 (1), 1287(2019).
- Lak, B., Li, S., Belevich, I., Sree, S. Specific subdomain localization of ER resident proteins and membrane contact sites resolved by electron microscopy. European Journal of Cell Biology. 100 (7), 151180(2021).
- Collado, J., Kalemanov, M., Campelo, F., Bourgoint, C. Tricalbin-mediated contact sites control ER curvature to maintain plasma membrane integrity. Developmental Cell. 51 (4), 476-487 (2019).
- West, M., Zurek, N., Hoenger, A., Voeltz, G. K. A 3D analysis of yeast ER structure reveals how ER domains are organized by membrane curvature. Journal of Cell Biology. 193 (2), 333-346 (2011).
- Ilacqua, N., Anastasia, I., Raimondi, A., Lemieux, P. A three-organelle complex made by wrappER contacts with peroxisomes and mitochondria responds to liver lipid flux changes. Journal of Cell Science. 135 (5), 259091(2022).
- Cardona, A., Saalfeld, S., Schindelin, J., Arganda-Carreras, I. TrakEM2 software for neural circuit reconstruction. PLoS One. 7 (6), 38011(2012).
- Stalling, D., Westerhoff, M., Hege, H. -C. Amira: A highly interactive system for visual data analysis. The Visualization Handbook. 38, 749-767 (2005).
- Hsieh, T. S., Chen, Y. J., Chang, C. L., Lee, W. R., Liou, J. Cortical actin contributes to spatial organization of ER-PM junctions. Molecular Biology of the Cell. 28 (23), 3171-3180 (2017).
- Anastasia, I., Ilacqua, N., Raimondi, A., Lemieux, P. Mitochondria-rough-ER contacts in the liver regulate systemic lipid homeostasis. Cell Reports. 34 (11), 108873(2021).
- Cattin, A. L., Burden, J. J., Van Emmenis, L., Mackenzie, F. E. Macrophage-Induced Blood Vessels Guide Schwann Cell-Mediated Regeneration of Peripheral Nerves. Cell. 162 (5), 1127-1139 (2015).
- Lopes-da-Silva, M., et al. A GBF1-dependent mechanism for environmentally responsive regulation of ER-Golgi transport. Developmental Cell. 49 (5), 786-801 (2019).
- Banushi, B., Forneris, F., Straatman-Iwanowska, A., Strange, A. Regulation of post-Golgi LH3 trafficking is essential for collagen homeostasis. Nature Communications. 7, 12111(2016).
- Rey, S. A., et al. Ultrastructural and functional fate of recycled vesicles in hippocampal synapses. Nature Communications. 6, 8043(2015).
- Belicova, L., Repnik, U., Delpierre, J., Gralinska, E. Anisotropic expansion of hepatocyte lumina enforced by apical bulkheads. Journal of Cell Biology. 220 (10), 202303003(2021).
- Kizilyaprak, C., Daraspe, J., Humbel, B. M. Focused ion beam scanning electron microscopy in biology. Journal of Microscopy. 254 (3), 109-114 (2014).
- Xu, C. S., Hayworth, K. J., Lu, Z., Grob, P. Enhanced FIB-SEM systems for large-volume 3D imaging. Elife. 6, 1-36 (2017).
- Parlakgül, G., Arruda, A. P., Cagampan, E., Pang, S. High resolution 3D imaging of liver reveals a central role for subcellular architectural organization in metabolism. bioRxiv. , (2020).
- Guerin, C. J., Kremer, A., Borghgraef, P., Lippens, S. Targeted studies using serial block face and focused ion beam scan electron microscopy. The Journal of Visualized Experiments: JoVE. (150), e59480(2019).
- Kremer, A., et al. A workflow for 3D-CLEM investigating liver tissue. Journal of Microscopy. 281 (3), 231-242 (2021).
- Hayat, M. Principles and techniques of electron microscopy: biological applications. , Cambridge University Press. (2000).
- Wisse, E., Braet, F., Duimel, H., Vreuls, C. Fixation methods for electron microscopy of human and other liver. World Journal of Gastroenterology. 16 (23), 2851-2866 (2010).
- Hanley, J., Dhar, D. K., Mazzacuva, F., Fiadeiro, R. Vps33b is crucial for structural and functional hepatocyte polarity. Journal of Hepatology. 66 (5), 1001-1011 (2017).
- Deerinck, T. J., Bushong, E. A., Thor, A., Ellisman, M. H. NCMIR methods for 3D EM: a new protocol for preparation of biological specimens for serial block face scanning electron microscopy. Microscopy. 1, 6-8 (2010).
- Miranda, K., Girard-Dias, W., Attias, M., de Souza, W., Ramos, I. Three dimensional reconstruction by electron microscopy in the life sciences: An introduction for cell and tissue biologists. Molecular Reproduction and Development. 82 (7-8), 530-547 (2015).
- Yamaguchi, M., Chibana, H. A method for obtaining serial ultrathin sections of microorganisms in transmission electron microscopy. The Journal of Visualized Experiments: JoVE. (131), e56235(2018).
- Hall, D. H., Hartwieg, E., Nguyen, K. C. Modern electron microscopy methods for C. elegans. Methods in Cell Biology. 107, 93-149 (2012).
- Hagler, H. K. Ultramicrotomy for biological electron microscopy. Methods in Molecular Biology. 369, 67-96 (2007).
- Arganda-Carreras, I., et al. Consistent and elastic registration of histological sections using vector-spline regularization. Computer vision approaches to medical image analysis, CVAMIA 2006, Lecture Notes in Computer Science. Beichel, R. R., Sonka, M., et al. 4241, Springer Berlin Heidelberg. Berlin, Heidelberg. 85-95 (2006).
- Belevich, I., Joensuu, M., Kumar, D., Vihinen, H., Jokitalo, E. Microscopy image browser: a platform for segmentation and analysis of multidimensional datasets. PLoS Biology. 14 (1), 1002340(2016).
- Fiala, J. C. Reconstruct: a free editor for serial section microscopy. Journal of Microscopy. 218, 52-61 (2005).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD). Journal of Structural Biology. 116 (1), 71-76 (1996).
- Iudin, A., Korir, P. K., Salavert-Torres, J., Kleywegt, G. J., Patwardhan, A. EMPIAR: a public archive for raw electron microscopy image data. Nature Methods. 13 (5), 387-388 (2016).
- Xu, C. S., Pang, S., Shtengel, G., Muller, A. An open-access volume electron microscopy atlas of whole cells and tissues. Nature. 599 (7883), 147-151 (2021).
- Karabag, C., et al. Semantic segmentation of HeLa cells: An objective comparison between one traditional algorithm and four deep-learning architectures. PLoS One. 15 (10), 0230605(2020).
- Heinrich, L., Bennett, D., Ackerman, D., Park, W. Whole-cell organelle segmentation in volume electron microscopy. Nature. 599 (7883), 141-146 (2021).
- Kim, J. S., Greene, M. J., Zlateski, A., Lee, K. Space-time wiring specificity supports direction selectivity in the retina. Nature. 509 (7500), 331-336 (2014).
- Spiers, H., Songhurst, H., Nightingale, L., de Folter, J. Deep learning for automatic segmentation of the nuclear envelope in electron microscopy data, trained with volunteer segmentations. Traffic. 22 (7), 240-253 (2021).
- Hasan, N. M., Gupta, A., Polishchuk, E., Yu, C. H. Molecular events initiating exit of a copper-transporting ATPase ATP7B from the trans-Golgi network. The Journal of Biological Chemistry. 287 (43), 36041-36050 (2012).
- Stoeck, I. K., Lee, J. Y., Tabata, K., Romero-Brey, I. Hepatitis C virus replication depends on endosomal cholesterol homeostasis. The Journal of Virology. 92 (1), 01196(2018).
- Ma, X., Qian, H., Chen, A., Ni, H. M., Ding, W. X. Perspectives on mitochondria-ER and mitochondria-lipid droplet contact in hepatocytes and hepatic lipid metabolism. Cells. 10 (9), 2273(2021).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유