Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Ensayo de cribado de alto contenido para la identificación de compuestos modificadores de citotoxicidad celular dependientes de anticuerpos
* Estos autores han contribuido por igual
En este artículo
Resumen
Este protocolo presenta una técnica automatizada de alto rendimiento basada en imágenes para identificar compuestos que modulan la destrucción de células de cáncer de mama mediada por células asesinas naturales en presencia de un anticuerpo terapéutico anti-HER-2.
Resumen
La inmunoterapia con anticuerpos antígenos específicos o inhibidores de puntos de control inmunitario ha revolucionado la terapia del cáncer de mama. Las células de cáncer de mama que expresan el receptor del factor de crecimiento epidérmico HER2 pueden ser atacadas por el anticuerpo anti-HER-2 trastuzumab. La citotoxicidad celular dependiente de anticuerpos (ADCC) es un mecanismo importante implicado en la acción antitumoral de HER-2. El trastuzumab unido a las células cancerosas puede ser reconocido por los receptores Fc de las células efectoras ADCC (por ejemplo, células asesinas naturales [NK], macrófagos y granulocitos), lo que desencadena la actividad citotóxica de estas células inmunitarias que conduce a la muerte de las células cancerosas. Nos propusimos desarrollar un ensayo basado en imágenes para la cuantificación de ADCC para identificar nuevos compuestos moduladores de ADCC mediante cribado de alto contenido. En el ensayo, las células de cáncer de mama JIMT-1 que sobreexpresan HER2 se cultivan conjuntamente con células NK-92 en presencia de trastuzumab, y la muerte de las células diana se cuantifica mediante microscopía automatizada y análisis cuantitativo de imágenes. Las células diana se distinguen de las células efectoras en función de su fluorescencia EGFP. Mostramos cómo se pueden probar las bibliotecas de compuestos en el ensayo para identificar fármacos moduladores de ADCC. Para este propósito, se instaló una placa de prueba de biblioteca compuesta utilizando productos químicos finos seleccionados al azar del estante del laboratorio. También se incluyeron en la biblioteca de pruebas tres compuestos desestabilizadores de microtúbulos (colchicina, vincristina, podofilotoxina) que se esperaba que interfirieran con la migración y desgranulación de las células NK. La pantalla de prueba identificó los tres compuestos de control positivo como éxitos que demuestran la idoneidad del método para identificar fármacos modificadores de ADCC en una biblioteca química. Con este ensayo, se pueden realizar exámenes de biblioteca de compuestos para identificar compuestos que mejoran el ADCC que podrían usarse como agentes terapéuticos adyuvantes para el tratamiento de pacientes que reciben inmunoterapias contra el cáncer. Además, el método también se puede utilizar para identificar cualquier efecto secundario inhibidor de ADCC indeseable de los fármacos terapéuticos tomados por pacientes con cáncer para diferentes indicaciones.
Introducción
La inmunoterapia con anticuerpos contra el cáncer, inhibidores de puntos de control inmunitario o células T que expresan receptores de antígenos quiméricos (CAR-T) representa un enfoque poderoso para el tratamiento del cáncer 1,2,3. Trastuzumab es un anticuerpo monoclonal humanizado anti-HER-2 (receptor 2 del factor de crecimiento epidérmico humano) utilizado para tratar el cáncer de mama en estadio temprano o metastásico HER-2 positivo, así como el cáncer gástrico metastásico HER-2 positivo 4,5,6. Actúa principalmente inhibiendo el efecto estimulante de la proliferación del factor de crecimiento epidérmico4. Sin embargo, se ha informado que trastuzumab desencadena eficientemente la muerte de las células cancerosas, incluso si las células cancerosas han perdido su capacidad de respuesta a la estimulación de HER-27. Este efecto paradójico del anticuerpo se debe a la citotoxicidad mediada por células dependientes de anticuerpos (ADCC)7. El ADCC puede ser mediado por células asesinas naturales (NK), granulocitos y macrófagos conocidos colectivamente como las células efectoras de ADCC 8,9. Si un anticuerpo, como trastuzumab, se une a las células tumorales, entonces estas células efectoras usan sus receptores Fc para unirse a la región constante (Fc) del anticuerpo. El anticuerpo une las células tumorales y las células efectoras portadoras del receptor Fc, desencadenando la liberación de sus mediadores citotóxicos10. Las células asesinas naturales liberan la carga citotóxica de sus gránulos que contienen perforina para generar poros en la membrana celular diana y granzima (desencadenando vías de señalización de muerte celular) en la sinapsis inmune que conduce a la apoptosis de las células cancerosas (ver Figura 1).
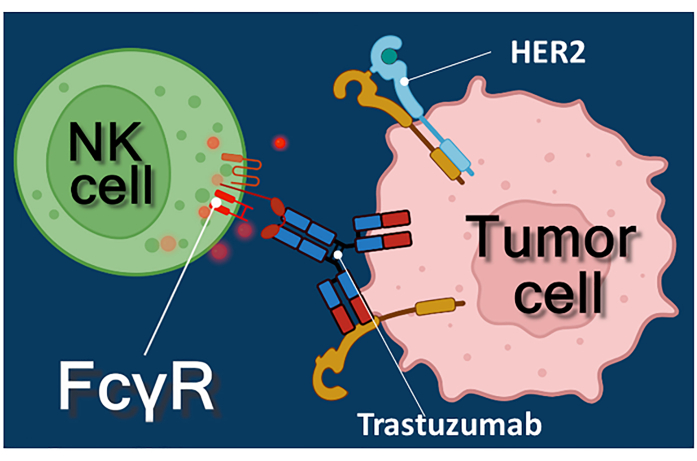
Figura 1: Interacciones entre células efectoras y diana en ADCC. El receptor Fcγ de la superficie celular de la célula NK efectora reconoce la región Fc del anticuerpo anti-HER2 trastuzumab específico para la molécula HER2 expresada en la superficie de la célula tumoral. Así, se establece la llamada sinapsis inmunológica entre las dos células, induciendo la exocitosis dirigida de gránulos citotóxicos de la célula efectora. Las moléculas de perforina y granzima liberadas eventualmente resultan en la apoptosis de la célula diana. Haga clic aquí para ver una versión más grande de esta figura.
Se han desarrollado previamente varios ensayos para cuantificar la citotoxicidad, incluido el ADCC. El estándar de oro es el método de liberación de cromo radiactivo, donde las células diana se marcan con isótopo radiactivo 51Cr, y el ADCC se cuantifica midiendo la radiactividad del sobrenadante de las células diana lisadas11. Debido a los problemas obvios debido al manejo, almacenamiento y eliminación estrictamente regulados de farmacones y desechos radiactivos, este método se ha vuelto cada vez más impopular entre los científicos de la vida. Además, tampoco es susceptible de aplicaciones de alto rendimiento. La medición de la actividad de las enzimas (por ejemplo, lactato-deshidrogenasa) liberadas de las células diana muertas puede proporcionar una alternativa no radiactiva al ensayo 51Cr12. Estos ensayos, sin embargo, no distinguen entre muertes de células diana y efectoras. La detección de impedancia de sustrato celular eléctrico (ECIS) demostró ser adecuada para la cuantificación de ADCC13, pero el equipo ECIS no está disponible en la mayoría de los laboratorios y la técnica no es compatible con aplicaciones / cribado de alto rendimiento. Las células marcadas con fluorescencia representan una alternativa popular en muchos ensayos de biología celular y se utilizan a menudo en citometría de flujo o aplicaciones basadas en lectores de placas14,15,16. Sin embargo, estos ensayos a menudo contienen pasos de lavado o son incompatibles con aplicaciones de alto rendimiento (por ejemplo, técnicas basadas en citometría de flujo). Algunos ensayos populares de citotoxicidad, que en teoría deberían ser adecuados para la cuantificación del ADCC, no logran determinar de manera confiable la eficiencia del ADCC13. Recientemente, con la difusión de la microscopía confocal fluorescente, los ensayos basados en imágenes y de alto contenido se están volviendo cada vez más populares en diversas áreas de las ciencias de la vida17. Por un lado, los equipos de imágenes celulares son ahora bastante ubicuos, mientras que, por otro lado, se pueden recopilar parámetros morfológicos prácticamente infinitos a partir de las imágenes adquiridas. Por lo tanto, nos propusimos desarrollar un ensayo ADCC compatible con el cribado de alto contenido y demostrar su idoneidad para el cribado de bibliotecas compuestas.
Aquí, presentamos un ensayo ADCC basado en imágenes y demostramos cómo se puede usar este ensayo para la detección de alto contenido (HCS) para identificar compuestos moduladores de ADCC. El modelo se basa en células diana de carcinoma de mama JIMT-1, células efectoras CD16.176V.NK-92 y el anticuerpo monoclonal humanizado anti-HER2 trastuzumab. Con este método, es posible identificar fármacos que pueden mejorar la acción destructora de tumores de las células NK o obtener información sobre el mecanismo del ADCC mediado por células NK mediante la identificación de moléculas pequeñas que interfieren con el ADCC. Sugerimos que los científicos de la vida que buscan cuantificar la citotoxicidad mediada por células con especial atención al ADCC pueden beneficiarse del uso de este ensayo, ya sea para la ciencia del descubrimiento o el desarrollo de fármacos. Este ensayo puede ser una alternativa si un laboratorio tiene acceso y cierta experiencia en imágenes fluorescentes y análisis cuantitativo de imágenes.
Protocolo
NOTA: Los pasos clave del flujo de trabajo del ensayo se presentan en la Figura 2.
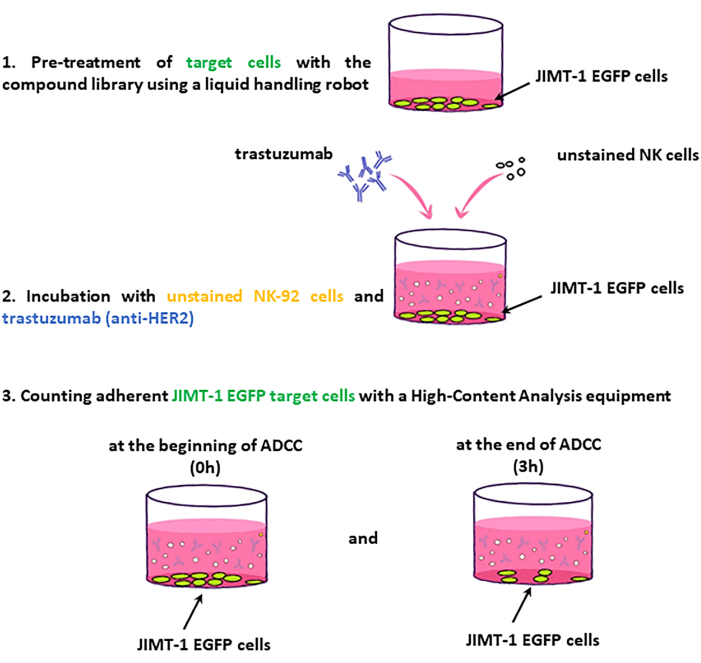
Figura 2: Flujo de trabajo de la pantalla ADCC. Las células diana JIMT-1-EGFP sembradas en placas HCS de 96 pocillos se tratan con fármacos de la biblioteca de compuestos. A su vez, se agregan células NK (efectoras) no teñidas y trastuzumab, y se obtiene una imagen de la placa en un punto de tiempo 0 y después de 3 h de incubación. La evaluación ADCC se basa en el cambio en el número de células diana viables (adherentes a la superficie). Haga clic aquí para ver una versión más grande de esta figura.
1. Recubrimiento de la placa HCS
- Cubra las placas de cribado de alto contenido (HSC) de 96 pocillos con medio JIMT-1 de 50 μL/pocillo (medio DMEM/F-12 suplementado con suero bovino fetal al 20% (FBS), insulina 0.3 U/ml (100 UI/ml, Humulin R y 1% de penicilina-estreptomicina).
- Coloque la placa en una incubadora deCO2 durante 1 h.
NOTA: El recubrimiento es crucial para unir las celdas JIMT-1 a la superficie de vidrio de la placa.
2. Siembra de células de proteína fluorescente verde mejorada (EGFP) JIMT-1
NOTA: Las células JIMT-1 que expresan EGFP se generaron en nuestro trabajo anterior18, y las células se cultivaron en matraces de cultivo de tejidos T25 en medios JIMT-1 (ver composición en el paso 1.1).
- Lave las celdas con 2 ml de PBS estéril.
- Añadir 1 ml de tripsina-EDTA al matraz y volver a colocar el matraz en una incubadora deCO2 durante 10 min.
- Después de la incubación, golpee el matraz para comprobar si las células JIMT-1 están desprendidas.
- Detenga la digestión con 2 ml de medios JIMT-1 y recoja la suspensión celular en un tubo de 15 ml.
- Contar las células con azul de tripano al 0,4% (80 μL del colorante + 20 μL de la suspensión celular) en una cámara de Bürker y ajustar el número de células a 133.000 células/ml.
- Aspirar el medio de recubrimiento de la placa de 96 pocillos (paso 1.2).
- Pipetear 75 μL de la suspensión celular a cada pocillo de las placas HCS (ver Tabla de materiales).
- Dejar que las células se adhieran durante una incubación nocturna a 37 °C en una incubadora deCO2 .
3. Pretratamiento de células JIMT-1 EGFP con la biblioteca de compuestos
- Aspirar el medio de las células JIMT-1 y añadir 50 μL/pocillo de medio JIMT-1 fresco a los pocillos. Transfiera la placa al laboratorio de cribado de alto rendimiento.
NOTA: El uso de un robot de manejo de líquidos hace que la adición de bibliotecas de compuestos sea más eficiente y reproducible. - Transfiera los compuestos de prueba de la placa de biblioteca de compuestos a la placa de ensayo con una herramienta de pasador calibrada a un volumen de 25 nL. Realice esto cuatro veces. Cuatro rondas de transferencia dan un volumen final de 100 nL (y una concentración final de 20 μM).
- Entre cada paso, lave la herramienta del pasador, primero con 50% de DMSO y luego con 70% de etanol.
- Incubar las placas durante 1 h en una incubadora deCO2 a 37 °C.
4. Inicio del ensayo ADCC añadiendo las células efectoras
NOTA: Las células CD16.176V.NK92 (en adelante, células NK92) se cultivaron en α-MEM suplementadas con 20% de FBS, 1% de MEM-NEAA, 1% de Na-piruvato, 1% de glutamina, 1% de penicilina-estreptomicina y 100 UI/ml de IL-2.
- Contar células NK92 con azul de tripano (80 μL del colorante + 20 μL de la suspensión celular). Ajuste el número de células a 400.000 células/ml.
- Centrifugar 4 ml de la suspensión celular a 150 x g durante 3 min a temperatura ambiente.
- Prepare el medio ADCC agregando 20 μg/ml de anticuerpo anti-HER2 (trastuzumab) al medio JIMT-1.
- Resuspender el pellet de células NK en medio ADCC de 5 ml.
- Pipetear 20.000 células NK en 50 μL de medio ADCC a las células JIMT-1 objetivo. El volumen final es de 100 μL, y la concentración final de trastuzumab es de 10 μg/ml.
- Coloque la placa de ensayo en el equipo de análisis de alto contenido con una incubadora incorporada a 37 °C.
5. Imágenes
NOTA: Las placas deben ser fotografiadas en dos puntos de tiempo, primero, inmediatamente después de la adición de las células efectoras a las células diana y segundo, a las 3 h después de la adición de células NK. Para la obtención de imágenes, se puede utilizar el analizador de alto contenido y su software o alternativas adecuadas (consulte la Tabla de materiales).
- Seleccione Tipo de placa (portador de celda ultra de 96 pocillos) de la lista de placas.
- Seleccione el enfoque automático Dos picos si el ensayo se realiza en placas.
- Utilice un objetivo 10x en modo no confocal.
- Seleccione Binning 2 para duplicar la relación señal/ruido.
- Tome imágenes de campo claro a 650-760 nm e imágenes fluorescentes de las células JIMT-1 transducidas por EGFP a longitudes de onda de 488 nm (excitación) y 500-550 nm (emisión).
- Seleccione el número de campos y el número de puntos de tiempo para la creación de imágenes.
6. Análisis de imágenes
NOTA: Para analizar la eficiencia de ADCC, se cuentan las células JIMT-1 viables. Las células diana eliminadas por ADCC se desprenden de la superficie y se alejan del plano focal del microscopio. Por lo tanto, la diferencia entre el número de células viables al principio y al final de la reacción ADCC corresponde a las células diana eliminadas por ADCC. Para mostrar cómo construir la secuencia de evaluación, se muestra un pozo ADCC de control en el video.
- Utilice el módulo Buscar celdas para detectar regiones en la imagen que corresponden a celdas.
NOTA: Cada celda se detecta como una región en la imagen con una intensidad de fluorescencia más alta que su entorno. - Seleccione celdas utilizando el algoritmo M incorporado con un mínimo de 80 μm de diámetro.
- Establezca Sensibilidad de división, que divide un objeto grande en objetos más pequeños, en 0,5.
- Establezca el umbral común (el nivel más bajo de intensidad de píxeles) en 0.
- Excluir la detección de área de fondo con alta intensidad de fluorescencia EGFP en dos pasos.
- En primer lugar, utilice la función Calcular propiedades de intensidad para determinar la intensidad de fluorescencia EGFP en la región de celdas seleccionada anteriormente.
- Establezca el umbral de intensidad mínima y máxima mediante la opción Seleccionar población .
Resultados
Para demostrar cómo funciona el ensayo en la vida real, creamos una biblioteca de pruebas de 16 compuestos seleccionados al azar de los estantes del laboratorio (Figura 3). Además, el DMSO también se incluyó como control negativo y tres compuestos inhibidores de la polimerización de microtúbulos (colchicina, vincristina y podofilotoxina) como controles positivos. Se esperaba que estos últimos inhibieran el ADCC al interferir con la migración de células NK a las células cancerosas y...
Discusión
La reacción del ADCC se ha descrito hace relativamente mucho tiempo. También se han descrito eventos moleculares clave del proceso19. Los métodos para medir el ADCC van desde el ensayo de liberación de cromo radiactivo estándar de oro, los ensayos de liberación de enzimas citoplasmáticas hasta varios ensayos de citometría de flujo o microplacas basados en fluorescencia20. Sin embargo, una limitación común de estos ensayos es que no son susceptibles de aplicaciones...
Divulgaciones
Los autores no informan ningún conflicto de intereses.
Agradecimientos
LV recibió fondos de la Oficina Nacional de Investigación, Desarrollo e Innovación subvenciones GINOP-2.3.2-15-2016-00010 TUMORDNS", GINOP-2.3.2-15-2016-00048-STAYALIVE y OTKA K132193, K147482. Las células CD16.176V.NK-92 se obtuvieron del Dr. Kerry S. Campbell (Fox Chase Center, Philapedlphia, PA, en nombre de Brink Biologics, lnc. San Diego, CA), están protegidos por patentes en todo el mundo, y fueron licenciados por Nantkwest, lnc. Los autores agradecen a György Vereb y Árpád Szöőr por su ayuda con el uso de la línea celular NK-92 y por el asesoramiento técnico.
Materiales
| Name | Company | Catalog Number | Comments |
| 5-fluorouracil | Applichem | A7686 | in compound library |
| 96-well Cell Carrier Ultra plate | PerkinElmer | LLC 6055302 | |
| Betulin | Sigma | B9757 | in compound library |
| CD16.176V.NK92 cells | Nankwest Inc. | ||
| Cerulenin | ChemCruz | sc-396822 | in compound library |
| Cisplatin | Santa Cruz Biotechnology | sc-200896 | in compound library |
| Colchicine | Sigma | C9754 | in compound library |
| Concanavalin-A | Calbiochem | 234567 | in compound library |
| Dexamethasone | Sigma | D4902 | in compound library |
| DMEM/F-12 medium | Sigma | D8437 | in JIMT-1 EGFP medium |
| DMSO | Sigma | D2650 | in compound library |
| Etoposide | Sigma | E1383 | E1383 |
| Fetal bovine serum (FBS) | Biosera | FB-1090/500 | JIMT-1 EGFP and NK medium |
| Fisetin | Sigma | F4043 | in compound library |
| Freedom EVO liquid handling robot | TECAN | ||
| Gallotannin | Fluka Chemical Corp. | 16201 | in compound library |
| Glutamine | Gibco | 35,050–061 | in NK medium |
| Harmony software | PerkinElmer | ||
| Humanized anti-HER2 monoclonal antibody (Herzuma) | EGIS Pharmaceuticals, Budapest Hungary | N/A | |
| Humulin R (insulin) | Eli Lilly | HI0219 | JIMT-1 EGFP medium |
| IL-2 | Novartis Hungária Kft. | PHC0026 | in NK medium |
| Isatin | Sigma | 114618 | in compound library |
| MEM Non-essential Amino Acids (MEM-NEAA) | Gibco | 11,140–050 | in NK medium |
| Na-pyruvate | Lonza | BE13-115E | in NK medium |
| Naringenin | Sigma | N5893 | in compound library |
| NQDI-1 | Sigma | SML0185 | in compound library |
| Opera Phenix High-Content Analysis equipment | PerkinElmer | ||
| Penicillin–streptomycin | Biosera | LM-A4118 | JIMT-1 EGFP and NK medium |
| Pentoxyfilline | Sigma | P1784 | in compound library |
| Phosphate buffered saline (PBS) | Lonza | BE17-517Q | to wash the cells |
| Podophyllotoxin | Sigma | P4405 | in compound library |
| Quercetin | Sigma | Q4951 | in compound library |
| Tannic acid | Sigma | T8406 | in compound library |
| Temozolomide | Sigma | T2577 | in compound library |
| Trypan blue 0.4% solution | Sigma | T8154 | for cell counting |
| Vincristine sulfate | Sigma | V0400000 | in compound library |
| α-MEM | Sigma | M8042 | in NK medium |
Referencias
- Gupta, S. L., Basu, S., Soni, V., Jaiswal, R. K. Immunotherapy: an alternative promising therapeutic approach against cancers. Molecular Biology Reports. 49 (10), 9903-9913 (2022).
- Moretti, A., et al. The past, present, and future of non-viral CAR T cells. Frontiers in Immunology. 13, 867013 (2022).
- June, C. H., O'Connor, R. S., Kawalekar, O. U., Ghassemi, S., Milone, M. C. CAR T cell immunotherapy for human cancer. Science. 359 (6382), 1361-1365 (2018).
- Ross, J. S., et al. The HER-2 receptor and breast cancer: ten years of targeted anti-HER-2 therapy and personalized medicine. Oncologist. 14 (4), 320-368 (2009).
- Shitara, K., et al. Discovery and development of trastuzumab deruxtecan and safety management for patients with HER2-positive gastric cancer. Gastric Cancer. 24 (4), 780-789 (2021).
- Gianni, L., et al. Efficacy and safety of neoadjuvant pertuzumab and trastuzumab in women with locally advanced, inflammatory, or early HER2-positive breast cancer (NeoSphere): a randomised multicentre, open-label, phase 2 trial. Lancet Oncology. 13 (1), 25-32 (2012).
- Barok, M., et al. Trastuzumab causes antibody-dependent cellular cytotoxicity-mediated growth inhibition of submacroscopic JIMT-1 breast cancer xenografts despite intrinsic drug resistance. Molecular and Cancer Therapy. 6 (7), 2065-2072 (2007).
- Gauthier, M., Laroye, C., Bensoussan, D., Boura, C., Decot, V. Natural Killer cells and monoclonal antibodies: Two partners for successful antibody dependent cytotoxicity against tumor cells. Crit Rev Oncol Hematol. 160, 103261 (2021).
- Gruijs, M., Sewnath, C. A. N., van Egmond, M. Therapeutic exploitation of neutrophils to fight cancer. Semin Immunol. 57, 101581 (2021).
- Mando, P., Rivero, S. G., Rizzo, M. M., Pinkasz, M., Levy, E. M. Targeting ADCC: A different approach to HER2 breast cancer in the immunotherapy era. Breast. 60, 15-25 (2021).
- van der Haar Avila, I., Marmol, P., Kiessling, R., Pico de Coana, Y. Evaluating antibody-dependent cell-mediated cytotoxicity by chromium release assay. Methods in Molecular Biology. 1913, 167-179 (2019).
- Broussas, M., Broyer, L., Goetsch, L. Evaluation of antibody-dependent cell cytotoxicity using lactate dehydrogenase (LDH) measurement. Methods in Molecular Biology. 988, 305-317 (2013).
- Toth, G., Szollosi, J., Vereb, G. Quantitating ADCC against adherent cells: Impedance-based detection is superior to release, membrane permeability, or caspase activation assays in resolving antibody dose response. Cytometry A. 91 (10), 1021-1029 (2017).
- Chung, S., Nguyen, V., Lin, Y. L., Kamen, L., Song, A. Thaw-and-use target cells pre-labeled with calcein AM for antibody-dependent cell-mediated cytotoxicity assays. Journal of Immunological Methods. 447, 37-46 (2017).
- Lee-MacAry, A. E., et al. Development of a novel flow cytometric cell-mediated cytotoxicity assay using the fluorophores PKH-26 and TO-PRO-3 iodide. Journal of Immunological Methods. 252 (1-2), 83-92 (2001).
- Tanito, K., et al. Comparative evaluation of natural killer cell-mediated cell killing assay based on the leakage of an endogenous enzyme or a pre-loaded fluorophore. Analytical Science. 37 (11), 1571-1575 (2021).
- Lin, S., Schorpp, K., Rothenaigner, I., Hadian, K. Image-based high-content screening in drug discovery. Drug Discovery Today. 25 (8), 1348-1361 (2020).
- Guti, E., et al. The multitargeted receptor tyrosine kinase inhibitor sunitinib induces resistance of HER2 positive breast cancer cells to trastuzumab-mediated ADCC. Cancer Immunology, Immunotherapy. 71 (9), 2151-2168 (2022).
- Li, F., Liu, S. Focusing on NK cells and ADCC: A promising immunotherapy approach in targeted therapy for HER2-positive breast cancer. Frontiers in Immunology. 13, 1083462 (2022).
- Perussia, B., Loza, M. J. Assays for antibody-dependent cell-mediated cytotoxicity (ADCC) and reverse ADCC (redirected cytotoxicity) in human natural killer cells. Methods in Molecular Biology. 121, 179-192 (2000).
- Garcia-Alonso, S., Ocana, A., Pandiella, A. Trastuzumab emtansine: Mechanisms of action and resistance, clinical progress, and beyond. Trends in Cancer. 6 (2), 130-146 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados