Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Medición en tiempo real del perfil bioenergético mitocondrial de neutrófilos
En este artículo
Resumen
Describimos protocolos paso a paso que miden la respiración mitocondrial de neutrófilos de ratón y humanos y células HL60 utilizando el analizador de flujo extracelular metabólico.
Resumen
Los neutrófilos son la primera línea de defensa y los leucocitos más abundantes en los humanos. Estas células efectoras realizan funciones como la fagocitosis y el estallido oxidativo, y crean trampas extracelulares de neutrófilos (NET) para la eliminación microbiana. Nuevos conocimientos sobre las actividades metabólicas de los neutrófilos desafían el concepto inicial de que se basan principalmente en la glucólisis. La medición precisa de las actividades metabólicas puede desplegar diferentes requisitos metabólicos de los neutrófilos, incluido el ciclo del ácido tricarboxílico (TCA) (también conocido como ciclo de Krebs), la fosforilación oxidativa (OXPHOS), la vía de la pentosa fosfato (PPP) y la oxidación de ácidos grasos (FAO) en condiciones fisiológicas y en estados de enfermedad. Este documento describe un protocolo paso a paso y requisitos previos para medir la tasa de consumo de oxígeno (OCR) como un indicador de la respiración mitocondrial en neutrófilos derivados de la médula ósea del ratón, neutrófilos derivados de la sangre humana y la línea celular HL60 similar a los neutrófilos, utilizando el análisis de flujo metabólico en un analizador de flujo extracelular metabólico. Este método se puede utilizar para cuantificar las funciones mitocondriales de los neutrófilos en condiciones normales y de enfermedad.
Introducción
Las mitocondrias juegan un papel importante en la bioenergética celular, que genera trifosfato de adenosina (ATP) por fosforilación oxidativa (OXPHOS). Además de esto, el papel de las mitocondrias se extiende a la generación y desintoxicación de especies reactivas de oxígeno, la regulación del calcio de la matriz citoplasmática y mitocondrial, la síntesis celular, el catabolismo y el transporte de metabolitos dentro de la célula1. La respiración mitocondrial es esencial en todas las células, ya que su disfunción puede dar lugar a problemas metabólicos 2, incluyendo enfermedades cardiovasculares3 y una amplia variedad de enfermedades neurodegenerativas, como la degeneración macular relacionada con la edad4, las enfermedades de Parkinson y Alzheimer5, y la enfermedad de Charcot-Marie-Tooth2 A (CMT2A)6.
Los estudios microscópicos electrónicos sobre neutrófilos revelaron que hay relativamente pocas mitocondrias7, y dependen en gran medida de la glucólisis para su producción de energía, ya que las tasas de respiración mitocondrial son muy bajas8. Sin embargo, las mitocondrias son cruciales para las funciones de los neutrófilos, como la quimiotaxis9 y la apoptosis10,11,12. Un estudio previo reveló una compleja red mitocondrial en neutrófilos humanos con alto potencial de membrana. La pérdida de potencial de la membrana mitocondrial es un indicador temprano de apoptosis de neutrófilos10. El tratamiento con cianuro de carbonilo mitocondrial con cianuro de carbonilo m-clorofenil hidrazona (CCCP) mostró inhibición significativa en la quimiotaxis, junto con un cambio en la morfología mitocondrial 9,10.
Aunque la principal fuente de energía para los neutrófilos es la glucólisis, las mitocondrias proporcionan el ATP que inicia la activación de los neutrófilos al alimentar la primera fase de la señalización purinérgica, lo que aumenta la señalización de Ca2+, amplifica la producción de ATP mitocondrial e inicia las respuestas funcionales de los neutrófilos13. La disfunción de la cadena respiratoria mitocondrial resulta en una producción excesiva de especies tóxicas reactivas de oxígeno (ROS) y conduce a daños patógenos14,15,16. La NETosis, que es el proceso de formación de trampas extracelulares de neutrófilos (TNE), es una propiedad crítica de los neutrófilos que les ayuda a luchar contra los patógenos17 y contribuye a muchas condiciones patológicas, incluyendo cáncer, trombosis y trastornos autoinmunes18. Las ROS derivadas de mitocondrial contribuyen a NETosis19, el ADN mitocondrial puede ser un componente de NETs18, y la homeostasis mitocondrial alterada afecta NETosis 20,21,22,23,24. Además, durante la diferenciación o maduración normal, la reprogramación metabólica de los neutrófilos se invierte al limitar la actividad glucolítica, y participan en la respiración mitocondrial y movilizan los lípidos intracelulares25,26.
El analizador de flujo extracelular metabólico puede monitorear y cuantificar continuamente la respiración mitocondrial de células vivas y la glucólisis. El analizador utiliza un cartucho sensor de formato placa de 96 pocillos y dos fluoróforos para cuantificar la concentración de oxígeno (O2) y los cambios de pH. El cartucho sensor está por encima de la monocapa celular durante el ensayo y forma una microcámara de ~200 nm de altura. Los haces de fibra óptica en el analizador se utilizan para excitar los fluoróforos y detectar los cambios de intensidad fluorescente. Los cambios en tiempo real en la concentración deO2 y el pH se calculan automáticamente y se muestran como tasa de consumo de oxígeno (OCR) y tasa de acidificación extracelular (ECAR). Hay cuatro puertos en el cartucho del sensor que permiten cargar hasta cuatro compuestos en cada pocillo durante las mediciones del ensayo. Este protocolo se centra en cuantificar la respiración mitocondrial de neutrófilos humanos y de ratón, así como las células HL60 similares a los neutrófilos, utilizando el analizador de flujo extracelular metabólico.
Protocolo
Las muestras de sangre total heparinizadas se obtuvieron de donantes humanos sanos después de obtener el consentimiento informado, según lo aprobado por la Junta de Revisión Institucional de UConn Health de acuerdo con la Declaración de Helsinki. Todos los experimentos con animales siguieron las pautas del Comité Institucional de Cuidado y Uso de Animales de UConn Health (IACUC), y la aprobación para el uso de roedores se obtuvo del IACUC de UConn Health de acuerdo con los criterios descritos en la Guía para el Cuidado y Uso de Animales de Laboratorio de los Institutos Nacionales de Salud. En este estudio se utilizaron ratones machos C57BL / 6 a las 6 semanas de edad.
1. Preparación de la placa de 96 pocillos para el ensayo de flujo extracelular metabólico
- Un cartucho sensor está empaquetado en la parte superior de la placa de 96 pocillos especialmente diseñada para el ensayo de flujo extracelular metabólico. Hidratar el cartucho levantándolo con cuidado y colocar 200 μL/pocillo de medio de calibración en cada pocillo de la placa subyacente. Coloque el cartucho sobre la placa con calibrante en una incubadora humidificada a 37 °C sinCO2 durante la noche para hidratar.
- Según el tipo de célula, utilice un recubrimiento específico para la placa de cultivo para garantizar la adhesión celular. Para los neutrófilos humanos, las células HL60 indiferenciadas y diferenciadas recubren la placa de 96 pocillos con solución salina estéril tamponada con fosfato (PBS) que contiene 50 μL de 5 μg / ml de anticuerpo antihumano antihumano purificado (Clon TS1/18) a 4 °C durante la noche. Para neutrófilos de ratón, cubra la placa de 96 pocillos con 25 μL de Cell Tak de 22,4 μg/ml a pH 8,0 en NaHCO3 a 0,1 M a temperatura ambiente (RT) durante 20 min.
- Lave las placas con 200 μL de PBS estéril dos veces.
- Agregue medio completo a los pocillos de esquina (A1, A12, H1 y H12) de la placa de cultivo celular (sin células) para la corrección de fondo en las placas de cultivo celular durante la siembra.
NOTA: La evaporación durante el recubrimiento y la calibración puede afectar el volumen de los medios de calibración y la normalización. Use una bandeja o cámara con pañuelo estéril humedecido con agua y coloque la placa con el calibrante encima para evitar la evaporación.
2. Preparación y siembra de células
- Preparación del medio de ensayo
- Prepare el medio de ensayo agregando 1 mM de piruvato, 10 mM de glucosa y 2 mM de glutamina al medio Eagle modificado (DMEM) de Dulbecco.
- Aislamiento de neutrófilos de ratón de la médula ósea
- Aísle los neutrófilos de ratón de la médula ósea utilizando el kit de enriquecimiento de neutrófilos de ratón, de acuerdo con las instrucciones del fabricante.
- Anestesiar a los ratones mediante inyección intraperitoneal (i.p.) de ketamina (125 mg/kg) y xilazina (12,5 mg/kg) y luego sacrificar a los ratones por luxación cervical.
- Cosechar los fémures y tibias, como se describió anteriormente27. Brevemente, corte la piel para exponer la pierna con los músculos. Corte la articulación de la cadera para extraer la pierna del cuerpo. Luego, retire los músculos para recoger el fémur y la tibia.
- Cortar los extremos más pequeños de los fémures y tibias. Sujetando los extremos cortados hacia abajo, colóquelos en un tubo de centrífuga de 0,5 ml con dos orificios perforados con aguja de jeringa de 25 g en la parte inferior, y coloque el tubo de 0,5 ml en un tubo de centrífuga de 1,5 ml (Figura 1A). Agregue 50 μL de PBS en el tubo de 0,5 ml para evitar el secado de las células de la médula ósea.
- Centrifugar a 5.900 × g a RT durante 15 s para recoger la médula ósea en el fondo del tubo de 1,5 ml. Resuspender las células de la médula ósea con 1 ml de DMEM. Agregue 50 μL de suero de rata del kit, mezcle suavemente por pipeteo y transfiera la suspensión celular a un tubo de ensayo de cultivo de poliestireno de 5 ml.
- Añadir 50 μL de cóctel de enriquecimiento del kit de enriquecimiento de neutrófilos de ratón e incubar durante 15 min a RT. Centrifugar a 300 × g a RT durante 5 min.
- Resuspender las células con 1 ml de DMEM, añadir 50 μL de cóctel de selección de biotina del kit, mezclar suavemente por pipeteo e incubar durante 15 min en RT.
- Añadir 150 μL de partículas magnéticas de vórtice del kit, mezclar suavemente por pipeteo e incubar durante 10 min a RT.
- Agregue ~ 1.3 ml de DMEM, mezcle suavemente por pipeteo, coloque el tubo en el imán durante 3 minutos y transfiera el sobrenadante que contiene neutrófilos purificados de ratón a un nuevo tubo de ensayo de cultivo de poliestireno de 5 ml invirtiendo el tubo original junto con el imán.
- Centrifugar a 300 × g a RT durante 5 min. Después de retirar el sobrenadante por aspiración al vacío, resuspender las células con 1 ml de medio de ensayo.
- Contar las células manualmente con un hemocitómetro.
- Ajustar la densidad celular a 1,1 × 106 células/ml añadiendo medio de ensayo, sembrar 180 μL de la suspensión de neutrófilos de ratón (2 × 105 células) por pocillo en la placa de 96 pocillos preparada (pasos 1.2-1.4), y centrifugar la placa a 300 × g a RT durante 3 minutos sin freno para asegurar la adherencia adecuada de las células en la parte inferior de la placa.
- Incubar la placa en una incubadora humidificada a 37 °C sinCO2 durante 1 hora para preequilibrar las células con el medio de ensayo.
NOTA: La pureza de los neutrófilos es crítica para el ensayo, ya que es un sesgo potencial. La pureza del aislamiento de neutrófilos de ratón es del 69,9%-88,7% mediante este protocolo. Existen otros métodos para aislar neutrófilos de la médula ósea del ratón, como la centrifugación por gradiente de densidad28. También hay kits alternativos de aislamiento de neutrófilos de otros proveedores, basados en la selección magnética negativa utilizando los anticuerpos monoclonales contra antígenos que no se expresan en los neutrófilos.
- Aislamiento de neutrófilos humanos de sangre periférica
- Agregue 8 ml de solución de polisacárido en un tubo de centrífuga de 15 ml, luego coloque una capa sobre 4 ml de sangre periférica sobre la solución de polisacárido sin mezclar.
- Centrifugadora a 550 × g a 20 °C durante 30 min. Haga que el rotor desacelere a 1.
NOTA: El tiempo de separación de neutrófilos puede variar entre los donantes. Es un mínimo de 30 minutos, y se pueden necesitar 10-20 minutos adicionales si la separación de neutrófilos no tiene éxito. - Observe la separación de plasma/plaquetas y células mononucleares después de la centrifugación, como se muestra en la Figura 1B. Retire con cuidado el líquido amarillo superior en la parte superior (plasma y plaquetas) y la banda turbia superior (células mononucleares) sin alterar la banda turbia inferior (neutrófilos) con una pipeta de 1 ml.
- Recoja la banda turbia inferior y ~3-4 ml del líquido transparente debajo en un nuevo tubo de centrífuga de 15 ml que contenga 10 ml de PBS.
- Centrifugar a 400 × g a 20 °C durante 10 min y extraer el sobrenadante por aspiración al vacío.
- Resuspender las células con 5 ml de PBS y centrifugar a 300 × g a 20 °C durante 5 min.
- Después de retirar el sobrenadante, resuspender las células con 1 ml de medio de ensayo.
- Contar las células manualmente con un hemocitómetro.
NOTA: Dado que los eritrocitos en la sangre no tienen mitocondrias, la contaminación globular no afecta el ensayo de la prueba de esfuerzo mitocondrial y previene la activación/cebado de neutrófilos29,30. Los eritrocitos necesitan ser lisados durante el recuento celular para obtener una concentración precisa de neutrófilos. Agregue 10 μL de la suspensión celular a 891 μL de agua desionizada durante 10-30 s para lisar los eritrocitos, luego agregue 99 μL de 10x PBS para equilibrar la presión osmótica, evitando la lisis de los neutrófilos. - Ajustar la densidad celular a 2,2 × 106 células/ml añadiendo medio de ensayo, sembrar 180 μL de neutrófilos humanos (~4 × 105) por pocillo en la placa preparada de 96 pocillos y centrifugar la placa a 300 × g a RT durante 3 minutos sin freno para garantizar la adherencia adecuada de las células en la parte inferior de la placa.
- Incubar la placa en una incubadora humidificada a 37 °C sinCO2 durante 1 hora para preequilibrar las células con el medio de ensayo.
NOTA: La pureza de los neutrófilos es crítica para el ensayo, ya que es un sesgo potencial. La pureza del aislamiento de neutrófilos humanos es del 86,6% -96,8%. Existen otros métodos de centrifugación por gradiente de densidad para aislar neutrófilos de la sangre humana, incluido el aislamiento de Percoll, así como una combinación de aislamiento de Ficoll y sedimentación dextrano31.
- Cultivo celular HL60 y diferenciación dirigida a neutrófilos
- Mantener células HL60 en medio Roswell Park Memorial Institute (RPMI)-1640 que contiene 10% de suero bovino fetal (FBS), 100 μg/ml de penicilina, 100 μg/ml de estreptomicina y 250 ng/ml de anfotericina B a 37 °C y 5% deCO2.
- Para la diferenciación dirigida a neutrófilos, mantener las células HL60 (células suspendidas) en un matraz T25 a una densidad de 1 × 105 células/ml en medio RPMI-1640 que contenga 10% de FBS, 100 μg/ml de penicilina, 100 μg/ml de estreptomicina, 250 ng/ml de anfotericina B y 1,3% de dimetilsulfóxido (DMSO) a 37 °C y 5% deCO2 durante 6 días.
- El día del ensayo, contar las células manualmente con el hemocitómetro, centrifugar las células HL60 diferenciadas o indiferenciadas a 300 × g a RT durante 5 min, lavar con DMEM una vez y resuspender las células con el medio de ensayo para obtener una densidad celular de 1,39 × 106 células/ml.
- Sembrar 180 μL de células HL60 diferenciadas o indiferenciadas (~2,5 × 105) por pocillo en la placa preparada de 96 pocillos y centrifugar la placa a 300 × g a RT durante 3 minutos sin freno para asegurar la adherencia adecuada de las células en el fondo de la placa.
- Incubar la placa en una incubadora humidificada a 37 °C sinCO2 durante 1 hora para preequilibrar las células con el medio de ensayo.
NOTA: Confirme la adhesión completa de las células utilizando un microscopio antes del siguiente paso.
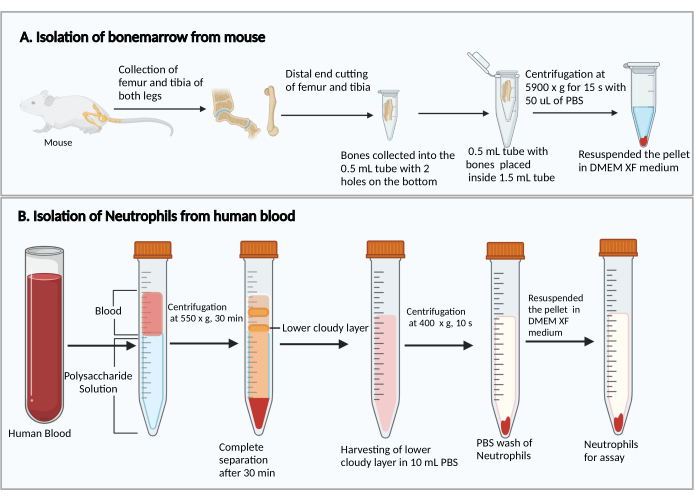
Figura 1: Diagrama esquemático del aislamiento de células de médula ósea y neutrófilos. (A) Recolección de células de médula ósea de un ratón y (B) aislamiento de neutrófilos de sangre humana. Haga clic aquí para ver una versión más grande de esta figura.
| Tipo de célula | Células por pocillo | Compuestos/Reactivos | Concentración de la solución de trabajo | Volumen de inyección a puertos | Concentración final en pozos |
| Neutrófilos de ratón | 2 × 105 | Oligomicina | 25 μM | 20 μL | 2,5 μM |
| FCCP | 7,5 μM | 17,6 μL | 0,61 μM | ||
| Rotenone Antimicina Una mezcla | 10 μM | 24 μL | 1 μM | ||
| Neutrófilos humanos | 4 × 105 | Oligomicina | 10 μM | 20 μL | 1 μM |
| FCCP | 12,5 μM | 22 μL | 1,25 μM | ||
| Rotenone Antimicina Una mezcla | 10 μM | 24 μL | 1 μM | ||
| Células HL60 indiferenciadas o diferenciadas | 2,5 × 105 | Oligomicina | 25 μM | 20 μL | 2,5 μM |
| FCCP | 15 μM | 22 μL | 1,5 μM | ||
| Rotenone Antimicina Una mezcla | 10 μM | 24 μL | 1 μM |
Tabla 1: Número de células y concentraciones de reactivos para la prueba de esfuerzo mitocondrial.
3. Preparación de compuestos en el kit de prueba de esfuerzo mitocondrial
- Abra el kit de prueba de esfuerzo mitocondrial y prepare los reactivos.
NOTA: Las diferentes concentraciones de solución de trabajo, el volumen de inyección en los pocillos y las concentraciones finales en los pocillos de oligomicina, carbonilcianuro p-trifluorometoxifenilhidrazona (FCCP) y mezcla de rotenona/antimicina A utilizada para neutrófilos de ratón, neutrófilos humanos y células HL60 se muestran en la Tabla 1.- Preparar la solución madre de oligomicina reconstituyendo oligomicina con 630 μL de medio de ensayo para obtener una solución madre de 100 μM.
NOTA: Se recomienda utilizar la solución madre el mismo día.- Preparar la solución de trabajo de oligomicina para ensayos de neutrófilos de ratón y células HL60 mezclando 630 μL de solución madre con 1.890 μL de medio de ensayo para obtener una solución de trabajo de 25 μM.
- Preparar la solución de trabajo de oligomicina para el ensayo de neutrófilos humanos mezclando 300 μL de solución madre con 2.700 μL de medio de ensayo para obtener una solución de trabajo de 10 μM.
NOTA: La unión de la oligomicina a la subunidad Fo de la placa base de la FO/F1 ATPasa (ATP sintasa) impide la reentrada de protones en las mitocondrias e inhibe la síntesis de ATP32. Esto reduce significativamente el flujo de electrones a través de la cadena de transporte de electrones y el OCR. Sin embargo, el flujo de electrones no se detiene completamente debido a la fuga de protones a través de la membrana mitocondrial interna33.
- Preparar la solución madre de FCCP reconstituyendo FCCP con 720 μL de medio de ensayo para obtener una solución madre de 100 μM.
- Preparar la solución de trabajo para el ensayo de neutrófilos de ratón mezclando 300 μL de solución madre con 3.700 μL de medio de ensayo para obtener una solución de trabajo de 7,5 μM.
- Preparar la solución de trabajo para el ensayo de neutrófilos humanos mezclando 375 μL de solución madre con 2.625 μL de medio de ensayo para obtener una solución de trabajo de 12,5 μM.
- Preparar la solución de trabajo para el ensayo de células HL60 mezclando 720 μL de solución madre con 4.080 μL de medio de ensayo para obtener una solución de trabajo de 15 μM.
NOTA: La adición de FCCP revela la capacidad máxima de las mitocondrias para usar OXPHOS. Es un ácido soluble en lípidos, débil permeable a las mitocondrias que da como resultado la disipación del potencial transmembrana. La descarga del gradiente de protones a través de la membrana interna mitocondrial y el desvío del flujo de protones de la FO/F1 ATP sintasa da como resultado el desacoplamiento mitocondrial. Este efecto de desacoplamiento aumenta abruptamente el consumo de oxígeno mitocondrial para preservar el gradiente de protones34.
- Preparar la solución madre de rotenona/antimicina A reconstituyendo la mezcla rotenona/antimicina A con 540 μL de medio de ensayo para obtener una solución madre de 50 μM.
- Preparar rotenona/antimicina Una solución de trabajo para todos los ensayos mezclando 540 μL de solución madre con 2.160 μL de medio de ensayo para obtener una solución de trabajo de 10 μM.
NOTA: La rotenona bloquea el complejo I inhibiendo la transferencia de electrones desde los centros de hierro-azufre en el complejo I a la ubiquinona, mientras que la antimicina A bloquea el complejo III de la cadena de transporte de electrones, lo que lleva a un bloqueo de OXPHOS con una síntesis limitada de ATP35. De este modo, esto revela respiración no mitocondrial36,37.
- Preparar rotenona/antimicina Una solución de trabajo para todos los ensayos mezclando 540 μL de solución madre con 2.160 μL de medio de ensayo para obtener una solución de trabajo de 10 μM.
- Preparar la solución madre de oligomicina reconstituyendo oligomicina con 630 μL de medio de ensayo para obtener una solución madre de 100 μM.
- Cargue el cartucho con reactivos; cargar la mezcla preparada de oligomicina, FCCP y rotenona/antimicina A en los puertos A, B y C, respectivamente, en el cartucho para preparaciones inyectables (Figura 2). Utilice el inserto de plantilla disponible junto con el cartucho para facilitar la carga de reactivos en los puertos. Llene los puertos no utilizados con 20 μL de medio de ensayo.
- Para el ensayo de neutrófilos de ratón, cargue 20 μL de oligomicina (25 μM; cada pocillo de la placa de 96 pocillos tiene 180 μL de medio de ensayo al principio, por lo que la concentración final es de 2,5 μM) en los puertos A. Cargue 17,6 μL de FCCP (7,5 μM; concentración final: ~0,61 μM) en los puertos B. Cargue 24 μL de mezcla de rotenona/antimicina A (10 μM; concentración final: ~1 μM) en los puertos C.
- Para el ensayo de neutrófilos humanos, cargar 20 μL de oligomicina (10 μM; cada pocillo de la placa de 96 pocillos tiene 180 μL de medio de ensayo al principio, por lo que la concentración final es de 1 μM) en los puertos A. Cargue 22 μL de FCCP (12,5 μM; concentración final: ~1,25 μM) en los puertos B. Cargue 24 μL de mezcla de rotenona/antimicina A (10 μM; concentración final: ~1 μM) en los puertos C.
- Para el ensayo de células HL60 y dHL60, cargue 20 μL de oligomicina (25 μM; cada pocillo de la placa de 96 pocillos tiene 180 μL de medio de ensayo al principio, por lo que la concentración final es de 2,5 μM) en los puertos A. Cargue 22 μL de FCCP (15 μM; concentración final: ~1.5 μM) en los puertos B. Cargue 24 μL de mezcla de rotenona/antimicina A (10 μM; concentración final: ~1 μM) en los puertos C.
NOTA: Pipetear la solución del medicamento en el puerto sin tocar el fondo del puerto. No golpee la placa después de la carga para evitar fugas, ya que los líquidos son retenidos por las fuerzas capilares. Los pocillos de fondo de la placa de cultivo y los puertos del cartucho sensor se cargan con medio de ensayo o con el mismo puerto de carga de reactivos que en los pocillos de muestra para normalizar el efecto de los reactivos sobre los valores de fondo. Los reactivos deben agregarse a los puertos respectivos sin levantar el cartucho de la placa de utilidad que contiene el calibrante para evitar cualquier trampa de aire. Llene el último puerto de todos los pozos con el medio como se muestra en la Figura 2.

Figura 2: El cartucho de ensayo de esfuerzo mitocondrial y sus puertos de inyección. La imagen muestra el cartucho del ensayo de estrés mitocondrial y una imagen ampliada que muestra la carga de fármacos individuales / medio a los puertos. Abreviatura: FCCP = carbonilcianuro p-trifluorometoxifenilhidrazona. Haga clic aquí para ver una versión más grande de esta figura.
4. Ejecución del ensayo de esfuerzo mitocondrial
- Encienda el analizador metabólico y la computadora, abra el software Wave y haga clic en Heater On para configurar la máquina a 37 °C con al menos 5 horas de anticipación. Después de alcanzar la temperatura, la esquina inferior izquierda del software de la ola se muestra lista.
- Abra una plantilla para el kit de ensayo de estrés mitocondrial en el software. Haga clic en Definición de grupo en la barra de menú superior.
- Preconfigure por separado cada definición en el lado izquierdo, como estrategias de inyección, pretratamientos, medios de ensayo y tipos de células (biomaterial utilizado).
- Haga clic en estrategias de inyección en el lado izquierdo, luego haga clic en Agregar y nombre la condición de inyección como Neutrófilos humanos / Neutrófilos de ratón / HL60. Seleccione el puerto A-D haciendo clic en cada uno y haga clic en Agregar compuesto e ingrese Oligomicina/FCCP/Rotenona Antimicina A con las concentraciones respectivas (Tabla 1).
- Haga clic en el pretratamiento en el lado izquierdo, luego haga clic en Agregar y asígneles el nombre CD18 y Cell Tak por separado. Haga clic en el biomaterial utilizado en el lado izquierdo, luego haga clic en Agregar y nómbrelos como Neutrófilos humanos, Neutrófilos de ratón y HL60 / dHL60 con densidad de siembra.
- Defina los grupos haciendo clic en Agregar grupo (por ejemplo, para el ensayo de neutrófilos humanos) y haciendo clic en la definición subyacente (por ejemplo, Estrategias de inyección según la Tabla 1-Neutrófilos humanos, Pretratamiento como CD18 y tipo de célula como neutrófilos humanos).
- Haga clic en Mapa de placas en el menú superior para observar todos los grupos, con definiciones en el lado izquierdo y el mapa de placas a la derecha. Arrastre y suelte para agregar cada pozo al grupo mientras mantiene los cuatro pozos de esquina como Fondo (predeterminado).
- Configure el protocolo haciendo clic en Protocolo en el menú superior y defina tres ciclos de mezcla de línea de base, oligomicina, FCCP, rotenona y antimicina A estableciendo el tiempo en Mezclar como 3 min, Resto como 0 min y Medición como 7 min.
- Vaya a la página Ejecutar ensayo , proporcione la información de resumen del proyecto como referencia y haga clic en Iniciar ejecución.
- Proporcione la ubicación para guardar los archivos, de modo que todos los resultados se guarden después de completar el ensayo.
- Después de la carga automática del ensayo, espere a que se abra la bandeja para colocar el cartucho del sensor y la placa con 200 μL de calibrante (paso 1.1). Coloque la dirección del código de barras del cartucho mirando hacia la derecha. Ejecute la calibración haciendo clic en Estoy listo, lo que tarda aproximadamente 20 minutos.
- Después de la calibración, haga clic en Abrir bandeja. Reemplace la placa con la placa sembrada de celdas y haga clic en Cargar placa de celda para continuar el ensayo.
- Después de completar el ensayo, los datos se guardan automáticamente. Haga clic en Ver resultados y expórtelo como una hoja de cálculo u otro archivo de software de análisis.
- Grafique y analice los datos (Figura 3 y Figura 4).
- Calcular los parámetros respiratorios, incluida la respiración mitocondrial basal, la respiración ligada a fugas de protones, la respiración vinculada al ATP, la respiración máxima, la capacidad respiratoria sobrante y la respiración no mitocondrial (Figura 4A)38,39,40,41.
- Calcular la respiración mitocondrial basal restando el valor de OCR medido después de agregar la mezcla de rotenona/antimicina A del OCR antes de inyectar oligomicina (Figura 4A, a).
- Calcule la respiración ligada a fugas de protones restando el valor de OCR después de la inyección de mezcla de rotenona/antimicina A del valor de OCR medido después de la inyección de oligomicina (Figura 4A, b).
- Estimar la respiración ligada al ATP calculando la diferencia entre la respiración mitocondrial basal y la respiración ligada a fugas de protones. Reste el primer valor de OCR medido después de la inyección de oligomicina del primer valor de OCR antes de la inyección de oligomicina (Figura 4A,c).
- La respiración máxima es la frecuencia respiratoria máxima que una célula puede alcanzar después de agregar FCCP. Calcule esto restando el valor de OCR después de la inyección de mezcla de rotenona/antimicina A del valor de OCR medido después de la inyección de FCCP (Figura 4A, d).
- La capacidad respiratoria sobrante se refiere a la capacidad de una célula para satisfacer la mayor demanda de energía a través de OXPHOS. Calcule esto encontrando la diferencia entre la respiración máxima y la respiración mitocondrial basal (Figura 4A, e).
- La respiración no mitocondrial es la cantidad de oxígeno consumido por fuentes no mitocondriales. Mida esto después de la adición de la mezcla de rotenona/antimicina A (Figura 4A,f).
- Realizar análisis estadísticos utilizando la prueba t de Student para comparar diferentes parámetros de respiración de células HL60 indiferenciadas y diferenciadas. Considere p < 0,05 como estadísticamente significativo.
NOTA: Las réplicas con valores de OCR o ECAR por debajo de cero se consideran un error en la preparación de la muestra, la inyección del compuesto o la medición. Se excluyen de futuros análisis.
Resultados
Se muestra una dinámica representativa de OCR que indica los cambios en la respiración mitocondrial en respuesta a la mezcla de oligomicina, FCCP y rotenona/antimicina A de neutrófilos de ratón (Figura 3A), neutrófilos humanos (Figura 3B) y células HL60 indiferenciadas y diferenciadas (Figura 3C). En todas las células, el tratamiento con oligomicina disminuye el valor de OCR al inhibir el canal de protones de la ATP sintasa; ...
Discusión
El procedimiento estándar que mide la respiración mitocondrial de los neutrófilos utilizando el analizador de flujo extracelular metabólico está limitado por muchos factores, incluido el número de células, el crecimiento celular y la viabilidad. Cada concentración de compuesto varía entre el tipo y la fuente de células en este ensayo. La oligomicina y la rotenona/antimicina A se utilizan principalmente en una concentración similar entre la mayoría de los tipos de células. Sin embargo, como la frecuencia res...
Divulgaciones
Los autores declaran que no hay intereses financieros contrapuestos.
Agradecimientos
Agradecemos al Dr. Anthony T. Vella y a la Dra. Federica Aglianoin del Departamento de Inmunología de UConn Health por su capacitación en el uso del analizador de flujo extracelular metabólico, y a la Dra. Lynn Puddington en el Departamento de Inmunología de UConn Health por su apoyo a los instrumentos. Agradecemos a la Dra. Geneva Hargis de la Facultad de Medicina de UConn por su ayuda con la redacción científica y la edición de este manuscrito. Esta investigación fue apoyada por subvenciones de los Institutos Nacionales de Salud, el Instituto Nacional del Corazón, los Pulmones y la Sangre (R01HL145454), el Instituto Nacional de Ciencias Médicas Generales (R35GM147713 y P20GM139763), un fondo inicial de UConn Health y una beca de reingreso profesional de la Asociación Americana de Inmunólogos.
Materiales
| Name | Company | Catalog Number | Comments |
| 37 °C non-CO2 incubator | Precision | Economy Model 2EG | Instrument |
| Biorender | Software Application | ||
| Centrifuge | Eppendorf | Model 5810R | Instrument |
| Corning Cell-Tak Cell and Tissue Adhesive | Corning | 102416-100 | Reagent |
| EasySep Magnet | STEMCELL | 18000 | Magnet |
| EasySepMouse Neutrophil Enrichment kit | STEMCELL | 19762A | Reagents |
| Graphpad Prism 9 | Software Application | ||
| Human Serum Albumin Solution (25%) | GeminiBio | 800-120 | Reagents |
| Ketamine (VetaKet) | DAILYMED | NDC 59399-114-10 | Anesthetic |
| PBS | Cytiva | SH30256.01 | Reagents |
| Plate buckets | Eppendorf | UL155 | Accessory |
| PolymorphPrep | PROGEN | 1895 (previous 1114683) | polysaccharide solution |
| Purified mouse anti-human CD18 antibody | Biolegend | 302102 | Clone TS1/18 |
| RPMI 1640 Medium | Gibco | 11-875-093 | Reagents |
| Seahorse metabolic extracellular flux analyzer | Agilent | XFe96 | Instrument |
| Seahorse XF Cell Mito Stress Test Kit | Agilent | 103015-100 | mitochondrial stress test Kit |
| Swing-bucket rotor | Eppendorf | A-4-62 | Rotor |
| Vactrap 2 Vacum Trap | Fox Lifesciences | 3052101-FLS | Instrument |
| Wave | Software Application | ||
| XF 1.0 M Glucose Solution | Agilent | 103577-100 | Reagent |
| XF 100 mM Pyruvate Solution | Agilent | 103578-100 | Reagent |
| XF 200 mM Glutamine Solution | Agilent | 103579-100 | Reagent |
| XF DMEM medium | Agilent | 103575-100 | Reagent |
| XFe96 FluxPak | Agilent | 102601-100 | Material |
| Xylazine (AnaSed Injection) | DAILYMED | NDC 59399-110-20 | Anesthetic |
Referencias
- Demine, S., Renard, P., Arnould, T. Mitochondrial uncoupling: a key controller of biological processes in physiology and diseases. Cells. 8 (8), 795 (2019).
- Noguchi, M., Kasahara, A. Mitochondrial dynamics coordinate cell differentiation. Biochemical and Biophysical Research Communications. 500 (1), 59-64 (2018).
- Zhu, L., et al. Correlation between mitochondrial dysfunction, cardiovascular diseases, and traditional Chinese medicine. Evidence-Based Complementary and Alternative Medicine. 2020, e2902136 (2020).
- Kaarniranta, K., et al. Mechanisms of mitochondrial dysfunction and their impact on age-related macular degeneration. Progress in Retinal and Eye Research. 79, 100858 (2020).
- Onyango, I. G., Khan, S. M., Bennett, J. P. Mitochondria in the pathophysiology of Alzheimer's and Parkinson's diseases. Frontiers in Bioscience. 22 (5), 854-872 (2017).
- Loiseau, D., et al. Mitochondrial coupling defect in Charcot-Marie-Tooth type 2A disease. Annals of Neurology. 61 (4), 315-323 (2007).
- Zucker-Franklin, D. Electron microscopic studies of human granulocytes: structural variations related to function. Seminars in Hematology. 5 (2), 109-133 (1968).
- Karnovsky, M. L. The metabolism of leukocytes. Seminars in Hematology. 5 (2), 156-165 (1968).
- Bao, Y., et al. mTOR and differential activation of mitochondria orchestrate neutrophil chemotaxis. The Journal of Cell Biology. 210 (7), 1153-1164 (2015).
- Fossati, G., et al. The mitochondrial network of human neutrophils: role in chemotaxis, phagocytosis, respiratory burst activation, and commitment to apoptosis. Journal of Immunology. 170 (4), 1964-1972 (2003).
- Pryde, J. G., Walker, A., Rossi, A. G., Hannah, S., Haslett, C. Temperature-dependent arrest of neutrophil apoptosis. Failure of Bax insertion into mitochondria at 15 degrees C prevents the release of cytochrome c. The Journal of Biological Chemistry. 275 (43), 33574-33584 (2000).
- Maianski, N. A., Mul, F. P. J., van Buul, J. D., Roos, D., Kuijpers, T. W. Granulocyte colony-stimulating factor inhibits the mitochondria-dependent activation of caspase-3 in neutrophils. Blood. 99 (2), 672-679 (2002).
- Bao, Y., et al. Mitochondria regulate neutrophil activation by generating ATP for autocrine purinergic signaling. The Journal of Biological Chemistry. 289 (39), 26794-26803 (2014).
- Chouchani, E. T., et al. Ischaemic accumulation of succinate controls reperfusion injury through mitochondrial ROS. Nature. 515 (7527), 431-435 (2014).
- Hayashi, G., Cortopassi, G. Oxidative stress in inherited mitochondrial diseases. Free Radical Biology and Medicine. 88, 10-17 (2015).
- Mailloux, R. J. An update on mitochondrial reactive oxygen species production. Antioxidants. 9 (6), 472 (2020).
- Abuaita, B. H., et al. The IRE1α stress signaling axis is a key regulator of neutrophil antimicrobial effector function. Journal of Immunology. 207 (1), 210-220 (2021).
- Lood, C., et al. Neutrophil extracellular traps enriched in oxidized mitochondrial DNA are interferogenic and contribute to lupus-like disease. Nature Medicine. 22 (2), 146-153 (2016).
- Douda, D. N., Khan, M. A., Grasemann, H., Palaniyar, N. SK3 channel and mitochondrial ROS mediate NADPH oxidase-independent NETosis induced by calcium influx. Proceedings of the National Academy of Sciencesa. 112 (9), 2817-2822 (2015).
- Monteith, A. J., et al. Altered mitochondrial homeostasis during systemic lupus erythematosus impairs neutrophil extracellular trap formation rendering neutrophils ineffective at combating Staphylococcus aureus. Journal of Immunology. 208 (2), 454-463 (2022).
- Monteith, A. J., Miller, J. M., Beavers, W. N., Juttukonda, L. J., Skaar, E. P. Increased dietary manganese impairs neutrophil extracellular trap formation rendering neutrophils ineffective at combating Staphylococcus aureus. Infection and Immunity. 90 (3), 0068521 (2022).
- Monteith, A. J., et al. Mitochondrial calcium uniporter affects neutrophil bactericidal activity during Staphylococcus aureus infection. Infection and Immunity. 90 (2), 0055121 (2022).
- Cao, Z., et al. Roles of mitochondria in neutrophils. Frontiers in Immunology. 13, 934444 (2022).
- Papayannopoulos, V., Metzler, K. D., Hakkim, A., Zychlinsky, A. Neutrophil elastase and myeloperoxidase regulate the formation of neutrophil extracellular traps. The Journal of Cell Biology. 191 (3), 677-691 (2010).
- Fan, Z., Ley, K. Developing neutrophils must eat…themselves. Immunity. 47 (3), 393-395 (2017).
- Riffelmacher, T., et al. Autophagy-dependent generation of free fatty acids is critical for normal neutrophil differentiation. Immunity. 47 (3), 466-480 (2017).
- Amend, S. R., Valkenburg, K. C., Pienta, K. J. Murine hind limb long bone dissection and bone marrow isolation. Journal of Visualized Experiments. (110), e53936 (2016).
- Swamydas, M., Isolation Lionakis, M. S. purification and labeling of mouse bone marrow neutrophils for functional studies and adoptive transfer experiments. Journal of Visualized Experiments. (77), e50586 (2013).
- Gerner, M. C., et al. Packed red blood cells inhibit T-cell activation via ROS-dependent signaling pathways. The Journal of Biological Chemistry. 296, 100487 (2021).
- Zhang, Z. -. W., et al. Red blood cell extrudes nucleus and mitochondria against oxidative stress. IUBMB Life. 63 (7), 560-565 (2011).
- Kuhns, D. B., Priel, D. A. L., Chu, J., Zarember, K. A. Isolation and functional analysis of human neutrophils. Current Protocols in Immunology. 111, 1-16 (2015).
- Hearne, A., Chen, H., Monarchino, A., Wiseman, J. S. Oligomycin-induced proton uncoupling. Toxicology In Vitro. 67, 104907 (2020).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-Protocol. 8 (10), e2850 (2018).
- Nath, S. The molecular mechanism of ATP synthesis by F1F0-ATP synthase: a scrutiny of the major possibilities. Advances in Biochemical Engineering/Biotechnology. 74, 65-98 (2002).
- Heinz, S., et al. Mechanistic investigations of the mitochondrial complex I inhibitor rotenone in the context of pharmacological and safety evaluation. Scientific Reports. 7 (1), 45465 (2017).
- Hytti, M., et al. Antimycin A-induced mitochondrial damage causes human RPE cell death despite activation of autophagy. Oxidative Medicine and Cellular Longevity. 2019, 1583656 (2019).
- Malecki, M., Kamrad, S., Ralser, M., Bähler, J. Mitochondrial respiration is required to provide amino acids during fermentative proliferation of fission yeast. EMBO Reports. 21 (11), e50845 (2020).
- Divakaruni, A. S., Paradyse, A., Ferrick, D. A., Murphy, A. N., Jastroch, M. Analysis and interpretation of microplate-based oxygen consumption and pH data. Methods in Enzymology. 547, 309-354 (2014).
- Marchetti, P., Fovez, Q., Germain, N., Khamari, R., Kluza, J. Mitochondrial spare respiratory capacity: Mechanisms, regulation, and significance in non-transformed and cancer cells. The FASEB Journal. 34 (10), 13106-13124 (2020).
- Nicholas, D., et al. Advances in the quantification of mitochondrial function in primary human immune cells through extracellular flux analysis. PLoS One. 12 (2), e0170975 (2017).
- Tur, J., et al. Mitofusin 2 in macrophages links mitochondrial ROS production, cytokine release, phagocytosis, autophagy, and bactericidal activity. Cell Reports. 32 (8), 108079 (2020).
- Benz, R., McLaughlin, S. The molecular mechanism of action of the proton ionophore FCCP (carbonylcyanide p-trifluoromethoxyphenylhydrazone). Biophysical Journal. 41 (3), 381-398 (1983).
- Wettmarshausen, J., Perocchi, F. Assessing calcium-stimulated mitochondrial bioenergetics using the seahorse XF96 analyzer. Methods in Molecular Biology. 1925, 197-222 (2019).
- Forkink, M., et al. Mitochondrial hyperpolarization during chronic complex I inhibition is sustained by low activity of complex II, III, IV and V. Biochimica et Biophysica Acta. 1837 (8), 1247-1256 (2014).
- . Methods for Reducing Cell Growth Edge Effects in Agilent Seahorse XF Cell Culture Microplates Available from: https://www.agilent.com/cs/library/usermanuals/public/user-manual-methods-for-reducing-cell-growth-edge-effect-cell-analysis-5994-0240en-agilent.pdf (2019)
- Lundholt, B. K., Scudder, K. M., Pagliaro, L. A simple technique for reducing edge effect in cell-based assays. Journal of Biomolecular Screening. 8 (5), 566-570 (2003).
- Wu, D., Yotnda, P. Induction and testing of hypoxia in cell culture. Journal of Visualized Experiments. (54), e2899 (2011).
- Normalisation of Seahorse XFe96 metabolic assaysto cell number with Hoechst stain using well-scan mode on the CLARIOstar Plus. BMG Labtech Available from: https://www.bmglabtech.com/cn/normalisation-of-seahorse-xfe96-metabolic-assays-to-cell-number-with-hoechst-stain/ (2020)
- Yetkin-Arik, B., et al. The role of glycolysis and mitochondrial respiration in the formation and functioning of endothelial tip cells during angiogenesis. Scientific Reports. 9 (1), 12608 (2019).
- Jastroch, M., Divakaruni, A. S., Mookerjee, S., Treberg, J. R., Brand, M. D. Mitochondrial proton and electron leaks. Essays in Biochemistry. 47, 53-67 (2010).
- Jandl, R. C., et al. Termination of the respiratory burst in human neutrophils. The Journal of Clinical Investigation. 61 (5), 1176-1185 (1978).
- Azevedo, E. P., et al. A metabolic shift toward pentose phosphate pathway is necessary for amyloid fibril- and phorbol 12-myristate 13-acetate-induced neutrophil extracellular trap (NET) formation. The Journal of Biological Chemistry. 290 (36), 22174-22183 (2015).
- Six, E., et al. AK2 deficiency compromises the mitochondrial energy metabolism required for differentiation of human neutrophil and lymphoid lineages. Cell Death & Disease. 6 (8), e1856 (2015).
- Kumar, S., Dikshit, M. Metabolic insight of neutrophils in health and disease. Frontiers in Immunology. 10, 2099 (2019).
- Rodríguez-Espinosa, O., Rojas-Espinosa, O., Moreno-Altamirano, M. M. B., López-Villegas, E. O., Sánchez-García, F. J. Metabolic requirements for neutrophil extracellular traps formation. Immunology. 145 (2), 213-224 (2015).
- Invernizzi, F., et al. Microscale oxygraphy reveals OXPHOS impairment in MRC mutant cells. Mitochondrion. 12 (2), 328-335 (2012).
- Zenaro, E., et al. Neutrophils promote Alzheimer's disease-like pathology and cognitive decline via LFA-1 integrin. Nature Medicine. 21 (8), 880-886 (2015).
- Maianski, N. A., et al. Functional characterization of mitochondria in neutrophils: a role restricted to apoptosis. Cell Death and Differentiation. 11 (2), 143-153 (2004).
- Bergman, O., Ben-Shachar, D. Mitochondrial oxidative phosphorylation system (OXPHOS) deficits in schizophrenia. Canadian Journal of Psychiatry. 61 (8), 457-469 (2016).
- Zhou, W., Qu, J., Xie, S., Sun, Y., Yao, H. Mitochondrial dysfunction in chronic respiratory diseases: implications for the pathogenesis and potential therapeutics. Oxidative Medicine and Cellular Longevity. 2021, 5188306 (2021).
- Hirano, M., Emmanuele, V., Quinzii, C. M. Emerging therapies for mitochondrial diseases. Essays in Biochemistry. 62 (3), 467-481 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados