È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Misurazione in tempo reale del profilo bioenergetico mitocondriale dei neutrofili
In questo articolo
Riepilogo
Descriviamo protocolli graduali che misurano la respirazione mitocondriale di neutrofili di topo e umani e cellule HL60 utilizzando l'analizzatore di flusso extracellulare metabolico.
Abstract
I neutrofili sono la prima linea di difesa e i leucociti più abbondanti nell'uomo. Queste cellule effettrici svolgono funzioni come la fagocitosi e l'esplosione ossidativa e creano trappole extracellulari di neutrofili (NET) per la clearance microbica. Nuove conoscenze sulle attività metaboliche dei neutrofili sfidano il concetto iniziale che si basano principalmente sulla glicolisi. La misurazione precisa delle attività metaboliche può dispiegare diversi requisiti metabolici dei neutrofili, tra cui il ciclo dell'acido tricarbossilico (TCA) (noto anche come ciclo di Krebs), la fosforilazione ossidativa (OXPHOS), la via del pentoso fosfato (PPP) e l'ossidazione degli acidi grassi (FAO) in condizioni fisiologiche e in stati patologici. Questo articolo descrive un protocollo passo-passo e i prerequisiti per misurare il tasso di consumo di ossigeno (OCR) come indicatore della respirazione mitocondriale sui neutrofili derivati dal midollo osseo di topo, sui neutrofili derivati dal sangue umano e sulla linea cellulare HL60 simile ai neutrofili, utilizzando l'analisi del flusso metabolico su un analizzatore di flusso extracellulare metabolico. Questo metodo può essere utilizzato per quantificare le funzioni mitocondriali dei neutrofili in condizioni normali e di malattia.
Introduzione
I mitocondri svolgono un ruolo importante nella bioenergetica cellulare, che genera adenosina trifosfato (ATP) mediante fosforilazione ossidativa (OXPHOS). Oltre a questo, il ruolo dei mitocondri si estende alla generazione e alla disintossicazione delle specie reattive dell'ossigeno, alla regolazione del calcio della matrice citoplasmatica e mitocondriale, alla sintesi cellulare, al catabolismo e al trasporto di metaboliti all'interno della cellula1. La respirazione mitocondriale è essenziale in tutte le cellule, poiché la loro disfunzione può causare problemi metabolici 2, comprese le malattie cardiovascolari3 e un'ampia varietà di malattie neurodegenerative, come la degenerazione maculare legata all'età4, il morbo di Parkinson e l'Alzheimer5 e la malattia di Charcot-Marie-Tooth2 A (CMT2A)6.
Studi al microscopio elettronico sui neutrofili hanno rivelato che ci sono relativamente pochi mitocondri7 e fanno molto affidamento sulla glicolisi per la loro produzione di energia poiché i tassi di respirazione mitocondriale sono molto bassi8. Tuttavia, i mitocondri sono cruciali per le funzioni dei neutrofili, come la chemiotassi9 e l'apoptosi10,11,12. Uno studio precedente ha rivelato una complessa rete mitocondriale nei neutrofili umani con alto potenziale di membrana. La perdita di potenziale della membrana mitocondriale è un indicatore precoce dell'apoptosi dei neutrofili10. Il trattamento con disaccoppiatore mitocondriale cianuro di carbonile m-clorofenil idrazono (CCCP) ha mostrato una significativa inibizione della chemiotassi, insieme a un cambiamento nella morfologia mitocondriale 9,10.
Sebbene la fonte primaria di energia per i neutrofili sia la glicolisi, i mitocondri forniscono l'ATP che avvia l'attivazione dei neutrofili alimentando la prima fase della segnalazione purinergica, che aumenta la segnalazione Ca2+, amplifica la produzione di ATP mitocondriale e avvia le risposte funzionali dei neutrofili13. La disfunzione della catena respiratoria mitocondriale provoca un'eccessiva produzione di specie tossiche reattive dell'ossigeno (ROS) e porta a danni patogeni14,15,16. La NETosi, che è il processo di formazione di trappole extracellulari di neutrofili (NET), è una proprietà critica dei neutrofili che li aiuta a combattere contro i patogeni17 e contribuisce a molte condizioni patologiche, tra cui cancro, trombosi e disturbi autoimmuni18. I ROS di derivazione mitocondriale contribuiscono alla NETosi19, il DNA mitocondriale può essere un componente di NETs18 e l'omeostasi mitocondriale alterata compromette NETosis 20,21,22,23,24. Inoltre, durante la normale differenziazione o maturazione, la riprogrammazione metabolica dei neutrofili viene invertita limitando l'attività glicolitica, e si impegnano nella respirazione mitocondriale e mobilitano i lipidi intracellulari25,26.
L'analizzatore del flusso metabolico extracellulare può monitorare e quantificare continuamente la respirazione mitocondriale e la glicolisi delle cellule vive. L'analizzatore utilizza una cartuccia sensore a 96 pozzetti e due fluorofori per quantificare la concentrazione di ossigeno (O2) e le variazioni di pH. La cartuccia del sensore si trova sopra il monostrato cellulare durante il test e forma una microcamera alta ~ 200 nm. I fasci di fibre ottiche nell'analizzatore vengono utilizzati per eccitare i fluorofori e rilevare le variazioni di intensità fluorescente. Le variazioni in tempo reale della concentrazione di O2 e del pH vengono calcolate automaticamente e mostrate come tasso di consumo di ossigeno (OCR) e tasso di acidificazione extracellulare (ECAR). Ci sono quattro porte sulla cartuccia del sensore che consentono di caricare fino a quattro composti in ciascun pozzetto durante le misurazioni del test. Questo protocollo si concentra sulla quantificazione della respirazione mitocondriale dei neutrofili di topo e umani, nonché delle cellule HL60 simili ai neutrofili, utilizzando l'analizzatore del flusso extracellulare metabolico.
Protocollo
I campioni di sangue intero eparinizzato sono stati ottenuti da donatori umani sani dopo aver ottenuto il consenso informato, come approvato dall'Institutional Review Board di UConn Health in conformità con la Dichiarazione di Helsinki. Tutti gli esperimenti sugli animali hanno seguito le linee guida dell'UConn Health Institutional Animal Care and Use Committee (IACUC) e l'approvazione per l'uso dei roditori è stata ottenuta dall'UConn Health IACUC secondo i criteri delineati nella Guida per la cura e l'uso degli animali da laboratorio del National Institutes of Health. In questo studio sono stati utilizzati topi maschi C57BL / 6 a 6 settimane di età.
1. Preparazione della piastra a 96 pozzetti per il saggio del flusso metabolico extracellulare
- Una cartuccia sensore è confezionata sulla parte superiore della piastra a 96 pozzetti appositamente progettata per il saggio del flusso extracellulare metabolico. Idratare la cartuccia sollevandola con cautela e posizionare 200 μL/pozzetto di mezzo di calibrazione in ciascun pozzetto della piastra sottostante. Posizionare la cartuccia sulla piastra con calibrante in un incubatore non CO2, umidificato, a 37 °C per tutta la notte per idratare.
- In base al tipo di cellula, utilizzare un rivestimento specifico per la piastra di coltura per garantire l'adesione cellulare. Per le cellule HL60 indifferenziate e differenziate di neutrofili umani rivestire la piastra a 96 pozzetti con soluzione salina sterile tamponata fosfato (PBS) contenente 50 μL di 5 μg/mL di anticorpo CD18 anti-umano purificato di topo (Clone TS1/18) a 4 °C durante la notte. Per i neutrofili di topo, rivestire la piastra a 96 pozzetti con 25 μL di 22,4 μg/mL di Cell Tak a pH 8,0 in 0,1 M NaHCO3 a temperatura ambiente (RT) per 20 minuti.
- Lavare le piastre con 200 μL di PBS sterile due volte.
- Aggiungere il mezzo completo ai pozzetti angolari (A1, A12, H1 e H12) della piastra di coltura cellulare (senza celle) per la correzione dello sfondo nelle piastre di coltura cellulare durante la semina.
NOTA: L'evaporazione durante il rivestimento e la calibrazione può influire sul volume del mezzo di calibrazione e sulla normalizzazione. Utilizzare un vassoio o una camera con tessuto sterile bagnato con acqua e posizionare la piastra con il calibrante sopra di essa per evitare l'evaporazione.
2. Preparazione e semina delle cellule
- Preparazione del mezzo di analisi
- Preparare il terreno di analisi aggiungendo 1 mM di piruvato, 10 mM di glucosio e 2 mM di glutammina al mezzo Eagle modificato (DMEM) di Dulbecco.
- Isolamento dei neutrofili di topo dal midollo osseo
- Isolare i neutrofili di topo dal midollo osseo utilizzando il kit di arricchimento dei neutrofili del topo, secondo le istruzioni del produttore.
- Anestetizzare i topi mediante iniezione intraperitoneale (i.p.) di ketamina (125 mg/kg) e xilazina (12,5 mg/kg) e quindi eutanasia dei topi mediante lussazione cervicale.
- Raccogliere i femori e le tibie, come descritto in precedenza27. Brevemente, tagliare la pelle per esporre la gamba con i muscoli. Tagliare l'articolazione dell'anca per rimuovere la gamba dal corpo. Quindi, rimuovere i muscoli per raccogliere il femore e la tibia.
- Taglia le estremità più piccole dei femori e delle tibie. Tenendo le estremità tagliate verso il basso, metterle in una provetta da centrifuga da 0,5 mL con due fori per siringhe da 25 G perforati ad ago sul fondo e inserire la provetta da 0,5 mL in una provetta da centrifuga da 1,5 mL (Figura 1A). Aggiungere 50 μL di PBS nel tubo da 0,5 mL per prevenire l'essiccazione delle cellule del midollo osseo.
- Centrifugare a 5.900 × g a RT per 15 s per raccogliere il midollo osseo sul fondo del tubo da 1,5 ml. Risospendere le cellule del midollo osseo con 1 mL di DMEM. Aggiungere 50 μL di siero di ratto dal kit, mescolare delicatamente mediante pipettaggio e trasferire la sospensione cellulare in una provetta per colture di polistirene da 5 ml.
- Aggiungere 50 μL di cocktail di arricchimento dal kit di arricchimento dei neutrofili del topo e incubare per 15 minuti a RT. Centrifugare a 300 × g a RT per 5 minuti.
- Risospendere le cellule con 1 mL di DMEM, aggiungere 50 μL di cocktail di selezione di biotina dal kit, mescolare delicatamente mediante pipettaggio e incubare per 15 minuti a RT.
- Aggiungere 150 μL di particelle magnetiche a vortice dal kit, mescolare delicatamente mediante pipettaggio e incubare per 10 minuti a RT.
- Aggiungere ~ 1,3 ml di DMEM, mescolare delicatamente mediante pipettaggio, posizionare il tubo nel magnete per 3 minuti e trasferire il surnatante contenente neutrofili di topo purificati in una nuova provetta di coltura in polistirene da 5 mL invertendo il tubo originale insieme al magnete.
- Centrifugare a 300 × g a RT per 5 min. Dopo aver rimosso il surnatante mediante aspirazione sotto vuoto, risospendere le cellule con 1 mL di mezzo di analisi.
- Contare le cellule manualmente utilizzando un emocitometro.
- Regolare la densità cellulare a 1,1 × 106 cellule/ml aggiungendo il mezzo di analisi, seminare 180 μL della sospensione di neutrofili di topo (2 × 105 cellule) per pozzetto nella piastra preparata a 96 pozzetti (fasi 1,2-1,4) e centrifugare la piastra a 300 × g a RT per 3 minuti senza freno per garantire la corretta aderenza delle cellule sul fondo della piastra.
- Incubare la piastra in un incubatore non CO2, umidificato, a 37 °C per 1 ora per preequilibrare le cellule con il mezzo di analisi.
NOTA: La purezza dei neutrofili è fondamentale per il test poiché è un potenziale pregiudizio. La purezza dell'isolamento dei neutrofili del topo è 69,9% -88,7% secondo questo protocollo. Esistono altri metodi per isolare i neutrofili dal midollo osseo di topo, come la centrifugazione a gradiente di densità28. Esistono anche kit alternativi di isolamento dei neutrofili di altri fornitori, basati sulla selezione magnetica negativa utilizzando gli anticorpi monoclonali contro antigeni che non sono espressi sui neutrofili.
- Isolamento dei neutrofili umani dal sangue periferico
- Aggiungere 8 ml di soluzione di polisaccaridi in una provetta da centrifuga da 15 ml, quindi sovrapporre 4 ml di sangue periferico sopra la soluzione di polisaccaridi senza mescolare.
- Centrifugare a 550 × g a 20 °C per 30 min. Fai decelerare il rotore a 1.
NOTA: Il tempo di separazione dei neutrofili può variare tra i donatori. È un minimo di 30 minuti e potrebbero essere necessari altri 10-20 minuti se la separazione dei neutrofili non ha successo. - Osservare la separazione di plasma/piastrine e cellule mononucleate dopo la centrifugazione, come mostrato nella Figura 1B. Rimuovere con cautela il liquido giallo superiore sulla parte superiore (plasma e piastrine) e la banda torbida superiore (cellule mononucleate) senza disturbare la banda torbida inferiore (neutrofili) utilizzando una pipetta da 1 ml.
- Raccogliere la banda torbida inferiore e ~ 3-4 ml del liquido trasparente sottostante in una nuova provetta da centrifuga da 15 ml contenente 10 ml di PBS.
- Centrifugare a 400 × g a 20 °C per 10 minuti e rimuovere il surnatante mediante aspirazione sotto vuoto.
- Risospendere le cellule con 5 mL di PBS e centrifugare a 300 × g a 20 °C per 5 minuti.
- Dopo aver rimosso il surnatante, risospendere le cellule con 1 mL di terreno di analisi.
- Contare le cellule manualmente utilizzando un emocitometro.
NOTA: Poiché gli eritrociti nel sangue non hanno mitocondri, la contaminazione eritrocitaria non influisce sul test da sforzo mitocondriale e impedisce l'attivazione/priming dei neutrofili29,30. Gli eritrociti devono essere lisati durante il conteggio delle cellule per ottenere una concentrazione accurata di neutrofili. Aggiungere 10 μL della sospensione cellulare a 891 μL di acqua deionizzata per 10-30 s per lisare gli eritrociti, quindi aggiungere 99 μL di 10x PBS per bilanciare la pressione osmotica, evitando la lisi dei neutrofili. - Regolare la densità cellulare a 2,2 × 106 cellule / ml aggiungendo terreno di analisi, seminare 180 μL di neutrofili umani (~ 4 × 105) per pozzetto nella piastra a 96 pozzetti preparata e centrifugare la piastra a 300 × g a RT per 3 minuti senza freno per garantire la corretta aderenza delle cellule sul fondo della piastra.
- Incubare la piastra in un incubatore non CO2, umidificato, a 37 °C per 1 ora per preequilibrare le cellule con il mezzo di analisi.
NOTA: La purezza dei neutrofili è fondamentale per il test poiché è un potenziale pregiudizio. La purezza dell'isolamento dei neutrofili umani è 86,6%-96,8%. Esistono altri metodi di centrifugazione a gradiente di densità per isolare i neutrofili dal sangue umano, incluso l'isolamento di Percoll e una combinazione di isolamento di Ficoll e sedimentazione del destrano31.
- Coltura cellulare HL60 e differenziazione diretta dai neutrofili
- Mantenere le cellule HL60 nel terreno di coltura del Roswell Park Memorial Institute (RPMI)-1640 contenente il 10% di siero bovino fetale (FBS), 100 μg/mL di penicillina, 100 μg/mL di streptomicina e 250 ng/mL di amfotericina B a 37 °C e 5% di CO2.
- Per la differenziazione diretta dai neutrofili, mantenere le cellule HL60 (cellule sospese) in un matraccio T25 ad una densità di 1 × 10 5 cellule/ml in terreno RPMI-1640 contenente il 10% di FBS, 100 μg/mL di penicillina, 100 μg/mL di streptomicina, 250 ng/mL di amfotericina B e 1,3% di dimetil solfossido (DMSO) a 37 °C e5 % di CO2 per 6 giorni.
- Il giorno del test, contare le cellule manualmente utilizzando l'emocitometro, centrifugare le cellule HL60 differenziate o indifferenziate a 300 × g a RT per 5 minuti, lavare con DMEM una volta e risospendere le cellule con il mezzo di analisi per ottenere una densità cellulare di 1,39 × 106 cellule / ml.
- Seminare 180 μL di cellule HL60 differenziate o indifferenziate (~2,5 × 105) per pozzetto nella piastra a 96 pozzetti preparata e centrifugare la piastra a 300 × g a RT per 3 minuti senza freno per garantire la corretta aderenza delle cellule sul fondo della piastra.
- Incubare la piastra in un incubatore non CO2, umidificato, a 37 °C per 1 ora per preequilibrare le cellule con il mezzo di analisi.
NOTA: Confermare la completa adesione delle cellule utilizzando un microscopio prima del passaggio successivo.
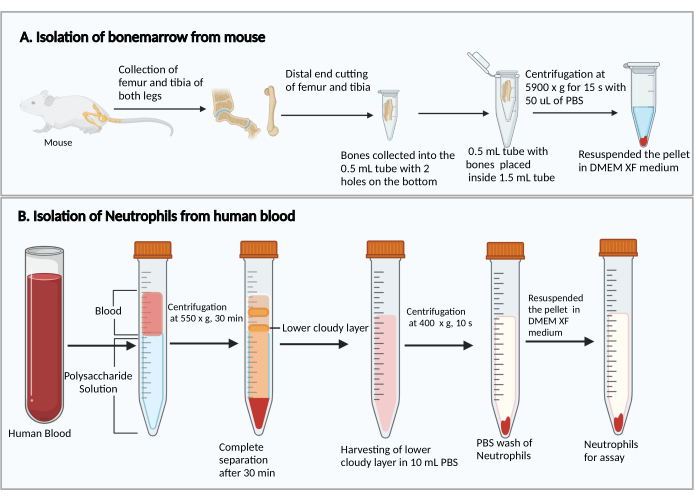
Figura 1: Diagramma schematico dell'isolamento delle cellule del midollo osseo e dei neutrofili. (A) Raccolta di cellule del midollo osseo da un topo e (B) isolamento dei neutrofili dal sangue umano. Fare clic qui per visualizzare una versione ingrandita di questa figura.
| Tipo di cella | Celle per pozzetto | Compound/Reagenti | Concentrazione della soluzione di lavoro | Volume di iniezione alle porte | Concentrazione finale nei pozzi |
| Neutrofili di topo | 2 × 105 | Oligomicina | 25 μM | 20 μL | 2,5 μM |
| FCCP | 7,5 μM | 17,6 μL | 0,61 μM | ||
| Rotenone Antimicina Una miscela | 10 μM | 24 μL | 1 μM | ||
| Neutrofili umani | 4 × 105 | Oligomicina | 10 μM | 20 μL | 1 μM |
| FCCP | 12,5 μM | 22 μL | 1,25 μM | ||
| Rotenone Antimicina Una miscela | 10 μM | 24 μL | 1 μM | ||
| Cellule HL60 indifferenziate o differenziate | 2.5 × 105 | Oligomicina | 25 μM | 20 μL | 2,5 μM |
| FCCP | 15 μM | 22 μL | 1,5 μM | ||
| Rotenone Antimicina Una miscela | 10 μM | 24 μL | 1 μM |
Tabella 1: Numero di cellule e concentrazioni di reagenti per il test da sforzo mitocondriale.
3. Preparazione dei composti nel kit di test da sforzo mitocondriale
- Aprire il kit per il test da sforzo mitocondriale e preparare i reagenti.
NOTA: Le diverse concentrazioni della soluzione di lavoro, il volume di iniezione nei pozzetti e le concentrazioni finali nei pozzetti di oligomicina, carbonilcianuro p-trifluorometossifenilidrazone (FCCP) e rotenone/antimicina Una miscela utilizzata per neutrofili di topo, neutrofili umani e cellule HL60 sono mostrati nella Tabella 1.- Preparare la soluzione madre di oligomicina ricostituendo oligomicina con 630 μL di terreno di analisi per ottenere una soluzione madre da 100 μM.
NOTA: si consiglia di utilizzare la soluzione madre lo stesso giorno.- Preparare la soluzione di lavoro di oligomicina per i saggi di neutrofili di topo e cellule HL60 miscelando 630 μL di soluzione madre con 1.890 μL di terreno di analisi per ottenere una soluzione di lavoro da 25 μM.
- Preparare la soluzione di lavoro di oligomicina per il saggio dei neutrofili umani miscelando 300 μL di soluzione madre con 2.700 μL di mezzo di analisi per ottenere una soluzione di lavoro da 10 μM.
NOTA: Il legame dell'oligomicina alla subunità della piastra di base Fo della Fo/F1 ATPasi (ATP sintasi) impedisce il rientro dei protoni nei mitocondri e inibisce la sintesi di ATP32. Ciò riduce significativamente il flusso di elettroni attraverso la catena di trasporto degli elettroni e l'OCR. Tuttavia, il flusso di elettroni non si ferma completamente a causa della perdita di protoni attraverso la membrana mitocondriale interna33.
- Preparare la soluzione madre FCCP ricostituendo FCCP con 720 μL di terreno di analisi per ottenere una soluzione madre da 100 μM.
- Preparare la soluzione di lavoro per il saggio dei neutrofili di topo mescolando 300 μL di soluzione madre con 3.700 μL di terreno di analisi per ottenere una soluzione di lavoro da 7,5 μM.
- Preparare la soluzione di lavoro per il saggio dei neutrofili umani miscelando 375 μL di soluzione madre con 2.625 μL di terreno di analisi per ottenere una soluzione di lavoro da 12,5 μM.
- Preparare la soluzione di lavoro per il test della cella HL60 miscelando 720 μL di soluzione madre con 4.080 μL di terreno di analisi per ottenere una soluzione di lavoro da 15 μM.
NOTA: L'aggiunta di FCCP rivela la capacità massima dei mitocondri di utilizzare OXPHOS. È un acido liposolubile, debole permeabile ai mitocondri provoca la dissipitazione del potenziale transmembrana. La scarica del gradiente protonico attraverso la membrana interna mitocondriale e la deviazione del flusso protonico dalla fostasi Fo/F1 ATP provoca il disaccoppiamento mitocondriale. Questo effetto disaccoppiante aumenta bruscamente il consumo di ossigeno mitocondriale per preservare il gradiente protonico34.
- Preparare rotenone/antimicina A ricostituendo la miscela rotenone/antimicina A con 540 μL di terreno di analisi per ottenere una soluzione madre da 50 μM.
- Preparare rotenone/antimicina Una soluzione di lavoro per tutti i saggi miscelando 540 μL di soluzione madre con 2.160 μL di terreno di analisi per ottenere una soluzione di lavoro da 10 μM.
NOTA: Il rotenone blocca il complesso I inibendo il trasferimento di elettroni dai centri ferro-zolfo nel complesso I all'ubichinone, mentre l'antimicina A blocca il complesso III della catena di trasporto degli elettroni, portando ad un blocco di OXPHOS con una sintesi limitata di ATP35. In tal modo, questo rivela la respirazione non mitocondriale36,37.
- Preparare rotenone/antimicina Una soluzione di lavoro per tutti i saggi miscelando 540 μL di soluzione madre con 2.160 μL di terreno di analisi per ottenere una soluzione di lavoro da 10 μM.
- Preparare la soluzione madre di oligomicina ricostituendo oligomicina con 630 μL di terreno di analisi per ottenere una soluzione madre da 100 μM.
- Caricare la cartuccia con i reagenti; caricare la miscela preparata di oligomicina, FCCP e rotenone/antimicina A rispettivamente nelle porte A, B e C nella cartuccia per iniezioni (Figura 2). Utilizzare l'inserto del modello disponibile insieme alla cartuccia per facilitare il caricamento dei reagenti alle porte. Riempire le porte inutilizzate con 20 μL di mezzo di analisi.
- Per il test dei neutrofili di topo, caricare 20 μL di oligomicina (25 μM; ogni pozzetto della piastra a 96 pozzetti ha 180 μL di mezzo di analisi all'inizio, quindi la concentrazione finale è di 2,5 μM) nelle porte A. Caricare 17,6 μL di FCCP (7,5 μM; concentrazione finale: ~0,61 μM) nelle porte B. Caricare 24 μL di miscela rotenone/antimicina A (10 μM; concentrazione finale: ~1 μM) nelle porte C.
- Per il saggio dei neutrofili umani, caricare 20 μL di oligomicina (10 μM; ogni pozzetto della piastra a 96 pozzetti ha 180 μL di mezzo di analisi all'inizio, quindi la concentrazione finale è di 1 μM) nelle porte A. Caricare 22 μL di FCCP (12,5 μM; concentrazione finale: ~1,25 μM) nelle porte B. Caricare 24 μL di miscela rotenone/antimicina A (10 μM; concentrazione finale: ~1 μM) nelle porte C.
- Per il saggio delle cellule HL60 e dHL60, caricare 20 μL di oligomicina (25 μM; ogni pozzetto della piastra a 96 pozzetti ha 180 μL di mezzo di saggio all'inizio, quindi la concentrazione finale è di 2,5 μM) nelle porte A. Caricare 22 μL di FCCP (15 μM; concentrazione finale: ~1,5 μM) nelle porte B. Caricare 24 μL di miscela rotenone/antimicina A (10 μM; concentrazione finale: ~1 μM) nelle porte C.
NOTA: pipettare la soluzione farmacologica nella porta senza toccare il fondo della porta. Non toccare la piastra dopo il caricamento per evitare perdite, poiché i liquidi sono trattenuti dalle forze capillari. I pozzetti di fondo della piastra di coltura e le porte sulla cartuccia del sensore sono caricati con il mezzo di analisi o con la stessa porta di caricamento dei reagenti come nei pozzetti campione per normalizzare l'effetto dei reagenti sui valori di fondo. I reagenti devono essere aggiunti alle rispettive porte senza sollevare la cartuccia dalla piastra contenente il calibrante per evitare trappole d'aria. Riempire l'ultima porta di tutti i pozzi con il mezzo come mostrato nella Figura 2.

Figura 2: La cartuccia per il saggio dello stress mitocondriale e le loro porte di iniezione. L'immagine mostra la cartuccia del saggio di stress mitocondriale e un'immagine ingrandita che mostra il caricamento di singoli farmaci / mezzo alle porte. Abbreviazione: FCCP = carbonilcianuro p-trifluorometossi fenilidrazone. Fare clic qui per visualizzare una versione ingrandita di questa figura.
4. Esecuzione del test di stress mitocondriale
- Accendere l'analizzatore metabolico e il computer, aprire il software Wave e fare clic su Riscaldatore acceso per impostare la macchina a 37 °C con almeno 5 ore di anticipo. Dopo aver raggiunto la temperatura, l'angolo in basso a sinistra del software wave mostra pronto.
- Aprire un modello per il kit di analisi dello stress mitocondriale nel software. Fai clic su Definizione gruppo nella barra dei menu in alto.
- Preimpostare separatamente ogni definizione sul lato sinistro, come strategie di iniezione, pretrattamenti, mezzi di analisi e tipi di cellule (biomateriale utilizzato).
- Fare clic sulle strategie di iniezione sul lato sinistro, quindi fare clic su Aggiungi e nominare la condizione di iniezione come neutrofili umani / neutrofili di topo / HL60. Selezionare la porta A-D facendo clic su ciascuna e fare clic su Aggiungi composto e inserire Oligomicina / FCCP / Rotenone Antimicina A con le rispettive concentrazioni (Tabella 1).
- Fare clic sul pretrattamento sul lato sinistro, quindi fare clic su Aggiungi e nominarli separatamente come CD18 e Cell Tak. Fare clic sul biomateriale utilizzato sul lato sinistro, quindi fare clic su Aggiungi e nominarli come neutrofili umani, neutrofili di topo e HL60 / dHL60 con densità di semina.
- Definire i gruppi facendo clic su Aggiungi gruppo (ad esempio, per il test dei neutrofili umani) e facendo clic sulla definizione sottostante (ad esempio, Strategie di iniezione secondo la Tabella 1-Neutrofili umani, Pretrattamento come CD18 e tipo di cellula come Neutrofili umani).
- Clicca su Mappa delle piastre nel menu in alto per osservare tutti i gruppi, con le definizioni sul lato sinistro e la mappa della targa sulla destra. Trascinate e rilasciate per aggiungere ogni pozzetto al gruppo mantenendo i quattro pozzetti angolari come Sfondo (impostazione predefinita).
- Imposta il protocollo facendo clic su Protocollo nel menu in alto e definisci tre cicli di basale, oligomicina, FCCP, rotenone e antimicina A impostando il tempo su Mescolare come 3 min, Riposo come 0 min e Misurazione come 7 min.
- Passare alla pagina Esegui analisi , fornire le informazioni di riepilogo del progetto come riferimento e fare clic su Avvia esecuzione.
- Indicare la posizione in cui salvare i file, in modo che tutti i risultati vengano salvati dopo il completamento del test.
- Dopo il caricamento automatico del test, attendere che il vassoio si apra per posizionare la cartuccia del sensore e la piastra con 200 μL di calibrante (passaggio 1.1). Impostare la direzione del codice a barre della cartuccia rivolta verso destra. Esegui la calibrazione facendo clic su Sono pronto, che richiede circa 20 minuti.
- Dopo la calibrazione, fare clic su Apri vassoio. Sostituire la piastra con la piastra seminata dalla cellula e fare clic su Load Cell Plate per continuare il test.
- Dopo aver completato il test, i dati vengono salvati automaticamente. Fare clic su Visualizza risultati ed esportarlo come foglio di calcolo o altro file software di analisi.
- Rappresentare graficamente e analizzare i dati (Figura 3 e Figura 4).
- Calcolare i parametri respiratori, tra cui la respirazione mitocondriale basale, la respirazione legata alla perdita di protoni, la respirazione legata all'ATP, la respirazione massima, la capacità respiratoria di riserva e la respirazione non mitocondriale (Figura 4A) 38,39,40,41.
- Calcolare la respirazione mitocondriale basale sottraendo il valore OCR misurato dopo aver aggiunto rotenone/antimicina A miscela dall'OCR prima di iniettare oligomicina (Figura 4A, a).
- Calcolare la respirazione legata alla perdita di protoni sottraendo il valore OCR dopo iniezione di miscela rotenone/antimicina A dal valore OCR misurato dopo iniezione di oligomicina (Figura 4A,b).
- Stimare la respirazione legata all'ATP calcolando la differenza tra la respirazione mitocondriale basale e la respirazione legata alla perdita di protoni. Sottrarre il primo valore OCR misurato dopo l'iniezione di oligomicina dal primo valore OCR prima dell'iniezione di oligomicina (Figura 4A,c).
- La respirazione massima è la velocità di respirazione massima che una cellula può raggiungere dopo l'aggiunta di FCCP. Calcolarlo sottraendo il valore OCR dopo iniezione di miscela rotenone/antimicina A dal valore OCR misurato dopo iniezione FCCP (Figura 4A,d).
- La capacità respiratoria di riserva si riferisce alla capacità di una cellula di soddisfare la maggiore domanda di energia attraverso OXPHOS. Calcola questo trovando la differenza tra la respirazione massima e la respirazione mitocondriale basale (Figura 4A, e).
- La respirazione non mitocondriale è la quantità di ossigeno consumata da fonti non mitocondriali. Misurare questo dopo l'aggiunta di miscela rotenone/antimicina A (Figura 4A,f).
- Eseguire analisi statistiche utilizzando il t-test di Student per confrontare diversi parametri respiratori di cellule HL60 indifferenziate e differenziate. Considera p < 0,05 statisticamente significativo.
NOTA: le repliche con valori OCR o ECAR inferiori allo zero sono considerate un errore nella preparazione del campione, nell'iniezione di composti o nella misurazione. Sono esclusi dall'analisi futura.
Risultati
Le dinamiche OCR rappresentative sono mostrate indicando i cambiamenti della respirazione mitocondriale in risposta a oligomicina, FCCP e rotenone / antimicina Una miscela di neutrofili di topo (Figura 3A), neutrofili umani (Figura 3B) e cellule HL60 indifferenziate e differenziate (Figura 3C). In tutte le cellule, il trattamento con oligomicina diminuisce il valore OCR inibendo il canale protonico dell'ATP sintasi; Il trattamento F...
Discussione
La procedura standard che misura la respirazione mitocondriale dei neutrofili utilizzando l'analizzatore del flusso extracellulare metabolico è limitata da molti fattori, tra cui il numero di cellule, la crescita cellulare e la vitalità. Ogni concentrazione di composto varia tra il tipo e la fonte di cellule in questo test. Oligomicina e rotenone / antimicina A sono per lo più utilizzati in una concentrazione simile tra la maggior parte dei tipi di cellule. Tuttavia, poiché la frequenza respiratoria massima indotta ...
Divulgazioni
Gli autori non dichiarano alcun interesse finanziario concorrente.
Riconoscimenti
Riconosciamo il Dr. Anthony T. Vella e la Dr. Federica Aglianoin del Dipartimento di Immunologia di UConn Health per la loro formazione nell'uso dell'analizzatore di flusso extracellulare metabolico e la Dr. Lynn Puddington del Dipartimento di Immunologia di UConn Health per il suo supporto agli strumenti. Riconosciamo la dottoressa Geneva Hargis della UConn School of Medicine per il suo aiuto nella scrittura scientifica e nella revisione di questo manoscritto. Questa ricerca è stata supportata da sovvenzioni del National Institutes of Health, National Heart, Lung, and Blood Institute (R01HL145454), National Institute of General Medical Sciences (R35GM147713 e P20GM139763), un fondo di avvio di UConn Health e una borsa di rientro professionale dell'American Association of Immunologists.
Materiali
| Name | Company | Catalog Number | Comments |
| 37 °C non-CO2 incubator | Precision | Economy Model 2EG | Instrument |
| Biorender | Software Application | ||
| Centrifuge | Eppendorf | Model 5810R | Instrument |
| Corning Cell-Tak Cell and Tissue Adhesive | Corning | 102416-100 | Reagent |
| EasySep Magnet | STEMCELL | 18000 | Magnet |
| EasySepMouse Neutrophil Enrichment kit | STEMCELL | 19762A | Reagents |
| Graphpad Prism 9 | Software Application | ||
| Human Serum Albumin Solution (25%) | GeminiBio | 800-120 | Reagents |
| Ketamine (VetaKet) | DAILYMED | NDC 59399-114-10 | Anesthetic |
| PBS | Cytiva | SH30256.01 | Reagents |
| Plate buckets | Eppendorf | UL155 | Accessory |
| PolymorphPrep | PROGEN | 1895 (previous 1114683) | polysaccharide solution |
| Purified mouse anti-human CD18 antibody | Biolegend | 302102 | Clone TS1/18 |
| RPMI 1640 Medium | Gibco | 11-875-093 | Reagents |
| Seahorse metabolic extracellular flux analyzer | Agilent | XFe96 | Instrument |
| Seahorse XF Cell Mito Stress Test Kit | Agilent | 103015-100 | mitochondrial stress test Kit |
| Swing-bucket rotor | Eppendorf | A-4-62 | Rotor |
| Vactrap 2 Vacum Trap | Fox Lifesciences | 3052101-FLS | Instrument |
| Wave | Software Application | ||
| XF 1.0 M Glucose Solution | Agilent | 103577-100 | Reagent |
| XF 100 mM Pyruvate Solution | Agilent | 103578-100 | Reagent |
| XF 200 mM Glutamine Solution | Agilent | 103579-100 | Reagent |
| XF DMEM medium | Agilent | 103575-100 | Reagent |
| XFe96 FluxPak | Agilent | 102601-100 | Material |
| Xylazine (AnaSed Injection) | DAILYMED | NDC 59399-110-20 | Anesthetic |
Riferimenti
- Demine, S., Renard, P., Arnould, T. Mitochondrial uncoupling: a key controller of biological processes in physiology and diseases. Cells. 8 (8), 795 (2019).
- Noguchi, M., Kasahara, A. Mitochondrial dynamics coordinate cell differentiation. Biochemical and Biophysical Research Communications. 500 (1), 59-64 (2018).
- Zhu, L., et al. Correlation between mitochondrial dysfunction, cardiovascular diseases, and traditional Chinese medicine. Evidence-Based Complementary and Alternative Medicine. 2020, e2902136 (2020).
- Kaarniranta, K., et al. Mechanisms of mitochondrial dysfunction and their impact on age-related macular degeneration. Progress in Retinal and Eye Research. 79, 100858 (2020).
- Onyango, I. G., Khan, S. M., Bennett, J. P. Mitochondria in the pathophysiology of Alzheimer's and Parkinson's diseases. Frontiers in Bioscience. 22 (5), 854-872 (2017).
- Loiseau, D., et al. Mitochondrial coupling defect in Charcot-Marie-Tooth type 2A disease. Annals of Neurology. 61 (4), 315-323 (2007).
- Zucker-Franklin, D. Electron microscopic studies of human granulocytes: structural variations related to function. Seminars in Hematology. 5 (2), 109-133 (1968).
- Karnovsky, M. L. The metabolism of leukocytes. Seminars in Hematology. 5 (2), 156-165 (1968).
- Bao, Y., et al. mTOR and differential activation of mitochondria orchestrate neutrophil chemotaxis. The Journal of Cell Biology. 210 (7), 1153-1164 (2015).
- Fossati, G., et al. The mitochondrial network of human neutrophils: role in chemotaxis, phagocytosis, respiratory burst activation, and commitment to apoptosis. Journal of Immunology. 170 (4), 1964-1972 (2003).
- Pryde, J. G., Walker, A., Rossi, A. G., Hannah, S., Haslett, C. Temperature-dependent arrest of neutrophil apoptosis. Failure of Bax insertion into mitochondria at 15 degrees C prevents the release of cytochrome c. The Journal of Biological Chemistry. 275 (43), 33574-33584 (2000).
- Maianski, N. A., Mul, F. P. J., van Buul, J. D., Roos, D., Kuijpers, T. W. Granulocyte colony-stimulating factor inhibits the mitochondria-dependent activation of caspase-3 in neutrophils. Blood. 99 (2), 672-679 (2002).
- Bao, Y., et al. Mitochondria regulate neutrophil activation by generating ATP for autocrine purinergic signaling. The Journal of Biological Chemistry. 289 (39), 26794-26803 (2014).
- Chouchani, E. T., et al. Ischaemic accumulation of succinate controls reperfusion injury through mitochondrial ROS. Nature. 515 (7527), 431-435 (2014).
- Hayashi, G., Cortopassi, G. Oxidative stress in inherited mitochondrial diseases. Free Radical Biology and Medicine. 88, 10-17 (2015).
- Mailloux, R. J. An update on mitochondrial reactive oxygen species production. Antioxidants. 9 (6), 472 (2020).
- Abuaita, B. H., et al. The IRE1α stress signaling axis is a key regulator of neutrophil antimicrobial effector function. Journal of Immunology. 207 (1), 210-220 (2021).
- Lood, C., et al. Neutrophil extracellular traps enriched in oxidized mitochondrial DNA are interferogenic and contribute to lupus-like disease. Nature Medicine. 22 (2), 146-153 (2016).
- Douda, D. N., Khan, M. A., Grasemann, H., Palaniyar, N. SK3 channel and mitochondrial ROS mediate NADPH oxidase-independent NETosis induced by calcium influx. Proceedings of the National Academy of Sciencesa. 112 (9), 2817-2822 (2015).
- Monteith, A. J., et al. Altered mitochondrial homeostasis during systemic lupus erythematosus impairs neutrophil extracellular trap formation rendering neutrophils ineffective at combating Staphylococcus aureus. Journal of Immunology. 208 (2), 454-463 (2022).
- Monteith, A. J., Miller, J. M., Beavers, W. N., Juttukonda, L. J., Skaar, E. P. Increased dietary manganese impairs neutrophil extracellular trap formation rendering neutrophils ineffective at combating Staphylococcus aureus. Infection and Immunity. 90 (3), 0068521 (2022).
- Monteith, A. J., et al. Mitochondrial calcium uniporter affects neutrophil bactericidal activity during Staphylococcus aureus infection. Infection and Immunity. 90 (2), 0055121 (2022).
- Cao, Z., et al. Roles of mitochondria in neutrophils. Frontiers in Immunology. 13, 934444 (2022).
- Papayannopoulos, V., Metzler, K. D., Hakkim, A., Zychlinsky, A. Neutrophil elastase and myeloperoxidase regulate the formation of neutrophil extracellular traps. The Journal of Cell Biology. 191 (3), 677-691 (2010).
- Fan, Z., Ley, K. Developing neutrophils must eat…themselves. Immunity. 47 (3), 393-395 (2017).
- Riffelmacher, T., et al. Autophagy-dependent generation of free fatty acids is critical for normal neutrophil differentiation. Immunity. 47 (3), 466-480 (2017).
- Amend, S. R., Valkenburg, K. C., Pienta, K. J. Murine hind limb long bone dissection and bone marrow isolation. Journal of Visualized Experiments. (110), e53936 (2016).
- Swamydas, M., Isolation Lionakis, M. S. purification and labeling of mouse bone marrow neutrophils for functional studies and adoptive transfer experiments. Journal of Visualized Experiments. (77), e50586 (2013).
- Gerner, M. C., et al. Packed red blood cells inhibit T-cell activation via ROS-dependent signaling pathways. The Journal of Biological Chemistry. 296, 100487 (2021).
- Zhang, Z. -. W., et al. Red blood cell extrudes nucleus and mitochondria against oxidative stress. IUBMB Life. 63 (7), 560-565 (2011).
- Kuhns, D. B., Priel, D. A. L., Chu, J., Zarember, K. A. Isolation and functional analysis of human neutrophils. Current Protocols in Immunology. 111, 1-16 (2015).
- Hearne, A., Chen, H., Monarchino, A., Wiseman, J. S. Oligomycin-induced proton uncoupling. Toxicology In Vitro. 67, 104907 (2020).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-Protocol. 8 (10), e2850 (2018).
- Nath, S. The molecular mechanism of ATP synthesis by F1F0-ATP synthase: a scrutiny of the major possibilities. Advances in Biochemical Engineering/Biotechnology. 74, 65-98 (2002).
- Heinz, S., et al. Mechanistic investigations of the mitochondrial complex I inhibitor rotenone in the context of pharmacological and safety evaluation. Scientific Reports. 7 (1), 45465 (2017).
- Hytti, M., et al. Antimycin A-induced mitochondrial damage causes human RPE cell death despite activation of autophagy. Oxidative Medicine and Cellular Longevity. 2019, 1583656 (2019).
- Malecki, M., Kamrad, S., Ralser, M., Bähler, J. Mitochondrial respiration is required to provide amino acids during fermentative proliferation of fission yeast. EMBO Reports. 21 (11), e50845 (2020).
- Divakaruni, A. S., Paradyse, A., Ferrick, D. A., Murphy, A. N., Jastroch, M. Analysis and interpretation of microplate-based oxygen consumption and pH data. Methods in Enzymology. 547, 309-354 (2014).
- Marchetti, P., Fovez, Q., Germain, N., Khamari, R., Kluza, J. Mitochondrial spare respiratory capacity: Mechanisms, regulation, and significance in non-transformed and cancer cells. The FASEB Journal. 34 (10), 13106-13124 (2020).
- Nicholas, D., et al. Advances in the quantification of mitochondrial function in primary human immune cells through extracellular flux analysis. PLoS One. 12 (2), e0170975 (2017).
- Tur, J., et al. Mitofusin 2 in macrophages links mitochondrial ROS production, cytokine release, phagocytosis, autophagy, and bactericidal activity. Cell Reports. 32 (8), 108079 (2020).
- Benz, R., McLaughlin, S. The molecular mechanism of action of the proton ionophore FCCP (carbonylcyanide p-trifluoromethoxyphenylhydrazone). Biophysical Journal. 41 (3), 381-398 (1983).
- Wettmarshausen, J., Perocchi, F. Assessing calcium-stimulated mitochondrial bioenergetics using the seahorse XF96 analyzer. Methods in Molecular Biology. 1925, 197-222 (2019).
- Forkink, M., et al. Mitochondrial hyperpolarization during chronic complex I inhibition is sustained by low activity of complex II, III, IV and V. Biochimica et Biophysica Acta. 1837 (8), 1247-1256 (2014).
- . Methods for Reducing Cell Growth Edge Effects in Agilent Seahorse XF Cell Culture Microplates Available from: https://www.agilent.com/cs/library/usermanuals/public/user-manual-methods-for-reducing-cell-growth-edge-effect-cell-analysis-5994-0240en-agilent.pdf (2019)
- Lundholt, B. K., Scudder, K. M., Pagliaro, L. A simple technique for reducing edge effect in cell-based assays. Journal of Biomolecular Screening. 8 (5), 566-570 (2003).
- Wu, D., Yotnda, P. Induction and testing of hypoxia in cell culture. Journal of Visualized Experiments. (54), e2899 (2011).
- Normalisation of Seahorse XFe96 metabolic assaysto cell number with Hoechst stain using well-scan mode on the CLARIOstar Plus. BMG Labtech Available from: https://www.bmglabtech.com/cn/normalisation-of-seahorse-xfe96-metabolic-assays-to-cell-number-with-hoechst-stain/ (2020)
- Yetkin-Arik, B., et al. The role of glycolysis and mitochondrial respiration in the formation and functioning of endothelial tip cells during angiogenesis. Scientific Reports. 9 (1), 12608 (2019).
- Jastroch, M., Divakaruni, A. S., Mookerjee, S., Treberg, J. R., Brand, M. D. Mitochondrial proton and electron leaks. Essays in Biochemistry. 47, 53-67 (2010).
- Jandl, R. C., et al. Termination of the respiratory burst in human neutrophils. The Journal of Clinical Investigation. 61 (5), 1176-1185 (1978).
- Azevedo, E. P., et al. A metabolic shift toward pentose phosphate pathway is necessary for amyloid fibril- and phorbol 12-myristate 13-acetate-induced neutrophil extracellular trap (NET) formation. The Journal of Biological Chemistry. 290 (36), 22174-22183 (2015).
- Six, E., et al. AK2 deficiency compromises the mitochondrial energy metabolism required for differentiation of human neutrophil and lymphoid lineages. Cell Death & Disease. 6 (8), e1856 (2015).
- Kumar, S., Dikshit, M. Metabolic insight of neutrophils in health and disease. Frontiers in Immunology. 10, 2099 (2019).
- Rodríguez-Espinosa, O., Rojas-Espinosa, O., Moreno-Altamirano, M. M. B., López-Villegas, E. O., Sánchez-García, F. J. Metabolic requirements for neutrophil extracellular traps formation. Immunology. 145 (2), 213-224 (2015).
- Invernizzi, F., et al. Microscale oxygraphy reveals OXPHOS impairment in MRC mutant cells. Mitochondrion. 12 (2), 328-335 (2012).
- Zenaro, E., et al. Neutrophils promote Alzheimer's disease-like pathology and cognitive decline via LFA-1 integrin. Nature Medicine. 21 (8), 880-886 (2015).
- Maianski, N. A., et al. Functional characterization of mitochondria in neutrophils: a role restricted to apoptosis. Cell Death and Differentiation. 11 (2), 143-153 (2004).
- Bergman, O., Ben-Shachar, D. Mitochondrial oxidative phosphorylation system (OXPHOS) deficits in schizophrenia. Canadian Journal of Psychiatry. 61 (8), 457-469 (2016).
- Zhou, W., Qu, J., Xie, S., Sun, Y., Yao, H. Mitochondrial dysfunction in chronic respiratory diseases: implications for the pathogenesis and potential therapeutics. Oxidative Medicine and Cellular Longevity. 2021, 5188306 (2021).
- Hirano, M., Emmanuele, V., Quinzii, C. M. Emerging therapies for mitochondrial diseases. Essays in Biochemistry. 62 (3), 467-481 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon