É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Mensuração em Tempo Real do Perfil Bioenergético Mitocondrial de Neutrófilos
Neste Artigo
Resumo
Descrevemos protocolos stepwise medindo a respiração mitocondrial de neutrófilos humanos e camundongos e células HL60 usando o analisador de fluxo extracelular metabólico.
Resumo
Os neutrófilos são a primeira linha de defesa e os leucócitos mais abundantes em humanos. Essas células efetoras desempenham funções como fagocitose e explosão oxidativa, e criam armadilhas extracelulares de neutrófilos (NETs) para depuração microbiana. Novos conhecimentos sobre as atividades metabólicas dos neutrófilos desafiam o conceito inicial de que eles dependem principalmente da glicólise. A medição precisa das atividades metabólicas pode desdobrar diferentes exigências metabólicas dos neutrófilos, incluindo o ciclo do ácido tricarboxílico (TCA) (também conhecido como ciclo de Krebs), fosforilação oxidativa (OXPHOS), via das pentoses fosfato (PPP) e oxidação de ácidos graxos (FAO) sob condições fisiológicas e em estados patológicos. Este artigo descreve um protocolo passo-a-passo e pré-requisitos para medir a taxa de consumo de oxigênio (OCR) como um indicador da respiração mitocondrial em neutrófilos derivados da medula óssea de camundongos, neutrófilos derivados do sangue humano e a linhagem celular HL60 semelhante a neutrófilos, usando análise de fluxo metabólico em um analisador de fluxo extracelular metabólico. Este método pode ser usado para quantificar as funções mitocondriais de neutrófilos em condições normais e de doença.
Introdução
As mitocôndrias desempenham um papel importante na bioenergética celular, que gera trifosfato de adenosina (ATP) por fosforilação oxidativa (OXPHOS). Além disso, o papel das mitocôndrias se estende à geração e desintoxicação de espécies reativas de oxigênio, regulação do cálcio da matriz citoplasmática e mitocondrial, síntese celular, catabolismo e transporte de metabólitos dentro da célula1. A respiração mitocondrial é essencial em todas as células, pois sua disfunção pode resultar em problemas metabólicos 2, incluindo doenças cardiovasculares3 e uma grande variedade de doenças neurodegenerativas, como degeneração macular relacionada à idade4, Parkinson e Alzheimer5 e doença de Charcot-Marie-Tooth2 A (CMT2A)6.
Estudos de microscopia eletrônica em neutrófilos revelaram que há relativamente poucas mitocôndrias7, e elas dependem fortemente da glicólise para sua produção de energia, uma vez que as taxas de respiração mitocondrial são muito baixas8. Entretanto, as mitocôndrias são cruciais para as funções dos neutrófilos, como quimiotaxia9 e apoptose10,11,12. Um estudo anterior revelou uma complexa rede mitocondrial em neutrófilos humanos com alto potencial de membrana. A perda do potencial de membrana mitocondrial é um indicador precoce de apoptose neutrofílica10. O tratamento com o desacoplador mitocondrial carbonil cianeto m-clorofenil hidrazona (CCCP) mostrou inibição significativa da quimiotaxia, juntamente com alteração na morfologia mitocondrial 9,10.
Embora a principal fonte de energia para os neutrófilos seja a glicólise, as mitocôndrias fornecem o ATP que inicia a ativação dos neutrófilos alimentando a primeira fase da sinalização purinérgica, que aumenta a sinalização de Ca2+, amplifica a produção de ATP mitocondrial e inicia as respostas funcionais dos neutrófilos13. A disfunção da cadeia respiratória mitocondrial resulta na produção excessiva de espécies reativas tóxicas de oxigênio (EROs) e leva a danos patogênicos14,15,16. NETosis, que é o processo de formação de armadilhas extracelulares de neutrófilos (NETs), é uma propriedade crítica dos neutrófilos que os ajuda a lutar contra patógenos17 e contribui para muitas condições patológicas, incluindo câncer, trombose e doenças autoimunes18. As ERO derivadas da mitocôndria contribuem para a NETosis19, o DNA mitocondrial pode ser um componente das NETs18 e a homeostase mitocondrial alterada prejudica a NETosis 20,21,22,23,24. Além disso, durante a diferenciação ou maturação normal, a reprogramação metabólica dos neutrófilos é revertida pela limitação da atividade glicolítica, que se engaja na respiração mitocondrial e mobiliza lipídios intracelulares25,26.
O analisador de fluxo extracelular metabólico pode monitorar e quantificar continuamente a respiração mitocondrial de células vivas e a glicólise. O analisador utiliza um cartucho sensor em formato de placa de 96 poços e dois fluoróforos para quantificar a concentração de oxigênio (O2) e as mudanças de pH. O cartucho do sensor está acima da monocamada da célula durante o ensaio e forma uma microcâmara de ~200 nm de altura. Os feixes de fibras ópticas no analisador são usados para excitar os fluoróforos e detectar as mudanças de intensidade fluorescente. Mudanças em tempo real na concentração de O2 e pH são automaticamente calculadas e mostradas como taxa de consumo de oxigênio (OCR) e taxa de acidificação extracelular (ECAR). Há quatro portas no cartucho do sensor que permitem carregar até quatro compostos em cada poço durante as medições de ensaio. Este protocolo concentra-se em quantificar a respiração mitocondrial de neutrófilos humanos e camundongos, bem como as células HL60 semelhantes a neutrófilos, usando o analisador de fluxo extracelular metabólico.
Protocolo
Amostras de sangue total heparinizadas foram obtidas de doadores humanos saudáveis após a obtenção do consentimento informado, conforme aprovado pelo Comitê de Revisão Institucional da UConn Health de acordo com a Declaração de Helsinque. Todos os experimentos com animais seguiram as diretrizes do Comitê Institucional de Cuidados e Uso de Animais da UConn Health (IACUC), e a aprovação para o uso de roedores foi obtida da IACUC da UConn Health de acordo com os critérios descritos no Guide for the Care and Use of Laboratory Animals do National Institutes of Health. Camundongos C57BL/6 machos com 6 semanas de idade foram utilizados neste estudo.
1. Preparo da placa de 96 poços para o ensaio do fluxo metabólico extracelular
- Um cartucho sensor é embalado sobre a placa de 96 poços especialmente projetada para o ensaio de fluxo extracelular metabólico. Hidrate o cartucho levantando-o cuidadosamente e coloque 200 μL/poço de meio de calibração em cada poço da placa subjacente. Coloque o cartucho sobre a placa com calibrador em uma incubadora não-CO2, umidificada, a 37 °C durante a noite para hidratar.
- Com base no tipo de célula, utilizar um revestimento específico para a placa de cultura para garantir a adesão celular. Para neutrófilos humanos - células HL60 indiferenciadas e diferenciadas - revestem a placa de 96 poços com solução salina tamponada com fosfato estéril (PBS) contendo 50 μL de 5 μg/mL de anticorpo anti-humano CD18 purificado de camundongo (Clone TS1/18) a 4 °C durante a noite. Para neutrófilos de camundongo, revestir a placa de 96 poços com 25 μL de 22,4 μg/mL de Cell Tak em pH 8,0 em NaHCO3 0,1 M à temperatura ambiente (TR) por 20 min.
- Lave as placas com 200 μL de PBS estéril duas vezes.
- Adicione meio completo aos poços de canto (A1, A12, H1 e H12) da placa de cultura celular (sem células) para correção de fundo nas placas de cultura de células durante a semeadura.
NOTA: A evaporação durante o revestimento e a calibração podem afetar o volume do meio de calibração e a normalização. Use uma bandeja ou câmara com tecido estéril molhado com água e coloque a placa com o calibrador acima para evitar a evaporação.
2. Preparação e semeadura de células
- Preparo do meio de ensaio
- Preparar o meio de ensaio adicionando 1 mM de piruvato, 10 mM de glicose e 2 mM de glutamina ao meio Eagle modificado (DMEM) de Dulbecco.
- Isolamento de neutrófilos de camundongos da medula óssea
- Isole neutrófilos de camundongos da medula óssea usando o Kit de Enriquecimento de Neutrófilos de Camundongos, de acordo com as instruções do fabricante.
- Anestesiar os camundongos por injeção intraperitoneal (i.p.) de cetamina (125 mg/kg) e xilazina (12,5 mg/kg) e, em seguida, sacrificar os camundongos por deslocamento cervical.
- Colher os fêmures e tíbias, conforme descrito anteriormente27. Resumidamente, corte a pele para expor a perna com os músculos. Corte a articulação do quadril para remover a perna do corpo. Em seguida, remova os músculos para coletar o fêmur e a tíbia.
- Corte as extremidades menores dos fêmures e tíbias. Segurando as extremidades do corte, coloque-as em um tubo centrífugo de 0,5 mL com dois orifícios perfurados com agulha de seringa de 25 G no fundo e coloque o tubo de 0,5 mL em um tubo de centrífuga de 1,5 mL (Figura 1A). Adicionar 50 μL de PBS no tubo de 0,5 mL para evitar o ressecamento das células da medula óssea.
- Centrifugar a 5.900 × g em TR por 15 s para coletar a medula óssea no fundo do tubo de 1,5 mL. Ressuspender as células da medula óssea com 1 mL de DMEM. Adicionar 50 μL de soro de rato do kit, misturar suavemente por pipetagem e transferir a suspensão celular para um tubo de ensaio de cultura de poliestireno de 5 mL.
- Adicionar 50 μL de cocktail de enriquecimento do Kit de Enriquecimento de Neutrófilos de Rato e incubar durante 15 min em RT. Centrifugar a 300 × g em RT durante 5 min.
- Ressuspender as células com 1 mL de DMEM, adicionar 50 μL de coquetel de seleção de biotina do kit, misturar suavemente por pipetagem e incubar por 15 min em TR.
- Adicione 150 μL de partículas magnéticas em vórtice do kit, misture suavemente por pipetagem e incube por 10 min no RT.
- Adicione ~1,3 mL de DMEM, misture suavemente por pipetagem, coloque o tubo no ímã por 3 min e transfira o sobrenadante contendo neutrófilos purificados de camundongo para um novo tubo de ensaio de cultura de poliestireno de 5 mL invertendo o tubo original junto com o ímã.
- Centrifugar a 300 × g em RT por 5 min. Após a remoção do sobrenadante por aspiração a vácuo, ressuspender as células com 1 mL de meio de ensaio.
- Conte as células manualmente usando um hemocitômetro.
- Ajustar a densidade celular para 1,1 × 106 células/mL adicionando meio de ensaio, semente de 180 μL da suspensão de neutrófilos de camundongo (2 × 105 células) por poço na placa preparada de 96 poços (passos 1.2-1.4) e centrifugar a placa a 300 × g em RT por 3 min sem freio para garantir a aderência adequada das células no fundo da placa.
- Incubar a placa em estufa não CO2, umidificada, a 37 °C por 1 h para pré-equilibrar as células com o meio de ensaio.
NOTA: A pureza dos neutrófilos é crítica para o ensaio, uma vez que é um viés potencial. A pureza do isolamento de neutrófilos em camundongos é de 69,9%-88,7% por este protocolo. Existem outros métodos para isolar neutrófilos da medula óssea de camundongos, como a centrifugação por gradiente de densidade28. Existem também kits alternativos de isolamento de neutrófilos de outros fornecedores, baseados na seleção magnética negativa usando os anticorpos monoclonais contra antígenos que não são expressos nos neutrófilos.
- Isolamento de neutrófilos humanos do sangue periférico
- Adicionar 8 ml de solução de polissacarídeo num tubo de centrifugação de 15 ml e, em seguida, colocar mais de 4 ml de sangue periférico sobre a solução de polissacarídeo sem misturar.
- Centrifugar a 550 × g a 20 °C durante 30 min. Faça o rotor desacelerar em 1.
NOTA: O tempo de separação dos neutrófilos pode variar entre os doadores. É um mínimo de 30 min, e um adicional de 10-20 min pode ser necessário se a separação de neutrófilos não for bem-sucedida. - Observe a separação plasma/plaquetas e células mononucleares após a centrifugação, como mostra a Figura 1B. Remova cuidadosamente o líquido amarelo superior na parte superior (plasma e plaquetas) e a banda turva superior (células mononucleares) sem perturbar a banda turva inferior (neutrófilos) usando uma pipeta de 1 mL.
- Coletar a faixa turva inferior e ~3-4 mL do líquido claro abaixo em um novo tubo de centrífuga de 15 mL contendo 10 mL de PBS.
- Centrifugar a 400 × g a 20 °C durante 10 min e remover o sobrenadante por aspiração a vácuo.
- Ressuspender as células com 5 mL de PBS e centrifugar a 300 × g a 20 °C por 5 min.
- Após a remoção do sobrenadante, ressuspender as células com 1 mL de meio de ensaio.
- Conte as células manualmente usando um hemocitômetro.
NOTA: Como os eritrócitos no sangue não possuem mitocôndrias, a contaminação eritrocitária não afeta o teste de estresse mitocondrial e impede a ativação/priming neutrofílico29,30. Os eritrócitos precisam ser lisados durante a contagem celular para obter uma concentração precisa de neutrófilos. Adicionar 10 μL da suspensão celular a 891 μL de água deionizada por 10-30 s para lisar os eritrócitos e, em seguida, adicionar 99 μL de 10x PBS para equilibrar a pressão osmótica, evitando a lise dos neutrófilos. - Ajustar a densidade celular para 2,2 × 106 células/mL adicionando meio de ensaio, semente de 180 μL de neutrófilos humanos (~4 × 105) por poço na placa preparada de 96 poços e centrifugar a placa a 300 × g em RT por 3 min sem freio para garantir a aderência adequada das células no fundo da placa.
- Incubar a placa em estufa umidificada a 37 °C não -CO2 por 1 h para pré-equilibrar as células com o meio de ensaio.
NOTA: A pureza dos neutrófilos é crítica para o ensaio, uma vez que é um viés potencial. A pureza do isolamento de neutrófilos humanos é de 86,6%-96,8%. Existem outros métodos de centrifugação por gradiente de densidade para isolar neutrófilos do sangue humano, incluindo o isolamento de Percoll, bem como uma combinação de isolamento de Ficoll e sedimentação com dextran31.
- Cultura de células HL60 e diferenciação dirigida por neutrófilos
- Manter células HL60 em meio Roswell Park Memorial Institute (RPMI)-1640 contendo 10% de soro fetal bovino (SFB), 100 μg/mL de penicilina, 100 μg/mL de estreptomicina e 250 ng/mL de anfotericina B a 37 °C e 5% de CO2.
- Para diferenciação dirigida por neutrófilos, manter as células HL60 (células em suspensão) em frasco T25 a uma densidade de 1 × 10 5 células/mL em meio RPMI-1640 contendo FBS a 10%, penicilina 100 μg/mL, estreptomicina 100 μg/mL, anfotericina B 250 ng/mL e dimetilsulfóxido (DMSO) a 37 °C e5 % CO2 por 6 dias.
- No dia do ensaio, contar as células manualmente usando o hemocitômetro, centrifugar células HL60 diferenciadas ou indiferenciadas a 300 × g em TR por 5 min, lavar com DMEM uma vez e ressuspender as células com o meio de ensaio para obter uma densidade celular de 1,39 × 106 células/mL.
- Semear 180 μL de células HL60 diferenciadas ou indiferenciadas (~2,5 × 105) por poço na placa preparada de 96 poços e centrifugar a placa a 300 × g em RT por 3 min sem freio para garantir a aderência adequada das células no fundo da placa.
- Incubar a placa em estufa não CO2, umidificada, a 37 °C por 1 h para pré-equilibrar as células com o meio de ensaio.
NOTA: Confirme a adesão completa das células usando um microscópio antes da próxima etapa.
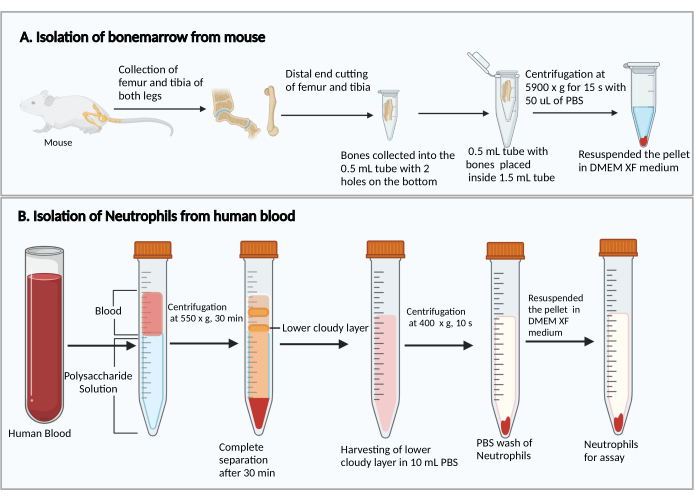
Figura 1: Diagrama esquemático do isolamento das células da medula óssea e neutrófilos. (A) Coleta de células da medula óssea de um camundongo e (B) isolamento de neutrófilos do sangue humano. Clique aqui para ver uma versão maior desta figura.
| Tipo de célula | Células por poço | Compostos/Reagentes | Concentração da solução de trabalho | Volume de injeção nos portos | Concentração final em poços |
| Neutrófilos de camundongo | 2 × 105 | Oligomicina | 25 μM | 20 μL | 2,5 μM |
| FCCP | 7,5 μM | 17,6 μL | 0,61 μM | ||
| Rotenone Antimicina A mistura | 10 μM | 24 μL | 1 μM | ||
| Neutrófilos humanos | 4 × 105 | Oligomicina | 10 μM | 20 μL | 1 μM |
| FCCP | 12,5 μM | 22 μL | 1,25 μM | ||
| Rotenone Antimicina A mistura | 10 μM | 24 μL | 1 μM | ||
| Células HL60 indiferenciadas ou diferenciadas | 2.5 × 105 | Oligomicina | 25 μM | 20 μL | 2,5 μM |
| FCCP | 15 μM | 22 μL | 1,5 μM | ||
| Rotenone Antimicina A mistura | 10 μM | 24 μL | 1 μM |
Tabela 1: Número de células e concentrações de reagentes para o teste de estresse mitocondrial.
3. Preparação de compostos no kit de teste de estresse mitocondrial
- Abra o kit de teste de estresse mitocondrial e prepare os reagentes.
NOTA: As diferentes concentrações da solução de trabalho, o volume de injeção nos poços e as concentrações finais nos poços de oligomicina, carbonilcianeto p-trifluorometoxifenilhidrazona (FCCP) e mistura de rotenona/antimicina A usada para neutrófilos de camundongo, neutrófilos humanos e células HL60 são mostrados na Tabela 1.- Preparar a solução-mãe de oligomicina reconstituindo a oligomicina com 630 μL de meio de ensaio para obter uma solução-mãe de 100 μM.
NOTA: Recomenda-se usar a solução estoque no mesmo dia.- Preparar solução de trabalho de oligomicina para ensaios de neutrófilos de camundongo e células HL60 misturando 630 μL de solução-mãe com 1.890 μL de meio de ensaio para obter uma solução de trabalho de 25 μM.
- Preparar a solução de trabalho de oligomicina para o ensaio de neutrófilos humanos misturando 300 μL de solução-mãe com 2.700 μL de meio de ensaio para obter uma solução de trabalho de 10 μM.
NOTA: A ligação da oligomicina à subunidade da placa de base Fo da ATPase Fo/F1 (ATP sintase) impede a reentrada de prótons nas mitocôndrias e inibe a síntese de ATP32. Isso reduz significativamente o fluxo de elétrons através da cadeia de transporte de elétrons e do OCR. No entanto, o fluxo de elétrons não cessa completamente devido ao vazamento de prótons através da membrana mitocondrial interna33.
- Preparar a solução-mãe FCCP reconstituindo FCCP com 720 μL de meio de ensaio para obter uma solução-mãe de 100 μM.
- Preparar a solução de trabalho para o ensaio de neutrófilos no ratinho misturando 300 μL de solução-mãe com 3.700 μL de meio de ensaio para obter uma solução de trabalho de 7,5 μM.
- Preparar a solução de trabalho para o ensaio de neutrófilos humanos misturando 375 μL de solução-mãe com 2,625 μL de meio de ensaio para obter uma solução de trabalho de 12,5 μM.
- Preparar a solução de trabalho para o ensaio da célula HL60 misturando 720 μL de solução-mãe com 4.080 μL de meio de ensaio para obter uma solução de trabalho de 15 μM.
NOTA: A adição de FCCP revela a capacidade máxima das mitocôndrias para usar OXPHOS. É um ácido fraco lipossolúvel permeável às mitocôndrias resulta na dissipação do potencial transmembrana. A descarga do gradiente de prótons através da membrana interna mitocondrial e o desvio do fluxo de prótons da Fo/F1 ATP sintase resultam em desacoplamento mitocondrial. Esse efeito de desacoplamento aumenta abruptamente o consumo de oxigênio mitocondrial para preservar o gradiente de prótons34.
- Preparar a solução-mãe de rotenona/antimicina A, reconstituindo a mistura de rotenona/antimicina A com 540 μL de meio de ensaio para obter uma solução-mãe de 50 μM.
- Preparar a rotenona/antimicina Uma solução de trabalho para todos os ensaios, misturando 540 μL de solução-mãe com 2.160 μL de meio de ensaio para obter uma solução de trabalho de 10 μM.
NOTA: A rotenona bloqueia o complexo I inibindo a transferência de elétrons dos centros ferro-enxofre do complexo I para a ubiquinona, enquanto a antimicina A bloqueia o complexo III da cadeia de transporte de elétrons, levando a um bloqueio de OXPHOS com uma síntese limitada de ATP35. Desse modo, isso revela respiração não mitocondrial36,37.
- Preparar a rotenona/antimicina Uma solução de trabalho para todos os ensaios, misturando 540 μL de solução-mãe com 2.160 μL de meio de ensaio para obter uma solução de trabalho de 10 μM.
- Preparar a solução-mãe de oligomicina reconstituindo a oligomicina com 630 μL de meio de ensaio para obter uma solução-mãe de 100 μM.
- Carregue o cartucho com reagentes; preparar a mistura de oligomicina, FCCP e rotenona/antimicina A nas portas A, B e C, respectivamente, no cartucho para preparações injetáveis (Figura 2). Use a inserção do modelo disponível junto com o cartucho para facilitar o carregamento de reagentes para as portas. Encher as portas não utilizadas com 20 μL de meio de ensaio.
- Para o ensaio de neutrófilos em camundongos, carregar 20 μL de oligomicina (25 μM; cada poço da placa de 96 poços tem 180 μL de meio de ensaio no início, portanto, a concentração final é de 2,5 μM) nas portas A. Carregar 17,6 μL de FCCP (7,5 μM; concentração final: ~0,61 μM) nas portas B. Carregar 24 μL da mistura rotenona/antimicina A (10 μM; concentração final: ~1 μM) nas portas C.
- Para o ensaio de neutrófilos humanos, carregar 20 μL de oligomicina (10 μM; cada poço da placa de 96 poços tem 180 μL de meio de ensaio no início, portanto, a concentração final é de 1 μM) nos portos A. Carregar 22 μL de FCCP (12,5 μM; concentração final: ~1,25 μM) nas portas B. Carregar 24 μL da mistura rotenona/antimicina A (10 μM; concentração final: ~1 μM) nas portas C.
- Para o ensaio celular HL60 e dHL60, carregar 20 μL de oligomicina (25 μM; cada poço da placa de 96 poços tem 180 μL de meio de ensaio no início, portanto, a concentração final é de 2,5 μM) nos portos A. Carregar 22 μL de FCCP (15 μM; concentração final: ~1,5 μM) nas portas B. Carregar 24 μL da mistura rotenona/antimicina A (10 μM; concentração final: ~1 μM) nas portas C.
NOTA: Pipetar a solução do medicamento no porto sem tocar no fundo do porto. Não bata na placa após o carregamento para evitar vazamentos, pois os líquidos são retidos pelas forças capilares. Os poços de fundo da placa de cultura e as portas do cartucho do sensor são carregados com meio de ensaio ou com a mesma porta de carregamento de reagentes como nos poços de amostra para normalizar o efeito dos reagentes sobre os valores de fundo. Os reagentes devem ser adicionados às respectivas portas sem levantar o cartucho da placa de serviço público que contém o calibrador, a fim de evitar qualquer armadilha de ar. Preencha a última porta de todos os poços com o meio como mostrado na Figura 2.

Figura 2: O cartucho de ensaio de estresse mitocondrial e suas portas de injeção. A imagem mostra o cartucho do ensaio de estresse mitocondrial e uma imagem ampliada mostrando o carregamento de drogas/meio individuais para os portos. Abreviatura: FCCP = carbonilcianeto p-trifluorometoxifenilhidrazona. Clique aqui para ver uma versão maior desta figura.
4. Execução do ensaio de estresse mitocondrial
- Ligue o analisador metabólico e o computador, abra o software Wave e clique em Aquecedor ligado para configurar a máquina a 37 °C com pelo menos 5 horas de antecedência. Depois de atingir a temperatura, o canto inferior esquerdo do software de onda mostra-se pronto.
- Abra um modelo para o kit de ensaio de estresse mitocondrial no software. Clique em Definição de Grupo na barra de menu superior.
- Pré-configurar separadamente cada definição no lado esquerdo, como estratégias de injeção, pré-tratamentos, meios de ensaio e tipos celulares (biomaterial usado).
- Clique em estratégias de injeção no lado esquerdo, em seguida, clique em Adicionar e nomeie a condição de injeção como neutrófilos humanos/neutrófilos de camundongo/HL60. Selecione a porta A-D clicando em cada uma e clique em Adicionar composto e digite Oligomicina/FCCP/Rotenone Antimicina A com as respectivas concentrações (Tabela 1).
- Clique em pré-tratamento no lado esquerdo, em seguida, clique em Adicionar e nomeá-los como CD18 e Cell Tak separadamente. Clique no biomaterial usado no lado esquerdo, depois clique em Adicionar e nomeie-os como Neutrófilos humanos, neutrófilos de camundongo e HL60 /dHL60 com densidade de semeadura.
- Defina os grupos clicando em Adicionar grupo (por exemplo, para o ensaio de neutrófilos humanos) e clicando na definição subjacente (por exemplo, Estratégias de injeção conforme Tabela 1 – Neutrófilos humanos, Pré-tratamento como CD18 e tipo de célula como neutrófilos humanos).
- Clique em Mapa de Placas no menu superior para observar todos os grupos, com definições no lado esquerdo e o mapa de placas à direita. Arraste e solte para adicionar cada poço ao grupo, mantendo os quatro poços de canto como Plano de fundo (padrão).
- Configure o protocolo clicando em Protocolo no menu superior e defina três ciclos de mistura de linha de base, oligomicina, FCCP, rotenona e antimicina A, definindo o tempo como Mix como 3 min, Rest como 0 min e Measurement como 7 min.
- Vá para a página Executar ensaio , forneça as informações de resumo do projeto para referência e clique em Iniciar execução.
- Forneça o local para salvar os arquivos, para que todos os resultados sejam salvos após a conclusão do ensaio.
- Após o carregamento automático do ensaio, aguarde a abertura da bandeja para colocar o cartucho e a placa do sensor com 200 μL de calibrador (passo 1.1). Defina a direção do código de barras do cartucho voltada para a direita. Execute a calibração clicando em Estou pronto, o que leva aproximadamente 20 min.
- Após a calibração, clique em Abrir bandeja. Substitua a placa pela placa semeada em célula e clique em Placa de célula de carga para continuar o ensaio.
- Após a conclusão do ensaio, os dados são salvos automaticamente. Clique em Exibir resultados e exporte-os como uma planilha ou outro arquivo de software de análise.
- Gráfico e análise dos dados (Figura 3 e Figura 4).
- Calcular parâmetros respiratórios, incluindo respiração mitocondrial basal, respiração ligada a vazamento de prótons, respiração ligada a ATP, respiração máxima, capacidade respiratória ociosa e respiração não mitocondrial (Figura 4A)38,39,40,41.
- Calcular a respiração mitocondrial basal subtraindo o valor de OCR medido após a adição de mistura de rotenona/antimicina A do OCR antes de injetar oligomicina (Figura 4A, a).
- Calcular a respiração ligada ao vazamento de prótons subtraindo o valor de OCR após injeção de mistura de rotenona/antimicina A do valor de OCR medido após injeção de oligomicina (Figura 4A,b).
- Estimar a respiração ligada ao ATP calculando a diferença entre a respiração mitocondrial basal e a respiração ligada ao extravasamento de prótons. Subtrair o primeiro valor de OCR medido após a injeção de oligomicina do primeiro valor de OCR antes da injeção de oligomicina (Figura 4A,c).
- A respiração máxima é a taxa respiratória máxima que uma célula pode atingir após a adição do FCCP. Calcule isso subtraindo o valor de OCR após a injeção da mistura de rotenona/antimicina A do valor de OCR medido após a injeção de FCCP (Figura 4A,d).
- A capacidade respiratória ociosa refere-se à capacidade de uma célula de atender à maior demanda de energia através do OXPHOS. Calcule isso encontrando a diferença entre a respiração máxima e a respiração mitocondrial basal (Figura 4A, e).
- A respiração não mitocondrial é a quantidade de oxigênio consumida por fontes não mitocondriais. Medir isso após a adição da mistura de rotenona/antimicina A (Figura 4A,f).
- Realizar análise estatística utilizando o teste t de Student para comparar diferentes parâmetros respiratórios de células HL60 indiferenciadas e diferenciadas. Considere-se que p < 0,05 é estatisticamente significante.
NOTA: Réplicas com valores de OCR ou ECAR abaixo de zero são consideradas um erro na preparação da amostra, injeção de composto ou medição. Eles são excluídos de análises futuras.
Resultados
Dinâmica representativa de OCR é mostrada indicando as alterações da respiração mitocondrial em resposta à oligomicina, FCCP e mistura de rotenona/antimicina A de neutrófilos de camundongos (Figura 3A), neutrófilos humanos (Figura 3B) e células HL60 indiferenciadas e diferenciadas (Figura 3C). Em todas as células, o tratamento com oligomicina diminui o valor de OCR inibindo o canal de prótons da ATP sintase; O tratamento...
Discussão
O procedimento padrão que mede a respiração mitocondrial de neutrófilos usando o analisador de fluxo extracelular metabólico é limitado por muitos fatores, incluindo número celular, crescimento celular e viabilidade. Cada concentração de composto varia entre o tipo e a fonte de células neste ensaio. Oligomicina e rotenona/antimicina A são usadas principalmente em uma concentração semelhante entre a maioria dos tipos celulares. No entanto, como a frequência respiratória máxima induzida pelo FCCP varia ent...
Divulgações
Os autores declaram não haver interesse financeiro concorrente.
Agradecimentos
Agradecemos ao Dr. Anthony T. Vella e à Dra. Federica Aglianoin do Departamento de Imunologia da UConn Health por seu treinamento no uso do analisador de fluxo extracelular metabólico, e à Dra. Lynn Puddington no Departamento de Imunologia da UConn Health por seu apoio aos instrumentos. Agradecemos à Dra. Geneva Hargis, da UConn School of Medicine, por sua ajuda na redação científica e edição deste manuscrito. Esta pesquisa foi apoiada por subsídios dos Institutos Nacionais de Saúde, Instituto Nacional do Coração, Pulmão e Sangue (R01HL145454), Instituto Nacional de Ciências Médicas Gerais (R35GM147713 e P20GM139763), um fundo de inicialização da UConn Health e uma bolsa de reentrada na carreira da Associação Americana de Imunologistas.
Materiais
| Name | Company | Catalog Number | Comments |
| 37 °C non-CO2 incubator | Precision | Economy Model 2EG | Instrument |
| Biorender | Software Application | ||
| Centrifuge | Eppendorf | Model 5810R | Instrument |
| Corning Cell-Tak Cell and Tissue Adhesive | Corning | 102416-100 | Reagent |
| EasySep Magnet | STEMCELL | 18000 | Magnet |
| EasySepMouse Neutrophil Enrichment kit | STEMCELL | 19762A | Reagents |
| Graphpad Prism 9 | Software Application | ||
| Human Serum Albumin Solution (25%) | GeminiBio | 800-120 | Reagents |
| Ketamine (VetaKet) | DAILYMED | NDC 59399-114-10 | Anesthetic |
| PBS | Cytiva | SH30256.01 | Reagents |
| Plate buckets | Eppendorf | UL155 | Accessory |
| PolymorphPrep | PROGEN | 1895 (previous 1114683) | polysaccharide solution |
| Purified mouse anti-human CD18 antibody | Biolegend | 302102 | Clone TS1/18 |
| RPMI 1640 Medium | Gibco | 11-875-093 | Reagents |
| Seahorse metabolic extracellular flux analyzer | Agilent | XFe96 | Instrument |
| Seahorse XF Cell Mito Stress Test Kit | Agilent | 103015-100 | mitochondrial stress test Kit |
| Swing-bucket rotor | Eppendorf | A-4-62 | Rotor |
| Vactrap 2 Vacum Trap | Fox Lifesciences | 3052101-FLS | Instrument |
| Wave | Software Application | ||
| XF 1.0 M Glucose Solution | Agilent | 103577-100 | Reagent |
| XF 100 mM Pyruvate Solution | Agilent | 103578-100 | Reagent |
| XF 200 mM Glutamine Solution | Agilent | 103579-100 | Reagent |
| XF DMEM medium | Agilent | 103575-100 | Reagent |
| XFe96 FluxPak | Agilent | 102601-100 | Material |
| Xylazine (AnaSed Injection) | DAILYMED | NDC 59399-110-20 | Anesthetic |
Referências
- Demine, S., Renard, P., Arnould, T. Mitochondrial uncoupling: a key controller of biological processes in physiology and diseases. Cells. 8 (8), 795 (2019).
- Noguchi, M., Kasahara, A. Mitochondrial dynamics coordinate cell differentiation. Biochemical and Biophysical Research Communications. 500 (1), 59-64 (2018).
- Zhu, L., et al. Correlation between mitochondrial dysfunction, cardiovascular diseases, and traditional Chinese medicine. Evidence-Based Complementary and Alternative Medicine. 2020, e2902136 (2020).
- Kaarniranta, K., et al. Mechanisms of mitochondrial dysfunction and their impact on age-related macular degeneration. Progress in Retinal and Eye Research. 79, 100858 (2020).
- Onyango, I. G., Khan, S. M., Bennett, J. P. Mitochondria in the pathophysiology of Alzheimer's and Parkinson's diseases. Frontiers in Bioscience. 22 (5), 854-872 (2017).
- Loiseau, D., et al. Mitochondrial coupling defect in Charcot-Marie-Tooth type 2A disease. Annals of Neurology. 61 (4), 315-323 (2007).
- Zucker-Franklin, D. Electron microscopic studies of human granulocytes: structural variations related to function. Seminars in Hematology. 5 (2), 109-133 (1968).
- Karnovsky, M. L. The metabolism of leukocytes. Seminars in Hematology. 5 (2), 156-165 (1968).
- Bao, Y., et al. mTOR and differential activation of mitochondria orchestrate neutrophil chemotaxis. The Journal of Cell Biology. 210 (7), 1153-1164 (2015).
- Fossati, G., et al. The mitochondrial network of human neutrophils: role in chemotaxis, phagocytosis, respiratory burst activation, and commitment to apoptosis. Journal of Immunology. 170 (4), 1964-1972 (2003).
- Pryde, J. G., Walker, A., Rossi, A. G., Hannah, S., Haslett, C. Temperature-dependent arrest of neutrophil apoptosis. Failure of Bax insertion into mitochondria at 15 degrees C prevents the release of cytochrome c. The Journal of Biological Chemistry. 275 (43), 33574-33584 (2000).
- Maianski, N. A., Mul, F. P. J., van Buul, J. D., Roos, D., Kuijpers, T. W. Granulocyte colony-stimulating factor inhibits the mitochondria-dependent activation of caspase-3 in neutrophils. Blood. 99 (2), 672-679 (2002).
- Bao, Y., et al. Mitochondria regulate neutrophil activation by generating ATP for autocrine purinergic signaling. The Journal of Biological Chemistry. 289 (39), 26794-26803 (2014).
- Chouchani, E. T., et al. Ischaemic accumulation of succinate controls reperfusion injury through mitochondrial ROS. Nature. 515 (7527), 431-435 (2014).
- Hayashi, G., Cortopassi, G. Oxidative stress in inherited mitochondrial diseases. Free Radical Biology and Medicine. 88, 10-17 (2015).
- Mailloux, R. J. An update on mitochondrial reactive oxygen species production. Antioxidants. 9 (6), 472 (2020).
- Abuaita, B. H., et al. The IRE1α stress signaling axis is a key regulator of neutrophil antimicrobial effector function. Journal of Immunology. 207 (1), 210-220 (2021).
- Lood, C., et al. Neutrophil extracellular traps enriched in oxidized mitochondrial DNA are interferogenic and contribute to lupus-like disease. Nature Medicine. 22 (2), 146-153 (2016).
- Douda, D. N., Khan, M. A., Grasemann, H., Palaniyar, N. SK3 channel and mitochondrial ROS mediate NADPH oxidase-independent NETosis induced by calcium influx. Proceedings of the National Academy of Sciencesa. 112 (9), 2817-2822 (2015).
- Monteith, A. J., et al. Altered mitochondrial homeostasis during systemic lupus erythematosus impairs neutrophil extracellular trap formation rendering neutrophils ineffective at combating Staphylococcus aureus. Journal of Immunology. 208 (2), 454-463 (2022).
- Monteith, A. J., Miller, J. M., Beavers, W. N., Juttukonda, L. J., Skaar, E. P. Increased dietary manganese impairs neutrophil extracellular trap formation rendering neutrophils ineffective at combating Staphylococcus aureus. Infection and Immunity. 90 (3), 0068521 (2022).
- Monteith, A. J., et al. Mitochondrial calcium uniporter affects neutrophil bactericidal activity during Staphylococcus aureus infection. Infection and Immunity. 90 (2), 0055121 (2022).
- Cao, Z., et al. Roles of mitochondria in neutrophils. Frontiers in Immunology. 13, 934444 (2022).
- Papayannopoulos, V., Metzler, K. D., Hakkim, A., Zychlinsky, A. Neutrophil elastase and myeloperoxidase regulate the formation of neutrophil extracellular traps. The Journal of Cell Biology. 191 (3), 677-691 (2010).
- Fan, Z., Ley, K. Developing neutrophils must eat…themselves. Immunity. 47 (3), 393-395 (2017).
- Riffelmacher, T., et al. Autophagy-dependent generation of free fatty acids is critical for normal neutrophil differentiation. Immunity. 47 (3), 466-480 (2017).
- Amend, S. R., Valkenburg, K. C., Pienta, K. J. Murine hind limb long bone dissection and bone marrow isolation. Journal of Visualized Experiments. (110), e53936 (2016).
- Swamydas, M., Isolation Lionakis, M. S. purification and labeling of mouse bone marrow neutrophils for functional studies and adoptive transfer experiments. Journal of Visualized Experiments. (77), e50586 (2013).
- Gerner, M. C., et al. Packed red blood cells inhibit T-cell activation via ROS-dependent signaling pathways. The Journal of Biological Chemistry. 296, 100487 (2021).
- Zhang, Z. -. W., et al. Red blood cell extrudes nucleus and mitochondria against oxidative stress. IUBMB Life. 63 (7), 560-565 (2011).
- Kuhns, D. B., Priel, D. A. L., Chu, J., Zarember, K. A. Isolation and functional analysis of human neutrophils. Current Protocols in Immunology. 111, 1-16 (2015).
- Hearne, A., Chen, H., Monarchino, A., Wiseman, J. S. Oligomycin-induced proton uncoupling. Toxicology In Vitro. 67, 104907 (2020).
- Plitzko, B., Loesgen, S. Measurement of oxygen consumption rate (OCR) and extracellular acidification rate (ECAR) in culture cells for assessment of the energy metabolism. Bio-Protocol. 8 (10), e2850 (2018).
- Nath, S. The molecular mechanism of ATP synthesis by F1F0-ATP synthase: a scrutiny of the major possibilities. Advances in Biochemical Engineering/Biotechnology. 74, 65-98 (2002).
- Heinz, S., et al. Mechanistic investigations of the mitochondrial complex I inhibitor rotenone in the context of pharmacological and safety evaluation. Scientific Reports. 7 (1), 45465 (2017).
- Hytti, M., et al. Antimycin A-induced mitochondrial damage causes human RPE cell death despite activation of autophagy. Oxidative Medicine and Cellular Longevity. 2019, 1583656 (2019).
- Malecki, M., Kamrad, S., Ralser, M., Bähler, J. Mitochondrial respiration is required to provide amino acids during fermentative proliferation of fission yeast. EMBO Reports. 21 (11), e50845 (2020).
- Divakaruni, A. S., Paradyse, A., Ferrick, D. A., Murphy, A. N., Jastroch, M. Analysis and interpretation of microplate-based oxygen consumption and pH data. Methods in Enzymology. 547, 309-354 (2014).
- Marchetti, P., Fovez, Q., Germain, N., Khamari, R., Kluza, J. Mitochondrial spare respiratory capacity: Mechanisms, regulation, and significance in non-transformed and cancer cells. The FASEB Journal. 34 (10), 13106-13124 (2020).
- Nicholas, D., et al. Advances in the quantification of mitochondrial function in primary human immune cells through extracellular flux analysis. PLoS One. 12 (2), e0170975 (2017).
- Tur, J., et al. Mitofusin 2 in macrophages links mitochondrial ROS production, cytokine release, phagocytosis, autophagy, and bactericidal activity. Cell Reports. 32 (8), 108079 (2020).
- Benz, R., McLaughlin, S. The molecular mechanism of action of the proton ionophore FCCP (carbonylcyanide p-trifluoromethoxyphenylhydrazone). Biophysical Journal. 41 (3), 381-398 (1983).
- Wettmarshausen, J., Perocchi, F. Assessing calcium-stimulated mitochondrial bioenergetics using the seahorse XF96 analyzer. Methods in Molecular Biology. 1925, 197-222 (2019).
- Forkink, M., et al. Mitochondrial hyperpolarization during chronic complex I inhibition is sustained by low activity of complex II, III, IV and V. Biochimica et Biophysica Acta. 1837 (8), 1247-1256 (2014).
- . Methods for Reducing Cell Growth Edge Effects in Agilent Seahorse XF Cell Culture Microplates Available from: https://www.agilent.com/cs/library/usermanuals/public/user-manual-methods-for-reducing-cell-growth-edge-effect-cell-analysis-5994-0240en-agilent.pdf (2019)
- Lundholt, B. K., Scudder, K. M., Pagliaro, L. A simple technique for reducing edge effect in cell-based assays. Journal of Biomolecular Screening. 8 (5), 566-570 (2003).
- Wu, D., Yotnda, P. Induction and testing of hypoxia in cell culture. Journal of Visualized Experiments. (54), e2899 (2011).
- Normalisation of Seahorse XFe96 metabolic assaysto cell number with Hoechst stain using well-scan mode on the CLARIOstar Plus. BMG Labtech Available from: https://www.bmglabtech.com/cn/normalisation-of-seahorse-xfe96-metabolic-assays-to-cell-number-with-hoechst-stain/ (2020)
- Yetkin-Arik, B., et al. The role of glycolysis and mitochondrial respiration in the formation and functioning of endothelial tip cells during angiogenesis. Scientific Reports. 9 (1), 12608 (2019).
- Jastroch, M., Divakaruni, A. S., Mookerjee, S., Treberg, J. R., Brand, M. D. Mitochondrial proton and electron leaks. Essays in Biochemistry. 47, 53-67 (2010).
- Jandl, R. C., et al. Termination of the respiratory burst in human neutrophils. The Journal of Clinical Investigation. 61 (5), 1176-1185 (1978).
- Azevedo, E. P., et al. A metabolic shift toward pentose phosphate pathway is necessary for amyloid fibril- and phorbol 12-myristate 13-acetate-induced neutrophil extracellular trap (NET) formation. The Journal of Biological Chemistry. 290 (36), 22174-22183 (2015).
- Six, E., et al. AK2 deficiency compromises the mitochondrial energy metabolism required for differentiation of human neutrophil and lymphoid lineages. Cell Death & Disease. 6 (8), e1856 (2015).
- Kumar, S., Dikshit, M. Metabolic insight of neutrophils in health and disease. Frontiers in Immunology. 10, 2099 (2019).
- Rodríguez-Espinosa, O., Rojas-Espinosa, O., Moreno-Altamirano, M. M. B., López-Villegas, E. O., Sánchez-García, F. J. Metabolic requirements for neutrophil extracellular traps formation. Immunology. 145 (2), 213-224 (2015).
- Invernizzi, F., et al. Microscale oxygraphy reveals OXPHOS impairment in MRC mutant cells. Mitochondrion. 12 (2), 328-335 (2012).
- Zenaro, E., et al. Neutrophils promote Alzheimer's disease-like pathology and cognitive decline via LFA-1 integrin. Nature Medicine. 21 (8), 880-886 (2015).
- Maianski, N. A., et al. Functional characterization of mitochondria in neutrophils: a role restricted to apoptosis. Cell Death and Differentiation. 11 (2), 143-153 (2004).
- Bergman, O., Ben-Shachar, D. Mitochondrial oxidative phosphorylation system (OXPHOS) deficits in schizophrenia. Canadian Journal of Psychiatry. 61 (8), 457-469 (2016).
- Zhou, W., Qu, J., Xie, S., Sun, Y., Yao, H. Mitochondrial dysfunction in chronic respiratory diseases: implications for the pathogenesis and potential therapeutics. Oxidative Medicine and Cellular Longevity. 2021, 5188306 (2021).
- Hirano, M., Emmanuele, V., Quinzii, C. M. Emerging therapies for mitochondrial diseases. Essays in Biochemistry. 62 (3), 467-481 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados