Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Mejora de la enumeración de esporas de Bacillus subtilis y el análisis de etiquetas en citometría de flujo
En este artículo
Resumen
Este protocolo se centra en el uso de citometría de flujo y perlas de recuento para cuantificar las esporas bacterianas marcadas con bromuro de etidio. El método también es eficiente para analizar el acoplamiento covalente de proteínas en la superficie de esporas intactas.
Resumen
Las esporas de Bacillus subtilis ya han sido propuestas para diferentes aplicaciones biotecnológicas e inmunológicas; Sin embargo, cada vez es más necesario desarrollar metodologías que mejoren la detección de antígenos inmovilizados en la superficie de las esporas junto con su cuantificación. Los análisis basados en citometría de flujo se han propuesto previamente como enfoques rápidos, fiables y específicos para detectar células marcadas de B. subtilis. En este trabajo, proponemos el uso de la citometría de flujo para evaluar la eficiencia de visualización de un anticuerpo fluorescente (AF) en la superficie de la espora y cuantificar el número de esporas mediante perlas de recuento.
Para ello, utilizamos bromuro de etidio como marcador de ADN y un anticuerpo marcado con aloficocianina (APC), que se acopló a las esporas, como marcador de superficie. La cuantificación de las esporas se realizó mediante perlas de conteo, ya que esta técnica demuestra una alta precisión en la detección de células. Las esporas marcadas se analizaron con un citómetro de flujo, lo que confirmó el acoplamiento. Como resultado, se demostró que el marcaje de ADN mejoró la precisión de la cuantificación por citometría de flujo, para la detección de esporas germinadas. Se observó que el bromuro de etidio no era capaz de marcar las esporas latentes; Sin embargo, esta técnica proporciona una determinación más precisa del número de esporas con proteína fluorescente acoplada a su superficie, lo que ayuda en el desarrollo de estudios que se centran en el uso de esporas como plataforma biotecnológica en diferentes aplicaciones.
Introducción
Bacillus subtilis es una bacteria grampositiva en forma de bastoncillo que es capaz de producir esporas quiescentes cuando las condiciones ambientales no permiten el crecimiento celular1. Las esporas son formas celulares extremadamente estables y las de varias especies, incluida B. subtilis, se utilizan ampliamente como probióticos para uso humano y animal2. Debido a sus propiedades de resistencia y seguridad, la espora de B. subtilis, que presenta proteínas heterólogas, ha sido propuesta como adyuvante de la mucosa, sistema de administración de vacunas y plataforma de inmovilización enzimática 3,4.
Para obtener esporas de B. subtilis, es necesario exponerlo a la privación de nutrientes utilizando un medio de cultivo especial. Después de obtener y purificar estas esporas, se deben cuantificar para mejorar la eficiencia de la prueba 5,6. Así, se aplican ciertos métodos para analizar la concentración de las esporas obtenidas. Se puede utilizar el conteo de placas y una cámara de Petroff-Hausser, también conocida como cámara de conteo. Este último se desarrolló originalmente para determinar la concentración de células sanguíneas; sin embargo, es posible utilizarlo en el campo de la microbiología para el recuento de esporas 7,8. A pesar de ser el método estándar utilizado para el conteo de células, la lectura es laboriosa ya que este método es completamente manual y su precisión depende de la experiencia del operador.
Los análisis basados en citometría de flujo (FC) se han propuesto previamente como enfoques rápidos, confiables y específicos para detectar células marcadas de Bacillus spp. El uso de perlas de recuento por citometría de flujo ha garantizado la reproducibilidad en el recuento celular, en los exámenes rutinarios (recuento absoluto de linfocitos T CD4 y CD8) y en el desarrollo de investigaciones con partículas capaces de ser detectadas y contadas mediante citometría de flujo9. Godjafrey y Alsharif sugirieron el uso de perlas de conteo para la cuantificación de FC de esporas no marcadas10. Se describió el uso de citometría de flujo para el monitoreo de la esporulación en Bacillus spp. a través del marcaje del ADN de las esporas 10,11,12,13. Otro estudio utilizó FC para evaluar la cantidad de proteínas marcadas con fluorescencia en la superficie de las esporas15.
Este estudio buscó utilizar perlas de conteo comerciales para asegurar un estándar de reproducibilidad con respecto al conteo de eventos mediante citometría de flujo. En este artículo, sugerimos el uso de perlas de recuento para el recuento de células en FC para refinar la enumeración de esporas y evaluar la eficiencia de acoplamiento de anticuerpos marcados con fluorescencia en la superficie de las esporas.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Consulte la Tabla de materiales para obtener detalles relacionados con todos los materiales, instrumentos y software utilizados en este protocolo.
1. Configuración de citometría de flujo
- Alineación de los parámetros ópticos de un citómetro de flujo acoplado a un ordenador
- Inicie sesión en el software Cytometer.
- En el espacio de trabajo de software, seleccione Citómetro | Inicie y espere unos minutos | Modo limpio | Siéntese líquidos.
NOTA: Las burbujas de aire y las obstrucciones se eliminaron durante el proceso de iniciación del citómetro, antes de la adquisición de la muestra.
- Control de calidad
- Utilice el reactivo de control de calidad para ajustar los voltajes de los tubos fotomultiplicadores y evaluar su sensibilidad.
- Prepare el reactivo de control de calidad en un tubo de poliestireno.
- Para definir la línea de base, prepare las perlas de suspensión agregando 0,5 mL de diluyente (10 mM de PBS filtrado, pH 7) y 3 gotas de perlas al tubo marcado.
- Mezcle el vial de perlas invirtiendo suavemente.
- En el espacio de trabajo de software, seleccione Citómetro | CST para conectarse a la interfaz CST. Espere unos minutos y observe el mensaje del citómetro desconectado.
- Conecte el tubo de poliestireno que contiene el reactivo de control de calidad a la sonda del citómetro de flujo.
- Utilice el reactivo de control de calidad para ajustar los voltajes de los tubos fotomultiplicadores y evaluar su sensibilidad.
- Verificación de la configuración del citómetro
- En la ventana Resumen del sistema , compruebe que la configuración del citómetro es adecuada para el experimento.
- Seleccione el ID de lote de cuentas de configuración correspondiente para verificar que el ID de lote de cuentas de configuración seleccionado coincida con el lote actual de cuentas de investigación CS&T.
- Comprobación del rendimiento
- Seleccione la opción Comprobar rendimiento y haga clic en Ejecutar.
- Una vez completada la comprobación de rendimiento, se mostrarán los resultados de rendimiento. Vea el informe de rendimiento del citómetro. Haga clic en Finalizar para completar la comprobación de rendimiento o retire el tubo del citómetro y revise los resultados en la ventana Resumen del sistema .
- Observe el resultado final del análisis morfométrico y de sensibilidad a la fluorescencia que aparecerá en el | resumen del sistema| resultado del rendimiento del citómetro| con el estado: APROBADO
2. Preparación de las esporas
- Después de la esporulación, enjuague las esporas 3 veces con agua helada ultrapura por centrifugación a 17.949 × g durante 10 min.
- Resuspender el pellet obtenido de la última centrifugación con 10 mL de agua ultrapura.
- Autoclave de las esporas durante 45 min a 121 ºC para la inactivación de las células vegetativas.
NOTA: Pruebas previas realizadas por nuestro grupo comparando diferentes métodos de inactivación demostraron que las condiciones mencionadas en el paso 2.3 presentaron alta eficiencia, no mostrando formación de colonias en el método de análisis de siembra en el medio agar LB.
3. Cuantificación de esporas esterilizadas en autoclave mediante citometría de flujo
- Tomar 50 μL de esporas esterilizadas en autoclave e incubarlas protegidas de la luz con bromuro de etidio (EtBr, 10 mg/mL diluido en agua) a un factor de dilución del 0,05% v/v durante 30 min (Figura 1A).
- Lavar las esporas 3 veces con 1 solución salina tamponada con fosfato (PBS) por centrifugación a 17.949 × g durante 10 minutos y volver a suspender en 1 PBS.
- Añadir 10 μL de perlas, siguiendo las recomendaciones del fabricante para la dilución.
- Analice la muestra con un citómetro de flujo como se describe en el paso 4.
NOTA: Es importante recalcar que las cuentas deben pipetearse de la forma más homogénea posible ya que se observaron disparidades en los cálculos cuando este paso no se ejecutó bien.
4. Análisis mediante citometría de flujo
- Inicie sesión en el software Cytometer.
- En el espacio de trabajo de software, seleccione Citómetro | Modo limpio | Siéntese líquidos.
- Ajústelo al espacio de trabajo para determinar el análisis en citometría de flujo.
- Defina las puertas para los análisis (Figura 2).
- Definir la estrategia de compuerta en función de las características morfométricas y de fluorescencia de las partículas en función del control negativo (esporas no marcadas).
- Para determinar la morfometría celular, elija Gráfico de diagrama de puntos para el análisis de los parámetros FSC-A en el eje x y SSC-A en el eje y.
- Para determinar la fluorescencia, elija gráficos de diagrama de puntos FL3 en el eje y utilizando FL5 en el eje x y cree las puertas en cuatro cuadrantes.
- Defina las puertas para los análisis (Figura 2).
- Utilice un tubo de poliestireno con tapón de 12 x 75 mm que contenga muestras sin etiquetar, previamente etiquetadas con EtBr de un solo color, APC de un solo color y APC EtBr+ multicolor.
- Mezcle muy suavemente el tubo que contiene el control negativo y conéctelo a la sonda del citómetro de flujo.
- Haga clic en Adquirir.
- Configure la potencia de los láseres navegando a Citómetro | Parámetros | FSC (375) y SSC (275).
- Establezca el umbral en (500) Citómetro | Umbral.
- Para eliminar la autofluorescencia, analice la muestra sin colorante que contenga solo esporas.
- Ajustar los voltajes de los detectores de filtros: FL3 (603) y FL5 (538) para discriminar poblaciones negativas y positivas con respecto a la fluorescencia elegida en los controles. Citómetro | Parámetros.
- Citómetro | Compensación | Establezca el desplazamiento de configuración de FL5/FL3 en 1.0.
- Después de la morfometría y el análisis fluorescente, configure el dispositivo para eventos de adquisición 30.000 | Experimento | Diseño del experimento | Adquisición |30.000 eventos....
- Después de ajustar los parámetros, adquiera datos para las muestras que están etiquetadas y contienen perlas en el citómetro de flujo.
- Calcule la concentración de esporas como se describe en las instrucciones del fabricante utilizando la ecuación (1).
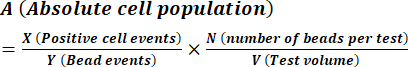 (1)
(1)
NOTA: Para este estudio, el método de cuantificación de perlas se comparó con el análisis de la cámara de conteo de Petroff-Hausser. Además, se comparó el etiquetado de las esporas esterilizadas en autoclave con el etiquetado de las esporas no esterilizadas en autoclave.
5. Estimación del índice de acoplamiento de proteínas en la superficie de una espora mediante citometría de flujo
- Cosechar por centrifugación de 50 μL de esporas (103/μL) 17.949 × g durante 10 min y luego resuspender con 25 μL de 1-etil-3-(3-dimetilaminopropil)carbodiimida (EDC) (300 mM)).
- Incubar durante 15 min a temperatura ambiente.
- Añadir 25 μL de N-hidroxisulfosuccinimida (NHS) (50 mM) a la suspensión de esporas e incubar a temperatura ambiente durante 30 min.
- Lave las esporas 3 veces con 1x PBS por centrifugación como se describe en el paso 5.1.
- Añadir proteína fluorescente y dejar las muestras durante la noche a 15 °C.
NOTA: La proteína fluorescente utilizada en este trabajo fue un anticuerpo anti-IL-10 humano marcado con APC. Este anticuerpo se utilizó como modelo para el estudio, aunque es posible la aplicación de otras moléculas fluorescentes ya que EDC/NHS promueve una ligadura covalente entre los grupos -COOH y -NH2 presentes en las proteínas de la superficie de las esporas. - Lave las esporas según el paso 5.3.
- Añadir 10 mg/mL de bromuro de etidio diluido a 1:50, dejar actuar 1 h en hielo y protegido de la luz (Figura 1B).
- Lave las esporas según el paso 5.3.
- Repita el paso 4.
- Para determinar la fluorescencia, cambie los parámetros FL3 en el eje X y FL5 en el eje Y del diagrama de puntos.
- Analice la muestra utilizando un citómetro de flujo con el láser azul (488 nm) en FL3 (filtro 670 LP) y el láser rojo (633 nm) en FL5 (filtro 660/20), como se describe en el paso 4.
NOTA: Las mismas muestras se analizaron para inmunofluorescencia en portaobjetos bajo un microscopio, en un rango de aumento de 1.000 x y utilizando los siguientes filtros: FITC 480/30 nm (verde) y TRITC 540/25 nm (rojo).
Access restricted. Please log in or start a trial to view this content.
Resultados
En muestras de esporas (AS) esterilizadas en autoclave, se detectaron 2 × 103 esporas/μl y 1 × 103 esporas/μl mediante el uso de perlas de recuento y el método de Petroff-Hausser, respectivamente (Figura 2).
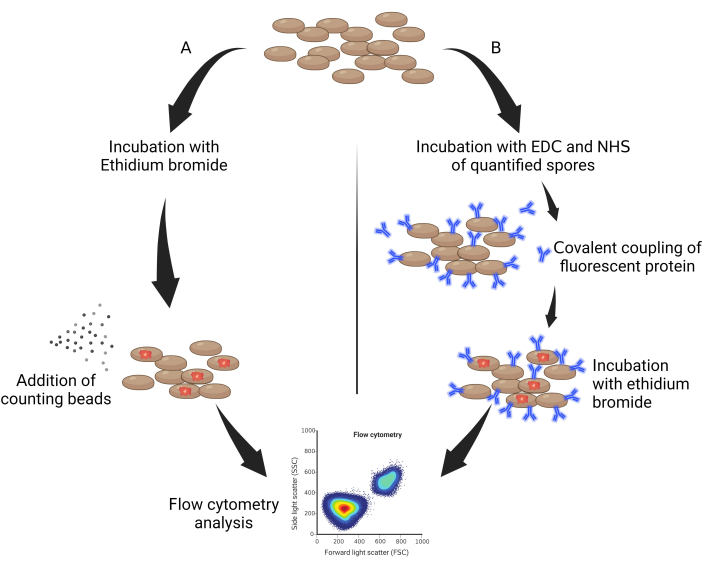
Figura 1: Esquema general de cuantificación de esporas. (A
Access restricted. Please log in or start a trial to view this content.
Discusión
Los métodos tradicionales, como el recuento en placa de las colonias, no solo consumen mucho tiempo, sino que también necesitan células viables y no permiten la cuantificación de esporas inactivadas5. La cámara de Petroff-Hausser es una metodología alternativa, pero requiere de un microscopista experimentado para realizarla. La citometría de flujo ha demostrado ser una alternativa útil para este propósito.
Genovese et al.12 describieron ...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Este estudio fue financiado en parte por la Coordenação de Aperfeiçoamento de Pessoal de Nível Superior-Brasil (CAPES)-Código de Finanzas 001; Governo do Estado do Amazonas con recursos de la Fundação de Amparo à Pesquisa do Estado do Amazonas-FAPEAM; Consejo Nacional de Desarrollo Científico y Tecnológico (CNPq). Los autores agradecen al Programa de Desarrollo Tecnológico en Herramientas para la Salud PDTIS-FIOCRUZ por el uso de sus instalaciones.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| (N-hydroxysuccinimide) (NHS) | Sigma | 130672 | |
| Anti-human fluorescent antibody | BioLegend | 501410 | APC anti-human IL-10 |
| Anti-mouse fluorescent antibody | Thermo Scientific | A32723 | Alexa Fluor Plus 488 |
| BD FACSCanto II | BD | Flow cytometer | |
| BD FACSDiva Cytometer Setup & Tracking Beads Kit (use with BD FACSDiva software v 6.x) | BD | 642412 | Quality control reagent |
| BD FACSDiva Software v. 6.1.3 | BD | 643629 | Software |
| Centrifuge MegaFuge 8R | Thermo Scientific | 75007213 | |
| Counting Beads | BD | 340334 | TruCount Tubes |
| Eclipse 80i | Nikon | Fluorescent Microsope | |
| Ethidium Bromide | Ludwig Biotec | ||
| Phosphate buffered saline | Sigma-Aldrich | A4503 | |
| Plastic Microtubes | Eppendorf | ||
| Polystyrene tube | Falcon | 352008 | 5 mL polystyrene tube, 12 x 75 mm, without lid, non-sterile |
Referencias
- McKenney, P. T., Driks, A., Eichenberger, P. The Bacillus subtilis endospore: assembly and functions of the multilayered coat. Nature Reviews. Microbiology. 11 (1), 33-44 (2013).
- Cutting, S. M. Bacillus probiotics. Food Microbiology. 28 (2), 214-220 (2011).
- Ricca, E., Baccigalupi, L., Cangiano, G., De Felice, M., Isticato, R. Mucosal vaccine delivery by non-recombinant spores of Bacillus subtilis. Microbial Cell Factories. 13, 115(2014).
- Falahati-Pour, S. K., Lotfi, A. S., Ahmadian, G., Baghizadeh, A. Covalent immobilization of recombinant organophosphorus hydrolase on spores of Bacillus subtilis. Journal of Applied Microbiology. 118 (4), 976-988 (2015).
- Harrold, Z., Hertel, M., Gorman-Lewis, D. Optimizing Bacillus subtilis spore isolation and quantifying spore harvest purity. Journal of Microbiological Methods. 87 (3), 325-329 (2011).
- Nicholson, W. L., Setlow, P. Sporulation, germination and outgrowth. Molecular biological methods for Bacillus. , John Wiley and Sons, Chichester, UK. (1990).
- Mora-Uribe, P., et al. Characterization of the adherence of Clostridium difficile spores: the integrity of the outermost layer affects adherence properties of spores of the epidemic strain R20291 to components of the intestinal mucosa. Frontiers in Cellular and Infection Microbiology. 6, 99(2016).
- Paidhungat, M., Setlow, P. Role of ger proteins in nutrient and nonnutrient triggering of spore germination in Bacillus subtilis. Journal of Bacteriology. 182 (9), 2513-2519 (2000).
- Schnizlein-Bick, C., Spritzler, J., Wilkening, C., Nicholson, J., O'Gorman, M. Evaluation of TruCount absolute-count tubes for determining CD4 and CD8 cell numbers in human immunodeficiency virus-positive adults. Site Investigators and The NIAID DAIDS New Technologies Evaluation Group. Clinical and Diagnostic Laboratory Immunology. 7 (3), 336-343 (2000).
- Rapid enumeration of viable spores by flow cytometry. US Patent. , US20040023319A1 https://patentimages.storage.googleapis.com/fa/3c/dc/9e08d9ecd1b315/US20040023319A1.pdf (2003).
- Karava, M., Bracharz, F., Kabisch, J. Quantification and isolation of Bacillus subtilis spores using cell sorting and automated gating. PLoS ONE. 14 (7), e021989(2019).
- Genovese, M., Poulain, E., Doppler, F., Toussaint, R., Boyer, M. Bacillus spore enumeration using flow cytometry: A proof of concept for probiotic application. Journal of Microbiological Methods. 190, 106336(2021).
- Trunet, C., Ngo, H., Coroller, L. Quantifying permeabilization and activity recovery of Bacillus spores in adverse conditions for growth. Food Microbiology. 81, 115-120 (2019).
- Tehri, N., Kumar, N., Raghu, H., Vashishth, A. Biomarkers of bacterial spore germination. Annals of Microbiology. 68, 513-523 (2018).
- Isticato, R., Ricca, E., Baccigalupi, L. Spore adsorption as a nonrecombinant display system for enzymes and antigens. Journal of Visualized Experiments. 145, e59102(2019).
- Song, M., et al. Killed Bacillus subtilis spores as a mucosal adjuvant for an H5N1 vaccine. Vaccine. 30 (22), 3266-3277 (2012).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados