È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Miglioramento dell'enumerazione delle spore di Bacillus subtilis e analisi dell'etichetta nella citometria a flusso
In questo articolo
Riepilogo
Questo protocollo si concentra sull'uso della citometria a flusso e del conteggio delle microsfere per quantificare le spore batteriche marcate con bromuro di etidio. Il metodo è anche efficiente per analizzare l'accoppiamento covalente di proteine sulla superficie di spore intatte.
Abstract
Le spore di Bacillus subtilis sono già state proposte per diverse applicazioni biotecnologiche ed immunologiche; Tuttavia, vi è una crescente necessità di sviluppare metodologie che migliorino la rilevazione degli antigeni immobilizzati sulla superficie delle spore insieme alla loro quantificazione. Le analisi basate sulla citometria a flusso sono state precedentemente proposte come approcci veloci, affidabili e specifici per la rilevazione di cellule marcate di B. subtilis. In questo articolo, proponiamo l'uso della citometria a flusso per valutare l'efficienza di visualizzazione di un anticorpo fluorescente (FA) sulla superficie della spora e quantificare il numero di spore utilizzando le perle di conteggio.
Per questo, abbiamo usato il bromuro di etidio come marcatore del DNA e un anticorpo marcato con alloficocianina (APC), che è stato accoppiato alle spore, come marcatore di superficie. La quantificazione delle spore è stata eseguita utilizzando le perle di conteggio poiché questa tecnica dimostra un'elevata precisione nel rilevamento delle cellule. Le spore marcate sono state analizzate utilizzando un citometro a flusso, che ha confermato l'accoppiamento. Di conseguenza, è stato dimostrato che la marcatura del DNA ha migliorato l'accuratezza della quantificazione mediante citometria a flusso, per la rilevazione delle spore germinate. È stato osservato che il bromuro di etidio non era in grado di marcare le spore dormienti; Tuttavia, questa tecnica fornisce una determinazione più precisa del numero di spore con proteina fluorescente accoppiata alla loro superficie, aiutando così nello sviluppo di studi che si concentrano sull'uso delle spore come piattaforma biotecnologica in diverse applicazioni.
Introduzione
Il Bacillus subtilis è un batterio gram-positivo a forma di bastoncino in grado di produrre spore quiescenti quando le condizioni ambientali non consentono la crescita cellulare1. Le spore sono forme cellulari estremamente stabili e quelle di diverse specie, tra cui B. subtilis, sono ampiamente utilizzate come probiotici per uso umano e animale2. Grazie alle sue proprietà di resistenza e sicurezza, la spora di B. subtilis, che presenta proteine eterologhe, è stata proposta come adiuvante della mucosa, sistema di somministrazione di vaccini e piattaforma di immobilizzazione enzimatica 3,4.
Per ottenere spore da B. subtilis, è necessario esporlo alla privazione di nutrienti utilizzando uno speciale terreno di coltura. Dopo aver ottenuto e purificato queste spore, è necessario quantificarle per migliorare l'efficienza del test 5,6. Pertanto, vengono applicati alcuni metodi per analizzare la concentrazione delle spore ottenute. È possibile utilizzare il conteggio delle piastre e una camera Petroff-Hausser, nota anche come camera di conteggio. Quest'ultimo è stato originariamente sviluppato per determinare la concentrazione delle cellule del sangue; tuttavia, è possibile utilizzarlo nel campo della microbiologia per il conteggio delle spore 7,8. Nonostante sia il metodo standard utilizzato per il conteggio delle cellule, la lettura è laboriosa poiché questo metodo è completamente manuale e la sua precisione dipende dall'esperienza dell'operatore.
Le analisi basate sulla citometria a flusso (FC) sono state precedentemente proposte come approcci veloci, affidabili e specifici per rilevare cellule marcate di Bacillus spp. L'uso di microsfere di conteggio con citometria a flusso ha garantito la riproducibilità nella conta cellulare negli esami di routine (conta assoluta dei linfociti T CD4 e CD8) e nello sviluppo della ricerca che coinvolge particelle in grado di essere rilevate e contate utilizzando la citometria a flusso9. Godjafrey e Alsharif hanno suggerito l'uso di perline di conteggio per la quantificazione FC di spore non marcate10. L'uso della citometria a flusso è stato descritto per il monitoraggio della sporulazione in Bacillus spp. tramite l'etichettatura del DNA delle spore 10,11,12,13. Un altro studio ha utilizzato FC per valutare la quantità di proteine marcate in modo fluorescente sulla superficie delle spore15.
Questo studio ha cercato di utilizzare perle di conteggio commerciali per garantire uno standard di riproducibilità rispetto al conteggio degli eventi utilizzando la citometria a flusso. In questo articolo, suggeriamo l'uso di perline di conteggio per il conteggio delle cellule in FC per perfezionare l'enumerazione delle spore e valutare l'efficienza di accoppiamento di anticorpi marcati con fluorescenza sulla superficie delle spore.
Access restricted. Please log in or start a trial to view this content.
Protocollo
Vedere la Tabella dei materiali per i dettagli relativi a tutti i materiali, gli strumenti e il software utilizzati in questo protocollo.
1. Impostazione della citometria a flusso
- Allineamento dei parametri ottici di un citometro a flusso accoppiato ad un computer
- Accedere al software del citometro.
- Dall'area di lavoro del software, selezionare Citometro | Avviare e attendere qualche minuto | Modalità di pulizia | Liquidi seduti.
NOTA: Le bolle d'aria e le ostruzioni sono state rimosse durante il processo di avvio del citometro, prima dell'acquisizione del campione.
- Controllo di qualità
- Utilizzare il reagente per il controllo qualità per regolare le tensioni dei tubi fotomoltiplicatori e valutarne la sensibilità.
- Preparare il reagente per il controllo qualità in una provetta di polistirolo.
- Per definire la linea di base, preparare le perle di sospensione aggiungendo 0,5 mL di diluente (PBS filtrato 10 mM, pH 7) e 3 gocce di microsfere nella provetta marcata.
- Mescolare il flaconcino di perline con una leggera inversione.
- Dall'area di lavoro del software, selezionare Citometro | CST per connettersi all'interfaccia CST. Attendere qualche minuto e osservare il messaggio citometro disconnesso.
- Collegare il tubo di polistirolo contenente il reagente per il controllo qualità alla sonda del citometro a flusso.
- Utilizzare il reagente per il controllo qualità per regolare le tensioni dei tubi fotomoltiplicatori e valutarne la sensibilità.
- Verifica della configurazione del citometro
- Nella finestra Riepilogo sistema , verificare che la configurazione del citometro sia appropriata per l'esperimento.
- Selezionare l'ID del lotto di perline di setup corrispondente per verificare che l'ID del lotto di perline di setup selezionato corrisponda al lotto corrente di perline di ricerca CS&T.
- Controllo delle prestazioni
- Selezionare l'opzione Controlla prestazioni e fare clic su Esegui.
- Al termine del controllo delle prestazioni, verranno visualizzati i risultati delle prestazioni. Visualizza il rapporto sulle prestazioni del citometro. Fare clic su Fine per completare il controllo delle prestazioni oppure rimuovere la provetta dal citometro ed esaminare i risultati nella finestra Riepilogo sistema .
- Osservare il risultato finale dell'analisi morfometrica e di sensibilità alla fluorescenza che apparirà nel | riepilogo del sistema| risultato delle prestazioni del citometro| con lo stato: SUPERATO
2. Preparazione delle spore
- Dopo la sporulazione, sciacquare le spore 3 volte con acqua ghiacciata ultrapura mediante centrifugazione a 17.949 × g per 10 min.
- Risospendere il pellet ottenuto dall'ultima centrifugazione con 10 mL di acqua ultrapura.
- Autoclavare le spore per 45 minuti a 121 ºC per l'inattivazione delle cellule vegetative.
NOTA: Precedenti test effettuati dal nostro gruppo confrontando diversi metodi di inattivazione hanno dimostrato che le condizioni menzionate nel passaggio 2.3 presentavano un'elevata efficienza, non mostrando formazione di colonie nel metodo di analisi della placcatura sul terreno di agar LB.
3. Quantificazione delle spore sterilizzate in autoclave mediante citometria a flusso
- Prelevare 50 μL di spore sterilizzate in autoclave e incubarle al riparo dalla luce con bromuro di etidio (EtBr, 10 mg/mL diluito in acqua) con un fattore di diluizione dello 0,05% v/v per 30 minuti (Figura 1A).
- Lavare le spore 3 volte con 1 soluzione salina tamponata con fosfato (PBS) mediante centrifugazione a 17.949 × g per 10 minuti e risospendere in 1x PBS.
- Aggiungere 10 μL di perline, seguendo le raccomandazioni del produttore per la diluizione.
- Analizzare il campione utilizzando un citometro a flusso come descritto al punto 4.
NOTA: È importante sottolineare che le perle devono essere pipettate nel modo più omogeneo possibile poiché sono state osservate disparità nei calcoli quando questo passaggio non è stato eseguito correttamente.
4. Analisi mediante citometria a flusso
- Accedere al software del citometro.
- Dall'area di lavoro del software, selezionare Citometro | Modalità di pulizia | Liquidi seduti.
- Regolare l'area di lavoro per determinare l'analisi nella citometria a flusso.
- Definire i gate per le analisi (Figura 2).
- Definire la strategia di gating in base alle caratteristiche morfometriche e di fluorescenza delle particelle in base al controllo negativo (spore non marcate).
- Per determinare la morfometria cellulare, scegliere Dotplot graphic per le analisi dei parametri FSC-A sull'asse x e SSC-A sull'asse y.
- Per determinare la fluorescenza, scegliete i grafici FL3 Dotplot sull'asse y utilizzando FL5 sull'asse x e create i gate in quattro quadranti.
- Definire i gate per le analisi (Figura 2).
- Utilizzare una provetta in polistirene con tappo da 12 x 75 mm contenente campioni non etichettati, precedentemente etichettati con EtBr monocolore, APC monocolore e APC EtBr+ multicolore.
- Mescolare molto delicatamente la provetta contenente il controllo negativo e collegarla alla sonda del citometro a flusso.
- Fare clic su Acquisisci.
- Imposta la potenza dei laser navigando su Citometro | Parametri | FSC (375) e SSC (275).
- Impostare la soglia su (500) Citometro | Soglia.
- Per rimuovere l'autofluorescenza, analizzare il campione privo di coloranti contenente solo spore.
- Regolare le tensioni per i rivelatori filtro: FL3 (603) e FL5 (538) per discriminare le popolazioni negative e positive rispetto alla fluorescenza scelta nei controlli. Citometro | Parametri.
- Citometro | Compensazione | Impostare l'offset di impostazione FL5/FL3 su 1.0.
- Dopo la morfometria e l'analisi fluorescente, configurare il dispositivo per l'acquisizione di 30.000 eventi | Esperimento | Layout dell'esperimento | Acquisizione |30.000 eventi....
- Dopo aver regolato i parametri, acquisire i dati per i campioni etichettati e contenenti microsfere nel citometro a flusso.
- Calcolare la concentrazione di spore come descritto dalle istruzioni del produttore utilizzando l'equazione (1).
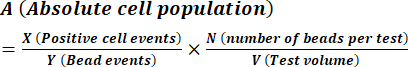 (1)
(1)
NOTA: Per questo studio, il metodo di quantificazione delle perle è stato confrontato con l'analisi della camera di conteggio di Petroff-Hausser. Inoltre, l'etichettatura delle spore sterilizzate in autoclave è stata confrontata con l'etichettatura delle spore non sterilizzate in autoclave.
5. Stima dell'indice di accoppiamento proteico sulla superficie di una spora mediante citometria a flusso
- Raccogliere per centrifugazione 50 μL di spore (103/μL) 17.949 × g per 10 minuti e quindi risospendere con 25 μL di 1-etil-3-(3-dimetilamminopropil)carbodiimmide (EDC) (300 mM)).
- Incubare per 15 minuti a temperatura ambiente.
- Aggiungere 25 μL di N-idrossisolfosuccinimide (NHS) (50 mM) alla sospensione di spore e incubarla a temperatura ambiente per 30 minuti.
- Lavare le spore 3 volte con 1x PBS mediante centrifugazione come descritto al punto 5.1.
- Aggiungere proteine fluorescenti e lasciare i campioni per una notte a 15 °C.
NOTA: La proteina fluorescente utilizzata in questo lavoro era un anticorpo anti-IL-10 anti-umano marcato con APC. Questo anticorpo è stato utilizzato come modello per lo studio, anche se l'applicazione di altre molecole fluorescenti è possibile poiché EDC/NHS promuovono una legatura covalente tra i gruppi -COOH e -NH2 presenti nelle proteine sulla superficie della spora. - Lavare le spore come da punto 5.3.
- Aggiungere 10 mg/mL di bromuro di etidio diluito a 1:50, quindi lasciare per 1 ora su ghiaccio e al riparo dalla luce (Figura 1B).
- Lavare le spore come da punto 5.3.
- Ripetere il passaggio 4.
- Per determinare la fluorescenza, modificare i parametri FL3 sull'asse X e FL5 sull'asse Y del dotplot.
- Analizzare il campione utilizzando un citometro a flusso con il laser blu (488 nm) a FL3 (filtro 670 LP) e il laser rosso (633 nm) in FL5 (filtro 660/20), come descritto al punto 4.
NOTA: Gli stessi campioni sono stati analizzati per l'immunofluorescenza su vetrini al microscopio, ad un intervallo di ingrandimento di 1.000 x e utilizzando i seguenti filtri: FITC 480/30 nm (verde) e TRITC 540/25 nm (rosso).
Access restricted. Please log in or start a trial to view this content.
Risultati
Nei campioni di spore autoclavate (AS), sono state rilevate rispettivamente 2 × 103 spore/μl e 1 ×10 3 spore/μl utilizzando le microsfere di conteggio e il metodo Petroff-Hausser (Figura 2).
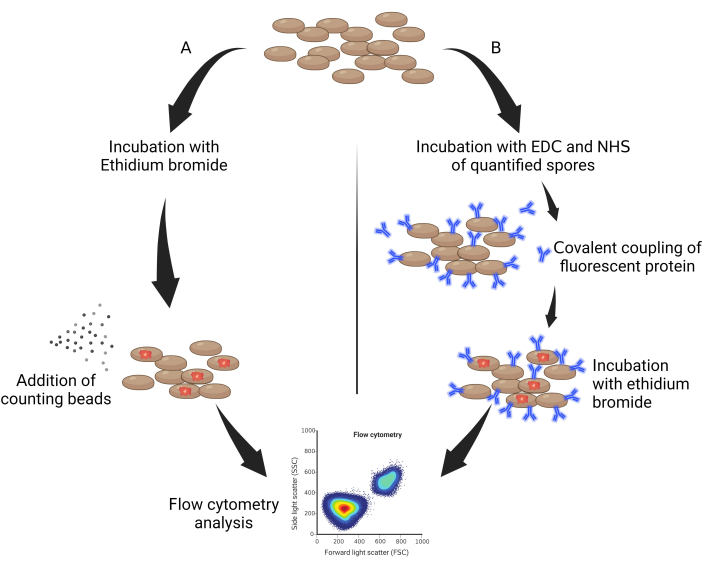
Figura 1: Schema generale di quantificazione delle spore. (A) spore marcate ...
Access restricted. Please log in or start a trial to view this content.
Discussione
I metodi tradizionali, come il conteggio su piastra delle colonie, non solo richiedono molto tempo, ma richiedono anche cellule vitali e non consentono la quantificazione delle spore inattivate5. La camera di Petroff-Hausser è una metodologia alternativa, ma richiede un microscopista esperto per eseguirla. La citometria a flusso si è dimostrata un'utile alternativa a questo scopo.
Genovese et al.12 hanno descritto l'uso della citometria a fluss...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Questo studio è stato finanziato in parte dal Coordenação de Aperfeiçoamento de Pessoal de Nível Superior-Brasil (CAPES)-Codice finanziario 001; Governo do Estado do Amazonas con risorse della Fundação de Amparo à Pesquisa do Estado do Amazonas-FAPEAM; Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq). Gli autori ringraziano il Programma per lo Sviluppo Tecnologico in Strumenti per la Salute PDTIS-FIOCRUZ per l'uso delle sue strutture.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| (N-hydroxysuccinimide) (NHS) | Sigma | 130672 | |
| Anti-human fluorescent antibody | BioLegend | 501410 | APC anti-human IL-10 |
| Anti-mouse fluorescent antibody | Thermo Scientific | A32723 | Alexa Fluor Plus 488 |
| BD FACSCanto II | BD | Flow cytometer | |
| BD FACSDiva Cytometer Setup & Tracking Beads Kit (use with BD FACSDiva software v 6.x) | BD | 642412 | Quality control reagent |
| BD FACSDiva Software v. 6.1.3 | BD | 643629 | Software |
| Centrifuge MegaFuge 8R | Thermo Scientific | 75007213 | |
| Counting Beads | BD | 340334 | TruCount Tubes |
| Eclipse 80i | Nikon | Fluorescent Microsope | |
| Ethidium Bromide | Ludwig Biotec | ||
| Phosphate buffered saline | Sigma-Aldrich | A4503 | |
| Plastic Microtubes | Eppendorf | ||
| Polystyrene tube | Falcon | 352008 | 5 mL polystyrene tube, 12 x 75 mm, without lid, non-sterile |
Riferimenti
- McKenney, P. T., Driks, A., Eichenberger, P. The Bacillus subtilis endospore: assembly and functions of the multilayered coat. Nature Reviews. Microbiology. 11 (1), 33-44 (2013).
- Cutting, S. M. Bacillus probiotics. Food Microbiology. 28 (2), 214-220 (2011).
- Ricca, E., Baccigalupi, L., Cangiano, G., De Felice, M., Isticato, R. Mucosal vaccine delivery by non-recombinant spores of Bacillus subtilis. Microbial Cell Factories. 13, 115(2014).
- Falahati-Pour, S. K., Lotfi, A. S., Ahmadian, G., Baghizadeh, A. Covalent immobilization of recombinant organophosphorus hydrolase on spores of Bacillus subtilis. Journal of Applied Microbiology. 118 (4), 976-988 (2015).
- Harrold, Z., Hertel, M., Gorman-Lewis, D. Optimizing Bacillus subtilis spore isolation and quantifying spore harvest purity. Journal of Microbiological Methods. 87 (3), 325-329 (2011).
- Nicholson, W. L., Setlow, P. Sporulation, germination and outgrowth. Molecular biological methods for Bacillus. , John Wiley and Sons, Chichester, UK. (1990).
- Mora-Uribe, P., et al. Characterization of the adherence of Clostridium difficile spores: the integrity of the outermost layer affects adherence properties of spores of the epidemic strain R20291 to components of the intestinal mucosa. Frontiers in Cellular and Infection Microbiology. 6, 99(2016).
- Paidhungat, M., Setlow, P. Role of ger proteins in nutrient and nonnutrient triggering of spore germination in Bacillus subtilis. Journal of Bacteriology. 182 (9), 2513-2519 (2000).
- Schnizlein-Bick, C., Spritzler, J., Wilkening, C., Nicholson, J., O'Gorman, M. Evaluation of TruCount absolute-count tubes for determining CD4 and CD8 cell numbers in human immunodeficiency virus-positive adults. Site Investigators and The NIAID DAIDS New Technologies Evaluation Group. Clinical and Diagnostic Laboratory Immunology. 7 (3), 336-343 (2000).
- Rapid enumeration of viable spores by flow cytometry. US Patent. , US20040023319A1 https://patentimages.storage.googleapis.com/fa/3c/dc/9e08d9ecd1b315/US20040023319A1.pdf (2003).
- Karava, M., Bracharz, F., Kabisch, J. Quantification and isolation of Bacillus subtilis spores using cell sorting and automated gating. PLoS ONE. 14 (7), e021989(2019).
- Genovese, M., Poulain, E., Doppler, F., Toussaint, R., Boyer, M. Bacillus spore enumeration using flow cytometry: A proof of concept for probiotic application. Journal of Microbiological Methods. 190, 106336(2021).
- Trunet, C., Ngo, H., Coroller, L. Quantifying permeabilization and activity recovery of Bacillus spores in adverse conditions for growth. Food Microbiology. 81, 115-120 (2019).
- Tehri, N., Kumar, N., Raghu, H., Vashishth, A. Biomarkers of bacterial spore germination. Annals of Microbiology. 68, 513-523 (2018).
- Isticato, R., Ricca, E., Baccigalupi, L. Spore adsorption as a nonrecombinant display system for enzymes and antigens. Journal of Visualized Experiments. 145, e59102(2019).
- Song, M., et al. Killed Bacillus subtilis spores as a mucosal adjuvant for an H5N1 vaccine. Vaccine. 30 (22), 3266-3277 (2012).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon