Method Article
Obtención de imágenes de ATG9A, una proteína de membrana de expansión múltiple
En este artículo
Resumen
Este protocolo describe varios métodos que pueden ayudar en el estudio de la biología de ATG9A, incluida la inmunofluorescencia seguida del análisis de imágenes, las consideraciones de sobreexpresión transitoria y la investigación del estado de glicosilación de ATG9A mediante Western blot.
Resumen
La autofagia es una vía altamente conservada que la célula utiliza para mantener la homeostasis, degradar los orgánulos dañados, combatir los patógenos invasores y sobrevivir a condiciones patológicas. Un conjunto de proteínas, llamadas proteínas ATG, comprenden la maquinaria central de la autofagia y trabajan juntas en una jerarquía definida. Los estudios realizados en los últimos años han mejorado nuestro conocimiento de la vía de la autofagia. Más recientemente, se ha propuesto que las vesículas ATG9A están en el corazón de la autofagia, ya que controlan la rápida síntesis de novo de un orgánulo llamado fagóforo. El estudio de ATG9A ha demostrado ser un reto, ya que ATG9A es una proteína transmembrana y está presente en diferentes compartimentos de membrana. Por lo tanto, comprender su tráfico es un elemento importante para comprender la autofagia. En este trabajo se presentan métodos detallados que pueden ser utilizados para estudiar ATG9A y, en particular, su localización mediante técnicas de inmunofluorescencia, que pueden ser evaluados y cuantificados. También se abordan los escollos de la sobreexpresión transitoria. La correcta caracterización de la función ATG9A y la estandarización de las técnicas para analizar su tráfico son cruciales para caracterizar mejor los eventos que gobiernan el inicio de la autofagia.
Introducción
ATG9A es la única proteína transmembrana de la maquinaria central de autofagia y es traficada entre el Golgi y un compartimiento de vesícula ATG9A citosólico, transitando a través del compartimiento endosomal1. Después de haber sido enigmático durante mucho tiempo, recientemente se ha descrito que ATG9A funciona como una scramblasa lipídica, ya que equilibra los lípidos a través de las bicapasde membrana 2,3. Ahora está claro que ATG9A reside en la parte superior de la jerarquía en la formación de autofagosomas y, por lo tanto, su estudio es vital para comprender la autofagia 4,5. Como tal, las vesículas ATG9A han sido propuestas recientemente como la "semilla" del autofagosoma 6,7. Sin embargo, estudios previos han demostrado que ATG9A solo interactúa transitoriamente con el autofagosoma en formación en diferentes etapas de su maduración y no se integra en la membrana autofágica 6,8,9,10,11. Por lo tanto, se necesitan más investigaciones para desentrañar por completo el papel y las múltiples funciones potenciales de ATG9A en la formación de autofagosomas. Sin embargo, la discrepancia entre los modelos actuales y los datos anteriores solo puede resolverse mediante experimentos dirigidos que aborden el tráfico de ATG9A utilizando enfoques cuantitativos validados y marcadores intracelulares.
Existen varias herramientas en uso para estudiar ATG9A, cada una con ventajas y desventajas, y el uso de estas herramientas se complica por la estructura de ATG9A, su función molecular y el tráfico celular 2,8,12. ATG9A forma un homotrímero, se glicosila y es transportado a través de la célula a compartimentos como el Golgi, los endosomas y la membrana plasmática13,14. Dado su complejo itinerario, existen varios desafíos en la interpretación de lecturas como la dispersión de ATG9A desde el Golgi tras tratamientos o estímulos específicos (como la inanición de nutrientes y suero). ATG9A es extremadamente dinámico en términos de tráfico vesicular; de hecho, las vesículas que contienen ATG9A se han definido como el compartimento ATG9A en el contexto de la autofagia inducida por inanición. El compartimento ATG9A, formado por estas vesículas dinámicas, interactúa transitoriamente con varios orgánulos intracelulares 8,15,16,17. Las técnicas descritas aquí, incluida la inmunofluorescencia, las imágenes en vivo y los ensayos de glicosilación, deberían ayudar en la detección y comprensión de la biología de ATG9A. En particular, los enfoques descritos en este artículo ayudarán a abordar cuestiones sobre la localización en compartimentos celulares específicos y las interacciones con socios proteicos específicos y/o compartimentos de membrana. Dado que el dominio central hidrofóbico conservado ATG9A (dominio PFAM PF04109) tiene una topología única y ATG9A circula entre varios compartimentos de membrana, los investigadores deben tener en cuenta ciertos escollos y artefactos cuando se sobreexpresa transitoriamente ATG9A, incluida, entre otras, la retención del retículo endoplásmico (RE). Otros posibles problemas pueden surgir debido a un mal plegamiento de la proteína, agregación artifactual en condiciones normales de crecimiento o detección insuficiente del compartimento vesicular debido a protocolos de permeabilización subóptimos para la inmunofluorescencia.
Al obtener imágenes endógenas de ATG9A, se debe tener cuidado en la preparación de la muestra y la adquisición de imágenes para garantizar la calidad del análisis cuantitativo posterior y la correcta interpretación de los datos. La combinación de las técnicas descritas en este artículo con enfoques bioquímicos estándar (como la inmunoprecipitación o los experimentos de pull-down no descritos aquí) debería mejorar nuestra comprensión de la función de ATG9A. Este conjunto de herramientas experimentales está destinado a ayudar a los nuevos investigadores a navegar por algunos de los ensayos necesarios para determinar la función de ATG9A en su sistema biológico.
Protocolo
Todos los reactivos utilizados en este estudio están disponibles comercialmente, excepto las construcciones de ADN ATG9A y el anticuerpo STO-215 casero (ver Tabla de materiales), que están disponibles a pedido. Las herramientas de análisis aquí descritas se basan en software de código abierto (FIJI/ImageJ)18.
1. Cultivo celular
- Mantener HEK293A células en un matraz tratado con cultivo de tejidos T150 hasta una confluencia del 80%-90% en DMEM (medio Eagle modificado de Dulbecco) con alto contenido de glucosa suplementado con 10% de FBS (suero fetal bovino) y 4 mM de L-glutamina (ver Tabla de materiales). Incubar las células a 37 °C en una incubadora de cultivo de tejidos humidificada al 10% de CO2.
NOTA: HEK293A células se utilizan en el presente estudio, ya que tienen una respuesta de autofagia canónica robusta ante la inanición de aminoácidos, que se detecta, en particular, por un aumento de la CL3 1,9,19 lipídica asociada a la membrana, y son adecuadas para la obtención de imágenes. - Pasar las células aspirando el medio del matraz con una pipeta serológica. Lave las células una vez con 15 ml de 1x solución salina tamponada con fosfato (PBS) o una solución similar antes de agregar 2 ml de solución de tripsina-EDTA (ácido etilendiaminotetraacético) para separar las células. Recoja las células desprendidas con 8 ml de DMEM y vuelva a sembrar un número de células para que se alcance una confluencia del 80%-90% después de 2 días para los experimentos descritos aquí.
2. Tinción endógena de ATG9A
- Siembre HEK293A células (o células de elección) en cubreobjetos de vidrio estériles No. 1.5 colocados en una placa de 24 pocillos para que sean ~80% confluentes al día siguiente. Por lo general, esto da 7 x 104 células/pocillo en 500 μL de DMEM.
NOTA: Para las células HEK293A u otras líneas celulares poco adheridas, los cubreobjetos deben recubrirse con poli-D-lisina a 0,1 mg/ml en agua desionizada. Añadir 500 μL de poli-D-lisina (ver Tabla de Materiales) en la parte superior de los cubreobjetos colocados en los pocillos durante 10 min a temperatura ambiente (RT), seguido de tres lavados con agua desionizada y un último lavado en DMEM. Proceda a sembrar las células después del recubrimiento y tenga cuidado de que se distribuyan uniformemente en la placa de cultivo. - Tratar las células de acuerdo con las condiciones experimentales específicas (es decir, inanición durante 2 h en EBSS [solución salina equilibrada de Earle]).
NOTA: La composición de EBSS es de 1 g/L de D-glucosa, 6,8 g/L de NaCl, 0,4 g/L de KCl, 0,151 g/L de CaCl 2,2H 2 O, 0,2 g/L mM de MgSO 4,7H 2 O, 0,124 g/L de NaH 2 HPO4,2H 2 O y 2,2 g/L de NaHCO 3 (véase la Tabla de Materiales) disueltos en agua destilada. - Aspire el medio y reemplácelo cuidadosamente con 500 μL de solución de formaldehído al 4% en PBS suplementado con 0,1 mM de CaCl 2 y 0,1 mM de MgCl2 durante 20 min en RT para fijar las células.
- Aspirar la solución de formaldehído al 4% de cada pocillo, sustituyéndola por 500 μL de PBS. Repite este paso de lavado tres veces. No deje que los cubreobjetos se sequen o se queden sin líquido.
- Enfriar los grupos aldehídos libres utilizando 500 μL de solución de 50 mM de NH4Cl en PBS durante 10 min a RT.
- Aspirar el PBS y reemplazarlo con 500 μL de una solución de digitonina de 50 μg/mL en PBS (solución madre 1 mg/mL en agua desionizada, ver Tabla de Materiales) durante 5 min a RT para permeabilizar las células.
- Aspire la solución de digitonina de cada pocillo y reemplácela con 500 μL de PBS. Repite este paso de lavado tres veces.
- Aspirar el PBS de cada pocillo y sustituirlo por 500 μL de solución de bloqueo (5% de BSA [albúmina sérica bovina] diluida en PBS) durante 30 min a RT.
- Aspire la solución de bloqueo y reemplácela con 500 μL de PBS.
- Con unas pinzas, recoja los cubreobjetos del pocillo, retire con cuidado el exceso de solución de PBS con toallitas de papel tisú fino o papel de filtro de celulosa, y coloque suavemente cada cubreobjetos, con la célula hacia abajo, sobre una gota de 50 μL de solución de anticuerpos primarios (p. ej., hámster armenio 14F2 diluido a 0,9 μg/ml en solución de BSA/PBS al 1%, consulte la Tabla de materiales). Incubar en una cámara humidificada durante 1 h a RT.
NOTA: Para facilitar su manipulación, las gotas de solución de anticuerpos se pueden colocar en una lámina de película termoplástica autosellante (consulte la Tabla de materiales) dentro de otro recipiente en lugar de directamente sobre una superficie sólida. - Recoja los cubreobjetos con pinzas y, después de drenar suavemente el exceso de solución de anticuerpos primarios con toallitas de tejido fino, vuelva a colocar los cubreobjetos (con la célula hacia arriba) en la placa de 24 pocillos y lávelos tres veces con PBS.
- Repita el paso 2.10. Con unas pinzas, recoja los cubreobjetos de cada pocillo, drenando con cuidado el exceso de PBS con toallitas de tejido fino, y coloque suavemente cada cubreobjetos (con la célula hacia abajo) sobre una gota de 50 μl de solución secundaria de anticuerpos diluida 1:1.000 en una solución BSA/PBS al 1% (p. ej., Cy3 Goat Anti-Armenian Hamster IgG, que tiene una reactividad cruzada mínima con bovinos, humanos, proteínas séricas de ratón, conejo y rata, véase la Tabla de Materiales). Incubar en una cámara humidificada durante 1 h a RT.
NOTA: Opcional: Se puede utilizar un marcador de citoesqueleto además del anticuerpo secundario para el análisis de imagen posterior (es decir, Alexa Fluor 647 Phalloidin diluido 1:1.000, consulte la Tabla de materiales). Para facilitar su manipulación, las gotas de solución de anticuerpos se pueden colocar en una hoja de película de sellado dentro de otro recipiente en lugar de directamente sobre una superficie sólida. - Recoja los cubreobjetos con unas pinzas y, después de drenar el exceso de solución de anticuerpos secundarios, colóquelos en la placa de 24 pocillos (con la célula hacia arriba) y lávelos tres veces con 500 μL de PBS.
- Con unas pinzas, recoja los cubreobjetos, drene con cuidado el exceso de PBS con toallitas de papel fino, y coloque suavemente cada cubreobjetos (con la célula hacia abajo) sobre una gota de 50 μL de solución de Hoechst 1:4.000 (Hoechst 33342) en PBS. Incubar en una cámara de humedad durante 5 min a RT.
NOTA: Para facilitar su manipulación, las gotas de solución de anticuerpos se pueden colocar en una hoja de película de sellado dentro de otro recipiente en lugar de directamente sobre una superficie sólida. - Recoja los cubreobjetos con unas pinzas y, después de escurrir suavemente el exceso de solución de Hoechst con toallitas de papel fino, vuelva a colocar los cubreobjetos en la placa de 24 pocillos y lávelos tres veces con PBS y una vez con agua desionizada (500 μL por lavado).
- Drene con cuidado el exceso de agua desionizada con toallitas de tejido fino y coloque suavemente cada cubreobjetos (con la célula hacia abajo) sobre una gota de 10-20 μL de solución de montaje (consulte la Tabla de materiales) sobre un portaobjetos de microscopio para inmunofluorescencia, evitando la formación de burbujas de aire.
NOTA: El medio de montaje utilizado aquí tiene el mismo índice de refracción que el aceite de inmersión y se endurece después de unas horas a RT o durante la noche a 4 °C. Se pueden utilizar soluciones de montaje que no se endurezcan, pero se debe tener cuidado de sellar el cubreobjetos con esmalte de uñas. - Retire el exceso de solución de montaje por aspiración y deje que las muestras se sequen mientras permanecen planas en RT durante la noche en la oscuridad, ya sea en un soporte para portaobjetos o cubiertas con papel de aluminio.
3. Adquisición de imágenes
- Encienda el microscopio confocal. Abra el software de adquisición de imágenes (consulte Tabla de materiales) para iniciar la configuración de adquisición de imágenes.
- En la pestaña Función , seleccione la lente objetivo Plan-Apochromat 63x/1.4 Oil DIC M27 para capturar las imágenes para su análisis.
- En la pestaña Función de adquisición , encienda los láseres apropiados en la zona Láser del panel de control (Argón, Diodo-405-30, DPSS 561-10 y HeNe633).
- En la zona Configuración de imágenes del Panel de control, cree cuatro pistas, cada una de las cuales corresponde a un canal, para realizar la adquisición secuencial.
- Establezca la resolución adecuada de las imágenes en la ventana Modo de adquisición . Establezca la resolución en 1.024 x 1.024 (tamaño de fotograma) y seleccione una profundidad de bits de 16 bits.
- Para cada canal, ajuste la potencia y la ganancia del láser para obtener una buena señal sin píxeles saturados, lo que impediría el análisis de la intensidad de la imagen. Con el botón Live, ajuste el nivel de salida del láser y la ganancia (maestro).
NOTA: Se recomienda el uso de la opción de indicador de rango para la detección de píxeles saturados. Mantenga la potencia del láser entre el 1% y el 10% y la ganancia (maestra) por debajo de 850 para evitar el ruido de fondo. - En la zona Canales del Panel de control, establezca la misma apertura estenopeica para cada canal, considerando 1 Unidad de Airy (UA) para el canal con la longitud de onda más alta.
- Adquiera 10 campos aleatorios que contengan cantidades similares de celdas (20-30 celdas por campo). La adquisición de 100-200 células por condición proporcionará una buena potencia para el posterior análisis de imágenes.
4. Análisis de imágenes de dispersión de ATG9A
- Descargue el software FIJI de Internet (consulte la Tabla de materiales). Abra la imagen con FIJI haciendo clic en Plugins > Bio-Formats > BioFormats Importer.
- Haga clic en Analizar > Establecer mediciones y seleccione el valor de gris medio en la ventana de mediciones para el análisis de intensidad.
- Haga clic en Imagen > Color > Dividir canales y separe los canales en tres imágenes (marcador de Golgi, citoesqueleto, señal ATG9A).
- Vaya a Imagen > Ajustar > umbral y defina un umbral en el canal correspondiente al marcador de Golgi.
- Haga clic en Editar > selección > Crear selección y cree una selección a partir de la imagen binaria.
- Vaya a Editar > selección > Agregar al administrador > Cambiar nombre (Golgi), guarde la selección en el administrador de ROI y cámbiele el nombre a Golgi.
- Haga clic en Imagen > Ajustar > Umbral y defina un umbral en el canal correspondiente al marcador del citoesqueleto para definir el contorno celular.
- Vaya a Editar > selección > Crear selección y cree una selección a partir de la imagen binaria.
- Haga clic en Editar > selección > Agregar al administrador > Cambiar nombre (Total), guarde la selección en el administrador de ROI y cámbiele el nombre a Total.
- Seleccione la imagen correspondiente a la tinción ATG9A.
- En Analizar > medir, aplique el ROI de Golgi haciendo clic en él y mida la intensidad de la región de Golgi .
- Haga clic en Analizar > medir, aplique el ROI Total haciendo clic en él y mida la intensidad de la región Total.
- Repita el procedimiento en todas las imágenes, guarde los resultados como archivos .csv y procese los datos mediante la siguiente fórmula:
Velocidad de dispersión ATG9A = Intensidad de Golgi/Intensidad total
5. Obtención de imágenes de células vivas de construcciones ATG9A
- Siembre HEK293A células (o células de elección) en 2 ml de medio en una placa de cultivo de tejidos de 60 mm para que alcancen ~65%-70% de confluencia al día siguiente; Por lo general, esto da 1 x 10 6-2 x 106 celdas.
- Al día siguiente, prepare mezclas de lipofectamina:ADN (o un reactivo de transfección de ADN alternativo adecuado, consulte la Tabla de materiales) de la siguiente manera:
- Dependiendo de la eficiencia de la expresión del constructo, diluir 0,5-2 μg de ADN plasmídico en 100 μL de un medio adecuado sin suero y mezclar suavemente la solución pipeteando hacia arriba y hacia abajo. Incubar la mezcla durante 5 min a RT.
NOTA: La construcción del ADN plasmídico es específica del experimento. Los investigadores pueden clonar por sí mismos u obtener de los autores si así lo solicitan. - Diluir el reactivo de transfección Lipofectamine 2000 en una proporción de 3:1 Lipofectamine:DNA en 100 μL de un medio adecuado sin suero y mezclar suavemente la solución pipeteando hacia arriba y hacia abajo. Incubar esta mezcla durante 5 min a RT.
- Mezcle ambas soluciones pipeteando suavemente hacia arriba y hacia abajo e incube durante 20 minutos a RT.
- Dependiendo de la eficiencia de la expresión del constructo, diluir 0,5-2 μg de ADN plasmídico en 100 μL de un medio adecuado sin suero y mezclar suavemente la solución pipeteando hacia arriba y hacia abajo. Incubar la mezcla durante 5 min a RT.
- Agregue la mezcla de lipofectamina:ADN a cada placa de cultivo celular que contenga 4 ml de medio de crecimiento, y mueva suavemente la placa hacia adelante y hacia atrás para distribuir la mezcla de manera uniforme. Incubar las células a 37 °C en una incubadora de cultivo celular humidificada al 10% deCO2.
- Reemplace el medio con medio de crecimiento fresco 4 h después de la transfección e incube las células a 37 °C en una incubadora de cultivo celular humidificada al 10% de CO2 durante la noche.
- Al día siguiente, tripsinizar, contar y resembrar las células en placas de cultivo adecuadas para microscopía de células vivas (placas de cultivo con un cubreobjetos de vidrio n.º 1,5, véase la tabla de materiales). Siembre 0,4 x 10 6-0,7 x 106 células en los platos para alcanzar ~60%-75% de confluencia en el cubreobjetos en el momento de la toma de imágenes.
NOTA: Se recomienda el recubrimiento con poli-D-lisina, como se explica en el paso 2.1, para las células HEK293A. - Al día siguiente (48 h después de la transfección), tome imágenes de las células.
6. Investigación del estado de glicosilación de ATG9A
- Siembre HEK293A células (o células de elección) en una placa de cultivo de tejidos de 10 cm para que alcancen ~80% de confluencia al día siguiente si planea investigar ATG9A endógeno, que generalmente da 1.5 x 10 6-2.5 x 106 células en 10 mL. Alternativamente, siembre las células para que alcancen ~ 65%-70% de confluencia si transfecta construcciones ATG9A, lo que generalmente da 1 x 10 6-1.5 x 106 células en 10 mL.
- Trate las células con 100 μg/mL de ciclohexamida (CHX, ver Tabla de materiales) o vehículo al día siguiente. Incubar durante 24 h (o hasta el punto de tiempo deseado).
- Retire las células de la incubadora, aspire el medio y colóquelo en hielo. Reemplácelo con 5 ml de 1x PBS helado. Separe físicamente las células de la placa con un raspador de células y pipetee la solución de células PBS en un tubo de fondo cónico de 15 ml. Centrifugar a 800 x g durante 5 min a 4 °C para granular las células.
- Aspirar el sobrenadante y resuspender las células en ~100 μL (dependiendo del tamaño del gránulo celular) de tampón de lisis de TNTE helado (Triton al 1%, 150 mM de NaCl, 20 mM de Tris-HCl, pH 7,4, 0,5 mM de EDTA) suplementado con comprimidos de cóctel de inhibidores de la proteasa (ver Tabla de materiales), y transferir a un tubo de microcentrífuga de 1,5 mL.
- Incubar el lisado en hielo durante 15 min. Después de esto, centrifugue el lisado a 20,000 x g durante 10 min a 4 °C para sedimentar los núcleos y los desechos insolubles, y transfiera el sobrenadante a un tubo de microcentrífuga nuevo.
- Cuantificar la concentración de proteínas del lisado utilizando el método de Bradford20 y un espectrofotómetro que puede medir a una longitud de onda de 595 nm.
- Normalice las cantidades de proteína y combine 16 μL del lisado (que no contenga más de 100 μg de proteína en total) con 5 veces tampón PNGasa F (Péptido:N-glicosidasa F) de acuerdo con las instrucciones del fabricante antes de agregar 1 μL de enzima PNGasa F (consulte la Tabla de materiales). Incubar según las instrucciones del fabricante de PNGase F.
- Añadir un volumen de tampón de muestra Laemmli 3x para lograr una concentración 1x, e incubar a 65 °C durante 5 min antes de cargar para electroforesis utilizando un gel de acetato de tris para maximizar la separación de proteínas. Transfiera las proteínas del gel a una membrana adecuada (es decir, PVDF [difluoruro de polivinilideno]) utilizando los protocolos estándar de Western blot21 (ver Tabla de materiales).
NOTA: Hervir las muestras a 95 °C hará que ATG9A se agregue, reduciendo así la detección de ATG9A. - Realizar el western blot utilizando anticuerpos específicos para ATG9A (anticuerpo STO-215, de producción propia1) (ver Tabla de Materiales). Deje la sección de mayor peso molecular de la membrana sin cortar para visualizar las especies de mayor peso molecular ATG9A.
Resultados
ATG9A es una proteína transmembrana asociada a varios compartimentos de membrana intracelular 8,17,22,23,24. En condiciones basales, ATG9A se localiza principalmente en la red trans-Golgi (TGN), como lo indica la inmunofluorescencia de la proteína endógena y los solapamientos con GM130, un marcador cis-Golgi (Figura 1A), así como en pequeñas vesículas que se superponen parcialmente con el compartimento de reciclaje endocítico (ERC)23. La localización de ATG9A en el Golgi se puede detectar utilizando diferentes protocolos de inmunofluorescencia. Sin embargo, la fracción vesicular de ATG9A, así como su cambio de localización, en particular el aumento de la reserva vesicular, en respuesta a estímulos específicos como la inanición de nutrientes y suero, puede ser bastante variable en intensidad y difícil de visualizar con los enfoques de imagen convencionales. La relación entre ATG9A localizada en el Golgi y ATG9A localizada en una fracción vesicular se denomina velocidad de dispersión de ATG9A. Para detectar cambios en la tasa de dispersión de ATG9A, por ejemplo, tras el tratamiento con EBSS, que se utiliza para agotar tanto el suero como los aminoácidos, un marcador de Golgi como GM130 o TGN46 y un marcador de citoesqueleto como la faloidina, que tiñe el contorno celular25, son útiles para cuantificar fácilmente la dispersión de ATG9A (Figura 1B). Es importante destacar que el análisis de la relación de fluorescencia media solo puede interpretarse como una medida comparativa entre condiciones en lugar de como una tasa fija de dispersión. La relación entre compartimentos depende en gran medida de factores biológicos y no biológicos, como la línea celular utilizada, la calidad de la tinción o los métodos de umbral aplicados (Figura 1B). Por esta razón, el investigador necesita establecer una tubería que sea capaz de detectar el enriquecimiento de ATG9A Golgi en sus condiciones experimentales específicas y luego extender el análisis con los mismos parámetros a todas las imágenes del conjunto a analizar. Las imágenes binarias representativas y las áreas seleccionadas para el análisis de la fluorescencia media de ATG9A se muestran como guía en la Figura 1B.
ATG9A alberga varios dominios transmembrana flanqueados por dos dominios N- y C-terminal relativamente flexibles y no estructurados, de los cuales la secuencia C-terminal abarca casi la mitad de la proteína12. Es importante destacar que el patrón de localización de la sobreexpresión de ATG9A puede verse influenciado por el extremo de la proteína que está marcado (Figura 2A). En particular, cuando se utilizan sistemas de expresión transitoria y se marca ATG9A directamente en su extremo N-terminal con una etiqueta fluorescente (p. ej., eGFP, mRFP o derivados), su localización de Golgi puede verse parcialmente comprometida, con menos enriquecimiento observado en condiciones basales (es decir, alimentadas), mientras que las vesículas ATG9A siguen siendo fácilmente visibles (Figura 2A). El marcaje de ATG9A en su extremo C parece inducir ligeramente grupos positivos de GFP más grandes que podrían agregarse. Finalmente, una versión monomérica de mRFP-ATG9A también muestra grupos similares de vesículas fluorescentes y poca tinción de Golgi en células sobreexpresadas (Figura 2A).
ATG9A se pliega en la membrana del RE antes de ser traficado a las vesículas de Golgi y ATG9A. Durante su residencia en el RE, ATG9A se modifica por glicanos ligados a N en la asparagina 99, y luego, al llegar al Golgi, adquiere glicanos ligados a N complejosy maduros 1,14. Esta modificación por glicosilación puede ser detectada a través de Western blot por la aparición de una doble banda14. De acuerdo con su localización intracelular, la mayor parte del ATG9A endógeno alberga glicanos complejos ligados a N y, por lo tanto, predomina la banda de mayor peso molecular, con una débil banda de menor peso molecular también visible (Figura 2B). La presencia de una doble banda se ve más fácilmente cuando se utilizan geles de acetato de tris para mejorar la resolución de proteínas de mayor peso molecular (Figura 2B, control, t = 0). Cuando la proteína endógena se somete al tratamiento con PNGasa F (péptido:N-glicosidasa F), que elimina la mayor parte de los glicanos complejos ligados a N, la proteína funciona como una sola banda (Figura 2B, PNGasa F, t = 0). Por lo tanto, el estado de glicosilación ligado a N de ATG9A se puede utilizar como proxy para monitorear la salida de ATG9A del RE al Golgi, que se refleja en la relación relativa entre las dos bandas.
Cuando la transfección de mRFP-ATG9A se construye transitoriamente, la proteína sobreexpresada se acumula inicialmente en el RE, posiblemente porque la maquinaria de tráfico es incapaz de plegar y traficar todo el ATG9A, y la banda de menor peso molecular es predominante (Figura 2C, control t = 0). En particular, después de 24 h de expresión de mRFP-ATG9A, hay aproximadamente una distribución igual entre las bandas superior e inferior, lo que sugiere que el grupo mRFP-ATG9A se está moviendo hacia el Golgi (Figura 2C, Control, t = 24). Si las células se tratan con cicloheximida (CHX), que bloquea la síntesis de proteínas de novo 26, se puede aclarar el plegamiento y la salida de ATG9A del RE. Como la proteína endógena está plegada, glicosilada y residente en el Golgi, el tratamiento con CHX no altera significativamente la proporción de las bandas de menor y mayor peso molecular (Figura 2B, Control). Sin embargo, utilizando la expresión transitoria de mRFP-ATG9A, el tratamiento con CHX promueve la acumulación de la banda de mayor peso molecular (Figura 2C, Control, CHX t = 24). La banda mRFP-ATG9A sobreexpresada de mayor peso molecular colapsa en la banda inferior después del tratamiento con PNGasa F (Figura 2C, PNGasa F, t = 24). Estos datos muestran que la proteína endógena adquiere rápidamente glicanos maduros, como se refleja en el predominio de la banda de mayor peso molecular, y la persecución de CHX no afecta la proporción de las bandas dobles (Figura 2B). En el caso de la sobreexpresión transitoria de mRFP-ATG9A, el tratamiento con CHX induce la acumulación de la banda superior, lo que indica que se adquieren glicanos más maduros a medida que el grupo de RE se pliega y sale del RE hacia el Golgi (Figura 2C).
La adición de un enlazador entre la secuencia ATG9A y las etiquetas fluorescentes puede ser útil para promover una localización y un tráfico más fisiológicos de la proteína. La fusión de una secuencia 3x-FLAG (24 aminoácidos) entre un fluoróforo N-terminal y ATG9A ayuda a que la proteína sobreexpresada se comporte de manera similar a la endógena (Figura 3). De hecho, la sobreexpresión de mCherry-3xFLAG-ATG9A se colocaliza con el marcador de Golgi GM130 en condiciones de alimentación (Figura 3A). Es importante destacar que esta localización y el compartimento vesicular de ATG9A se conservan a lo largo del tiempo, lo que permite el estudio espacio-temporal del tráfico de ATG9A (Figura 3B).
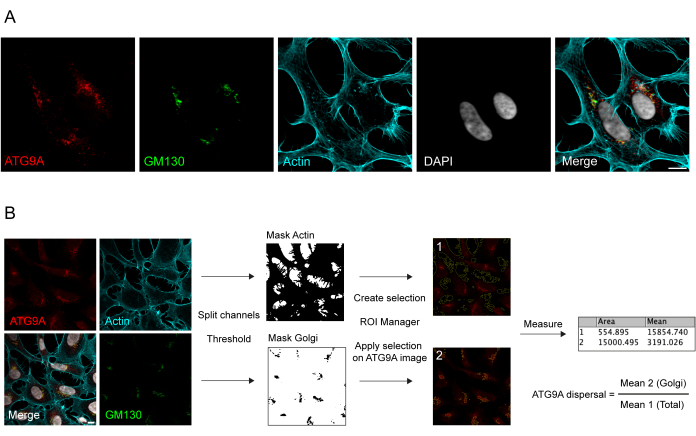
Figura 1: Análisis de imágenes de la localización endógena de ATG9A. (A) Imagen representativa de inmunofluorescencia de ATG9A endógeno (rojo), GM130 como marcador de Golgi (verde) y faloidina para visualizar el citoesqueleto de actina (cian). Barra de escala = 10 μm. (B) Flujo de trabajo del análisis de imágenes para determinar la fracción de ATG9A endógena que se localiza en el área de Golgi. Barra de escala = 10 μm. Haga clic aquí para ver una versión más grande de esta figura.
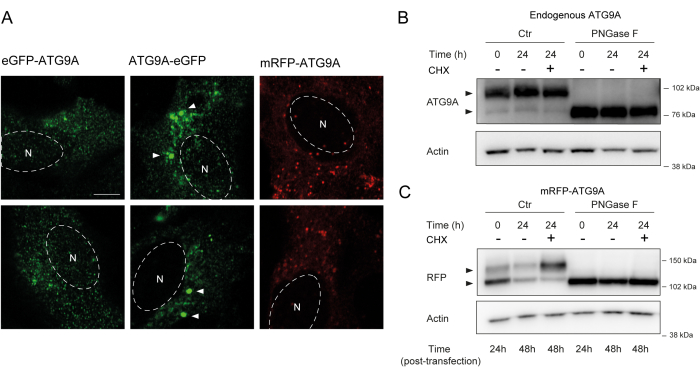
Figura 2: Análisis de las construcciones de ATG9A marcadas con fluorescencia por localización y glicosilación. (A) El ATG9A marcado con eGFP N-terminal está menos localizado en el Golgi y reside principalmente en las vesículas. El ATG9A marcado con eGFP C-terminal exhibe agregados dentro de la celda (algunos ejemplos están marcados con puntas de flecha blancas; el eGFP-ATG9A y el ATG9A-eGFP están en verde). El ATG9A marcado con mRFP N-terminal está menos localizado en el Golgi y reside principalmente en las vesículas. N denota la ubicación aproximada del núcleo de la célula, y el mRFP-ATG9A está en rojo. Barra de escala = 5 μm. (B) El ATG9A endógeno aparece como dos bandas cuando se analiza mediante Western blot (puntas de flecha): una banda superior (glicanos unidos a N complejos) y una banda inferior (sin glicanos maduros ligados a N). El tratamiento con ciclohexamida (CHX) no afecta a la relación entre las bandas superior e inferior. El tratamiento con PNGasa F provoca la desaparición de la banda superior. (C) Después de la transfección transitoria de ATG9A marcado con mRFP en células HEK293A, dos bandas prominentes son visibles en Western blot (puntas de flecha). El tratamiento con PNGasa F provoca la desaparición de la banda superior. El tratamiento con CHX después de la transfección conduce a un aumento de la glicosilación, ya que el grupo de ATG9A transfectado se transporta desde el RE hasta el Golgi. Haga clic aquí para ver una versión más grande de esta figura.
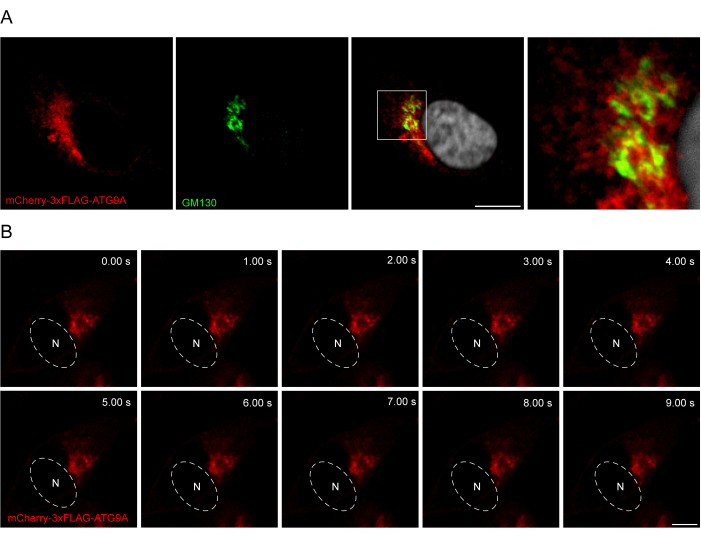
Figura 3: Análisis de la localización de mCherry-3xFLAG-ATG9A mediante inmunofluorescencia e imágenes en vivo. (A) Experimentos de inmunofluorescencia de células HEK293A sobreexpresando transitoriamente mCherry-3xFLAG-ATG9A y teñidas con el marcador de Golgi GM130. Barra de escala = 10 μm. El mCherry-3xFLAG-ATG9A está en rojo y el marcador GM130 Golgi está en verde. (B) Montaje a partir de experimentos de imágenes en vivo en células HEK293A que sobreexpresan transitoriamente mCherry-3xFLAG-ATG9A. N denota la ubicación aproximada del núcleo. Período de tiempo = 1 fps. Barra de escala = 10 μm. El mCherry-3xFLAG-ATG9A está en rojo. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Este estudio ilustra las diversas herramientas que se pueden utilizar para investigar la localización de ATG9A. En primer lugar, este estudio describe cómo se puede visualizar ATG9A mediante inmunofluorescencia y cómo se puede cuantificar. En segundo lugar, se comparan las estrategias que se pueden utilizar para marcar ATG9A con un marcador fluorescente para su visualización en células fijas o vivas. Finalmente, este trabajo describe cómo investigar y utilizar el estado de glicosilación de ATG9A para determinar si ATG9A ha salido del RE y ha traficado a través del Golgi.
En cuanto a la caracterización de la localización endógena de ATG9A por inmunofluorescencia, se debe tener cuidado con los métodos de fijación y permeabilización empleados para el experimento. De acuerdo con los procedimientos estándar aquí descritos, la fijación de paraformaldehído junto con la permeabilización de digitonina son buenas condiciones para visualizar vesículas ATG9A asociadas a Golgi y ATG9A positivas 7. Junto con la fijación y la permeabilización, el momento de la incubación con la solución primaria de anticuerpos también es crítico. Hemos observado, pero no documentado, que concentraciones más altas de solución de anticuerpos primarios y tiempos de incubación más largos pueden conducir a un aumento de tergiversación en la tinción de Golgi de ATG9A, lo que finalmente compromete la detección de la redistribución de ATG9A a otros compartimentos de membrana. Además, dado que ATG9A está presente en muchos compartimentos intracelulares 1,13,17,22,23,24,27,28, es importante utilizar marcadores de membrana específicos, junto con ATG9A, para identificar dónde se encuentra ATG9A. En el pasado se han utilizado varios enfoques para cuantificar la localización de ATG9A, incluido el coeficiente de correlación de Pearson para la colocalización29. Sin embargo, la superposición parcial de ATG9A con el compartimento vesicular de Golgi y distinto conduce a un alto número de valores atípicos de píxeles, lo que puede sesgar la interpretación del coeficiente de correlación. Por esta razón, se prefiere un enfoque más simplista basado en la relación de la fluorescencia media en los dos compartimentos que se van a analizar, y este enfoque es menos sensible a la variabilidad célula por célula. Para obtener más información sobre el análisis de imágenes a través de microscopía, se dirige a los lectores al capítulo30 de este libro.
Al investigar el estado de glicosilación de ATG9A, es importante la selección de geles para ejecutar las transferencias de Western. Para este protocolo, se prefieren los geles de trisacetato al 3%-8% porque ofrecen la resolución más alta para proteínas más grandes, pero también se pueden usar composiciones de gel alternativas o tampones de funcionamiento que ofrecen una buena separación de proteínas de alto peso molecular. El experimentador puede asegurar la máxima separación de proteínas aumentando el tiempo de electroforesis.
Al preparar las muestras para visualizar ATG9A en Western blot, se debe tener cuidado de no hervir las muestras después de agregar el tampón Laemmli; la ebullición a 95 °C induce la formación de agregados de ATG9A y, posteriormente, ATG9A no migra de manera eficiente al gel1. Se recomienda calentar las muestras a 65 °C durante 5 min27.
Los niveles altos de transfección generalmente conducen a una mayor acumulación de ATG9A en el RE, mientras que los niveles moderados de expresión ayudan a la localización fisiológica de la proteína. Como anécdota, los tiempos de incubación de 72 h en lugar de 48 h a menudo ayudan a reducir los artefactos de localización del RE. En particular, mRFP-ATG9A puede informar con precisión sobre el tráfico y la función de ATG9A si los niveles se controlan a través de niveles de expresión o mediante el uso de líneas celulares estables 8,9,22,27.
El hecho de que una población de ATG9A sobreexpresada no adquiera glicanos maduros ligados a N puede utilizarse como lectura para detectar el tráfico de ATG9A perturbado. Al mutar o eliminar ciertas regiones de ATG9A, existe el riesgo de un aumento de la retención de RE, lo que puede conducir a la imposibilidad de adquirir glicanos maduros ligados a N y, por lo tanto, a una banda de ATG9A de migración más rápida en Western blot. Los investigadores que trabajan con construcciones ATG9A truncadas deben comprobar la retención del RE, los estados de glicosilación y la localización de Golgi.
Para la obtención de imágenes de células vivas de ATG9A, un microscopio Airyscan, que se basa en la función rápida Airyscan, proporciona una resolución óptima de unos 120 nm. Para la precisión de la localización, las velocidades de fotogramas de alrededor de 1-2 fotogramas por segundo (fps) en el modo de superresolución son óptimas en función de cuántos canales se visualicen. También se pueden utilizar microscopios confocales similares que pueden obtener imágenes a alta velocidad para obtener imágenes de vesículas ATG9A; Sin embargo, hay que tener en cuenta que la velocidad de obtención de imágenes puede afectar directamente a la detección de eventos y, por tanto, afectar a la interpretación de los datos.
En resumen, los protocolos presentados describen formas de cuantificar y caracterizar la localización de ATG9A mediante inmunofluorescencia, microscopía de células vivas y su estado de glicosilación. Estos protocolos pueden ayudar a los investigadores que trabajan con ATG9A y ayudar a evitar algunos escollos.
Divulgaciones
S.A.T. forma parte del consejo asesor científico de Casma Therapeutics.
Agradecimientos
Los autores agradecen a Rocco D'Antuono por los aspectos de corrección del manuscrito, así como a todos los miembros actuales y pasados del laboratorio de Biología Celular Molecular de la Autofagia (MCBA) por las discusiones que condujeron al refinamiento de estos protocolos. A. v.V., S.d.T., E.A., S.A.T, contaron con el apoyo del Instituto Francis Crick, que recibe su financiación principal de Cancer Research UK (CC2134), el Consejo de Investigación Médica del Reino Unido (CC2134). Esta investigación fue financiada en su totalidad o en parte por el Wellcome Trust (CC2134). A los efectos del acceso abierto, el autor ha aplicado una licencia pública de derechos de autor CC BY a cualquier versión del Manuscrito Aceptado por el Autor que surja de esta presentación.
Materiales
| Name | Company | Catalog Number | Comments |
| 24 multiwell plates | Falcon | 353047 | For tissue culture |
| 35 mm Dish | No. 1.5 Coverslip | 14 mm Glass Diameter | Uncoated | MATTEK | P35G-1.5-14-C | Cell culture dish for live-cell microscopy |
| 4x Laemmli Sample Buffer | Bio-Rad | 1610747 | |
| 60 mm tissue culture dish | Thermofisher Scientific | 10099170 | For tissue culture |
| Alexa Fluor 647 Phalloidin | Thermofisher Scientific | A22287 | Actin stain |
| anti-ATG9A antibody | home made | STO-215 | Rabbit anti N-terminal peptide ATG9A |
| anti-Rabbit IgG, peroxidase-linked | Invitrogen | 10794347/NA934-1ml | Secondary antibody for rabbit polyclonal STO-215 |
| anti-RFP antibody | Evrogen | AB233 | for Western Blot |
| ATG9A Monoclonal Antibody (14F2 8B1), Invitrogen | Invitrogen | 15232826 | Antibody for immunofluorescence |
| ATG9A-eGFP | home made | Construct which expresses tagged ATG9A | |
| Bemis Parafilm | Thermofisher Scientific | 11747487 | self-sealing thermoplastic film |
| Bio-Rad Protein Assay Dye Reagent Concentrate | Bio-Rad | 5000006 | For determining protein concentration |
| Bovine serum albumin (BSA) | Merck | 10735086001 | For blocking non-specific labelling |
| CaCl2.2H2O | / | For PBS and EBSS | |
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | Supplement in lysis buffer to prevent protein degradation |
| Cy3 AffiniPure Goat Anti-Armenian Hamster IgG | Jackson ImmunoResearch | 127-165-099 | Secondary antibody for i14F2 8B1 antibody for mmunofluorescence |
| Cyclohexamide | Sigma Aldrich | 66-81-9 | To stop protein translation |
| D-Glucose | / | For EBSS | |
| Digitonin | Merck | 300410 | For permeabilizing cells |
| DMEM | Merck | D6546-6x500ml | For tissue culture |
| eGFP-ATG9A | home made | Construct which expresses tagged ATG9A | |
| Fetal Bovine Serum | Gibco | 10270-106 | Supplement for DMEM for cell culture |
| FIJI (ImageJ) | / | https:/fiji.sc/ | Open source image analysis software |
| Hoechst | Thermofisher Scientific | H3570 | Stains the nucleus |
| KCl | / | For EBSS | |
| L-glutamine | Sigma | 67513 | For tissue culture |
| Lipofectamine 2000 | Invitrogen | 11668-019 | For Cell Transfection |
| LSM880 Airyscan microscope | Zeiss | / | Confocal microscopy |
| MgCl2 | / | For PBS | |
| MgSO4.7H2O | / | For EBSS | |
| Mowiol mounting solution | Millipore | 475904 | for permanent mounting glass coverslips |
| NaCl | / | For EBSS | |
| NaH2PO4.2H2O | / | For EBSS | |
| NaHCO3 | / | For EBSS | |
| NuPAGE 3 to 8%, Tris-Acetate, 1.5 mm, Mini Protein Gels | Thermofisher Scientific | EA0378BOX | for Western Blotting |
| NuPAGE MES SDS Running Buffer (20x) | Life Tech | NP0002 | for Western Blotting |
| Opti-MEM I Reduced Serum Medium | Thermofisher Scientific | 31985062 | For Cell Transfection |
| Paraformaldehyde | Agar Scientific | R1026 | For fixing cells |
| pcDNA3.1-mCherry-3xFlag-ATG9A | home made | Construct which expresses tagged ATG9A | |
| Phosphate Buffered Saline (PBS) | / | For tissue culture | |
| PNGaseF | NEB | P0710S | To remove N-linked glycans |
| Poly-D-lysine hydrobromide mol wt 70,000-150,000 | Merck | P0899 | For coating coverslips |
| Rapid PNGase F enzyme | NEB | P07105 | To remove N-linked glycans |
| RFP-ATG9A | home made | Construct which expresses tagged ATG9A | |
| Triton X-100 | Thermofisher Scientific | 13454259 | Detergent for Cell lysis |
| Trypsin-EDTA solution | Sigma | T4049 | For tissue culture |
| Whatman Filter Paper | Merck | WHA1001325 | For Western Blot and IF |
| XCell SureLock Mini-Cell Electrophoresis System | Invitrogen | EI0001 | For Western Blotting |
| Zen Black edition | Zeiss | / | Used to operate the LSM 880 |
Referencias
- Young, A. R., et al. Starvation and ULK1-dependent cycling of mammalian Atg9 between the TGN and endosomes. Journal of Cell Science. 119, 3888-3900 (2006).
- Maeda, S., et al. lipid scrambling activity and role in autophagosome formation of ATG9A. Nature Structural & Molecular Biology. 27 (12), 1194-1201 (2020).
- Matoba, K., et al. Atg9 is a lipid scramblase that mediates autophagosomal membrane expansion. Nature Structural & Molecular Biology. 27 (12), 1185-1193 (2020).
- Mercer, T. J., Gubas, A., Tooze, S. A. A molecular perspective of mammalian autophagosome biogenesis. Journal of Biological Chemistry. 293 (15), 5386-5395 (2018).
- Yamamoto, H., Zhang, S., Mizushima, N. Autophagy genes in biology and disease. Nature Reviews: Genetics. , (2023).
- Sawa-Makarska, J., et al. Reconstitution of autophagosome nucleation defines Atg9 vesicles as seeds for membrane formation. Science. 369 (6508), (2020).
- Melia, T. J., Lystad, A. H., Simonsen, A. Autophagosome biogenesis: From membrane growth to closure. Journal of Cell Biology. 219 (6), 202002085 (2020).
- Orsi, A., et al. Dynamic and transient interactions of Atg9 with autophagosomes, but not membrane integration, are required for autophagy. Molecular Biology of the Cell. 23 (10), 1860-1873 (2012).
- Judith, D., et al. ATG9A shapes the forming autophagosome through Arfaptin 2 and phosphatidylinositol 4-kinase IIIbeta. Journal of Cell Biology. 218 (5), 1634-1652 (2019).
- Karanasios, E., et al. Autophagy initiation by ULK complex assembly on ER tubulovesicular regions marked by ATG9 vesicles. Nature Communications. 7, 12420 (2016).
- Koyama-Honda, I., Itakura, E., Fujiwara, T. K., Mizushima, N. Temporal analysis of recruitment of mammalian ATG proteins to the autophagosome formation site. Autophagy. 9 (10), 1491-1499 (2013).
- Guardia, C. M., et al. Structure of human ATG9A, the only transmembrane protein of the core autophagy machinery. Cell Reports. 31 (13), 107837 (2020).
- Soreng, K., et al. SNX18 regulates ATG9A trafficking from recycling endosomes by recruiting Dynamin-2. EMBO Reports. 19 (4), e44837 (2018).
- Staudt, C., Gilis, F., Boonen, M., Jadot, M. Molecular determinants that mediate the sorting of human ATG9A from the endoplasmic reticulum. Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1863 (9), 2299-2310 (2016).
- Knaevelsrud, H., Carlsson, S. R., Simonsen, A. SNX18 tubulates recycling endosomes for autophagosome biogenesis. Autophagy. 9 (10), 1639-1641 (2013).
- Takahashi, Y., et al. The Bif-1-Dynamin 2 membrane fission machinery regulates Atg9-containing vesicle generation at the Rab11-positive reservoirs. Oncotarget. 7 (15), 20855-20868 (2016).
- Imai, K., et al. Atg9A trafficking through the recycling endosomes is required for autophagosome formation. Journal of Cell Science. 129 (20), 3781-3791 (2016).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- van Vliet, A. R., et al. ATG9A and ATG2A form a heteromeric complex essential for autophagosome formation. Molecular Cell. 82 (22), 4324-4339 (2022).
- Kielkopf, C. L., Bauer, W., Urbatsch, I. L. Bradford assay for determining protein concentration. Cold Spring Harbor Protocols. 2020 (4), 102269 (2020).
- Kurien, B. T., Scofield, R. H. Validating antibody specificities for immunohistochemistry by protein blotting. Methods in Molecular Biology. 2593, 21-33 (2023).
- Lamb, C. A., et al. TBC1D14 regulates autophagy via the TRAPP complex and ATG9 traffic. EMBO Journal. 35 (3), 281-301 (2016).
- Longatti, A., et al. TBC1D14 regulates autophagosome formation via Rab11- and ULK1-positive recycling endosomes. Journal of Cell Biology. 197 (5), 659-675 (2012).
- Ravussin, A., Brech, A., Tooze, S. A., Stenmark, H. The phosphatidylinositol 3-phosphate-binding protein SNX4 controls ATG9A recycling and autophagy. Journal of Cell Science. 134 (3), (2021).
- DesMarais, V., Eddy, R. J., Sharma, V. P., Stone, O., Condeelis, J. S. Optimizing leading edge F-actin labeling using multiple actin probes, fixation methods and imaging modalities. BioTechniques. 66 (3), 113-119 (2019).
- Ennis, H. L., Lubin, M. Cycloheximide: Aspects of inhibition of protein synthesis in mammalian cells. Science. 146 (3650), 1474-1476 (1964).
- Webber, J. L., Tooze, S. A. Coordinated regulation of autophagy by p38alpha MAPK through mAtg9 and p38IP. EMBO Journal. 29 (1), 27-40 (2010).
- Claude-Taupin, A., et al. ATG9A protects the plasma membrane from programmed and incidental permeabilization. Nature Cell Biology. 23 (8), 846-858 (2021).
- Aaron, J. S., Taylor, A. B., Chew, T. L. Image co-localization - Co-occurrence versus correlation. Journal of Cell Science. 131 (3), 211847 (2018).
- D'Antuono, R., Nechyporuk-Zloy, V. Basic digital image acquisition, design, processing, analysis, management, and presentation. Principles of Light Microscopy: From Basic to Advanced. , 77-104 (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados