Method Article
Imaging di ATG9A, una proteina di membrana multi-spanning
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo descrive vari metodi che possono aiutare nello studio della biologia di ATG9A, tra cui l'immunofluorescenza seguita da analisi delle immagini, considerazioni sulla sovraespressione transitoria e lo studio dello stato di glicosilazione di ATG9A utilizzando il western blot.
Abstract
L'autofagia è un percorso altamente conservato che la cellula utilizza per mantenere l'omeostasi, degradare gli organelli danneggiati, combattere gli agenti patogeni invasori e sopravvivere a condizioni patologiche. Un insieme di proteine, chiamate proteine ATG, comprende il meccanismo principale dell'autofagia e lavora insieme in una gerarchia definita. Gli studi degli ultimi anni hanno migliorato la nostra conoscenza del percorso dell'autofagia. Più recentemente, è stato proposto che le vescicole ATG9A siano al centro dell'autofagia, in quanto controllano la rapida sintesi de novo di un organello chiamato fagoforo. Lo studio di ATG9A si è dimostrato impegnativo, poiché ATG9A è una proteina transmembrana ed è presente in diversi compartimenti di membrana. In quanto tale, comprendere il suo traffico è un elemento importante per comprendere l'autofagia. Qui vengono presentati metodi dettagliati che possono essere utilizzati per studiare ATG9A e, in particolare, la sua localizzazione utilizzando tecniche di immunofluorescenza, che possono essere valutate e quantificate. Vengono anche affrontate le insidie della sovraespressione transitoria. La corretta caratterizzazione della funzione di ATG9A e la standardizzazione delle tecniche per analizzare il suo traffico sono cruciali per caratterizzare ulteriormente gli eventi che regolano l'inizio dell'autofagia.
Introduzione
ATG9A è l'unica proteina transmembrana del meccanismo di autofagia del nucleo ed è trafficata tra il Golgi e un compartimento citosolico della vescicola ATG9A, transitando attraverso il compartimento endosomiale1. Essendo stato a lungo enigmatico, ATG9A è stato recentemente descritto per funzionare come uno scramblasi lipidico, in quanto equilibra i lipidi attraverso i doppi stratidi membrana 2,3. È ormai chiaro che ATG9A risiede in cima alla gerarchia nella formazione degli autofagosomi, e il suo studio è, quindi, vitale per comprendere l'autofagia 4,5. Come tali, le vescicole ATG9A sono state recentemente proposte come il "seme" dell'autofagosoma 6,7. Tuttavia, studi precedenti hanno dimostrato che ATG9A interagisce solo transitoriamente con l'autofagosoma in formazione in diverse fasi della sua maturazione e non si integra nella membrana autofagica 6,8,9,10,11. Pertanto, sono necessarie ulteriori indagini per svelare completamente il ruolo e le potenziali funzioni multiple di ATG9A nella formazione degli autofagosomi. Tuttavia, la discrepanza tra i modelli attuali e i dati precedenti può essere risolta solo attraverso esperimenti mirati che affrontino il traffico di ATG9A utilizzando approcci quantitativi convalidati e marcatori intracellulari.
Ci sono vari strumenti in uso per studiare ATG9A, ognuno con vantaggi e svantaggi, e l'uso di questi strumenti è complicato dalla struttura di ATG9A, dalla sua funzione molecolare e dal traffico cellulare 2,8,12. ATG9A forma un omotrimero, è glicosilato, e viene trafficato in tutta la cellula verso compartimenti come il Golgi, gli endosomi e la membrana plasmatica13,14. Dato il suo complesso itinerario, ci sono diverse sfide nell'interpretare letture come la dispersione di ATG9A dal Golgi su trattamenti o stimoli specifici (come la fame di nutrienti e siero). ATG9A è estremamente dinamico in termini di traffico vescicolare; infatti, le vescicole contenenti ATG9A sono state definite come il compartimento ATG9A nel contesto dell'autofagia indotta dalla fame. Il compartimento ATG9A, formato da queste vescicole dinamiche, interagisce transitoriamente con diversi organelli intracellulari 8,15,16,17. Le tecniche qui descritte, tra cui l'immunofluorescenza, l'imaging dal vivo e i saggi di glicosilazione, dovrebbero aiutare nel rilevamento e nella comprensione della biologia di ATG9A. In particolare, gli approcci descritti in questo articolo aiuteranno a rispondere a domande sulla localizzazione in specifici compartimenti cellulari e sulle interazioni con specifici partner proteici e/o compartimenti di membrana. Poiché il dominio centrale conservato idrofobico ATG9A (dominio PFAM PF04109) ha una topologia unica e ATG9A si muove tra diversi compartimenti di membrana, i ricercatori dovrebbero essere consapevoli di alcune insidie e artefatti quando sovraesprimono transitoriamente ATG9A, tra cui, ma non solo, la ritenzione del reticolo endoplasmatico (ER). Altri possibili problemi possono sorgere a causa di un errato ripiegamento della proteina, di un'aggregazione artefatta in condizioni di crescita normali o di un rilevamento insufficiente del compartimento vescicolare a causa di protocolli di permeabilizzazione non ottimali per l'immunofluorescenza.
Quando si esegue l'imaging endogeno di ATG9A, è necessario prestare attenzione nella preparazione del campione e nell'acquisizione dell'immagine per garantire la qualità della successiva analisi quantitativa e la corretta interpretazione dei dati. La combinazione delle tecniche descritte in questo articolo con approcci biochimici standard (come l'immunoprecipitazione o gli esperimenti di pull-down non descritti qui) dovrebbe migliorare la nostra comprensione della funzione di ATG9A. Questo toolkit sperimentale ha lo scopo di aiutare i nuovi ricercatori a navigare in alcuni dei saggi necessari per determinare la funzione di ATG9A nel loro sistema biologico.
Protocollo
Tutti i reagenti utilizzati in questo studio sono disponibili in commercio, ad eccezione dei costrutti di DNA ATG9A e dell'anticorpo STO-215 fatto in casa (vedi Tabella dei materiali), che sono disponibili su richiesta. Gli strumenti di analisi qui descritti si basano su software open source (FIJI/ImageJ)18.
1. Colture cellulari
- Mantenere HEK293A cellule in un matraccio trattato con coltura tissutale T150 all'80%-90% di confluenza in DMEM ad alto contenuto di glucosio (Dulbecco's Modified Eagle Medium) integrato con il 10% di FBS (siero fetale bovino) e 4 mM di L-glutammina (vedere Tabella dei materiali). Incubare le cellule a 37 °C in un incubatore per colture tissutali umidificato al 10% di CO2 .
NOTA: HEK293A cellule sono utilizzate nel presente studio, in quanto hanno una robusta risposta canonica di autofagia alla fame di aminoacidi, che è, in particolare, rilevata da un aumento di LC3 1,9,19 lipidato associato alla membrana e sono adatte per l'imaging. - Far passare le cellule aspirando il terreno dal matraccio utilizzando una pipetta sierologica. Lavare le cellule una volta con 15 ml di 1x PBS (soluzione salina tamponata con fosfato) o una soluzione simile prima di aggiungere 2 ml di soluzione di tripsina-EDTA (acido etilendiamminotetraacetico) per staccare le cellule. Raccogliere le cellule staccate con 8 mL di DMEM e riseminare un certo numero di cellule in modo che l'80%-90% di confluenza sia raggiunto dopo 2 giorni per gli esperimenti qui descritti.
2. Colorazione endogena di ATG9A
- Seminare le cellule HEK293A (o cellule a scelta) su vetrini coprioggetti sterili n. 1,5 posti in una piastra da 24 pozzetti in modo che siano confluenti al ~80% il giorno successivo. In questo modo si ottengono in genere 7 x 104 cellule/pozzetto in 500 μL di DMEM.
NOTA: Per le cellule HEK293A o altre linee cellulari debolmente aderenti, i vetrini coprioggetti devono essere rivestiti con poli-D-lisina a 0,1 mg/mL in acqua deionizzata. Aggiungere 500 μL di poli-D-lisina (vedi Tabella dei materiali) sulla parte superiore dei vetrini coprioggetti posti nei pozzetti per 10 minuti a temperatura ambiente (RT), seguiti da tre lavaggi con acqua deionizzata e un ultimo lavaggio in DMEM. Procedere alla semina delle cellule dopo il rivestimento e fare attenzione che siano distribuite uniformemente nella piastra di coltura. - Trattare le cellule in base alle specifiche condizioni sperimentali (ad esempio, morire di fame per 2 ore in EBSS [soluzione salina bilanciata di Earle]).
NOTA: La composizione EBSS è 1 g/L D-glucosio, 6,8 g/L NaCl, 0,4 g/L KCl, 0,151 g/L CaCl 2,2H 2 O, 0,2 g/L mM MgSO 4,7H 2 O, 0,124 g/L NaH 2 HPO4,2H 2 O e 2,2 g/L NaHCO3 (vedi Tabella dei materiali) disciolti in acqua distillata. - Aspirare il terreno e sostituirlo con cura con 500 μL di soluzione di formaldeide al 4% in PBS integrata con 0,1 mM di CaCl 2 e 0,1 mM di MgCl2 per 20 minuti a RT per fissare le cellule.
- Aspirare la soluzione di formaldeide al 4% da ciascun pozzetto, sostituendola con 500 μL di PBS. Ripetere questa fase di lavaggio tre volte. Non lasciare che i vetrini coprioggetti si asciughino o rimangano senza liquidi.
- Estinguere i gruppi di aldeidi libere utilizzando 500 μL di soluzione 50 mM di NH4Cl in PBS per 10 minuti a RT.
- Aspirare il PBS e sostituirlo con 500 μL di una soluzione di digitonina da 50 μg/mL in PBS (soluzione madre 1 mg/mL in acqua deionizzata, vedere la tabella dei materiali) per 5 minuti a RT per permeabilizzare le cellule.
- Aspirare la soluzione di digitonina da ciascun pozzetto e sostituirla con 500 μL di PBS. Ripetere questa fase di lavaggio tre volte.
- Aspirare il PBS da ciascun pozzetto e sostituirlo con 500 μL di soluzione bloccante (5% di BSA [albumina sierica bovina] diluita in PBS) per 30 minuti a RT.
- Aspirare la soluzione bloccante e sostituirla con 500 μL di PBS.
- Usando una pinzetta, raccogliere i vetrini coprioggetti dal pozzetto, rimuovere con cautela la soluzione di PBS in eccesso utilizzando salviette sottili o carta da filtro di cellulosa e adagiare delicatamente ciascun vetrino coprioggetti, con il lato cellulare rivolto verso il basso, su una goccia da 50 μL di soluzione anticorpale primaria (ad esempio, criceto armeno 14F2 diluito a 0,9 μg/mL in soluzione BSA/PBS all'1%, vedere la tabella dei materiali). Incubare in camera umidificata per 1 h a RT.
NOTA: Per una facile manipolazione, le gocce di soluzione anticorpale possono essere posizionate su un foglio di pellicola termoplastica autosigillante (vedere la tabella dei materiali) all'interno di un altro contenitore invece che direttamente su una superficie solida. - Raccogliere i vetrini coprioggetti con una pinzetta e, dopo aver drenato delicatamente la soluzione anticorpale primaria in eccesso utilizzando salviette per tessuti sottili, riposizionare i vetrini coprioggetti (con il lato cellulare rivolto verso l'alto) nella piastra a 24 pozzetti e lavarli tre volte con PBS.
- Ripetere il passaggio 2.10. Usando una pinzetta, raccogliere i vetrini coprioggetti da ciascun pozzetto, drenando accuratamente il PBS in eccesso con salviette sottili per fazzoletti, e adagiare delicatamente ciascun vetrino coprioggetti (lato cellulare rivolto verso il basso) su una goccia di 50 μL di soluzione anticorpale secondaria diluita 1:1.000 in soluzione BSA/PBS all'1% (ad esempio, Cy3 Goat Anti-Armenian Hamster IgG, che ha una minima crossreattività con bovini, umani, proteine sieriche di topo, coniglio e ratto, vedi Tabella dei materiali). Incubare in camera umidificata per 1 h a RT.
NOTA: Opzionale: è possibile utilizzare un marcatore del citoscheletro in aggiunta all'anticorpo secondario per la successiva analisi dell'immagine (ad esempio, Alexa Fluor 647 Phalloidin diluito 1:1.000, vedere la tabella dei materiali). Per una facile manipolazione, le gocce di soluzione anticorpale possono essere posizionate su un foglio di pellicola sigillante all'interno di un altro contenitore invece che direttamente su una superficie solida. - Raccogliere i vetrini coprioggetti con una pinzetta e, dopo aver drenato la soluzione anticorpale secondaria in eccesso, posizionare i vetrini coprioggetti nella piastra a 24 pozzetti (lato cellulare rivolto verso l'alto) e lavarli tre volte con PBS da 500 μL.
- Usando una pinzetta, raccogliere i vetrini coprioggetti, drenando accuratamente il PBS in eccesso con salviette di tessuto sottile, e adagiare delicatamente ciascun vetrino coprioggetti (lato cellulare rivolto verso il basso) su una goccia da 50 μL di soluzione di Hoechst 1:4.000 (Hoechst 33342) in PBS. Incubare in una camera umida per 5 minuti a RT.
NOTA: Per una facile manipolazione, le gocce di soluzione anticorpale possono essere posizionate su un foglio di pellicola sigillante all'interno di un altro contenitore invece che direttamente su una superficie solida. - Raccogliere i vetrini coprioggetti con una pinzetta e, dopo aver drenato delicatamente la soluzione di Hoechst in eccesso con salviette per tessuti sottili, riposizionare i vetrini coprioggetti nella piastra a 24 pozzetti e lavare tre volte con PBS e una volta con acqua deionizzata (500 μL per lavaggio).
- Drenare accuratamente l'acqua deionizzata in eccesso utilizzando salviette sottili per fazzoletti e adagiare delicatamente ciascun vetrino coprioggetto (lato cellulare rivolto verso il basso) su una goccia da 10-20 μL di soluzione di montaggio (vedere Tabella dei materiali) individuata su un vetrino da microscopio per l'immunofluorescenza, evitando la formazione di bolle d'aria.
NOTA: Il mezzo di montaggio utilizzato qui ha lo stesso indice di rifrazione dell'olio da immersione e si indurisce dopo alcune ore a RT o durante la notte a 4 °C. È possibile utilizzare soluzioni di montaggio non indurenti, ma è necessario prestare attenzione a sigillare il vetrino coprioggetto con smalto per unghie. - Rimuovere la soluzione di montaggio in eccesso mediante aspirazione e lasciare asciugare i campioni mentre si trovano in piano a RT per una notte al buio, in un supporto per vetrini o coperti con un foglio di alluminio.
3. Acquisizione delle immagini
- Accendere il microscopio confocale. Aprire il software di imaging (vedere Tabella dei materiali) per avviare la configurazione dell'acquisizione delle immagini.
- Nella scheda Funzione , selezionare l'obiettivo Plan-Apochromat 63x/1.4 Oil DIC M27 per acquisire le immagini da analizzare.
- Nella scheda Funzione di acquisizione , accendere i laser appropriati nella zona Laser del pannello di controllo (Argon, Diode-405-30, DPSS 561-10 e HeNe633).
- Nell'area Imaging Setup (Impostazione imaging ) del Pannello di controllo, creare quattro tracce, ciascuna delle quali corrisponde a un canale, per eseguire l'acquisizione sequenziale.
- Impostare la risoluzione appropriata delle immagini nella finestra Modalità di acquisizione . Impostare la risoluzione su 1.024 x 1.024 (Dimensione fotogramma) e selezionare una profondità di bit di 16 bit.
- Per ogni canale, regolare la potenza e il guadagno del laser per ottenere un buon segnale senza pixel saturi, che ostacolerebbero l'analisi dell'intensità dell'immagine. Utilizzando il pulsante Live, impostare il livello di uscita del laser e il guadagno (Master).
NOTA: L'uso dell'opzione dell'indicatore di portata è consigliato per il rilevamento di pixel saturi. Mantenere la potenza del laser tra l'1% e il 10% e il guadagno (master) al di sotto di 850 per evitare il rumore di fondo. - Nella zona Canali del Pannello di controllo, impostare la stessa apertura stenopeica per ciascun canale, considerando 1 Airy Unit (AU) per il canale con la lunghezza d'onda più alta.
- Acquisisci 10 campi casuali contenenti quantità simili di celle (20-30 celle per campo). L'acquisizione di 100-200 cellule per condizione fornirà una buona potenza per la successiva analisi dell'immagine.
4. Analisi delle immagini della dispersione di ATG9A
- Scaricare il software FIJI da Internet (vedere la Tabella dei materiali). Apri l'immagine con FIJI cliccando su Plugins > Bio-Formats > BioFormats Importer.
- Fare clic su Analizza > Imposta misurazioni e selezionare il valore di grigio medio nella finestra delle misurazioni per l'analisi dell'intensità.
- Fare clic su Immagine > Colore > Dividi canali e separare i canali in tre immagini (marcatore di Golgi, citoscheletro, segnale ATG9A).
- Vai su Immagine > Regola > Soglia e definisci una soglia nel canale corrispondente al marcatore di Golgi.
- Fare clic su Modifica > selezione > Crea selezione e creare una selezione dall'immagine binaria.
- Vai a Modifica > selezione > Aggiungi a Manager > Rinomina (Golgi), salva la selezione in Gestione ROI e rinominala in Golgi.
- Fare clic su Immagine > Regola > soglia e definire una soglia nel canale corrispondente al marcatore del citoscheletro per definire il contorno della cella.
- Vai a Modifica > selezione > Crea selezione e crea una selezione dall'immagine binaria.
- Fare clic su Modifica > selezione > Aggiungi a Manager > Rinomina (totale), salvare la selezione nel gestore ROI e rinominarla Totale.
- Selezionare l'immagine corrispondente alla colorazione ATG9A.
- In Analizza > Misura, applica il ROI Golgi facendo clic su di esso e misura l'intensità della regione del Golgi .
- Fare clic su Analizza > Misura, applicare il ROI Totale facendo clic su di esso e misurare l'intensità della regione Totale.
- Ripetere la procedura in tutte le immagini, salvare i risultati come file .csv ed elaborare i dati utilizzando la seguente formula:
Tasso di dispersione ATG9A = Intensità del Golgi/Intensità totale
5. Imaging di cellule vive di costrutti ATG9A
- Seminare HEK293A cellule (o cellule a scelta) in 2 mL di terreno in un piatto di coltura tissutale da 60 mm in modo che raggiungano una confluenza del ~65%-70% il giorno successivo; Questo in genere dà 1 x 10 6-2 x 106 celle.
- Il giorno seguente, preparare le miscele di Lipofectamina:DNA (o un reagente alternativo adatto per la trasfezione del DNA, vedere la tabella dei materiali) come segue:
- A seconda dell'efficienza di espressione del costrutto, diluire 0,5-2 μg di DNA plasmidico in 100 μL di un terreno adatto privo di siero e mescolare delicatamente la soluzione pipettando su e giù. Incubare la miscela per 5 minuti a RT.
NOTA: Il costrutto di DNA plasmidico è specifico per l'esperimento. I ricercatori possono clonare da soli o ottenere dagli autori su richiesta. - Diluire il reagente di trasfezione Lipofectamine 2000 in un rapporto di 3:1 Lipofetectamina:DNA in 100 μL di un terreno iero idoneo e mescolare delicatamente la soluzione pipettando su e giù. Incubare questa miscela per 5 minuti a RT.
- Miscelare entrambe le soluzioni pipettando delicatamente su e giù e incubando per 20 minuti a RT.
- A seconda dell'efficienza di espressione del costrutto, diluire 0,5-2 μg di DNA plasmidico in 100 μL di un terreno adatto privo di siero e mescolare delicatamente la soluzione pipettando su e giù. Incubare la miscela per 5 minuti a RT.
- Aggiungere la miscela Lipofectamine:DNA a ciascuna piastra di coltura cellulare contenente 4 ml di terreno di coltura e far oscillare delicatamente la piastra avanti e indietro per distribuire uniformemente la miscela. Incubare le cellule a 37 °C in un incubatore per colture cellulari umidificato al 10% di CO2 .
- Sostituire il terreno con terreno di coltura fresco 4 ore dopo la trasfezione e incubare le cellule a 37 °C in un incubatore per colture cellulari umidificato al 10% di CO2 per una notte.
- Il giorno successivo, tripsinizzare, contare e riseminare le cellule su piastre di coltura adatte alla microscopia di cellule vive (piastre di coltura con vetrino coprioggetti n. 1,5, vedere Tabella dei materiali). Seminare 0,4 x 10 6-0,7 x 106 cellule sulle piastre per raggiungere ~60%-75% di confluenza sul vetrino coprioggetto al momento dell'imaging.
NOTA: Per le celle HEK293A si consiglia il rivestimento con poli-D-lisina, come spiegato al punto 2.1. - Il giorno successivo (48 ore dopo la trasfezione), eseguire l'imaging delle cellule.
6. Studio dello stato di glicosilazione di ATG9A
- Seminare le cellule HEK293A (o le cellule a scelta) in un piatto di coltura tissutale da 10 cm in modo che raggiungano ~80% di confluenza il giorno successivo se si prevede di studiare ATG9A endogeno, che in genere fornisce 1,5 x 10 6-2,5 x 106 cellule in 10 mL. In alternativa, seminare le cellule in modo che raggiungano ~65%-70% di confluenza se si trasfettano i costrutti ATG9A, che in genere danno 1 x 10 6-1,5 x 106 cellule in 10 mL.
- Trattare le cellule con cicloesamemide (CHX) 100 μg/mL (CHX, vedere la tabella dei materiali) o con il veicolo il giorno successivo. Incubare per 24 ore (o fino a un punto temporale desiderato).
- Rimuovere le cellule dall'incubatrice, aspirare il terreno e metterle sul ghiaccio. Sostituiscilo con 5 ml di PBS 1x ghiacciato. Staccare fisicamente le cellule dalla capsula utilizzando un raschietto per cellule e pipettare la soluzione di cellule PBS in una provetta a fondo conico da 15 mL. Centrifugare a 800 x g per 5 minuti a 4 °C per pellettare le cellule.
- Aspirare il surnatante e risospendere le cellule in ~100 μL (a seconda delle dimensioni del pellet cellulare) di tampone di lisi TNTE ghiacciato (1% Triton, 150 mM NaCl, 20 mM Tris-HCl, pH 7,4, 0,5 mM EDTA) integrato con compresse cocktail di inibitori della proteasi (vedere Tabella dei materiali) e trasferire in una provetta per microcentrifuga da 1,5 mL.
- Incubare il lisato su ghiaccio per 15 min. Successivamente, centrifugare il lisato a 20.000 x g per 10 minuti a 4 °C per sedimentare i nuclei e i detriti insolubili e trasferire il surnatante in una nuova provetta per microcentrifuga.
- Quantificare la concentrazione proteica del lisato utilizzando il metodo Bradford20 e uno spettrofotometro in grado di misurare a una lunghezza d'onda di 595 nm.
- Normalizzare le quantità di proteine e combinare 16 μL di lisato (contenente non più di 100 μg di proteine in totale) con 5 tamponi PNGasi F (peptide:N-glicosidasi F) secondo le istruzioni del produttore prima di aggiungere 1 μL di enzima PNGasi F (vedere la tabella dei materiali). Incubare secondo le istruzioni del produttore di PNGase F.
- Aggiungere un volume di tampone campione Laemmli 3x per ottenere una concentrazione 1x e incubare a 65 °C per 5 minuti prima del caricamento per elettroforesi utilizzando un gel di Tris-acetato per massimizzare la separazione delle proteine. Trasferire le proteine dal gel a una membrana adatta (ad esempio, PVDF [difluoruro di polivinilidene]) utilizzando protocolli standard di western blot21 (vedere la tabella dei materiali).
NOTA: L'ebollizione dei campioni a 95 °C causerà l'aggregazione di ATG9A, riducendo così il rilevamento di ATG9A. - Eseguire il western blot utilizzando anticorpi specifici per ATG9A (anticorpo STO-215, prodotto internamente1) (vedere Tabella dei materiali). Lasciare intatta la sezione della membrana a peso molecolare più elevato per visualizzare le specie a peso molecolare ATG9A più elevato.
Risultati
ATG9A è una proteina transmembrana associata a diversi compartimenti di membrana intracellulare 8,17,22,23,24. In condizioni basali, ATG9A è localizzato principalmente nella rete trans-Golgi (TGN), come indicato dall'immunofluorescenza della proteina endogena e dalle sovrapposizioni con GM130, un marcatore cis-Golgi (Figura 1A), così come in piccole vescicole che si sovrappongono parzialmente al compartimento di riciclaggio endocitico (ERC)23. La localizzazione di ATG9A sul Golgi può essere rilevata utilizzando diversi protocolli di immunofluorescenza. Tuttavia, la frazione vescicolare di ATG9A, così come il suo cambiamento di localizzazione, in particolare l'aumento del pool vescicolare, in risposta a stimoli specifici come la carenza di nutrienti e siero, può essere molto variabile in intensità e difficile da visualizzare con approcci di imaging convenzionali. Il rapporto tra ATG9A localizzato al Golgi e ATG9A localizzato in una frazione vescicolare è definito tasso di dispersione di ATG9A. Per rilevare i cambiamenti nel tasso di dispersione di ATG9A, ad esempio durante il trattamento con EBSS, che viene utilizzato per esaurire sia il siero che gli amminoacidi, un marcatore di Golgi come GM130 o TGN46 e un marcatore del citoscheletro come la falloidina, che colora il contorno cellulare25, sono utili per quantificare prontamente la dispersione di ATG9A (Figura 1B). È importante sottolineare che l'analisi del rapporto medio di fluorescenza può essere interpretata solo come una misura comparativa tra le condizioni piuttosto che come un tasso fisso di dispersione. Il rapporto tra i compartimenti dipende fortemente da fattori biologici e non biologici come la linea cellulare utilizzata, la qualità della colorazione o i metodi di soglia applicati (Figura 1B). Per questo motivo, il ricercatore ha la necessità di mettere a punto una pipeline che sia in grado di rilevare l'arricchimento di Golgi di ATG9A nelle loro specifiche condizioni sperimentali e quindi estendere l'analisi con gli stessi parametri a tutte le immagini del set da analizzare. Le immagini binarie rappresentative e le aree selezionate per l'analisi della fluorescenza media ATG9A sono mostrate come guida nella Figura 1B.
ATG9A ospita diversi domini transmembrana affiancati da due domini N- e C-terminali relativamente flessibili e non strutturati, di cui la sequenza C-terminale comprende quasi la metà della proteina12. È importante sottolineare che il modello di localizzazione di ATG9A sovraespresso può essere influenzato da quale estremità proteica è marcata (Figura 2A). In particolare, quando si utilizzano sistemi di espressione transitoria e si marca ATG9A direttamente sul suo N-terminale con un tag fluorescente (ad esempio, eGFP, mRFP o derivati), la sua localizzazione del Golgi può essere parzialmente compromessa, con un minore arricchimento osservato in condizioni basali (cioè alimentate), mentre le vescicole ATG9A sono ancora prontamente visibili (Figura 2A). L'etichettatura di ATG9A sul suo C-terminale sembra indurre leggermente cluster positivi GFP più grandi che potrebbero essere aggregati. Infine, una versione monomerica di mRFP-ATG9A mostra anche cluster fluorescenti simili di vescicole e una piccola colorazione di Golgi nelle cellule sovraesprimenti (Figura 2A).
ATG9A si ripiega nella membrana dell'ER prima di essere trasportato alle vescicole del Golgi e dell'ATG9A. Durante la sua permanenza nel reticolo endoplasmatico, ATG9A viene modificato dai glicani legati all'N sull'Asparagina 99, e poi, dopo aver raggiunto il Golgi, acquisisce glicani complessi e maturi legati all'N 1,14. Questa modificazione per glicosilazione può essere rilevata attraverso il western blot dalla comparsa di una doppia banda14. Coerentemente con la sua localizzazione intracellulare, la maggior parte degli ATG9A endogeni ospita glicani complessi legati all'N e, pertanto, la banda di peso molecolare più elevato è predominante, con una debole banda di peso molecolare inferiore visibile anche (Figura 2B). La presenza di una doppia banda è più facilmente osservabile quando si utilizzano gel di Tris-acetato per migliorare la risoluzione di proteine a peso molecolare più elevato (Figura 2B, controllo, t = 0). Quando la proteina endogena viene sottoposta al trattamento con PNGasi F (peptide:N-glicosidasi F), che rimuove la maggior parte dei glicani complessi legati all'N, la proteina funziona come una singola banda (Figura 2B, PNGasi F, t = 0). Pertanto, lo stato di glicosilazione legato all'N di ATG9A può essere utilizzato come proxy per monitorare l'uscita di ATG9A dall'ER al Golgi, che si riflette nel rapporto relativo tra le due bande.
Quando la trasfezione di mRFP-ATG9A si costruisce in modo transitorio, la proteina sovraespressa si accumula inizialmente nel reticolo endoplasmatico, potenzialmente perché il meccanismo di traffico non è in grado di ripiegare e trafficare tutto l'ATG9A e la banda di peso molecolare inferiore è predominante (Figura 2C, controllo t = 0). In particolare, dopo 24 ore di espressione di mRFP-ATG9A, c'è una distribuzione approssimativamente uguale tra le bande superiore e inferiore, suggerendo che il pool di mRFP-ATG9A si sta muovendo nel Golgi (Figura 2C, Controllo, t = 24). Se le cellule sono trattate con cicloesimide (CHX), che blocca la sintesi proteica de novo 26, il ripiegamento e l'uscita di ATG9A dall'ER possono essere chiariti. Poiché la proteina endogena è ripiegata, glicosilata e residente nel Golgi, il trattamento con CHX non altera significativamente il rapporto tra le bande di peso molecolare inferiore e superiore (Figura 2B, controllo). Tuttavia, utilizzando l'espressione transitoria di mRFP-ATG9A, il trattamento con CHX promuove l'accumulo della banda di peso molecolare più elevato (Figura 2C, controllo, CHX t = 24). La banda mRFP-ATG9A sovraespressa a peso molecolare più elevato collassa nella banda inferiore dopo il trattamento con PNGasi F (Figura 2C, PNGasi F, t = 24). Questi dati mostrano che la proteina endogena acquisisce rapidamente glicani maturi, come si evince dalla predominanza della banda di peso molecolare più elevato, e l'inseguimento di CHX non influisce sul rapporto tra le doppie bande (Figura 2B). Nel caso di mRFP-ATG9A sovraespresso transitoriamente, il trattamento con CHX induce l'accumulo della banda superiore, indicando che i glicani più maturi vengono acquisiti quando il pool di ER si ripiega ed esce dal ER verso il Golgi (Figura 2C).
L'aggiunta di un linker tra la sequenza ATG9A e i tag fluorescenti può essere utile per promuovere una localizzazione e un traffico più fisiologici della proteina. La fusione di una sequenza 3x-FLAG (24 amminoacidi) tra un fluoroforo N-terminale e ATG9A aiuta la proteina sovraespressa a comportarsi in modo simile a quella endogena (Figura 3). Infatti, mCherry-3xFLAG-ATG9A sovraespresso colocalizza con il marcatore di Golgi GM130 in condizioni di alimentazione (Figura 3A). È importante sottolineare che questa localizzazione e il compartimento vescicolare di ATG9A sono preservati nel tempo, consentendo lo studio spazio-temporale del traffico di ATG9A (Figura 3B).
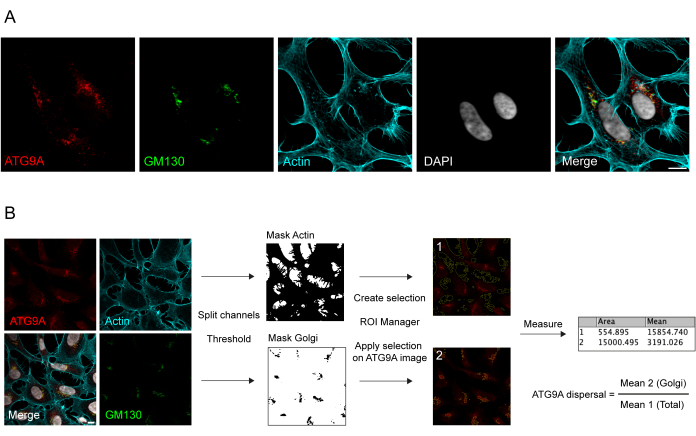
Figura 1: Analisi dell'immagine della localizzazione endogena di ATG9A. (A) Immagine rappresentativa in immunofluorescenza di ATG9A endogeno (rosso), GM130 come marcatore di Golgi (verde) e falloidina per visualizzare il citoscheletro di actina (ciano). Barra della scala = 10 μm. (B) Flusso di lavoro dell'analisi dell'immagine per determinare la frazione di ATG9A endogena che si localizza nell'area del Golgi. Barra della scala = 10 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
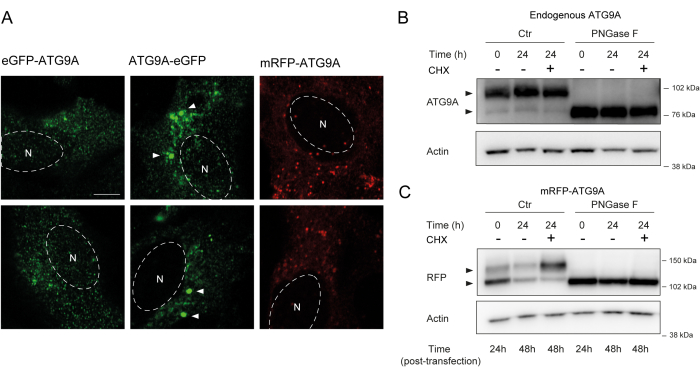
Figura 2: Analisi di costrutti ATG9A marcati in fluorescenza mediante localizzazione e glicosilazione. (A) L'ATG9A marcato con eGFP N-terminale è meno localizzato al Golgi e risiede principalmente nelle vescicole. L'ATG9A marcato terminalmente con eGFP C mostra aggregati all'interno della cella (alcuni esempi sono contrassegnati da punte di freccia bianche; eGFP-ATG9A e ATG9A-eGFP sono in verde). L'ATG9A marcato con mRFP N-terminale è meno localizzato al Golgi e risiede principalmente nelle vescicole. N indica la posizione approssimativa del nucleo cellulare e l'mRFP-ATG9A è in rosso. Barra della scala = 5 μm. (B) L'ATG9A endogeno appare come due bande quando analizzato mediante western blot (punte di freccia): una banda superiore (glicani N-legati complessi) e una banda inferiore (nessun glicani N-legato maturo). Il trattamento con cicloesameide (CHX) non influisce sul rapporto tra la banda superiore e quella inferiore. Il trattamento con PNGasi F provoca la scomparsa della banda superiore. (C) Dopo la trasfezione transitoria di ATG9A marcato con mRFP in cellule HEK293A, due bande prominenti sono visibili sul western blot (punte di freccia). Il trattamento con PNGasi F provoca la scomparsa della banda superiore. Il trattamento con CHX dopo la trasfezione porta ad un aumento della glicosilazione poiché il pool di ATG9A trasfettato viene trasferito dal reticolo endoplasmatico al Golgi. Fare clic qui per visualizzare una versione più grande di questa figura.
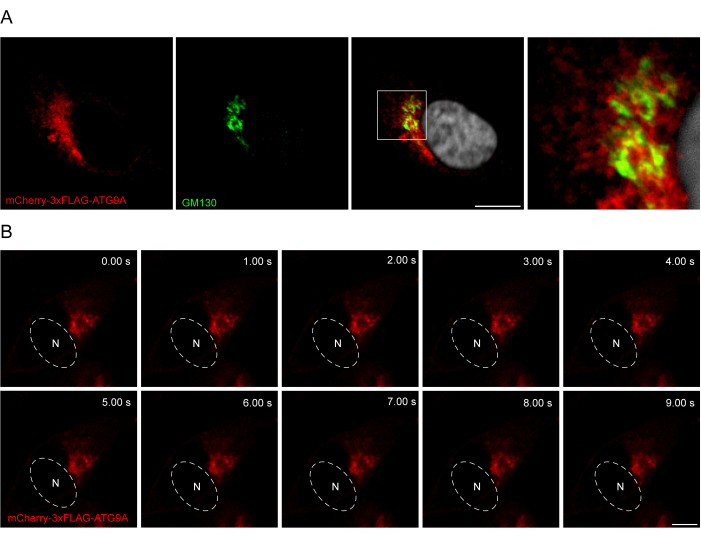
Figura 3: Analisi della localizzazione di mCherry-3xFLAG-ATG9A mediante immunofluorescenza e live imaging. (A) Esperimenti di immunofluorescenza di cellule HEK293A transientemente sovraesprimenti mCherry-3xFLAG-ATG9A e colorate con il marcatore di Golgi GM130. Barra della scala = 10 μm. Il mCherry-3xFLAG-ATG9A è in rosso e l'indicatore GM130 Golgi è in verde. (B) Montaggio da esperimenti di imaging dal vivo in cellule HEK293A che sovraesprimono transitoriamente mCherry-3xFLAG-ATG9A. N indica la posizione approssimativa del nucleo. Intervallo di tempo = 1 fps. Barra della scala = 10 μm. Il mCherry-3xFLAG-ATG9A è in rosso. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
Questo studio illustra i vari strumenti che possono essere utilizzati per studiare la localizzazione di ATG9A. In primo luogo, questo studio descrive come ATG9A può essere visualizzato mediante immunofluorescenza e come questo può essere quantificato. In secondo luogo, vengono confrontate le strategie che possono essere utilizzate per etichettare ATG9A con un marcatore fluorescente per la visualizzazione in cellule fisse o vive. Infine, questo lavoro descrive come studiare e utilizzare lo stato di glicosilazione di ATG9A per determinare se ATG9A è uscito dall'ER e ha trafficato attraverso il Golgi.
Per quanto riguarda la caratterizzazione della localizzazione endogena di ATG9A mediante immunofluorescenza, è necessario prestare attenzione ai metodi di fissazione e permeabilizzazione impiegati per l'esperimento. Secondo le procedure standard qui descritte, la fissazione della paraformaldeide accoppiata alla permeabilizzazione della digitonina sono buone condizioni per visualizzare sia le vescicole ATG9A associate al Golgi che le vescicole ATG9A-positive7. Insieme alla fissazione e alla permeabilizzazione, anche i tempi di incubazione con la soluzione anticorpale primaria sono fondamentali. Abbiamo osservato, ma non documentato, che concentrazioni più elevate di soluzione anticorpale primaria e tempi di incubazione più lunghi possono portare a un aumento fuorviante della colorazione di Golgi di ATG9A, che alla fine compromette la rilevazione della ridistribuzione di ATG9A ad altri compartimenti di membrana. Inoltre, poiché ATG9A è presente in molti compartimenti intracellulari 1,13,17,22,23,24,27,28, è importante utilizzare specifici marcatori di membrana, insieme ad ATG9A, per identificare dove si trova ATG9A. Diversi approcci sono stati utilizzati in passato per quantificare la localizzazione di ATG9A, incluso il coefficiente di correlazione di Pearson per la colocalizzazione29. Tuttavia, la parziale sovrapposizione di ATG9A con il Golgi e il distinto compartimento vescicolare porta a un elevato numero di valori anomali dei pixel, che possono influenzare l'interpretazione del coefficiente di correlazione. Per questo motivo, si preferisce un approccio più semplicistico basato sul rapporto della fluorescenza media nei due compartimenti da analizzare, e questo approccio è meno sensibile alla variabilità cellula-per-cellula. Per ulteriori informazioni sull'analisi delle immagini al microscopio, i lettori sono indirizzati al capitolo30 di questo libro.
Quando si studia lo stato di glicosilazione di ATG9A, è importante la selezione dei gel per l'esecuzione dei western blot. Per questo protocollo, sono preferiti gel di Tris-acetato al 3%-8% perché offrono la massima risoluzione per le proteine più grandi, ma possono essere utilizzate anche composizioni di gel alternative o tamponi in esecuzione che offrono una buona separazione delle proteine ad alto peso molecolare. Lo sperimentatore può garantire la massima separazione delle proteine aumentando il tempo di elettroforesi.
Quando si preparano i campioni per visualizzare ATG9A sul western blot, si deve fare attenzione a non far bollire i campioni dopo aver aggiunto il tampone Laemmli; l'ebollizione a 95 °C induce la formazione di aggregati di ATG9A e, successivamente, ATG9A non migra in modo efficiente nel gel1. Si consiglia di riscaldare i campioni a 65 °C per 5 minuti27.
Alti livelli di trasfezione di solito portano ad un maggiore accumulo di ATG9A nel reticolo endoplasmatico, mentre livelli moderati di espressione aiutano la fisiologica localizzazione della proteina. Aneddoticamente, i tempi di incubazione di 72 ore invece di 48 ore spesso aiutano a ridurre gli artefatti di localizzazione del pronto soccorso. In particolare, mRFP-ATG9A può riferire con precisione sul traffico e sulla funzione di ATG9A se i livelli sono controllati attraverso i livelli di espressione o utilizzando linee cellulari stabili 8,9,22,27.
L'incapacità di una popolazione di ATG9A sovraespressa di acquisire glicani N-linked maturi può essere utilizzata come lettura per il traffico perturbato di ATG9A. Quando si mutano o si eliminano alcune regioni di ATG9A, c'è il rischio di un aumento della ritenzione ER, che può portare alla mancata acquisizione di glicani N-legati maturi e, quindi, a una banda ATG9A che migra più velocemente sul western blot. I ricercatori che lavorano con costrutti ATG9A troncati dovrebbero verificare la ritenzione dell'ER, gli stati di glicosilazione e la localizzazione del Golgi.
Per l'imaging di cellule vive di ATG9A, un microscopio Airyscan, basandosi sulla funzione Airyscan veloce, fornisce una risoluzione ottimale di circa 120 nm. Per la precisione della localizzazione, frame rate di circa 1-2 fotogrammi al secondo (fps) in modalità super-risoluzione sono ottimali a seconda del numero di canali ripresi. Microscopi confocali simili in grado di eseguire immagini ad alta velocità possono essere utilizzati anche per l'imaging delle vescicole ATG9A; Tuttavia, va notato che la velocità di imaging può influenzare direttamente il rilevamento degli eventi e, quindi, influenzare l'interpretazione dei dati.
In sintesi, i protocolli presentati descrivono i modi per quantificare e caratterizzare la localizzazione di ATG9A mediante immunofluorescenza, microscopia su cellule vive e il suo stato di glicosilazione. Questi protocolli possono aiutare i ricercatori che lavorano con ATG9A e aiutare a evitare alcune insidie.
Divulgazioni
S.A.T. fa parte del comitato consultivo scientifico di Casma Therapeutics.
Riconoscimenti
Gli autori ringraziano Rocco D'Antuono per la correzione di bozze di alcuni aspetti del manoscritto, così come tutti i membri presenti e passati del laboratorio di Biologia Cellulare Molecolare dell'Autofagia (MCBA) per le discussioni che hanno portato al perfezionamento di questi protocolli. A. V.V., S.D.T., E.A., S.A.T, sono stati sostenuti dal Francis Crick Institute che riceve il suo finanziamento principale dal Cancer Research UK (CC2134), dal Consiglio per la ricerca medica del Regno Unito (CC2134). Questa ricerca è stata finanziata in tutto o in parte dal Wellcome Trust (CC2134). Ai fini dell'accesso aperto, l'autore ha applicato una licenza di copyright pubblico CC BY a qualsiasi versione del manoscritto accettato dall'autore derivante da questa presentazione.
Materiali
| Name | Company | Catalog Number | Comments |
| 24 multiwell plates | Falcon | 353047 | For tissue culture |
| 35 mm Dish | No. 1.5 Coverslip | 14 mm Glass Diameter | Uncoated | MATTEK | P35G-1.5-14-C | Cell culture dish for live-cell microscopy |
| 4x Laemmli Sample Buffer | Bio-Rad | 1610747 | |
| 60 mm tissue culture dish | Thermofischer Scientific | 10099170 | For tissue culture |
| Alexa Fluor 647 Phalloidin | Thermofischer Scientific | A22287 | Actin stain |
| anti-ATG9A antibody | home made | STO-215 | Rabbit anti N-terminal peptide ATG9A |
| anti-Rabbit IgG, peroxidase-linked | Invitrogen | 10794347/NA934-1ml | Secondary antibody for rabbit polyclonal STO-215 |
| anti-RFP antibody | Evrogen | AB233 | for Western Blot |
| ATG9A Monoclonal Antibody (14F2 8B1), Invitrogen | Invitrogen | 15232826 | Antibody for immunofluorescence |
| ATG9A-eGFP | home made | Construct which expresses tagged ATG9A | |
| Bemis Parafilm | Thermofischer Scientific | 11747487 | self-sealing thermoplastic film |
| Bio-Rad Protein Assay Dye Reagent Concentrate | Bio-Rad | 5000006 | For determining protein concentration |
| Bovine serum albumin (BSA) | Merck | 10735086001 | For blocking non-specific labelling |
| CaCl2.2H2O | / | For PBS and EBSS | |
| cOmplete Protease Inhibitor Cocktail | Roche | 11697498001 | Supplement in lysis buffer to prevent protein degradation |
| Cy3 AffiniPure Goat Anti-Armenian Hamster IgG | Jackson ImmunoResearch | 127-165-099 | Secondary antibody for i14F2 8B1 antibody for mmunofluorescence |
| Cyclohexamide | Sigma Aldrich | 66-81-9 | To stop protein translation |
| D-Glucose | / | For EBSS | |
| Digitonin | Merck | 300410 | For permeabilizing cells |
| DMEM | Merck | D6546-6x500ml | For tissue culture |
| eGFP-ATG9A | home made | Construct which expresses tagged ATG9A | |
| Fetal Bovine Serum | Gibco | 10270-106 | Supplement for DMEM for cell culture |
| FIJI (ImageJ) | / | https:/fiji.sc/ | Open source image analysis software |
| Hoechst | Thermofischer Scientific | H3570 | Stains the nucleus |
| KCl | / | For EBSS | |
| L-glutamine | Sigma | 67513 | For tissue culture |
| Lipofectamine 2000 | Invitrogen | 11668-019 | For Cell Transfection |
| LSM880 Airyscan microscope | Zeiss | / | Confocal microscopy |
| MgCl2 | / | For PBS | |
| MgSO4.7H2O | / | For EBSS | |
| Mowiol mounting solution | Millipore | 475904 | for permanent mounting glass coverslips |
| NaCl | / | For EBSS | |
| NaH2PO4.2H2O | / | For EBSS | |
| NaHCO3 | / | For EBSS | |
| NuPAGE 3 to 8%, Tris-Acetate, 1.5 mm, Mini Protein Gels | Thermofischer Scientific | EA0378BOX | for Western Blotting |
| NuPAGE MES SDS Running Buffer (20x) | Life Tech | NP0002 | for Western Blotting |
| Opti-MEM I Reduced Serum Medium | Thermo | 31985062 | For Cell Transfection |
| Paraformaldehyde | Agar Scientific | R1026 | For fixing cells |
| pcDNA3.1-mCherry-3xFlag-ATG9A | home made | Construct which expresses tagged ATG9A | |
| Phosphate Buffered Saline (PBS) | / | For tissue culture | |
| PNGaseF | NEB | P0710S | To remove N-linked glycans |
| Poly-D-lysine hydrobromide mol wt 70,000-150,000 | Merck | P0899 | For coating coverslips |
| Rapid PNGase F enzyme | NEB | P07105 | To remove N-linked glycans |
| RFP-ATG9A | home made | Construct which expresses tagged ATG9A | |
| Triton X-100 | Thermofischer Scientific | 13454259 | Detergent for Cell lysis |
| Trypsin-EDTA solution | Sigma | T4049 | For tissue culture |
| Whatman Filter Paper | Merck | WHA1001325 | For Western Blot and IF |
| XCell SureLock Mini-Cell Electrophoresis System | Invitrogen | EI0001 | For Western Blotting |
| Zen Black edition | Zeiss | / | Used to operate the LSM 880 |
Riferimenti
- Young, A. R., et al. Starvation and ULK1-dependent cycling of mammalian Atg9 between the TGN and endosomes. Journal of Cell Science. 119, 3888-3900 (2006).
- Maeda, S., et al. lipid scrambling activity and role in autophagosome formation of ATG9A. Nature Structural & Molecular Biology. 27 (12), 1194-1201 (2020).
- Matoba, K., et al. Atg9 is a lipid scramblase that mediates autophagosomal membrane expansion. Nature Structural & Molecular Biology. 27 (12), 1185-1193 (2020).
- Mercer, T. J., Gubas, A., Tooze, S. A. A molecular perspective of mammalian autophagosome biogenesis. Journal of Biological Chemistry. 293 (15), 5386-5395 (2018).
- Yamamoto, H., Zhang, S., Mizushima, N. Autophagy genes in biology and disease. Nature Reviews: Genetics. , (2023).
- Sawa-Makarska, J., et al. Reconstitution of autophagosome nucleation defines Atg9 vesicles as seeds for membrane formation. Science. 369 (6508), (2020).
- Melia, T. J., Lystad, A. H., Simonsen, A. Autophagosome biogenesis: From membrane growth to closure. Journal of Cell Biology. 219 (6), 202002085 (2020).
- Orsi, A., et al. Dynamic and transient interactions of Atg9 with autophagosomes, but not membrane integration, are required for autophagy. Molecular Biology of the Cell. 23 (10), 1860-1873 (2012).
- Judith, D., et al. ATG9A shapes the forming autophagosome through Arfaptin 2 and phosphatidylinositol 4-kinase IIIbeta. Journal of Cell Biology. 218 (5), 1634-1652 (2019).
- Karanasios, E., et al. Autophagy initiation by ULK complex assembly on ER tubulovesicular regions marked by ATG9 vesicles. Nature Communications. 7, 12420 (2016).
- Koyama-Honda, I., Itakura, E., Fujiwara, T. K., Mizushima, N. Temporal analysis of recruitment of mammalian ATG proteins to the autophagosome formation site. Autophagy. 9 (10), 1491-1499 (2013).
- Guardia, C. M., et al. Structure of human ATG9A, the only transmembrane protein of the core autophagy machinery. Cell Reports. 31 (13), 107837 (2020).
- Soreng, K., et al. SNX18 regulates ATG9A trafficking from recycling endosomes by recruiting Dynamin-2. EMBO Reports. 19 (4), e44837 (2018).
- Staudt, C., Gilis, F., Boonen, M., Jadot, M. Molecular determinants that mediate the sorting of human ATG9A from the endoplasmic reticulum. Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1863 (9), 2299-2310 (2016).
- Knaevelsrud, H., Carlsson, S. R., Simonsen, A. SNX18 tubulates recycling endosomes for autophagosome biogenesis. Autophagy. 9 (10), 1639-1641 (2013).
- Takahashi, Y., et al. The Bif-1-Dynamin 2 membrane fission machinery regulates Atg9-containing vesicle generation at the Rab11-positive reservoirs. Oncotarget. 7 (15), 20855-20868 (2016).
- Imai, K., et al. Atg9A trafficking through the recycling endosomes is required for autophagosome formation. Journal of Cell Science. 129 (20), 3781-3791 (2016).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- van Vliet, A. R., et al. ATG9A and ATG2A form a heteromeric complex essential for autophagosome formation. Molecular Cell. 82 (22), 4324-4339 (2022).
- Kielkopf, C. L., Bauer, W., Urbatsch, I. L. Bradford assay for determining protein concentration. Cold Spring Harbor Protocols. 2020 (4), 102269 (2020).
- Kurien, B. T., Scofield, R. H. Validating antibody specificities for immunohistochemistry by protein blotting. Methods in Molecular Biology. 2593, 21-33 (2023).
- Lamb, C. A., et al. TBC1D14 regulates autophagy via the TRAPP complex and ATG9 traffic. EMBO Journal. 35 (3), 281-301 (2016).
- Longatti, A., et al. TBC1D14 regulates autophagosome formation via Rab11- and ULK1-positive recycling endosomes. Journal of Cell Biology. 197 (5), 659-675 (2012).
- Ravussin, A., Brech, A., Tooze, S. A., Stenmark, H. The phosphatidylinositol 3-phosphate-binding protein SNX4 controls ATG9A recycling and autophagy. Journal of Cell Science. 134 (3), (2021).
- DesMarais, V., Eddy, R. J., Sharma, V. P., Stone, O., Condeelis, J. S. Optimizing leading edge F-actin labeling using multiple actin probes, fixation methods and imaging modalities. BioTechniques. 66 (3), 113-119 (2019).
- Ennis, H. L., Lubin, M. Cycloheximide: Aspects of inhibition of protein synthesis in mammalian cells. Science. 146 (3650), 1474-1476 (1964).
- Webber, J. L., Tooze, S. A. Coordinated regulation of autophagy by p38alpha MAPK through mAtg9 and p38IP. EMBO Journal. 29 (1), 27-40 (2010).
- Claude-Taupin, A., et al. ATG9A protects the plasma membrane from programmed and incidental permeabilization. Nature Cell Biology. 23 (8), 846-858 (2021).
- Aaron, J. S., Taylor, A. B., Chew, T. L. Image co-localization - Co-occurrence versus correlation. Journal of Cell Science. 131 (3), 211847 (2018).
- D'Antuono, R., Nechyporuk-Zloy, V. Basic digital image acquisition, design, processing, analysis, management, and presentation. Principles of Light Microscopy: From Basic to Advanced. , 77-104 (2022).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon