Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Generación de la vista de regulación transcripcional de características transcriptómicas para la tarea de predicción y la detección de biomarcadores oscuros en conjuntos de datos pequeños
En este artículo
Resumen
Aquí, presentamos un protocolo para convertir datos transcriptómicos en una vista mqTrans, lo que permite la identificación de biomarcadores oscuros. Si bien no se expresan diferencialmente en los análisis transcriptómicos convencionales, estos biomarcadores exhiben una expresión diferencial en la vista mqTrans. El enfoque sirve como una técnica complementaria a los métodos tradicionales, revelando biomarcadores que antes se pasaban por alto.
Resumen
El transcriptoma representa los niveles de expresión de muchos genes en una muestra y se ha utilizado ampliamente en la investigación biológica y la práctica clínica. Por lo general, los investigadores se centraron en biomarcadores transcriptómicos con representaciones diferenciales entre un grupo fenotípico y un grupo de control de muestras. Este estudio presentó un marco de aprendizaje multitarea de la red de atención gráfica (GAT) para aprender las complejas interacciones intergénicas de las muestras de referencia. Se entrenó previamente un modelo de referencia demostrativo en las muestras sanas (HealthModel), que podría usarse directamente para generar la vista de regulación transcripcional cuantitativa basada en modelos (mqTrans) de los transcriptomas de prueba independientes. La vista mqTrans generada de los transcriptomas se demostró mediante tareas de predicción y detección de biomarcadores oscuros. El término acuñado "biomarcador oscuro" se deriva de su definición de que un biomarcador oscuro mostraba una representación diferencial en la vista mqTrans, pero no una expresión diferencial en su nivel de expresión original. Un biomarcador oscuro siempre se pasó por alto en los estudios tradicionales de detección de biomarcadores debido a la ausencia de expresión diferencial. El código fuente y el manual de la canalización HealthModelPipe se pueden descargar desde http://www.healthinformaticslab.org/supp/resources.php.
Introducción
El transcriptoma consiste en la expresión de todos los genes en una muestra y puede ser perfilado por tecnologías de alto rendimiento como microarrays y RNA-seq1. Los niveles de expresión de un gen en un conjunto de datos se denominan característica transcriptómica, y la representación diferencial de una característica transcriptómica entre el fenotipo y los grupos control define a este gen como un biomarcador de este fenotipo 2,3. Los biomarcadores transcriptómicos se han utilizado ampliamente en las investigaciones del diagnóstico de enfermedades4, el mecanismo biológico5 y el análisis de supervivencia 6,7, etc.
Los patrones de actividad de los genes en los tejidos sanos contienen información crucial sobre las vidas 8,9. Estos patrones ofrecen información invaluable y actúan como referencias ideales para comprender las complejas trayectorias de desarrollo de los trastornos benignos10,11 y las enfermedades letales12. Los genes interactúan entre sí, y los transcriptomas representan los niveles finales de expresión después de sus complicadas interacciones. Tales patrones se formulan como la red de regulación transcripcional13 y la red metabólica14, etc. Las expresiones de ARN mensajeros (ARNm) pueden ser reguladas transcripcionalmente por factores de transcripción (TF) y ARN intergénicos largos no codificantes (lincRNAs)15,16,17. El análisis convencional de la expresión diferencial ignoró estas complejas interacciones génicas con el supuesto de independencia entre características18,19.
Los avances recientes en las redes neuronales de grafos (GNN) demuestran un potencial extraordinario en la extracción de información importante de los datos basados en OMIC para estudios de cáncer20, por ejemplo, la identificación de módulos de coexpresión21. La capacidad innata de las GNN las hace ideales para modelar las intrincadas relaciones y dependencias entre los genes22,23.
Los estudios biomédicos a menudo se centran en predecir con precisión un fenotipo en comparación con el grupo de control. Tales tareas se formulan comúnmente como clasificaciones binarias 24,25,26. Aquí, las dos etiquetas de clase se codifican normalmente como 1 y 0, verdadero y falso, o incluso positivo y negativo27.
Este estudio tuvo como objetivo proporcionar un protocolo fácil de usar para generar la vista de regulación transcripcional (mqTrans) de un conjunto de datos de transcriptoma basado en el modelo de referencia de la red de atención de grafos (GAT) preentrenado. Se utilizó el marco GAT multitarea de un trabajo publicado anteriormente26 para transformar las características transcriptómicas en las características mqTrans. Se utilizó un gran conjunto de datos de transcriptomas sanos de la plataforma Xena28 de la Universidad de California, Santa Cruz (UCSC) para preentrenar el modelo de referencia (HealthModel), que midió cuantitativamente las regulaciones de transcripción desde los factores reguladores (TF y lincRNAs) hasta los ARNm diana. La vista mqTrans generada podría utilizarse para construir modelos de predicción y detectar biomarcadores oscuros. Este protocolo utiliza el conjunto de datos de pacientes con adenocarcinoma de colon (COAD) de la base de datos del Atlas del Genoma del Cáncer (TCGA)29 como ejemplo ilustrativo. En este contexto, los pacientes en estadios I o II se clasifican como muestras negativas, mientras que los que se encuentran en estadios III o IV se consideran muestras positivas. También se comparan las distribuciones de los biomarcadores oscuros y tradicionales en los 26 tipos de cáncer TCGA.
Descripción de la canalización de HealthModel
La metodología empleada en este protocolo se basa en el marco26 publicado anteriormente, como se describe en la Figura 1. Para comenzar, los usuarios deben preparar el conjunto de datos de entrada, introducirlo en la canalización propuesta de HealthModel y obtener características de mqTrans. Las instrucciones detalladas de preparación de datos se proporcionan en la sección 2 de la sección de protocolo. Posteriormente, los usuarios tienen la opción de combinar las características de mqTrans con las características transcriptómicas originales o continuar solo con las características de mqTrans generadas. A continuación, el conjunto de datos producido se somete a un proceso de selección de características, en el que los usuarios tienen la flexibilidad de elegir su valor preferido para k en la validación cruzada de k-fold para la clasificación. La principal métrica de evaluación utilizada en este protocolo es la precisión.
HealthModel26 clasifica las características transcriptómicas en tres grupos distintos: TF (factor de transcripción), lincRNA (ARN intergénico largo no codificante) y ARNm (ARN mensajero). Las características de TF se definen en función de las anotaciones disponibles en el Atlas de Proteínas Humanas30,31. Este trabajo utiliza las anotaciones de lincRNAs del conjunto de datos GTEx32. Los genes que pertenecen a las vías de tercer nivel en la base de datos KEGG33 se consideran características de ARNm. Vale la pena señalar que si una característica de ARNm exhibe funciones reguladoras para un gen diana, como se documenta en la base de datos TRRUST34, se reclasifica en la clase TF.
Este protocolo también genera manualmente los dos archivos de ejemplo para las identificaciones de genes de los factores reguladores (regulatory_geneIDs.csv) y el ARNm diana (target_geneIDs.csv). La matriz de distancia por pares entre las características reguladoras (TFs y lincRNAs) se calcula mediante los coeficientes de correlación de Pearson y se agrupa mediante la popular herramienta de análisis de redes de coexpresión génica ponderada (WGCNA)36 (adjacent_matrix.csv). Los usuarios pueden utilizar directamente la canalización HealthModel junto con estos archivos de configuración de ejemplo para generar la vista mqTrans de un conjunto de datos transcriptómicos.
Detalles técnicos de HealthModel
HealthModel representa las intrincadas relaciones entre TF y lincRNAs como un grafo, con las características de entrada que sirven como los vértices denotados por V y una matriz de borde entre vértices designada como E. Cada muestra se caracteriza por características reguladoras K, simbolizadas como VK×1. En concreto, el conjunto de datos abarcó 425 TF y 375 lincRNAs, lo que dio como resultado una dimensionalidad de la muestra de K = 425 + 375 = 800. Para establecer la matriz de bordes E, en este trabajo se empleó la popular herramienta WGCNA35. El peso por pares que une dos vértices representados como 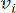 y
y 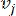 , está determinado por el coeficiente de correlación de Pearson. La red reguladora de genes exhibe una topología libre de escala36, caracterizada por la presencia de genes centrales con roles funcionales fundamentales. Calculamos la correlación entre dos características o vértices,
, está determinado por el coeficiente de correlación de Pearson. La red reguladora de genes exhibe una topología libre de escala36, caracterizada por la presencia de genes centrales con roles funcionales fundamentales. Calculamos la correlación entre dos características o vértices, 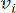 y
y 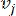 , utilizando la medida de superposición topológica (TOM) de la siguiente manera:
, utilizando la medida de superposición topológica (TOM) de la siguiente manera:
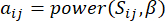 (1)
(1)
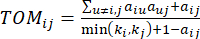 (2)
(2)
El β de umbral suave se calcula utilizando la función 'pickSoft Threshold' del paquete WGCNA. Se aplica la función exponencial de potencia aij , donde 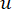 representa un gen excluyendo i y j, y
representa un gen excluyendo i y j, y 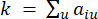 representa la conectividad de los vértices. WGCNA agrupa los perfiles de expresión de las características transcriptómicas en múltiples módulos utilizando una medida de disimilitud comúnmente empleada (
representa la conectividad de los vértices. WGCNA agrupa los perfiles de expresión de las características transcriptómicas en múltiples módulos utilizando una medida de disimilitud comúnmente empleada (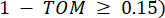 37.
37.
El marco HealthModel se diseñó originalmente como una arquitectura de aprendizaje multitarea26. Este protocolo solo utiliza la tarea de preentrenamiento del modelo para la construcción de la vista transcriptómica mqTrans. El usuario puede optar por refinar aún más el HealthModel previamente entrenado en la red de atención de grafos multitarea con muestras transcriptómicas adicionales específicas de la tarea.
Detalles técnicos de la selección y clasificación de características
El grupo de selección de características implementa once algoritmos de selección de características (FS). Entre ellos, tres son algoritmos de FS basados en filtros: selección de K mejores características utilizando el Coeficiente de Información Máxima (SK_mic), selección de K características basadas en el FPR de MIC (SK_fpr) y selección de K características con la tasa de falso descubrimiento más alta de MIC (SK_fdr). Además, tres algoritmos FS basados en árboles evalúan características individuales utilizando un árbol de decisión con el índice de Gini (DT_gini), árboles de decisión potenciados adaptativos (AdaBoost) y bosque aleatorio (RF_fs). El grupo también incorpora dos métodos contenedores: la eliminación de características recursivas con el clasificador de vectores de soporte lineal (RFE_SVC) y la eliminación de características recursivas con el clasificador de regresión logística (RFE_LR). Por último, se incluyen dos algoritmos de incrustación: el clasificador SVC lineal con los valores de importancia de la característica L1 mejor clasificados (lSVC_L1) y el clasificador de regresión logística con los valores de importancia de la característica L1 mejor clasificados (LR_L1).
El grupo de clasificadores emplea siete clasificadores diferentes para crear modelos de clasificación. Estos clasificadores comprenden la máquina de vectores de soporte lineal (SVC), Gaussian Naïve Bayes (GNB), el clasificador de regresión logística (LR), el k-vecino más cercano, con k establecido en 5 de forma predeterminada (KNN), XGBoost, bosque aleatorio (RF) y árbol de decisión (DT).
La división aleatoria del conjunto de datos en los subconjuntos train: test se puede establecer en la línea de comandos. En el ejemplo demostrado se utiliza la relación de train: test = 8:2.
Access restricted. Please log in or start a trial to view this content.
Protocolo
NOTA: El siguiente protocolo describe los detalles del procedimiento analítico informático y los comandos de Python de los módulos principales. La Figura 2 ilustra los tres pasos principales con comandos de ejemplo utilizados en este protocolo y se refieren a los trabajos publicados anteriormente26,38 para obtener más detalles técnicos. Realice el siguiente protocolo en una cuenta de usuario normal en un sistema informático y evite usar la cuenta de administrador o raíz. Este es un protocolo computacional y no tiene factores biomédicos peligrosos.
1. Preparar el entorno de Python
- Crear un entorno virtual.
- En este estudio se utilizó el lenguaje de programación Python y un entorno virtual (VE) Python con Python 3.7. Siga estos pasos (Figura 3A):
Conda create -n healthmodel python=3.7
conda create es el comando para crear un nuevo VE. El parámetro -n especifica el nombre del nuevo entorno, en este caso, healthmodel. Y python=3.7 especifica la versión de Python que se va a instalar. Elija cualquier nombre preferido y una versión de Python que admita el comando anterior. - Después de ejecutar el comando, el resultado es similar al de la figura 3B. Ingrese y y espere a que se complete el proceso.
- En este estudio se utilizó el lenguaje de programación Python y un entorno virtual (VE) Python con Python 3.7. Siga estos pasos (Figura 3A):
- Activar el entorno virtual
- En la mayoría de los casos, active el VE creado con el siguiente comando (Figura 3C):
Conda Activate HealthModel - Siga las instrucciones específicas de la plataforma para la activación de VE, si algunas plataformas requieren que el usuario cargue los archivos de configuración específicos de la plataforma para la activación.
- En la mayoría de los casos, active el VE creado con el siguiente comando (Figura 3C):
- Instalar PyTorch 1.13.1
- PyTorch es un popular paquete de Python para algoritmos de inteligencia artificial (IA). Utilice PyTorch 1.13.1, basado en la plataforma de programación de GPU CUDA 11.7, como ejemplo. Encuentre otras versiones en https://pytorch.org/get-started/previous-versions/. Utilice el siguiente comando (Figura 3D):
PIP3 Instalar antorcha de visiónAudio
NOTA: Se recomienda encarecidamente el uso de PyTorch versión 1.12 o posterior. De lo contrario, instalar el paquete requerido torch_geometric puede ser un desafío, como se indica en el sitio web oficial de torch_geometric: https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html.
- PyTorch es un popular paquete de Python para algoritmos de inteligencia artificial (IA). Utilice PyTorch 1.13.1, basado en la plataforma de programación de GPU CUDA 11.7, como ejemplo. Encuentre otras versiones en https://pytorch.org/get-started/previous-versions/. Utilice el siguiente comando (Figura 3D):
- Instalación de paquetes adicionales para torch-geometric
- Siguiendo las instrucciones de https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html, instale los siguientes paquetes: torch_scatter, torch_sparse, torch_cluster y torch_spline_conv mediante el comando (Figura 3E):
pip install pyg_lib torch_scatter torch_sparse torch_cluster torch_spline_conv -f https://data.pyg.org/whl/torch-1.13.0+cu117.html
- Siguiendo las instrucciones de https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html, instale los siguientes paquetes: torch_scatter, torch_sparse, torch_cluster y torch_spline_conv mediante el comando (Figura 3E):
- Instale el paquete antorcha-geométrica .
- Este estudio requiere una versión específica, 2.2.0, del paquete antorcha-geométrica . Ejecute el comando (Figura 3F):
pip install torch_geometric==2.2.0
- Este estudio requiere una versión específica, 2.2.0, del paquete antorcha-geométrica . Ejecute el comando (Figura 3F):
- Instale otros paquetes.
- Los paquetes como pandas suelen estar disponibles de forma predeterminada. Si no es así, instálelos usando el comando pip. Por ejemplo, para instalar pandas y xgboost, ejecute:
PIP Install Pandas
pip install xgboost
- Los paquetes como pandas suelen estar disponibles de forma predeterminada. Si no es así, instálelos usando el comando pip. Por ejemplo, para instalar pandas y xgboost, ejecute:
2. Uso del HealthModel previamente entrenado para generar las características de mqTrans
- Descargue el código y el modelo previamente entrenado.
- Descargue el código y el HealthModel previamente entrenado desde el sitio web: http://www.healthinformaticslab.org/supp/resources.php, que se denomina HealthModel-mqTrans-v1-00.tar.gz (Figura 4A). El archivo descargado se puede descomprimir a una ruta especificada por el usuario. La formulación detallada y los datos de apoyo del protocolo implementado se pueden encontrar en26.
- Introduzca los parámetros para ejecutar HealthModel.
- En primer lugar, cambie el directorio de trabajo a la carpeta HealthModel-mqTrans en la línea de comandos. Use la siguiente sintaxis para ejecutar el código:
Python main.py
Los detalles relativos a cada parámetro y a las carpetas de datos, modelo y salida son los siguientes:
carpeta de datos: Esta es la carpeta de datos de origen y cada archivo de datos está en formato csv. Esta carpeta de datos tiene dos archivos (consulte las descripciones detalladas en los pasos 2.3 y 2.4). Estos archivos deben ser reemplazados por datos personales.
data.csv: El archivo de matriz transcriptómica. En la primera fila se enumeran los identificadores de características (o genes) y en la primera columna se muestran los identificadores de muestra. La lista de genes incluye los factores reguladores (TF y lincRNAs) y los genes de ARNm regulados.
label.csv: El archivo de etiqueta de ejemplo. La primera columna enumera los identificadores de muestra y la columna con el nombre "label" proporciona la etiqueta de ejemplo.
carpeta del modelo: La carpeta para guardar información sobre el modelo:
HealthModel.pth: el HealthModel previamente entrenado.
regulatory_geneIDs.csv: Las identificaciones de genes reguladores utilizados en este estudio.
target_geneIDs.csv: Los genes diana utilizados en este estudio.
adjacent_matrix.csv: La matriz adyacente de genes reguladores.
Carpeta de salida: Los archivos de salida se escriben en esta carpeta, creada por el código.
test_target.csv: El valor de expresión génica de los genes diana después de la normalización e imputación de Z.
pred_target.csv: El valor de expresión génica predicho de los genes diana.
mq_target.csv: El valor de expresión génica predicho de los genes diana.
- En primer lugar, cambie el directorio de trabajo a la carpeta HealthModel-mqTrans en la línea de comandos. Use la siguiente sintaxis para ejecutar el código:
- Prepare el archivo de matriz transcriptómica en formato csv.
- Cada fila representa una muestra y cada columna representa un gen (Figura 4B). Asigne al archivo de matriz de datos transcriptómicos el nombre data.csv en la carpeta de datos .
NOTA: Este archivo se puede generar guardando manualmente una matriz de datos en el formato .csv de un software como Microsoft Excel. La matriz transcriptómica también puede ser generada por programación informática.
- Cada fila representa una muestra y cada columna representa un gen (Figura 4B). Asigne al archivo de matriz de datos transcriptómicos el nombre data.csv en la carpeta de datos .
- Prepare el archivo de etiqueta en formato csv.
- De forma similar al archivo de matriz transcriptómica, asigne al archivo de etiquetas el nombre label.csv en la carpeta de datos (Figura 4C).
NOTA: La primera columna proporciona los nombres de las muestras, y la etiqueta de clase de cada muestra se proporciona en la columna titulada etiqueta. El valor 0 en la columna de la etiqueta significa que esta muestra es negativa, 1 significa una muestra positiva.
- De forma similar al archivo de matriz transcriptómica, asigne al archivo de etiquetas el nombre label.csv en la carpeta de datos (Figura 4C).
- Genere las características de mqTrans.
- Ejecute el siguiente comando para generar las características de mqTrans y obtener los resultados que se muestran en la Figura 4D. Las características de mqTrans se generan como el archivo ./output/mq_targets.csv, y el archivo de etiquetas se vuelve a guardar como el archivo ./output/label.csv. Para facilitar un análisis posterior, los valores de expresión originales de los genes de ARNm también se extraen como archivo ./output/ test_target.csv.
python ./Get_mqTrans/code/main.py ./data ./Get_mqTrans/model ./output
- Ejecute el siguiente comando para generar las características de mqTrans y obtener los resultados que se muestran en la Figura 4D. Las características de mqTrans se generan como el archivo ./output/mq_targets.csv, y el archivo de etiquetas se vuelve a guardar como el archivo ./output/label.csv. Para facilitar un análisis posterior, los valores de expresión originales de los genes de ARNm también se extraen como archivo ./output/ test_target.csv.
3. Seleccione las características de mqTrans
- Sintaxis del código de selección de características
- En primer lugar, cambie el directorio de trabajo a la carpeta HealthModel-mqTrans . Utilice la siguiente sintaxis:
python ./FS_classification/testMain.py
Los detalles de cada parámetro son los siguientes:
in-data-file: El archivo de datos de entrada
in-label-file: La etiqueta del archivo de datos de entrada
Carpeta de salida: En esta carpeta se guardan dos archivos de salida, incluidos Output-score.xlsx (el método de selección de características y la precisión del clasificador correspondiente) y Output-SelectedFeatures.xlsx (los nombres de características seleccionadas para cada algoritmo de selección de características).- select_feature_number: seleccione el número de entidades, que va desde 1 hasta el número de entidades del archivo de datos.
- test_size: Establezca la proporción de la muestra de prueba que se va a dividir. Por ejemplo, 0,2 significa que el conjunto de datos de entrada se divide aleatoriamente en los subconjuntos de prueba train: test en la proporción de 0,8:0,2.
- combine: Si es true, combine dos archivos de datos para la selección de características, es decir, los valores de expresión originales y las características de mqTrans. Si es falso, utilice un archivo de datos para la selección de características, es decir, los valores de expresión originales o las características de mqTrans.
- combine file: Si combine es true, proporcione este nombre de archivo para guardar la matriz de datos combinada.
NOTA: Esta canalización tiene como objetivo demostrar cómo se comportan las características de mqTrans generadas en las tareas de clasificación, y utiliza directamente el archivo generado por la sección 2 para las siguientes operaciones.
- En primer lugar, cambie el directorio de trabajo a la carpeta HealthModel-mqTrans . Utilice la siguiente sintaxis:
- Ejecute el algoritmo de selección de características para la selección de características de mqTrans.
- Gire combinar =False si el usuario selecciona características de mqTrans o características originales.
- En primer lugar, seleccione 800 características originales y divida el conjunto de datos en train: test=0.8:0.2:
python ./FS_classification/testMain.py ./output/test_target.csv ./output/label.csv ./result 800 0.2 False - Gire combine =True, si el usuario desea combinar las características de mqTrans con los valores de expresión originales para seleccionar características. Aquí, el ejemplo demostrativo es seleccionar 800 características y dividir el conjunto de datos en train: test=0.8:0.2:
python ./FS_classification/testMain.py ./output/mq_targets.csv ./output/label.csv ./result_combine 800 0.2 True ./output/test_target.csv
NOTA: La Figura 5 muestra la información de salida. Los archivos suplementarios necesarios para este protocolo se encuentran en HealthModel-mqTrans-v1-00.tar carpeta (Archivo de codificación suplementaria 1).
Access restricted. Please log in or start a trial to view this content.
Resultados
Evaluación de la vista mqTrans del conjunto de datos transcriptómicos
El código de prueba utiliza once algoritmos de selección de características (FS) y siete clasificadores para evaluar cómo la vista mqTrans generada del conjunto de datos transcriptómicos contribuye a la tarea de clasificación (Figura 6). El conjunto de datos de prueba consta de 317 adenocarcinomas de colon (COAD) de la base de datos The Cancer Genome Atlas (TCGA)29. Los p...
Access restricted. Please log in or start a trial to view this content.
Discusión
La sección 2 (Uso del modelo de salud previamente entrenado para generar las características de mqTrans) del protocolo es el paso más crítico dentro de este protocolo. Después de preparar el entorno de trabajo computacional en la sección 1, la sección 2 genera la vista mqTrans de un conjunto de datos transcriptómico basado en el modelo de referencia grande previamente entrenado. La sección 3 es un ejemplo demostrativo de la selección de las características mqTrans generadas para tareas de predicción y detecci...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo contó con el apoyo del Equipo de Innovación Tecnológica Senior y Junior (20210509055RQ), los Proyectos de Ciencia y Tecnología de la Provincia de Guizhou (ZK2023-297), la Fundación de Ciencia y Tecnología de la Comisión de Salud de la Provincia de Guizhou (gzwkj2023-565), el Proyecto de Ciencia y Tecnología del Departamento de Educación de la Provincia de Jilin (JJKH20220245KJ y JJKH20220226SK), la Fundación Nacional de Ciencias Naturales de China (U19A2061), el Laboratorio Clave Provincial de Jilin de Computación Inteligente de Big Data (20180622002JC), y los Fondos de Investigación Fundamental para las Universidades Centrales, JLU. Extendemos nuestro más sincero agradecimiento al editor de la revisión y a los tres revisores anónimos por sus críticas constructivas, que han sido fundamentales para mejorar sustancialmente el rigor y la claridad de este protocolo.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
Referencias
- Mutz, K. -O., Heilkenbrinker, A., Lönne, M., Walter, J. -G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516(2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684(2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352(2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528(2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. Analysis of Survival Data. , Chapman and Hall/CRC. London. (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866(2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307(2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119(2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239(2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334(2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338(2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , Springer. Cham. (2022).
- Muzio, G., O'Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738(2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559(2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5(2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883(2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582(2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801(2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138(2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308(2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados