È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Generazione della vista di regolazione trascrizionale delle caratteristiche trascrittomiche per l'attività di predizione e il rilevamento di biomarcatori oscuri su set di dati di piccole dimensioni
In questo articolo
Riepilogo
Qui, introduciamo un protocollo per convertire i dati trascrittomici in una vista mqTrans, consentendo l'identificazione di biomarcatori oscuri. Sebbene non siano espressi in modo differenziale nelle analisi trascrittomiche convenzionali, questi biomarcatori mostrano un'espressione differenziale nella vista mqTrans. L'approccio funge da tecnica complementare ai metodi tradizionali, svelando biomarcatori precedentemente trascurati.
Abstract
Il trascrittoma rappresenta i livelli di espressione di molti geni in un campione ed è stato ampiamente utilizzato nella ricerca biologica e nella pratica clinica. I ricercatori di solito si sono concentrati sui biomarcatori trascrittomici con rappresentazioni differenziali tra un gruppo fenotipico e un gruppo di controllo di campioni. Questo studio ha presentato un framework di apprendimento GAT (Graph-Attention Network) multitasking per apprendere le complesse interazioni intergeniche dei campioni di riferimento. Un modello di riferimento dimostrativo è stato pre-addestrato sui campioni sani (HealthModel), che potrebbe essere utilizzato direttamente per generare la vista di regolazione trascrizionale quantitativa basata su modelli (mqTrans) dei trascrittomi di test indipendenti. La vista mqTrans generata dei trascrittomi è stata dimostrata da attività di predizione e rilevamento di biomarcatori oscuri. Il termine coniato "biomarcatore oscuro" deriva dalla sua definizione che un biomarcatore oscuro mostrava una rappresentazione differenziale nella vista mqTrans ma nessuna espressione differenziale nel suo livello di espressione originale. Un biomarcatore oscuro è sempre stato trascurato negli studi tradizionali di rilevamento dei biomarcatori a causa dell'assenza di espressione differenziale. Il codice sorgente e il manuale della pipeline HealthModelPipe possono essere scaricati da http://www.healthinformaticslab.org/supp/resources.php.
Introduzione
Il trascrittoma è costituito dall'espressione di tutti i geni in un campione e può essere profilato mediante tecnologie ad alto rendimento come il microarray e l'RNA-seq1. I livelli di espressione di un gene in un set di dati sono chiamati caratteristica trascrittomica e la rappresentazione differenziale di una caratteristica trascrittomica tra il fenotipo e i gruppi di controllo definisce questo gene come biomarcatore di questo fenotipo 2,3. I biomarcatori trascrittomici sono stati ampiamente utilizzati nelle indagini sulla diagnosi di malattia4, sul meccanismo biologico5 e sull'analisi di sopravvivenza 6,7, ecc.
I modelli di attività genica nei tessuti sani trasportano informazioni cruciali sulla vita 8,9. Questi modelli offrono informazioni preziose e fungono da riferimento ideali per comprendere le complesse traiettorie di sviluppo dei disturbi benigni 10,11 e delle malattie letali12. I geni interagiscono tra loro e i trascrittomi rappresentano i livelli di espressione finale dopo le loro complicate interazioni. Tali modelli sono formulati come rete di regolazione trascrizionale13 e rete metabolica14, ecc. L'espressione degli RNA messaggeri (mRNA) può essere regolata trascrizionalmente da fattori di trascrizione (TF) e lunghi RNA intergenici non codificanti (lincRNAs)15,16,17. L'analisi convenzionale dell'espressione differenziale ha ignorato tali complesse interazioni geniche con l'assunzione di indipendenza inter-caratteristica18,19.
I recenti progressi nelle reti neurali a grafo (GNN) dimostrano un potenziale straordinario nell'estrazione di informazioni importanti dai dati basati su OMIC per gli studi sul cancro20, ad esempio identificando i moduli di co-espressione21. La capacità innata dei GNN li rende ideali per modellare le intricate relazioni e dipendenze tra i geni22,23.
Gli studi biomedici spesso si concentrano sulla previsione accurata di un fenotipo rispetto al gruppo di controllo. Tali compiti sono comunemente formulati come classificazioni binarie 24,25,26. In questo caso, le due etichette di classe sono in genere codificate come 1 e 0, vero e falso o anche positivo e negativo27.
Questo studio mirava a fornire un protocollo di facile utilizzo per la generazione della vista di regolazione trascrizionale (mqTrans) di un set di dati di trascrittoma basato sul modello di riferimento GAT (graph-attention network) pre-addestrato. Il framework GAT multitasking di un lavoroprecedentemente pubblicato 26 è stato utilizzato per trasformare le caratteristiche trascrittomiche nelle caratteristiche mqTrans. Un ampio set di dati di trascrittomi sani della piattaforma Xena28 dell'Università della California, Santa Cruz (UCSC) è stato utilizzato per pre-addestrare il modello di riferimento (HealthModel), che ha misurato quantitativamente le regolazioni di trascrizione dai fattori regolatori (TF e lincRNA) agli mRNA bersaglio. La vista mqTrans generata potrebbe essere utilizzata per costruire modelli di previsione e rilevare biomarcatori oscuri. Questo protocollo utilizza il set di dati dei pazienti con adenocarcinoma del colon (COAD) dal database The Cancer Genome Atlas (TCGA)29 come esempio illustrativo. In questo contesto, i pazienti in stadio I o II sono classificati come campioni negativi, mentre quelli in stadio III o IV sono considerati campioni positivi. Vengono inoltre confrontate le distribuzioni dei biomarcatori oscuri e tradizionali nei 26 tipi di cancro TCGA.
Descrizione della pipeline HealthModel
La metodologia impiegata in questo protocollo si basa sul framework26 precedentemente pubblicato, come delineato nella Figura 1. Per iniziare, gli utenti devono preparare il set di dati di input, inserirlo nella pipeline HealthModel proposta e ottenere le funzionalità mqTrans. Le istruzioni dettagliate per la preparazione dei dati sono fornite nella sezione 2 della sezione relativa al protocollo. Successivamente, gli utenti hanno la possibilità di combinare le funzionalità mqTrans con le caratteristiche trascrittomiche originali o di procedere solo con le caratteristiche mqTrans generate. Il set di dati prodotto viene quindi sottoposto a un processo di selezione delle caratteristiche, con gli utenti che hanno la flessibilità di scegliere il valore preferito per k nella convalida incrociata k-fold per la classificazione. La metrica di valutazione principale utilizzata in questo protocollo è l'accuratezza.
HealthModel26 classifica le caratteristiche trascrittomiche in tre gruppi distinti: TF (fattore di trascrizione), lincRNA (RNA non codificante intergenico lungo) e mRNA (RNA messaggero). Le caratteristiche del TF sono definite in base alle annotazioni disponibili nell'Atlante delle Proteine Umane30,31. Questo lavoro utilizza le annotazioni dei lincRNA dal set di dati GTEx32. I geni appartenenti alle vie di terzo livello nel database KEGG33 sono considerati come caratteristiche dell'mRNA. Vale la pena notare che se una caratteristica dell'mRNA mostra ruoli regolatori per un gene bersaglio, come documentato nel database TRRUST34, viene riclassificata nella classe TF.
Questo protocollo genera anche manualmente i due file di esempio per gli ID dei geni dei fattori regolatori (regulatory_geneIDs.csv) e dell'mRNA bersaglio (target_geneIDs.csv). La matrice di distanza a coppie tra le caratteristiche regolatorie (TF e lincRNA) è calcolata dai coefficienti di correlazione di Pearson e raggruppata mediante la popolare analisi della rete di co-espressione genica pesata su strumenti (WGCNA)36 (adjacent_matrix.csv). Gli utenti possono utilizzare direttamente la pipeline HealthModel insieme a questi file di configurazione di esempio per generare la vista mqTrans di un set di dati trascrittomico.
Dettagli tecnici di HealthModel
HealthModel rappresenta le intricate relazioni tra TF e lincRNA come un grafo, con le caratteristiche di input che fungono da vertici indicati da V e una matrice di bordi inter-vertice designata come E. Ogni campione è caratterizzato da caratteristiche regolatorie K , simboleggiate da VK×1. In particolare, il set di dati comprendeva 425 TF e 375 lincRNA, risultando in una dimensionalità del campione di K = 425 + 375 = 800. Per stabilire la matrice dei bordi E, questo lavoro ha utilizzato il popolare strumento WGCNA35. Il peso a coppie che collega due vertici rappresentati come 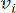 e , è determinato dal coefficiente
e , è determinato dal coefficiente 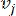 di correlazione di Pearson. La rete di regolazione genica presenta una topologia scale-free36, caratterizzata dalla presenza di geni hub con ruoli funzionali cardine. Calcoliamo la correlazione tra due caratteristiche o vertici,
di correlazione di Pearson. La rete di regolazione genica presenta una topologia scale-free36, caratterizzata dalla presenza di geni hub con ruoli funzionali cardine. Calcoliamo la correlazione tra due caratteristiche o vertici, 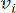 e
e 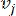 , usando la misura di sovrapposizione topologica (TOM) come segue:
, usando la misura di sovrapposizione topologica (TOM) come segue:
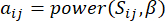 (1)
(1)
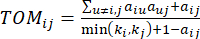 (2)
(2)
Il β di soglia soft viene calcolato utilizzando la funzione 'pickSoft Threshold' del pacchetto WGCNA. Viene applicata la funzione esponenziale di potenza aij , dove 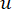 rappresenta un gene escludendo i e j, e
rappresenta un gene escludendo i e j, e 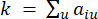 rappresenta la connettività dei vertici. WGCNA raggruppa i profili di espressione delle caratteristiche trascrittomiche in più moduli utilizzando una misura di dissimilarità comunemente impiegata (
rappresenta la connettività dei vertici. WGCNA raggruppa i profili di espressione delle caratteristiche trascrittomiche in più moduli utilizzando una misura di dissimilarità comunemente impiegata (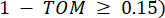 37.
37.
Il framework HealthModel è stato originariamente progettato come un'architettura di apprendimento multitasking26. Questo protocollo utilizza solo l'attività di pre-addestramento del modello per la costruzione della vista mqTrans trascrittomica. L'utente può scegliere di perfezionare ulteriormente l'HealthModel pre-addestrato nell'ambito della rete di attenzione del grafico multitask con ulteriori campioni trascrittomici specifici dell'attività.
Dettagli tecnici della selezione e della classificazione delle funzionalità
Il pool di selezione delle funzionalità implementa undici algoritmi di selezione delle funzionalità (FS). Tra questi, tre sono algoritmi FS basati su filtri: la selezione delle migliori caratteristiche K utilizzando il coefficiente di informazione massima (SK_mic), la selezione delle caratteristiche K in base all'FPR della MIC (SK_fpr) e la selezione delle caratteristiche K con il più alto tasso di falsa scoperta della MIC (SK_fdr). Inoltre, tre algoritmi FS basati su alberi valutano le singole funzionalità utilizzando un albero decisionale con l'indice di Gini (DT_gini), gli alberi decisionali potenziati adattivi (AdaBoost) e la foresta casuale (RF_fs). Il pool incorpora anche due metodi wrapper: l'eliminazione ricorsiva delle funzionalità con il classificatore del vettore di supporto lineare (RFE_SVC) e l'eliminazione ricorsiva delle funzionalità con il classificatore di regressione logistica (RFE_LR). Infine, sono inclusi due algoritmi di incorporamento: classificatore SVC lineare con i valori di importanza delle funzionalità L1 (lSVC_L1) più alti e classificatore di regressione logistica con i valori di importanza delle funzionalità L1 (LR_L1).
Il pool di classificatori utilizza sette classificatori diversi per compilare modelli di classificazione. Questi classificatori comprendono la macchina a vettori di supporto lineare (SVC), la naïve Bayes gaussiana (GNB), il classificatore di regressione logistica (LR), il vicino più vicino k, con k impostato su 5 per impostazione predefinita (KNN), XGBoost, foresta casuale (RF) e albero decisionale (DT).
La suddivisione casuale del set di dati nei sottoinsiemi di test train: può essere impostata nella riga di comando. Nell'esempio illustrato viene utilizzato il rapporto train: test = 8: 2.
Access restricted. Please log in or start a trial to view this content.
Protocollo
NOTA: Il seguente protocollo descrive i dettagli della procedura analitica informatica e dei comandi Python dei moduli principali. La Figura 2 illustra i tre passaggi principali con i comandi di esempio utilizzati in questo protocollo e fa riferimento ai lavori pubblicati in precedenza26,38 per maggiori dettagli tecnici. Eseguire il seguente protocollo con un normale account utente in un sistema informatico ed evitare di utilizzare l'account amministratore o root. Questo è un protocollo computazionale e non ha fattori di rischio biomedico.
1. Preparare l'ambiente Python
- Creare un ambiente virtuale.
- Questo studio ha utilizzato il linguaggio di programmazione Python e un ambiente virtuale Python (VE) con Python 3.7. Attenersi alla seguente procedura (Figura 3A):
conda create -n healthmodel python=3.7
conda create è il comando per creare un nuovo VE. Il parametro -n specifica il nome del nuovo ambiente, in questo caso healthmodel. E python=3.7 specifica la versione di Python da installare. Scegli il nome preferito e la versione di Python che supporta il comando precedente. - Dopo aver eseguito il comando, l'output è simile a quello della Figura 3B. Immettere y e attendere il completamento del processo.
- Questo studio ha utilizzato il linguaggio di programmazione Python e un ambiente virtuale Python (VE) con Python 3.7. Attenersi alla seguente procedura (Figura 3A):
- Attivare l'ambiente virtuale
- Nella maggior parte dei casi, attivare il VE creato con il seguente comando (Figura 3C):
Conda attiva HealthModel - Seguire le istruzioni specifiche della piattaforma per l'attivazione di VE, se alcune piattaforme richiedono all'utente di caricare i file di configurazione specifici della piattaforma per l'attivazione.
- Nella maggior parte dei casi, attivare il VE creato con il seguente comando (Figura 3C):
- Installare PyTorch 1.13.1
- PyTorch è un popolare pacchetto Python per algoritmi di intelligenza artificiale (AI). Usare PyTorch 1.13.1, basato sulla piattaforma di programmazione GPU CUDA 11.7, come esempio. Altre versioni sono disponibili su https://pytorch.org/get-started/previous-versions/. Utilizzare il comando seguente (Figura 3D):
pip3 installare torcia torcia torchvision torchaudio
NOTA: Si consiglia vivamente di utilizzare PyTorch versione 1.12 o successiva. In caso contrario, l'installazione del pacchetto richiesto torch_geometric potrebbe essere difficile, come indicato sul sito Web ufficiale torch_geometric: https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html.
- PyTorch è un popolare pacchetto Python per algoritmi di intelligenza artificiale (AI). Usare PyTorch 1.13.1, basato sulla piattaforma di programmazione GPU CUDA 11.7, come esempio. Altre versioni sono disponibili su https://pytorch.org/get-started/previous-versions/. Utilizzare il comando seguente (Figura 3D):
- Installare pacchetti aggiuntivi per torch-geometric
- Seguendo le linee guida riportate in https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html, installare i seguenti pacchetti: torch_scatter, torch_sparse, torch_cluster e torch_spline_conv utilizzando il comando (Figura 3E):
pip install pyg_lib torch_scatter torch_sparse torch_cluster torch_spline_conv -f https://data.pyg.org/whl/torch-1.13.0+cu117.html
- Seguendo le linee guida riportate in https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html, installare i seguenti pacchetti: torch_scatter, torch_sparse, torch_cluster e torch_spline_conv utilizzando il comando (Figura 3E):
- Installare il pacchetto torch-geometrico .
- Questo studio richiede una versione specifica, la 2.2.0, del pacchetto torch-geometrico . Eseguire il comando (Figura 3F):
pip install torch_geometric==2.2.0
- Questo studio richiede una versione specifica, la 2.2.0, del pacchetto torch-geometrico . Eseguire il comando (Figura 3F):
- Installare altri pacchetti.
- Pacchetti come panda sono di solito disponibili per impostazione predefinita. In caso contrario, installarli utilizzando il comando pip. Ad esempio, per installare panda e xgboost, eseguire:
pip installare panda
pip install xgboost
- Pacchetti come panda sono di solito disponibili per impostazione predefinita. In caso contrario, installarli utilizzando il comando pip. Ad esempio, per installare panda e xgboost, eseguire:
2. Utilizzo di HealthModel pre-addestrato per generare le funzionalità mqTrans
- Scaricare il codice e il modello pre-addestrato.
- Scaricare il codice e l'HealthModel pre-addestrato dal sito Web: http://www.healthinformaticslab.org/supp/resources.php, denominato HealthModel-mqTrans-v1-00.tar.gz (Figura 4A). Il file scaricato può essere decompresso in un percorso specificato dall'utente. La formulazione dettagliata e i dati di supporto del protocollo implementato sono disponibili in26.
- Introdurre i parametri per eseguire HealthModel.
- Innanzitutto, modificare la directory di lavoro nella cartella HealthModel-mqTrans nella riga di comando. Utilizzare la sintassi seguente per l'esecuzione del codice:
Cartella main.py
Di seguito sono riportati i dettagli relativi a ciascun parametro e alle cartelle di dati, modello e output:
cartella dei dati: questa è la cartella dei dati di origine e ogni file di dati è in formato csv. Questa cartella di dati contiene due file (vedere le descrizioni dettagliate nei passaggi 2.3 e 2.4). Questi file devono essere sostituiti con dati personali.
data.csv: Il file della matrice trascrittomica. La prima riga elenca gli ID delle funzionalità (o dei geni) e la prima colonna fornisce gli ID di esempio. L'elenco dei geni include i fattori regolatori (TF e lincRNA) e i geni mRNA regolati.
label.csv: il file di etichetta di esempio. La prima colonna elenca gli ID di esempio e la colonna con il nome "label" fornisce l'etichetta di esempio.
cartella del modello: la cartella in cui salvare le informazioni sul modello:
HealthModel.pth: HealthModel pre-addestrato.
regulatory_geneIDs.csv: Gli ID dei geni regolatori utilizzati in questo studio.
target_geneIDs.csv: I geni bersaglio utilizzati in questo studio.
adjacent_matrix.csv: La matrice adiacente dei geni regolatori.
cartella di output: i file di output vengono scritti in questa cartella, creata dal codice.
test_target.csv: Il valore di espressione genica dei geni bersaglio dopo la normalizzazione Z e l'imputazione.
pred_target.csv: Il valore di espressione genica previsto dei geni bersaglio.
mq_target.csv: Il valore di espressione genica previsto dei geni bersaglio.
- Innanzitutto, modificare la directory di lavoro nella cartella HealthModel-mqTrans nella riga di comando. Utilizzare la sintassi seguente per l'esecuzione del codice:
- Preparare il file della matrice trascrittomica in formato csv.
- Ogni riga rappresenta un campione e ogni colonna rappresenta un gene (Figura 4B). Assegnare al file Data Matrix trascrittomico il nome data.csv nella cartella dei dati .
NOTA: Questo file può essere generato salvando manualmente un data matrix in formato .csv da software come Microsoft Excel. La matrice trascrittomica può anche essere generata dalla programmazione informatica.
- Ogni riga rappresenta un campione e ogni colonna rappresenta un gene (Figura 4B). Assegnare al file Data Matrix trascrittomico il nome data.csv nella cartella dei dati .
- Preparare il file dell'etichetta in formato csv.
- Analogamente al file della matrice trascrittomica, assegnare al file di etichetta il nome label.csv nella cartella dei dati (Figura 4C).
NOTA: la prima colonna fornisce i nomi dei campioni e l'etichetta della classe di ciascun campione è indicata nella colonna intitolata etichetta. Il valore 0 nella colonna dell'etichetta indica che il campione è negativo, mentre 1 indica un campione positivo.
- Analogamente al file della matrice trascrittomica, assegnare al file di etichetta il nome label.csv nella cartella dei dati (Figura 4C).
- Generare le feature mqTrans.
- Eseguire il comando seguente per generare le feature mqTrans e ottenere gli output mostrati nella Figura 4D. Le feature mqTrans vengono generate come file ./output/mq_targets.csv e il file di etichetta viene salvato nuovamente come file ./output/label.csv. Per comodità di ulteriori analisi, i valori di espressione originali dei geni mRNA vengono estratti anche come file ./output/ test_target.csv.
python ./Get_mqTrans/code/main.py ./data ./Get_mqTrans/model ./output
- Eseguire il comando seguente per generare le feature mqTrans e ottenere gli output mostrati nella Figura 4D. Le feature mqTrans vengono generate come file ./output/mq_targets.csv e il file di etichetta viene salvato nuovamente come file ./output/label.csv. Per comodità di ulteriori analisi, i valori di espressione originali dei geni mRNA vengono estratti anche come file ./output/ test_target.csv.
3. Selezionare Caratteristiche mqTrans
- Sintassi del codice di selezione delle funzionalità
- Innanzitutto, modificare la directory di lavoro nella cartella HealthModel-mqTrans . Utilizzare la seguente sintassi:
python ./FS_classification/testMain.py
I dettagli di ogni parametro sono i seguenti:
in-data-file: il file di dati di input
in-label-file: l'etichetta del file di dati di input
cartella di output: in questa cartella vengono salvati due file di output, tra cui Output-score.xlsx (il metodo di selezione delle feature e la precisione del classificatore corrispondente) e Output-SelectedFeatures.xlsx (i nomi delle feature selezionate per ogni algoritmo di selezione delle feature).- select_feature_number: selezionare il numero di elementi, compreso tra 1 e il numero di elementi del file di dati.
- test_size: Impostare il rapporto del campione di prova da dividere. Ad esempio, 0,2 significa che il set di dati di input viene suddiviso in modo casuale nei sottoinsiemi di test train: in base al rapporto 0,8:0,2.
- combine: se true, combina due file di dati per la selezione delle feature, ovvero i valori dell'espressione originale e le feature mqTrans. Se false, è sufficiente utilizzare un file di dati per la selezione delle feature, ovvero i valori dell'espressione originale o le feature mqTrans.
- combine file: se combine è true, specificare il nome del file per salvare la matrice di dati combinata.
NOTA: Questa pipeline ha lo scopo di dimostrare come le funzionalità mqTrans generate si comportano nelle attività di classificazione e utilizza direttamente il file generato dalla sezione 2 per le seguenti operazioni.
- Innanzitutto, modificare la directory di lavoro nella cartella HealthModel-mqTrans . Utilizzare la seguente sintassi:
- Eseguire l'algoritmo di selezione delle caratteristiche per la selezione delle funzionalità mqTrans.
- Turn combine =False se l'utente seleziona le feature mqTrans o le feature originali.
- Innanzitutto, seleziona 800 funzionalità originali e suddividi il set di dati in train: test=0.8:0.2:
python ./FS_classification/testMain.py ./output/test_target.csv ./output/label.csv ./result 800 0.2 False - Turn combine =True, se l'utente desidera combinare le feature mqTrans con i valori dell'espressione originale per selezionare le feature. In questo caso, l'esempio dimostrativo consiste nel selezionare 800 funzionalità e suddividere il set di dati in training: test=0.8:0.2:
python ./FS_classification/testMain.py ./output/mq_targets.csv ./output/label.csv ./result_combine 800 0.2 True ./output/test_target.csv
NOTA: La Figura 5 mostra le informazioni sull'output. I file supplementari necessari per questo protocollo si trovano in HealthModel-mqTrans-v1-00.tar cartella (Supplementary Coding File 1).
Access restricted. Please log in or start a trial to view this content.
Risultati
Valutazione della vista mqTrans del dataset trascrittomico
Il codice di test utilizza undici algoritmi di selezione delle caratteristiche (FS) e sette classificatori per valutare in che modo la vista mqTrans generata del set di dati trascrittomico contribuisce all'attività di classificazione (Figura 6). Il set di dati del test è costituito da 317 adenocarcinoma del colon (COAD) dal database The Cancer Genome Atlas (TCGA)29. I pazienti COAD in sta...
Access restricted. Please log in or start a trial to view this content.
Discussione
La sezione 2 (Utilizzo di HealthModel pre-addestrato per generare le funzionalità mqTrans) del protocollo è il passaggio più critico all'interno di questo protocollo. Dopo aver preparato l'ambiente di lavoro computazionale nella sezione 1, la sezione 2 genera la vista mqTrans di un set di dati trascrittomico basato sul modello di riferimento di grandi dimensioni pre-addestrato. La sezione 3 è un esempio dimostrativo di selezione delle caratteristiche mqTrans generate per i rilevamenti di biomarcatori e le attività d...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato supportato dal team di innovazione tecnologica senior e junior (20210509055RQ), dai progetti scientifici e tecnologici della provincia di Guizhou (ZK2023-297), dalla Fondazione per la scienza e la tecnologia della Commissione sanitaria della provincia di Guizhou (gzwkj2023-565), dal progetto di scienza e tecnologia del Dipartimento dell'istruzione della provincia di Jilin (JJKH20220245KJ e JJKH20220226SK), dalla National Natural Science Foundation of China (U19A2061), dal Jilin Provincial Key Laboratory of Big Data Intelligent Computing (20180622002JC) e i Fondi di Ricerca di Base per le Università Centrali, JLU. Estendiamo il nostro più sincero apprezzamento al revisore e ai tre revisori anonimi per le loro critiche costruttive, che sono state determinanti nel migliorare sostanzialmente il rigore e la chiarezza di questo protocollo.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
Riferimenti
- Mutz, K. -O., Heilkenbrinker, A., Lönne, M., Walter, J. -G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516(2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684(2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352(2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528(2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. Analysis of Survival Data. , Chapman and Hall/CRC. London. (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866(2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307(2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119(2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239(2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334(2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338(2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , Springer. Cham. (2022).
- Muzio, G., O'Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738(2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559(2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5(2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883(2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582(2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801(2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138(2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308(2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon