É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Gerando a Exibição de Regulação Transcricional de Recursos Transcriptômicos para Tarefa de Previsão e Detecção de Biomarcadores Escuros em Pequenos Conjuntos de Dados
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para converter dados transcriptômicos em uma visão mqTrans, permitindo a identificação de biomarcadores escuros. Embora não expressos diferencialmente em análises transcriptômicas convencionais, esses biomarcadores exibem expressão diferencial na visão mqTrans. A abordagem serve como uma técnica complementar aos métodos tradicionais, revelando biomarcadores antes negligenciados.
Resumo
O transcriptoma representa os níveis de expressão de muitos genes em uma amostra e tem sido amplamente utilizado em pesquisas biológicas e na prática clínica. Os pesquisadores geralmente se concentraram em biomarcadores transcriptômicos com representações diferenciais entre um grupo fenotípico e um grupo controle de amostras. Este estudo apresentou uma estrutura de aprendizagem de rede de atenção gráfica multitarefa (GAT) para aprender as complexas interações intergênicas das amostras de referência. Um modelo de referência demonstrativo foi pré-treinado nas amostras saudáveis (HealthModel), que poderia ser usado diretamente para gerar a visão de regulação transcricional quantitativa baseada em modelo (mqTrans) dos transcriptomas de teste independentes. A visão mqTrans gerada dos transcriptomas foi demonstrada por tarefas de predição e detecção de biomarcadores escuros. O termo cunhado "biomarcador escuro" surgiu de sua definição de que um biomarcador escuro apresentava representação diferencial na visão mqTrans, mas nenhuma expressão diferencial em seu nível de expressão original. Um biomarcador escuro sempre foi negligenciado nos estudos tradicionais de detecção de biomarcadores devido à ausência de expressão diferencial. O código-fonte e o manual do pipeline HealthModelPipe podem ser baixados do http://www.healthinformaticslab.org/supp/resources.php.
Introdução
O transcriptoma consiste na expressão de todos os genes de uma amostra e pode ser perfilado por tecnologias de alto rendimento, como microarray e RNA-seq1. Os níveis de expressão de um gene em um conjunto de dados são chamados de característica transcriptômica, e a representação diferencial de uma característica transcriptômica entre os grupos fenótipo e controle define esse gene como um biomarcador desse fenótipo 2,3. Biomarcadores transcriptômicos têm sido extensivamente utilizados em investigações de diagnóstico dedoenças4, mecanismobiológico5, análise desobrevida6,7, etc.
Os padrões de atividade gênica nos tecidos sadios carregam informações cruciais sobre as vidas 8,9. Esses padrões oferecem insights inestimáveis e funcionam como referências ideais para a compreensão das complexas trajetórias de desenvolvimento das doençasbenignas10,11 eletais12. Os genes interagem entre si, e os transcriptomas representam os níveis finais de expressão após suas complicadas interações. Tais padrões são formulados como rede de regulação transcricional13 e rede de metabolismo14, etc. A expressão de RNAs mensageiros (mRNAs) pode ser regulada transcricionalmente por fatores de transcrição (FTs) e RNAs não codificadores intergênicos longos (lincRNAs)15,16,17. A análise convencional de expressão diferencial ignorou interações gênicas tão complexas com a suposição de independência inter-características18,19.
Avanços recentes em redes neurais de grafos (GNNs) demonstram um potencial extraordinário na extração de informações importantes de dados baseados em OMIC para estudos de câncer20, por exemplo, identificando módulos de co-expressão21. A capacidade inata dos GNNs os torna ideais para modelar as intrincadas relações e dependências entre genes22,23.
Estudos biomédicos geralmente se concentram em prever com precisão um fenótipo contra o grupo controle. Tais tarefas são comumente formuladas como classificações binárias 24,25,26. Aqui, os dois rótulos de classe são tipicamente codificados como 1 e 0, verdadeiro e falso, ou mesmo positivo e negativo27.
Este estudo teve como objetivo fornecer um protocolo fácil de usar para gerar a visão de regulação transcricional (mqTrans) de um conjunto de dados do transcriptoma baseado no modelo de referência de rede grafo-atenção (GAT) pré-treinado. O framework GAT multitarefa de um trabalho publicadoanteriormente26 foi usado para transformar características transcriptômicas para características mqTrans. Um grande conjunto de dados de transcriptomas saudáveis da plataforma Xena28 da Universidade da Califórnia, Santa Cruz (UCSC) foi usado para pré-treinar o modelo de referência (HealthModel), que mediu quantitativamente as regulamentações de transcrição dos fatores regulatórios (FTs e lincRNAs) para os mRNAs alvo. A visualização mqTrans gerada pode ser usada para construir modelos de previsão e detectar biomarcadores escuros. Este protocolo utiliza o conjunto de dados de pacientes com adenocarcinoma de cólon (COAD) do banco de dados The Cancer Genome Atlas (TCGA)29 como exemplo ilustrativo. Nesse contexto, os pacientes nos estágios I ou II são categorizados como amostras negativas, enquanto aqueles nos estágios III ou IV são considerados positivos. As distribuições de biomarcadores escuros e tradicionais entre os 26 tipos de câncer TCGA também são comparadas.
Descrição do pipeline HealthModel
A metodologia empregada neste protocolo baseia-se no referencial previamentepublicado26, conforme descrito na Figura 1. Para começar, os usuários precisam preparar o conjunto de dados de entrada, alimentá-lo no pipeline HealthModel proposto e obter recursos mqTrans. Instruções detalhadas de preparação de dados são fornecidas na seção 2 da seção de protocolo. Posteriormente, os usuários têm a opção de combinar os recursos do mqTrans com os recursos transcriptômicos originais ou prosseguir apenas com os recursos do mqTrans gerados. O conjunto de dados produzido é então submetido a um processo de seleção de recursos, com os usuários tendo a flexibilidade de escolher seu valor preferido para k em validação cruzada k-fold para classificação. A principal métrica de avaliação utilizada neste protocolo é a acurácia.
O HealthModel26 categoriza as características transcriptômicas em três grupos distintos: TF (Transcription Factor), lincRNA (Long Intergenic non-coding RNA) e mRNA (RNA mensageiro). As características dos FT são definidas com base nas anotações disponíveis no Atlas de Proteínas Humanas30,31. Este trabalho utiliza as anotações de lincRNAs do conjunto de dados GTEx32. Genes pertencentes às vias de terceiro nível no banco de dados KEGG33 são considerados como características de RNAm. É importante notar que, se uma característica de RNAm exibe funções regulatórias para um gene alvo, conforme documentado no banco de dados TRRUST34, ela é reclassificada na classe TF.
Este protocolo também gera manualmente os dois arquivos de exemplo para as IDs de gene de fatores regulatórios (regulatory_geneIDs.csv) e mRNA alvo (target_geneIDs.csv). A matriz de distância par a par entre as características regulatórias (FTs e lincRNAs) é calculada pelos coeficientes de correlação de Pearson e agrupada pela popular ferramenta de análise de rede de coexpressão gênica ponderada (WGCNA)36 (adjacent_matrix.csv). Os usuários podem utilizar diretamente o pipeline HealthModel junto com esses arquivos de configuração de exemplo para gerar a exibição mqTrans de um conjunto de dados transcriptômico.
Detalhes técnicos do HealthModel
HealthModel representa as intrincadas relações entre FTs e lincRNAs como um grafo, com as características de entrada servindo como os vértices denotados por V e uma matriz de borda intervértice designada como E. Cada amostra é caracterizada por características regulatórias K , simbolizadas como VK×1. Especificamente, o conjunto de dados abrangeu 425 FTs e 375 lincRNAs, resultando em uma dimensionalidade da amostra de K = 425 + 375 = 800. Para estabelecer a matriz de borda E, este trabalho empregou a popular ferramenta WGCNA35. O peso par a par ligando dois vértices representados como 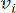 e
e 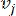 , é determinado pelo coeficiente de correlação de Pearson. A rede de regulação gênica exibe uma topologia livre de escalas36, caracterizada pela presença de genes hub com papéis funcionais fundamentais. Calculamos a correlação entre duas feições ou vértices,
, é determinado pelo coeficiente de correlação de Pearson. A rede de regulação gênica exibe uma topologia livre de escalas36, caracterizada pela presença de genes hub com papéis funcionais fundamentais. Calculamos a correlação entre duas feições ou vértices, 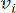 e
e 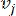 , usando a medida de sobreposição topológica (TOM) da seguinte forma:
, usando a medida de sobreposição topológica (TOM) da seguinte forma:
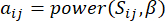 (1)
(1)
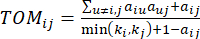 (2)
(2)
O β de limite suave é calculado usando a função 'pickSoft Threshold' do pacote WGCNA. A função exponencial de potência aij é aplicada, onde 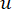 representa um gene excluindo i e j, e
representa um gene excluindo i e j, e 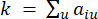 representa a conectividade de vértices. O WGCNA agrupa os perfis de expressão das características transcriptômicas em múltiplos módulos usando uma medida de dissimilaridade comumente empregada (
representa a conectividade de vértices. O WGCNA agrupa os perfis de expressão das características transcriptômicas em múltiplos módulos usando uma medida de dissimilaridade comumente empregada (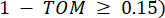 37.
37.
O framework HealthModel foi originalmente projetado como uma arquitetura de aprendizagem multitarefa26. Este protocolo utiliza apenas a tarefa de pré-treinamento do modelo para a construção da visão transcriptômica mqTrans. O usuário pode optar por refinar ainda mais o HealthModel pré-treinado sob a rede de atenção gráfica multitarefa com amostras transcriptômicas específicas de tarefas adicionais.
Detalhes técnicos da seleção e classificação de recursos
O pool de seleção de recursos implementa onze algoritmos de seleção de recursos (FS). Entre eles, três são algoritmos FS baseados em filtro: selecionando os melhores recursos K usando o Coeficiente de Informação Máxima (SK_mic), selecionando os recursos K com base no FPR do MIC (SK_fpr) e selecionando os recursos K com a maior taxa de descoberta falsa do MIC (SK_fdr). Além disso, três algoritmos FS baseados em árvore avaliam características individuais usando uma árvore de decisão com o índice de Gini (DT_gini), árvores de decisão adaptativas impulsionadas (AdaBoost) e floresta aleatória (RF_fs). O pool também incorpora dois métodos wrapper: eliminação de feições recursivas com o classificador vetorial de suporte linear (RFE_SVC) e eliminação de recursos recursivos com o classificador de regressão logística (RFE_LR). Finalmente, dois algoritmos de incorporação são incluídos: classificador linear SVC com os valores de importância de característica L1 (lSVC_L1) mais bem classificados e classificador de regressão logística com os valores de importância de característica L1 (LR_L1).
O pool de classificadores emprega sete classificadores diferentes para construir modelos de classificação. Esses classificadores compreendem máquina de vetor de suporte linear (SVC), Gaussian Naïve Bayes (GNB), classificador de regressão logística (LR), k-vizinho mais próximo, com k definido como 5 por padrão (KNN), XGBoost, floresta aleatória (RF) e árvore de decisão (DT).
A divisão aleatória do conjunto de dados no trem: subconjuntos de teste pode ser definida na linha de comando. O exemplo demonstrado usa a razão trem: teste = 8: 2.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Observação : o protocolo a seguir descreve os detalhes do procedimento analítico de informática e comandos Python dos módulos principais. A Figura 2 ilustra os três passos principais com exemplos de comandos utilizados neste protocolo e remete a trabalhos publicadosanteriormente26,38 para maiores detalhes técnicos. Faça o seguinte protocolo em uma conta de usuário normal em um sistema de computador e evite usar a conta de administrador ou root. Este é um protocolo computacional e não tem fatores biomédicos perigosos.
1. Preparar o ambiente Python
- Crie um ambiente virtual.
- Este estudo utilizou a linguagem de programação Python e um ambiente virtual Python (VE) com Python 3.7. Siga estas etapas (Figura 3A):
conda create -n healthmodel python=3.7
conda create é o comando para criar um novo VE. O parâmetro -n especifica o nome do novo ambiente, neste caso, healthmodel. E python=3.7 especifica a versão do Python a ser instalada. Escolha qualquer nome preferido e versão do Python que suporte o comando acima. - Depois de executar o comando, a saída é semelhante à Figura 3B. Digite y e aguarde a conclusão do processo.
- Este estudo utilizou a linguagem de programação Python e um ambiente virtual Python (VE) com Python 3.7. Siga estas etapas (Figura 3A):
- Ativar o ambiente virtual
- Na maioria dos casos, ative o VE criado com o seguinte comando (Figura 3C):
conda ativar healthmodel - Siga as instruções específicas da plataforma para a ativação do VE, se algumas plataformas exigirem que o usuário carregue os arquivos de configuração específicos da plataforma para ativação.
- Na maioria dos casos, ative o VE criado com o seguinte comando (Figura 3C):
- Instalar PyTorch 1.13.1
- PyTorch é um pacote Python popular para algoritmos de inteligência artificial (IA). Use o PyTorch 1.13.1, baseado na plataforma de programação CUDA 11.7 GPU, como exemplo. Encontre outras versões em https://pytorch.org/get-started/previous-versions/. Use o seguinte comando (Figura 3D):
pip3 instalar tocha torchvision torchaudio
Observação : usando PyTorch versão 1.12 ou mais recente é altamente recomendado. Caso contrário, instalar o pacote necessário torch_geometric pode ser um desafio, como observado no site oficial da torch_geometric: https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html.
- PyTorch é um pacote Python popular para algoritmos de inteligência artificial (IA). Use o PyTorch 1.13.1, baseado na plataforma de programação CUDA 11.7 GPU, como exemplo. Encontre outras versões em https://pytorch.org/get-started/previous-versions/. Use o seguinte comando (Figura 3D):
- Instalar pacotes adicionais para tocha-geométrica
- Seguindo as diretrizes em https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html, instale os seguintes pacotes: torch_scatter, torch_sparse, torch_cluster e torch_spline_conv usando o comando (Figura 3E):
pip install pyg_lib torch_scatter torch_sparse torch_cluster torch_spline_conv -f https://data.pyg.org/whl/torch-1.13.0+cu117.html
- Seguindo as diretrizes em https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html, instale os seguintes pacotes: torch_scatter, torch_sparse, torch_cluster e torch_spline_conv usando o comando (Figura 3E):
- Instale o pacote tocha-geométrico .
- Este estudo requer uma versão específica, 2.2.0, do pacote tocha-geométrico . Execute o comando (Figura 3F):
pip instalar torch_geometric==2.2.0
- Este estudo requer uma versão específica, 2.2.0, do pacote tocha-geométrico . Execute o comando (Figura 3F):
- Instale outros pacotes.
- Pacotes como pandas geralmente estão disponíveis por padrão. Caso contrário, instale-os usando o comando pip. Por exemplo, para instalar pandas e xgboost, execute:
pip instalar pandas
pip instalar xgboost
- Pacotes como pandas geralmente estão disponíveis por padrão. Caso contrário, instale-os usando o comando pip. Por exemplo, para instalar pandas e xgboost, execute:
2. Usando o HealthModel pré-treinado para gerar os recursos do mqTrans
- Faça o download do código e do modelo pré-treinado.
- Baixe o código e o HealthModel pré-treinado no site: http://www.healthinformaticslab.org/supp/resources.php, que é chamado de HealthModel-mqTrans-v1-00.tar.gz (Figura 4A). O arquivo baixado pode ser descompactado para um caminho especificado pelo usuário. A formulação detalhada e os dados de apoio do protocolo implementado podem ser encontrados em26.
- Introduza os parâmetros para executar HealthModel.
- Em primeiro lugar, altere o diretório de trabalho para a pasta HealthModel-mqTrans na linha de comando. Use a seguinte sintaxe para executar o código:
Python main.py
Os detalhes sobre cada parâmetro e as pastas de dados, modelo e saída são os seguintes:
pasta de dados: Esta é a pasta de dados de origem e cada arquivo de dados está no formato csv. Essa pasta de dados tem dois arquivos (consulte descrições detalhadas nas etapas 2.3 e 2.4). Esses arquivos precisam ser substituídos por dados pessoais.
data.csv: O arquivo de matriz transcriptômica. A primeira linha lista os IDs de recurso (ou gene) e a primeira coluna fornece os IDs de exemplo. A lista de genes inclui os fatores regulatórios (FTs e lincRNAs) e os genes de RNAm regulados.
label.csv: O arquivo de rótulo de exemplo. A primeira coluna lista os IDs de exemplo e a coluna com o nome "label" fornece o rótulo de exemplo.
pasta model: A pasta para salvar informações sobre o modelo:
HealthModel.pth: O HealthModel pré-treinado.
regulatory_geneIDs.csv: As identificações gênicas reguladoras utilizadas neste estudo.
target_geneIDs.csv: Os genes-alvo utilizados neste estudo.
adjacent_matrix.csv: A matriz adjacente de genes reguladores.
pasta de saída: Os arquivos de saída são gravados nesta pasta, criada pelo código.
test_target.csv: Valor de expressão gênica de genes-alvo após normalização e imputação Z.
pred_target.csv: Valor predito de expressão gênica de genes-alvo.
mq_target.csv: Valor predito de expressão gênica de genes-alvo.
- Em primeiro lugar, altere o diretório de trabalho para a pasta HealthModel-mqTrans na linha de comando. Use a seguinte sintaxe para executar o código:
- Prepare o arquivo de matriz transcriptômica no formato csv.
- Cada linha representa uma amostra e cada coluna representa um gene (Figura 4B). Nomeie o arquivo de matriz de dados transcriptômicos como data.csv na pasta de dados .
Observação : esse arquivo pode ser gerado salvando manualmente uma matriz de dados no formato .csv de software como o Microsoft Excel. A matriz transcriptômica também pode ser gerada por programação computacional.
- Cada linha representa uma amostra e cada coluna representa um gene (Figura 4B). Nomeie o arquivo de matriz de dados transcriptômicos como data.csv na pasta de dados .
- Prepare o arquivo de rótulo no formato csv.
- Semelhante ao arquivo de matriz transcriptômica, nomeie o arquivo de rótulo como label.csv na pasta de dados (Figura 4C).
Observação : a primeira coluna fornece os nomes de exemplo e o rótulo de classe de cada exemplo é fornecido na coluna intitulada rótulo. O valor 0 na coluna do rótulo significa que esta amostra é negativa, 1 significa uma amostra positiva.
- Semelhante ao arquivo de matriz transcriptômica, nomeie o arquivo de rótulo como label.csv na pasta de dados (Figura 4C).
- Gere os recursos mqTrans.
- Execute o comando a seguir para gerar os recursos mqTrans e obter as saídas mostradas na Figura 4D. Os recursos mqTrans são gerados como o arquivo ./output/mq_targets.csv, e o arquivo label é salvo novamente como o arquivo ./output/label.csv. Para a conveniência de análises adicionais, os valores de expressão originais dos genes de RNAm também são extraídos como o arquivo ./output/ test_target.csv.
python ./Get_mqTrans/código/main.py ./data ./Get_mqTrans/modelo ./output
- Execute o comando a seguir para gerar os recursos mqTrans e obter as saídas mostradas na Figura 4D. Os recursos mqTrans são gerados como o arquivo ./output/mq_targets.csv, e o arquivo label é salvo novamente como o arquivo ./output/label.csv. Para a conveniência de análises adicionais, os valores de expressão originais dos genes de RNAm também são extraídos como o arquivo ./output/ test_target.csv.
3. Selecione os recursos do mqTrans
- Sintaxe do código de seleção de recurso
- Em primeiro lugar, altere o diretório de trabalho para a pasta HealthModel-mqTrans . Use a seguinte sintaxe:
python ./FS_classification/testMain.py
Os detalhes de cada parâmetro são os seguintes:
in-data-file: O arquivo de dados de entrada
in-label-file: O rótulo do arquivo de dados de entrada
pasta de saída: Dois arquivos de saída são salvos nessa pasta, incluindo Output-score.xlsx (o método de seleção de recursos e a precisão do classificador correspondente) e Output-SelectedFeatures.xlsx (os nomes de recursos selecionados para cada algoritmo de seleção de recursos).- select_feature_number: selecione o número de recursos, variando de 1 ao número dos recursos do arquivo de dados.
- test_size: Defina a proporção da amostra de teste para dividir. Por exemplo, 0,2 significa que o conjunto de dados de entrada é dividido aleatoriamente nos subconjuntos de teste: trem pela proporção de 0,8:0,2.
- combine: Se true, combine dois arquivos de dados juntos para a seleção de recursos, ou seja, os valores de expressão originais e os recursos mqTrans. Se false, use apenas um arquivo de dados para a seleção de recursos, ou seja, os valores de expressão originais ou os recursos mqTrans.
- combinar arquivo: se combinar for true, forneça esse nome de arquivo para salvar a matriz de dados combinada.
NOTA: Este pipeline visa demonstrar como os recursos mqTrans gerados são executados em tarefas de classificação e usa diretamente o arquivo gerado pela seção 2 para as operações a seguir.
- Em primeiro lugar, altere o diretório de trabalho para a pasta HealthModel-mqTrans . Use a seguinte sintaxe:
- Execute o algoritmo de seleção de recursos para a seleção de recursos mqTrans.
- Ative combinar =False se o usuário selecionar recursos mqTrans ou recursos originais.
- Em primeiro lugar, selecione 800 recursos originais e divida o conjunto de dados em trem: test=0.8:0.2:
python ./FS_classification/testMain.py ./output/test_target.csv ./output/label.csv ./result 800 0.2 Falso - Turn combine =True, se o usuário quiser combinar os recursos mqTrans com os valores de expressão originais para selecionar recursos. Aqui, o exemplo demonstrativo é selecionar 800 recursos e dividir o conjunto de dados em trem: test=0.8:0.2:
python ./FS_classification/testMain.py ./output/mq_targets.csv ./output/label.csv ./result_combine 800 0.2 True ./output/test_target.csv
NOTA: A Figura 5 mostra as informações de saída. Os arquivos suplementares necessários para este protocolo estão em HealthModel-mqTrans-v1-00.tar pasta (Arquivo de codificação suplementar 1).
Access restricted. Please log in or start a trial to view this content.
Resultados
Avaliação da visão mqTrans do conjunto de dados transcriptômicos
O código de teste usa onze algoritmos de seleção de recursos (FS) e sete classificadores para avaliar como a visão mqTrans gerada do conjunto de dados transcriptômicos contribui para a tarefa de classificação (Figura 6). O conjunto de dados do teste consiste em 317 adenocarcinoma de cólon (COAD) do banco de dados The Cancer Genome Atlas (TCGA)29. Os pacientes com COAD nos ...
Access restricted. Please log in or start a trial to view this content.
Discussão
A seção 2 (Use o HealthModel pré-treinado para gerar os recursos mqTrans) do protocolo é a etapa mais crítica dentro desse protocolo. Depois de preparar o ambiente de trabalho computacional na seção 1, a seção 2 gera a visualização mqTrans de um conjunto de dados transcriptômicos baseado no modelo de referência grande pré-treinado. A seção 3 é um exemplo demonstrativo da seleção dos recursos mqTrans gerados para detecções de biomarcadores e tarefas de previsão. Os usuários podem realizar outras an?...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho foi apoiado pela Equipe de Inovação Tecnológica Sênior e Júnior (20210509055RQ), Projetos Provinciais de Ciência e Tecnologia de Guizhou (ZK2023-297), pela Fundação de Ciência e Tecnologia da Comissão de Saúde da Província de Guizhou (gzwkj2023-565), pelo Projeto de Ciência e Tecnologia do Departamento de Educação da Província de Jilin (JJKH20220245KJ e JJKH20220226SK), pela Fundação Nacional de Ciências Naturais da China (U19A2061), pelo Laboratório Chave Provincial de Jilin de Computação Inteligente de Big Data (20180622002JC), e os Fundos de Pesquisa Fundamental para as Universidades Centrais, JLU. Estendemos nosso mais sincero agradecimento ao editor de revisão e aos três revisores anônimos por suas críticas construtivas, que foram fundamentais para aumentar substancialmente o rigor e a clareza deste protocolo.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
Referências
- Mutz, K. -O., Heilkenbrinker, A., Lönne, M., Walter, J. -G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516(2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684(2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352(2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528(2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. Analysis of Survival Data. , Chapman and Hall/CRC. London. (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866(2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307(2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119(2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239(2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334(2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338(2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , Springer. Cham. (2022).
- Muzio, G., O'Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738(2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559(2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5(2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883(2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582(2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801(2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138(2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308(2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados