JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
작은 데이터 세트에서 예측 작업 및 다크 바이오마커 검출을 위한 전사체 특징의 전사체 조절 보기 생성
요약
여기에서는 전사체 데이터를 mqTrans 보기로 변환하여 다크 바이오마커를 식별할 수 있는 프로토콜을 소개합니다. 기존의 전사체 분석에서는 차등적으로 발현되지 않지만, 이러한 바이오마커는 mqTrans 보기에서 차등 발현을 나타냅니다. 이 접근법은 전통적인 방법을 보완하는 기술 역할을 하며, 이전에 간과되었던 바이오마커를 밝혀냅니다.
초록
전사체는 샘플에서 많은 유전자의 발현 수준을 나타내며 생물학 연구 및 임상 실습에서 널리 사용되었습니다. 연구자들은 일반적으로 표현형 그룹과 샘플의 대조군 사이에 차이 표현을 가진 전사체 바이오마커에 초점을 맞췄습니다. 이 연구는 참조 샘플의 복잡한 유전자 간 상호 작용을 학습하기 위한 멀티태스킹 그래프-주의 네트워크(GAT) 학습 프레임워크를 제시했습니다. 시범적 참조 모델은 건강한 샘플(HealthModel)에 대해 사전 학습되었으며, 이는 독립적인 테스트 전사체의 모델 기반 정량적 전사 조절(mqTrans) 보기를 생성하는 데 직접 사용할 수 있습니다. 생성된 전사체의 mqTrans 보기는 예측 작업 및 다크 바이오마커 검출에 의해 입증되었습니다. "다크 바이오마커(dark biomarker)"라는 용어는 다크 바이오마커가 mqTrans 뷰에서 차등 표현을 나타내지만 원래 발현 수준에서는 차등 발현을 나타내지 않는다는 정의에서 비롯되었습니다. 어두운 바이오마커는 차등 발현이 없기 때문에 전통적인 바이오마커 검출 연구에서 항상 간과되었습니다. 파이프라인 HealthModelPipe의 소스 코드 및 매뉴얼은 http://www.healthinformaticslab.org/supp/resources.php 에서 다운로드할 수 있습니다.
서문
전사체는 샘플에 있는 모든 유전자의 발현으로 구성되며 마이크로어레이 및 RNA-seq1과 같은 고처리량 기술로 프로파일링할 수 있습니다. 데이터 세트에서 한 유전자의 발현 수준을 전사체 특징이라고 하며, 표현형과 대조군 간의 전사체 특징의 차등 표현은 이 유전자를 이 표현형 2,3의 바이오마커로 정의합니다. 전사체 바이오마커는 질병 진단4, 생물학적 메커니즘5, 생존 분석6,7 등의 연구에 광범위하게 활용되어 왔다.
건강한 조직의 유전자 활동 패턴은 생명에 대한 중요한 정보를 담고 있다 8,9. 이러한 패턴은 귀중한 통찰력을 제공하며, 양성 질환(benign disorders)10,11 및 치명적 질환(lethal diseases)12의 복잡한 발달 궤적을 이해하기 위한 이상적인 참고 자료 역할을 한다. 유전자는 서로 상호 작용하며, 전사체는 복잡한 상호 작용 후 최종 발현 수준을 나타냅니다. 이러한 패턴은 전사 조절 네트워크(transcriptional regulation network)13 및 대사 네트워크(metabolism network)14 등으로 공식화된다. 메신저 RNA(mRNA)의 발현은 전사 인자(TF) 및 긴 유전자 간 비코딩 RNA(lincRNA)에 의해 전사적으로 조절될 수 있습니다15,16,17. 종래의 차등 발현 분석은 특징간 독립성(inter-feature independence)을 가정한 이러한 복잡한 유전자 상호작용을 무시하였다18,19.
그래프 신경망(GNN)의 최근 발전은 암 연구(20)를 위한 OMIC 기반 데이터로부터 중요한 정보를 추출하는 데 있어 비범한 잠재력을 보여주는데, 예를 들어, 공발현 모듈(21)을 식별한다. GNN의 타고난 능력은 유전자22,23 사이의 복잡한 관계와 의존성을 모델링하는 데 이상적입니다.
생물의학 연구는 종종 대조군에 대한 표현형을 정확하게 예측하는 데 중점을 둡니다. 이러한 작업은 일반적으로 이진 분류(binary classifications)24,25,26으로 공식화된다. 여기서 두 클래스 레이블은 일반적으로 1과 0, true 및 false 또는 양수 및 음수27로 인코딩됩니다.
이 연구는 사전 훈련된 GAT(Graph-Attention Network) 참조 모델을 기반으로 전사체 데이터 세트의 전사 조절(mqTrans) 보기를 생성하기 위한 사용하기 쉬운 프로토콜을 제공하는 것을 목표로 했습니다. 이전에 공개된 작업(26 )으로부터의 멀티태스킹 GAT 프레임워크를 사용하여 전사체 특징을 mqTrans 특징들로 변환하였다. UCSC(University of California, Santa Cruz) Xena 플랫폼28 의 건강한 전사체에 대한 대규모 데이터 세트를 사용하여 조절 인자(TF 및 lincRNA)에서 표적 mRNA까지의 전사 조절을 정량적으로 측정한 참조 모델(HealthModel)을 사전 훈련했습니다. 생성된 mqTrans 뷰는 예측 모델을 구축하고 다크 바이오마커를 감지하는 데 사용할 수 있습니다. 이 프로토콜은 TCGA(The Cancer Genome Atlas) 데이터베이스(29 )의 결장 선암종(COAD) 환자 데이터 세트를 예시로 사용합니다. 이러한 맥락에서 I기 또는 II기의 환자는 음성 샘플로 분류되고 III 또는 IV기의 환자는 양성 샘플로 간주됩니다. 26개의 TCGA 암 유형에 대한 어두운 바이오마커와 전통적인 바이오마커의 분포도 비교됩니다.
HealthModel 파이프라인에 대한 설명
이 프로토콜에 사용된 방법론은 그림 1에 요약된 바와 같이 이전에 발표된 프레임워크26을 기반으로 합니다. 시작하려면 사용자가 입력 데이터 세트를 준비하고, 제안된 HealthModel 파이프라인에 공급하고, mqTrans 기능을 가져와야 합니다. 자세한 데이터 준비 지침은 프로토콜 섹션의 섹션 2에 나와 있습니다. 그 후, 사용자는 mqTrans 기능을 원래 전사체 기능과 결합하거나 생성된 mqTrans 기능만 진행할 수 있습니다. 생성된 데이터 세트는 기능 선택 프로세스를 거치며, 사용자는 분류를 위한 k-폴드 교차 검증에서 k에 대해 선호하는 값을 유연하게 선택할 수 있습니다. 이 프로토콜에서 사용되는 기본 평가 메트릭은 정확도입니다.
HealthModel26은 전사체 특징을 TF(전사 인자), lincRNA(긴 유전자 간 비코딩 RNA) 및 mRNA(메신저 RNA)의 세 가지 그룹으로 분류합니다. TF 특징은 Human Protein Atlas30,31에서 사용할 수 있는 주석을 기반으로 정의됩니다. 이 작업은 GTEx 데이터 세트32의 lincRNA 주석을 활용합니다. KEGG 데이터베이스(33)에서 3단계 경로에 속하는 유전자는 mRNA 특징으로서 고려된다. mRNA 특징이 TRRUST 데이터베이스(34)에 문서화된 바와 같이 표적 유전자에 대한 조절 역할을 나타내면, TF 클래스로 재분류된다는 점에 주목할 필요가 있다.
이 프로토콜은 또한 조절 인자(regulatory_geneIDs.csv)와 표적 mRNA(target_geneIDs.csv)의 유전자 ID에 대한 두 개의 예제 파일을 수동으로 생성합니다. 조절 특징(TF 및 lincRNA) 간의 쌍별 거리 매트릭스는 Pearson 상관 계수에 의해 계산되고 널리 사용되는 도구인 WGCNA(Weighted Gene Co-Expression Network Analysis)36 (adjacent_matrix.csv)에 의해 클러스터링됩니다. 사용자는 이러한 예제 구성 파일과 함께 HealthModel 파이프라인을 직접 활용하여 전사체 데이터 세트의 mqTrans 보기를 생성할 수 있습니다.
HealthModel의 기술적 세부 사항
HealthModel은 TF와 lincRNA 간의 복잡한 관계를 그래프로 나타내며, 입력 특징은 V로 표시된 꼭짓점과 E로 지정된 꼭짓점 간 가장자리 행렬 역할을 합니다. 각 샘플은 VK×1로 기호화된 K 조절 특징이 특징입니다. 특히, 데이터 세트에는 425개의 TF와 375개의 lincRNA가 포함되어 있어 K = 425 + 375 = 800의 샘플 차원을 얻을 수 있었습니다. 에지 매트릭스 E를 확립하기 위해 이 작업에서는 널리 사용되는 도구인 WGCNA35를 사용했습니다. 및 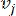 로 표시된
로 표시된 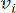 두 꼭짓점을 연결하는 쌍별 가중치는 Pearson 상관 계수에 의해 결정됩니다. 유전자 조절 네트워크(gene regulatory network)는 중추적인 기능적 역할을 하는 허브 유전자(hub gene)의 존재를 특징으로 하는 비늘 없는 토폴로지(scale-free topology)(36)를 나타낸다. 두 피처 또는 꼭짓점 간의 상관 관계를 계산하고
두 꼭짓점을 연결하는 쌍별 가중치는 Pearson 상관 계수에 의해 결정됩니다. 유전자 조절 네트워크(gene regulatory network)는 중추적인 기능적 역할을 하는 허브 유전자(hub gene)의 존재를 특징으로 하는 비늘 없는 토폴로지(scale-free topology)(36)를 나타낸다. 두 피처 또는 꼭짓점 간의 상관 관계를 계산하고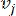 ,
, 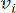 다음과 같이 TOM(Topological Overlap Measure)을 사용합니다.
다음과 같이 TOM(Topological Overlap Measure)을 사용합니다.
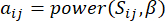 (1)
(1)
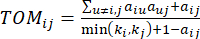 (2)
(2)
소프트 임계값 β는 WGCNA 패키지의 'pickSoft Threshold' 함수를 사용하여 계산됩니다. 거듭제곱 지수 함수 a ij가 적용되며, 여기서 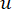 는 i와 j를 제외한 유전자를 나타내고
는 i와 j를 제외한 유전자를 나타내고 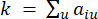 꼭짓점 연결을 나타냅니다. WGCNA는 일반적으로 사용되는 비유사성 측정(dissimilarity measure)을 사용하여 전사체 특징의 발현 프로파일을 여러 모듈로 클러스터링합니다(
꼭짓점 연결을 나타냅니다. WGCNA는 일반적으로 사용되는 비유사성 측정(dissimilarity measure)을 사용하여 전사체 특징의 발현 프로파일을 여러 모듈로 클러스터링합니다(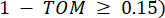 37.
37.
HealthModel 프레임워크는 원래 멀티태스킹 학습 아키텍처(multitask learning architecture)로 설계되었다(26). 이 프로토콜은 트랜스크립토믹 mqTrans 보기의 구성을 위해 모델 사전 학습 작업만 사용합니다. 사용자는 추가 작업별 전사체 샘플을 사용하여 멀티태스킹 그래프 주의 네트워크 하에서 사전 훈련된 HealthModel을 더욱 구체화하도록 선택할 수 있습니다.
기능 선택 및 분류에 대한 기술적 세부 사항
기능 선택 풀은 11개의 FS(기능 선택) 알고리즘을 구현합니다. 그 중 SK_mic(Maximal Information Coefficient)를 사용하여 K 최상의 특징을 선택하고, MIC의 FPR을 기반으로 K 특징을 선택하고(SK_fpr), MIC의 오탐지율이 가장 높은 K 특징을 선택(SK_fdr)하는 세 가지 필터 기반 FS 알고리즘입니다. 또한 세 가지 트리 기반 FS 알고리즘은 지니 지수(DT_gini), 적응형 부스트 의사 결정 트리(AdaBoost) 및 랜덤 포레스트(RF_fs)가 있는 의사 결정 트리를 사용하여 개별 기능을 평가합니다. 또한 풀은 선형 서포트 벡터 분류자(RFE_SVC)를 사용한 재귀적 기능 제거와 로지스틱 회귀 분류자(RFE_LR)를 사용한 재귀적 기능 제거라는 두 가지 래퍼 방법을 통합합니다. 마지막으로, 최상위 L1 기능 중요도 값(lSVC_L1)이 있는 선형 SVC 분류자와 최상위 L1 기능 중요도 값(LR_L1)이 있는 로지스틱 회귀 분류자의 두 가지 임베딩 알고리즘이 포함됩니다.
분류자 풀은 7개의 서로 다른 분류자를 사용하여 분류 모델을 빌드합니다. 이러한 분류기는 선형 서포트 벡터 머신(SVC), 가우스 나이브 베이즈(GNB), 로지스틱 회귀 분류기(LR), k-최근접이웃(k가 기본적으로 5로 설정됨(KNN), XGBoost, 랜덤 포레스트(RF) 및 의사 결정 트리(DT)로 구성됩니다.
데이터셋을 train: test 서브셋으로 임의로 분할하는 것은 명령줄에서 설정할 수 있습니다. 시연된 예제에서는 train: test = 8:2의 비율을 사용합니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
참고: 다음 프로토콜은 주요 모듈의 정보학 분석 절차 및 Python 명령에 대한 세부 정보를 설명합니다. 그림 2는 이 프로토콜에서 사용되는 예제 명령과 함께 세 가지 주요 단계를 설명하며 자세한 기술 세부 사항은 이전에 발표된 저작물26,38을 참조하십시오. 컴퓨터 시스템의 일반 사용자 계정으로 다음 프로토콜을 수행하고 관리자 또는 루트 계정을 사용하지 마십시오. 이것은 계산 프로토콜이며 생물 의학적 위험 요소가 없습니다.
1. Python 환경 준비
- 가상 환경을 만듭니다.
- 이 연구는 Python 3.7에서 Python 프로그래밍 언어와 Python 가상 환경(VE)을 사용했습니다. 다음 단계를 수행합니다(그림 3A).
conda create -n healthmodel 파이썬=3.7
conda create 는 새 VE를 만드는 명령입니다. 매개 변수 -n은 새 환경의 이름(이 경우 healthmodel)을 지정합니다. 그리고 python=3.7 은 설치할 Python 버전을 지정합니다. 위의 명령을 지원하는 원하는 이름과 Python 버전을 선택합니다. - 명령을 실행한 후 출력은 그림 3B와 유사합니다. y 를 입력하고 프로세스가 완료될 때까지 기다립니다.
- 이 연구는 Python 3.7에서 Python 프로그래밍 언어와 Python 가상 환경(VE)을 사용했습니다. 다음 단계를 수행합니다(그림 3A).
- 가상 환경 활성화
- 대부분의 경우 다음 명령을 사용하여 생성된 VE를 활성화합니다(그림 3C).
conda 활성화 healthmodel - 일부 플랫폼에서 사용자가 활성화를 위해 플랫폼별 구성 파일을 업로드해야 하는 경우 VE 활성화에 대한 플랫폼별 지침을 따릅니다.
- 대부분의 경우 다음 명령을 사용하여 생성된 VE를 활성화합니다(그림 3C).
- PyTorch 1.13.1 설치
- PyTorch는 인공 지능(AI) 알고리즘을 위한 인기 있는 Python 패키지입니다. CUDA 11.7 GPU 프로그래밍 플랫폼을 기반으로 하는 PyTorch 1.13.1을 예로 사용합니다. https://pytorch.org/get-started/previous-versions/ 에서 다른 버전을 찾으십시오. 다음 명령을 사용합니다(그림 3D).
pip3 토치 설치 torchvision torchaudio
참고: PyTorch 버전 1.12 이상을 사용하는 것이 좋습니다. 그렇지 않으면 공식 torch_geometric 웹 사이트 https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html 에 명시된 것처럼 필요한 패키지를 torch_geometric 설치하는 것이 어려울 수 있습니다.
- PyTorch는 인공 지능(AI) 알고리즘을 위한 인기 있는 Python 패키지입니다. CUDA 11.7 GPU 프로그래밍 플랫폼을 기반으로 하는 PyTorch 1.13.1을 예로 사용합니다. https://pytorch.org/get-started/previous-versions/ 에서 다른 버전을 찾으십시오. 다음 명령을 사용합니다(그림 3D).
- torch-geometric을 위한 추가 패키지 설치
- https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html 의 지침에 따라 다음 명령을 사용하여 torch_scatter, torch_sparse, torch_cluster 및 torch_spline_conv 패키지를 설치합니다(그림 3E).
pip 설치 pyg_lib torch_scatter torch_sparse torch_cluster torch_spline_conv -f https://data.pyg.org/whl/torch-1.13.0+cu117.html
- https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html 의 지침에 따라 다음 명령을 사용하여 torch_scatter, torch_sparse, torch_cluster 및 torch_spline_conv 패키지를 설치합니다(그림 3E).
- torch-geometric 패키지를 설치합니다.
- 이 연구에는 torch-geometric 패키지의 특정 버전 2.2.0이 필요합니다. 명령을 실행합니다(그림 3F).
pip 설치 torch_geometric==2.2.0
- 이 연구에는 torch-geometric 패키지의 특정 버전 2.2.0이 필요합니다. 명령을 실행합니다(그림 3F).
- 다른 패키지를 설치합니다.
- pandas와 같은 패키지는 일반적으로 기본적으로 사용할 수 있습니다. 그렇지 않은 경우 pip 명령을 사용하여 설치합니다. 예를 들어 pandas 및 xgboost를 설치하려면 다음을 실행합니다.
핍 설치 팬더
pip 설치 xgboost
- pandas와 같은 패키지는 일반적으로 기본적으로 사용할 수 있습니다. 그렇지 않은 경우 pip 명령을 사용하여 설치합니다. 예를 들어 pandas 및 xgboost를 설치하려면 다음을 실행합니다.
2. 사전 훈련된 HealthModel을 사용하여 mqTrans 기능 생성
- 코드 및 미리 학습된 모델을 다운로드합니다.
- http://www.healthinformaticslab.org/supp/resources.php(HealthModel-mqTrans-v1-00.tar.gz)라는 웹 사이트에서 코드와 사전 훈련된 HealthModel을 다운로드합니다(그림 4A). 다운로드한 파일은 사용자가 지정한 경로로 압축을 풀 수 있습니다. 구현된 프로토콜의 자세한 공식화 및 지원 데이터는26에서 찾을 수 있습니다.
- HealthModel을 실행하기 위한 파라미터를 소개합니다.
- 먼저 명령행에서 작업 디렉토리를 HealthModel-mqTrans 폴더로 변경하십시오. 다음 구문을 사용하여 코드를 실행합니다.
파이썬 main.py <데이터 폴더> <모델 폴더> <출력 폴더>
각 매개 변수와 데이터, 모델 및 출력 폴더에 대한 세부 정보는 다음과 같습니다.
데이터 폴더: 원본 데이터 폴더이며 각 데이터 파일은 csv 형식입니다. 이 데이터 폴더에는 두 개의 파일이 있습니다(2.3단계 및 2.4단계의 자세한 설명 참조). 이러한 파일은 개인 데이터로 바꿔야 합니다.
data.csv: 전사체 매트릭스 파일입니다. 첫 번째 행에는 특징(또는 유전자) ID가 나열되고 첫 번째 열에는 샘플 ID가 표시됩니다. 유전자 목록에는 조절 인자(TF 및 lincRNA)와 조절된 mRNA 유전자가 포함됩니다.
label.csv: 샘플 레이블 파일입니다. 첫 번째 열에는 샘플 ID가 나열되고 이름이 "label"인 열에는 샘플 레이블이 제공됩니다.
model folder: 모델에 대한 정보를 저장할 폴더입니다.
HealthModel.pth: 미리 학습된 HealthModel입니다.
regulatory_geneIDs.csv: 이 연구에 사용된 조절 유전자 ID입니다.
target_geneIDs.csv: 본 연구에서 사용된 표적 유전자.
adjacent_matrix.csv: 조절 유전자의 인접 매트릭스.
output folder: 출력 파일은 코드에 의해 생성된 이 폴더에 기록됩니다.
test_target.csv: Z-Normalization 및 imputation 후 표적 유전자의 유전자 발현 값.
pred_target.csv: 표적 유전자의 예측된 유전자 발현 값.
mq_target.csv: 표적 유전자의 예측된 유전자 발현 값.
- 먼저 명령행에서 작업 디렉토리를 HealthModel-mqTrans 폴더로 변경하십시오. 다음 구문을 사용하여 코드를 실행합니다.
- csv 형식의 전사체 매트릭스 파일을 준비합니다.
- 각 행은 샘플을 나타내고 각 열은 유전자를 나타냅니다(그림 4B). 트랜스크립토믹 데이터 매트릭스 파일의 이름을 데이터 폴더에 data.csv으로 지정합니다.
알림: 이 파일은 Microsoft Excel과 같은 소프트웨어에서 데이터 매트릭스를 .csv 형식으로 수동으로 저장하여 생성할 수 있습니다. 전사체 매트릭스는 또한 컴퓨터 프로그래밍에 의해 생성될 수 있다.
- 각 행은 샘플을 나타내고 각 열은 유전자를 나타냅니다(그림 4B). 트랜스크립토믹 데이터 매트릭스 파일의 이름을 데이터 폴더에 data.csv으로 지정합니다.
- csv 형식의 레이블 파일을 준비합니다.
- 전사체 매트릭스 파일과 마찬가지로 레이블 파일의 이름을 데이터 폴더의 label.csv로 지정합니다(그림 4C).
참고: 첫 번째 열은 샘플 이름을 제공하고 각 샘플의 클래스 레이블은 레이블이라는 열에 제공됩니다. 레이블 열의 0 값은 이 샘플이 음수임을 의미하고 1은 양성 샘플을 의미합니다.
- 전사체 매트릭스 파일과 마찬가지로 레이블 파일의 이름을 데이터 폴더의 label.csv로 지정합니다(그림 4C).
- mqTrans 기능을 생성하십시오.
- 다음 명령을 실행하여 mqTrans 기능을 생성하고 그림 4D에 표시된 출력을 가져오십시오. mqTrans 기능은 ./output/mq_targets.csv 파일로 생성되고 레이블 파일은 ./output/label.csv 파일로 다시 저장됩니다. 추가 분석의 편의를 위해 mRNA 유전자의 원래 발현 값도 ./output/ test_target.csv 파일로 추출됩니다.
파이썬 ./Get_mqTrans/code/main.py ./data ./Get_mqTrans/model ./output
- 다음 명령을 실행하여 mqTrans 기능을 생성하고 그림 4D에 표시된 출력을 가져오십시오. mqTrans 기능은 ./output/mq_targets.csv 파일로 생성되고 레이블 파일은 ./output/label.csv 파일로 다시 저장됩니다. 추가 분석의 편의를 위해 mRNA 유전자의 원래 발현 값도 ./output/ test_target.csv 파일로 추출됩니다.
3. mqTrans 기능 선택
- 기능 선택 코드의 구문
- 먼저 작업 디렉토리를 HealthModel-mqTrans 폴더로 변경하십시오. 다음 구문을 사용합니다.
파이썬 ./FS_classification/testMain.py
각 매개변수의 세부 정보는 다음과 같습니다.
in-data-file: 입력 데이터 파일
in-label-file: 입력 데이터 파일의 레이블
output 폴더: 이 폴더에는 Output-score.xlsx (기능 선택 방법 및 해당 분류자의 정확도) 및 Output-SelectedFeatures.xlsx (각 기능 선택 알고리즘에 대해 선택한 기능 이름)를 포함하여 두 개의 출력 파일이 저장됩니다.- select_feature_number: 1에서 데이터 파일의 기능 수까지 기능 수를 선택합니다.
- test_size: 분할할 테스트 샘플의 비율을 설정합니다. 예를 들어, 0.2는 입력 데이터셋이 0.8:0.2의 비율로 train: test 부분 집합으로 임의로 분할됨을 의미합니다.
- combine: true인 경우, 기능 선택(예: 원래 표현식 값 및 mqTrans 기능)을 위해 두 개의 데이터 파일을 함께 결합합니다. false인 경우, 기능 선택에 하나의 데이터 파일(예: 원래 표현식 값 또는 mqTrans 기능)만 사용하십시오.
- combine file: comparent가 true이면 이 파일 이름을 제공하여 결합된 데이터 행렬을 저장합니다.
참고: 이 파이프라인은 생성된 mqTrans 기능이 분류 태스크에서 어떻게 수행되는지 보여주는 것을 목표로 하며, 다음 조작을 위해 섹션 2에서 생성된 파일을 직접 사용합니다.
- 먼저 작업 디렉토리를 HealthModel-mqTrans 폴더로 변경하십시오. 다음 구문을 사용합니다.
- mqTrans 기능 선택에 대한 기능 선택 알고리즘을 실행하십시오.
- 사용자가 mqTrans 기능 또는 원래 기능을 선택하는 경우 결합 =False 를 설정하십시오.
- 먼저 800개의 원래 기능을 선택하고 데이터 세트를 학습(test=0.8:0.2)으로 분할합니다.
파이썬 ./FS_classification/testMain.py ./output/test_target.csv ./output/label.csv ./result 800 0.2 거짓 - 사용자가 mqTrans 기능을 원래 표현식 값과 결합하여 기능을 선택하려는 경우 combine =True를 설정하십시오. 여기서 시연 예제는 800개의 기능을 선택하고 데이터 세트를 train으로 분할하는 것입니다(test=0.8:0.2).
파이썬 ./FS_classification/testMain.py ./output/mq_targets.csv ./output/label.csv ./result_combine 800 0.2 True ./output/test_target.csv
참고: 그림 5 는 출력 정보를 보여줍니다. 이 프로토콜에 필요한 보충 파일은 HealthModel-mqTrans-v1-00.tar 폴더(보충 코딩 파일 1)에 있습니다.
Access restricted. Please log in or start a trial to view this content.
결과
전사체 데이터 세트의 mqTrans 보기 평가
테스트 코드는 11개의 기능 선택(FS) 알고리즘과 7개의 분류자를 사용하여 전사체 데이터 세트의 생성된 mqTrans 보기가 분류 작업에 어떻게 기여하는지 평가합니다(그림 6). 테스트 데이터 세트는 TCGA(The Cancer Genome Atlas) 데이터베이스29의 317개 결장 선암종(COAD)으로 구성됩니다. I기 또는 II기의 COAD 환자는 ?...
Access restricted. Please log in or start a trial to view this content.
토론
프로토콜의 섹션 2(사전 학습된 HealthModel을 사용하여 mqTrans 기능 생성)는 이 프로토콜 내에서 가장 중요한 단계입니다. 섹션 1에서 계산 작업 환경을 준비한 후 섹션 2에서는 사전 훈련된 대형 참조 모델을 기반으로 전사체 데이터 세트의 mqTrans 보기를 생성합니다. 섹션 3은 바이오마커 검출 및 예측 작업을 위해 생성된 mqTrans 기능을 선택하는 데모 예제입니다. 사용자는 자신의 도구 또는 코드를 사?...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 공개할 것이 없습니다.
감사의 말
이 작업은 시니어 및 주니어 기술 혁신팀(20210509055RQ), 구이저우성 과학기술프로젝트(ZK2023-297), 구이저우성 위생건강위원회 과학기술재단(gzwkj2023-565), 길림성 교육부 과학기술프로젝트(JJKH20220245KJ, JJKH20220226SK), 중국 국가자연과학재단(U19A2061), 길림성 빅데이터 지능컴퓨팅 중점연구소의 지원을 받았다 (20180622002JC) 및 중앙 대학을 위한 기초 연구 기금, JLU. 우리는 이 프로토콜의 엄격함과 명확성을 실질적으로 향상시키는 데 중요한 역할을 한 건설적인 비평에 대해 리뷰 편집자와 세 명의 익명 리뷰어에게 진심으로 감사를 표합니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
참고문헌
- Mutz, K. -O., Heilkenbrinker, A., Lönne, M., Walter, J. -G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516(2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684(2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352(2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528(2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. Analysis of Survival Data. , Chapman and Hall/CRC. London. (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866(2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307(2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119(2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239(2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334(2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338(2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , Springer. Cham. (2022).
- Muzio, G., O'Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738(2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559(2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5(2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883(2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582(2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801(2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138(2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308(2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유