A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
יצירת תצוגת ויסות התמלול של תכונות תמלול למשימת חיזוי וזיהוי סמנים ביולוגיים כהים במערכי נתונים קטנים
In This Article
Summary
כאן, אנו מציגים פרוטוקול להמרת נתוני שעתוק לתצוגת mqTrans, המאפשר זיהוי של סמנים ביולוגיים כהים. בעוד שסמנים ביולוגיים אלה אינם באים לידי ביטוי באופן דיפרנציאלי בניתוחי שעתוק קונבנציונליים, הם מציגים ביטוי דיפרנציאלי בתצוגת mqTrans. הגישה משמשת כטכניקה משלימה לשיטות מסורתיות, וחושפת סמנים ביולוגיים שבעבר התעלמו מהם.
Abstract
שעתוק מייצג את רמות הביטוי של גנים רבים בדגימה ונמצא בשימוש נרחב במחקר ביולוגי ובפרקטיקה קלינית. החוקרים התמקדו בדרך כלל בסמנים ביולוגיים שעתוק עם ייצוגים דיפרנציאליים בין קבוצת פנוטיפ לקבוצת ביקורת של דגימות. מחקר זה הציג מסגרת למידה מרובת משימות של רשת גרף-קשב (GAT) כדי ללמוד את האינטראקציות הבין-גניות המורכבות של דגימות הייחוס. מודל ייחוס מדגים הוכשר מראש על הדגימות הבריאות (HealthModel), אשר ניתן להשתמש בו ישירות כדי ליצור את תצוגת ויסות השעתוק הכמותי מבוסס המודל (mqTrans) של תעתיקי הבדיקה הבלתי תלויים. תצוגת mqTrans שנוצרה של תעתיקים הודגמה על ידי משימות חיזוי וזיהוי סמנים ביולוגיים כהים. המונח שנטבע "סמן ביולוגי כהה" נבע מהגדרתו כי סמן ביולוגי כהה הראה ייצוג דיפרנציאלי בתצוגת mqTrans אך לא ביטוי דיפרנציאלי ברמת הביטוי המקורית שלו. סמן ביולוגי כהה תמיד התעלם במחקרים מסורתיים לזיהוי סמנים ביולוגיים בשל היעדר ביטוי דיפרנציאלי. ניתן להוריד את קוד המקור ואת המדריך של הצינור HealthModelPipe מ http://www.healthinformaticslab.org/supp/resources.php.
Introduction
שעתוק מורכב מהביטויים של כל הגנים בדגימה ועשוי להיות פרופיל על ידי טכנולוגיות תפוקה גבוהה כמו microarray ו- RNA-seq1. רמות הביטוי של גן אחד במערך נתונים נקראות תכונת שעתוק (transcriptomic feature), והייצוג הדיפרנציאלי של תכונת שעתוק בין קבוצת הפנוטיפ וקבוצת הביקורת מגדיר גן זה כסמן ביולוגי של פנוטיפזה 2,3. סמנים ביולוגיים שעתוק שימשו באופן נרחב בחקירות של אבחון מחלות4, מנגנון ביולוגי5, וניתוח הישרדות 6,7 וכו '.
דפוסי הפעילות הגנטית ברקמות הבריאות נושאים מידע חיוני על החיים 8,9. דפוסים אלה מציעים תובנות שלא יסולא בפז ומשמשים כמקורות אידיאליים להבנת מסלולי ההתפתחות המורכבים של הפרעות שפירות10,11 ומחלות קטלניות12. גנים מתקשרים זה עם זה, ותעתיקים מייצגים את רמות הביטוי הסופיות לאחר האינטראקציות המסובכות שלהם. דפוסים אלה מנוסחים כרשת ויסות שעתוק13 ורשת מטבוליזם14 וכו '. הביטויים של רנ"א שליח (mRNAs) ניתנים לשעתוק על ידי גורמי שעתוק (TFs) ורנ"א אינטרגני ארוך שאינו מקודד (lincRNAs)15,16,17. ניתוח ביטוי דיפרנציאלי קונבנציונלי התעלם מאינטראקציות גנים מורכבות כאלה עם ההנחה של עצמאות בין תכונות18,19.
ההתקדמות האחרונה ברשתות עצביות גרפיות (GNNs) מדגימה פוטנציאל יוצא דופן בחילוץ מידע חשוב מנתונים מבוססי OMIC עבור מחקרי סרטן20, למשל, זיהוי מודולי ביטוי משותף21. היכולת המולדת של GNNs הופכת אותם לאידיאליים למידול היחסים המורכבים והתלות בין גנים22,23.
מחקרים ביו-רפואיים מתמקדים לעתים קרובות בניבוי מדויק של פנוטיפ כנגד קבוצת הביקורת. משימות כאלה מנוסחות בדרך כלל כסיווגים בינאריים 24,25,26. כאן, שתי תוויות המחלקה מקודדות בדרך כלל כ- 1 ו- 0, נכון ולא נכון, או אפילו חיובי ושלילי27.
מחקר זה נועד לספק פרוטוקול קל לשימוש ליצירת תצוגת ויסות התמלול (mqTrans) של ערכת נתוני תמלול המבוססת על מודל הייחוס של רשת תשומת הלב הגרפים (GAT) שהוכשר מראש. מסגרת GAT מרובת המשימות מעבודה26 שפורסמה בעבר שימשה להמרת תכונות תעתיק לתכונות mqTrans. מערך נתונים גדול של תעתיקים בריאים מאוניברסיטת קליפורניה, סנטה קרוז (UCSC) Xenaפלטפורמה 28 שימש לאימון מראש של מודל הייחוס (HealthModel), אשר מדד כמותית את תקנות השעתוק מהגורמים הרגולטוריים (TFs ו- lincRNAs) ל- mRNA המטרה. תצוגת mqTrans שנוצרה יכולה לשמש לבניית מודלים לחיזוי ולזיהוי סמנים ביולוגיים כהים. פרוטוקול זה משתמש במערך הנתונים של חולי אדנוקרצינומה של המעי הגס (COAD) מתוך מסד הנתונים29 של אטלס גנום הסרטן (TCGA) כדוגמה להמחשה. בהקשר זה, חולים בשלבים I או II מסווגים כדגימות שליליות, ואילו אלה בשלבים III או IV נחשבים לדגימות חיוביות. כמו כן מושווים ההתפלגויות של סמנים ביולוגיים כהים ומסורתיים על פני 26 סוגי סרטן TCGA.
תיאור הצינור של HealthModel
המתודולוגיה המיושמת בפרוטוקול זה מבוססת על מסגרת26 שפורסמה בעבר, כמתואר באיור 1. כדי להתחיל, המשתמשים נדרשים להכין את ערכת נתוני הקלט, להזין אותו לתוך צינור HealthModel המוצע, ולקבל תכונות mqTrans. הוראות מפורטות להכנת נתונים מפורטות מפורטות בסעיף 2 בסעיף הפרוטוקול. לאחר מכן, למשתמשים יש אפשרות לשלב תכונות mqTrans עם תכונות התמליל המקוריות או להמשיך עם תכונות mqTrans שנוצרו בלבד. לאחר מכן, ערכת הנתונים המופקת כפופה לתהליך בחירת תכונות, כאשר למשתמשים יש את הגמישות לבחור את הערך המועדף עליהם עבור k באימות צולב k-fold לסיווג. מדד ההערכה העיקרי המשמש בפרוטוקול זה הוא דיוק.
HealthModel26 מסווג את תכונות השעתוק לשלוש קבוצות נפרדות: TF (גורם שעתוק), lincRNA (RNA אינטרגני ארוך שאינו מקודד) ו- mRNA (רנ"א שליח). תכונות TF מוגדרות בהתבסס על הביאורים הזמינים באטלס החלבונים האנושי30,31. עבודה זו משתמשת בביאורים של lincRNAs מתוך מערך הנתונים GTEx32. גנים השייכים למסלולים ברמה השלישית במסד הנתוניםKEGG 33 נחשבים לתכונות mRNA. ראוי לציין כי אם תכונת mRNA מציגה תפקידי בקרה עבור גן מטרה כפי שמתועד במסד הנתונים TRRUST34, הוא מסווג מחדש למחלקת TF.
פרוטוקול זה גם מייצר באופן ידני את שני קבצי הדוגמה עבור מזהי הגנים של גורמים רגולטוריים (regulatory_geneIDs.csv) ו- mRNA מטרה (target_geneIDs.csv). מטריצת המרחק הזוגי בין תכונות הבקרה (TFs ו- lincRNAs) מחושבת על ידי מקדמי המתאם של פירסון ומקובצת על ידי הכלי הפופולרי ניתוח רשת ביטוי משותף של גנים (WGCNA)36 (adjacent_matrix.csv). משתמשים יכולים להשתמש ישירות בצינור HealthModel יחד עם קבצי תצורה לדוגמה אלה כדי ליצור את תצוגת mqTrans של ערכת נתונים תמלולית.
פרטים טכניים של HealthModel
HealthModel מייצג את היחסים המורכבים בין TFs ו- lincRNA כגרף, כאשר תכונות הקלט משמשות כקודקודים המסומנים ב- V ומטריצת קצה בין קודקודים המסומנת כ- E. כל מדגם מאופיין בתכונות רגולטוריות K , המסומלות כ - VK×1. באופן ספציפי, מערך הנתונים הקיף 425 TFs ו 375 lincRNAs, וכתוצאה מכך ממדיות מדגם של K = 425 + 375 = 800. כדי להקים את מטריצת הקצה E, עבודה זו השתמשה בכלי הפופולרי WGCNA35. המשקל הזוגי המקשר בין שני קודקודים המיוצגים כ 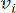 - ו
- ו 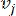 - , נקבע על ידי מקדם המתאם של פירסון. רשת הבקרה הגנטית מציגה טופולוגיה36 נטולת קנה מידה, המאופיינת בנוכחות גנים רכזתיים בעלי תפקידים פונקציונליים מרכזיים. אנו מחשבים את המתאם בין שתי תכונות או קודקודים,
- , נקבע על ידי מקדם המתאם של פירסון. רשת הבקרה הגנטית מציגה טופולוגיה36 נטולת קנה מידה, המאופיינת בנוכחות גנים רכזתיים בעלי תפקידים פונקציונליים מרכזיים. אנו מחשבים את המתאם בין שתי תכונות או קודקודים, 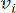 ו
ו 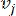 - , באמצעות מדד החפיפה הטופולוגית (TOM) באופן הבא:
- , באמצעות מדד החפיפה הטופולוגית (TOM) באופן הבא:
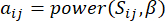 (1)
(1)
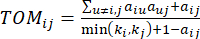 (2)
(2)
β הסף הרך מחושב באמצעות הפונקציה 'pickSoft Threshold' מחבילת WGCNA. הפונקציה מעריכית החזקה aij מוחלת, כאשר 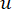 מייצג גן שאינו כולל i ו- j, ומייצג
מייצג גן שאינו כולל i ו- j, ומייצג 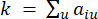 את קישוריות הקודקוד. WGCNA מקבץ את פרופילי הביטוי של תכונות התעתיק למודולים מרובים באמצעות מדד אי-דמיון נפוץ (
את קישוריות הקודקוד. WGCNA מקבץ את פרופילי הביטוי של תכונות התעתיק למודולים מרובים באמצעות מדד אי-דמיון נפוץ (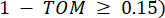 37.
37.
מסגרת HealthModel תוכננה במקור כארכיטקטורת למידה מרובת משימות26. פרוטוקול זה משתמש רק במשימת האימון מראש של המודל לבניית תצוגת mqTrans התעתוק. המשתמש יכול לבחור לחדד עוד יותר את HealthModel שהוכשר מראש תחת רשת הקשב של גרף ריבוי המשימות עם דגימות תמלול ספציפיות למשימה נוספות.
פרטים טכניים של בחירת תכונות וסיווגן
מאגר בחירת התכונות מיישם אחד-עשר אלגוריתמים לבחירת תכונות (FS). ביניהם, שלושה הם אלגוריתמי FS מבוססי מסנן: בחירת התכונות הטובות ביותר של K באמצעות מקדם המידע המרבי (SK_mic), בחירת תכונות K בהתבסס על FPR של מיקרופון (SK_fpr), ובחירת תכונות K עם שיעור גילוי השווא הגבוה ביותר של מיקרופון (SK_fdr). בנוסף, שלושה אלגוריתמי FS מבוססי עצים מעריכים תכונות בודדות באמצעות עץ החלטה עם מדד ג'יני (DT_gini), עצי החלטה מוגברת אדפטיבית (AdaBoost) ויער אקראי (RF_fs). המאגר משלב גם שתי שיטות עטיפה: חיסול תכונות רקורסיבי עם מסווג וקטור תמיכה ליניארי (RFE_SVC) וביטול תכונות רקורסיבי עם מסווג רגרסיה לוגיסטית (RFE_LR). לבסוף, שני אלגוריתמי הטבעה כלולים: מסווג SVC ליניארי עם ערכי חשיבות התכונות L1 המדורגים במקום הראשון (lSVC_L1) ומסווג רגרסיה לוגיסטית עם ערכי חשיבות התכונות L1 המדורגים במקום הראשון (LR_L1).
מאגר המסווגים מעסיק שבעה מסווגים שונים לבניית מודלים לסיווג. מסווגים אלה כוללים מכונת וקטור תמיכה ליניארית (SVC), גאוסיאן נאיבי בייס (GNB), מסווג רגרסיה לוגיסטית (LR), k השכן הקרוב ביותר, כאשר k מוגדר ל- 5 כברירת מחדל (KNN), XGBoost, יער אקראי (RF) ועץ החלטה (DT).
הפיצול האקראי של מערך הנתונים לרכבת: ניתן להגדיר תת-קבוצות בדיקה בשורת הפקודה. הדוגמה המודגמת משתמשת ביחס של רכבת: מבחן = 8: 2.
Access restricted. Please log in or start a trial to view this content.
Protocol
הערה: הפרוטוקול הבא מתאר את הפרטים של הפרוצדורה האנליטית של אינפורמטיקה ופקודות Python של המודולים העיקריים. איור 2 ממחיש את שלושת השלבים העיקריים באמצעות פקודות לדוגמה המשמשות בפרוטוקול זה, ומתייחס לעבודות שפורסמו בעבר26,38 לקבלת פרטים טכניים נוספים. בצע את הפרוטוקול הבא תחת חשבון משתמש רגיל במערכת מחשב והימנע משימוש בחשבון מנהל המערכת או הבסיס. זהו פרוטוקול חישובי ואין בו גורמים מסוכנים ביו-רפואיים.
1. הכינו סביבת Python
- צור סביבה וירטואלית.
- מחקר זה השתמש בשפת התכנות Python ובסביבה וירטואלית של Python (VE) עם Python 3.7. בצע את השלבים הבאים (איור 3A):
קונדה ליצור -n HealthModel Python=3.7
conda create היא הפקודה ליצירת VE חדש. הפרמטר -n מציין את שם הסביבה החדשה, במקרה זה, healthmodel. ו - python=3.7 מציין את גרסת Python שתותקן. בחר כל שם מועדף וגרסת Python התומכת בפקודה לעיל. - לאחר הפעלת הפקודה, הפלט דומה לאיור 3B. הזן y והמתן להשלמת התהליך.
- מחקר זה השתמש בשפת התכנות Python ובסביבה וירטואלית של Python (VE) עם Python 3.7. בצע את השלבים הבאים (איור 3A):
- הפעלת הסביבה הווירטואלית
- ברוב המקרים, הפעל את VE שנוצר באמצעות הפקודה הבאה (איור 3C):
Conda Activate HealthModel - בצע את ההוראות הספציפיות לפלטפורמה עבור הפעלת VE, אם פלטפורמות מסוימות דורשות מהמשתמש להעלות את קבצי התצורה הספציפיים לפלטפורמה להפעלה.
- ברוב המקרים, הפעל את VE שנוצר באמצעות הפקודה הבאה (איור 3C):
- התקן את PyTorch 1.13.1
- PyTorch היא חבילת Python פופולרית עבור אלגוריתמים של בינה מלאכותית (AI). השתמש PyTorch 1.13.1, מבוסס על פלטפורמת תכנות CUDA 11.7 GPU, כדוגמה. מצא גירסאות אחרות ב- https://pytorch.org/get-started/previous-versions/. השתמשו בפקודה הבאה (איור 3D):
PIP3 התקן לפיד TorchVision TorchAudio
הערה: מומלץ מאוד להשתמש ב-PyTorch גרסה 1.12 ומעלה. אחרת, התקנת torch_geometric החבילה הנדרשת עשויה להיות מאתגרת, כפי שצוין באתר הרשמי torch_geometric: https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html.
- PyTorch היא חבילת Python פופולרית עבור אלגוריתמים של בינה מלאכותית (AI). השתמש PyTorch 1.13.1, מבוסס על פלטפורמת תכנות CUDA 11.7 GPU, כדוגמה. מצא גירסאות אחרות ב- https://pytorch.org/get-started/previous-versions/. השתמשו בפקודה הבאה (איור 3D):
- התקנת חבילות נוספות עבור לפיד-גיאומטרי
- בהתאם להנחיות בשלב https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html, התקן את החבילות הבאות: torch_scatter, torch_sparse, torch_cluster ו- torch_spline_conv באמצעות הפקודה (איור 3E):
התקנת פיפ pyg_lib torch_scatter torch_sparse torch_cluster torch_spline_conv -f https://data.pyg.org/whl/torch-1.13.0+cu117.html
- בהתאם להנחיות בשלב https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html, התקן את החבילות הבאות: torch_scatter, torch_sparse, torch_cluster ו- torch_spline_conv באמצעות הפקודה (איור 3E):
- התקן חבילה גיאומטרית של לפיד .
- מחקר זה דורש גרסה ספציפית, 2.2.0, של חבילת הלפיד-גיאומטרי . הפעל את הפקודה (איור 3F):
התקנת PIP torch_geometric==2.2.0
- מחקר זה דורש גרסה ספציפית, 2.2.0, של חבילת הלפיד-גיאומטרי . הפעל את הפקודה (איור 3F):
- התקן חבילות אחרות.
- חבילות כמו פנדות זמינות בדרך כלל כברירת מחדל. אם לא, התקן אותם באמצעות הפקודה pip. לדוגמה, כדי להתקין פנדות ו- xgboost, הפעל:
פיפ להתקין פנדות
התקנת PIP XGBOOST
- חבילות כמו פנדות זמינות בדרך כלל כברירת מחדל. אם לא, התקן אותם באמצעות הפקודה pip. לדוגמה, כדי להתקין פנדות ו- xgboost, הפעל:
2. שימוש ב- HealthModel שהוכשר מראש כדי ליצור את תכונות mqTrans
- הורד את הקוד ואת המודל שהוכשר מראש.
- הורידו את הקוד ואת ה-HealthModel שהוכשר מראש מהאתר: http://www.healthinformaticslab.org/supp/resources.php, ששמו HealthModel-mqTrans-v1-00.tar.gz (איור 4A). ניתן לבטל את דחיסת הקובץ שהורדת לנתיב שצוין על-ידי המשתמש. את הניסוח המפורט ואת הנתונים התומכים של הפרוטוקול המיושם ניתן למצוא ב26.
- הצג את הפרמטרים להפעלת HealthModel.
- ראשית, שנה את ספריית העבודה לתיקייה HealthModel-mqTrans בשורת הפקודה. השתמש בתחביר הבא להפעלת הקוד:
Python main.py <תיקיית נתונים> <תיקיית דגם> <תיקיית פלט>
הפרטים לגבי כל פרמטר ותיקיות הנתונים, המודל והפלט הם כדלקמן:
תיקיית נתונים: זוהי תיקיית נתוני המקור וכל קובץ נתונים הוא בתבנית csv. תיקיית נתונים זו כוללת שני קבצים (ראה תיאורים מפורטים בשלבים 2.3 ו- 2.4). יש להחליף קבצים אלה בנתונים אישיים.
data.csv: קובץ המטריצה התמלול. השורה הראשונה מפרטת את מזהי התכונות (או הגנים), והעמודה הראשונה מציגה את מזהי הדגימה. רשימת הגנים כוללת את הגורמים הרגולטוריים (TFs ו- lincRNAs), ואת הגנים mRNA מוסדרים.
label.csv: קובץ התוויות לדוגמה. העמודה הראשונה מפרטת את המזהים לדוגמה, והעמודה עם השם "label" מעניקה את התווית לדוגמה.
תיקיית הדגם: התיקיה לשמירת מידע אודות הדגם:
HealthModel.pth: HealthModel שהוכשר מראש.
regulatory_geneIDs.csv: מזהי הגנים הרגולטוריים ששימשו במחקר זה.
target_geneIDs.csv: גני המטרה המשמשים במחקר זה.
adjacent_matrix.csv: המטריצה הסמוכה של גנים רגולטוריים.
תיקיית פלט: קבצי הפלט נכתבים בתיקייה זו, שנוצרו על ידי הקוד.
test_target.csv: ערך ביטוי הגנים של גני המטרה לאחר Z-נורמליזציה וזקיפה.
pred_target.csv: ערך ביטוי הגנים החזוי של גני המטרה.
mq_target.csv: ערך ביטוי הגנים החזוי של גני המטרה.
- ראשית, שנה את ספריית העבודה לתיקייה HealthModel-mqTrans בשורת הפקודה. השתמש בתחביר הבא להפעלת הקוד:
- הכן את קובץ המטריצה התמלול בפורמט csv.
- כל שורה מייצגת דגימה, וכל עמודה מייצגת גן (איור 4B). תן לקובץ מטריצת הנתונים התמליל את השם data.csv בתיקיית הנתונים .
הערה: ניתן ליצור קובץ זה על-ידי שמירה ידנית של מטריצת נתונים בתבנית .csv מתוכנות כגון Microsoft Excel. מטריצת התמלול עשויה להיווצר גם על ידי תכנות מחשבים.
- כל שורה מייצגת דגימה, וכל עמודה מייצגת גן (איור 4B). תן לקובץ מטריצת הנתונים התמליל את השם data.csv בתיקיית הנתונים .
- הכן את קובץ התווית בתבנית csv.
- בדומה לקובץ מטריצה תעתיק (transcriptomic metric), תן לקובץ התוויות את השם label.csv בתיקיית הנתונים (איור 4C).
הערה: העמודה הראשונה מציינת את שמות המדגמים, ותווית המחלקה של כל דוגמה ניתנת בעמודה שכותרתה תווית. הערך 0 בעמודת התווית פירושו שמדגם זה שלילי, 1 פירושו מדגם חיובי.
- בדומה לקובץ מטריצה תעתיק (transcriptomic metric), תן לקובץ התוויות את השם label.csv בתיקיית הנתונים (איור 4C).
- צור את תכונות mqTrans.
- הפעל את הפקודה הבאה כדי ליצור את תכונות mqTrans ולקבל את הפלטים המוצגים באיור 4D. תכונות mqTrans נוצרות כקובץ ./output/mq_targets.csv, וקובץ התווית נשמר מחדש כקובץ ./output/label.csv. לנוחות הניתוח הנוסף, ערכי הביטוי המקוריים של הגנים mRNA מופקים גם כקובץ ./output/ test_target.csv.
Python ./Get_mqTrans/code/main.py ./data ./Get_mqTrans/model ./output
- הפעל את הפקודה הבאה כדי ליצור את תכונות mqTrans ולקבל את הפלטים המוצגים באיור 4D. תכונות mqTrans נוצרות כקובץ ./output/mq_targets.csv, וקובץ התווית נשמר מחדש כקובץ ./output/label.csv. לנוחות הניתוח הנוסף, ערכי הביטוי המקוריים של הגנים mRNA מופקים גם כקובץ ./output/ test_target.csv.
3. בחר תכונות mqTrans
- תחביר קוד בחירת התכונות
- ראשית, שנה את ספריית העבודה לתיקייה HealthModel-mqTrans . השתמש בתחביר הבא:
Python ./FS_classification/testMain.py <תיקיית פלט> <שילוב> <שילוב קובץ>
הפרטים של כל פרמטר הם כדלקמן:
in-data-file: קובץ נתוני הקלט
in-label-file: התווית של קובץ נתוני הקלט
תיקיית פלט: שני קובצי פלט נשמרים בתיקייה זו, כולל Output-score.xlsx (שיטת בחירת התכונות והדיוק של המסווג המתאים) ו- Output-SelectedFeatures.xlsx (שמות התכונות שנבחרו עבור כל אלגוריתם לבחירת תכונות).- select_feature_number: בחר את מספר התכונות, החל מ- 1 ועד למספר התכונות של קובץ הנתונים.
- test_size: הגדר את היחס בין מדגם הבדיקה לפיצול. לדוגמה, 0.2 פירושו שערכת נתוני הקלט מפוצלת באופן אקראי לרכבת: תת-קבוצות בדיקה ביחס של 0.8:0.2.
- שילוב: אם נכון, שלב שני קבצי נתונים יחד לבחירת תכונות, כלומר, ערכי הביטוי המקוריים ותכונות mqTrans. אם false, פשוט השתמש בקובץ נתונים אחד לבחירת תכונות, כלומר, ערכי הביטוי המקוריים או תכונות mqTrans.
- שילוב קובץ: אם השילוב נכון, ספק שם קובץ זה כדי לשמור את מטריצת הנתונים המשולבת.
הערה: צינור זה נועד להדגים כיצד תכונות mqTrans שנוצרו לבצע במשימות סיווג, והוא משתמש ישירות בקובץ שנוצר על ידי סעיף 2 עבור הפעולות הבאות.
- ראשית, שנה את ספריית העבודה לתיקייה HealthModel-mqTrans . השתמש בתחביר הבא:
- הפעל אלגוריתם בחירת תכונות לבחירת תכונות mqTrans.
- הפוך לשלב = False אם המשתמש בוחר תכונות mqTrans או תכונות מקוריות.
- ראשית, בחר 800 תכונות מקוריות ופצל את מערך הנתונים לרכבת: test=0.8:0.2:
python ./FS_classification/testMain.py ./output/test_target.csv ./output/label.csv ./result 800 0.2 false - הפוך לשלב =True, אם המשתמש רוצה לשלב את תכונות mqTrans עם ערכי הביטוי המקוריים כדי לבחור תכונות. כאן, הדוגמה המדגימה היא לבחור 800 תכונות ולפצל את מערך הנתונים לרכבת: test=0.8:0.2:
python ./FS_classification/testMain.py ./output/mq_targets.csv ./output/label.csv ./result_combine 800 0.2 נכון ./פלט/test_target.csv
הערה: איור 5 מציג את פרטי הפלט. הקבצים המשלימים הדרושים לפרוטוקול זה נמצאים בתיקייה HealthModel-mqTrans-v1-00.tar (קובץ קידוד משלים 1).
Access restricted. Please log in or start a trial to view this content.
תוצאות
הערכת תצוגת mqTrans של מערך הנתונים התמלול
קוד הבדיקה משתמש באחד עשר אלגוריתמים לבחירת תכונות (FS) ובשבעה מסווגים כדי להעריך כיצד תצוגת mqTrans שנוצרה של מערך הנתונים התעתיק תורמת למשימת הסיווג (איור 6). מערך הנתונים של הבדיקה כולל 317 אדנוקרצינומה של המעי הגס (COAD) מתוך מסד...
Access restricted. Please log in or start a trial to view this content.
Discussion
סעיף 2 (השתמש ב- HealthModel שהוכשר מראש כדי ליצור את תכונות mqTrans) של הפרוטוקול הוא השלב הקריטי ביותר בפרוטוקול זה. לאחר הכנת סביבת העבודה החישובית בסעיף 1, סעיף 2 יוצר את תצוגת mqTrans של ערכת נתונים תעתיק המבוססת על מודל הייחוס הגדול שהוכשר מראש. סעיף 3 הוא דוגמה מדגימה לבחירת תכונות mqTrans שנוצרו עבור ז...
Access restricted. Please log in or start a trial to view this content.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
עבודה זו נתמכה על ידי צוות החדשנות הטכנולוגית הבכיר והזוטר (20210509055RQ), פרויקטי המדע והטכנולוגיה המחוזיים של גוויג'ואו (ZK2023-297), קרן המדע והטכנולוגיה של ועדת הבריאות של מחוז גוויג'ואו (gzwkj2023-565), פרויקט המדע והטכנולוגיה של מחלקת החינוך של מחוז ג'ילין (JJKH20220245KJ ו- JJKH20220226SK), הקרן הלאומית למדעי הטבע של סין (U19A2061), מעבדת המפתח המחוזית של ג'ילין למחשוב חכם של ביג דאטה (20180622002JC), וקרנות המחקר הבסיסיות של האוניברסיטאות המרכזיות, JLU. אנו מביעים את הערכתנו הכנה לעורך הביקורת ולשלושת הסוקרים האנונימיים על ביקורתם הבונה, אשר סייעו בשיפור משמעותי של ההקפדה והבהירות של פרוטוקול זה.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
References
- Mutz, K. -O., Heilkenbrinker, A., Lönne, M., Walter, J. -G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516(2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684(2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352(2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528(2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. Analysis of Survival Data. , Chapman and Hall/CRC. London. (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866(2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307(2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119(2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239(2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334(2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338(2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , Springer. Cham. (2022).
- Muzio, G., O'Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738(2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559(2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5(2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883(2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582(2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801(2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138(2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308(2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved