サインイン
このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Methods Article
小さなデータセットでの予測タスクとダークバイオマーカー検出のためのトランスクリプトーム特徴の転写制御ビューの生成
要約
ここでは、トランスクリプトームデータをmqTransビューに変換し、ダークバイオマーカーの同定を可能にするプロトコルを紹介します。これらのバイオマーカーは、従来のトランスクリプトーム解析では発現差が認められませんが、mqTransでは発現差が見られます。このアプローチは、従来の手法を補完する手法として機能し、これまで見過ごされていたバイオマーカーを明らかにします。
要約
トランスクリプトームは、サンプル中の多くの遺伝子の発現レベルを表し、生物学研究や臨床現場で広く使用されています。研究者は通常、サンプルの表現型グループとコントロールグループの間で異なる表現を持つトランスクリプトームバイオマーカーに焦点を当てました。本研究では、参照サンプルの複雑な遺伝子間相互作用を学習するためのマルチタスクグラフアテンションネットワーク(GAT)学習フレームワークを提示しました。実証的参照モデルは、健康なサンプル(HealthModel)で事前にトレーニングされており、独立したテストトランスクリプトームのモデルベースの定量的転写調節(mqTrans)ビューを生成するために直接使用できます。生成されたトランスクリプトームの mqTrans ビューは、予測タスクとダークバイオマーカー検出によって実証されました。「ダークバイオマーカー」という造語は、ダークバイオマーカーはmqTransビューでは差異表現を示すが、元の発現レベルでは差異的発現を示さないという定義に由来しています。ダークバイオマーカーは、発現差がないため、従来のバイオマーカー検出研究では常に見過ごされていました。パイプラインHealthModelPipeのソースコードとマニュアルは、http://www.healthinformaticslab.org/supp/resources.php からダウンロードできます。
概要
トランスクリプトームは、サンプル中のすべての遺伝子の発現で構成されており、マイクロアレイやRNA-seq1などのハイスループット技術によってプロファイリングすることができます。データセット内の1つの遺伝子の発現レベルはトランスクリプトーム特徴と呼ばれ、表現型と対照群の間でトランスクリプトームの特徴が異なって表現されることで、この遺伝子はこの表現型のバイオマーカーとして定義されます2,3。トランスクリプトームバイオマーカーは、疾患診断4、生物学的メカニズム5、生存解析6,7などの研究に広く利用されています。
健康な組織の遺伝子活性パターンは、生命に関する重要な情報を運びます8,9。これらのパターンは、非常に貴重な洞察を提供し、良性疾患10,11および致死性疾患12の複雑な発達の軌跡を理解するための理想的な参考資料として機能します。遺伝子は互いに相互作用し、トランスクリプトームは複雑な相互作用の後の最終的な発現レベルを表します。このようなパターンは、転写調節ネットワーク13および代謝ネットワーク14等として定式化される。メッセンジャーRNA(mRNA)の発現は、転写因子(TF)と長鎖遺伝子間ノンコーディングRNA(lincRNA)によって転写制御されている15,16,17。従来の差次的発現解析では、このような複雑な遺伝子相互作用は無視され、特徴間の独立性を仮定していた18,19。
グラフニューラルネットワーク(GNN)の最近の進歩は、がん研究20、例えば共発現モジュールの同定21など、OMICベースのデータから重要な情報を抽出する上で並外れた可能性を示しています。GNNの生来の能力は、遺伝子間の複雑な関係と依存関係をモデル化するのに理想的です22,23。
生物医学研究では、対照群に対する表現型を正確に予測することに重点が置かれることがよくあります。このようなタスクは、一般に二項分類24、25、26として定式化されます。ここで、2 つのクラス ラベルは通常、1 と 0、true と false、または正と負の27 としてエンコードされます。
この研究は、事前学習済みのグラフアテンションネットワーク(GAT)参照モデルに基づいて、トランスクリプトームデータセットの転写制御(mqTrans)ビューを生成するための使いやすいプロトコルを提供することを目的としています。以前に発表された研究26 のマルチタスクGATフレームワークを使用して、トランスクリプトームの特徴をmqTransの特徴に変換しました。カリフォルニア大学サンタクルーズ校(UCSC)のXenaプラットフォーム28 の健康なトランスクリプトームの大規模なデータセットを使用して、制御因子(TFおよびlincRNA)から標的mRNAまでの転写制御を定量的に測定する参照モデル(HealthModel)を事前トレーニングしました。生成されたmqTransビューは、予測モデルの構築やダークバイオマーカーの検出に使用できます。このプロトコルは、例示的な例として、がんゲノムアトラス(TCGA)データベース29 からの結腸腺癌(COAD)患者データセットを利用します。これに関連して、ステージIまたはIIの患者は陰性サンプルに分類され、ステージIIIまたはIVの患者は陽性サンプルと見なされます。また、26種類のTCGAがんにおけるダークバイオマーカーと従来のバイオマーカーの分布も比較しています。
HealthModel パイプラインの説明
このプロトコルで採用されている方法論は、図1に概説されているように、以前に公開されたフレームワーク26に基づいています。まず、ユーザーは入力データセットを準備し、提案されたHealthModelパイプラインにフィードし、mqTrans特徴を取得する必要があります。詳細なデータ準備手順は、プロトコルセクションのセクション2に記載されています。その後、ユーザーは、mqTransの特徴を元のトランスクリプトームの特徴と組み合わせるか、生成されたmqTransの特徴のみを続行するかを選択できます。生成されたデータセットは、特徴選択プロセスにかけられ、ユーザーは分類のためのk分割交差検証でkの優先値を柔軟に選択できます。このプロトコルで使用される主な評価指標は精度です。
HealthModel26は、トランスクリプトームの特徴をTF(転写因子)、lincRNA(長鎖遺伝子間ノンコーディングRNA)、mRNA(メッセンジャーRNA)の3つのグループに分類しています。TFの特徴は、Human ProteinAtlas30,31で利用可能なアノテーションに基づいて定義されています。この研究は、GTExデータセット32からのlincRNAのアノテーションを利用しています。KEGGデータベース33における第3レベルの経路に属する遺伝子は、mRNAの特徴と見なされる。TRRUSTデータベース34に記録されているように、mRNAの特徴が標的遺伝子に対する調節的役割を示す場合、それはTFクラスに再分類されることは注目に値する。
また、このプロトコルでは、規定因子(regulatory_geneIDs.csv)と標的mRNA(target_geneIDs.csv)の遺伝子IDの2つのサンプルファイルを手動で生成します。調節特性(TFおよびlincRNA)間のペアワイズ距離マトリックスは、ピアソン相関係数によって計算され、一般的なツール加重遺伝子共発現ネットワーク解析(WGCNA)36 (adjacent_matrix.csv)によってクラスタリングされます。ユーザーは、HealthModel パイプラインをこれらのサンプル構成ファイルと共に直接利用して、トランスクリプトーム データセットの mqTrans ビューを生成できます。
HealthModelの技術的詳細
HealthModel は、TF と lincRNA の間の複雑な関係をグラフとして表し、入力特徴は V で示される頂点として機能し、頂点間エッジ行列は E として示されます。各サンプルは、VK×1として表されるK個の調節特徴によって特徴付けられます。具体的には、データセットには 425 の TF と 375 の lincRNA が含まれ、サンプルの次元は K = 425 + 375 = 800 でした。エッジ行列 E を確立するために、この作業では一般的なツール WGCNA35 を使用しました。と 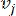 で表される
で表される 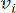 2 つの頂点をつなぐペアワイズ重みは、ピアソン相関係数によって決定されます。遺伝子調節ネットワークは、極めて重要な機能的役割を有するハブ遺伝子の存在を特徴とするスケールフリートポロジー36を呈する。2 つの特徴または頂点 と
2 つの頂点をつなぐペアワイズ重みは、ピアソン相関係数によって決定されます。遺伝子調節ネットワークは、極めて重要な機能的役割を有するハブ遺伝子の存在を特徴とするスケールフリートポロジー36を呈する。2 つの特徴または頂点 と 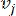 の間の相関関係は、
の間の相関関係は、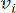 トポロジカル オーバーラップ メジャー (TOM) を使用して次のように計算します。
トポロジカル オーバーラップ メジャー (TOM) を使用して次のように計算します。
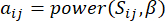 (1)
(1)
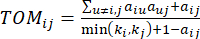 (2)
(2)
ソフトしきい値βは、WGCNA パッケージの 'pickSoft Threshold' 関数を使用して計算されます。べき乗指数関数 aij が適用され、ここで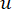 、 は i と j を除いた遺伝子を表し、
、 は i と j を除いた遺伝子を表し、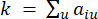 頂点接続性を表します。WGCNAは、トランスクリプトームの特徴の発現プロファイルを、一般的に採用されている非類似度尺度
頂点接続性を表します。WGCNAは、トランスクリプトームの特徴の発現プロファイルを、一般的に採用されている非類似度尺度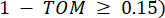 を用いて複数のモジュールにクラスタリングする(37.
を用いて複数のモジュールにクラスタリングする(37.
HealthModelフレームワークは、もともとマルチタスク学習アーキテクチャ26として設計されました。このプロトコルは、トランスクリプトーム mqTrans ビューの構築にモデルの事前トレーニング タスクのみを利用します。ユーザは、追加のタスク特異的トランスクリプトームサンプルを用いて、マルチタスクグラフアテンションネットワークの下で事前訓練されたHealthModelをさらに洗練させることを選択することができる。
機能の選択と分類に関する技術的な詳細
特徴選択プールには、11 個の特徴選択 (FS) アルゴリズムが実装されています。その中には、最大情報量係数(SK_mic)を使用したK個の最良の特徴量の選択、MICのFPRに基づくK個の特徴量の選択(SK_fpr)、MICの偽発見率が最も高いK個の特徴量の選択(SK_fdr)の3つがフィルタベースのFSアルゴリズムです。さらに、3つのツリーベースのFSアルゴリズムは、ジニ指数(DT_gini)、適応型ブースト決定木(AdaBoost)、およびランダムフォレスト(RF_fs)を備えた決定木を使用して個々の特徴を評価します。また、このプールには、線形サポートベクター分類器による再帰的特徴量消去法 (RFE_SVC) とロジスティック回帰分類器 (RFE_LR) による再帰的特徴量消去法の 2 つのラッパー手法も組み込まれています。最後に、最上位の L1 特徴重要度値 (lSVC_L1) を持つ線形 SVC 分類器と、最上位の L1 特徴重要度値 (LR_L1) を持つロジスティック回帰分類器の 2 つの埋め込みアルゴリズムが含まれています。
分類子プールでは、分類モデルを構築するために 7 つの異なる分類子が採用されています。これらの分類器は、線形サポート ベクター マシン (SVC)、ガウス単純ベイズ (GNB)、ロジスティック回帰分類器 (LR)、k 最近傍 (k は既定で 5 に設定されている) (KNN)、XGBoost、ランダム フォレスト (RF)、および決定木 (DT) で構成されます。
データセットをtrain:testサブセットにランダムに分割する方法は、コマンドラインで設定できます。この例では、train: test = 8:2 の比率を使用します。
プロトコル
注: 次のプロトコルでは、主要なモジュールのインフォマティクス解析手順と Python コマンドの詳細について説明します。 図2 は、このプロトコルで利用されるコマンドの例とともに3つの主要なステップを示しており、技術的な詳細については、以前に公開された作品26、38 を参照してください。コンピュータシステムの通常のユーザーアカウントで次のプロトコルを実行し、管理者またはrootアカウントの使用を避けてください。これは計算プロトコルであり、生物医学的危険因子はありません。
1. Python環境の準備
- 仮想環境を作成します。
- この研究では、Python プログラミング言語と Python 3.7 を使用した Python 仮想環境 (VE) を使用しました。以下の手順に従います(図3A)。
conda create -n healthmodel python=3.7
conda create は、新しい VE を作成するコマンドです。パラメーター -n は、新しい環境の名前 (この場合は healthmodel) を指定します。また、 python=3.7 はインストールする Python のバージョンを指定します。上記のコマンドをサポートする任意の名前と Python バージョンを選択します。 - コマンドを実行すると、出力は 図 3B のようになります。 「y 」と入力し、プロセスが完了するのを待ちます。
- この研究では、Python プログラミング言語と Python 3.7 を使用した Python 仮想環境 (VE) を使用しました。以下の手順に従います(図3A)。
- 仮想環境をアクティブ化する
- ほとんどの場合、作成したVEを以下のコマンドでアクティブにします(図3C)。
conda activate healthmodel - 一部のプラットフォームでは、アクティベーションのためにプラットフォーム固有の設定ファイルをアップロードする必要がある場合は、プラットフォーム固有の手順に従って、VE アクティベーションを行います。
- ほとんどの場合、作成したVEを以下のコマンドでアクティブにします(図3C)。
- PyTorch 1.13.1 をインストールする
- PyTorch は、人工知能 (AI) アルゴリズム用の一般的な Python パッケージです。例として、CUDA 11.7 GPU プログラミング プラットフォームに基づく PyTorch 1.13.1 を使用します。https://pytorch.org/get-started/previous-versions/ で他のバージョンを見つけてください。次のコマンドを使用します(図3D)。
pip3 torch torchvision torchaudio をインストールする
注: PyTorch バージョン 1.12 以降を使用することを強くお勧めします。そうしないと、torch_geometricの公式Webサイトに記載されているように、必要なパッケージ torch_geometric のインストールが困難になる可能性があります:https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html。
- PyTorch は、人工知能 (AI) アルゴリズム用の一般的な Python パッケージです。例として、CUDA 11.7 GPU プログラミング プラットフォームに基づく PyTorch 1.13.1 を使用します。https://pytorch.org/get-started/previous-versions/ で他のバージョンを見つけてください。次のコマンドを使用します(図3D)。
- トーチジオメトリックの追加パッケージをインストールする
- https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html のガイドラインに従って、次のコマンドを使用して、torch_scatter、torch_sparse、torch_cluster、およびtorch_spline_convパッケージをインストールします(図3E)。
pip install pyg_lib torch_scatter torch_sparse torch_cluster torch_spline_conv -f https://data.pyg.org/whl/torch-1.13.0+cu117.html を実行します。
- https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html のガイドラインに従って、次のコマンドを使用して、torch_scatter、torch_sparse、torch_cluster、およびtorch_spline_convパッケージをインストールします(図3E)。
- torch-geometric パッケージをインストールしてください。
- このスタディには、 トーチ ジオメトリック パッケージの特定のバージョン 2.2.0 が必要です。次のコマンドを実行します(図3F)。
pipインストールtorch_geometric==2.2.0
- このスタディには、 トーチ ジオメトリック パッケージの特定のバージョン 2.2.0 が必要です。次のコマンドを実行します(図3F)。
- 他のパッケージをインストールします。
- pandas のようなパッケージは、通常、デフォルトで利用可能です。そうでない場合は、pipコマンドを使用してインストールします。たとえば、pandas と xgboost をインストールするには、次のコマンドを実行します。
pipインストールパンダ
pipインストールxgboost
- pandas のようなパッケージは、通常、デフォルトで利用可能です。そうでない場合は、pipコマンドを使用してインストールします。たとえば、pandas と xgboost をインストールするには、次のコマンドを実行します。
2. 事前学習済みの HealthModel を使用して mqTrans 特徴を生成する
- コードと事前トレーニング済みモデルをダウンロードします。
- コードと事前トレーニング済みの HealthModel を Web サイト http://www.healthinformaticslab.org/supp/resources.php (HealthModel-mqTrans-v1-00.tar.gz という名前) からダウンロードします (図 4A)。ダウンロードしたファイルは、ユーザー指定のパスに解凍できます。実装されたプロトコルの詳細な定式化とサポートデータは、26にあります。
- HealthModel を実行するためのパラメーターを紹介します。
- まず、コマンドラインで作業ディレクトリを HealthModel-mqTrans フォルダに変更します。コードを実行するには、次の構文を使用します。
Python main.py <データ フォルダー> <モデル フォルダー> <出力フォルダー>
各パラメーターと、データ、モデル、および出力フォルダーに関する詳細は次のとおりです。
データフォルダ:これはソースデータフォルダで、各データファイルはcsv形式です。このデータフォルダには2つのファイルがあります(手順2.3と2.4の詳細な説明を参照)。これらのファイルは個人データに置き換える必要があります。
data.csv:トランスクリプトームマトリックスファイル。最初の行には特徴(または遺伝子)IDがリストされ、最初の列にはサンプルIDが示されます。遺伝子のリストには、制御因子(TFおよびlincRNA)と制御されたmRNA遺伝子が含まれています。
label.csv: サンプル ラベル ファイル。最初の列にはサンプル ID が一覧表示され、"label" という名前の列にはサンプル ラベルが表示されます。
model folder: モデルに関する情報を保存するフォルダー。
HealthModel.pth: 事前トレーニング済みの HealthModel。
regulatory_geneIDs.csv:この研究で使用した制御遺伝子ID。
target_geneIDs.csv:本研究で用いた標的遺伝子
adjacent_matrix.csv:制御遺伝子の隣接マトリックス。
出力フォルダー: 出力ファイルは、コードによって作成されたこのフォルダーに書き込まれます。
test_target.csv:Z-Normalizationおよびインピュテーション後の標的遺伝子の発現値。
pred_target.csv:標的遺伝子の予測遺伝子発現値。
mq_target.csv:標的遺伝子の予測遺伝子発現値。
- まず、コマンドラインで作業ディレクトリを HealthModel-mqTrans フォルダに変更します。コードを実行するには、次の構文を使用します。
- トランスクリプトームマトリックスファイルをcsv形式で準備します。
- 各行はサンプルを表し、各列は遺伝子を表します(図4B)。トランスクリプトームデータマトリックスファイルに、データフォルダー内のdata.csvとおりに名前を付けます。
注: このファイルは、Microsoft Excel などのソフトウェアからデータマトリクスを .csv 形式で手動で保存することで生成できます。トランスクリプトームマトリックスは、コンピュータプログラミングによって生成することもできる。
- 各行はサンプルを表し、各列は遺伝子を表します(図4B)。トランスクリプトームデータマトリックスファイルに、データフォルダー内のdata.csvとおりに名前を付けます。
- ラベルファイルをcsv形式で用意します。
- トランスクリプトームマトリックスファイルと同様に、データフォルダ内のlabel.csvラベルファイルに名前を付けます(図4C)。
注: 最初の列にはサンプル名が表示され、各サンプルのクラス ラベルは label というタイトルの列に示されます。ラベル列の 0 の値は、このサンプルが負であることを意味し、1 は正のサンプルを意味します。
- トランスクリプトームマトリックスファイルと同様に、データフォルダ内のlabel.csvラベルファイルに名前を付けます(図4C)。
- mqTrans 特徴を生成します。
- 次のコマンドを実行してmqTrans特徴を生成し、 図4Dに示す出力を取得します。mqTrans フィーチャーはファイル ./output/mq_targets.csv として生成され、ラベル・ファイルはファイル ./output/label.csv として再保存されます。さらなる解析の便宜上、mRNA遺伝子の元の発現値もファイル ./output/ test_target.csvとして抽出されます。
python ./Get_mqTrans/code/main.py ./data ./Get_mqTrans/model ./output
- 次のコマンドを実行してmqTrans特徴を生成し、 図4Dに示す出力を取得します。mqTrans フィーチャーはファイル ./output/mq_targets.csv として生成され、ラベル・ファイルはファイル ./output/label.csv として再保存されます。さらなる解析の便宜上、mRNA遺伝子の元の発現値もファイル ./output/ test_target.csvとして抽出されます。
3. mqTrans 機能の選択
- 機能選択コードの構文
- まず、作業ディレクトリを HealthModel-mqTrans フォルダに変更します。次の構文を使用します。
python ./FS_classification/testMain.py
各パラメータの詳細は次のとおりです。
in-data-file: 入力データファイル
in-label-file: 入力データファイルのラベル
出力フォルダー: このフォルダーには、 Output-score.xlsx (特徴選択方法と対応する分類器の精度) と Output-SelectedFeatures.xlsx (各特徴選択アルゴリズムで選択された特徴名) の 2 つの出力ファイルが保存されます。- select_feature_number:1からデータファイルの特徴の数までの範囲で、特徴の数を選択します。
- test_size:分割するテストサンプルの比率を設定します。たとえば、0.2 は、入力データセットが 0.8:0.2 の比率で train: test サブセットにランダムに分割されることを意味します。
- combine: trueの場合、2つのデータファイルを結合して特徴量(元の式値とmqTrans特徴量)を選択します。falseの場合、特徴量の選択に1つのデータファイル、つまり元の式の値またはmqTrans特徴量を使用します。
- combine file: combine が true の場合、このファイル名を指定して、結合されたデータ行列を保存します。
注: このパイプラインは、生成された mqTrans 特徴が分類タスクでどのように実行されるかを示すことを目的としており、セクション 2 で生成されたファイルを次の操作に直接使用します。
- まず、作業ディレクトリを HealthModel-mqTrans フォルダに変更します。次の構文を使用します。
- mqTrans 特徴選択のための特徴選択アルゴリズムを実行します。
- ユーザーが mqTrans 特徴または元の特徴を選択した場合は 、combine =False を回します。
- まず、800個の元の特徴量を選択し、データセットをtrain: test=0.8:0.2に分割します。
python ./FS_classification/testMain.py ./output/test_target.csv ./output/label.csv ./result 800 0.2 False - ユーザーが mqTrans 特徴を元の式の値と結合して特徴を選択する場合は、combine =True にします。ここでは、800 個の特徴量を選択し、データセットを train: test=0.8:0.2 に分割する例を示します。
python ./FS_classification/testMain.py ./output/mq_targets.csv ./output/label.csv ./result_combine 800 0.2 True ./output/test_target.csv
メモ: 図 5 に出力情報を示します。このプロトコルに必要な補足ファイルは、HealthModel-mqTrans-v1-00.tarフォルダ(Supplementary Coding File 1)にあります。
結果
トランスクリプトームデータセットのmqTransビューの評価
このテストコードでは、11 個の特徴選択 (FS) アルゴリズムと 7 個の分類器を使用して、トランスクリプトームデータセットの生成された mqTrans ビューが分類タスクにどのように寄与するかを評価します(図 6)。テストデータセットは、The Cancer Genome Atlas(TCGA)データベース29の317の結?...
ディスカッション
プロトコルのセクション 2 (事前トレーニング済みの HealthModel を使用して mqTrans 特徴を生成する) は、このプロトコル内で最も重要なステップです。セクション 1 で計算作業環境を準備した後、セクション 2 では、事前学習済みの大規模参照モデルに基づいて、トランスクリプトームデータセットの mqTrans ビューを生成します。セクション3は、バイオマーカーの検出と予測タスクのために生?...
開示事項
著者は何も開示していません。
謝辞
この研究は、シニアおよびジュニア技術革新チーム(20210509055RQ)、貴州省科学技術プロジェクト(ZK2023-297)、貴州省衛生委員会科学技術基金会(gzwkj2023-565)、吉林省教育部科学技術プロジェクト(JJKH20220245KJおよびJJKH20220226SK)、中国国家自然科学基金会(U19A2061)、吉林省ビッグデータインテリジェントコンピューティング重点実験室の支援を受けました(20180622002JC)、およびJLU中央大学基盤研究費。このプロトコルの厳密さと明瞭さを大幅に向上させるのに役立った建設的な批評について、査読編集者と3人の匿名の査読者に心から感謝します。
資料
| Name | Company | Catalog Number | Comments |
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
参考文献
- Mutz, K. -. O., Heilkenbrinker, A., Lönne, M., Walter, J. -. G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516 (2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684 (2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352 (2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528 (2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. . Analysis of Survival Data. , (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866 (2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307 (2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119 (2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239 (2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334 (2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338 (2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. . BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , (2022).
- Muzio, G., O'Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738 (2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559 (2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5 (2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883 (2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582 (2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801 (2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138 (2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308 (2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved
当社はcookieを使用しています。
「続行」をクリックすることで、当社のcookieへの同意となります。