Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Generierung der Transkriptionsregulationsansicht von transkriptomischen Merkmalen für die Vorhersageaufgabe und die Detektion dunkler Biomarker auf kleinen Datensätzen
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll zur Umwandlung von transkriptomischen Daten in eine mqTrans-Ansicht vor, das die Identifizierung dunkler Biomarker ermöglicht. Während diese Biomarker in konventionellen transkriptomischen Analysen nicht differentiell exprimiert werden, zeigen sie in der mqTrans-Ansicht eine differentielle Expression. Der Ansatz dient als komplementäre Technik zu herkömmlichen Methoden und enthüllt bisher übersehene Biomarker.
Zusammenfassung
Das Transkriptom repräsentiert die Expressionsniveaus vieler Gene in einer Probe und wird in der biologischen Forschung und in der klinischen Praxis häufig verwendet. Die Forscher konzentrierten sich in der Regel auf transkriptomische Biomarker mit unterschiedlichen Repräsentationen zwischen einer Phänotypgruppe und einer Kontrollgruppe von Proben. In dieser Studie wurde ein Multitasking-Graph-Aufmerksamkeits-Netzwerk (GAT) Lernrahmen vorgestellt, um die komplexen intergenen Interaktionen der Referenzproben zu erlernen. An den gesunden Proben wurde ein demonstratives Referenzmodell (HealthModel) vortrainiert, das direkt zur Generierung der modellbasierten quantitativen Transkriptionsregulation (mqTrans) der unabhängigen Testtranskriptome verwendet werden konnte. Die generierte mqTrans-Ansicht von Transkriptomen wurde durch Vorhersageaufgaben und die Detektion dunkler Biomarker demonstriert. Der geprägte Begriff "dunkler Biomarker" rührt von seiner Definition her, dass ein dunkler Biomarker in der mqTrans-Ansicht eine differentielle Repräsentation zeigte, aber keine differentielle Expression in seinem ursprünglichen Expressionsniveau. Ein dunkler Biomarker wurde in traditionellen Biomarker-Nachweisstudien aufgrund des Fehlens einer differentiellen Expression immer übersehen. Der Quellcode und das Handbuch der Pipeline HealthModelPipe können von http://www.healthinformaticslab.org/supp/resources.php heruntergeladen werden.
Einleitung
Das Transkriptom besteht aus der Expression aller Gene in einer Probe und kann mit Hochdurchsatztechnologien wie Microarray und RNA-seq1 profiliert werden. Die Expressionsniveaus eines Gens in einem Datensatz werden als transkriptomisches Merkmal bezeichnet, und die differentielle Repräsentation eines transkriptomischen Merkmals zwischen dem Phänotyp und der Kontrollgruppe definiert dieses Gen als Biomarker für diesen Phänotyp 2,3. Transkriptomische Biomarker wurden in großem Umfang bei der Untersuchung der Krankheitsdiagnose4, des biologischen Mechanismus5 und der Überlebensanalyse 6,7 usw. eingesetzt.
Genaktivitätsmuster in den gesunden Geweben enthalten wichtige Informationen über das Leben 8,9. Diese Muster bieten unschätzbare Erkenntnisse und dienen als ideale Referenzen für das Verständnis der komplexen Entwicklungsverläufe von gutartigen Erkrankungen10,11 und tödlichen Erkrankungen12. Gene interagieren miteinander, und Transkriptome stellen die endgültigen Expressionsniveaus nach ihren komplizierten Interaktionen dar. Solche Muster werden als transkriptionelles Regulationsnetzwerk13 und Stoffwechselnetzwerk14 usw. formuliert. Die Expression von Boten-RNAs (mRNAs) kann durch Transkriptionsfaktoren (TFs) und lange intergene nicht-kodierende RNAs (lincRNAs) transkriptionell reguliert werden15,16,17. Die konventionelle differentielle Expressionsanalyse ignorierte solche komplexen Geninteraktionen mit der Annahme der Unabhängigkeit zwischen den Merkmalen18,19.
Jüngste Fortschritte bei neuronalen Graphennetzen (GNNs) zeigen ein außerordentliches Potenzial bei der Extraktion wichtiger Informationen aus OMIC-basierten Daten für Krebsstudien20, z. B. die Identifizierung von Co-Expressionsmodulen21. Die angeborene Kapazität von GNNs macht sie ideal für die Modellierung der komplizierten Beziehungen und Abhängigkeiten zwischen Genen22,23.
Biomedizinische Studien konzentrieren sich oft auf die genaue Vorhersage eines Phänotyps im Vergleich zur Kontrollgruppe. Solche Aufgaben werden üblicherweise als binäre Klassifikationen24, 25, 26 formuliert. Hier werden die beiden Klassenbezeichnungen in der Regel als 1 und 0, wahr und falsch oder sogar positiv und negativ27 codiert.
Ziel dieser Studie war es, ein einfach zu verwendendes Protokoll zur Generierung der Transkriptionsregulation (mqTrans) eines Transkriptom-Datensatzes auf der Grundlage des vortrainierten Graph-Attention-Netzwerks (GAT) Referenzmodells bereitzustellen. Das Multitasking-GAT-Framework aus einer zuvor veröffentlichten Arbeit26 wurde verwendet, um transkriptomische Merkmale in die mqTrans-Merkmale umzuwandeln. Ein großer Datensatz gesunder Transkriptome der Xena-Plattform28 der University of California, Santa Cruz (UCSC) wurde verwendet, um das Referenzmodell (HealthModel) vorzutrainieren, das die Transkriptionsregulation von den regulatorischen Faktoren (TFs und lincRNAs) zu den Ziel-mRNAs quantitativ maß. Die generierte mqTrans-Ansicht könnte verwendet werden, um Vorhersagemodelle zu erstellen und dunkle Biomarker zu erkennen. Dieses Protokoll verwendet den Patientendatensatz für Dickdarmadenokarzinome (COAD) aus der Datenbank 29 des Cancer Genome Atlas (TCGA)29 als anschauliches Beispiel. In diesem Zusammenhang werden Patienten in den Stadien I oder II als negative Proben eingestuft, während Patienten in den Stadien III oder IV als positive Proben gelten. Die Verteilungen von dunklen und traditionellen Biomarkern über die 26 TCGA-Krebsarten hinweg werden ebenfalls verglichen.
Beschreibung der HealthModel-Pipeline
Die in diesem Protokoll verwendete Methodik basiert auf dem zuvor veröffentlichten Framework26, wie in Abbildung 1 skizziert. Zu Beginn müssen die Benutzer den Eingabedatensatz vorbereiten, ihn in die vorgeschlagene HealthModel-Pipeline einspeisen und mqTrans-Funktionen abrufen. Detaillierte Anweisungen zur Datenaufbereitung finden Sie in Abschnitt 2 des Protokollabschnitts. Anschließend haben Benutzer die Möglichkeit, mqTrans-Merkmale mit den ursprünglichen transkriptomischen Merkmalen zu kombinieren oder nur mit den generierten mqTrans-Merkmalen fortzufahren. Der erzeugte Datensatz wird dann einem Merkmalsauswahlprozess unterzogen, wobei die Benutzer die Flexibilität haben, ihren bevorzugten Wert für k in der k-fachen Kreuzvalidierung für die Klassifizierung zu wählen. Die primäre Bewertungsmetrik, die in diesem Protokoll verwendet wird, ist die Genauigkeit.
HealthModel26 kategorisiert die transkriptomischen Merkmale in drei verschiedene Gruppen: TF (Transkriptionsfaktor), lincRNA (lange intergene nicht-kodierende RNA) und mRNA (Boten-RNA). Die TF-Merkmale werden auf der Grundlage der im Human Protein Atlas30,31 verfügbaren Annotationen definiert. In dieser Arbeit werden die Annotationen von lincRNAs aus dem GTEx-Datensatz32 verwendet. Gene, die zu den Signalwegen der dritten Ebene in der KEGG-Datenbank33 gehören, werden als mRNA-Merkmale betrachtet. Es ist erwähnenswert, dass, wenn ein mRNA-Merkmal regulatorische Rollen für ein Zielgen aufweist, wie es in der TRRUST-Datenbank34 dokumentiert ist, es in die TF-Klasse umklassifiziert wird.
Dieses Protokoll generiert auch manuell die beiden Beispieldateien für die Gen-IDs der regulatorischen Faktoren (regulatory_geneIDs.csv) und der Ziel-mRNA (target_geneIDs.csv). Die paarweise Distanzmatrix zwischen den regulatorischen Merkmalen (TFs und lincRNAs) wird mit den Pearson-Korrelationskoeffizienten berechnet und mit der beliebten toolgewichteten Gen-Co-Expressions-Netzwerkanalyse (WGCNA)36 (adjacent_matrix.csv) geclustert. Benutzer können die HealthModel-Pipeline zusammen mit diesen Beispielkonfigurationsdateien direkt verwenden, um die mqTrans-Ansicht eines transkriptomischen Datensatzes zu generieren.
Technische Details von HealthModel
HealthModel stellt die komplizierten Beziehungen zwischen TFs und lincRNAs als Graph dar, wobei die Eingabemerkmale als die mit V bezeichneten Eckpunkte und eine als E bezeichnete Zwischenscheitelpunktkantenmatrix dienen. Jede Probe ist durch K-Regulationsmerkmale gekennzeichnet, die als VK×1 symbolisiert sind. Konkret umfasste der Datensatz 425 TFs und 375 lincRNAs, was zu einer Probendimensionalität von K = 425 + 375 = 800 führte. Um die Kantenmatrix E zu ermitteln, wurde in dieser Arbeit das beliebte Werkzeug WGCNA35 verwendet. Die paarweise Gewichtung, die zwei Scheitelpunkte verbindet, die als und 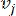 dargestellt werden
dargestellt werden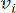 , wird durch den Pearson-Korrelationskoeffizienten bestimmt. Das genregulatorische Netzwerk weist eine skalenfreie Topologie36 auf, die durch das Vorhandensein von Hub-Genen mit zentralen funktionellen Rollen gekennzeichnet ist. Wir berechnen die Korrelation zwischen zwei Features oder Stützpunkten
, wird durch den Pearson-Korrelationskoeffizienten bestimmt. Das genregulatorische Netzwerk weist eine skalenfreie Topologie36 auf, die durch das Vorhandensein von Hub-Genen mit zentralen funktionellen Rollen gekennzeichnet ist. Wir berechnen die Korrelation zwischen zwei Features oder Stützpunkten 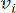 und
und 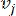 , indem wir das topologische Überlappungsmaß (TOM) wie folgt verwenden:
, indem wir das topologische Überlappungsmaß (TOM) wie folgt verwenden:
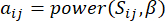 (1)
(1)
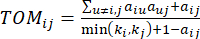 (2)
(2)
Der weiche Schwellenwert β wird mit der Funktion "pickSoft Threshold" aus dem WGCNA-Paket berechnet. Es wird die Potenzexponentialfunktion aij angewendet, wobei 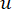 ein Gen ohne i und j steht und
ein Gen ohne i und j steht und 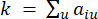 die Vertex-Konnektivität darstellt. WGCNA gruppiert die Expressionsprofile der transkriptomischen Merkmale in mehrere Module unter Verwendung eines häufig verwendeten Unähnlichkeitsmaßes (
die Vertex-Konnektivität darstellt. WGCNA gruppiert die Expressionsprofile der transkriptomischen Merkmale in mehrere Module unter Verwendung eines häufig verwendeten Unähnlichkeitsmaßes (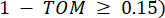 37.
37.
Das HealthModel-Framework wurde ursprünglich als Multitasking-Lernarchitekturkonzipiert 26. Dieses Protokoll verwendet nur die Modell-Pre-Training-Aufgabe für die Konstruktion der transkriptomischen mqTrans-Ansicht. Der Benutzer kann sich dafür entscheiden, das vortrainierte HealthModel im Rahmen des Multitasking-Graph-Aufmerksamkeitsnetzwerks mit zusätzlichen aufgabenspezifischen transkriptomischen Proben weiter zu verfeinern.
Technische Details der Merkmalsauswahl und -klassifizierung
Der Feature-Selection-Pool implementiert elf Feature-Selection-Algorithmen (FS). Darunter sind drei filterbasierte FS-Algorithmen: die Auswahl der besten K-Merkmale unter Verwendung des maximalen Informationskoeffizienten (SK_mic), die Auswahl von K-Merkmalen basierend auf der FPR von MIC (SK_fpr) und die Auswahl von K-Merkmalen mit der höchsten False-Discovery-Rate von MIC (SK_fdr). Darüber hinaus bewerten drei baumbasierte FS-Algorithmen einzelne Merkmale mithilfe eines Entscheidungsbaums mit dem Gini-Index (DT_gini), adaptiven verstärkten Entscheidungsbäumen (AdaBoost) und Random Forest (RF_fs). Der Pool enthält auch zwei Wrappermethoden: die rekursive Featureeliminierung mit dem linearen Unterstützungsvektorklassifikator (RFE_SVC) und die rekursive Featureeliminierung mit dem logistischen Regressionsklassifikator (RFE_LR). Schließlich sind zwei Einbettungsalgorithmen enthalten: der lineare SVC-Klassifikator mit den am höchsten bewerteten L1-Merkmalsbedeutungswerten (lSVC_L1) und der logistische Regressionsklassifikator mit den am höchsten bewerteten L1-Merkmalswichtigkeitswerten (LR_L1).
Der Klassifikatorpool verwendet sieben verschiedene Klassifikatoren, um Klassifizierungsmodelle zu erstellen. Zu diesen Klassifikatoren gehören Linear Support Vector Machine (SVC), Gaußian Naïve Bayes (GNB), Logistic Regression Classifier (LR), k-Nearest Neighbor, wobei k standardmäßig auf 5 gesetzt ist (KNN), XGBoost, Random Forest (RF) und Decision Tree (DT).
Die zufällige Aufteilung des Datensatzes in die train: Test-Teilmengen kann in der Befehlszeile festgelegt werden. Im gezeigten Beispiel wird das Verhältnis von train: test = 8:2 verwendet.
Access restricted. Please log in or start a trial to view this content.
Protokoll
HINWEIS: Das folgende Protokoll beschreibt die Details des Informatik-Analyseverfahrens und der Python-Befehle der Hauptmodule. Abbildung 2 veranschaulicht die drei Hauptschritte mit Beispielbefehlen, die in diesem Protokoll verwendet werden, und verweist auf die zuvor veröffentlichten Werke26,38 für weitere technische Details. Führen Sie das folgende Protokoll unter einem normalen Benutzerkonto in einem Computersystem aus und vermeiden Sie die Verwendung des Administrator- oder Root-Kontos. Dies ist ein computergestütztes Protokoll und enthält keine biomedizinischen Gefahrenfaktoren.
1. Python-Umgebung vorbereiten
- Erstellen Sie eine virtuelle Umgebung.
- In dieser Studie wurden die Programmiersprache Python und eine virtuelle Python-Umgebung (VE) mit Python 3.7 verwendet. Gehen Sie folgendermaßen vor (Abbildung 3A):
conda create -n healthmodel python=3.7
conda create ist der Befehl zum Erstellen eines neuen VE. Der Parameter -n gibt den Namen der neuen Umgebung an, in diesem Fall healthmodel. Und python=3.7 gibt die zu installierende Python-Version an. Wählen Sie einen beliebigen Namen und eine Python-Version aus, die den obigen Befehl unterstützt. - Nach dem Ausführen des Befehls ähnelt die Ausgabe Abbildung 3B. Geben Sie y ein, und warten Sie, bis der Vorgang abgeschlossen ist.
- In dieser Studie wurden die Programmiersprache Python und eine virtuelle Python-Umgebung (VE) mit Python 3.7 verwendet. Gehen Sie folgendermaßen vor (Abbildung 3A):
- Aktivieren der virtuellen Umgebung
- Aktivieren Sie in den meisten Fällen das erstellte VE mit dem folgenden Befehl (Abbildung 3C):
Conda Activate HealthModel - Befolgen Sie die plattformspezifischen Anweisungen für die VE-Aktivierung, wenn einige Plattformen erfordern, dass der Benutzer die plattformspezifischen Konfigurationsdateien für die Aktivierung hochlädt.
- Aktivieren Sie in den meisten Fällen das erstellte VE mit dem folgenden Befehl (Abbildung 3C):
- Installieren von PyTorch 1.13.1
- PyTorch ist ein beliebtes Python-Paket für Algorithmen der künstlichen Intelligenz (KI). Verwenden Sie als Beispiel PyTorch 1.13.1, das auf der GPU-Programmierplattform CUDA 11.7 basiert. Weitere Versionen finden Sie unter https://pytorch.org/get-started/previous-versions/. Verwenden Sie den folgenden Befehl (Abbildung 3D):
pip3 Taschenlampe installierenVision Torchaudio
HINWEIS: Es wird dringend empfohlen, PyTorch Version 1.12 oder höher zu verwenden. Andernfalls kann die Installation des erforderlichen Pakets torch_geometric eine Herausforderung darstellen, wie auf der offiziellen torch_geometric-Website angegeben ist: https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html.
- PyTorch ist ein beliebtes Python-Paket für Algorithmen der künstlichen Intelligenz (KI). Verwenden Sie als Beispiel PyTorch 1.13.1, das auf der GPU-Programmierplattform CUDA 11.7 basiert. Weitere Versionen finden Sie unter https://pytorch.org/get-started/previous-versions/. Verwenden Sie den folgenden Befehl (Abbildung 3D):
- Installieren Sie zusätzliche Pakete für torch-geometric
- Installieren Sie gemäß den Richtlinien in https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html die folgenden Pakete: torch_scatter, torch_sparse, torch_cluster und torch_spline_conv mit dem Befehl (Abbildung 3E):
pip install pyg_lib torch_scatter torch_sparse torch_cluster torch_spline_conv -f https://data.pyg.org/whl/torch-1.13.0+cu117.html
- Installieren Sie gemäß den Richtlinien in https://pytorch-geometric.readthedocs.io/en/latest/install/installation.html die folgenden Pakete: torch_scatter, torch_sparse, torch_cluster und torch_spline_conv mit dem Befehl (Abbildung 3E):
- Installieren Sie das Paket torch-geometric .
- Für diese Studie ist eine spezielle Version, 2.2.0, des Torch-geometric-Pakets erforderlich. Führen Sie den folgenden Befehl aus (Abbildung 3F):
pip install torch_geometric==2.2.0
- Für diese Studie ist eine spezielle Version, 2.2.0, des Torch-geometric-Pakets erforderlich. Führen Sie den folgenden Befehl aus (Abbildung 3F):
- Installieren Sie andere Pakete.
- Pakete wie Pandas sind in der Regel standardmäßig verfügbar. Ist dies nicht der Fall, installieren Sie sie mit dem Befehl pip. Um beispielsweise pandas und xgboost zu installieren, führen Sie Folgendes aus:
pip installieren pandas
pip install xgboost
- Pakete wie Pandas sind in der Regel standardmäßig verfügbar. Ist dies nicht der Fall, installieren Sie sie mit dem Befehl pip. Um beispielsweise pandas und xgboost zu installieren, führen Sie Folgendes aus:
2. Verwenden des vortrainierten HealthModel zum Generieren der mqTrans-Features
- Laden Sie den Code und das vortrainierte Modell herunter.
- Laden Sie den Code und das vortrainierte HealthModel von der Website herunter: http://www.healthinformaticslab.org/supp/resources.php, die den Namen HealthModel-mqTrans-v1-00.tar.gz hat (Abbildung 4A). Die heruntergeladene Datei kann in einen benutzerdefinierten Pfad dekomprimiert werden. Die detaillierte Formulierung und die unterstützenden Daten des implementierten Protokolls finden Sie in26.
- Stellen Sie die Parameter zum Ausführen von HealthModel vor.
- Ändern Sie zunächst das Arbeitsverzeichnis in der Befehlszeile in den Ordner HealthModel-mqTrans . Verwenden Sie die folgende Syntax zum Ausführen des Codes:
python main.py
Die Details zu den einzelnen Parametern und den Daten-, Modell- und Ausgabeordnern lauten wie folgt:
Datenordner: Dies ist der Quelldatenordner, und jede Datendatei liegt im CSV-Format vor. Dieser Datenordner enthält zwei Dateien (siehe detaillierte Beschreibungen in den Schritten 2.3 und 2.4). Diese Dateien müssen durch personenbezogene Daten ersetzt werden.
data.csv: Die Transkriptom-Matrix-Datei. In der ersten Zeile werden die Merkmals- (oder Gen-) IDs aufgelistet, und in der ersten Spalte sind die Beispiel-IDs aufgeführt. Die Liste der Gene umfasst die regulatorischen Faktoren (TFs und lincRNAs) und die regulierten mRNA-Gene.
label.csv: Die Beispiel-Etikettendatei. Die erste Spalte listet die Beispiel-IDs auf, und die Spalte mit dem Namen "label" gibt die Beispielbezeichnung an.
Modellordner: Der Ordner, in dem Informationen über das Modell gespeichert werden:
HealthModel.pth: Das vortrainierte HealthModel.
regulatory_geneIDs.csv: Die regulatorischen Gen-IDs, die in dieser Studie verwendet wurden.
target_geneIDs.csv: Die in dieser Studie verwendeten Zielgene.
adjacent_matrix.csv: Die benachbarte Matrix der regulatorischen Gene.
Ausgabeordner: Die Ausgabedateien werden in diesen Ordner geschrieben, der vom Code erstellt wurde.
test_target.csv: Der Genexpressionswert der Zielgene nach Z-Normalisierung und Imputation.
pred_target.csv: Der vorhergesagte Genexpressionswert von Zielgenen.
mq_target.csv: Der vorhergesagte Genexpressionswert von Zielgenen.
- Ändern Sie zunächst das Arbeitsverzeichnis in der Befehlszeile in den Ordner HealthModel-mqTrans . Verwenden Sie die folgende Syntax zum Ausführen des Codes:
- Bereiten Sie die Transkriptomik-Matrixdatei im csv-Format vor.
- Jede Zeile stellt eine Probe dar, und jede Spalte steht für ein Gen (Abbildung 4B). Benennen Sie die Transkriptomik-Datamatrix-Datei wie data.csv im Datenordner .
HINWEIS: Diese Datei kann durch manuelles Speichern einer Datenmatrix im .csv Format aus einer Software wie Microsoft Excel generiert werden. Die transkriptomische Matrix kann auch durch Computerprogrammierung erzeugt werden.
- Jede Zeile stellt eine Probe dar, und jede Spalte steht für ein Gen (Abbildung 4B). Benennen Sie die Transkriptomik-Datamatrix-Datei wie data.csv im Datenordner .
- Bereiten Sie die Etikettendatei im CSV-Format vor.
- Ähnlich wie bei der Transkriptomik-Matrixdatei benennen Sie die Bezeichnungsdatei wie label.csv im Datenordner (Abbildung 4C).
Hinweis: Die erste Spalte enthält die Beispielnamen, und die Klassenbezeichnung jedes Beispiels wird in der Spalte mit dem Titel Bezeichnung angegeben. Der Wert 0 in der Beschriftungsspalte bedeutet, dass diese Probe negativ ist, 1 bedeutet eine positive Stichprobe.
- Ähnlich wie bei der Transkriptomik-Matrixdatei benennen Sie die Bezeichnungsdatei wie label.csv im Datenordner (Abbildung 4C).
- Generieren Sie die mqTrans-Features.
- Führen Sie den folgenden Befehl aus, um die mqTrans-Features zu generieren und die in Abbildung 4D gezeigten Ausgaben zu erhalten. Die mqTrans-Features werden als Datei ./output/mq_targets.csv generiert, und die Beschriftungsdatei wird als Datei ./output/label.csv erneut gespeichert. Um die weitere Analyse zu erleichtern, werden auch die ursprünglichen Expressionswerte der mRNA-Gene als Datei extrahiert ./output/ test_target.csv.
python ./Get_mqTrans/code/main.py ./data ./Get_mqTrans/model ./output
- Führen Sie den folgenden Befehl aus, um die mqTrans-Features zu generieren und die in Abbildung 4D gezeigten Ausgaben zu erhalten. Die mqTrans-Features werden als Datei ./output/mq_targets.csv generiert, und die Beschriftungsdatei wird als Datei ./output/label.csv erneut gespeichert. Um die weitere Analyse zu erleichtern, werden auch die ursprünglichen Expressionswerte der mRNA-Gene als Datei extrahiert ./output/ test_target.csv.
3. mqTrans-Funktionen auswählen
- Syntax des Merkmalsauswahlcodes
- Ändern Sie zunächst das Arbeitsverzeichnis in den Ordner HealthModel-mqTrans . Verwenden Sie die folgende Syntax:
python ./FS_classification/testMain.py
Die Details der einzelnen Parameter lauten wie folgt:
in-data-file: Die Eingabedatendatei
in-label-file: Die Bezeichnung der Eingabedatendatei
Ausgabeordner: In diesem Ordner werden zwei Ausgabedateien gespeichert, darunter Output-score.xlsx (die Feature-Auswahlmethode und die Genauigkeit des entsprechenden Klassifikators) und Output-SelectedFeatures.xlsx (die ausgewählten Feature-Namen für jeden Feature-Auswahlalgorithmus).- select_feature_number: Wählen Sie die Anzahl der Features aus, die von 1 bis zur Anzahl der Features der Datendatei reicht.
- test_size: Legen Sie das Verhältnis der zu teilenden Testprobe fest. 0,2 bedeutet beispielsweise, dass das Eingabedataset nach dem Zufallsprinzip in die Testteilmengen des Zuges im Verhältnis 0,8:0,2 aufgeteilt wird.
- combine: Wenn true, werden zwei Datendateien für die Feature-Auswahl miteinander kombiniert, d.h. die ursprünglichen Ausdruckswerte und die mqTrans-Features. Wenn false, wird nur eine Datendatei für die Feature-Auswahl verwendet, d.h. die ursprünglichen Ausdruckswerte oder die mqTrans-Features.
- combine-Datei: Wenn combine true ist, geben Sie diesen Dateinamen an, um die kombinierte Datenmatrix zu speichern.
ANMERKUNG: Diese Pipeline soll zeigen, wie sich die generierten mqTrans-Features bei Klassifizierungsaufgaben verhalten, und sie verwendet direkt die in Abschnitt 2 generierte Datei für die folgenden Operationen.
- Ändern Sie zunächst das Arbeitsverzeichnis in den Ordner HealthModel-mqTrans . Verwenden Sie die folgende Syntax:
- Führen Sie den Feature-Auswahlalgorithmus für die mqTrans-Feature-Auswahl aus.
- Turn combine =False , wenn der Benutzer mqTrans-Features oder Original-Features auswählt.
- Wählen Sie zunächst 800 Original-Features aus, und teilen Sie das Dataset in train: test=0.8:0.2 auf:
python ./FS_classification/testMain.py ./output/test_target.csv ./output/label.csv ./result 800 0.2 False - Turn combine =True, wenn der Benutzer die mqTrans-Features mit den ursprünglichen Ausdruckswerten kombinieren möchte, um Features auszuwählen. Hier besteht das anschauliche Beispiel darin, 800 Features auszuwählen und das Dataset in train: test=0.8:0.2 aufzuteilen:
python ./FS_classification/testMain.py ./output/mq_targets.csv ./output/label.csv ./result_combine 800 0.2 True ./output/test_target.csv
HINWEIS: Abbildung 5 zeigt die Ausgabeinformationen. Die für dieses Protokoll benötigten Zusatzdateien befinden sich in HealthModel-mqTrans-v1-00.tar Ordner (Supplementary Coding File 1).
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Evaluierung der mqTrans-Ansicht des Transkriptomik-Datensatzes
Der Testcode verwendet elf Merkmalsauswahlalgorithmen (FS) und sieben Klassifikatoren, um zu bewerten, wie die generierte mqTrans-Ansicht des transkriptomischen Datensatzes zur Klassifizierungsaufgabe beiträgt (Abbildung 6). Der Testdatensatz besteht aus 317 Kolonadenokarzinomen (COAD) aus der Datenbank des Cancer Genome Atlas (TCGA)29. Die COAD-Patienten in den Stadien I oder II gelte...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Abschnitt 2 (Verwenden des vortrainierten HealthModel zum Generieren der mqTrans-Funktionen) des Protokolls ist der kritischste Schritt innerhalb dieses Protokolls. Nach der Vorbereitung der rechnerischen Arbeitsumgebung in Abschnitt 1 wird in Abschnitt 2 die mqTrans-Ansicht eines transkriptomischen Datensatzes auf der Grundlage des vortrainierten großen Referenzmodells generiert. Abschnitt 3 ist ein anschauliches Beispiel für die Auswahl der generierten mqTrans-Merkmale für Biomarker-Detektionen und Vorhersageaufgabe...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde unterstützt vom Senior and Junior Technological Innovation Team (20210509055RQ), den Guizhou Provincial Science and Technology Projects (ZK2023-297), der Science and Technology Foundation der Gesundheitskommission der Provinz Guizhou (gzwkj2023-565), dem Science and Technology Project des Bildungsministeriums der Provinz Jilin (JJKH20220245KJ und JJKH20220226SK), der National Natural Science Foundation of China (U19A2061), dem Jilin Provincial Key Laboratory of Big Data Intelligent Computing (20180622002JC) und den Mitteln für Grundlagenforschung der Zentralen Universitäten der JLU. Wir danken dem Herausgeber der Rezension und den drei anonymen Gutachtern aufrichtig für ihre konstruktive Kritik, die maßgeblich dazu beigetragen hat, die Strenge und Klarheit dieses Protokolls wesentlich zu verbessern.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Anaconda | Anaconda | version 2020.11 | Python programming platform |
| Computer | N/A | N/A | Any general-purpose computers satisfy the requirement |
| GPU card | N/A | N/A | Any general-purpose GPU cards with the CUDA computing library |
| pytorch | Pytorch | version 1.13.1 | Software |
| torch-geometric | Pytorch | version 2.2.0 | Software |
Referenzen
- Mutz, K. -O., Heilkenbrinker, A., Lönne, M., Walter, J. -G., Stahl, F. Transcriptome analysis using next-generation sequencing. Curr Opin in Biotechnol. 24 (1), 22-30 (2013).
- Meng, G., Tang, W., Huang, E., Li, Z., Feng, H. A comprehensive assessment of cell type-specific differential expression methods in bulk data. Brief Bioinform. 24 (1), 516(2023).
- Iqbal, N., Kumar, P. Integrated COVID-19 Predictor: Differential expression analysis to reveal potential biomarkers and prediction of coronavirus using RNA-Seq profile data. Comput Biol Med. 147, 105684(2022).
- Ravichandran, S., et al. VB(10), a new blood biomarker for differential diagnosis and recovery monitoring of acute viral and bacterial infections. EBioMedicine. 67, 103352(2021).
- Lv, J., et al. Targeting FABP4 in elderly mice rejuvenates liver metabolism and ameliorates aging-associated metabolic disorders. Metabolism. 142, 155528(2023).
- Cruz, J. A., Wishart, D. S. Applications of machine learning in cancer prediction and prognosis. Cancer Inform. 2, 59-77 (2007).
- Cox, D. R. Analysis of Survival Data. , Chapman and Hall/CRC. London. (2018).
- Newman, A. M., et al. Robust enumeration of cell subsets from tissue expression profiles. Nat Methods. 12 (5), 453-457 (2015).
- Ramilowski, J. A., et al. A draft network of ligand-receptor-mediated multicellular signalling in human. Nat Commun. 6 (1), 7866(2015).
- Xu, Y., et al. MiR-145 detection in urinary extracellular vesicles increase diagnostic efficiency of prostate cancer based on hydrostatic filtration dialysis method. Prostate. 77 (10), 1167-1175 (2017).
- Wang, Y., et al. Profiles of differential expression of circulating microRNAs in hepatitis B virus-positive small hepatocellular carcinoma. Cancer Biomark. 15 (2), 171-180 (2015).
- Hu, S., et al. Transcriptional response profiles of paired tumor-normal samples offer novel. Oncotarget. 8 (25), 41334-41347 (2017).
- Xu, H., Luo, D., Zhang, F. DcWRKY75 promotes ethylene induced petal senescence in carnation (Dianthus caryophyllus L). Plant J. 108 (5), 1473-1492 (2021).
- Niu, H., et al. Dynamic role of Scd1 gene during mouse oocyte growth and maturation. Int J Biol Macromol. 247, 125307(2023).
- Aznaourova, M., et al. Single-cell RNA sequencing uncovers the nuclear decoy lincRNA PIRAT as a regulator of systemic monocyte immunity during COVID-19. Proc Natl Acad Sci U S A. 119 (36), 2120680119(2022).
- Prakash, A., Banerjee, M. An interpretable block-attention network for identifying regulatory feature interactions. Brief Bioinform. 24 (4), (2023).
- Zhai, Y., et al. Single-cell RNA sequencing integrated with bulk RNA sequencing analysis reveals diagnostic and prognostic signatures and immunoinfiltration in gastric cancer. Comput Biol Med. 163, 107239(2023).
- Duan, L., et al. Dynamic changes in spatiotemporal transcriptome reveal maternal immune dysregulation of autism spectrum disorder. Comput Biol Med. 151, 106334(2022).
- Zolotareva, O., et al. Flimma: a federated and privacy-aware tool for differential gene expression analysis). Genome Biol. 22 (1), 338(2021).
- Su, R., Zhu, Y., Zou, Q., Wei, L. Distant metastasis identification based on optimized graph representation of gene. Brief Bioinform. 23 (1), (2022).
- Xing, X., et al. Multi-level attention graph neural network based on co-expression gene modules for disease diagnosis and prognosis. Bioinformatics. 38 (8), 2178-2186 (2022).
- Bongini, P., Pancino, N., Scarselli, F., Bianchini, M. BioGNN: How Graph Neural Networks Can Solve Biological Problems. Artificial Intelligence and Machine Learning for Healthcare: Vol. 1: Image and Data Analytics. , Springer. Cham. (2022).
- Muzio, G., O'Bray, L., Borgwardt, K. Biological network analysis with deep learning. Brief Bioinform. 22 (2), 1515-1530 (2021).
- Luo, H., et al. Multi-omics integration for disease prediction via multi-level graph attention network and adaptive fusion. bioRxiv. , (2023).
- Feng, X., et al. Selecting multiple biomarker subsets with similarly effective binary classification performances. J Vis Exp. (140), e57738(2018).
- Duan, M., et al. Orchestrating information across tissues via a novel multitask GAT framework to improve quantitative gene regulation relation modeling for survival analysis. Brief Bioinform. 24 (4), (2023).
- Chicco, D., Starovoitov, V., Jurman, G. The benefits of the Matthews correlation Coefficient (MCC) over the diagnostic odds ratio (DOR) in binary classification assessment. IEEE Access. 9, 47112-47124 (2021).
- Goldman, M. J., et al. Visualizing and interpreting cancer genomics data via the Xena platform. Nat Biotechnol. 38 (6), 675-678 (2020).
- Liu, J., et al. An integrated TCGA pan-cancer clinical data resource to drive high-quality survival outcome analytics. Cell. 173 (2), 400-416 (2018).
- Uhlen, M., et al. Towards a knowledge-based human protein atlas. Nat Biotechnol. 28 (12), 1248-1250 (2010).
- Hernaez, M., Blatti, C., Gevaert, O. Comparison of single and module-based methods for modeling gene regulatory. Bioinformatics. 36 (2), 558-567 (2020).
- Consortium, G. The genotype-tissue expression (GTEx) project. Nat Genet. 45 (6), 580-585 (2013).
- Kanehisa, M., et al. KEGG for taxonomy-based analysis of pathways and genomes. Nucleic Acids Res. 51, D587-D592 (2023).
- Han, H., et al. TRRUST v2: an expanded reference database of human and mouse transcriptional. Nucleic Acids Res. 46, D380-D386 (2018).
- Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics. 9, 559(2008).
- Sulaimanov, N., et al. Inferring gene expression networks with hubs using a degree weighted Lasso. Bioinformatics. 35 (6), 987-994 (2019).
- Kogelman, L. J. A., Kadarmideen, H. N. Weighted Interaction SNP Hub (WISH) network method for building genetic networks. BMC Syst Biol. 8, 5(2014).
- Duan, M., et al. Pan-cancer identification of the relationship of metabolism-related differentially expressed transcription regulation with non-differentially expressed target genes via a gated recurrent unit network. Comput Biol Med. 148, 105883(2022).
- Duan, M., et al. Detection and independent validation of model-based quantitative transcriptional regulation relationships altered in lung cancers. Front Bioeng Biotechnol. 8, 582(2020).
- Fiorini, N., Lipman, D. J., Lu, Z. Towards PubMed 2.0. eLife. 6, 28801(2017).
- Liu, J., et al. Maternal microbiome regulation prevents early allergic airway diseases in mouse offspring. Pediatr Allergy Immunol. 31 (8), 962-973 (2020).
- Childs, E. J., et al. Association of common susceptibility variants of pancreatic cancer in higher-risk patients: A PACGENE study. Cancer Epidemiol Biomarkers Prev. 25 (7), 1185-1191 (2016).
- Wang, C., et al. Thailandepsins: bacterial products with potent histone deacetylase inhibitory activities and broad-spectrum antiproliferative activities. J Nat Prod. 74 (10), 2031-2038 (2011).
- Lv, X., et al. Transcriptional dysregulations of seven non-differentially expressed genes as biomarkers of metastatic colon cancer. Genes (Basel). 14 (6), 1138(2023).
- Li, X., et al. Undifferentially expressed CXXC5 as a transcriptionally regulatory biomarker of breast cancer. Advanced Biology. , (2023).
- Yuan, W., et al. The N6-methyladenosine reader protein YTHDC2 promotes gastric cancer progression via enhancing YAP mRNA translation. Transl Oncol. 16, 101308(2022).
- Tanabe, A., et al. RNA helicase YTHDC2 promotes cancer metastasis via the enhancement of the efficiency by which HIF-1α mRNA is translated. Cancer Lett. 376 (1), 34-42 (2016).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten